沙门氏菌(Salmonella)是导致食源性疾病的主要原因之一,可引起胃肠炎和腹泻疾病,还会引起动物伤寒、副伤寒、霍乱等疾病发生。沙门氏菌广泛存在于自然环境中,因此在新鲜农产品和动物饲料生产过程中的任何环节都可能发生沙门氏菌污染。在世界各国发生的细菌性食物中毒中,沙门氏菌常位列榜首。因此,为防止和减少沙门氏菌对人类健康造成危害,建立沙门氏菌快速检测方法成为食品安全领域日益增长的需求。
目前,沙门氏菌的检测方法主要有:基于细菌理化差异的平板菌落计数(培养)法、基于抗原与抗体免疫反应的酶联免疫吸附试验(ELISA)、基于DNA扩增的聚合酶链反应(PCR)(常规PCR、定量PCR等)。平板菌落培养法是沙门氏菌检测的最传统、最常用检测方法,但其劳动强度大、耗时长,一般至少需要3~5 d,因此不能用于现场检测。ELISA具有特异性强、通量高、成本低等优点,在沙门氏菌检测中得到广泛应用。然而,这是一种多相检测方法,需要多次洗涤,可能会导致交叉污染和假阳性,另外,其缺乏足够的灵敏性,通常有一个低检出限104 CFU/mL[1]。PCR方法具有高的检测灵敏度、时间短,但其需要复杂的DNA提取过程,对技术人员要求较高。
近年来,生物传感技术以及纳米材料的迅速发展,推动了基于抗原/抗体、核酸适配体、酶、细胞、仿生受体、噬菌体等传感元件的光学、电化学、表面增强拉曼散射(Surface Eenhanced Raman Scattering,SERS)、石英晶体微量天平(Quartz Crystal Microbalance,QCM)等多种检测食源性致病菌的快速、高灵敏检测方法的迅速发展。其中,核酸适配体是一种短的核酸序列,具有特异性强、亲和力高、合成速度快、重现性好、修饰方便等优点。基于核酸适配体的生物传感器被称为核酸适配体传感器,已被广泛应用于食源性致病菌的定量检测,且正在逐步取代以抗原/抗体为基础的免疫生物传感器。本文就核酸适配体生物传感技术在沙门氏菌定量检测中的应用研究进展进行综述。
1 沙门氏菌核酸适配体
核酸适配体是利用指数富集配体系统进化技 术(systematic evolution of ligandsby exponential enrichment,SELEX)从一种人工合成的寡核苷酸文库中筛选得到的能与靶分子高亲和性和高特异性性结合的单链寡核苷酸。近年来,为提高筛选效率、简化筛选过程,开发了多种SELEX新技术,如亲和层析SELEX、毛细管电泳SELEX、全细胞SELEX和加尾SELEX技术等,这些新型筛选技术极大促进了核酸适配体在各个领域的发展和应用[2]。
在沙门氏菌核酸适配体的筛选(表1)方面,WANG等[3]利用全细胞SELEX技术,筛选得到了鼠伤寒沙门氏菌的ssDNA核酸适配体,Kd值为6.33(±0.58)nmol/L。Lee等[4]利用传统全细胞SELEX技术筛选得到鼠伤寒沙门氏菌的RNA适配体,这种RNA核酸适配体具有抗RNA酶的作用,其Kd值约为20 nmol/L。近年来,Li等[5]建立了一种基于石英晶体微天平(QCM)的SELEX技术,并筛选得到鼠伤寒沙门氏菌的ssDNA核酸适配体,Kd值为58.5 nmol/L。通过对比发现,通过传统的SELEX筛选技术得到的ssDNA核酸适配体的亲和力要高于RNA核酸适配体;而通过新型基于QCM的SELEX技术得到的ssDNA核酸适配体的亲和力要高于传统SELEX筛选技术,且新型筛选方法速度快、效率高、成本低。近年来,利用上述SELEX筛选技术得到的核酸适配体,科学家们已经建立了多种新型核酸适配体生物传感检测方法,并将其应用于沙门氏菌的定量检测中。本文基于核酸适配体的生物传感技术在沙门氏菌定量检测中的应用研究进展进行了详细概述,并对这些检测方法进行详细对比(表2)。
2 核酸适配体生物传感技术在沙门氏菌定量检测中的应用
2.1 比色检测
比色法(Colorimetry)是通过比较或测量有色物质溶液颜色深度来确定待测组分含量的方法。作为一种最简单的传感检测方法,已被广泛应用于沙门氏菌的定量检测。
表1 沙门氏菌核酸适配体筛选
Table 1 Screening of Salmonella aptamer

参考文献References全细胞SELEX Whole cell SELEX ssDNA 6.33±0.58 筛选时间长、亲和力低、成本高等 [3]全细胞SELEX Whole cell SELEX RNA 20 筛选时间长、亲和力低、成本高等 [4]石英晶体微天平(QCM)-SELEX QCM-SELEX ssDNA 58.5 筛选时间短、亲和力高、效率高、成本低等 [5]筛选方法Screening methods适配体类型Aptamer types Kd值Kd value(nmol/L)特点Characteristics
表2 适配体生物传感技术在沙门氏菌定量检测中的应用情况比较
Table 2 Comparison of application of aptamer biosensor technology in the quantitative detection of Salmonella
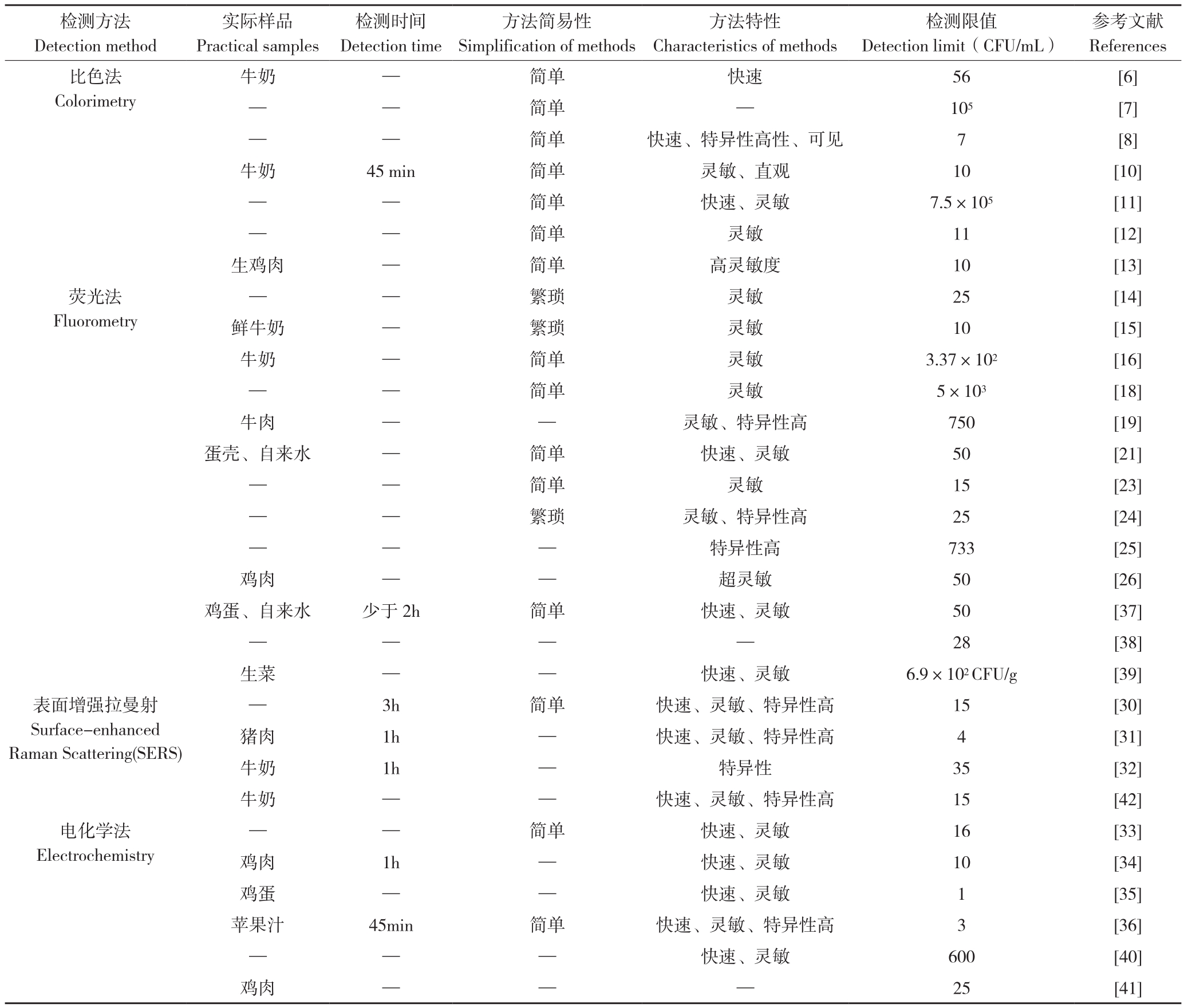
注:“—”表示未报道。
Note: “—”represents unreported.
检测方法Detection method参考文献References实际样品Practical samples检测时间Detection time方法简易性Simplification of methods方法特性Characteristics of methods检测限值Detection limit(CFU/mL)比色法Colorimetry牛奶 — 简单 快速 56 [6]——简单 — 105 [7]简单 快速、特异性高性、可见 7 [8]牛奶 45 min 简单 灵敏、直观 10 [10]——简单 快速、灵敏 7.5×105 [11]——简单 灵敏 11 [12]——生鸡肉 — 简单 高灵敏度 10 [13]荧光法Fluorometry繁琐 灵敏 25 [14]鲜牛奶 — 繁琐 灵敏 10 [15]牛奶 — 简单 灵敏 3.37×102 [16]——简单 灵敏 5×103 [18]牛肉 — — 灵敏、特异性高 750 [19]蛋壳、自来水 — 简单 快速、灵敏 50 [21]——简单 灵敏 15 [23]——繁琐 灵敏、特异性高 25 [24]——特异性高 733 [25]——鸡肉 — — 超灵敏 50 [26]鸡蛋、自来水 少于2h 简单 快速、灵敏 50 [37]——28 [38]生菜 — — 快速、灵敏 6.9×102 CFU/g [39]表面增强拉曼射Surface-enhanced Raman Scattering(SERS)3h 简单 快速、灵敏、特异性高 15 [30]猪肉 1h — 快速、灵敏、特异性高 4 [31]牛奶 1h — 特异性 35 [32]牛奶 — — 快速、灵敏、特异性高 15 [42]电化学法Electrochemistry—简单 快速、灵敏 16 [33]鸡肉 1h — 快速、灵敏 10 [34]鸡蛋 — — 快速、灵敏 1 [35]苹果汁 45min 简单 快速、灵敏、特异性高 3 [36]——快速、灵敏 600 [40]鸡肉 — — — 25 [41]——
2.1.1 盐诱导金纳米颗粒聚集 金纳米颗粒(Gold nanoparticles,GNPs)在分散和聚集状态下具有不同的外观颜色,根据这一特性,科学家们建立了多种基于盐诱导GNPs聚集的比色生物传感检测方法。如MA等[6]将沙门氏菌核酸适配体包被于GNPs表面,当样品中含有目标分子沙门氏菌时,适配体与沙门氏菌专一性结合,致使适配体从GNPs-适配体复合物的表面脱离,释放出GNPs。当加入NaCl时,则会导致GNPs的聚集,GNPs溶液的颜色由红色变成紫色甚至深蓝色。该方法得到沙门氏菌的检测范围为102~107 CFU/mL,最低检测限为56 CFU/mL。WU等[7]利用相同的检测原理,实现了沙门氏菌和大肠杆菌的定量检测,最低检测限为105 CFU/mL。该检测方法具有简单、快速、成本低的优点,但其检测灵敏度相对比较低。
2.1.2 银还原放大比色信号 为进一步提高灵敏度,Yuan等[8]提出了一种银还原的比色生物传感检测方法。通过生物素亲和素的相互作用,沙门氏菌的适配体被固定于微孔板;另外,巯基修饰的适配体被固定于GNPs。当沙门氏菌存在于样品中,形成适配体-沙门氏菌-适配体-GNPs三明治复合物;然后,利用银放大溶液增强比色信号,肉眼即可对沙门氏菌进行定量检测,得到沙门氏菌的最低检测限为7 CFU/mL。这种银还原放大比色信号的方法,可大大提高沙门氏菌的检测灵敏度,具有简单、快速、无需检测仪器的优点。
2.1.3 磁纳米颗粒对GNPs的高消光系数特性 磁纳米颗粒(Magnetic Nanoparticles,MNPs)除具有较大的比表面积、丰富的功能基团外,还具有良好的磁响应强度及对GNPs的高消光系数等优良特性[9]。根据这一特性,DUAN等[10]将巯基修饰适配体和生物素修饰适配体分别固定在于GNPs和MNPs表面;当沙门氏菌目标物存在于样品中时,适配体与沙门氏菌高专一性结合,形成GNPs-适配体-沙门氏菌-适配体-磁珠复合物;磁分离后,GNPs在悬浮液中的含量降低,因此悬浮液颜色变浅,相应其UV吸光度也会降低。利用该方法得到沙门氏菌的检测范围为25~105 CFU/mL,最低检测限为10 CFU/mL。与上述微孔板中银还原方法相比,这种在磁纳米颗粒上进行核酸适配体的固定、磁纳米复合物的分离纯化等过程都相对比较简单、快速,且降低交叉污染的风险。
2.1.4 酶催化 MNPs除具有高消光系数的特性外,还具有酶催化活性。Park等[11]利用MNPs的催化活性以及无标记核酸适配体,构建了一种核酸适配体生物传感检测方法。首先,表面带负电荷的核酸适配体被吸附到表面带正电荷的MNPs上,这种吸附作用会阻碍MNPs的催化活性;当沙门氏菌目标物存在时,适配体从磁珠上脱离,MNPs的催化活性恢复,导致底物四甲基联苯胺(TMB)溶液颜色变成蓝色,这种颜色变化可通过肉眼进行观察,最后得到沙门氏菌的最低检测限为7.5×105 CFU/mL。这种检测方法具有简单、快速的特点,但灵敏度低。
为提高检测灵敏度,一些具有酶催化活性的纳米复合材料也被应用于核酸适配体生物传感检测方法的构建。如Wu等[12]构建了一种ZnFe2O4还原的氧化石墨烯(ZnFe2O4-rGO)纳米结构,这种新型纳米结构具有过氧化氢酶的催化活性,可催化过氧化氢对TMB的氧化,产生蓝色产物;利用修饰有ZnFe2O4-rGO的适配体作为信号探针,生物素修饰适配体被固定于微孔板,作为捕获探针;当沙门氏菌目标物存在时,形成微孔板-适配体-沙门氏菌-适配体-的ZnFe2O4-rGO复合物三明治结构;ZnFe2O4-rGO催化底物TMB产生颜色变化,最后得到沙门氏菌的最低检测限为11 CFU/mL,检测范围为 11~1.10×105 CFU/mL。由此可见,复合纳米材料应用于生物传感检测方法中,可大大提高沙门氏菌的检测灵敏度。
2.1.5 过氧化氢还原Au3+ Au3+可被过氧化氢还原成红色非聚集Au原子。Zhu等[13]将沙门氏菌核酸适配体固定于微孔板,当样品中含有目标物沙门氏菌时,将会被核酸适配体捕获;然后再次加入生物素修饰的适配体,形成双适配体夹心结构;最后加入亲和素修饰的过氧化氢酶后,过氧化氢酶催化过氧化氢的降解,因此,导致晶体生长动力学反应减慢,形成聚集的纳米颗粒,溶液颜色变成蓝色;当样品中不含沙门氏菌时,过氧化氢则不被降解,Au3+在高浓度过氧化氢条件下,被快速还原形成非聚集的球型纳米颗粒,溶液变成红色。利用该方法实现了沙门氏菌的定量检测,检测范围为101~106 CFU/mL,最低检测限为10 CFU/mL。该方法具有快速、高灵敏度、低成本、高专一性以及高准确性的优点。
2.2 荧光检测
2.2.1 传统有机荧光染料 WANG等[14]利用适配体修饰的NaYF4:Ce/Tb纳米颗粒和一个荧光DNA标签,构建了一种均一的时间分辨荧光共振能量转移(TR-FRET)检测方法,实现了沙门氏菌的检测。其中,适配体功能化的NaYF4:Ce/Tb纳米颗粒作为能量供体,标记有羧基荧光素(5-Carboxyfluorescein,FAM)的互补寡核苷酸(cDNA)作为受体。当样品中不存在沙门氏菌时,适配体与cDNA杂交,光子能量从NaYF4:Ce/Tb纳米颗粒转移到FAM。通过这种方法得到沙门氏菌的线性检测范围为102~106CFU/mL,最低检测限为25 CFU/mL。Duan等[15]通过抗-ε-亚基抗体-生物素-亲和素-生物素-核酸适配体连接臂,将鼠伤寒沙门氏菌的核酸适配体与色素细胞中的F0F1-ATP酶的“旋转子”ε-亚基偶联,F-DHPE荧光探针标记于色素细胞的表面,建立了一种新型核酸适配体生物传感器,实现了沙门氏菌的定量检测,其线性检测范围为10~104CFU/mL,最低检测限为10 CFU/mL。尽管这些方法具有高的检测灵敏度,但其操作比较繁琐、耗时。
近年来,微流控芯片技术作为一种极具潜力的分析平台,具有集成化、微型化、高通量及自动化检测的特点,已被广泛应用于食源性致病菌的定量检测中。如ZHANG等[16]建立了一种微芯片毛细管电泳耦合激光诱导荧光技术(MCELIF),SYBR Gold标记的核酸适配体与沙门氏菌专一性结合后,利用核酸适配体-沙门氏菌复合物与核酸适配体的质荷比的不同,通过MCE将复合物与核酸适配体分离开,实现了牛奶样品中沙门氏菌的定量检测,得到了沙门氏菌的最低检测限为3.37×102 CFU/mL。这种微流控芯片检测方法具有高度的集成化、操作简便的优点。
尽管上述基于传统有机荧光染料的检测方法具有较高的检测灵敏度,但这些传统有机荧光染料的荧光强度相对较低,易漂白,对环境变化灵敏度高。为克服这些问题,一些具有高荧光强度、高稳定性的荧光纳米发光材料,如量 子 点(Quantum Dot,QDs)、 碳 点(Carbon Dots,CDs)、上转换纳米发光材料(Up-conversion Nanoparticles,UCNPs)等作为传统有机荧光染料的替代标签,被用于新型核酸适配体荧光生物传感器的构建中。
2.2.2 量子点(QDs) QDs具有宽的激发波长范围、窄的发射峰、宽大的斯托克斯位移、高荧光强度及光稳定性,已成为最最理想的荧光替代标签[17]。Hamula等[18]利用发射波长分别在535、585 nm处的QDs作为光学标签,并将其分别与副溶血性弧菌和鼠伤寒沙门氏菌的适配体偶联,建立了一种双色流式细胞传感分析方法,最终得到副溶血性弧菌和鼠伤寒沙门氏菌的最低检测限为5×103 CFU/mL。Xu等[19]首先分别利用修饰有大肠杆菌0157:H7、金黄色葡萄球菌、单增李斯特菌和鼠伤寒沙门氏菌抗体的免疫磁珠从食品样本中分离出4种目标菌,然后利用发射波长不同的4种量子点(528、572、621、668 nm)分别标记4种食源性致病菌的适配体,利用该标记的适配体专一性识别磁珠上的捕获的目标菌。利用该方法得到大肠杆菌、金黄色葡萄球菌、单增李斯特菌和鼠伤寒沙门氏菌纯菌样的最低检测限分别为80、100、47、160 CFU/mL;得到牛肉样品中4种菌的最低检测限分别为320、350、110、750 CFU/mL。与传统有机荧光染料相比,这些基于QDs的核酸适配体生物传感检测方法改善了荧光淬灭现象,具有更高的检测灵敏度;此外,还能同时实现多种食源性致病菌的高通量检测。除这些优势外,由于QDs的激发光仍然在紫外-可见光区,因此,被测生物样品同样会被激发,荧光背景值偏高,检测灵敏度仍会受到一定程度影响。
2.2.3 碳点(CDs) CDs是指粒径小于10 nm的碳纳米颗粒,是一类新型的光致发光纳米材料。CDs具有荧光量子产率高、合成简单、毒性低、原材料来源广泛、生物相容性好、荧光性能优等优点,受到科学家们的广泛关注[20]。近年来,Wang等[21]以柠檬酸为碳源,采用水热法,成功合成了CDs,并利用该CDs建立了一种基于aptamer-CDs的核酸适配体生物传感器,实现了蛋壳和自来水中的鼠伤寒沙门氏菌的定量检测,得到沙门氏菌的线性范围为103~105 CFU/mL,最低检测限为50 CFU/mL。该方法具有简单、快速、高灵敏的特点,可用于其他食源性致病菌的定量检测。
2.2.4 上转换纳米发光材料(UCNPs) UCNPs具有高的化学稳定性,不易被光漂白,不受外界环境(如湿度、酸度等)和被测物样品的影响的优点,此外,其使用近红外光(980、880 nm)作为激发光源,发射光在可见光区域分布,且具有单激发多谱带发射,具有低的荧光背景值的优异特性,特别适合作为复杂生物样本中的荧光标记物[22]。因此,UCNPs为核酸适配体荧光生物传感方法的构建提供了新的领域。Wu等[23]利用蓝、绿、红3种UCNPs分别作为金黄色葡萄球菌、副溶血性弧菌和鼠伤寒沙门氏菌核酸适配体的荧光标记,同时利用互补链功能化的磁珠进行分离纯化,建立了一种基于多色UCNPs的荧光传感器,实现了3种食源性致病菌的多重检测,得到金黄色葡萄球菌、副溶血性弧菌和鼠伤寒沙门氏菌的最低检测限分别为25、10、15 CFU/mL。这种基于UCNPs的核酸适配体荧光生物传感检测方法具有高灵敏、简单、高通量检测的优点。
2.2.5 无标记荧光核酸适配体传感器 以上描述的荧光标记核酸适配体传感器,都需要荧光基团或荧光纳米材料对核酸适配体进行荧光标记,因此,需要对标记后的分子进行复杂的分离和纯化,所以这种标记方法费时费力、成本昂贵。因此,开发方便、无需标记的核酸适配体荧光传感器,实现沙门氏菌的定量检测非常有研究意义。
虽然SYBR、PicoGreen和AccuBlue等传统有机荧光染料具有弱的荧光强度,但与双链DNA(dsDNA)结合后,会产生高的荧光强度,而与ssDNA结合后,却没有明显荧光强度变化。DUAN等[24]利用这一特性,建立了一种基于AccuBlue染料的无标记适配体荧光传感器,实现了鼠伤寒沙门氏菌的定量检测。其检测原理为:当沙门氏菌目标物存在于样本中,核酸适配体与沙门氏菌专一性结合,导致cDNA从cDNA-aptamer双链中脱离,因而导致AccuBlue的释放,荧光强度降低。利用该方法得到了沙门氏菌的线性检测范围为50~106CFU/mL,最低检测限为25 CFU/mL。近年来,Srinivasan等[25]也利用相同的实验原理,利用SYBR Green I作为荧光信号,实现了鼠伤寒沙门氏菌的定量检测,最低检出限为733 CFU/mL。此外,Srinivasan等[25]也建立了另外一种检测鼠伤寒沙门氏菌的无标记荧光核酸适配体传感检测方法,该方法利用罗丹明B(Rhodamine B,RB)和GNPs之间的荧光共振能量转移(Fluorescence Rresonance Eenergy Transfer,FRET)进行沙门氏菌的定量检测。其检测原理为:当适配体与GNPs与RB混合后,由于FRET,RB荧光被大大淬灭;当核酸适配体被吸附到GNPs的表面,以保护GNPs受盐诱导发生聚集现象,而这时存在的GNPs会导致RB荧光猝灭。当沙门氏菌目标物存在时,适配体与目标物专一性结合,导致GNPs不稳定,发生盐诱导聚集,进而导致淬灭的RB荧光的恢复。利用这种方法实现了沙门氏菌的定量检测,检测范围为1 530~96 938 CFU/mL,最低检出限为464 CFU/mL。此外,Zhang等[26]建立了一种基于三重触发序列可再生链取代放大-驱动荧光Ag纳米团簇的聚集的新型无标记、无修饰、无DNA萃取的荧光检测方法,实现了鼠伤寒沙门氏菌的定量检测,线性检测范围为102~107 CFU/mL,最低检测限为50 CFU/mL。上述这些无标记荧光核酸适配体生物传感检测方法,不需对标记的复合物进行分离纯化,其构建过程相对简单、快速,大大降低了检测成本。
2.3 表面增强拉曼散射(SERS)检测
SERS已被广泛应用于环境监测、医学诊断、食品安全等多个研究领域。SERS技术具有快速、高灵敏检测食源性致病菌的潜力,可为食品安全提供保障。常见的表面增强拉曼散射基质有银纳米粒子(AgNPs)、GNPs等重金属纳米材料[27-29]。ZHANG等[30]建立了一种GNPs增强的SERS核酸适配体生物传感器,实现了金黄色葡萄球菌和鼠伤寒沙门氏菌的同时检测。修饰有拉曼分子4-巯基苯甲酸(p-MBA)、5,5’-二硫基(2-硝基苯甲酸)和适配体的GNPs作为信号探针;固定有沙门氏菌和葡萄球菌适配体的Fe3O4磁性金纳米颗粒(Magnetic Gold Nanoparticles, MGNPs)作为捕获探针。沙门氏菌和葡萄球菌目标物加入到反应体系后,被捕获探针和信号探针上的适配体所捕获,形成三明治结构。最终得到沙门氏菌和葡萄球菌的的最低检测限分别为15、35 CFU/mL。该检测方法利用MGNPs进行分离纯化,因此简化了实验过程,且具有高灵敏、高专一性的优点;但该方法约需3 h才能实现沙门氏菌的定量检测,因此检测时间相对较长。
近年来,Ma等[31]构建了一种基于多刺状金纳米颗粒(Spiny Gold Nanoparticles, SGNPs)的SERS核酸适配体传感检测方法。SGNPs依次经4-巯基苯甲酸(p-MBA)和巯基化修饰适配体(SH-Aptamer)进行功能化修饰,其中p-MBA作为拉曼信号分子,巯基修饰的适配体作为SERS探针。此外,被固定在孔板上的生物素修饰的适配体用以专一性识别和捕获沙门氏菌。该方法得到了沙门氏菌的检测范围为101~105 CFU/mL,最低检测限为4 CFU/mL,检测时间约1 h。该检测方法具有超高的检测灵敏度和选择性,还具有低成本、快速、简易操作等优点,且检测时间相对较短。
Xu等[32]利用DNA组装的金纳米二聚体(Gold Nanodimer,GNDs),建立了一种SERS适配体传感检测方法,实现了沙门氏菌的定量检测。其中,适配体(ssDNA1)功能化的35 nm的GNPs作为捕获探针;Cy3修饰的互补链(ssDNA2)功能化的15 nm的GNPs作为信号探针。拉曼信号探针与捕获探针经互补的ssDNAs的杂交,形成不对称的GNDs。沙门氏菌目标物加入后与适配体专一性结合,从二聚体复合物中解离,因此,拉曼信号强度下降。最后得到沙门氏菌的检测范围在102~107 CFU/mL,最低检测限为35 CFU/mL。该检测方法具有高灵敏、稳定、均一的优点,且其检测时间也相对较短。
2.4 电化学检测
电化学生物传感器具有操作简单、灵敏度高、携带方便、成本低廉等优点,已成为一种潜在的POCT分析平台,被广泛应用于沙门氏菌的定量检测中。其中,电化学阻抗法可用于检测分析物与固定在电极表面的探针分子相互作用后电极界面性质变化,可提供一种快速、灵敏和无损的分析平台。近年来,出现了多种利用电化学阻抗法检测沙门氏菌的研究报道。GE等[33]通过结合诱导GNPs表面核酸适配体的取代以及滚环扩增(Rolling Circle Amplification, RCA),建立了一种超高灵敏的电化学阻传感检测方法,并将其用于沙门氏菌的定量检测中。沙门氏菌与核酸适配体的异质识别和特异性结合,会释放引物结合部分,导致在核酸适配体传感器表面锚定多个圆形模板;RCA反应产生具有多个串联重复序列的长DNA分子产物,这些产物可以与适配体传感器表面的生物素化的检测探针杂交,以输出酶放大的电化学信号。该方法得到沙门氏菌的线性检测范围为2×101~2×108 CFU/mL,最低检测限为16 CFU/mL。该检测方法不需要精密的温度循环仪器,具有快速、稳健、低成本、高灵敏、高专一性的优点。
Muniandy等[34]建立了一种基于还原的氧化石墨烯二氧化钛(Reduced Graphene Oxide Titanium Dioxider,GO-TiO2)纳米复合材料的适配体传感器,并将其用于沙门氏菌的定量检测。通过静电作用,无标记的适配体被固定于rGO-TiO2纳米复合材料表面,当DNA适配体与沙门氏菌细胞结合后,会对电极表面电子的转移产生阻碍;电极表面电导率的改变通过电分析方法进行表征;最后得到沙门氏菌的检测范围为101~108 CFU/mL,检测时间约1 h。该方法具有高灵敏、高选择性、快速、稳健的优点。
另外,Ranjbar等[35]建立了一种基于纳米多孔金(Nano-porous Gold, NPG)作为底物的新型鼠伤寒沙门氏菌电化学传感检测方法。采用金铜合金,通过电化学方法在Au/GCE表面合成NPG;硫醇功能化适配体通过自组装被有效的连接到NPG/Au/GCE表面;最后得到鼠伤寒沙门氏菌的线性检测范围为6.5×102~6.5×108 CFU/mL,定量限检测限为65 CFU/mL,最低检测限为1 CFU/mL。该适配体生物传感器具有高灵敏、高选择性、高重现性的优点,该方法具有可区分活菌细胞和死菌细胞的重要优势。
此外,无标记的方法也被应用于电化学核酸适配体生物传感方法的构建中。如Sheikhzadeh等[36]研制了一种基于聚[吡咯-3-羧基-吡咯]共聚物和适配体的无标记电化学阻抗生物传感器,并用于鼠伤寒沙门氏菌的检测。适配体与沙门氏菌的相互作用对聚[吡咯-3-羧基吡咯]共聚物内在的共轭及其随后的电性能的影响,通过阻抗进行测定。该方法得到沙门氏菌的检测范围为102~108CFU/mL,定量检测限为100 CFU/mL,最低检测限为3 CFU/mL。该无标记电化学核酸适配体生物传感检测方法,简化了实验过程,具有简单、快速(约45 min)、低成本、高灵敏的优点。
3 结语
综上所述,基于比色、荧光、SERS、电化学等的适配体生物传感技术已经引起人们的极大关注,并已被广泛应用于沙门氏菌的定量检测中。与沙门氏菌传统检测方法比较,适配体生物传感技术具有高灵敏、高专一性、快速等优点。目前,比色和荧光检测法是适配体生物传感检测中应用最广泛的方法。在荧光传感检测方法中,多种新型纳米材料,如量子点、碳点、上转换荧光纳米粒子等,都被广泛应用于沙门氏菌的定量检测中。其中,不同发射波长的量子点还被用于多种食源性致病菌的高通量检测中。虽然这些方法具有特异优势,但也存在需要对适配体进行标记的弊端。因而,又出现了多种免标记适配体传感检测方法的建立。
核酸适配体生物传感技术在沙门氏菌定量检测中存在着巨大挑战。首先,样品基质的复杂性。目标物的提取和纯化相对比较复杂繁琐,且适配体会非特异性地与样品基质结合,导致错误或不精确的结果的出现。其次,食源性致病菌的多样性。样品基质中往往会存在多种食源性致病菌,而目前现有的适配体生物传感检测方法的检测对象还比较单一,因此高通量检测也是目前的一大挑战。最后,目前应用于沙门氏菌定量检测的核酸适配体生物传感技术,还仅限于实验室内进行检测,不能实现实时实地的快速检测。因此,开发与这些生物传感技术相匹配的便携式检测仪器,实现沙门氏菌的实时实地快速检测是另一重大挑战。
[1]WUX L,WANGWB,LIU L Q,KUANGH,XU C L.Monoclonal antibodybased cross-reactive sandwich ELISA for the detection of Salmonella spp in milk samples[J].Analytical Methods,2015,7(21):9047-9053.doi:10.1039/C5AY01923K.
[2]满燕,吕雪飞,张玉奎,庆宏,邓玉林.核酸适配体及其在生物医学研究中的应用[J].航天医学与医学工程,2013,26(6):485-490.MAN Y, LV X F,ZHANG Y K,QING H, DENG Y L.Nucleic acid aptamers and their applications in biomedical research [J].Aerospace Medicine and Medical Engineering,2013,26(6):485-490.
[3]DUAN N, WU S J, CHEN X J, HUANG Y K, XIA Y, MA X Y, WANG Z P.Selection and characterization of aptamers against Salmonella typhimurium using whole-bacterium systemic evolution of ligands by exponential enrichment (SELEX)[J].Journal of Agricultural and Food Chemistry,2013,61(13):3229-3234.doi:10.1021/jf400767d.
[4]RYUL H S,LEE S W.In vitro selection of RNA aptamer specific to Salmonella typhimurium[J].Journal of microbiology and biotechnology,2013,23(6) 878-884.doi:10.4014/jmb.1212.12033.
[5]WANG L J,WANG R H,CHEN F,JIANG T S,WANG H,MICHAEL S,WEI H, LI Y B.QCM-basedaptamer selection and detection of Salmonella typhimurium[J].Food Chemistry,2017,221:776-782.doi:10.1016/j.foodchem.2016.11.104.
[6]MA X,SONG L,ZHOU,XIA Y,WANG Z.A novel aptasensor for the colorimetric detection of S.typhimurium based on gold nanoparticles[J].International Journal of Food Microbiology,2017,245:1-5.doi:10.1016/j.ijfoodmicro.2016.12.024.
[7]WU W H, LI M,WANG Y, OUYANG H X, WANG L,LI C X, CAO Y C, MENG Q H,LU J X.Aptasensors for rapid detection of Escherichia coli O157:H7 and Salmonella typhimurium[J].Nanoscale Research Letters,2012, 7(1):1-7.doi:10.1186/1556-276X-7-658.
[8]YUAN J,TAO Z,YU Y,MA X,XIAY,WANG L,WANG Z.A visual detection method for Salmonella Typhimurium based on aptamer recognition and nanogold labeling[J].Food Control,2014,37:188-192.doi:10.1016/j.foodcont.2013.09.046.
[9]MANY, REN J, LI B, JIN X, PAN L.A simple, highly sensitive colorimetric immunosensor for the detection of alternariol monomethyl ether in fruit by non-aggregated gold nanoparticles[J].Analytical and Bioanalytical Chemistry, 2018, 410(28):7511-7521.doi:10.1007/s00216-018-1369-z.
[10]DUAN N,XU B,WU S,WANG Z.Magnetic nanoparticlesbased aptasensorusing gold nanoparticles as colorimetricprobes for thedetection of Salmonella typhimurium[J].Analytical Sciences,2016,32(4):431-436.doi:10.2116/analsci.32.431.
[11]PARK J Y, JEONG H Y, KIM M I,PARK T J.Colorimetric detection system for Salmonella typhimurium based on peroxidase-like activity of magnetic nanoparticles with DNA aptamers[J].Journal of Nanomaterials,2015 (1):1-9.doi:10.1155/2015/527126.
[12]WU S,DUAN N,QIU Y,LI J,WANG Z.Colorimetric aptasensor for the detection of Salmonella enterica serovar typhimurium using ZnFe2O4-reduced graphene oxide nanostructures as an effective peroxidase mimetics[J].International Journal of Food Microbiology,2017,261:42-48.doi:10.1016/j.ijfoodmicro.2017.09.002.
[13]ZHU C Q,HONG Y,XIAO Z,ZHOU Y,JIANG Y,HUANG M,XU X L,ZHOU G H.Colorimetric determination of Salmonella typhimurium based on aptamer recognition[J].Analytical Methods,2016,8(35):6560-6565.doi:10.1039/c6ay01918h.
[14]WANG X L,NIAZIS, YUKUN H,SUN W J,WU S J,DUAN N,HUN X,WANG Z P.Homogeneous time-resolved FRET assay for the detection of Salmonella typhimurium using aptamer-modified NaYF4:Ce/Tb nanoparticles and a fluorescent DNA label[J].Microchimica Acta,2017,184(10):4021-4027.doi:10.1007/s00604-017-2399-5.
[15]DUAN N, SUN W, WU S, LIU L, HUN X.,WANG Z.Aptamer-based F0F1-ATPase biosensor for Salmonella typhimurium detection[J].Sensors and Actuators B:Chemical,2018,255:2582-2588.doi:10.1016/j.snb.2017.09.064.
[16]ZHANG Y, LUO F F, ZHANG Y T, ZHU L Q, LI Y, ZHAO S L, HE P G,WANG Q J.A sensitive assay based on specific aptamer binding for the detection of Salmonella enterica serovar Typhimurium in milk samples by microchip capillary electrophoresis[J].Journal of Chromatography A,2018,1534:188-194.doi:10.1016/j.chroma.2017.12.054.
[17]CHEN M Y, CHEN Z Z, CAI L, LIN Y, PANG D W, TANG H W.Indirect immunofluorescence detection of E.coli O157:H7 with fluorescent silica nanoparticles[J].Biosensors and Bioelectronics,2015,66:95-102.doi:10.1016/j.bios.2014.11.007.
[18]HAMULACL A,LEX C,LIX F.DNA aptamers binding to multiple prevalent M-types of streptococcus pyogenes[J].Analytical Chemistry,2011,83(10):3640-3647.doi:10.1021/ac200575e.
[19]XU L.A fluorescent aptasensorcoupled withnanobeadbased immunomagneticseparation for simultaneous detection of four foodbornepathogenic bacteria[J].Transactions of the ASABE,2015,58(3):891-906.doi:10.13031/trans.58.11089.
[21]QU Q,ZHU A W,SHAO X L,SHI G Y,TIAN Y.Development of a carbon quantum dots-based fluorescent Cu2+ probe suitable for living cell imaging[J].Chemical Communications,2012,48(44):5473-5475.doi:10.1039/c2cc31000g.
[21]WANG R J,XU Y,ZHANG T,JIANG Y.Rapid and sensitive detection of Salmonella typhimurium using aptamer-conjugated carbon dots as fluorescence probe[J].Analytical Methods,2015,7(5):1701-1706.doi:10.1039/C4AY02880E.
[22]GU B,ZHANG Q.Recent advances on functionalized upconversionnanoparticles for detection of small molecules and ions in biosystems[J].Advanced Science,2018,5(3):1700609-n/a.doi:10.1002/advs.201700609.
[23]WU S J, DUAN N, SHI Z, FANG C C,WANG Z P.Simultaneous aptasensor for multiplex pathogenic bacteria detection based on multicolor upconversionnanoparticles labels[J].Analytical Chemistry,2014,86(6): 3100-3107.doi:10.1021/ac404205c.
[24]DUAN N,WU S,YU Y, MA X, XIA Y, CHEN X, HUANG Y, WANG Z.A dual-color flow cytometry protocol for the simultaneous detection of Vibrio parahaemolyticus and Salmonella typhimurium using aptamer conjugated quantum dots as labels[J].Analytica Chimica Acta,2013,804: 151-158.doi.org/10.1016/j.aca.2013.09.047.
[25]SRINIVASAN S,RANGANATHAN V,DEROSAMC,MURARI B M.Label-free aptasensors based on fluorescent screening assays for the detection of Salmonella typhimurium[J].Analytical Biochemistry,2018,559:17-23.doi:10.1016/j.ab.2018.08.002.
[26]ZHANG P, LIU H, LI X, MAS, MEN S, WEI H, CUI J, WANG H.A label-free fluorescent direct detection of live Salmonella typhimurium using cascade triple trigger sequences-regenerated strand displacement amplification and hairpin template-generated-scaffolded silver nanoclusters[J].Biosensors and Bioelectronics,2017,87: 1044-1049.doi:10.1016/j.bios.2016.09.037.
[27]CAI WY, WANG X, YAN Y X.Controllable fabrication and sensitive detection based on SERS substrates with Au nanocubes coated Fe3O4[J].Materials Research Bulletin,2014,52:1-5.doi:10.1016/j.materresbull.2013.12.046.
[28]HUAN T N, KIM S, TUONG P V, CHUNG H.Au-Ag bimetallic nanodendrite synthesized via simultaneous co-electrodeposition and its application as a SERS substrate[J].RSC Advances,2014,4(8):3929-3933.doi:0.1039/C3RA44916E.
[29]QUYENTT B,CHANG C C,SUW N, UEN Y H, PAN C J,LIU J Y,RICK J,LIN K Y,HWANG B J.Self-focusing Au@SiO2 nanorods with rhodamine 6G as highly sensitive SERS substrate for carcinoembryonic antigen detection[J].Journal of Materials of Chemistry B,2014,2(6):629-636.doi:10.1039/C3TB21278E.
[30]ZHANG H,MA X, LIU Y, DUANN, WU S, WANGZ,XU B.Gold nanoparticles enhanced SERS aptasensor for the simultaneous detection of Salmonella typhimurium and Staphylococcus aureus[J].Biosensors and Bioelectronics,2015,74: 872-877.doi:10.1016/j.bios.2015.07.033.
[31]MA X,XU X, XIA Y, WANG Z.SERS aptasensor for Salmonella typhimurium detection based on spiny gold nanoparticles[J].Food Control,2018, 84: 232-237.doi:10.1016/j.foodcont.2017.07.016.
[32]XU X,MA X,WANG H,WANG Z.Aptamer based SERS detection of Salmonella typhimurium using DNA-assembled gold nanodimers[J].Microchimica Acta, 2018,185(7):325.doi: 10.1007/s00604-018-2852-0.
[33]GE C,YUAN R,L Y, YANG J L,ZHANG H W,LI X L, NIANW Q, GY I.Target-induced aptamer displacement on gold nanoparticles and rolling circle amplification for ultrasensitive live Salmonella typhimurium electrochemical biosensing[J].Journal of Electroanalytical Chemistry,2018,826:174-180.doi:10.1016/j.jelechem.2018.07.002.
[34]MUNIANDY S, TEH S J, APPATURIJ N, THONGKL, LAIC W, IBRAHIM F, LEO B F.A reduced graphene oxide-titanium dioxide nanocomposite based electrochemical aptasensor for rapid and sensitive detection of Salmonella enterica[J].Bioelectro chemistry,2019,127:136-144.doi:10.1016/j.bioelechem.2019.02.005.
[35]RANJBAR S, SHAHROKHIAN S,NURMOHAMMAD F.Nanoporous gold as a suitable substrate for preparation of a new sensitive electrochemical aptasensor for detection of Salmonella typhimurium[J].Sensors and Actuators B: Chemical, 2018, 255:1536-1544.doi:10.1016/j.snb.2017.08.160.
[36]SHEIKHZADEH E, CHAMSAZ M, TURNER A, JAGER E, BENI V.Label-free impedimetric biosensor for Salmonella Typhimurium detection based on poly [pyrrole-co-3-carboxyl-pyrrole]copolymer supported aptamer[J].Biosensors and Bioelectronics,2016, 80: 194-200.doi:10.1016/j.bios.2016.01.057.
[37]WANG R J,XU Y,ZHANG T,JIANG Y.Rapid and sensitive detection of Salmonella typhimurium using aptamer-conjugated carbon dots as fluorescence probe[J].Analytical Methods,2015,7(5):1701-1706.doi:10.1039/C4AY02880E.
[38]KURT H, YÜCE M, HUSSAIN B, BUDAK H.Dual-excitation upconverting nanoparticle and quantum dot aptasensor for multiplexed food pathogen detection[J].Biosensors and Bioelectronics,2016,81:280-286.doi:10.1016/j.bios.2016.03.005.
[39]YU S,TANG Y,YAN M,AGUILAR Z P,LAI W,XU H.A fluorescent cascade amplification method for sensitive detection of Salmonella based on magnetic Fe3O4 nanoparticles and hybridization chain reaction[J].Sensors and Actuators B: Chemical, 2019,279:31-37.doi:10.1016/j.snb.2018.09.091.
[40]LABIB M, ZAMAY A S, KOLOYSKAYA O S, RESHETNEVA I T,ZAMAY G S, KIBBEE R J, SATTAR S A, ZAMAY T N, BEREZOVSKI M V.Aptamer-based viability impedimetric sensor for bacteria[J].Analytical Chemistry,2012, 84(21): 8966-8969.doi:10.1021/ac203412m.
[41]JIA F, DUAN N, WU S J, DAI R T, WANG Z.P, LI X M.Impedimetric Salmonella aptasensor using a glassy carbon electrode modified with an electrodeposited composite consisting of reduced graphene oxide and carbon nanotubes[J].Microchimica Acta,2016,183(1): 337-344.doi:10.1007/s00604-015-1649-7.
[42]DUAN N,CHANG B,ZHANG H,WANG Z,WU S.Salmonella typhimurium detection using a surface-enhanced Raman scatteringbased aptasensor[J].International Journal of Food Microbiology,2016,218:38-43.doi:10.1016/j.ijfoodmicro.2015.11.006.