【研究意义】在植物逆境胁迫应答过程中,转录因子通过与启动子中的顺式作用元件特异性结合,调控靶基因的表达,从而可以提升植物的抗逆性[1]。以往植物抗逆育种研究通常只针对单一基因进行,由于植物逆境胁迫应答是一个非常复杂的过程,需要大量基因协同参与,单一基因对植株抗逆性状提升往往不明显,因此植物抗逆育种研究需要从多基因、多性状和多通路入手。【前人研究进展】AP2/ERF(APETALA 2/ethylene-responsive element binding factor)转录因子家族是植物最大的转录因子家族之一,根据结构域特征可以将AP2/ERF转录因子家族蛋白分为AP2、ERF、DREB、RAV亚家族。其中,RAV亚家族蛋白的特征是同时具有AP2和B3保守结构域,相比其他AP2/ERF转录因子亚家族,RAV家族成员数量较为有限,但已有研究表明RAV转录因子可以在乙烯[2]、油菜素内酯[3]和生物和非生物胁迫响应[4]过程中发挥重要作用。作为细胞膜的组成部分,膜脂通常可以在信号转导过程中发挥重要作用,而膜脂水解酶磷脂酰肌醇特异性磷脂酶C(phosphatidylinositolspecific phospholipase C,PI-PLC。以下简称PLC)家族蛋白是脂信号通路的重要成员,在植物中,PLC由EF手型结构域、X结构域、Y结构域和C2结构域组成,PLC通过对膜脂底物的水解以及相关代谢产物的合成,调控磷脂酰肌醇4,5二磷酸(PIP2)、三磷酸肌醇(IP3)、二酰甘油(DAG)、磷脂酸(PA)等信使类物质的含量以适应植物对生物胁迫及非生物胁迫的应答,其中OsPLC1参与介导钙离子信号调控叶片钠离子累积[5];OsPLC4通过水解膜脂底物PI生成的小分子信使参与钙离子和磷脂酸信号通路,进而提升水稻幼苗的耐盐及耐旱性[6];番茄PLC4和PLC6则被证实参与植物防卫及免疫信号通路[7]。由此可见,PI-PLC所参与的细胞活动与RAV转录因子类似,但目前尚未有研究证实二者在相关信号通路中有直接关联。【本研究切入点】本课题组前期对杨树PI-PLC家族基因鉴定过程中,发现了一个可编码RAV和PLC结构域的特殊基因。【拟解决的关键问题】利用生物信息学分析工具,分析该基因编码氨基酸的理化性质、保守结构域、磷酸化位点、亚细胞定位、信号肽、跨膜区、启动子序列、蛋白质功能网络、KEGG通路及三维结构,以期为该基因克隆及所参与信号通路分析提供参考。
1 材料与方法
1.1 毛果杨RAV/PLC基因和蛋白序列的获得
通过Phytozome数据库(https://phytozome.jgi.doe.gov/pz/portal.html#!search?show=BLAST&method=Org_Ptrichocarpa)检索“PLC”,查找并获得毛果杨RAV/PLC基因序列(Potri. 018G109200)。
1.2 分析方法
将RAV/PLC基因序列输入NCBI BLAST进 行 分 析(https://blast.ncbi.nlm.nih.gov/Blast.cgi);利用ExPaSy的ProtParam软件(https://web.expasy.org/protparam/)预测蛋白质分子量、理论等电点;通过Interpro(http://www.ebi.ac.uk/interpro/)进行蛋白保守区分析;使用Mega 7.0构建序列进化树;通过NetPhos3.1预测其磷酸化位点[8];利用 SignalP 3.0[9]预测其信号(http://www.cbs.dtu.dk/services/SignalP-3.0/); 采 用Plant-mPLoc[10-12](http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)进行亚细胞定位预测;通过 TMHMM Server 2.0[13](http://www.cbs.dtu.dk/services/TMHMM/)和 TMpred[14](https://embnet.vital-it.ch/software/TMPRED_form.html)预测其跨膜区;利用 PlantCARE[15](http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析其启动子序列;借助String软件(https://string-db.org/cgi/input.pl?sessionId=mWhORPeJ7Wtd&input_page_active_form=single_sequence)预测蛋白质功能网络并通过KEGG Blast分析该蛋白生物学功能;利用 I-TASSER[16-18](https://zhanglab.ccmb.med.umich.edu/I-TASSER/)进行同源建模预测三维结构。
2 结果与分析
2.1 RAV/PLC理化性质预测及结构域分析
在Phytozome中以“PLC”为关键字进行检索Populus trichocarpa v3.0数据库,结果包含一个可编码AP2结构域(45~107位氨基酸)、B3结构域(167~277位氨基酸)和PLC-X结构域(476~542位氨基酸)的基因(图1A),登录号为Potri. 0180G109200,根据结构特征将其命名为RAV/PLC基因。RAV/PLC基因定位于18号染色体,基因全长2 866 bp,其ORF区域长度为1 650 bp,编码氨基酸549个,含有4个外显子及3个内含子。ExPaSy的ProtParam预测结果表明,RAV/PLC编码的氨基酸分子量为62.72338 ku;理论等电点为9.16;其分子式为C2771H4372N782O832S24,不稳定系数为48.70,说明该蛋白为不稳定蛋白。
NCBI Blast检索确认RAV/PLC基因序列与 POP043-C12(GenBank:AC215840.1) 部 分序列比对完全一致,选取BLAST结果中得分较高序列构建序列进化树,结果表明除Potri.0180G109200.1、Potri. 0180G109200.2两个转录本外,RAV/PLC与XM_024590588.1的亲缘关系较为接近(图1B),这主要是由于该基因也含有PLC-X结构域的编码序列,但XM_024590588.1在RAV结构域之后提前出现了终止密码子,使得该基因ORF区并不包含PLC结构域。
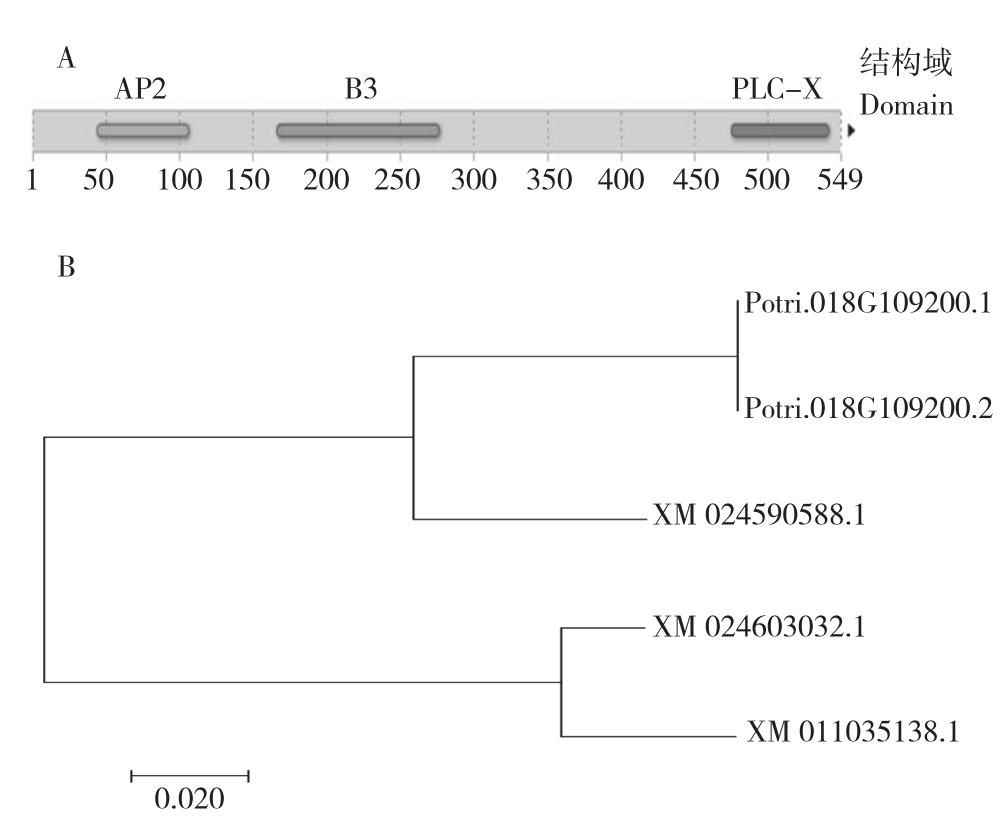
图1 RAV/PLC结构域预测及进化树
Fig. 1 Domain prediction and the constructed phylogenetic tree for RAV/PLC
A:RAV/PLC结构域分布;B:序列进化树
A: Domain distribution for RAV/PLC; B: Phylogenetic tree
Phytozome数据库中RAV/PLC基因在杨树中表达情况如图2(彩插一)所示,该基因在杨树芽、叶、根、茎中均有表达,在茎中整体表达量较高,芽中表达量次之,叶和根部相对表达量较少。
2.2 RAV/PLC信号肽、亚细胞定位、磷酸化位点、跨膜区预测
通过SignlP 3.0对RAV/PLC进行信号肽预测,结果表明RAV/PLC不含有信号肽。由于RAV转录因子家族与PLC家族蛋白行使功能不同,其定位位置通常存在差异,因此分别对RAV/PLC N端(1~300位氨基酸,包含RAV结构域)和C端(450~549位氨基酸,包含PLC-X结构域)进行亚细胞定位预测,预测结果表明,RAV/PLC N端定位于细胞核,而C端定位于细胞膜。通过NetPhos 3.1预测得到14个潜在的磷酸化位点,各位点信息如表1所示。通过TMHMM Server 2.0和TMpred预测其跨膜区并确认RAV/PLC N端、C端之间存在一个跨膜区(膜内到膜外),其中TMHMM Server 2.0预测跨膜区位于第455~477位氨基酸(图3A中间区域,彩插一),TMpred预测跨膜区位于第455~483位氨基酸(图3B分值高于500区域,彩插一)。

图2 RAV/PLC在杨树中表达情况热图
Fig. 2 Heatmap displaying the expression patterns of RAV/PLC in poplar
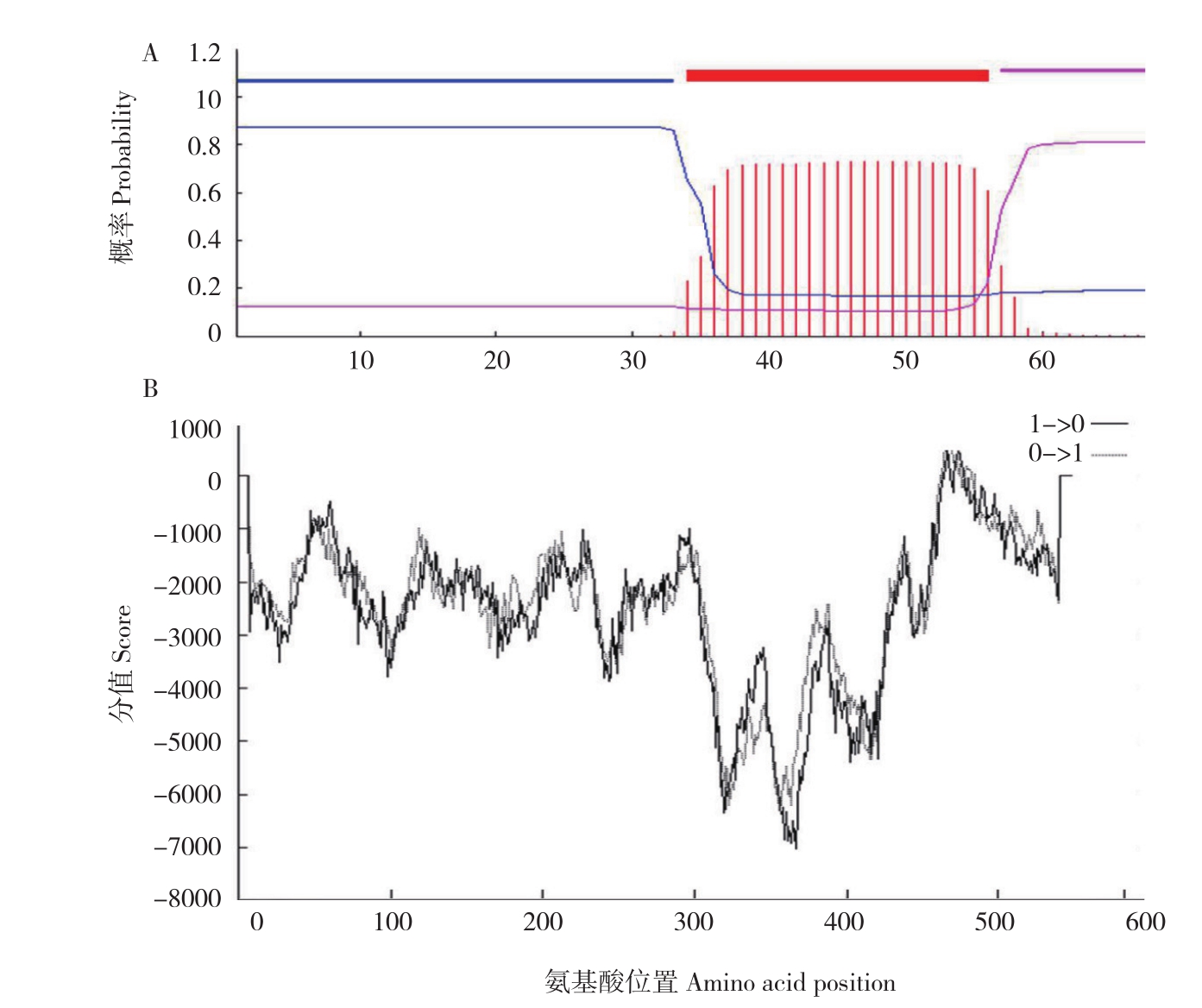
图3 RAV/PLC跨膜区预测结果
Fig. 3 Transmembrane regions prediction results for RAV/PLC
A:TMHMM Server 2.0预测结果,蓝色为膜内、粉色为膜外、红色为跨膜部分;
B:TMpred预测结果
A: Results predicted by TMHMM Server 2.0, the inside region is marked by blue, the outside region is marked by pink and the transmembrane region is marked by red;
B: Results predicted by TMpred

图4 RAV/PLC蛋白网络和同源建模
Fig. 4 Protein functional association networks and homology modeling for RAV/PLC
A:String预测RAV/PLC蛋白功能网络;
B:I-TASSER预测RAV/PLC 3D结构
A: RAV/PLC protein networks predicted by String;
B: 3D structure of RAV/PLC predicted by I-TASSER
表1 RAV/PLC磷酸化位点预测结果
Table 1 Prediction results of phosphorylation sites of RAV/PLC
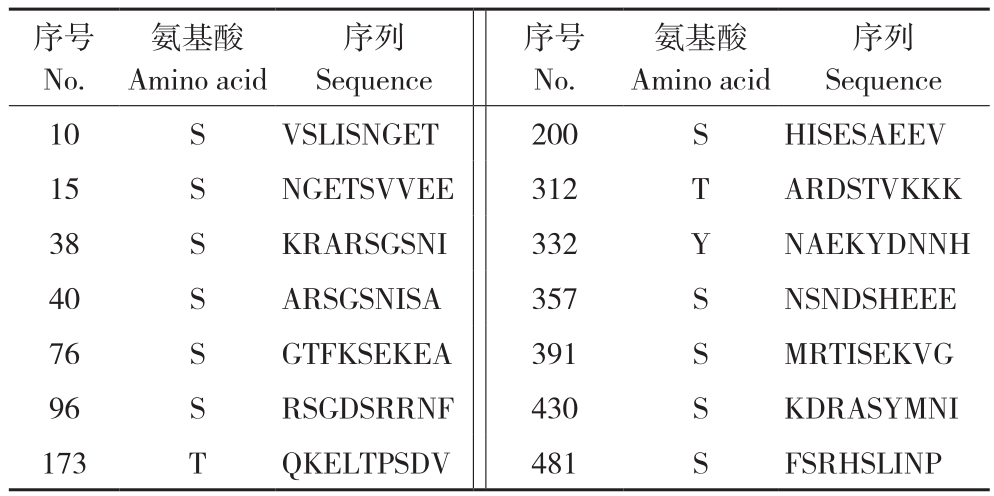
序号No.序列Sequence 10 S VSLISNGET 200 S HISESAEEV 15 S NGETSVVEE 312 T ARDSTVKKK 38 S KRARSGSNI 332 Y NAEKYDNNH 40 S ARSGSNISA 357 S NSNDSHEEE 76 S GTFKSEKEA 391 S MRTISEKVG 96 S RSGDSRRNF 430 S KDRASYMNI 173 T QKELTPSDV 481 S FSRHSLINP氨基酸Amino acid序列Sequence序号No.氨基酸Amino acid
2.3 RAV/PLC启动子分析、蛋白质功能网络预测
将RAV/PLC基因上游3 000 bp序列输入PlantCARE进行顺式元件预测,结果如表2所示。RAV/PLC基因启动子中包含多个重要的顺式作用元件,其中TATA-box为转录起始点上游-30~-50 bp核心启动元件;CAAT-box是常见的转录效率调控元件;AE-box、I-box、Box 4、GATA-motif、MRE、GA-motif、TCT-motif均与光响应有关;GARE-motif、TGACG-motif、TCA-element分别与赤霉素、茉莉酸甲酯、水杨酸等植物激素应答相关;TC-rich repeats可以参与植物防卫反应和胁迫应答;MBS顺式元件与干旱诱导调控有关,同时MBS和MRE均为MYB转录因子的结合位点。以上结果表明,RAV/PLC基因表达可能受到上述条件诱导,并由MYB类转录因子直接参与调控。
将RAV/PLC氨基酸序列输入String分析其蛋白质相互作用网络,结果表明,RAV/PLC与String数据库中POPTR_0018s11780.1蛋白序列有极高同源性,与RAV/PLC序列相比,POPTR_0018s11780.1缺失了第207~479位氨基酸。作用网络预测结果表明,该蛋白可能与毛果杨 MYB103(POPTR_0006s12400.1)、WRKY49(POPTR_0006s08730.1,POPTR_0016s10610.1)、ABI5(POPTR_0006s08300.1)、DXS2(POPTR_0007s09490.1)存在功能关联(图4A,彩插一),RAV/PLC及网络中蛋白GO(Gene ontology)功能注释结果见表3,KEGG通路分析表明该基因符合RAV转录因子特征(KO entry :K09287),RAV/PLC可能与上述蛋白协同在相应途径中发挥其生物学功能。
表2 RAV/PLC的PlantCARE预测结果
Table 2 PlantCARE prediction results of RAV/PLC
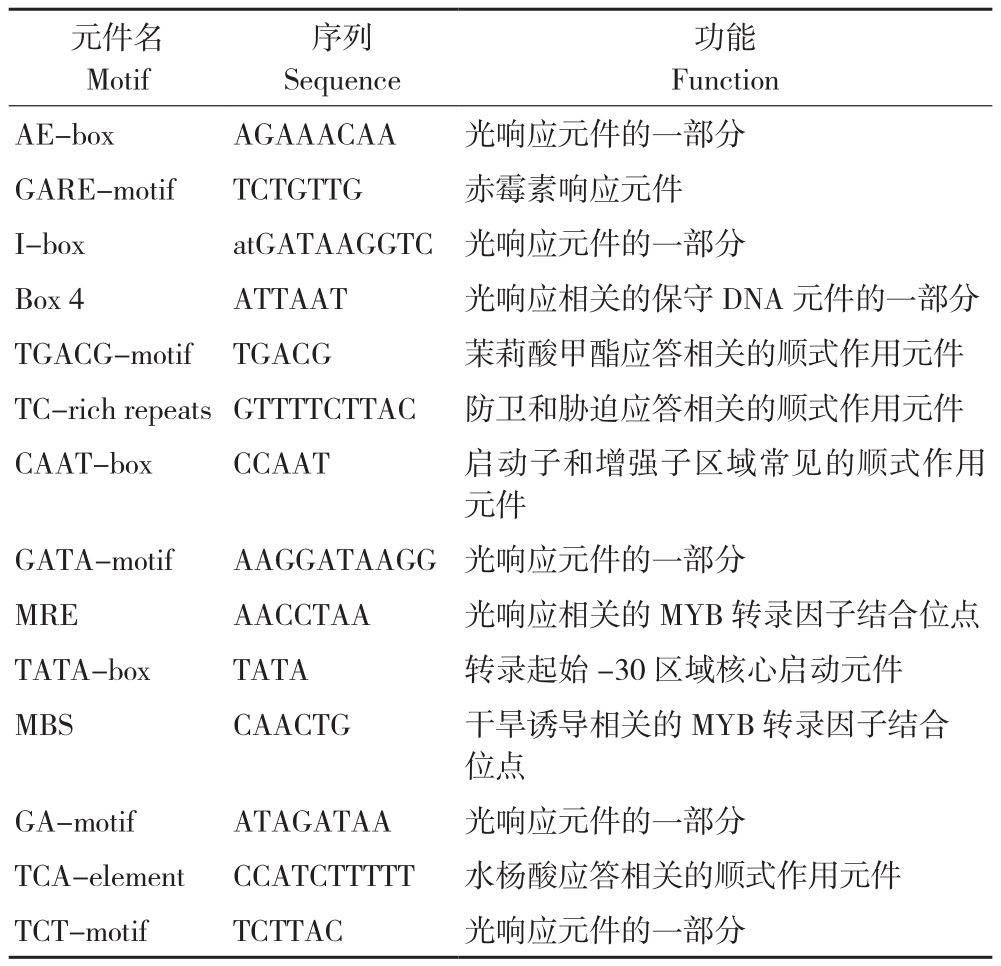
功能Function AE-box AGAAACAA 光响应元件的一部分GARE-motif TCTGTTG 赤霉素响应元件I-box atGATAAGGTC 光响应元件的一部分Box 4 ATTAAT 光响应相关的保守DNA元件的一部分TGACG-motif TGACG 茉莉酸甲酯应答相关的顺式作用元件TC-rich repeatsGTTTTCTTAC 防卫和胁迫应答相关的顺式作用元件CAAT-box CCAAT 启动子和增强子区域常见的顺式作用元件GATA-motif AAGGATAAGG 光响应元件的一部分MRE AACCTAA 光响应相关的MYB转录因子结合位点TATA-box TATA 转录起始-30区域核心启动元件MBS CAACTG 干旱诱导相关的MYB转录因子结合位点GA-motif ATAGATAA 光响应元件的一部分TCA-element CCATCTTTTT 水杨酸应答相关的顺式作用元件TCT-motif TCTTAC 光响应元件的一部分元件名Motif序列Sequence
2.4 同源建模
利用I-TASSER对RAV/PLC三维结构进行同源建模,其建模结果如图4B(彩插一)所示。
表3 RAV/PLC蛋白网络基因功能注释
Table 3 GO function annotations for RAV/PLC networks
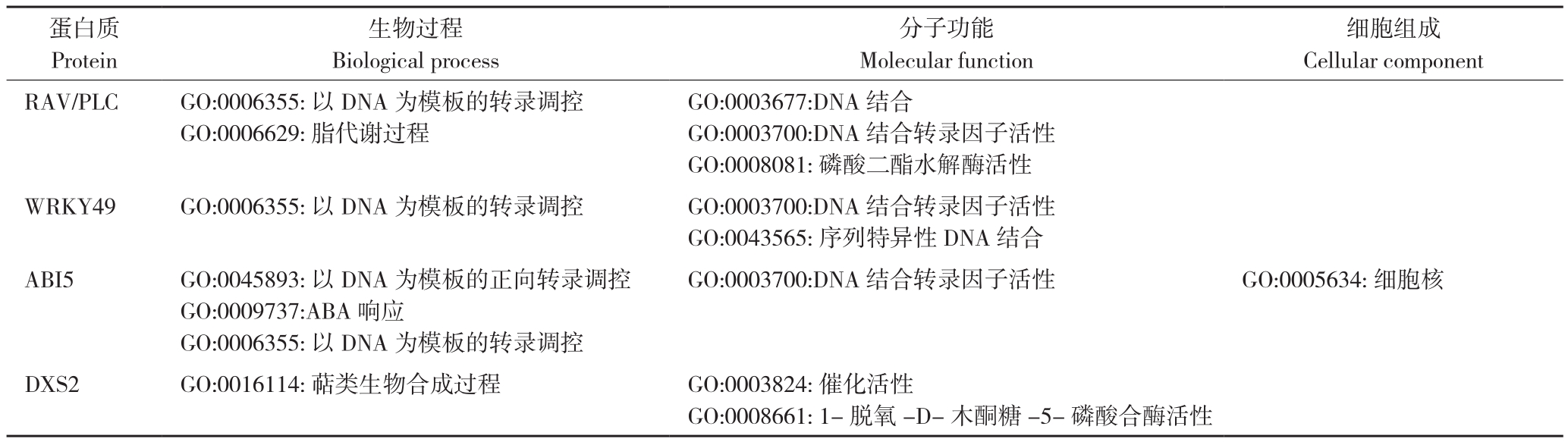
细胞组成Cellular component RAV/PLC GO:0006355:以DNA为模板的转录调控GO:0006629:脂代谢过程蛋白质Protein生物过程Biological process分子功能Molecular function GO:0003677:DNA结合GO:0003700:DNA结合转录因子活性GO:0008081:磷酸二酯水解酶活性WRKY49 GO:0006355:以DNA为模板的转录调控 GO:0003700:DNA结合转录因子活性GO:0043565:序列特异性DNA结合ABI5 GO:0045893:以DNA为模板的正向转录调控GO:0009737:ABA响应GO:0006355:以DNA为模板的转录调控GO:0003700:DNA结合转录因子活性 GO:0005634:细胞核DXS2 GO:0016114:萜类生物合成过程 GO:0003824:催化活性GO:0008661: 1-脱氧-D-木酮糖-5-磷酸合酶活性
3 讨论
杨树具有分布广、适应性强、生长速度快的特点,是我国重要的造林和用材树种,也是基因工程研究中的模式植物,筛选杨树抗逆基因可以为优良适应性树种选育提供分子依据。在胁迫条件下,转录因子与顺势作用元件结合,启动相关基因表达以提升植株对逆境的适应能力。与此同时,在感知逆境信号后,细胞膜可以通过脂信号向胞内进一步传递,以启动下游相关代谢通路应答。RAV是AP2/ERF转录因子亚家族成员,研究表明RAV可以在植物激素处理和生物非生物胁迫应答过程中发挥作用,尽管已有大量研究探讨杨树AP2/ERF转录因子功能,但相比其他亚家族,RAV亚家族成员数量较少,对其在杨树中的功能探讨较为有限[19]。而PI-PLC作为脂信号的重要水解酶,其参与的细胞活动与RAV转录因子相似,但并未有文献报道证实二者有直接关系。
本研究发现了一个可编码PLC-X结构域、AP2结构域和B3结构域的基因RAV/PLC,Interpro确认其编码氨基酸序列符合RAV转录因子和PLC-X催化结构域的特征,XM_024590588.1与RAV/PLC整体亲缘关系较为接近,但该基因ORF区在RAV结构域后出现提前终止,因而并不包含PLC结构域。RAV/PLC基因表达在茎部最为显著,在根部和叶部表达量相对有限。其编码肽段不含信号肽,含有14个潜在的丝氨酸、苏氨酸和酪氨酸磷酸化位点。对RAV/PLC N端(1~300位氨基酸包含RAV结构域)和C端(450~549位氨基酸包含PLC-X结构域)分别进行亚细胞定位预测,结果表明该肽段N端定位于细胞核,而C端定位于细胞膜。这一定位结果与RAV和PLC生物学功能相符合,一般转录因子定位在细胞核内发挥转录激活功能,而磷脂酶定位于细胞膜参与水解脂类底物。通过定位结果可以进一步推测,RAV结构域与PLC-X结构域之间可能存在一个跨膜区,TMHMM Server 2.0和TMpred均确认了这一跨膜区的存在,结合亚细胞定位可以预测RAV和PLC-X结构域分别位于核膜两侧,二者可能在特定条件下分别发挥其生物学功能。启动子序列分析表明,RAV/PLC基因启动子中包含TATA-box、CAAT-box等关键转录调控元件,同时包含光响应元件、植物激素应答元件、植物防卫和胁迫应答以及干旱调控元件,部分元件可以同MYB转录因子特异性结合,以上结果表明RAV/PLC基因表达可能在上述特定条件下诱导发生并受MYB转录因子家族成员直接调控。通过String软件分析RAV/PLC蛋白功能网络,Blast结果表明RAV/PLC与POPTR_0018s11780.1具有较高的同源性,序列比对证明二者属于不同转录本,与RAV/PLC相比POPTR_0018s11780.1缺失了第207~479位氨基酸,这一缺失包含了部分B3结构域和跨膜区序列,缺失部分可能与选择性剪切调控有关,但缺失后其GO功能注释结果并未受到影响。功能网络表明该蛋白可能与MYB103、WRKY49、ABI5、DXS2存在功能关联,RAV/PLC及其关联蛋白GO功能注释见表3,KO通路分析表明该基因与RAV信号通路相关。研究表明拟南芥MYB103基因在植物防卫反应激发子几丁质处理30 min后,其表达水平上调[20];TaWRKY49可以通过水杨酸、茉莉酸、乙烯以及ROS介导信号,对小麦条锈菌抗性进行负调控[21];ABI5是脱落酸介导的种子萌发及萌发后幼苗生长的关键调节子[22],它通常与 ABI3协同作用[23];在萜类生物合成反应MEP通路当中,DXS是前两步反应所需的酶之一,它也在整个通路中发挥重要调控作用[24]。由此可见,蛋白网络各成员功能与胁迫应答、激素响应有密切相关。同时利用I-TASSER获得了RAV/PLC编码蛋白的3D模型,对于RAV/PLC而言,绝大多数同源建模工具只会通过输入序列比对情况对同源序列部分进行预测,因此在此类预测工具中,只有RAV结构域和PLC结构域的3D模型会生成。相比之下,I-TASSER则可以对RAV/PLC完整序列进行建模。
本研究所预测RAV/PLC生物学功能主要基于相关生物信息学分析手段,对于亚细胞定位、跨膜区等相关预测结果仍需要实验验证;启动子序列中的核心调控元件仍需要进一步鉴定;基因表达模式、组织特异性表达规律、涉及的代谢通路等将是后续研究的主要内容。
4 结论
本研究发现的RAV/PLC基因编码氨基酸序列符合RAV转录因子和PLC催化活性区域的结构特征,通过生物信息学手段预测该序列N端定位于细胞核,C端定位于细胞膜,二者之间存在一个跨核膜区域。通过启动子序列分析和蛋白质功能网络预测RAV/PLC基因可能与光响应、植物激素、生物和非生物胁迫应答相关。本研究为该基因克隆及参与信号通路预测提供参考。
[1] SCHWECHHEIMER C, BEVAN M. The regulation of transcription factor activity in plants[J]. Trend in Plant Science, 1998, 3(10):278-283.
[2] ALONSO J M, STEPANOVA A N, LEISSE T J, KIM C J, CHEN H,SHINN P, STEVENSON D K, ZIMMERMAN J, BARAJAS P, CHEUK R, GADRINAB C, HELLER C, JESKE A, KOESEMA E, MEYERS CC, PARKER H, PREDNIS L, ANSARI Y, CHOY N, DEEN H,GERALT M, HAZARI N, HOM E, KARNES M, MULHOLLAND C,NDUBAKU R, SCHMIDT I, GUZMAN P, AGUILAR-HENONIN L,SCHMID M, WEIGEL D, CARTER D E, MARCHAND T, RISSEEUW E, BROGDEN D, ZEKO A, CROSBY W L, BERRY C C, ECKER J R.Genome-wide insertional mutagenesis of Arabidopsis thaliana[J].Science, 2003, 301(5633):653-657.
[3] HU Y, WANG Y H, LIU X F, LI J Y. Arabidopsis RAV1 is downregulated by brassinosteroid and may act as a negative regulator during plant development[J]. Cell Research, 2004, 14:8-15.
[4] SOHN K H, LEE S C, JUNG H W, HONG J K, HWANG B K.Expression and functional roles of the pepper pathogen-induced transcription factor RAV1 in bacterial disease resistance, and drought and salt stress tolerance[J]. Plant Molecular Biology, 2006, 61(6):897-915.
[5] LI L, WANG F, YAN P, JING W, ZHANG C, KUDLA J, ZHANG W.A phosphoinositide-specific phospholipase C pathway elicits stressinduced Ca2+, signals and confers salt tolerance to rice[J]. New Phytologist, 2017, 214(3):1172-1187.
[6] DENG X J, YUAN S, CAO H S, LAM S M, SHUI G H, HONG Y Y, WANG X M. Phosphatidylinositol-hydrolyzing phospholipase C4 modulates rice response to salt and drought[J].Plant, Cell &Environment, 2019, 42(2):536-548.
[7] VOSSEN J H, ABD-EL-HALIEM A, FRADIN E F, VAN DEN BERG G C, EKENGREN S K, MEIJER H J, SEIFI A, BAI Y, TEN HAVE A,MUNNIK T, THOMMA B P, JOOSTEN M H. Identification of tomato phosphatidylinositol-specific phospholipase-C(PI-PLC) family members and the role of PLC4 and PLC6 in HR and disease resistance[J]. The Plant Journal, 2010, 62(2):224-239.
[8] BLOM N, GAMMELOFT S, BRUNAK S. Sequence- and structurebased prediction of eukaryotic protein phosphorylation sites[J].Journal of Molecular Biology, 1999,294(5): 1351-1362.
[9] BENDTSEN J D, NIELSEN H, VON HEIJNE G, BRUNAK S. Improved prediction of signal peptides: SignaIP 3.0[J]. Journal of Molecular Biology, 2004, 340(4):783-795.
[10] CHOU K C, SHEN H B. Plant-mPLoc: A top-down strategy to augment the power for predicting plant protein subcellular localization[J].PLoS One, 2010, 5(6):e11335.
[11] CHOU K C, SHEN H B. Cell-PLoc: a package of Web servers for predicting subcellular localization of proteins in various organisms[J].Nature Protocols, 2008, 3(2):153-162.
[12] CHOU K C, SHEN H B. Large-scale plant protein subcellular location prediction[J]. Journal of Cellular Biochemistry, 2007, 100(3):665-678.
[13] KROGH A, LARSSON B, HEIJNE G V, ERIK L L S. Predicting transmembrane protein topology with a hidden markov model:application to complete genomes[J]. Journal of Molecular Biology,2001, 305(3):567-580.
[14] HOFMANN K, STOEL W. TMbase—A database of membrane spanning proteins segments[J]. Biological Chemistry, 1993, 374(166).
[15] LESCOT M, DÉHAIS P, THIJS G, MARCHAL K, MOREAU Y, VAN DE PEER Y, ROUZÉ P, ROMBAUTS S. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research, 2002, 30(1):325-327.
[16] ROY A, KUCUKURAL A, ZHANG Y. I-TASSER: a unified platform for automated protein structure and function prediction[J]. Nature Protocols, 2010, 5(4):725-738.
[17] YANG J, YAN R, ROY A, XU D, POISSON J, ZHANG Y. The I-TASSER Suite: protein structure and function prediction[J]. Nature Methods, 2014, 12(1):7-8.
[18] YANG J, ZHANG Y. I-TASSER server: new development for protein structure and function predictions[J]. Nucleic Acids Research, 2015,43: 174-181.
[19] 赵金玲,姚文静,王升级,姜廷波,周博如.杨树AP2/ERF转录因子家族生物信息学分析[J].东北林业大学学报,2015(10):23-31.ZHAO J L, YAO W J, WANG S J, JIANG T B, ZHOU B R. AP2/ERF gene family in Populus trichocarpa by bioinformatics[J].Journal of Northeast Forestry University, 2015,43(10):21-29.
[20] LIBAULT M, WAN J, CZECHOWSKI T, UDVARDI M, STACEY G.Identification of 118 Arabidopsis transcription factor and 30 ubiquitinligase genes responding to chitin, a plant-defense elicitor[J].Molecular Plant-microbe Interactions, 2007, 20(8):900-911.
[21] WANG J, TAO F, TIAN W, GUO Z, CHEN X, XU X, SHANG H, HU X.The wheat WRKY transcription factors TaWRKY49 and TaWRKY62 confer differential high-temperature seedling-plant resistance to Puccinia striiformis f. sp. tritici[J]. PLoS One, 2017, 12(7):e0181963.
[22] DAI M, XUE Q, MCCRAY T, MARGAVAGE K, CHEN F, LEE J H,NEZAMES C D, GUO L, TERZAGHI W, WAN J, DENG X W, WANG H.The PP6 phosphatase regulates ABI5 phosphorylation and abscisic acid signaling in Arabidopsis[J]. Plant Cell, 2013, 25(2):517-534.
[23] LOPEZ-MOLINA L, MONGRAND S, MCLACHLIN D T, CHAIT B T, CHUA N H. ABI5 acts downstream of ABI3 to execute an ABA-dependent growth arrest during germination[J]. The Plant Journal,2002, 32(3):317-328.
[24] MAO J P, HE Z D, HAO J, LIU T Y, CHEN J H, HUANG S W.Identification, expression, and phylogenetic analyses of terpenoid biosynthesis-related genes in secondary xylem of loblolly pine(Pinus taeda L.) based on transcriptome analyses[J]. Peer J, 2019(7):e6124.