【研究意义】 大白口蘑(Tricholoma giganteum)又称为巨大口蘑、洛巴伊口蘑,是一种大型簇生的食、药用真菌,隶属于担子菌门、伞菌纲、伞菌目、口蘑科、口蘑属[1]。大白口蘑的营养价值极高,其粗蛋白和多糖含量分别是115.9 g/kg和365.9 g/kg,含有18种氨基酸,其中人体必需氨基酸74.4 g/kg,满足FAO/WHO最高蛋白质的需求[2]。大白口蘑含有丰富的矿物质元素,如锌、钾、钙、铁、钠、镁等[3];还具有防突变、增强免疫力、抗肿瘤、抗氧化、抗艾滋病毒、抗真菌、降血糖血脂血压和保护肝脏等功能[4-9]。大白口蘑是一种簇生的野生珍稀食药用菌,菇体肥大、菌肉鲜美、口感极佳,具有很高的食药用价值,而且不易褐变和腐烂,耐贮运性好,有很好的开发价值和应用前景[10]。大白口蘑属于高温品种,可在夏季市场短缺菇的情况下抢占市场,具有良好的发展前景[11]。韶关学院“食用菌生态循环栽培技术及产业化创新服务团队”在韶关学院校园内的荒地杂草丛中(旁有苦楝树和海南蒲桃树)发现 1 株疑似大白口蘑野生菌。本研究结合形态学特征与ITS序列分析鉴定其分类学地位,并筛选该菌株适宜的菌丝培养基,旨在丰富其种质资源,也为其驯化、栽培和育种提供依据和参考。【前人研究进展】 大白口蘑是由法国真菌学家Hiem发现于非洲,于1970年定名,被称为“菌中新秀”[12]。文献报道了从不同地方分离纯化得到的野生大白口蘑新品系:卯晓岚[13]于1992年在香港中文大学校内凤凰木树旁草地采集到 1 株;郭翠英等[14]在福建省热带植物研究所(厦门)、上官舟建等[15]在荆西地区、汤洪敏等[16]在云南楚雄、邓优锦等[17]在福建福州、黎金锋等[18]在广西南宁、张国广等[19]在厦门大学、刘月廉等[20]在湛江海洋大学校园内均采集到野生菌株,并进行了分离鉴定和驯化等方面的研究。莫美华等[10]在广州先后分离到 4 株大白口蘑新品系,分别命名为SCAUI、Dongguanzhuang、SCAU2和SCAU3,并对它们的生物学特性、栽培学特性和品质特性等进行了研究,经过驯化及优化栽培,获得了4.5万~7.5万kg/hm2的产量,而且得到抗杂菌能力强的菌株。粤北具有丰富的野生大型真菌种质资源,毕志树等在粤北地区采集并鉴定包括担子菌和子囊菌等大型真菌529种,其中野生食药用菌113种[21]。据报道,目前粤北地区已知野生食药用菌共有145种[22],但没有发现野生大白口蘑的文献报道。大白口蘑的商业化栽培范围较窄,大部分地区仍处于试栽培阶段,生产规模较小,制约了该菌的大规模推广[23]。【本研究切入点】 本团队在粤北韶关学院校园内中发现 1 株疑似大白口蘑野生菌株,旁有苦楝和海南蒲桃树。采集并从子实体组织中分离纯化得到纯菌丝体,菌株命名为Tsg1。本研究结合形态学特征与 ITS 序列分析进行鉴定,并通过在PSA培养基、酵母膏培养基和棉籽壳培养基中的培养试验,筛选该菌株菌丝培养最合适的培养基。【拟解决的关键问题】 通过ITS序列克隆和分析,结合形态学,对野生菌株Tsg1进行鉴定,丰富其生物种质资源;并通过菌丝培养基筛选试验和野生环境分析,为其开发利用提供参考。
1 材料与方法
1.1 试验材料
1.1.1 供试菌株 野生大白口蘑Tsg1,采自广东韶关学院校园内荒地杂草丛中,通过组织分离法分离培养得到纯化的菌丝体。
1.1.2 试剂 PSA培养基:马铃薯200 g,蔗糖20 g,琼脂粉15 g,蒸馏水定容至1 L,自然pH。酵母膏培养基:麦芽糖26 g,酵母膏2 g,MgSO4 2.7 g,KH2PO4 1.8 g,维生素B 116 mg,琼脂粉15 g,蒸馏水定容至1 L,自然pH。棉籽壳培养基:棉籽壳150 g,麸皮20 g,蒸馏水1 L,煮沸20 min,过滤;滤液中加葡萄糖20 g,琼脂粉20 g,煮沸后蒸馏水定容至1 L,自然pH。
1.1.3 引物设计与选择 菌株Tsg1的ITS序列PCR扩增采用真菌ITS通用引物:ITS1,5'-TCCGTAGGTGAACCTGCGG-3';ITS4,5'-TCCTCCGCTTATTGATATGC-3'。
1.2 试验方法
1.2.1 野生菌株的采集和分离 2016年7月16日,在韶关学院西区的荒地上采集到 1 株野生大型真菌,采用食用菌组织分离法:取1块黄豆大小的菌盖与菌柄交界部位的菌肉接种于PSA固体培养基上,30 ℃下培养6 d,然后将得到的纯菌丝体转入PSA斜面培养基中,命名为Tsg1,备用。
1.2.2 野生菌株的鉴定 (1)形态观察和鉴定。观察野生菌株子实体的菌盖、菌褶、菌柄、菌环和菌托等主要构成部分,观察菌丝体在固体培养基上菌落的形状、大小、味道、颜色、生活力等,同时采用平板斜插盖玻片法[24-25]观测菌丝细胞大小、形态、锁状联合结构等。(2)ITS鉴定。采用FDEB法[26]提取Tsg1基因组DNA,ITS区序列PCR扩增主要参考Ahlawat等[27]的方法。以真菌ITS通用引物ITS1和ITS4进行PCR扩增:10×buffer(无 Mg2+ )5 μL,MgCl2(25 mmol/L)2.0 μL,ddH2O 33 μL,Primer(IST1,4)4 μL,dNTPs(各 2.5 mmol/L)2 μL,DNA 模板 3 μL,Taq 酶(5 U/μL)1 μL,总体积 50.0 μL。PCR扩增程序:预变性94 ℃ 5 min,变性94 ℃ 45 s,退火56 ℃ 1 min,延伸72 ℃ 1 min,以上共32个循环,最后72 ℃延伸10 min。
将PCR产物送交生工生物工程(上海)股份有限公司进行正反序列测定。将该序列在GenBank核酸数据库中进行BLAST,在DNA序列数据库中搜索同源性高的序列进行比对分析,并用MEGA 7.2软件对Tsg1的ITS序列进行NJ法聚类分析[28]。构建系统发育树,分析G含量、C含量、遗传距离和遗传进化关系等[29]。
1.2.3 菌丝培养基的筛选 选取一管生长良好的母种,活化后再进行转接。用接种钩划取一黄豆大小的菌块,接入PSA培养基、酵母膏培养基和棉籽壳培养基中,30 ℃下培养12 d后,观察测量菌丝的生长速度、色泽、密度和爬壁能力等情况。
试验数据采用SPSS19.0软件进行t检验统计学分析。
2 结果与分析
2.1 形态观察和鉴定
该野生蘑菇大型,簇生,初期为半球形或伞形,菌盖厚,边缘平滑内卷;后期平展,表面光滑,棕白色,菌盖直径5.0~16.0 cm、厚1.9~6.1 cm,平滑或稍粗糙,微粘。菌肉白色至微棕色、厚实、坚韧、嫩脆、有浓厚的蘑菇香味,伤不变色。菌褶白色至浅黄色,直生或弯生,稍密,初期窄后变宽。菌柄中生或偏生,基部往往连合成一丛,实心粗壮,圆柱或倒棒状,长10.0~25.0 cm、粗3.5~9.0 cm,棕白色,基部肥大略弯,无菌环与菌托(图1)。孢子表面光滑,球圆或椭圆形,孢子印白色。
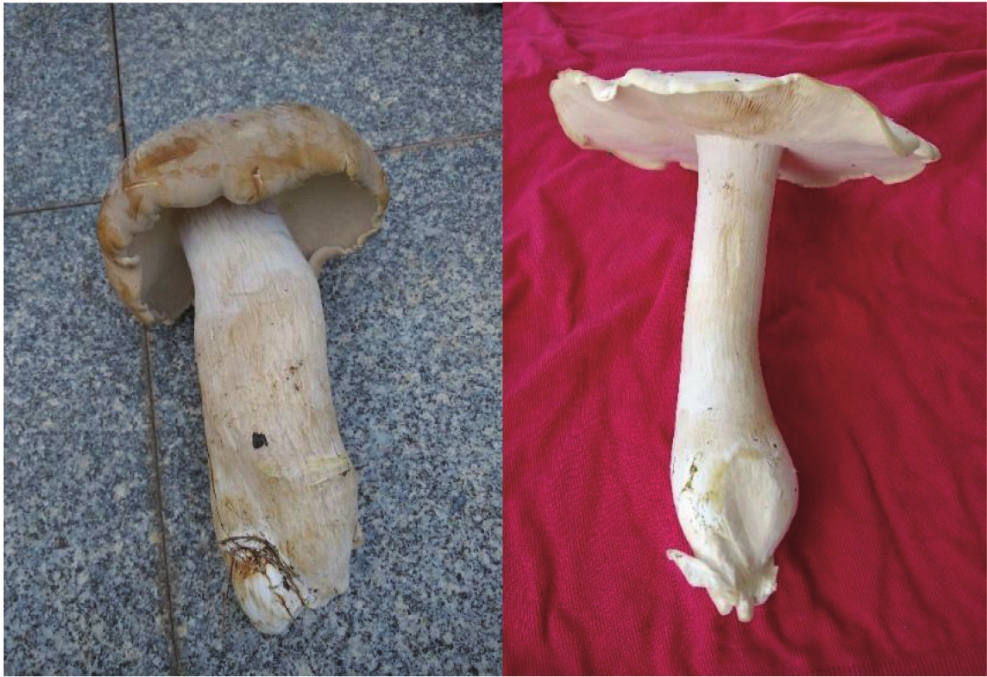
图1 野生大白口蘑(Tsg1)子实体
Fig.1 The fruiting bodies of wild Tricholoma giganteum
由图2可知,在PSA平板中菌丝体呈白色,絮状,气生菌丝较多且较致密,但菌丝生长较缓慢,菌丝体3 d后开始扩展。显微观察显示菌丝细胞有隔膜,无色透明,均一,分枝均匀,菌丝直径2.5~4.0 μm,锁状联合明显。综上所述,根据菌株的形态学特征,并参考《中国大型真菌》[30],初步判断Tsg1与大白口蘑相似,暂定为“拟野生大白口蘑”。

图2 野生大白口蘑菌丝锁状联合
Fig.2 Clamp connection of wild Tricholoma giganteum (1 000×)
2.2 ITS序列分析与鉴定
采用真菌ITS通用引物ITS1和ITS4,对疑似野生大白口蘑菌株Tsg1基因组DNA的ITS序列进行PCR扩增,扩增结果用1%琼脂糖凝胶电泳进行检测(图3)。PCR产物送至生工生物工程(上海)股份有限公司进行正反序列测定。将获得的正反向序列使用DNAStar软件包中的SeqMan进行拼接、编辑,获得完整的ITS序列(序列长度为650 bp,GenBank登记号为MK660795)。
通过NCBI在线BLAST N检索,该序列与GenBank中报道的大白口蘑JX041888.1、JX068526.1、MK024240.1、MH053153.1、MG867660.1、JX193694.1、KY744346.1、KJ463732.1、JN006792.1、KJ463731.1等菌株序列相似性较高,其Ident约为90%,且E value为0。
通过软件MEGA7.2分析Tsg1与GenBank中报道的大白口蘑(共25株)的GC含量,结果显示GC含量在39.0~45.3之间,而Tsg1的GC含量较低,与HM120872.1极为相近、皆为41.8。选择“Krima2-Paramenter”核酸距离模式进行数据处理,获得遗传距离矩阵(图4)。显示Tsg1与其他大白口蘑菌株之间的遗传距离矩阵较小,有着很近的亲缘关系。采用N-J(Neighbor-Joining)法构建系统发育树(图5)进行聚类分析,结果显示Tsg1序列与GenBank中报道的大白口蘑(24株)序列具有较高的相似性、Ident在82.23%~90.06%之间,其中与序列JX041888.1相似性最高、Ident为90.06%,与序列KT154007.1相似性较低、Ident为82.23%。因此,结合形态学鉴定可确定Tsg1为大白口蘑新菌株。

图3 ITS序列PCR扩增结果
Fig.3 Result of ITS PCR amplification
M:DNA Marker 3,T:Tsg1
2.3 菌丝培养基的筛选
Tsg1菌丝在3种培养基中30 ℃培养12 d,生长情况如图6(彩版见封三)和表1。图6和表1显示,Tsg1菌丝在3种培养基中均能生长,菌丝的长势存在明显不同,其生长速度表现为棉籽壳培养基>酵母膏培养基>PSA培养基,爬壁能力表现为棉籽壳培养基>酵母膏培养基=PSA培养基,菌丝浓密和粗壮度表现为棉籽壳培养基>PSA培养基>酵母膏培养基。其中在棉籽壳培养基中菌丝的生长速度最快、长势最好,菌丝雪白,爬壁能力强,致密健壮。

图4 大白口蘑的遗传距离矩阵
Fig.4 Genetic distance matrix of Tricholoma giganteum

图5 新菌株Tsg1与其他大白口蘑的进化树
Fig.5 The phylogenetic tree of new strain Tsg1 and Tricholoma giganteum related species
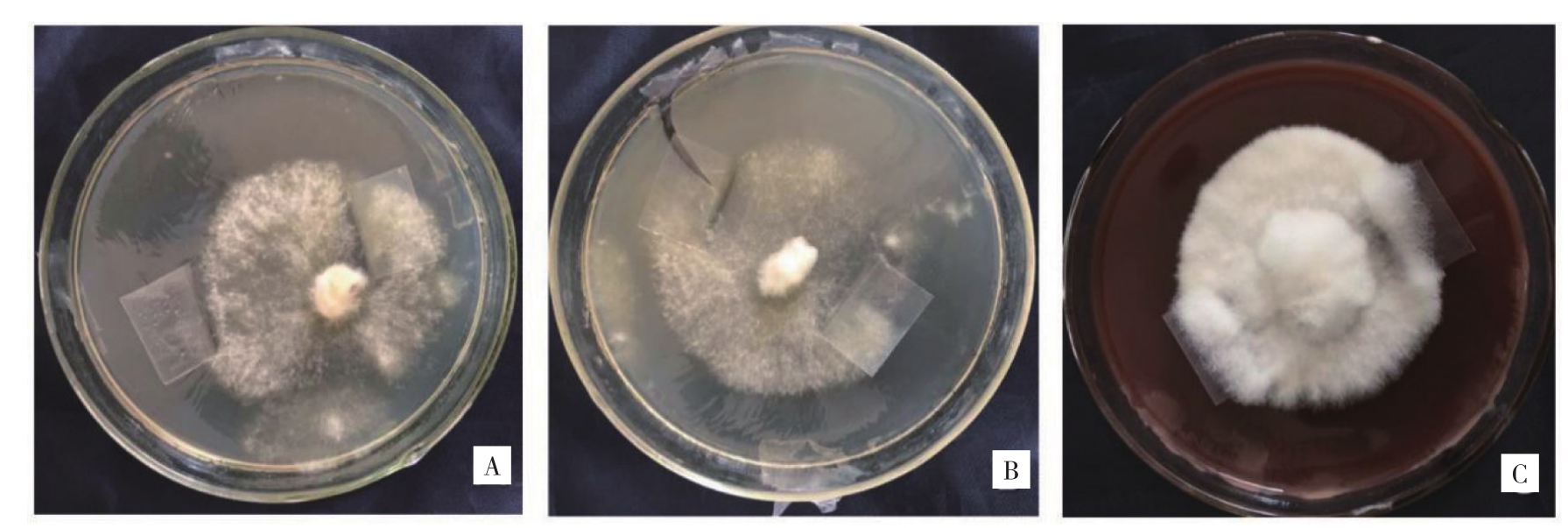
图6 Tsg1在3种培养基上的菌丝生长情况
Fig.6 The growth situation of Tsg1 in three mycelium culture media
A:PSA培养基,B:酵母膏培养基,C:棉籽壳培养基
A: PSA medium; B: Yeast extract medium; C: Cottonseed shell medium
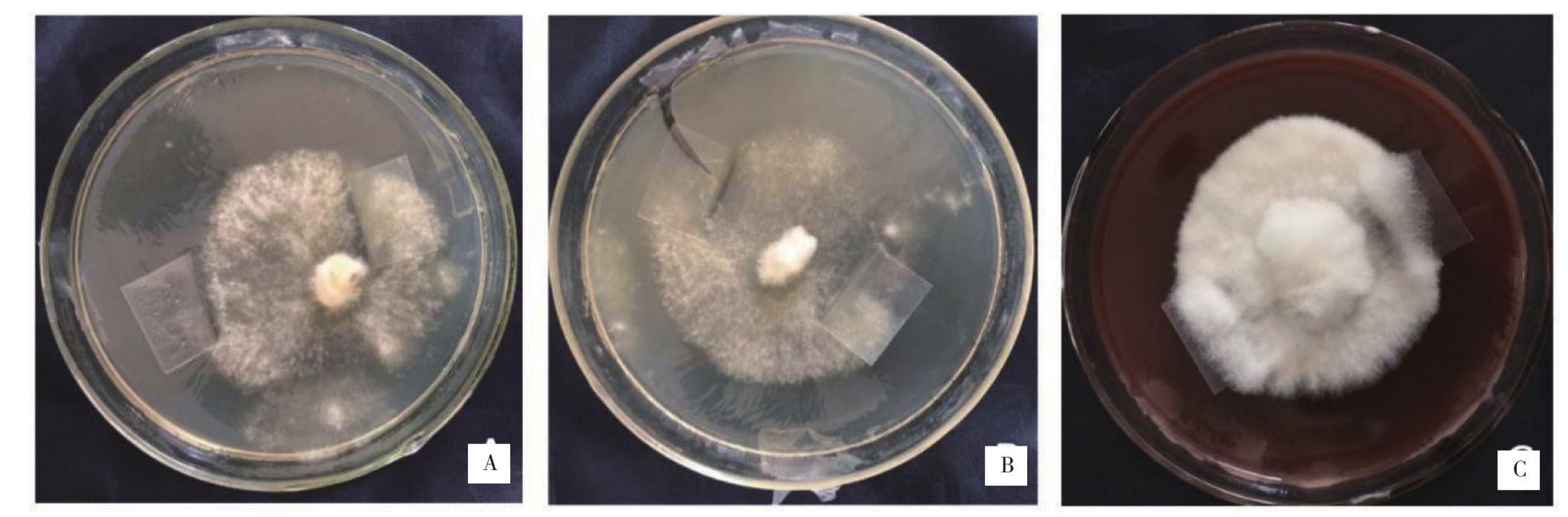
图6 Tsg1在3种培养基上的菌丝生长情况
Fig.6 The growth situation of Tsg1 in three mycelium culture media
A:PSA培养基,B:酵母膏培养基,C:棉籽壳培养基
A: PSA medium; B: Yeast extract medium; C: Cottonseed shell medium
表1 Tsg1在3种培养基上菌丝的生长情况
Table 1 The mycelium growth situation of Tsg1 in three culture media

注:同列数据后小写英文字母不同者表示差异显著。
Note: The different lowercase letters in the same column represent significant differences.
菌丝生长速度Mycelium growth rate(mm/d)PSA培养基 PSA medium 较浓 较白 较粗壮 较弱 2.32±0.18 a酵母膏培养基Yeast extract medium 较稀疏 较白 较纤细 较弱 2.95±0.18 b棉籽壳培养基Cottonseed shell medium 浓密 雪白 粗壮 强 3.68±0.23 c培养基Medium菌丝浓密度Mycelium density菌丝颜色Mycelium colour菌丝粗壮度Mycelium roughness菌丝爬壁能力Wall climbing ability of mycelium
3 讨论
对野生大型真菌种质资源的收集、驯化是当前科研工作的重点之一。然而,以往对野生大型真菌的鉴定大多依据形态学分类,由于这些依据指标存在一定的主观性,且有些特征会随着生长条件的改变而发生变化,这给传统分类带来了困难,特别是对于那些形态特征非常相近的种,更是难以准确区分[31]。分子生物学的快速发展为野生真菌的鉴定提供了准确而便捷的方法,采用分子方法进行物种间的分类和鉴定,分析物种间的同源性,更加贴近生物的本原。目前关于真核微生物的鉴定,通常利用其高度保守区rDNA间隔转录区间(ITS)特征进行鉴定[32],因为rDNA的ITS区同时具有保守区和多变区,在真菌系统发育分析以及物种鉴定方面,ITS序列也作为一个有效鉴定真菌种类的分子标记而被广泛采用[33]。18S、5.8S和28S的序列在进化中趋于保守、种间变化少;然而,内转录间隔区ITS1和ITS2作为非编码区,最终不加入成熟核糖体,受到的选择压力较小,进化速度快且变异速度存在较大的种间差异,适合种及种以下水平的分类研究[34]。
本研究在形态学的基础上,通过ITS序列分析,结合GenBank已有的大白口蘑基因序列特征,最终确定Tsg1为大白口蘑。近年在香港、云南、广东、湖南、福建等多个地方的夏秋季都发现了野生大白口蘑,在凤凰木树旁、羊蹄甲树旁、榕树下、竹林中的沃土或草地上丛生[14-20]。本次发现的野生大白口蘑是在苦楝和海南蒲桃树旁的杂草中,地处粤北的韶关学院校园内。ITS序列分析显示,该菌株与其他发现的野生菌株存在一定差异,与莫美华等[10,35]在广州发现的菌 株(JX041888.1、JX068526.1、JX068527.1和JX193694.1)较为相似,其Ident分别为90.06%、89.63%、89.47%和89.58%,这对丰富大白口蘑的野生资源具有积极意义,有利于对本地野生巨大口蘑资源的保护、开发和育种。同时,培养基筛选试验显示棉籽壳培养基是该菌株菌丝培养合适的培养基,这为后期相关研究奠定了基础。
4 结论
本研究团队在粤北韶关学院内苦楝和海南蒲桃树旁的杂草中采集到1株疑似大白口蘑野生菌株,采用组织分离法,从野生子实体中分离纯化得到纯菌丝体。通过ITS序列克隆与序列分析,结合传统形态学分析,鉴定该菌株为野生大白口蘑,命名为Tsg1。棉籽壳培养基是其菌丝培养适宜培养基。
[1] CHANG S T, MAO X L.Hong Kong Mushrooms[M].Hong Kong: The Chinese University of Hong Kong Press, 1995: 79-80.
[2] 王元忠,汤洪敏,虞泓,张振富.巨大口蘑子实体营养成分分析[J].食用菌学报,2005,12(2):24-26.doi:10.16488/j.cnki.1005-9873.2005.02.005.
WANG Y Z, TANG H M, YU H, ZHANG Z F.Analysis of main nutrition components in Tricholoma giganteum Fruitbodies[J].Acta Edulis Fungi, 2005, 12(2): 24-26.doi:10.16488/j.cnki.1005-9873.2005.02.005.
[3] 李涛,王元忠,刘鸿高.巨大口蘑中微量元素的光谱测定[J].光谱学与光谱分析,2008,28(2):450-452.
LI T, WANG Y Z, LIU H G.Spectrometric determination of trace elements in Tricholoma Giganteum Massee[J].Spectroscopy and Spectral Analysis, 2008, 28(2): 450-452.
[4] LIU F, OOI V C, LIU W K, CHANG S T.Immunomodulation and antitumor activity of polysaccharide-protein complex from the culture filtrates of a local edible mushroom, Tricholoma lobayense[J].General Pharmacology, 1996, 27(4): 621-624.
[5] MIZUNO T, KINOSHITA T, ZHUANG C, ITO H, MAYUZUMI Y.Antitumor-active heteroglycans from niohshimeji mushroom,Tricholoma giganteum[J].Bioscience, Biotechnology, and Biochemistry, 1995, 59(4): 568-571.doi:10.1271/bbb.59.568.
[6] MAU J L, LIN H C, SONG S E.Antioxidant properties of several specially mushrooms[J].Food Research International, 2002, 35(6):519-526.
[7] GUO Y, WANG H, NG T B.Isolation of trichogen, an antifungal protein from fresh fruiting bodies of the edible mushroom Tricholoma giganteum[J].Peptides, 2005, 26(4): 575-580.doi:10.1007/s10528-010-9347-y.
[8] LEE D H, KIM J H, PARK J S, CHOI Y J, LEE J S.Isolation and characterization of a novel angiotensin l-converting enzyme inhibitory Peptide derived from the edible mushroom Tricholoma giganteum[J].Peptides, 2004, 25(4): 621-627.doi: https://doi.org/10.1016/j.peptides.2004.01.015.
[9] YAUG J L, LIN H C, MAU J L.Non-volatile taste components of several Pecialty mushrooms[J].Food Chemistry, 2001, 72(4): 465-471.
[10] 杨水莲,聂健,叶运寿,莫美华,蒋鑫宇,黄友环,郑锦荣,刘英,袁菁艺.巨大口蘑的研究现状及前景展望[J].安徽农业科学,2016,44(10):9-12.doi:10.13989/j.cnki.0517-6611.2016.10.004.
YANG S L, NIE J, YE Y S, MO M H, JIANG X Y, HUANG Y H,ZHENG J R, LIU Y, YUAN J Y.Research status and prospect forecast of Tricholoma giganteum[J].Journal of Anhui Agricultural Science,2016, 44(10): 9-12.doi:10.13989/j.cnki.0517-6611.2016.10.004.
[11] 何志强,岑延新.金福菇夏季栽培技术[J].食用菌,2009(4):49.doi:cnki:sun:siyj.0.2009-04-036.
HE Z Q, CEN Y X.Cultivation techniques of Tricholoma lobayerse in summertime[J].Edible Fungi, 2009(4): 49.doi:cnki:sun:si yj.0.2009-04-036.
[12] 王桂芹,王志.金福菇菌株间品比试验[J].中国林副特产,2008(2):32-34.doi:10.3969/j.issn.1001-6902.2008.02.012.
WANG G Q, WANG Z.Comparative study of the cultural strains of Tricholoma lobayerse[J].Forest By-Product and Speciality in China,2008(2): 32-34.doi:10.3969/j.issn.1001-6902.2008.02.012.
[13] 李志生 .巨大口蘑及其栽培技术[J].食用菌,2007(5):59-61.doi:10.3969/j.issn.1000-8357.2007.04.050.
LI Z S.Cultivation techniques of Tricholoma giganteum[J].Edible Fungi, 2007(5): 59-61.doi:10.3969/j.issn.1000-8357.2007.04.050.
[14] 郭翠英,沈育芬.大白口蘑厦1菌株的特性研究[J].食用菌,2002,24(3):11-12.doi:cnki:sun:siyj.0.2002-03-009.
GUO C Y, SHEN Y F.Study of characteristics on Tricholoma giganteum strain XIA 1[J].Edible Fungi, 2002, 24(3): 11-12.doi:cnki:sun:si yj.0.2002-03-009.
[15] 上官舟建,林汝楷,吴宝芳.巨大口蘑生物学特性的研究[J].浙江食用菌,2007(1):16-21.doi:cnki:sun:zsyc.0.2007-01-011.
SHANGGUAN Z J, LIN R K, WU B F.Study on biological characteristics of Tricholoma giganteum[J].Zhejiang Shiyongjun,2007(1): 16-21.doi:cnki:sun:zsyc.0.2007-01-011.
[16] 汤洪敏,虞泓,李长利,张灼.大白口蘑生物学特性的观察[J].菌物学报, 2007, 26(2): 297-301.doi:cnki:sun:jwxt.0.2007-02-025.
TANG H M, YU H, LI C L, ZHANG Z.Biological characteristics of Tricholoma giganteum Massee[J].Mycosystema, 2007, 26(2): 297-301.doi:cnki:sun:jwxt.0.2007-02-025.
[17] 邓优锦,傅俊生,谢宝贵,林娓娓,郑域茹.野生金福菇的鉴定及其生物学特性研究[J].浙江食用菌,2008(6):10-14.
DENG Y J, FU J S, XIE B G, LIN W W, ZHENG Y R.Identification and study of biological characteristics on wild Tricholoma giganteum[J].Zhejiang Shiyongjun, 2008( 6) : 10-14.
[18] 黎金锋,覃培升.野生巨大口蘑生物学特性研究[J].中国食用菌,2008(3):43-45.doi:10.3969/j.issn.1003-8310.2008.03.016.
LI J F, QIN P S.Studies on biological characteristics of Tricholoma giganteum[J].Edible Fungi of China, 2008(3): 43-45.doi:10.3969/j.issn.1003-8310.2008.03.016.
[19] 张国广,邹金美,郑琳,史志敏,谢懿斌,陈祖华.一株野生大型真菌的ITS分析及生物学特征研究[J].云南民族大学学报(自然科学版),2010,19(6):395-399.doi:10.3969/j.issn.1672-8513.2010.06.002.
ZHANG G G, ZOU J M, ZHENG L, SHI Z M, XIE Y B,CHEN Z H.Study on a wild Macrofungi ITS and its biological characteristics[J].Journal of Yunnan University of Nationalities(Natural Sciences Edition), 2010, 19(6): 395-399.doi:10.3969/j.issn.1672-8513.2010.06.002.
[20] 刘月廉,谭树明,温美英,梁恩义.野生洛巴伊口蘑菌株的分离与鉴定[J].食用菌学报,2001,8(2):19-23.doi:10.3969/j.issn.1005-9873.2001.02.004.
LIU Y L, TAN S M, WEN M Y, LIANG E Y.A study on the isolation and identification of wild Tricholoma lobayense strain[J].Acta Edulis Fungi, 2001, 8(2): 19-23.doi:10.3969/j.issn.1005-9873.2001.02.004.
[21] 毕志树, 郑国杨, 李泰辉, 王又昭.粤北山区大型真菌志[M].广州:广东科学技术出版社, 1990.
BI Z S, ZHENG G Y, LI T H, WANG Y Z.Macrofungi in Mountainous Areas of Northern Guangdong[M].Guangzhou: Guangdong Science and Technology Press, 1990.
[22] 蔡爱群.粤北地区的抗肿瘤野生食用菌资源[J].安徽农业科学,2008, 36(1): 250-251, 267.doi:10.3969/j.issn.0517-6611.2008.01.107.
CAI A Q.Wild edible fungi resources of antitumor in the north region of Guangdong province[J].Journal of Anhui Agricultural Science, 2008,36(1): 250-251, 267.doi:10.3969/j.issn.0517-6611.2008.01.107.
[23] 马紫英,夏斌,倪焱,魏要武,聂健,莫美华.以椰子壳为主碳源的巨大口蘑原种培养基优化[J].北方园艺,2014(13):142-145.doi:cnki:sun:bfyy.0.2014-13-040.
MA Z Y, XIA B, NI Y, WEI Y W, NIE J, MO M H.Optimization of culture medium for second-class spawn of Tricholoma giganteum using coconut shell as main carbon source[J].Northern Horticulture,2014(13):142-145.doi:cnki:sun:bfyy.0.2014-13-040.
[24] LIU Z, LIN J F, GUO L Q, WANG W, YUAN J H, WU Y D.The migration of multipartial nuclear and its regulation by DCTN1 gene in the mycelium cells of Volvariella volvacea[J].Nanoscience and Nanotechnology Letters, 2017(9): 2061-2066.doi:https://doi.org/10.1166/nnl.2017.2582.
[25] 肖自添,刘明, 何焕清.一株野生乌芝的鉴定及其生物学特性研究[J].广东农业科学,2016,43(3):72-76.doi:10.16768/j.issn.1004-874X.2016.03.015.
XIAO Z T, LIU M, HE H Q.Research on identification and biological characteristics of a wild Amauroderma rugosum[J].Guangdong Agricultural Sciences, 2016, 43(3): 72-76.doi:10.16768/j.issn.1004-874X.2016.03.015.
[26] WANG J, GUO L Q, ZHANG K, WU Q, LIN J F.Highly efficient Agrobacterium-mediated transformation of Volvariella volvacea[J].Bioresource Technology, 2008, 99: 8524-8527.doi:10.1135/cccc20030105.
[27] AHLAWAT O P, GUPTA P, KAMAL S, DHAR B L.Variability in intra-specific and monosporous isolates of Volvariella volvacea based on enzyme activity, ITS and RAPD [J].Indian Journal of Microbiology, 2010, 50(2): 192-204.doi:https://doi.org/10.1007/s12088-010-0031-z.
[28] 兰玉菲,王庆武,唐丽娜,安秀荣.糙皮侧耳种质主要农业艺形状的遗传多样性及聚类分析[J].食用菌学报,2014(4):10-14.doi:cnki:sun:syjb.0.2014-04-003.
LAN Y F, WANG Q W, TANG L N, AN X R.Cluster analysis based on agronomic traits of PIeurotus ostreatus strains[J].Acta Edulis Fungi,2014(4): 10-14.doi:cnki:sun:syjb.0.2014-04-003.
[29] 饶冬梅.NCBI组数据库及其资源的获取[J].科技视界,2013(7):30-32.doi:cnki:sun:kjsj.0.2013-07-036.
RAO D M.NCBI database and the access of resources[J].Science &Technology Vision, 2013(7): 30-32.doi:cnki:sun:kjsj.0.2013-07-036.
[30] 卯晓岚.中国大型真菌[M].郑州:河南科学技术出版社,2000:128.
MAO X L.Chinese Macrofungi[M].Zhengzhou: Henan Science and Technology Press, 2000: 128.
[31] 刘主,蔡爱群,刘静华,何智丹,孟晓东.基于ITS序列的疑似野生“南华”草菇菌株的分子鉴定[J].北方园艺,2016(1):126-130.doi:10.11937/bfyy.201601033.
LIU Z, CAI A Q, LIU J H, HE Z D, MENG X D.Molecular identification of suspected wild Nanhua Volvariella volvacea strain based on internal transcribed spacer sequence[J].Northern Horticulture, 2016(1): 126-130.doi:10.11937/bfyy.201601033.
[32] 何凌冰,吴惠萍,游春平,陈炳旭,韩正洲,徐冰,兰金旭,邢建永.岗梅枝枯病病原鉴定[J].广东农业科学,2017,44(1):111-114.doi:10.16768/j.issn.1004-874X.2017.01.017.
HE L B, WU H P, YOU C P, CHEN B X, HAN Z Z, XU B, LAN J X,XING J Y.Identification of the pathogen causing branch blight of Ilex asprella[J].Guangdong Agricultural Sciences, 2017, 44(19): 111-114.doi:10.16768/j.issn.1004-874X.2017.01.017.
[33] 任翔,何德,李翠新,丁浩书.香菇细胞内ITS致同进化不完全及对分类鉴定和系统发育的影响[J].广东农业科学,2015,42(19):111-121.doi:10.16768/j.issn.1004-874X.2016.03.015.
REN X, HE D, LI C X, DING H S.Incomplete intracellular ITS concerted evolution in Lentinula edodes and its influence on identification and phylogenesis[J].Guangdong Agricultural Sciences,2015, 42(19): 111-121.doi:10.16768/j.issn.1004-874X.2016.03.015.
[34] LANDEWEERT R, LEEFLANG P, KUYPER T W, HOFFLAND E, ROSLING A, WERNARS K, SMIT E.Molecular identification of Ectomycorrhizal mycelium in soil horizons[J].Applied and Environmental Microbiology, 2003, 69(1): 327-333.doi:10.1128/aem.69.1.327-333.2003.
[35] 马紫英,倪焱,魏要武,聂健,杨水莲,昌毓嵩,莫美华.野生巨大口蘑1株新菌株ITS鉴定及菌丝培养基优化[J].华南农业大学学报,2015,36(3):98-103.doi:10.7671/j.issn1001-411X.2015.03.017.
MA Z Y, NI Y, WEI Y W, NIE J, YANG S L, CHANG Y S, MO M H.Identification of a new strain of Tricholoma giganteum by ITS and optimization of culture medium for mycelium[J].Journal of South China Agricultural University, 2015, 36(3): 98-103.doi:10.7671/j.issn1001-411X.2015.03.017.