【研究意义】塞内卡病毒(Senecavirus A,SVA),也称为塞内加谷病毒(Seneca valley virus,SVV),属于小RNA病毒科(Picornaviridae)塞内卡病毒属[1],与心病毒属(Cardiovirus)的亲缘关系较近。SVA病毒粒子的直径为20~30 nm ,呈正二十面体,无囊膜。SVA具有小RNA病毒科病毒基因组的共同特点,即呈现L-4-3-4分布[2]。SVA 基因组约7.2 kb,为单链正股RNA。虽然SVA造成的损失有限,但由于该病的临床症状症状与口蹄疫(foot-and-mouth disease,FMD)、猪水疱病 (swine vesicular disease,SVD)、猪水疱疹(vescular exanthema of swine,VES)和水疱性口炎(vesicular stomatitis,VS)等猪原发性水疱病(porcine idiopathic vesicular disease,PIVD)极为相似,能引起猪的鼻吻和蹄部冠状带、蹄部出现水疱样病变[3],临床上很难区分,甚至容易误诊,必须借助实验室方法[4-6]来鉴别。SVA危害养猪业的健康,能引起很大的经济损失。【前人研究进展】塞内卡病毒病最早于2002年在美国分离,此后美洲陆续报道了SVA引起猪水疱性疾病的病例。从2015年开始,巴西、美国、加拿大、哥伦比亚、泰国、越南[7-13]等多国相继发生SVA感染猪的疫情,且呈蔓延之势。2015年,我国广东省发生由SVA引起的水疱性疾病,这是我国首次发现SVA的存在[14],此后,SVA在黑龙江、福建、河南、湖北等省[15-20]陆续有相关报道。【本研究切入点】我们于2016—2018年从广东地区两个养殖场收集及保存了108份临床样品,包括口鼻部、蹄冠部位带有水疱样病变的水疱皮及水疱液病料,采用荧光RT-PCR的方法排查FMD、SVD,初步怀疑猪病由SVA感染引起。本研究采用细胞分离、电子显微镜检查、RT-PCR等鉴定方法,对病料进行分离和鉴定,最后分离得到两株SVA毒株,并进行基因序列测定及遗传进化分析。【拟解决的关键问题】目前,国内分离到的SVA流行毒株数量较少,对于SVA的研究还不够深入。本研究通过对样品进行筛选,从而分离鉴定出两株SVA,为研究SVA的生物学特性、发病机理、遗传进化提供试验材料。
1 材料与方法
1.1 试验材料
供试病料为2016—2018年于广东省两个养殖场收集的疑似SVA的108份病料组织,供试细胞为BHK-21细胞、ST细胞,均由广东省农业科学院动物卫生研究所保存。
主要试剂:高糖DMEM培养基、胎牛血清、0.25% 胰蛋白酶,均购自赛默飞世尔有限公司;PrimeScript ™ One Step RT- PCR Kit试 剂 盒、pMD19-T载体、DNA 胶回收试剂盒、DL2000 DNA Marker,均购自大连 TaKaRa 公司;感受态细胞TOP 10,购自天根生化(北京)公司;AxyPrepTM 体液病毒DNA/ RNA小量提取试剂盒,购自康宁生命科学有限公司;口蹄疫病毒通用型/猪水疱病毒/塞内卡病毒三重核酸检测试剂盒(PCR-荧光探针法),购自深圳澳东检验检测科技有限公司。
主要仪器:梯度PCR仪(美国BIO-RAD T100)、荧光定量PCR仪(Agilent Stratagene MX3000P)、CO2培养箱、超净工作台(苏州净化设备厂)、三目倒置显微镜(重庆光电XDS-1B)、电子显微镜(日立H-7650)。
1.2 试验方法
1.2.1 样品采集与处理 无菌采集病猪蹄部水泡皮(病料1)、鼻镜部水泡皮(病料2),-20 ℃保存。将病料剪碎、加入pH 7.4的PBS研磨,将制成的悬液反复冻融3次,12 000 g、4 ℃离心20 min ,取上清液。
1.2.2 病毒提取 根据AxyPrep体液病毒 DNA/RNA小量提取试剂盒的说明书对病料提取核酸,-80 ℃保存备用。
1.2.3 荧光定量PCR鉴定 提取核酸后,利用口蹄疫病毒通用型/猪水疱病毒/塞内卡病毒三重核酸检测试剂盒鉴定所采集的病料。反应体系制备:核酸扩增反应液16 μL,FMDV/SVD/SVA三重反应液2 μL,RT-PCR酶混液2 μL,待检样品RNA 2 μL,总体积为25 μL,分别做阴性对照、阳性对照。反应程序:50 ℃反转录30 min,95 ℃预变性5 min,95 ℃变性10 s、55 ℃退火/延伸40 s,共40个循环。设置荧光信号:荧光信号采集设定在退火/延伸时。对于多通道荧光PCR仪,口蹄疫病毒通用型选择荧光素ROX为信号采集通道,猪水疱病毒选择荧光素FAM为信号采集通道,SVA选择荧光素CY5为信号采集通道,淬灭基团选None,染料校正选None,记录实验结果。
1.2.4 病毒细胞分离 将经过研磨的病料1和病料2,4 ℃下12 000 g离心10 min,取上清液,用孔径0.22 μm的过滤器过滤。将处理好的病毒液分别接种到长至80%~90%的单层ST细胞、BHK-21细胞上,在37℃、5%CO2的条件下孵育1 h,弃上清液,加入10 mL含 2%胎牛血清的DMEM,培养48 h,每隔12 h观察细胞病变情况,盲传10代,同时设立正常ST细胞、BHK-21细胞为对照。
1.2.5 电子显微镜检查 收集出现细胞病变的病毒液,利用蔗糖梯度密度离心法[21]纯化,并进行病毒液处理,具体步骤如下:取5 μL纯化后的样品滴于铜网上,静置2 min后用滤纸从铜网边缘吸去多余液体;滴加1滴3%、pH为7.0的磷钨酸于铜网上,静置2 min后用滤纸从铜网边缘吸去多余的磷钨酸;滴加1滴纯水于铜网上,用滤纸从铜网边缘吸取多余水分,约20 min铜网干燥后,在电子显微镜下观察,拍照记录。
1.2.6 病毒的 RT-PCR鉴定 利用RT-PCR检测方法对纯化的病毒进行鉴定。采用文献[22]的SVA鉴定引物,上游引物F为5′-GTTCCACTC-CACCGACAA-3′,下游引物R为 5′-AAACCACCCTACAGCAAAT-3′,预期目的片段大小为429 bp。一步法反应体系:基因组RNA模板2 μL,PrimeScript 1 Step Enzyme Mix 1 μL,上游引物 F 0.5 μL,下游引物 R 0.5 μL,2×1 Step Buffer 12.5 uL、灭菌蒸馏水8.5 μL,共25 μL。反应程序:50 ℃反转录30 min,94 ℃预变性 5 min;94 ℃变性 30 s、55 ℃退火 30 s、72 ℃延伸60 s ,共 35 个循环;72 ℃延伸10 min。反应结束后,用1.2%琼脂糖凝胶对PCR产物进行电泳鉴定。将经胶回收纯化的PCR阳性产物克隆至pMD19-T载体,重组质粒送生工生物工程(上海)股份有限公司进行测序。
1.2.7 RT-PCR扩增序列分析 对扩增片段测序,测序结果提交至NCBI Blast进行搜索比对,对扩增的核苷酸序列与GenBank中登录的SVA序列进行同源性比对,并利用软件Megalign、DNAStar绘制系统进化树。
1.2.8 病毒的TCID50检测 利用半数组织细胞感染量(Tissue Culture Infective Dose,TCID50)的方法确定病毒液的滴度,并用Reed-Muench法计算。具体步骤如下:将长势良好的BHK-21细胞进行传代,待细胞长到单层,用2 mL pH 7.4 PBS洗涤细胞表面,重复3次,用0.25%胰酶消化长成单层的BHK-21细胞,用细胞培养基使细胞重新悬浮于液体中,制成细胞悬液;用全自动细胞计数器TC20测定细胞悬液的浓度,浓度约2×105个/mL时,将含有细胞的悬浮培养基接种到96孔板上,100 μL /孔,待细胞长成单层后,弃去旧的培养基;每孔各加入100 μL用细胞维持液以10倍倍比稀释的病毒液(10-1~10-8),每个稀释度重复8个孔,并以正常BHK-21细胞为阴性对照,37 ℃、5% CO2培养箱中培养72 h,每隔12 h观察1次; 通过三目倒置生物显微镜观察细胞病变情况,记录细胞病变的孔数和未有细胞病变的孔数,并用Reed-Muench法计算病毒的TCID50。
2 结果与分析
2.1 荧光RT-PCR检测结果
经荧光RT-PCR检测,CY5信号采集时,阳性对照的Ct值为22.58、病料1的Ct值为17.97、病料2的Ct值为19.37,病料1、病料2均为塞内卡病毒阳性,对口蹄疫病毒、猪水疱病病毒均为阴性(图1)。
2.2 病毒的分离与增殖
在倒置显微镜下观察,结果(图2、图3)发现,接种病料的ST细胞,分别盲传至第6代,培养 48 h 后开始出现细胞病变,表现为细胞变圆,脱落,大量细胞漂浮在培养基里,对照的ST细胞生长良好,没有出现任何病变(图2)。
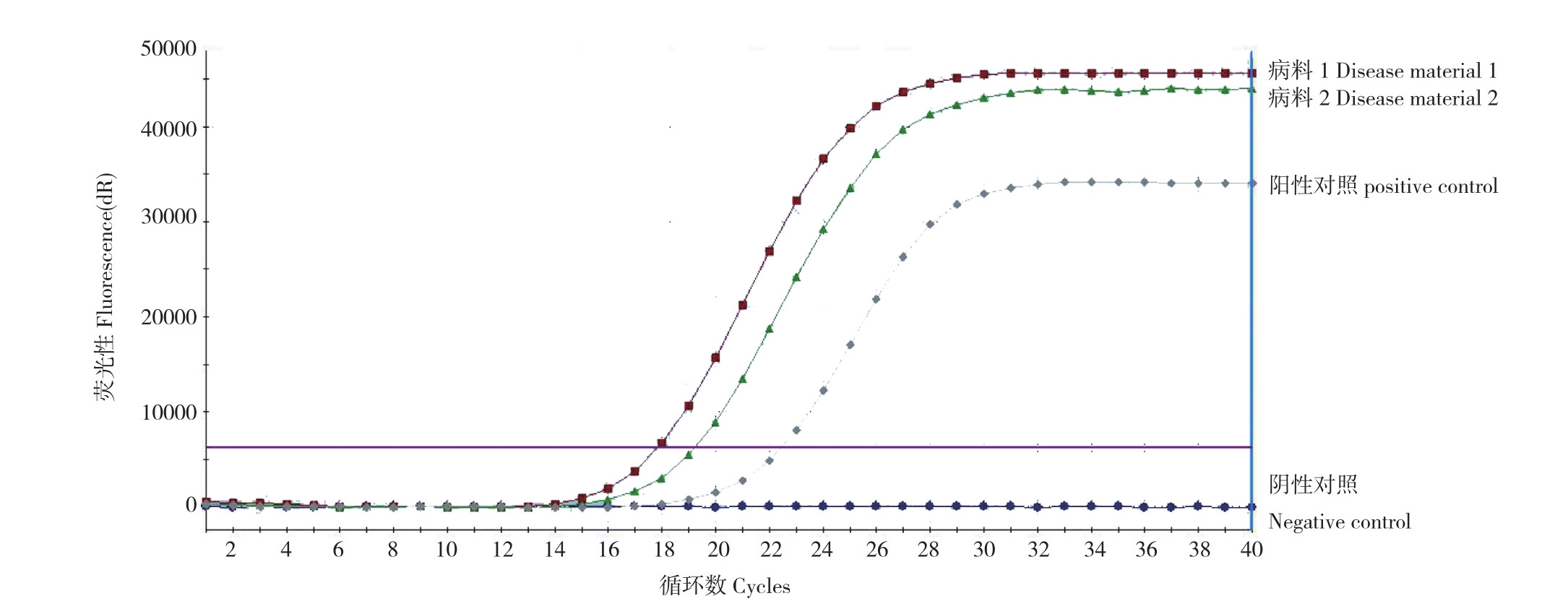
图1 荧光RT-PCR检测结果
Fig.1 Fluorescence RT-PCR results
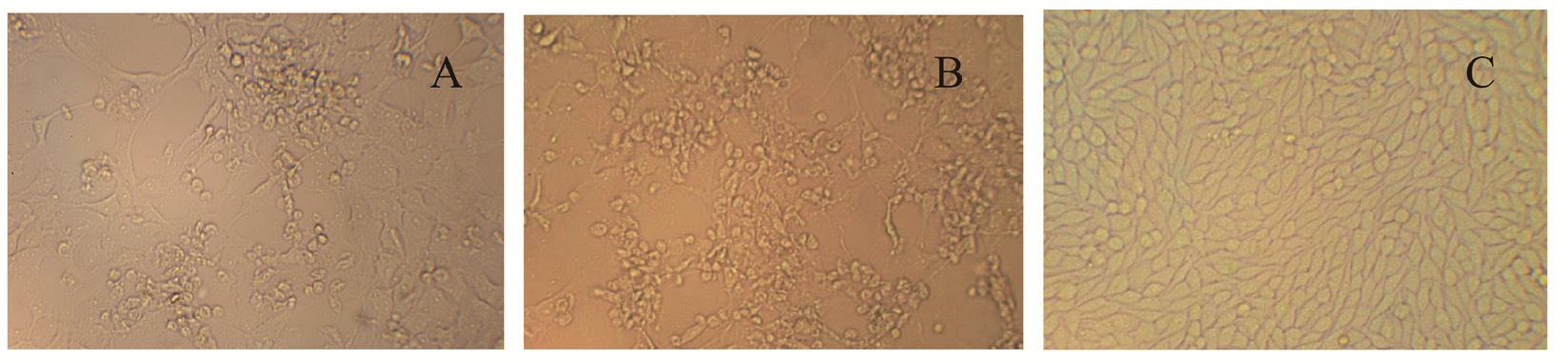
图2 病料接种ST细胞的细胞病变情况
Fig.2 The CPE of ST cells in clinical test
A:病料1;B:病料2;C:正常ST细胞(对照)
A:Disease material 1;B:Disease material 2;C:Normal ST cells (CK)
而病料接种到BHK-21细胞上,盲传至第6代,培养48 h后开始出现细胞病变,表现为细胞变圆,脱落,大量细胞漂浮于液面上,对照的BHK-21细胞生长良好,没有出现任何病变(图3)。
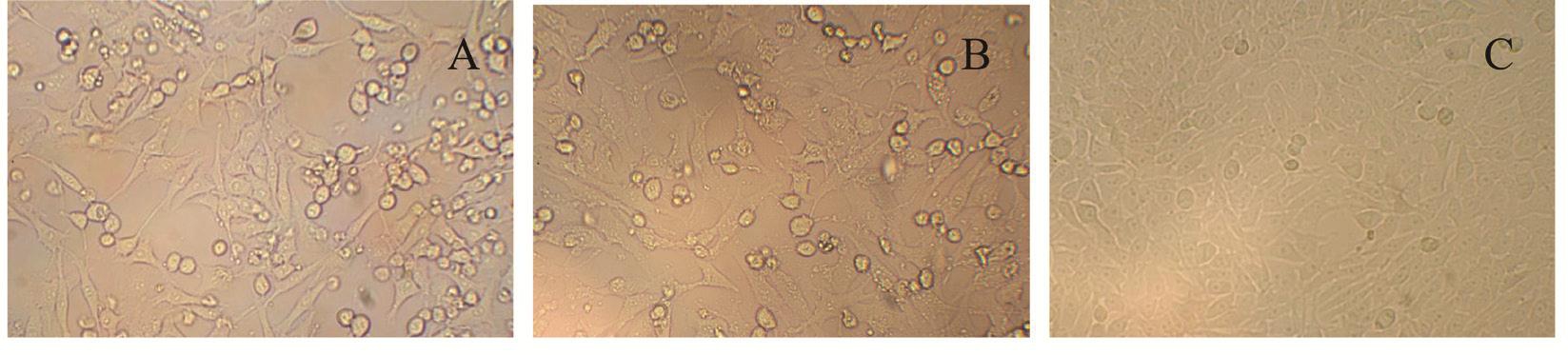
图3 病料接种BHK-21细胞的细胞病变情况
Fig.3 The CPE of BHK-21 cells in clinical test
A:病料1;B:病料2;C:正常BHK-21细胞(对照)
A:Disease material 1;B:Disease material 2;C:Normal BHK-21 cells (CK)
2.3 病毒的电子显微镜观察结果
将通过蔗糖浓度梯度离心纯化的病毒液,经磷钨酸负染后,在电子显微镜下可清晰观察到病毒粒子,直径大小约为25 nm(图4)。
2.4 病毒的RT-PCR鉴定
将两个病料纯化的病毒经过RT-PCR反应,PCR产物经1.2%琼脂糖凝胶电泳检测,两个病料病毒均在429 bp处出现明亮的条带,大小与预期结果相符(图5)。
2.5 NCBI Blastn序列比对结果
将扩增得到的片段测序,结果提交到NCBI Blastn进行搜索比对,两个毒株均鉴定为SVA片段,结果表明,本研究分离到两株塞内卡病毒,将病料1病毒命名为SVA CH-GDBL1-2016,病料2病毒命名为SVA CHGDBL2-2016。
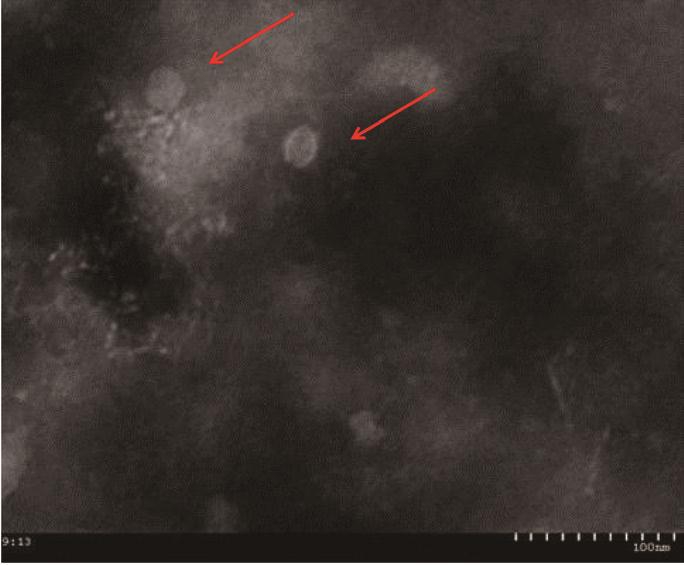
图4 SVA电镜图
Fig.4 Electron micrograph of SVA
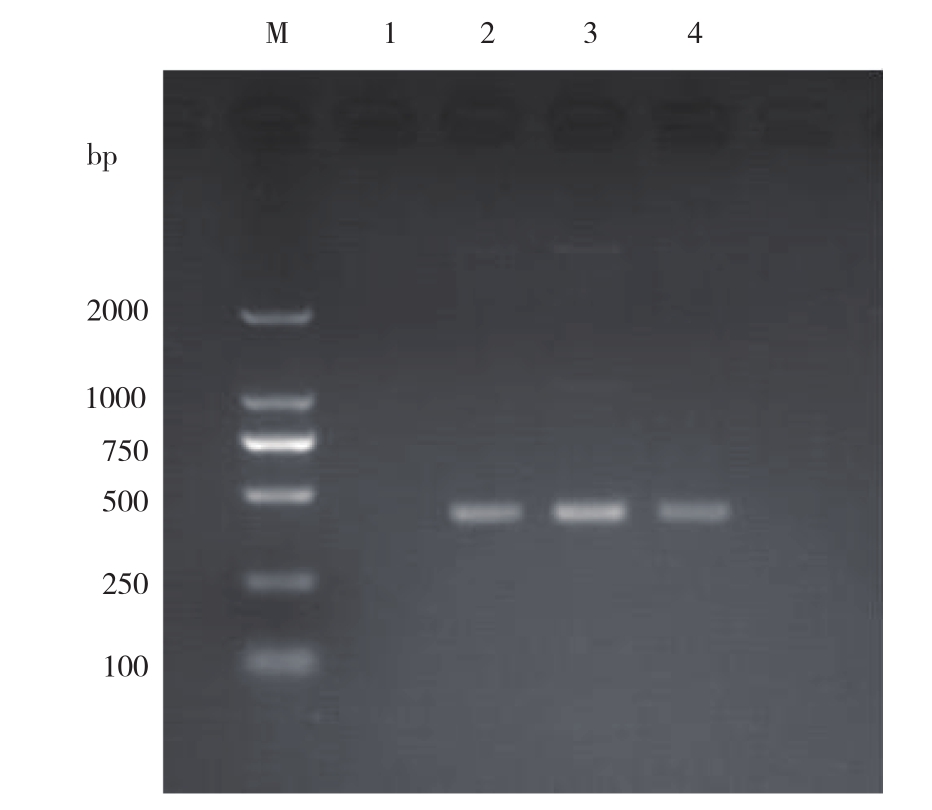
图5 纯化病毒的RT-PCR检测结果
Fig.5 RT-PCR results of purified virus
M: DL2000 Maker;1:阴性对照;2 :SVA阳性对照;3:病料1病毒;4:病料2病毒
M: Maker DL2000; 1: Negative control; 2: SVA positive control;3:Virus 1; 4 : Virus 2
2.6 病毒的同源性及遗传变化分析
利用软件Megalign、DNAStar进行序列分析,序列比对结果(图6)表明,本研究的分离株 SVA CH-GDBL1-2016、SVA CH-GDBL2-2016的序列与2002年美国分离株SVV-001相比,同源性分别为92.6%、92.3%;分离株SVA CHGDBL1-2016与美国分离株SVA-OH1、SVAOH2、SVA-715、MN15-308-M3、NC-011349、US-15-41901SD、USA-SD41901-2015、USA-INPurdue-1581-2016、USA-IN-Purdue-3698-2016的同源性在92.6%~96.3%之间;分离株SVA CH-GDBL2-2016与上述美国分离株的同源性在92.3%~97%之间;分离株SVA CHGDBL1-2016、SVA CH-GDBL2-2016与 加 拿 大分离株11-55910-3的同源性分别为97.7%、97%,与哥伦比亚分离株Colombia-2016的同源性分别为96.5%、95.8%;与巴西分离株SVABRA-GO3-2015、SVA-BRA-MG1-2015的 同 源性分别为96.5%、97.2%;与泰国分离株G103-SV-1-2016-Thailand、G103-SV-2-2016-Thailand的同源性分别为95.8%、95.1%;与越南分离株SVA-VIT-3187-2018的同源性分别为98.4%、97.7%;分离株SVA CH-GDBL1-2016与中国分 离 株 CH-GDLZ02-2017、CH-GDQC-2017、C H-G D Y D-2 0 1 7、C H-G X I 0 9-2 0 1 6、CH-HNSL-2017、CH-LX-01-2016、CHZW-01-2016、GD05-2017、HB-CH-2016、SVA-CHN-17-2017、SVA-HLJ-CHA-2016、CHDB-11-2015、CH-DL-01-2016、CH-FJ-2017、CH-GDLZ01-2017的同源性在94.9%~98.6%之间;分离株SVA CH-GDBL2-2016与上述中国分离株的同源性在95.6%~99.3%之间,其中与分离株CH-GXI09-2016、SVA-CHN-17-2017的同源性高达99.3%。
由图7可知,本研究分离的2株病毒在同一个小分支上,与中国分离株CH-GXI09-2016的亲缘关系较近,而与美国分离株SVV-001、NC-011349不在同一分支上,亲缘关系较远,存在较大差异。
2.7 病毒的TCID50检测结果
根据Reed-Muench法及病毒液在BHK-21细胞的病变情况,计算病毒液的TCID50,分别得到SVA CH-GDBL1-2016的 TCID50为 106.8,SVA CH-GDBL2-2016的TCID50为106.7。
3 讨论
3.1 新分离毒株的同源性及遗传变化
从同源性分析来看,本研究分离到的2个毒株与我国分离株CH-GXI09-2016、SVACHN-17-2017的同源性最高、达99.3%;与美国分离株SVV-001的同源性分别为92.6%、92.3%,差异较大;从系统进化树来看,本研究分离到的2个毒株与我国分离株CH-GXI09-2016、SVACHN-17-2017同属一个分支,与加拿大分离株存在一定差异,与美国分离的原型毒株 SVV-001不在一个分支上,且距离最远,这些差异可能与地域分布有关。SEGALÉS等[23]将SVA VP1的系统进化暂时划分为 3 个谱系:Ⅰ系为原型毒株 SVV-001,Ⅱ系主要为 1988—1997 年的美国分离株,Ⅲ系包括了 2001 年至今从巴西、加拿大、中国、泰国和美国分离的毒株。说明近30年来SVA VP1发生了变异,这些变异是否给发病机制、致病机理、致病性等带来变化,需要进一步研究。
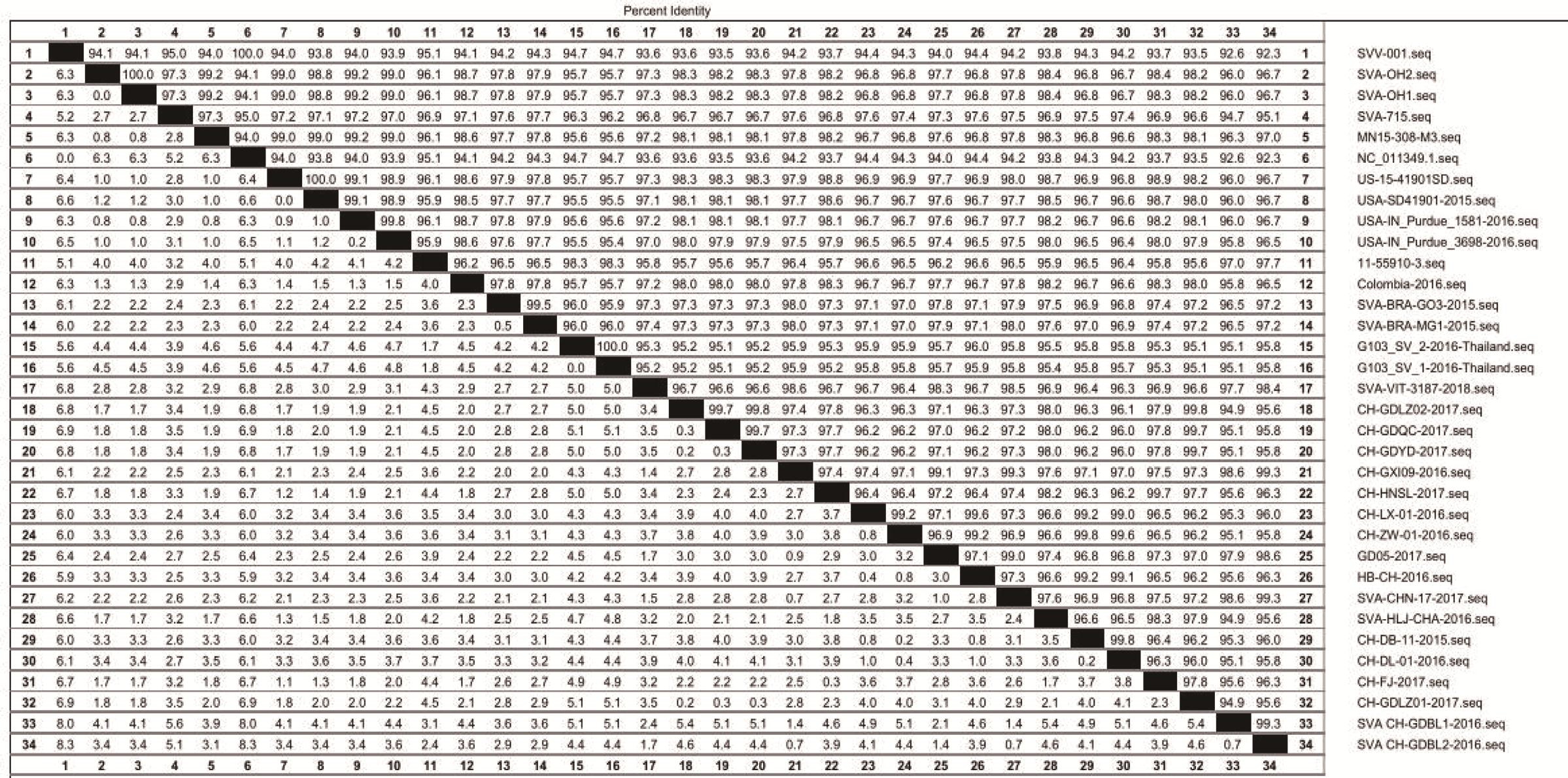
图6 SVA VP1核苷酸序列同源性分析
Fig.6 Homology analysis of the SVA VP1 according to nucleotide sequence
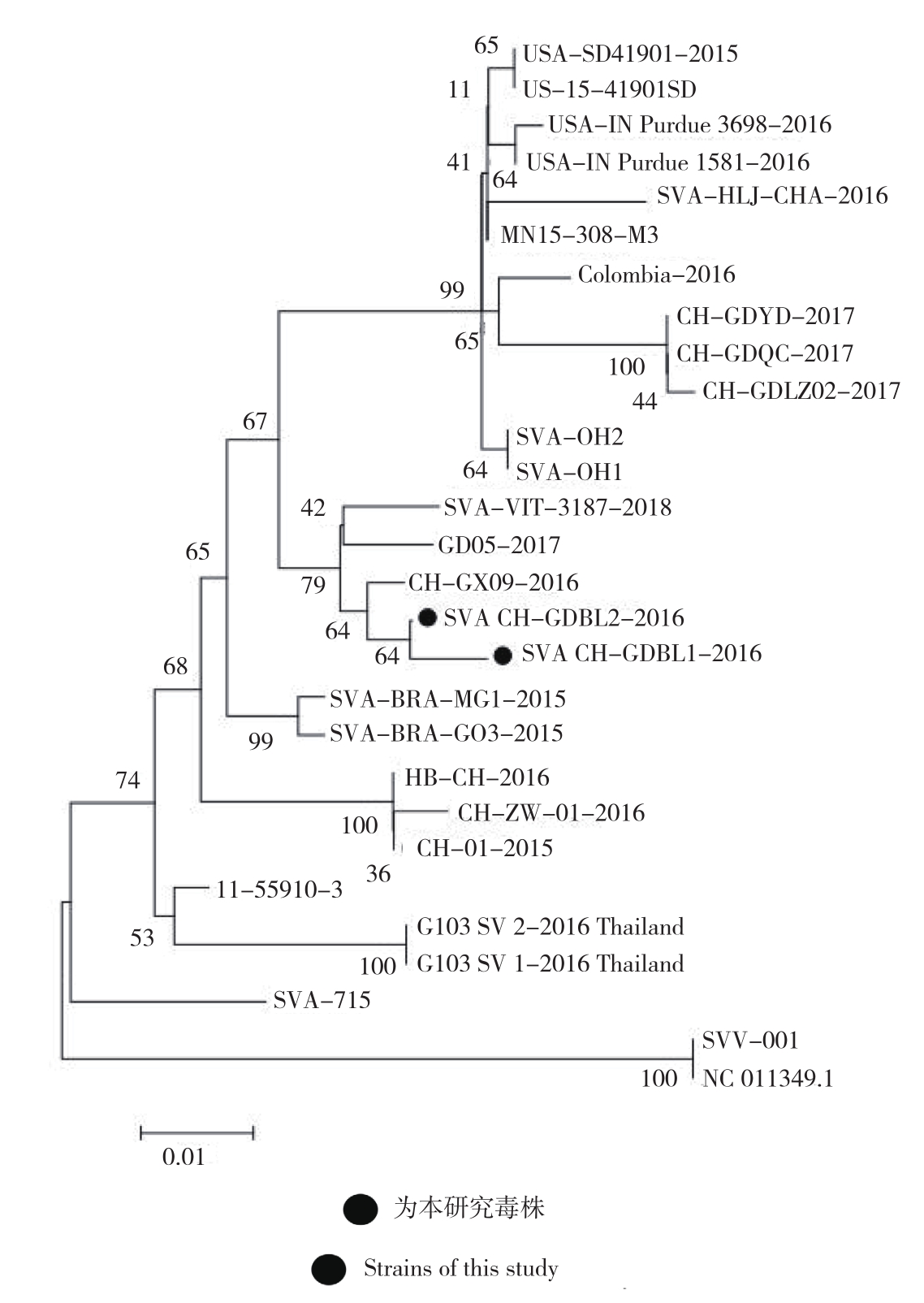
图7 SVA系统进化树
Fig.7 Phylogenetic tree of SVA
3.2 猪鼻镜与蹄冠水疱病的鉴别诊断
病毒的分离培养是研究病毒特性、病毒机理的基础。塞内卡病毒病与口蹄疫、猪水疱病、猪水疱疹及水疱性口炎等猪原发性水疱疾病的临床症状极为相似,必须借助实验室方法来鉴别。研究表明,在细胞分离方面,SVA能在NCI-H1299细胞、ST细胞、PK-15细胞、BHK-21细胞、IBRS-2细胞等细胞中增殖,并能产生相对明显的CPE。本研究选择了实验室比较常见的BHK-21细胞、ST细胞,对疑似SVA的水泡皮病料进行分离培养,自第6代起,病毒液在两种细胞中均能出现典型的CPE。
为了能进一步的判断,利用RT-PCR、荧光PCR能特异性地鉴定上述几种猪原发性水疱疾病。研究人员在RT-PCR、荧光RT-PCR等方面做出了大量研究。KNOWLES等[24]设计了特异性引物,上、下游引物分别位于VP3和2A区域上,利用RT-PCR诊断技术,扩增完整的SVA VP1基因,在猪群中能检测到SVA的存在。樊晓旭等[25]在SVA病毒保守的3D基因区域,设计Taq Man探针以及特异性引物,筛选两者的最优组合,建立 Taq Man荧光定量 PCR 检测方法,用于病毒的早期检测。刘健新等[6]根据SVA的VP1基因保守序列设计引物,建立了实时荧光定量PCR的检测方法,该法特异性好、灵敏度高、操作简单。耗时短。
3.3 塞内卡病毒病是不容忽视的新发病
塞内卡病毒病的危害性虽然没有猪口蹄疫高,但也不容忽视,防控该疾病非常必要。2015年,在我国广东省发现了首例由SVA感染引起的水疱性疾病[26]。在我国,塞内卡病毒是新传入的外来病,目前还没有将塞内卡病毒病归类,也不在最新一版的世界动物卫生组织(OIE)《陆生动物诊断试验与疫苗手册》中。对所有水泡特征的疾病都应在采取严格生物安全措施的情况下尽快确诊,必须与口蹄疫、猪水疱病、水疱性口炎和水疱疹相区别,防止与我国I类传染病口蹄疫和猪水疱性疾病混淆。
农业部办公厅2018年发布的《2018 年兽医工作要点》明确提出,要做好塞内卡病毒病等新传入疫病的防治工作。此公告引起了业界对塞内卡病毒病的高度关注,需要对SVA致病机理、流行情况、诊断和防控技术等进行深入研究。
4 结论
本研究从广东两个猪场疑似塞内卡病毒感染引起死亡的猪群中分离到2株病毒,经过普通RT-PCR鉴定、荧光RT-PCR鉴定、细胞分离培养以及电子显微镜形态学观察等方法检测,证实分离的这两株病毒均为塞内卡病毒新毒株,分别命名为SVA CH-GDBL1-2016和SVA CHGDBL2-2016。塞内卡病毒新毒株可为后续塞内卡病毒的诊断和疫苗研制提供试验材料。
[1] HALES L M,KNOWLES N J,REDDY P S,XU L,HAY C,HALLENBECK P L. Complete genome sequence analysis of Seneca Valley Virus-001, a novel oncolytic picornavirus[J]. Journal of General Virology, 2008, 89(5):1265-1275. doi :10.1099/vir.0.83570-0.
[2] BURKE M. Oncolytic Seneca Valley Virus: past perspectives and future directions[J]. Oncolytic Virotherapy, 2016,5:81-89. doi:10.2147/OV.S96915.
[3] VANNUCCI F A, LINHARES D, BARCELLOS D E S N, LAM H C,COLLINS J, MARTHALER D. Identification and complete genome of Seneca Valley Virus in vesicular fluid and sera of pigs affected with idiopathic vesicular disease, Brazil[J]. Transboundary and Emerging Diseases, 2015,62 (2015): 589-593. doi: 10.1111/tbed.12410.
[4] FOWLER V L, RANSBURGH R H, POULSEN E G, WADSWORTH J, KING D P, MIOULET V, KNOWLES N J, WILLIAMSON S, LIU X M, ANDERSON G A, FANG Y, BAI J. Development of a novel realtime RT-PCR assay to detect Seneca Valley Virus-1 associated with emerging cases of vesicular disease in pigs[J]. Journal of Virological Methods, 2017,239:34-37. doi:10.1016/j.jviromet.2016.10.012.
[5] BRACHT A J, O HEARN E S, FABIAN A W, BARRETTE R W,SAYED A. Real-Time reverse transcription PCR Assay for detection of Senecavirus A in swine vesicular diagnostic specimens[J]. PLOS ONE, 2016,11(1):e146211. doi:10.1371/journal.pone.0146211.
[6] 刘健新,查云峰,鲁荣,任旭皎,黎振标,李慧子,张彭涛,宁章勇.猪塞尼卡病毒荧光定量RT-PCR检测方法的建立及初步应用[J].中国兽医科学,2018,1-9. doi.org/10.16656/j.issn.1673-4696.2019.0041.LIU J X, ZHA Y F, LU R, REN X J, LI Z B, LI H Z, ZHANG P T,NING Z Y. Establishment and primary application of real-time RTPCR for the detection of porcine Senecavirus A[J]. Chinese Veterinary Science,2018:1-9. doi.org/10.16656/j.issn.1673-4696.2019.0041.
[7] HAUSE B M, OLIVIA M, DUFF J, HESSE R A. Senecavirus A in pigs,United States, 2015[J]. Emerging Infectious Diseases, 2016,22(7):1323-1325. doi: 10.3201/eid2207.151591.
[8] QIAN S, FAN W, QIAN P, LI X. Isolation and full-genome sequencing of Seneca Valley virus in piglets from China, 2016[J]. Virology Journal, 2016,13(1). doi:10.1186/s12985-016-0631-2.
[9] SUN D, VANNUCCI F, KNUTSON T P,CORZO C, MARTHALER D G.Emergence and whole-genome sequence of Senecavirus A in Colombia[J]. Transboundary and Emerging Diseases, 2017,64(5):1346-1349.doi:10.1111/tbed.12669.
[10] LEME R A, ZOTTI E, ALCÂNTARA B K, OLIVEIRA M V, FREITAS L A, ALFIERI A F, ALFIERI A A. Senecavirus A: An emerging vesicular infection in brazilian pig herds[J]. Transboundary and Emerging Diseases, 2015, 62:603-611. doi:10.1111/tbed.12430.
[11] SAENG-CHUTO K, RODTIAN P, TEMEEYASEN G, WEGNER M,NILUBOL D. The first detection of Senecavirus A in pigs in Thailand,2016[J]. Transboundary and Emerging Diseases, 2018,65(1):285-288. doi:10.1111/tbed.12654.
[12] ARZT J, BERTRAM M R, VU L T,PAUSZEK S J, HARTWIG E J,SMOLIGA G R,PALINSKI R, STENFELDT C, FISH I H, HOANG B H, PHUONG N T, HUNG V V, VU P P, DUNG N K, DONG P V, TIEN N N, DUNG D H. First detection and genome sequence of Senecavirus A in vietnam[J]. Microbiology resource announcements, 2019,8(3).doi:10.1128/MRA.01247-18.
[13] 贺东生,罗天霞,苏丹萍,徐帅飞,熊景峰. 规模化猪场暴发塞内加谷病及SVV HN16株的分离鉴定[J]. 猪业科学,2017,34(10):72-74.HE D S, LUO T X, SU D P, XU S F, XIONG J F. Separation and identification of Senegalosis and SVV HN16 strain in large-scale pig farms[J]. Swine Industry Science, 2017, 34(10):72-74.
[14] WU Q, ZHAO X, CHEN Y, HE X, ZHANG G, MA J. Complete genome sequence of Seneca Valley virus CH-01-2015[J].Genome Announcements, 2019, 4(1): e01509-15. doi:10.1128/genomeA.01509-15.
[15] ZHU Z, YANG F, CHEN P, LIU H, CAO W,ZHANG K, LIU X, ZHENG H. Emergence of novel Seneca Valley virus strains in China, 2017[J].Transboundary and Emerging Diseases, 2017;64:1024-1029. doi:10.1111/tbed.12662.
[16] ZHAO X, WU Q, BAI Y, CHEN G, ZHOU L, WU Z, LI Y, ZHOU W,YANG H, MA J. Phylogenetic and genome analysis of seven senecavirus A isolates in China[J]. Transboundary and Emerging Diseases,2017,64(6):2075-2082. doi:10.1111/tbed.12619.
[17] SUN Y, CHENG J, WU R, WU Z, CHEN J, LUO Y, XIE Q, MA J.Phylogenetic and genome analysis of 17 novel Senecavirus A isolates in Guangdong Province, 2017[J]. Frontiers in Veterinary Science,2018,5:314. doi:10.3389/fvets.2018.00314.
[18] LUO T, XU S, XIONG J, SU D, HE D. Complete genome sequence of Senecavirus A strain SVV HN16 identified China[J].Genome Announcements, 2017, 5(44): e01168-17. doi:10.1128/genomeA.01168-17.
[19] ZHANG X, XIAO J, BA L,WANG F, GAO D, ZHANG J, PAN C,QI P. Identification and genomic characterization of the emerging Senecavirus A in southeast China, 2017[J]. Transboundary and Emerging Diseases, 2018,65(2):297-302. doi:10.1111/tbed.12750.
[20] 黄元,刘涛,陈锦良,王晓虎,陈晶,梁培新,黄武,向华. 阳江地区某猪场塞内卡病毒抗原和抗体的检测与分析[J]. 广东农业科学,2018,45(12):107-111. doi: 10.16768/j.issn.1004-874X.2018.12.018.HUANG Y, LIU T, CHEN J L, WANG X H, CHEN J, LIANG P X,HUANG W, XIANG H. Detection and analysis of antigen and antibody of Seneca virus from a pig farm in Yangjiang[J]. Guangdong Agricultural Sciences, 2018,45(12):107-111. doi: 10.16768/j.issn.1004-874X.2018.12.018.
[21] 赵晓亚,伍绮文,伍子娴,陈桂华,白杨,马静云. 国内首株猪塞内加谷病毒(Seneca Valley virus)的分离鉴定[J].中国预防兽医学报,2016,38(11):839-843.doi:10.3969/j.issn.1008-0425.2016.11.01.ZHAO X Y, WU Q W, WU Z X, CHEN G H, BAI Y, MA J Y. Isolation and identification of the first Seneca Valley virus strain in China[J]. Chinese Journal of Preventive Veterinary Medicine, 2016,38(11).doi:10.3969/j.issn.1008-0425.2016.11.01.
[22] 杨彩娟,温肖会,任裕其,冼望强,刘暖华,谢乐新. 塞尼卡病毒A VP1基因遗传进化分析[J]. 安徽农业科学,2017,45(26):106-108,128. doi: 10.13989/j.cnki.0517-6611.2017.26.032.YANG C J,WEN X H,REN Y Q, XIE L Q, LIU N H, XIE L X.Phylogenetic analysis of VP1 gene of Senecavirus A[J]. Journal of Anhui Agricultural Science, 2017, 45(26): 106-108,128. doi: 10.13989/j.cnki.0517-6611.2017.26.032.
[23] SEGALÉS J, BARCELLOS D, ALFIERI A, BURROUGH E,MARTHALER D. Senecavirus A[J]. Veterinary Pathology,2016,54(1):11-21. doi:10.1177/0300985816653990.
[24] KNOWLES N J, HALES L M,JONES B H, LANDGRAF J G, HOUSE J A, SKELE K L, BURROUGHS K D, HALLENBECK P L. Epidemiology of Seneca Valley virus: Identification and characterization of isolates from pigs in the United States[C]//EUROPIC 2006: XIVth meeting of the European study group on the molecular biology of picornaviruses,Saariselkä, Inari, Finland. European Study Group on the Molecular Biology of Picornaviruses, p G2.2006.
[25] 樊晓旭,赵永刚,迟田英,哈登楚日亚,吴晓东,王志亮. 塞尼卡谷病毒TaqMan荧光定量PCR检测方法的建立[J].中国预防兽医学报,2016,38(12):959-962. doi: 10.3969/j.issn.1008-0425.FAN X X, ZHAO Y G, CHI T Y, HADENG C R Y,WU X D, WANG Z L. Establishment of a Taq Man real-time PCR assay for detection of Seneca Valley virus [J]. Chinese Journal of Preventive Veterinary Medicine, 2016,38(12): 959-962. doi: doi: 10.3969/j.issn.1008-0425.
[26] WU Q, ZHAO Q, BAI X, SUN Y, B, XIE Q, MA J. The first identification and complete genome of Senecavirus A affecting pig with idiopathic vesicular disease in China[J]. Transboundary and Emerging Diseases, 2016, 64(2017): 1633-1640. doi: 10.1111/tbed.12557.