【研究意义】水稻是世界上最重要的粮食作物之一,其产量占世界粮食总产量的50 %左右,世界上一半以上人口以稻米为主食[1-2]。水稻新品种的选育对于提升粮食安全水平具有重要意义,而种质资源的不断创新是水稻新品种选育的基础。近年来,分子标记辅助选择技术已经在水稻抗病及品质种质创新方面开展了大量研究,显著提升了水稻新品种选育的效率[3]。此外,诱变育种技术在丰富种质资源、加速育种进程方面也起到了重要作用[4]。将分子育种、诱变育种与传统育种技术结合,可以不断丰富水稻遗传资源,提升水稻新品种选育效率。对于分子育种或诱变育种获得的遗传分离群体,利用分子标记获取不同个体的基因型来说,是种质资源精确鉴定的强有力手段[5]。
【前人研究进展】对于植物基因分型来说,一般都需要从数量庞大的遗传分离群体中进行筛选,同时要实现单株与DNA样本一一对应,以便准确筛选出目标植株,而对某基因的定位也经常需要从成千上百份分离群体中提取DNA来筛选目标位点,其工作量之大显而易见[6]。目前,普遍采用的方法是剪取植物的叶片并提取DNA,但面对大批量样本采集时,该方法成本高、效率低且容易出现单株与DNA样本无法对应的错误[7]。此外,目前普遍使用的DNA 提取方法为 SDS法或CTAB法,虽然这些方法提取的 DNA 质量、产量和纯度均较高,但操作步骤多、耗时长、成本高,成为限制水稻大规模基因分型中提高效率和降低成本的关键因素[8-11]。
【本研究切入点】在大规模基于PCR的基因分型检测作业中,高效、快速、简便的模板DNA制备显得非常重要,针对目前组织样本获取和DNA提取效率低的问题,建立一种适用于高通量基因分型的简易、高效和低成本的水稻 DNA 提取方法十分迫切和必要。【拟解决的关键问题】为解决限制水稻DNA高效提取问题,探索快速有效的DNA提取方法,本研究从组织取样与DNA提取两个方面着手,设计了一次性可采集96个水稻植株的根部组织并快速提取DNA的方法,该方法克服了传统方法的工作量大、效率低的缺点,在水稻育种实践中具有较大价值,为水稻分子育种提供有效的技术支持。
1 材料与方法
1.1 试验材料
器材:水稻种子,96孔深孔板,96孔PCR板,金属球(d≈0.27 cm),金属盘(32 cm×22 cm× 3 cm),24微孔板金属热块(QBLock-0.2),高通量组织研磨机(2010 Genogrinder),玻璃棒(d≈0.35 cm)等。
试剂:干冰,乙醇 (95% V/V),2% CTAB提 取 液,500 mmol/L TRIS(pH=8),50 mmol/L EDTA(pH=9)等。
1.2 试验方法
1.2.1 种子处理及培养 将水稻种子消毒、浸种、催芽后,挑选胚芽长2.5 mm左右的种子备用(图1A)。在96深孔板每个孔底部钻出小孔(图1B),孔径约3 mm(图1C)。将萌发的水稻种子放入96深孔板中,胚向下,每孔1粒种子(图1D)。深孔板与PCR板上下叠放(图1E),深孔板在上、PCR板在下,两者的孔一一对应,即深孔板A1应对应PCR板A1孔,用橡皮筋将两端固定(图1F)。固定好的装置称为双层生长板,将生长板放入金属托盘中,加入蒸馏水漫过下部PCR板(图1G),水深约3 cm(图1H),使PCR板的每个孔中均充满蒸馏水。金属托盘及生长板(图1I)放置于培养箱中培养约9 d,培养条件:12 h光照、12 h黑暗,30 ℃恒温。培养过程中,注意观察液面高度,及时更换补充蒸馏水至3 cm深。
1.2.2 DNA提取及扩增 (1)CTAB法提取根部DNA,参考 CHEN等[12]的方法进行。(2)磁珠法提取样本DNA,对获取的根部组织进行研磨和不研磨两种处理,参考CHEN等[12]的方法分别提取样本DNA。(3)改良TRIS-EDTA法提取根部DNA。将 25 μ L TRIS-EDTA(500 mmol/L TRIS,pH=8;50 mmol/L EDTA,pH=9)溶液和根部组织放入含有250 μ L蒸馏水的PCR板孔中密封,组织研磨机粉碎后,3 600 r/min离心10 min,取上清液进行检测并进行PCR扩增。

图1 水稻种子的处理与幼苗培养
Fig.1 Treatment of rice seed treatment and seedling cultivation culture
A:催芽的水稻种子;B:96深孔板,即上部种子板;C:底部钻有小孔的96深孔板;D:催芽的水稻种子用镊子放入深孔板中,胚向下;E:96孔PCR板;F:96深孔板在上,PCR板在下,两者的孔一一对应,使用两个皮筋固定二者;G:双层生长板装置放入金属盘中并加入蒸馏水;H:金属盘液面位置;I:种子幼苗的培养
A:Germinated rice seed; B:96 deep-orifice plate, i.e.the upper seed plate; C:96 deep-orifice plate with small holes at the bottom; D:The germinated rice seeds are placed into the deep-orifice plate with tweezers, and the embryos are downward; E:96-orificel PCR plate; F:96-deepl orifice plate is on the top, PCR plate is at the bottom, the orifices of both are corresponding one by one, which are fixed by two rubber bands; G:A double-layer growth plate device is placed in a metal tray plates and distilled water is added; H:The position of the liquid level of the metal tray is indicated; I:Cultivation of seedlings
引物设计及PCR扩增方法:根据已报道的GS3基因第三外显子(45 bp)序列设计引物[13],引物序列由金唯智公司合成,正向序列:5′-GAACTTCGTCGATTGTGTGG-3′,反向序列:5′-GCTTCTCCGATGAACTGCTT-3′,扩增产物大小为250 bp。PCR扩增体系为 10 μL:DNA 模板 1 μL;Master Mix 5 μL;引物各 0.2 μL;ddH2O 3.6 μL。反应程序:95 ℃预变性5 min;然后按 95 ℃ 30 s,60 ℃ 30 s,72 ℃ 1 min,35个循环;最后72 ℃延伸5 min。取7 μL PCR产物,加入5 μL指示剂,在1%琼脂糖凝胶中电泳30 min,用凝胶成像仪扫描拍照。
2 结果与分析
2.1 水稻幼苗根部组织捕获效率分析
水稻种子培养约9 d后,下部PCR板的孔中存在大量的根,可以进行根部组织的捕获。具体方法如下:(1)从金属托盘中取出双层生长板(图2A),此时水稻幼苗已发育完全(图2B)。将橡皮筋留在原位,在两板之间打开一个间隙,小心地在上下板之间插入玻璃棒,在两个板之间形成一个间隙(图2C),从孔中可以清晰的观察到内部的根与苗(图2D)。(2)将4块24孔金属热块拼在一起放入金属盘中。小心地将95% (V/V)乙醇与干冰(乙醇500 mL,干冰3 kg)倒入玻璃盘中,直到混合液体的液面恰好低于金属热块的表面。金属热块冷却约30 min,使温度达到平衡(图2E)。(3)将双层生长板放置于金属热块上,下部PCR板插入金属热块对应孔中。双层生长板放在金属热块上约6 min,在此期间下部PCR板中的水将冷冻为冰(图2F)。6 min后将双层生长板从金属热块上移出,移除橡皮筋,并从板之间移除玻璃棒。向下按压上部深孔板,然后小心地分开上部深孔板与下部PCR板,清除下部PCR板表面的残冰(图2G)。此时,下部PCR板的孔中即保留了冷冻后的根部组织(图2H)。收获根部组织后,上部幼苗可转移到装满水或营养液的金属托盘中,以使幼苗继续生长 (图2I)。利用此方法,成功获得96个样本的根部组织,样本捕获的成功率为100%;此外,此方法获得的根部组织长度介于2~3 cm,重量约0.03~0.06 g,各样品组织均匀一致,为DNA提取奠定了良好基础。与传统剪取叶片法比较,本方法获取96个样本组织仅耗时约10 min,而剪取叶片法耗时约30 min。
2.2 DNA提取结果分析

图2 水稻幼根捕获与DNA提取
Fig.2 Rice root capture and DNA extraction
A:金属盘中培养9 d左右的水稻幼苗;B:培养9 d后水稻单株表型;C:上部种子板和下部根板之间插入玻璃棒,分隔两板,能快速高效捕获水稻根部的幼根;D:生长9 d后上部种子板孔中的幼苗与根;E:金属盘中放入干冰与95%酒精,中间放入4块24微孔板金属热块,进行冰浴降温捕获水稻幼根组织;F:在温度恒定后,把图C装置放入金属热块中,待下部结冰后,取出捕获的水稻幼根组织;G:冰浴后,分离的上部苗板与下部种子板;H:冰浴后,分离的下部种子板,内含捕获的幼根;I:水稻幼根根部组织被捕获后,把上部种子板放入金属盘中加入蒸馏水继续培养植株;J:钢珠分装器;K:PCR板与钢珠分装器组合装置,可把单个钢珠准确的放入PCR板的孔中;L:捕获的幼根与钢珠;M:高通量组织研磨机,用于容纳96孔PCR板,批量研磨根部
A:Rice seedlings cultivated for about 9 days in a metal tray; B: Rice phenotype cultivated after 9 days ; C:Inserting two glass rods between the upper seed plate and the lower root plate to separate the two plates, which can capture the rice roots quickly and efficiently; D:Seedlings and roots in the hole of the upper seed plate after 9 days of growth; E: Dry ice and 95% ethand is put in the metal tray, 4 pieces of 24 microporous plate metal thermal block are put in the middle, and the rice root tissue is captured by ice bath cooling; F:After the temperature is constant, the device of Figure C is placed in a metal thermal block, and after the lower portion is frozen, the captured young root tissue of the rice is taken out; G: The separated upper seedling plate and the lower seed plate after ice bath cooling; H: The separated lower seed plate with captured young toots after ice bath cooling; I:After the roots of the rice are captured, the upper seed plate is placed in a metal tray and distilled water is added to continue the cultivation of the plants; J:Steel ball dispenser; K:PCR plate and steel ball dispenser combination device, which can accurately put a single steel ball into the orifices of the PCR plate; L:The captured young roots and steel balls; M: High-throughput tissue grinder for holding 96-orifice PCR plates for batch grinding of roots
对获得的根部样本,采用4种方法进行DNA提取,并比较DNA提取效率。结果(表1)表明,4种方法均可提取水稻幼苗根部DNA,所获取的DNA可满足分子标记分析需求。从浓度与质量上来看,相比较其他3种方法,使用改良TRISEDTA法提取的 DNA个体间差异较小,整体浓度较高,浓度平均值为92.54 ng/vL,A260/280平均值为1.74,DNA纯度较高;基于磁珠的两种DNA提取方法,所获得的DNA浓度差别较大,其中根组织研磨后DNA提取浓度为55.68 ng/μL,显著高于不研磨的磁珠法。从效率上来看,改良TRIS-EDTA法批量提取DNA的时间明显低于其他3种方法,效率最高。此外,TRIS-EDTA法提取DNA的费用约为0.1元/样品,而CTAB法为0.8元/样品、磁珠法平均为0.5元/样品。可见,TRIS-EDTA法提取DNA的产量高、效率高、费用低,是比较理想的水稻根部DNA提取技术。
为了比较4种方法提取DNA的PCR扩增效果,利用GS3基因引物分别扩增4种方法获得的模板DNA。电泳检测结果(图3A)表明,4种方法所提取的DNA扩增产物均条带明亮、清晰,片段大小符合预期,说明4种方法所提取DNA均可用于分子标记分析。改良TRIS-EDTA法提取的基因组DNA在96个样本中均可扩增出清晰、明亮的目的条带,具有较高的稳定性(图3B)。
表1 不同提取方法的DNA浓度、质量及提取时间
Table 1 DNA concentration, quality and extraction time of different extraction methods
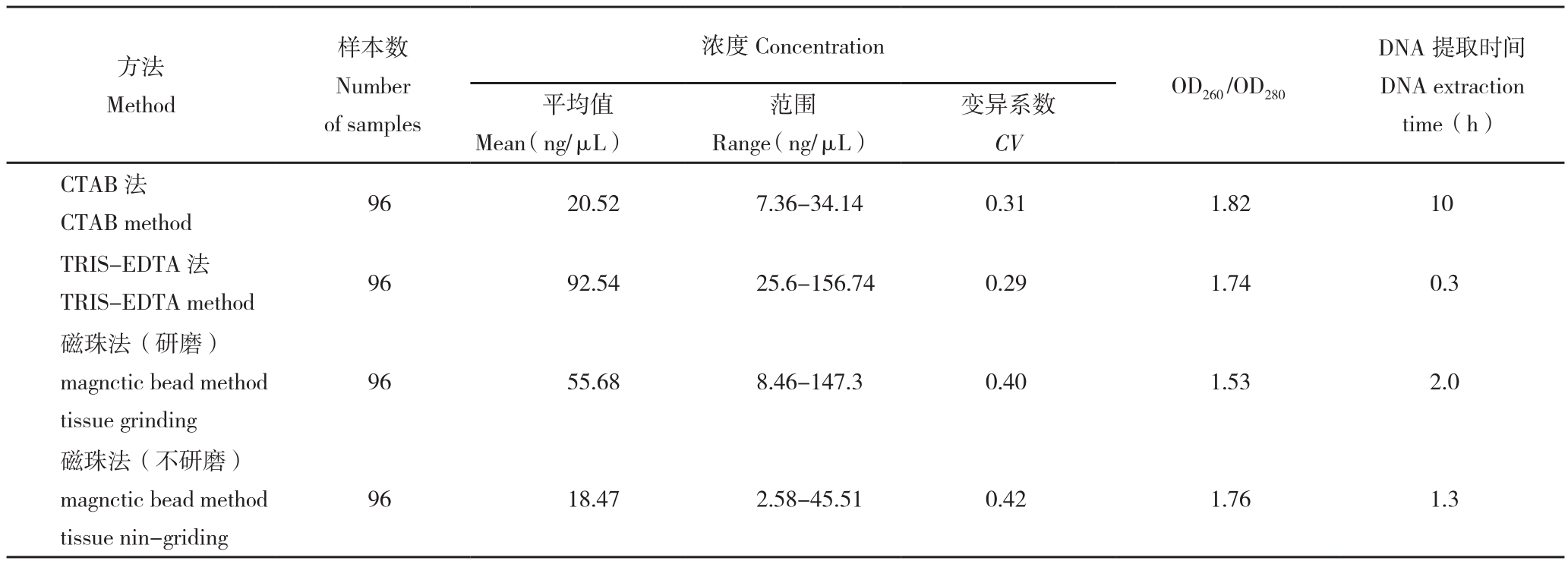
注:CTAB法、磁珠法(研磨)、磁珠法(不研磨)提取的DNA溶解于60 μL TE或蒸馏水中。
Note: DNA extracted by CTAB method, magnetic bead method (tissue grinding), magnetic bead method (tissue non- grinding) is dissolved in 60 μL TE or distilled water.
方法Method样本数Number of samples浓度Concentration OD260 /OD280 DNA提取时间DNA extraction time(h)平均值Mean (ng/μL)范围Range (ng/μL)变异系数CV CTAB法CTAB method 96 20.52 7.36-34.14 0.31 1.82 10 TRIS-EDTA法TRIS-EDTA method 96 92.54 25.6-156.74 0.29 1.74 0.3磁珠法(研磨)magnctic bead method tissue grinding 96 55.68 8.46-147.3 0.40 1.53 2.0磁珠法(不研磨)magnctic bead method tissue nin-griding 96 18.47 2.58-45.51 0.42 1.76 1.3
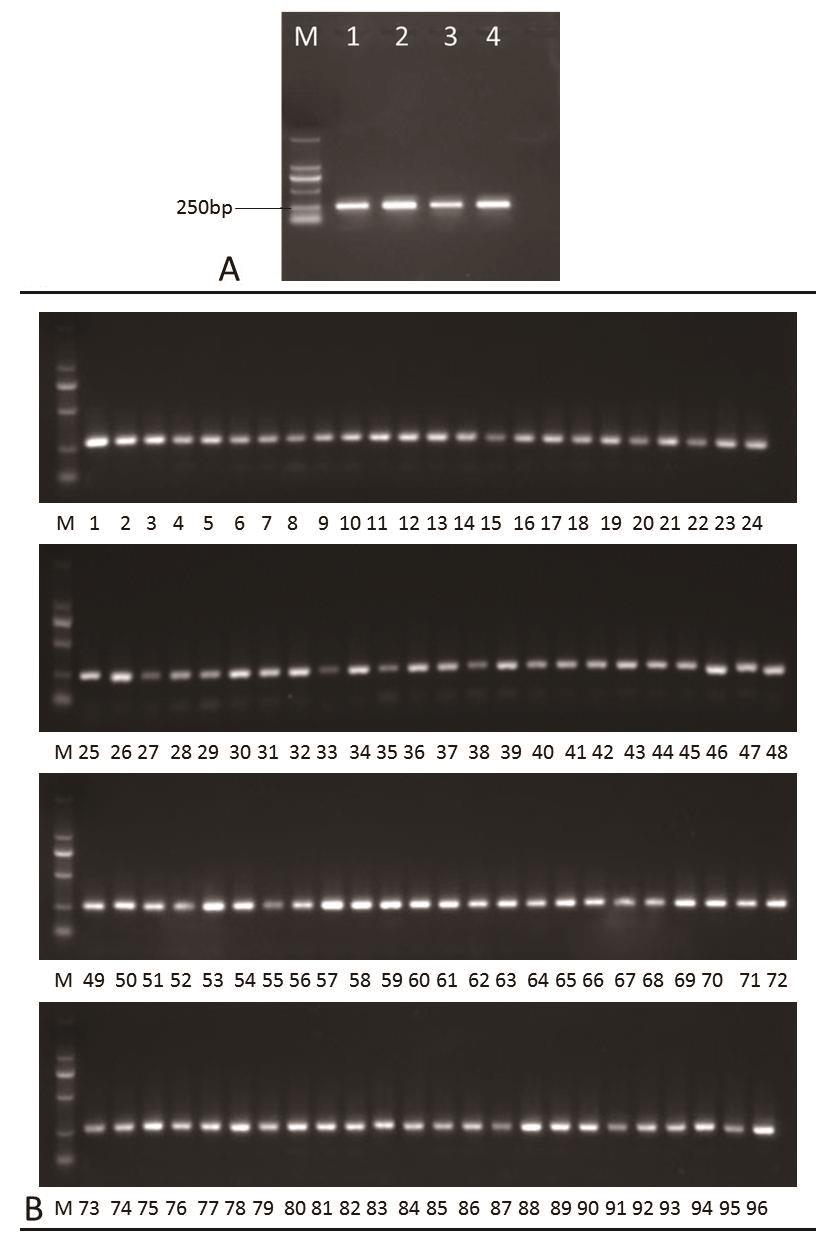
图3 PCR产物琼脂糖凝胶电泳结果
Fig.3 Agarose gel electrophoresis results of PCR product
A:4种方法提取DNA的PCR扩增产物检测,1:CTAB法,2:TRISEDTA法,3:磁珠法(组织研磨),4:磁珠法(组织不研磨);B:TRISEDTA法批量提取DNA的PCR扩增产物检测。M:DL2000 DNA Ladder Marker
A: Detection of PCR amplification products of DNA extracted by four methods,1: CTAB method,2: TRIS-EDTA method; 3: Magnetic bead method (tissue grinding); 4: Magnetic bead method (tissue non-grinding); B: Detection of PCR amplification products of DNA for batch extraction by TRIS-EDTA method.M: DL2000 DNA Ladder Marker
3 讨论
随着越来越多的分子标记可供植物育种者使用,迫切需要能对大量单株进行基因分型的高效方法[14-15]。而大量样本的种植管理、样本组织的高效获取及DNA的有效提取是高通量基因分型的关键环节[16-17]。
对于样本组织的获取,目前普遍采用的方法是技术人员在田间收取植物的叶片或其他组织器官并编号,但该方法过程烦琐,成本较高,较易出错,不适于大规模的样本收集[18-19]。针对传统方法缺点,本研究将水稻种植于96孔板中,实现根部组织的批量获取,同时继续保留样本活体,可以在获取基因型后把目标单株种植到田间。与前人方法相比,本研究的组织获取方法可显著降低田间劳作强度,且样品和DNA可以实现精确对应,具有高效便捷、省时省力的优点。限制高通量基因分型的另一个因素是DNA的快速提取,经典的CTAB或SDS可获取高质量的DNA,但提取过程烦琐,成本较高,不适于大规模的基因型检测[10,20]。目前也报道了各类简化DNA提取方法,但是这些方法仍然存在提取效率不高的问题[21-24],如赵国珍等[25]的方法提取96个样本DNA需耗时1 h。而本研究改良TRIS-EDTA法批量提取96个样本DNA的时间约为0.3 h,并且DNA质量完全满足分子标记需求。此外,改良TRIS-EDTA法提取DNA的费用约为0.1元/样品,而CTAB法为0.8元/样品。可见,本研究开发的TRIS-EDTA法提取DNA的产量高、效率高、费用低,是比较理想的水稻根部DNA提取技术,非常适合水稻的高通量基因分型。
4 结论
本研究开发的水稻根部组织DNA获取方法(TRIS-EDTA法)克服了传统水稻DNA样本获取工作量大、费时费力的缺点,可有效服务于水稻分子育种和突变体基因型筛选。此外,该方法还可用于收集水稻根部组织进行代谢组、转录组或蛋白质组的高通量分析,对于其他作物的DNA高通量提取也具有参考价值。
[1] 杨峰.水稻育种技术发展对水稻产量的影响[J].吉林农业,2016(8):48-49.doi:10.14025/j.cnki.jlny.2016.08.015.YANG F.Effects of rice breeding technology development on rice yield[J].Jilin Agriculture, 2016(8): 48-49.doi:10.14025/j.cnki.jlny.2016.08.015.
[2] 谢放鸣,彭少兵.杂交水稻在国外的发展历程与展望[J].科学通报,2016,61(35): 3858-3868.doi:CNKI:SUN:KXTB.0.2016- 35-020.XIE F M, PENG S B.Development and prospect of hybrid rice in foreign countries[J].Chinese Science Bulletin, 2016,61(35): 38583868.do i:CNKI:SUN:KXTB.0.2016-35-020.
[3] 魏凤娟,陈秀晨.分子标记技术及其在水稻育种中的应用[J].广东农业科学,2010,37(8): 185-187.doi:10.16768/j.issn.1004-874.2010.08.075.WEI F J, CHEN X C.Molecular marker technology and its application in rice breeding[J].Guangdong Agricultural Sciences, 2010, 37(8):185-187.doi:10.16768/j.issn.1004-874.2010.08.075.
[4] 杨瑰丽,陈莹,郭涛,黄明,黄翠红,王慧,李文建,陈志强.碳离子束辐照水稻诱变效应及突变体的筛选[J].华南农业大学学报,2018,39(2): 29-33.YANG G L, CHEN Y, GUO T, HUANG M, HUANG C H,WANG H,LI W J,CHEN Z Q.Mutagenic effects of rice by carbon beam irradiation and screening of mutants[J].Journal of South China Agricultural University, 2018, 39(2): 29-33.
[5] 斯琴图雅,高德玉,张玉宝,王强,梁宏斌,纪东清.我国水稻辐射诱变育种现状[J].黑龙江科学, 2013(5): 42-43.doi:10.3969/j.issn.1674-8646.2013.05.020.SI Q T Y, GAO D Y, ZHANG Y B, WANG Q, LIANG H B,JI D Q.Current status of radiation mutation breeding in rice in China[J].Heilongjiang Science, 2013(5): 42-43.doi:10.3969/j.issn.1674-8646.2013.05.020.
[6] CLARK K A, KRYSAN P J.Protocol: An improved high-throughput method for generating tissue samples in 96-well format for plant genotyping (Ice-Cap 2.0)[J]. Plant Methods, 2007 (3): 8-15.
[7] 刘毅,赵洪阳,唐金娟,王加红,刘国兰.一种高通量水稻DNA提取方法及其在种子纯度检测中的应用[J].上海农业学报, 2016,32(5):8-10.doi:10.15955/j.issn1000-3924.2016.05.02.LIU Y, ZHAO H Y, TANG J J, WANG J H,LIU G L.A high-throughput rice DNA extraction method and its application in seed purity detection[J].Shanghai Journal of Agricultural Sciences, 2016, 32(5): 8-10.doi:10.15955/j.issn1000-3924.2016.05.02.
[8] 马文东.水稻基因组DNA提取方法的研究[J].黑龙江农业科学,2014(1): 7-11.doi:10.3969/j.issn.1002-2767.2014.01.003.MA W D.Study on extraction methods of rice genomic DNA[J].Heilongjiang Agricultural Sciences,2014(1): 7-11.doi:10.3969/j.issn.1002-2767.2014.01.003.
[9] 袁云香,李海娟.水稻基因组DNA提取方法的研究进展[J].湖北农业科学, 2010,49(4): 968-971.doi:10.3969/j.issn.0439- 8114.2010.04.065.YUAN Y X, LI H J.Advances in the extraction of rice genomic DNA[J].Hubei Agricultural Sciences, 2010,49(4): 968-971.doi:10.3969/j.issn.0439-8114.2010.04.065.
[10] TAN S C, YIAP B C.DNA, RNA, and protein extraction: The past and the present[J].Journal of Biomedicine and Biotechnology, 2009,2009(5): 1-10.
[11] 母洪娜,曾继吾,易干军,陈金印.一种快速高效适于柑桔AFLP分析的DNA提取方法[J].广东农业科学, 2007(3): 25-26.doi:10.16768/j.issn.1004-874X.2007.03.009.MU H N, ZENG J W, YI G J, CHEN J Y.A rapid and efficient DNA extraction method for citrus AFLP analysis [J].Guangdong Agricultural Sciences, 2007 (3): 25-26.doi:10.16768/j.issn.1004-874X.2007.03.009.
[12] CHEN L K, GAO W W, CHEN S P, WANG L P,ZOU J Y,LIU Y Z,WANG H,CHEN Z Q,GUO T.High-resolution QTL mapping for grain appearance traits and co-localization of chalkiness-associated differentially expressed candidate genes in rice[J].Rice, 2016, 9(1):48-64.doi:10.1186/s12284-016-0121-6.
[13] 裔传灯,李玮,王德荣,蒋伟,王颖,周勇,梁国华,顾铭洪.水稻粒形基因GS3的功能标记开发与鉴定[J].江苏农业科学,2016,44(12): 64-67.doi:10.15889/j.issn.1002-13022016.12.016.SHI C D, LI W, WANG D R, JIANG W,WANG Y,ZHOU Y,LIANG G H,GU M H.Development and identification of functional markers of rice grain-shaped gene GS3 [J].Jiangsu Agricultural Sciences, 2016, 44(12): 64-67.doi:10.15889/j.issn.1002-1302.2016.12.016.
[14] 高美凤,王晓峰,夏莲红,曹月琴,顾明飞,杨建芳.3种水稻分子标记辅助育种技术的应用概况[J].上海农业科技, 2017(5): 49-51.doi: CNKI:SUN:SLYK.0.2017-05-022.GAO M F, WANG X F, XIA L H, CAO Y Q,GU M F,YANG J F.Application of three kinds of rice molecular marker-assisted breeding techniques[J].Shanghai Agricultural Science and Technology, 2017(5): 49-51.doi: CNKI:SUN:SLYK.0.2017-05-022.
[15] 姚姝,陈涛,张亚东,朱镇,赵庆勇,周丽慧,赵凌,赵春芳,王才林.利用分子标记辅助选择聚合水稻Pi-ta、Pi-b和Wx-mq基 因[J].作 物 学 报 , 2017,43(11): 1622-1631.doi:10.3724/SP.J.1006.2017.01622.YAO S, CHEN T, ZHANG Y D,ZHU Z,ZHAO Q Y,ZHOU L H,ZHAO L,ZHAO C F,WANG C L.Polymerization of Pi-ta, Pi-b and Wx-mq genes by molecular marker-assisted selection[J].Acta Agronomica Sinica, 2017, 43(11): 1622-1631.doi:10.3724/SP.J.1006.2017.01622.
[16] 田孟祥,张时龙,余本勋,何友勋,叶永印,李雪松.一种应用PCR缓冲液快速制备水稻DNA模板的方法[J].分子植物育种 ,2015,13(2): 438-442.TIAN M X, ZHANG S L, YU B X,HE Y X,YE Y Z,LI X S.A method for rapid preparation of rice DNA template using PCR buffer[J].Molecular Plant Breeding, 2015, 13(2): 438-442.
[17] 罗天宽,张小玲,朱世杨,徐谦,裘波音,卢华金,杨文清,唐征,刘庆,荆赞革,吴海涛.应用水稻叶片直接PCR扩增的方法[J].分子植物育种, 2015,13(11): 2590-2592.LUO T K, ZHANG X L, ZHI S Y,XU Q,QIU B Y,LU H J,YANG W Q,TANG Z,LIU Q,JING Z G,WU H T.Direct PCR amplification of rice leaves[J].Molecular Plant Breeding.2015, 13(11): 2590-2592.
[18] 李惠珠,陈仕军,刘朝东,陶计叁,傅荣富,王慧.优质水稻品种江航丝苗的选育及栽培技术要点[J].中国稻米, 2018, 24(6): 116-117.LI H Z, CHEN S J, LIU C D, TAO J S,FU R F,WANG H.Breeding and cultivation techniques of high quality rice variety Jianghang silk seedlings[J].China Rice,2018, 24(6): 116-117.
[19] 陈立凯,黄明,刘永柱,王慧,陈志强,郭涛.水稻开颖半不育突变体的观察、遗传分析和基因定位[J].中国农业科学, 2016,49(1):1-13.doi:10.3864/j.issn.0578-1752.2016.01.001.CHEN L K, HUANG M, LIU Y Z,WANG H,CHEN Z Q,GUO T.Observation、Genetic analysis and gene mapping of semi-sterile mutants in rice[J]. Scientia Agricultura Sinica, 2016, 49(1): 1-13.doi:10.3864/j.issn.0578-1752.2016.01.001.
[20] 张菊平,张长远,张树珍.苦瓜基因组DNA提取和RAPD分析[J].广东农业科学 , 2002(4): 18-20.doi:10.16768/j.issn.1004-874X.2002.04.008.ZHANG J P, ZHANG C Y, ZHANG S Z.Genomic DNA extraction and RAPD analysis of Momordica charantia[J].Guangdong Agricultural Sciences, 2002(4): 18-20.doi:10.16768/j.issn.1004-874X.2002.04.008.
[21] 孙川,陈 刚,饶玉春,张光恒,高振宇,刘坚,鞠培娜,胡江,郭龙彪,钱前,曾大力.水稻基因组DNA简易制备方法[J].中国水稻科学,2010,24(6): 677-680.doi:10.3969/j.issn.1001- 7216.2010.06.021.SUN C, CHEN G, RAO Y C, ZHANG G H,GAO Z Y,LIU J,JU P N,HU J,GUO L B,QIAN Q,ZENG D L.Simple preparation method of rice genomic DNA[J].China Rice Science, 2010, 24(6): 677-680.doi:10.3969/j.issn.1001-7216.2010.06.021.
[22] 孙川.水稻基因组DNA简易制备方法研究以及分子标记辅助选择改良稻米品质[D].扬州:扬州大学, 2010.doi: 10.7666/d.y1701963.SUN C.Simple preparation method of rice genomic DNA and molecular marker-assisted selection to improve rice quality [D].Yangzhou:Yangzhou University, 2010.doi:10.7666/d.y1701963.
[23] 邓力超,邱道寿,屠乃美,谭铭喜,张振臣,陈俊标,李淑玲,王泽清.PVP对烟草基因组DNA提取的影响[J].广东农业科学, 2009(5):37-39,57.doi:10.16768/j.issn.1004-874X.2009.05.017.DENG L C, LIU D S, TU N M, TAN M X,ZHANG Z C,CHEN J B,LI S L,WANG Z Q.Effects of PVP on the extraction of tobacco genomic DNA[J].Guangdong Agricultural Sciences, 2009(5): 37-39,57.doi:10.16768/j.issn.1004-874X.2009.05.017.
[24] 魏琦超,畅丽萍,周岩,宫俊丽,王慧娟.一种简便实用的玉米干种子基因组DNA提取方法[J].广东农业科学, 2009(6): 165-167.doi: 10.16768/j.issn.1004-874X.2009.06.052.WEI Q C, CHANG L P, ZHOU Y, GONG J L,WANG H J.A simple and practical method for extracting genomic DNA from dried corn seeds[J].Guangdong Agricultural Sciences, 2009(6): 165-167.doi:10.16768/j.issn.1004-874X.2009.06.052.
[25] 赵国珍,贾育林,严宗卜,CHRISTOPHER W DEREN,MELISSAH JIA,戴陆园.一种高效便捷的水稻DNA提取法及其应用[J].中国水稻科学, 2012,26(4): 495-499.doi:10.3969/j.issn.1001-7216.2012.04.016.ZHAO G Z, JIA Y L, YAN Z B, CHRISTOPHER W D,MELISSA H J,DAI L Y.An efficient and convenient method for rice DNA extraction and its application[J].Chinese Journal of Rice Science, 2012, 26(4):495-499.doi:10.3969/j.issn.1001-7216.2012.04.016.