【研究意义】 DNA条形码技术是利用基因组中一段公认的标准短序列来进行物种鉴定的分子生物学技术。DNA 条形码鉴定与其他分子鉴定方法比较具有三大优势:鉴定结果具有良好的重复性;鉴定方法通用性强;并可通过构建数据统一的鉴定平台,而易于推广和标准化[1-3]。近年来,随着对DNA 条形码技术的深入研究人们发现该技术在药用植物鉴定中具有广泛的应用前景,将是中药鉴定方法的革命性突破。目前,中药材DNA条形码分子鉴定指导原则建立了以 ITS2为核心、psbA-trnH为辅的植物类药材DNA条形码鉴定体系和以COI为主、ITS2为辅的动物类药材DNA条形码鉴定体系[4]。前者已纳入到2010版《中国药典》(增补本)。
【前人研究进展】 自2003年DNA条形码被提出以来,该技术已成为全球生物分类学的研究热点和方向。2003年,HEBERT 等对动物界11个门13 320个物种的CO1(cytochrome c oxidase subunit 1)基因序列进行分析,发现该序列种间变异能较好地区分除刺胞动物门以外的物种。随后的研究中,COI基因被公认为动物界中标准的DNA 条形码基因。植物由于在进化历程中容易发生杂交、网状进化等事件,而且同一基因在不同类群的进化速率可能不同,导致植物条形码的研究比动物要复杂得多。近几年提出了一些植物DNA条形码候选序列及其组合,但尚未达成一致意见。2009年,生命条形码联盟植物工作组(the plant working group of the consortium for thebarcode of life,CBOL)分析了550个物种907个样品后,推荐以rbcL+matK组成的复合序列作为整个植物界的DNA条形码序列的通用条形码序列,但由于扩增效率、鉴定成功率等问题,其在植物界的通用性仍受到质疑。此外,ITS2 片段在物种水平的变异较快,有更多的突变位点以区分不同的物种,因此在DNA条形码鉴定物种方面具有潜在的研究价值。
【本研究切入点】 黎族医药是黎族人民在长期与疾病斗争的医疗实践中不断积累的宝贵经验的总结,是中华民族传统医药的重要组成部分,由于黎族没有自己的文字,药物的名称和使用方法是靠语言代代相传,随着现代医药的普及,这一宝贵的历史医药文化遗产面临失传的危机,亟待我们进行抢救性发掘和保护[5]。据文献记载和民间调查,五指山区黎族使用的药用植物约有515种,隶属于125科360属[6-7],中大戟科植物多数种类有毒,有毒药用植物对治疗一些疾病有特殊疗效,具有不可替代性,但如果鉴定错误很容易引起中毒事件,重者甚至会危及患者生命,为确保用药安全,寻求准确快速的鉴定大戟科药用植物的方法是十分必要的。
本课题组之前的研究显示:候选DNA条形码ITS2序列对海南大戟科植物具有较强的鉴定能力[8],但是,其他热点DNA条形码序列对海南大戟科植物鉴定能力如何还需进一步认证。【拟解决的关键问题】 因此,本研究对ITS2、ITS、psbA-trnH、rbcL、matKDNA条形码序列对海南大戟科植物鉴定能力进行比较,以期筛选出用于鉴定海南大戟科植物最优的DNA条形码序列,从而为建立黎族药用大戟科植快速鉴定方法,确保用药安全及黎药的传承奠定基础。
1 材料与方法
1.1 试验材料
供试大戟科植物材料共29个种69个样本。样本形态学鉴定由海南医学院唐历波教授完成,凭证标本及数字影像信息保存于海南医学院基础医学与生命科学学院。样本来源见表1。
1.2 试验方法
1.2.1 植物叶片采集 每株采集健康无病斑叶片,用酒精棉球擦拭去除细菌等杂物,放入加有变色硅胶(青岛海洋化工有限公司)的密封袋中进行干燥。
1.2.2 DNA提取 利用MM300 DNA球磨仪(Retsch,德国)研磨30 s(2 000 r/min)。采用植物基因组DNA提取试剂盒(天根生化科技有限公司,中国)提取总DNA。利用核酸蛋白检测仪测定样本DNA的浓度,然后经1%的琼脂糖凝胶电泳进一步分析DNA提取结果。
表1 样本来源
Table 1 Source of samples
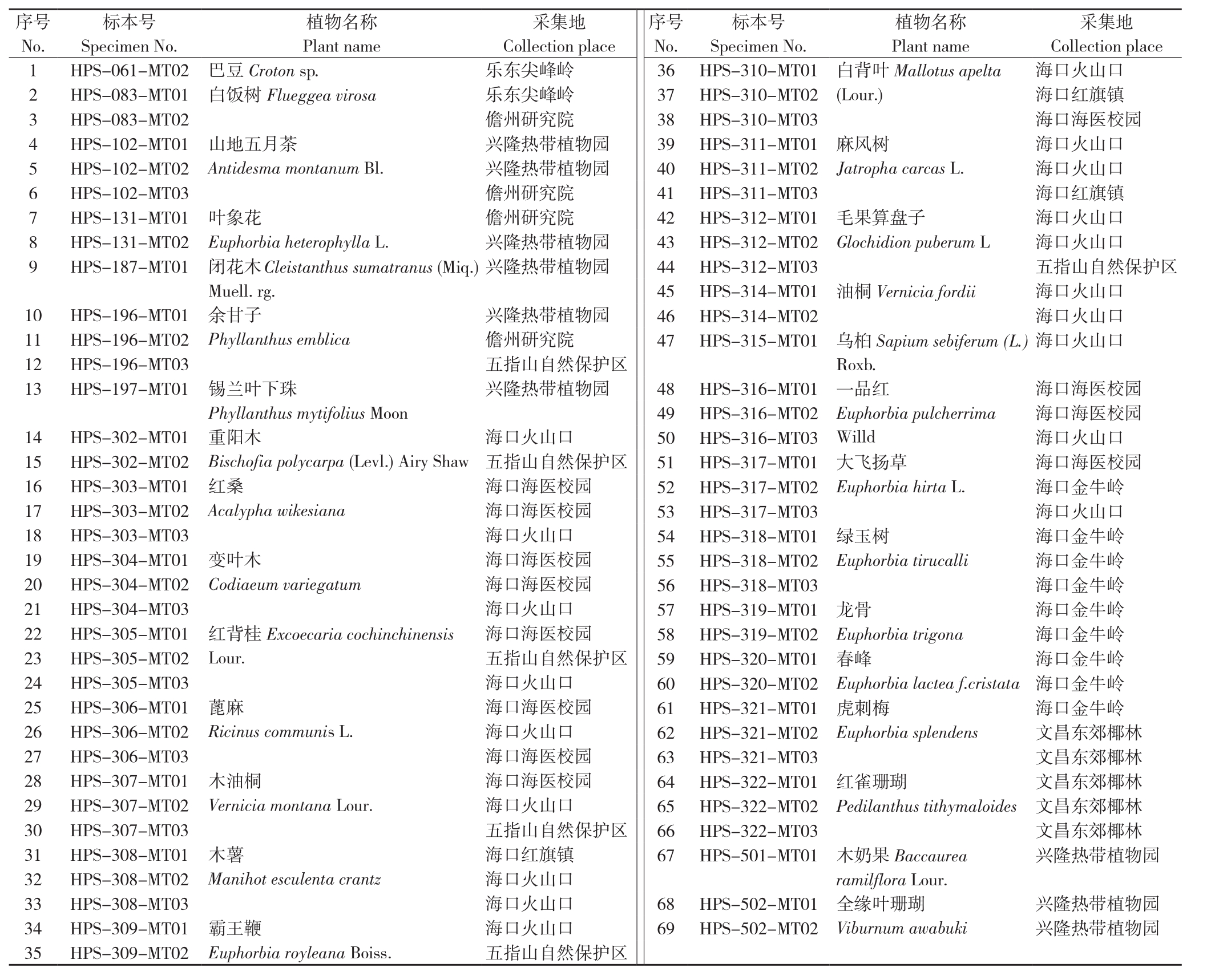
序号No.采集地Collection place 1 HPS-061-MT02 巴豆Croton sp. 乐东尖峰岭2 HPS-083-MT01 白饭树Flueggea virosa 乐东尖峰岭3 HPS-083-MT02 儋州研究院4 HPS-102-MT01 山地五月茶Antidesma montanum Bl.标本号Specimen No.植物名称Plant name兴隆热带植物园5 HPS-102-MT02 兴隆热带植物园6 HPS-102-MT03 儋州研究院7 HPS-131-MT01 叶象花Euphorbia heterophylla L.兴隆热带植物园10 HPS-196-MT01 余甘子Phyllanthus emblica儋州研究院8 HPS-131-MT02 兴隆热带植物园9 HPS-187-MT01 闭花木Cleistanthus sumatranus (Miq.)Muell.rg.兴隆热带植物园11 HPS-196-MT02 儋州研究院12 HPS-196-MT03 五指山自然保护区13 HPS-197-MT01 锡兰叶下珠Phyllanthus mytifolius Moon兴隆热带植物园14 HPS-302-MT01 重阳木Bischofia polycarpa (Levl.) Airy Shaw海口火山口15 HPS-302-MT02 五指山自然保护区16 HPS-303-MT01 红桑Acalypha wikesiana海口海医校园17 HPS-303-MT02 海口海医校园18 HPS-303-MT03 海口火山口19 HPS-304-MT01 变叶木Codiaeum variegatum海口海医校园20 HPS-304-MT02 海口海医校园21 HPS-304-MT03 海口火山口22 HPS-305-MT01 红背桂Excoecaria cochinchinensis Lour.海口海医校园23 HPS-305-MT02 五指山自然保护区24 HPS-305-MT03 海口火山口25 HPS-306-MT01 蓖麻Ricinus communis L.海口海医校园26 HPS-306-MT02 海口火山口27 HPS-306-MT03 海口海医校园28 HPS-307-MT01 木油桐Vernicia montana Lour.海口海医校园29 HPS-307-MT02 海口火山口30 HPS-307-MT03 五指山自然保护区31 HPS-308-MT01 木薯Manihot esculenta crantz海口红旗镇32 HPS-308-MT02 海口火山口33 HPS-308-MT03 海口火山口34 HPS-309-MT01 霸王鞭Euphorbia royleana Boiss.海口火山口35 HPS-309-MT02 五指山自然保护区序号No.采集地Collection place 36 HPS-310-MT01 白背叶Mallotus apelta(Lour.)标本号Specimen No.植物名称Plant name海口火山口37 HPS-310-MT02 海口红旗镇38 HPS-310-MT03 海口海医校园39 HPS-311-MT01 麻风树Jatropha carcas L.海口火山口40 HPS-311-MT02 海口火山口41 HPS-311-MT03 海口红旗镇42 HPS-312-MT01 毛果算盘子Glochidion puberum L海口火山口43 HPS-312-MT02 海口火山口44 HPS-312-MT03 五指山自然保护区45 HPS-314-MT01 油桐Vernicia fordii 海口火山口46 HPS-314-MT02 海口火山口47 HPS-315-MT01 乌桕Sapium sebiferum (L.)Roxb.海口火山口48 HPS-316-MT01 一品红Euphorbia pulcherrima Willd海口海医校园49 HPS-316-MT02 海口海医校园50 HPS-316-MT03 海口火山口51 HPS-317-MT01 大飞扬草Euphorbia hirta L.海口海医校园52 HPS-317-MT02 海口金牛岭53 HPS-317-MT03 海口火山口54 HPS-318-MT01 绿玉树Euphorbia tirucalli海口金牛岭55 HPS-318-MT02 海口金牛岭56 HPS-318-MT03 海口金牛岭57 HPS-319-MT01 龙骨Euphorbia trigona海口金牛岭58 HPS-319-MT02 海口金牛岭59 HPS-320-MT01 春峰Euphorbia lactea f.cristata海口金牛岭60 HPS-320-MT02 海口金牛岭61 HPS-321-MT01 虎刺梅Euphorbia splendens海口金牛岭62 HPS-321-MT02 文昌东郊椰林63 HPS-321-MT03 文昌东郊椰林64 HPS-322-MT01 红雀珊瑚Pedilanthus tithymaloides文昌东郊椰林65 HPS-322-MT02 文昌东郊椰林66 HPS-322-MT03 文昌东郊椰林67 HPS-501-MT01 木奶果Baccaurea ramilflora Lour.兴隆热带植物园68 HPS-502-MT01 全缘叶珊瑚Viburnum awabuki兴隆热带植物园69 HPS-502-MT02 兴隆热带植物园
1.2.3 PCR扩增及产物测序 引物与反应条件[9-13]:本研究引物的合成由上海生工生物工程股份有限公司完成,所使用引物及其相应的反应条件见表2。PCR反应体系为25 μL,体系包括MgCl2 2 μL(25 mmol/L),dNTP 2 μL(2.5 mmol/L),引物各1.0 μL(2.5 μmol/L),PCR缓冲液2.5 μL,聚合酶1.0 U(博彩生物科技有限公司,中国),总DNA 1 μL(约30 ng)。PCR仪型号:promega M7501 PCR Master Mix 10 reactions TAQ。琼脂糖凝胶电泳检测PCR扩增结果。PCR扩增产物的测序工作由深圳华大基因生物医学工程有限公司完成。
1.2.4 序列分析 采用PCR扩增效率、测序成功率和序列获得率来评价5对候选序列的获得情况。测序峰图文件中,ITS2序列依照KELLER等[14]的方法处理,psbA-trnH序列根据GenBank上的注释处理,其余序列采用CodonCode Aligner处理,剔除不确定碱基数目大于10个和长度小于100 bp的序列。将拼接后的序列用软件MEGA6.0进行分析比对[15],并基于K2P 模型进行遗传距离等分析,使用TAXON DNA软件分析序列种内、种间变异并作barcoding gap图。
2 结果与分析
2.1 PCR扩增率、测序成功率及序列获得率分析 本研究通过比较5条候选序列的PCR扩增率(出现明显PCR条带即判定为成功)、测序成功率(测序后获得高质量的序列即判定为成功)和序列获得率(序列获得率=PCR扩增效率×测序成功率)发现,ITS序列扩增效率最高、达98.4%,psbA-trnH序列居其次、为95.3%,matK序列最低、为62.5%;而测序成功率以psbA-trnH序列最高、为94.29%,其次是ITS2、为91.34%,ITS序列测序成功率最低、为78.06%;5条候选序列的序列获得率分别为86.96%、76.81%、89.86%、79.71%、49.28%(图1)。
表2 植物DNA条形码通用引物序列及PCR反应条件
Table 2 General primer sequence and PCR reaction conditions of plant DNA barcode

PCR反应条件PCR reaction conditions ITS2 S2F ATGCGATACTTGGTGTGAAT 94℃,5min S3R GACGCTTCTCCAGACTACAAT 94℃ ,30s;56℃ ,30s;72℃ ,45s 40cycles 72℃,10min ITS 5F GGAAGTAAAAGTCGTAACAAGG 94℃,5min 4R TCCTCCGCTTATTGATATGC 94℃ ,1min;50℃ ,1min;72℃ ,1.5min+3s/cycle 30cycles 72℃,7min rbcL rbcLa-F ATGTCACCACAAACAGAGACTAAAGC 95℃,4min rbcLa-R GTAAAATCAAGTCCACCRCG 94℃ ,30s;55℃ ,1min;72℃ ,1min 35cycles 72℃,10min psbA-trnH fwd GTTATGCATGAACGTAATGCTC 95℃,4min rev CGCGCATGGTGGATTCACAATCC 94℃ ,30s;55℃ ,1min;72℃ ,1min 35cycles 72℃,10min matK 3F-KIM CGTACAGTACTTTTGTGTTTACGAG 94℃,1min 1R-KIM ACCCAGTCCATCTGGAAATCTTGGTTC 94℃ ,30s;52℃ ,20s;72℃ ,50s 35cycles 72℃,5min序列Sequence引物Primer引物序列(5'-3')Priner sequence(5'-3')
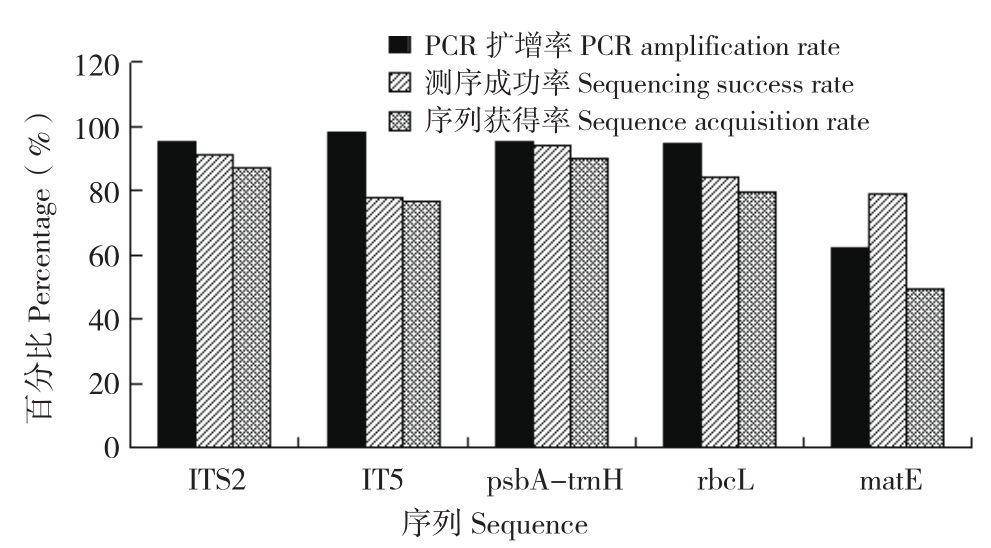
图1 5条序列PCR扩增率、测序成功率及序列获得率
Fig.1 PCR amplification rate,sequencing success rate and sequence acquisition rate of five sequences
2.2 序列长度和变异系数分析
用MEGA 6.0软件对所得序列进行比对分析,比较不同序列比对长度、变异位点数、信息位点数等参数。分析结果(表3)表明,ITS2序列的变异系数和信息位点系数均最大,其次为ITS序列,最小是rbcL序列;psbA-trnH序列的变异系数大于matK序列,但psbA-trnH序列的信息位点系数小于matK序列。
2.3 不同序列的Barcoding Gap分析
利用TAXON DNA软件对不同DNA条形码序列对供试材料种间、种内遗传距离进行分析,并作出barcoding gap图,结果(图2)表明,ITS2序列有明显的barcoding gap,种内变异、种间遗传距离具有明显差异,形成明显的种间与种内变异间隔区,该序列种内变异幅度较小,种间变异幅度较大,种内变异区间为0.05~0.10,种间变异区间为0.25~0.30,种间与种内变异分布呈两边分开的趋势,有利于物种鉴定;ITS序列存在barcoding gap,但不及ITS2序列显著,种内最大变异距离值1.1716,种间最小变异距离值0.2010,且种内变异和种间变异有较多的重叠区,种内和种间变异幅度都较大,不利于物种鉴定;psbA-trnH和matK序列barcoding gap均不明显,种内变异和种间变异有较多的重叠区,种内和种间变异幅度较大,不利于物种鉴定;rbcL序列barcoding gap不明显,但种内变异和种间变异重叠比例较小,该序列种内变异幅度较小,种间变异幅度较大,种内最大的变异区间为0.02~0.025。不能作为单独的条形码鉴定物种,但可以作为ITS2的补充条形码。
表3 5种序列的长度、变异位点和信息位点
Table 3 Length,variation sites and information sites of five sequences
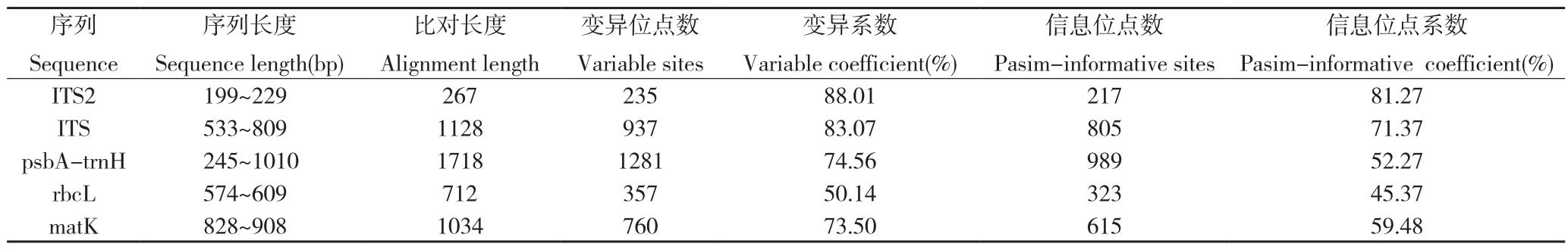
信息位点系数Pasim-informative coefficient(%)ITS2 199~229 267 235 88.01 217 81.27 ITS 533~809 1128 937 83.07 805 71.37 psbA-trnH 245~1010 1718 1281 74.56 989 52.27 rbcL 574~609 712 357 50.14 323 45.37 matK 828~908 1034 760 73.50 615 59.48序列Sequence序列长度Sequence length(bp)比对长度Alignment length变异位点数Variable sites变异系数Variable coefficient(%)信息位点数Pasim-informative sites
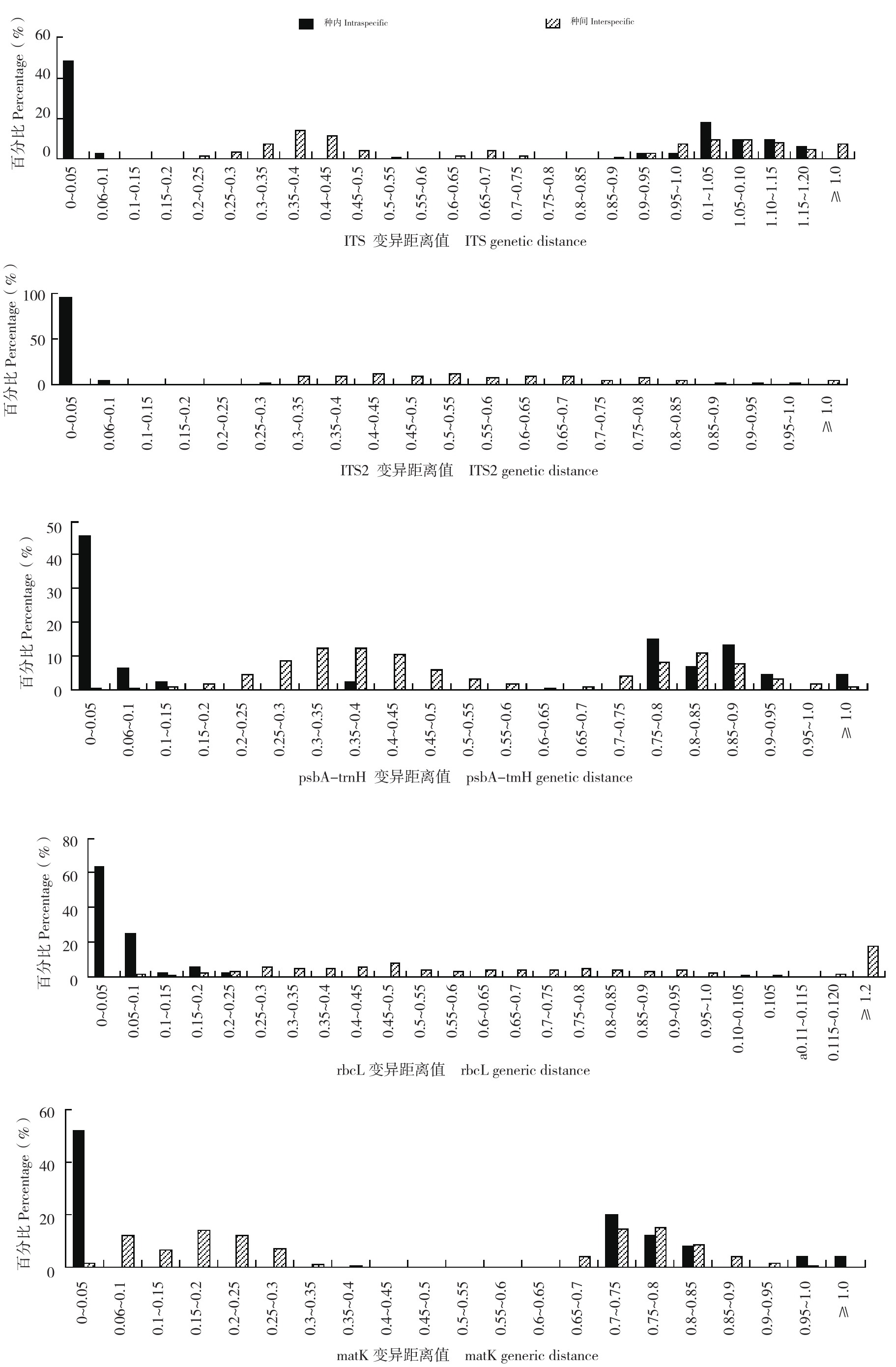
图2 5个候选序列的barcoding gap
Fig.2 Barcoding gap of 5 candidate sequences
3 讨论
DNA条形码的核心工作之一是寻找适合的条形码序列。 与线粒体CO1基因在动物条形码研究中的优异表现相比,植物DNA条形码研究进展相对缓慢[16-19]。由于植物线粒体基因组进化速率较慢,因此条形码序列主要在叶绿体和核基因上进行选择,被提议的候选序列主要有叶绿体编码区rbcL、rpoC1、ycf5、matK 和非编码区psbA-trnH序列,及核基因ITS、ITS2序列等[20-22]。
本研究通过对ITS2、ITS、psbA-trnH、rbcL、matK等热点条形码候选序列的分析比较发现,ITS序列种间变异要小于ITS2序列。其主要原因是ITS序列的变异主要集中在ITS1区段(平均长度为250 bp左右)和ITS2区段(平均长度250 bp左右),中间有大约160 bp的5.8 S高度保守区,因而降低了其种间变异[12],从公布的ITS通用引物来看,在各个科属的通用性较差。本研究中,ITS序列虽然具有较高的序列获得率,但其物种鉴定效率很低,因其具有较大的种内变异不利于分类和鉴定。
ITS2在海南大戟科物种鉴定方面具有以下优势:首先,无论在新鲜或降解组织中都较容易扩增和测序,具有很高的应用价值;其次,ITS2序列可根据不同物种类型基于隐马尔可夫模型的HMM 注释方法[14]去除两端5.8 S和26 S区段,获得标准ITS2间隔区序列,使后续数据处理更准确可靠,这也是目前其他候选条形码序列不具备的优势。另外,ITS2序列具有进化速率快的特点,较高的变异程度使其在区分物种方面能力较强。psbA-trnH间隔区在被子植物中具有高度变异性[23],其两端具有75 bp的保守区序列,便于设计通用引物[24],在大戟科中的扩增效率达95.3%,但其序列较长、存在大量的Poly(A)结构域[25],降低了测序的质量,同时在不同物种中普遍存在插入/缺失的情况,从而导致不同物种该片段长度变异较大,难以进行比对,给数据分析带来一定的困难。从本试验结果来看,psbA-trnH序列虽然具有较高的序列获得率,但其物种鉴定效率很低,因其具有较大的种内变异不利于分类和鉴定。
matK序列是植物叶绿体DNA中进化较快的一条编码区序列,目前对于matK作为DNA条形码对植物的鉴定能力的研究仍存在较大的分歧[13,17,26]。本研究中,matK基因序列获得率过低,会受到扩增和测序的限制,且种内与种间变异重叠比例大,物种鉴定效率很低,不适合作为大戟科DNA条形码鉴定的候选序列。
CBOL在最新的研究中推荐rbcL+matK作为植物条形码序列[27]。rbcL序列在GenBank中有大量的数据,并具有通用、易扩增、易比对的优点,尽管rbcL序列种内变异和种间变异barcoding gap不明显,但种内和种间遗传变异重叠较小,序列的种内变异幅度和变异样本数的比例明显较小,与其种间变异幅度和变异样本数比例较大形成鲜明的对照,有利于区分物种。尽管rbcL基因的PCR扩增率和序列获得率都小于ITS2序列,其在种的水平鉴定成功率也不及ITS2序列,但是可将其作为鉴别大戟科植物的DNA条形码的补充码,用以证明ITS2序列鉴定物种亲缘关系的准确性。
4 结论
本研究通过DNA条形吗技术,比较ITS2、ITS、psbA-trnH、rbcL、matK序列对海南大戟科植物的鉴定能力,结果表明,在以上候选DNA条形码中,ITS2条形码序列对海南大戟科植物鉴定能力强,可用于海南大戟科植物的快速鉴定。另外,rbcL序列不能作为单独的条形码鉴定物种,但可以作为ITS2的补充条形码。
[1]HEBERT P D,CYWINSKA A,BALL S L,WAARD J R..Biological identifications through DNA barcodes [J].Proc R Soc Lond B Biol Sci,2003,270: 313-321.doi:10.1098/rspb.2002.2218.
[2]GREGORY T R.DNA barcoding does not compete with taxonomy[J].Nature,2005,434: 1067.doi:10.1038/4341067b.
[3]MILLER S E.DNA barcoding and the renaissance of taxonomy[J].Proc Natl Acad Sci USA,2007,104: 4775-4776.doi:10.1073/pnas.0700466104.
[4]国家药典委员会.中华人民共和国药典2010年版(增补本)[M].北京:中国医药科技出版社,2014.
The National Pharmacopoeia Commission.Pharmacopoeia of the people’s Republic of China 2010 edition (updated)[M].Beijing: China Medical Science and Technology Press,2014.
[5]曾渝,刘明生,杨俊斌.海南黎族医药亟待抢救发掘[J].中国药业,2006 (11):18-19.doi:10.3969/j.issn.1006-4931.2006.11.015.
Zeng Y,Liu M S; Yang J B.Hainan Li nationality Medicine needs to be rescued and excavated urgently[J].China Pharmaceuticls,2006(11):18-19.doi:10.3969/j.issn.1006-4931.2006.11.015.
[6]刘明生.黎药学概论[M].北京:人民卫生出版社,2008.
LIU M S.Introduction to Li Pharmaceutical[M].Beijing: people’s Health Publishing House,2008.
[7]钟捷东.黎族医药[M].海口:海南出版社,2008.
ZHONG J D.Li Medicine[M].Haikou: Hainan Press,2008.
[8]李栎,徐腊红,唐历波.ITS2条形码序列对海南大戟科植物 的 鉴 定[J]. 中 草 药 .2016,44(9): 1572-1577.doi:10.7501/j.issn.0253-2670.2016.09.021.
LI L,XU L H,TANG L B.Identification of plants in Euphorbiaceae in Hainan by ITS2 barcoding sequence[J].Chinese Traditional and Herbal Drugs,2016,44(9): 1572-1577.doi:10.7501/j.issn.0253-2670.2016.09.021.
[9]李栎,肖憬,苏振宇,黄瑶,唐历波.ITS2条形码序列对茜草科黎药植物的鉴定[J].中草药,2013,44(13): 1814-1818.doi: 10.7501/j.issn.0253-2670.2013.13.021.
LI L,XIAO J,SU Z Y,HUANG Y,TANG L B.Identification of Li Medicine plants in Rubiaceae using ITS2 barcode sequence[J].Chinese Traditional and Herbal Drugs,2013,44(13): 1814-1818.doi:10.7501/j.issn.0253-2670.2013.13.021.
[10]CHEN S L,YAO H,HAN J P,LIU C,SONG J Y,SHI L C,ZHU Y J,MA X Y,GAO T,PANG X H,LUO K,LI Y,LI W,LEON C.Validation of the ITS2 Region as a novel DNA barcode for identifying medicinal plant species[J].PLoS ONE,2010,5: e8613.doi:10.1371/journal.pone.0008613.
[11]SASS C,LITTLE D P,STEVENSON D W,SPECHT C D.DNA barcoding in the Cycadales: testing the potential of proposed barcoding markers for species identification of Cycads[J].PLoS ONE,2007,2:e1154.doi:10.1371/journal.pone.0001154.
[12]SONG J Y,YAO H,LI Y,LI X W,LIN Y L,LIU C,HAN J P,XIE C X,CHEN S L.Authentication of the family Polygonaceae in Chinese pharmacopoeia by DNA barcoding technique[J].Ethnopharmacol,2009,124: 434-439.doi:10.1016/j.jep.2009.05.042.
[13]LAHAYE R,VANDER BANK M,BOGARIN D,BOGARIN D,WARNER J,PUPULIN F,GIGOT G,MAURIN O,BUTHOIT S,BARRACLOUGH T G,SAVOLAINEN V.DNA Barcoding the floras of biodiversity hotspots[J].Proc Natl Acad Sci USA,2008,105: 2923-2928.doi:10.1073/pnas.0709936105.
[14]KRESS W J,WURDACK K J,ZIMMER E A,WEIGT,L A,JANZEN,D H.Use of DNA barcodes to identify flowering plants[J].Proc Natl Acad Sci USA,2005,102: 8369-8374.doi:10.1073/pnas.0503123102.
[15]GAO T,YAO H,SONG J Y,LIU C,ZHUA Y J,MA X Y,PANG X H,XU H X,CHEN S L.Identification of medicinal plants in the family Fabaceae using a potential DNA barcode ITS2[J].Ethnopharmacol,2010,130: 116-121.doi:10.1016/j.jep.2010.04.026.
[16]HEBERT P D,RATNASINGHAM S,DE WAARD J R.Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species[J].Proc R Soc Biol Sci Ser B,2003,270: S96-S99.doi:10.1098/rsbl.2003.0025.
[17]VENCES M,THOMAS M,BONETT R M,VIEITES D R.Deciphering amphibian diversity through DNA barcoding: chances and challenges[J].Philos Trans R Soc Lond B Biol Sci,2005,360: 1859-1868.doi:10.1098/rstb.2005.1717.
[18]WARD R D,ZEMLAK T S,INNES B H,LAST P R,HEBERT P D.DNA barcoding Australia's fish species[J].Philos Trans R Soc Lond B Biol Sci,2005,360: 1847-1857.doi:10.1098/rstb.2005.1716.
[19]Newmaster S G,Fazekas A J,Ragupathy S.DNA barcoding in land plants: evaluation of rbcL in a multigene tiered approach[J].Can J Bot,2006,84: 335-341.doi:org/10.1139/b06-047.
[20]PENNISI E.Taxonomy.Wanted: a barcode for plants[J].Science,2007,318: 190-191.doi:10.1126/science.318.5848.190.
[21]CHASE M W,COWAN R S,HOLLINGSWORTH P M,BERG C V,MADRINAN S,PETERSEN G,SEBERG O,JORGSENSEN T,CAMERON K M,CARME M,PEDERSEN N,HEDDERSON T,CONRAD F,SALAZAR G A,RICHARDSON J E,HOLLINGSWORTH M L,BARRACLOUGH T G,KELLY L,WILKINSON Mike.A proposal for a standardised protocol to barcode all land plants[J].Taxon,2007,56: 295-299.doi:10.2307/25065788.
[22]KRESS W J,ERICKSON D L.A two-locus global DNA barcode for land plants: the coding rbcL gene complements the non-coding trn H-psb A spacer region.PLo S ONE,2007,2: e508.doi:10.1371/journal.pone.0000508.
[23]朱英杰,陈士林,姚辉,谭睿,宋经元,罗焜,鲁静.重楼属药用植物DNA条形码鉴定研究[J].药学学报,2010,45 (3):376-382.doi:10.16438/j.0513-4870.2010.03.004.
ZHU Y J,CHEN S L,YAO H,TAN R,SONG J Y,LUO K,LU J.DNA barcoding the medicinal plants of the genus Paris[J].Acta Pharmaceutica Sinica,2010,45 (3): 376-382.doi: 10.16438/j.0513-4870.2010.03.004.
[24]刘震,陈科力,罗焜,潘宏林,陈士林.忍冬科药用植物DNA条形码通用序列的筛选[J].中国中药杂志,2010,35(19): 2527-2532.doi:10.4268/cjcmm20101906.
LIU Z,CHEN K L,LUO K,PAN H L,CHEN S L.DNA barcoding in medicinal plants Caprifoliaceae[J].China Journal of Chinese Materia Medica,2010,35(19): 2527-2532.doi:10.4268/cjcmm20101906.
[25]罗焜,陈士林,陈科力,宋经元,姚辉,马新业,朱英杰,庞晓慧,余华,李西文,刘震.基于芸香科的植物通用DNA条形码研究[J].中国科学(生命科学),2010,40(4): 342 -358.doi:CNKI:SUN:JC XK.0.2010-04-007.
LUO K,CHEN S L,CHEN K L,SONG J Y,YAO H,MA X Y,ZHU Y J,PANG X H,YU H,LI X W,LIU Z.Assessment for universal plant DNA barcodes based on species of Rutaceae family[J].Chinese Science(Life Science) ,2010,40(4): 342 -358.doi:CNKI:SUN:JC XK.0.2010-04-007.
[26]FAZEKAS A J,BURGESS K S,KESANAKURTI P R,GRAHAM S W,MEWMASTER S G,HUSBAND B C,PERCY D M,HAJIBABAEI M,BARRERTT S.Multiple multiloeus DNA bareodes from the plastid genome diseriminate plant species equally well[J].PLo S One,2008,3,e2802.doi:10.1371/journal.pone.0002802.
[27]CBOL Plant Working Group.A DNA barcode for land plants[J].Proc Natl Acad Sci USA,2009,106: 12794-12797.doi:10.1073/pnas.0905845106.