【研究意义】目前我国乃至全世界动物源细菌存在耐药现象十分普遍,多重耐药、泛耐药的菌株不断出现,给公共卫生和食品安全造成了很大威胁[1-2]。实现耐药性控制的前提条件是实时、准确地进行细菌的耐药性监测。目前耐药监测的主要手段是对采样细菌进行药物敏感性试验,再利用试验结果获取监测范围内致病菌感染发生率及耐药动态。而这些结果是动态预测耐药趋势及抗菌药物治疗效果、指导临床用药、研究耐药机理的依据,同时也为政府耐药性监管、新药研发等工作提供支持[3]。细菌药物敏感性试验(antimicrobial susceptibility test,AST),简称药敏试验,是一种定量测定微生物对抗菌药物耐受程度的方法[2-8]。目前我国大部分养殖场,特别是中小型养殖场使用最多的AST试验方法是微量肉汤稀释法[2,6]。因此,研究提高药敏试验结果判读效率具有重要意义。【前人研究进展】微量肉汤稀释法主要过程是将分离培养后的菌株加入已配置不同浓度药剂的微孔板试剂盒的各个微孔中,孵育一段时间后,通过观察试剂盒中微孔的颜色或浑浊度变化来确定最低抑菌浓度(minimal inhibitory concentration,MIC)。目前,多数实验室通过实验人员肉眼观察来确定细菌MIC值。虽然近年国内外市场已出现了一些基于微量肉汤稀释法的自动快速检测系统,但仪器价格昂贵、试验条件要求复杂,无法支持我国基层的养殖场,特别是中小型养殖户批量测试的需求。而人工读取数据的方法效率低,且对实验人员眼力、体力是一个严重考验,无法长时间连续工作。近年来人工智能技术的突破引领计算机视觉的飞速发展,物体识别问题已初步解决,例如,ImageNet挑战赛的物体识别正确率已经达到人类水平。这方面研究成果正大规模应用于人们生产、生活场景,如刷脸支付、视频/监控分析等[9-11]。【本研究切入点】本研究将卷积神经网络在物体识别方面的研究成果应用于药敏试验中结果的判读,首先利用手机等设备采集药敏试验结果图像,再利用卷积神经网络进行试验结果识别,最终通过微孔坐标和药物浓度分布计算得出细菌耐药试验的MIC值。【拟解决的关键问题】本研究选择在图像分类领域表现优异的Inception_V4模型和MobileNet_V1模型进行药敏试验结果识别,通过对比分析选择更适合试验环境的卷积神经网络进行试验结果图像识别,提高MIC值计算的精度和判读效率。
1 研究基础
1.1 药敏试验
微量肉汤稀释法是目前最常用的药敏试验方法。该法采用96孔微量梯度稀释药敏板(简称96孔板),对每个微孔按顺序添加不同浓度的多种药物,用于测量某种细菌对指定药物的MIC值[4]。药敏试验结果中粉红色的圆孔代表有细菌生长,深蓝色或紫色的圆孔代表无细菌生长。由于高浓度的微孔中细菌不易生长而较低浓度的微孔中细菌容易生长,而且同一种药物的微孔从高浓度至低浓度按顺序排列,因此正常情况下微孔的颜色排列从蓝色逐渐变为粉色。第一个出现的粉色微孔的前一个紫色微孔对应的药物浓度即为所测细菌对所测药物的MIC值。如果出现同一种药物相邻微孔颜色从粉色突变为蓝色,或出现其他渐变的颜色排列,则认为发生“跳孔”现象,无法获得正确的MIC值,需要重新试验。
1.2 卷积神经网络
卷积神经网络能够对原始图像进行层次化抽象处理,自行抽取图像的特征包括颜色、纹理、形状等,从而取代手工选择图像特征的繁重工作[12]。与传统的神经网络相比,卷积神经网络具有适用性强、泛化能力强、全局优化训练参数少等优点[13]。卷积神经网络的结构一般为输入层-卷积层-池化层-卷积层-池化层-全连接层-输出层。其中输入层是整个神经网络的输入,在处理图像的卷积神经网络中,它是指图片的像素矩阵。卷积层通过深入分析神经网络中的每一个小块获取抽象程度更高的特征。池化层对卷积层输出的特征图传递进行特征选择和信息过滤。在多轮卷积层和池化层处理后,一般会由1个或2个全连接层来总结之前的特征并将其映射到样本空间中。输出层使用逻辑函数或归一化指数函数输出计算结果[10,12]。
1.3 Inception_V4网络模型
Inception网络源于2014年ILSVRC竞赛冠军神经网络GoogleNet(Inception_V1),Inception_V4是目前应用最广泛的版本。Inception_V4网络结构(图1)中的stem模块由多个卷积层和池化层组成,其作用是预处理数据。stem模块连 接3种Inception模 块,3种Inception模 块 间的Reduction模块起到池化作用。Inception-A模块使用两个3×3卷积,并且使用平均池化,主要处理35×35的特征图。Inception-B模块使用1×N和N×1卷积,同样使用平均池化,主要处理17×17的特征图。Inception-C模块用1×3卷积和3×1卷积。

图1 Inception_V4网络结构
Fig.1 Convolutional neural network model of Inception_V4
1.4 MobileNet_V1网络模型
MobileNet分类网络是Google研究团队针对嵌入式设备提出的一种轻量级的深度神经网络。MobileNet模型基于深度可分解的卷积,将标准卷积分解为1个深度卷积和1个点卷积(1×1卷积核)。这种分解使计算时间减少为分解前的1/9,计算参数减少为分解前的1/7。深度卷积层的每个滤波器都跟输入的每个通道进行卷积,点卷积层负责合并深度卷积层的结果[14]。标准卷积分解如图2所示。

图2 标准卷积分解过程
Fig.2 Decomposition process of standard convolution
MobileNet_V1的网络结构如表1所示。Moblienet_V1模型第一层为全卷积层,接着是13对深度卷积层和点卷积层组合,接下来是池化层、全连接层以及softmax层,共30层。所有层都包含批规格化操作且使用ReLU非线性激活函数,除了全连接层没有非线性激活函数直接送入softmax层进行分类。
2 试验设计与实现
国家兽医微生物耐药性风险评估实验室是国家发改委、农业部批准立项的国家重点实验室。畜禽重要病原菌耐药性监测是该实验室的一项重要工作,目前已积累从全国各省养殖场获取并分离到的细菌4万多株,且每年新增畜禽样品1万多个、新分离细菌2 000株左右。在药敏监测工作中,已保存大量药敏监测信息,包括样品、菌株、药品、药敏板图像和试验结果等信息。本研究依托该实验室开展。
首先对药敏试验图像进行校正、增强、切割形成适合卷积神经网络训练和验证的试验数据集。试验数据集中每张图像是1个微孔图像。标签分有菌、无菌两类,依据人工判读的试验结果为每张图片设置标签。
目前业界开发了许多成熟的深度学习框架包 括Tensorflow[15]、Caffe[16]、PyTorch[17]等,这些框架推动了机器学习和深度神经网络研究,同时更方便将卷积神经网络等模型应用于更多专业领域,引领、带动了应用的发展。本研究选择Tensorflow中用来构建、训练、评估复杂模型的轻量化库TF-Slim来开展试验工作。图像识别采用Inception_V4和MobileNet_V1两种神经网络模型,两个模型来自TF-Slim。对两个模型分别进行模型训练、图像识别,目标是挑选对药敏试验结果识别准确率、效率都比较高的模型。对识别结果结合96孔板药物浓度排列对照计算得到药敏试验的MIC值结果,并依据对获取的MIC值与人工判别结果进行对比,验证结果的可靠性。
表1 MobileNet_V1网络结构
Table 1 Convolutional neural network of MobileNet_V1
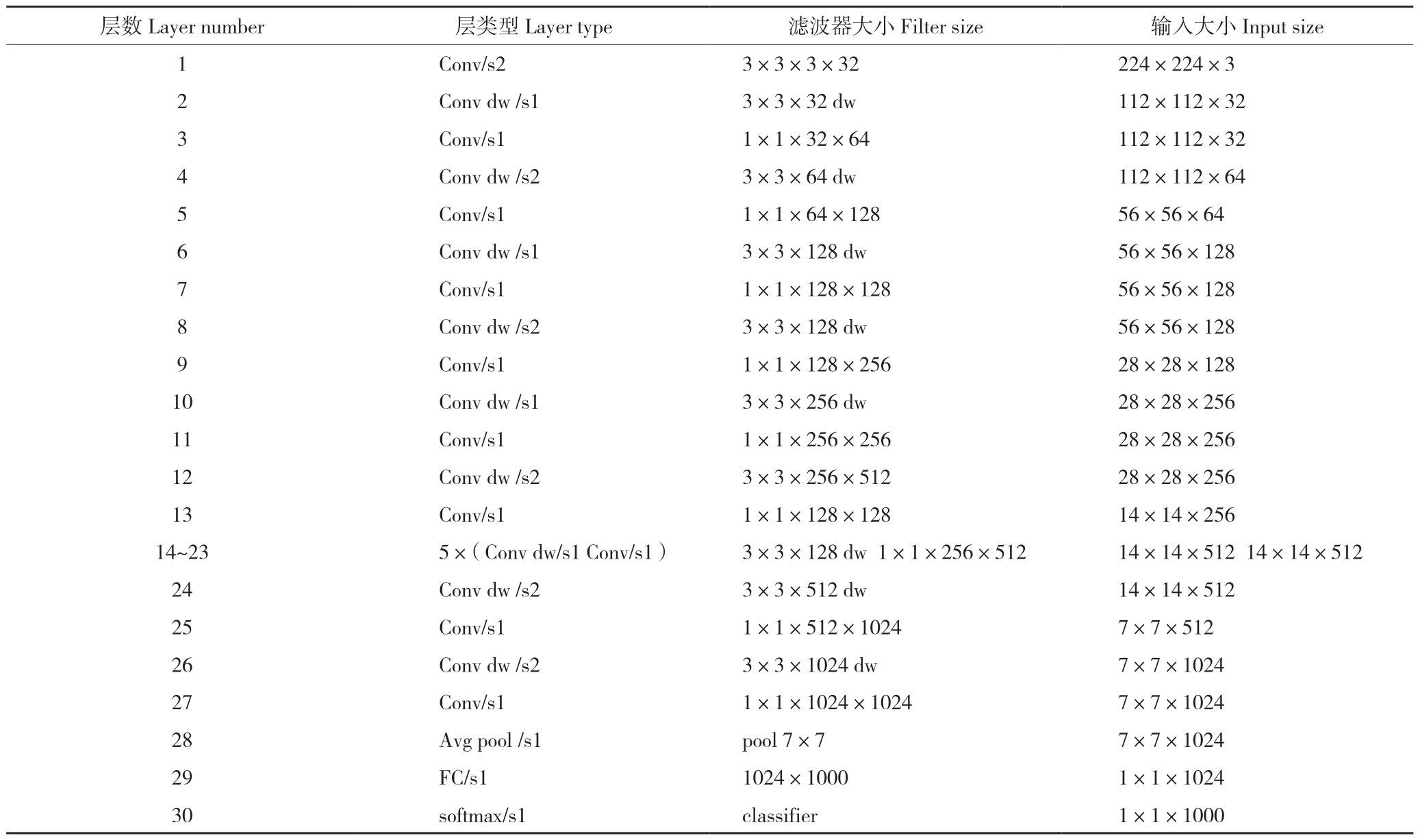
层数Layer number 层类型Layer type 滤波器大小Filter size 输入大小Input size 1 Conv/s2 3×3×3×32 224×224×3 2 Conv dw/s1 3×3×32 dw 112×112×32 3 Conv/s1 1×1×32×64 112×112×32 4 Conv dw/s2 3×3×64 dw 112×112×64 5 Conv/s1 1×1×64×128 56×56×64 6 Conv dw/s1 3×3×128 dw 56×56×128 7 Conv/s1 1×1×128×128 56×56×128 8 Conv dw/s2 3×3×128 dw 56×56×128 9 Conv/s1 1×1×128×256 28×28×128 10 Conv dw/s1 3×3×256 dw 28×28×256 11 Conv/s1 1×1×256×256 28×28×256 12 Conv dw/s2 3×3×256×512 28×28×256 13 Conv/s1 1×1×128×128 14×14×256 14~23 5×(Conv dw/s1 Conv/s1) 3×3×128 dw 1×1×256×512 14×14×512 14×14×512 24 Conv dw/s2 3×3×512 dw 14×14×512 25 Conv/s1 1×1×512×1024 7×7×512 26 Conv dw/s2 3×3×1024 dw 7×7×1024 27 Conv/s1 1×1×1024×1024 7×7×1024 28 Avg pool/s1 pool 7×7 7×7×1024 29 FC/s1 1024×1000 1×1×1024 30 softmax/s1 classifier 1×1×1000
本研究神经网络训练采用Ubuntu-MS-7A54服务器、GPU NVIDIA GeForce GTX 1080Ti,2块,操作系统为Ubuntu16.04,调用OpenCV3.4.2库进行图像处理(校正、增强和分割等),MIC计算采用Java1.8.0完成,神经网络训练用图像识别,使用Python3.5.4语言调用Tensorflow1.6.0(GPU版)完成。
2.1 试验数据集构建
国家兽医微生物耐药性风险评估实验室提供历年MIC试验有效96孔板图像约6 000张,图片由统一专业设备拍摄,背景为黑色底板。本研究随机抽取其中200张96孔板试验图像,裁剪得到微孔图像19 200张。去掉其中由于光线模糊、切割位置不准确等原因造成的不可用的微孔图像。对可用微孔图像通过旋转、翻转增加噪声等操作获取更多图像,方便神经网络训练。本试验所得数据情况如表2所示,其中训练集微孔图像100 000张,验证集微孔图像29 976张。
2.2 图像预处理
试验图像预处理是指将原始拍摄的药敏试验结果96孔板图像转换成神经网络模型可使用格式,得到试验数据集。图像预处理过程及各步骤处理结果见图3。首先对原始图像进行几何校正、大小调整,灰度化,得到96孔板的750×1 000正射灰度图;第2步进行高斯滤波处理,消除和抑制噪声;第3步进行Morphology形态学变换处理(开运算:先腐蚀再膨胀),进一步消除噪声;第4步进行Canny边缘检测处理,得到图像上轮廓的点集合图像;第5 步对点集集合进行直线拟合,拟合出96孔板4条最外围边界直线,从直线交点得到每个微孔的外切正方形4个顶点坐标,利用这些坐标在原图像上标注96孔板位置;第6步根据上一步得到的坐标将96孔板从原图中裁剪下来,通过透视变换得到标准矩形形式的96孔板图像;第7步根据孔位置相对不变性调整参数,在标准96孔板图像上定位96个微孔;第8步裁剪得到96个正方形微孔图像,对每个微孔图像去除4个角的冗余信息得到试验图像;最后一步对数据设置标签,本试验将数据集分为有菌、无菌两类标签。微孔颜色偏红色的表示有菌生长,标签标注为“有菌”,微孔颜色偏紫色表示无菌生长,标签标注为“无菌”。对数据的标注依据人工判读的药敏试验MIC值进行。
表2 试验数据分布情况
Table 2 Distribution of experimental data

数据集Data set有菌微孔数量Number of micropores with bacterial growth无菌微孔数量Number of micropores without bacterial growth样本总数Total number of samples训练集Training set 50000 50000 100000验证集Validation set 14976 15000 29976总样本Total number of samples 64976 65000 129976

图3 图像预处理过程
Fig.3 Preprocessing procedure of image
经过数据预处理后得到微孔图像19 200张,去掉不可用的微孔图像(原因可能是拍摄角度、变换失真等)后得到有效微孔图像15 573张,再经过图像旋转、翻转和添加噪声数据等变换将图像增加至186 876张,得到标注为“有菌”的图像64 976张,标注为“无菌”图像121 900张,为了平衡样本数量从紫色微孔图像中随机筛选65 000张,最后得到的样本总数为129 976张,将其中的100 000张图片作为训练集,29 976张图片作为验证集(表2)。
2.3 卷积神经网络训练
本研究使用Inception_V4和MobileNet_V1两个卷积神经网络进行图像识别。两个卷积神经网络模型训练过程相同,首先设置模型的超参数进行训练,依据损失值和准确率进行模型评估,若收敛到稳定损失值和准确率则停止试验,否则重复训练与模型评估过程直到符合停止条件。
模型超参数设置:初始学习率为0.01,权重衰减为0.00004,训练和验证时batch_size为32,采用均方根反向传播(RMSProp)学习策略提高收敛速度。
对两个卷积神经网络每训练5万步进行1次模型评估,2个模型都训练了50万步。模型的损失曲线如图4所示。由图4可见,MobileNet_V1模型在训练步数20万步左右达到收敛状态;Inception_V4模型在训练步数30万步左右达到敛状态,收敛速度稍慢。由图5可知,两个模型的验证准确率均随着训练次数增加而上升,在相同的验证集且验证集数量达到29 976张图片的情况下,MobileNet_V1验证准确率达到99.9733%,而Inception_V4则达到了99.9966%,两个模型验证准确率都非常高,但略有差异。
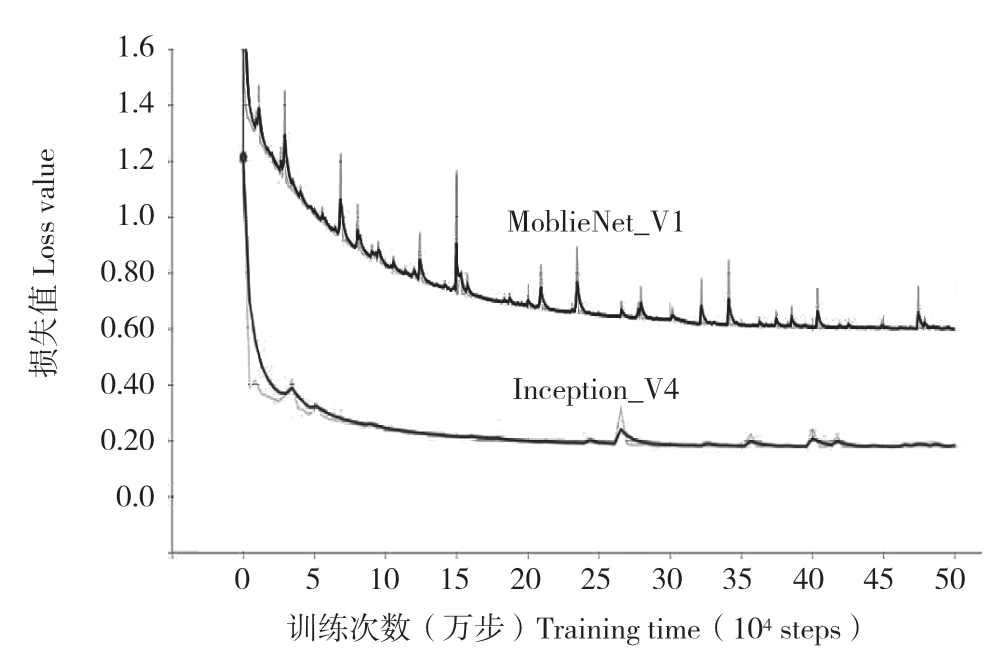
图4 两种模型的损失曲线
Fig.4 Loss curve of two models
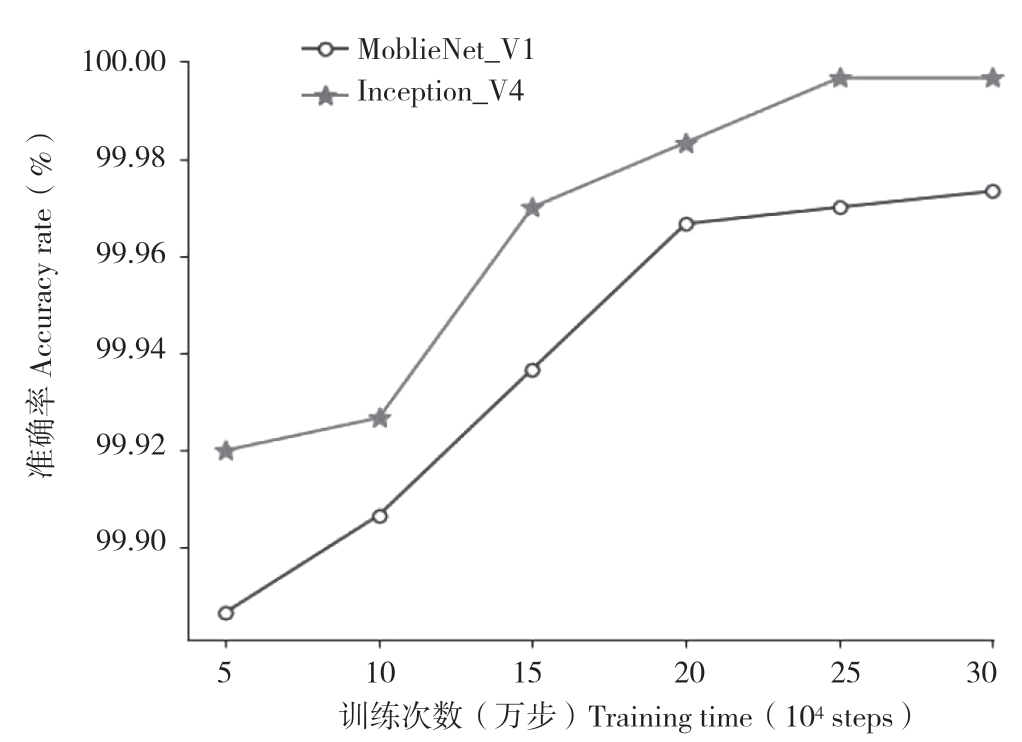
图5 两种模型的验证曲线
Fig.5 Validation curve of two models
2.4 MIC计算及试验异常判定
上述图像识别是针对单个微孔的,若要用于药敏检测结果识别还需要依据微孔坐标及96孔板药物及浓度排列进行判定。首先将每个微孔的分类结果依据坐标转换形成一个01矩阵(“ 0”表示图像识别结果为“无菌”,“1”表示图像识别结果为“有菌”)。对每种药物,因为微孔板上药物深度从高到低排列,因此正确的识别结果应为全0、全1,或连续多个0之后连续多个1(例如0001111)。若出现了从1变为0的序列(例如0011101)则说明发生了识别错误或“跳孔”现象。计算MIC值的流程如图6所示。
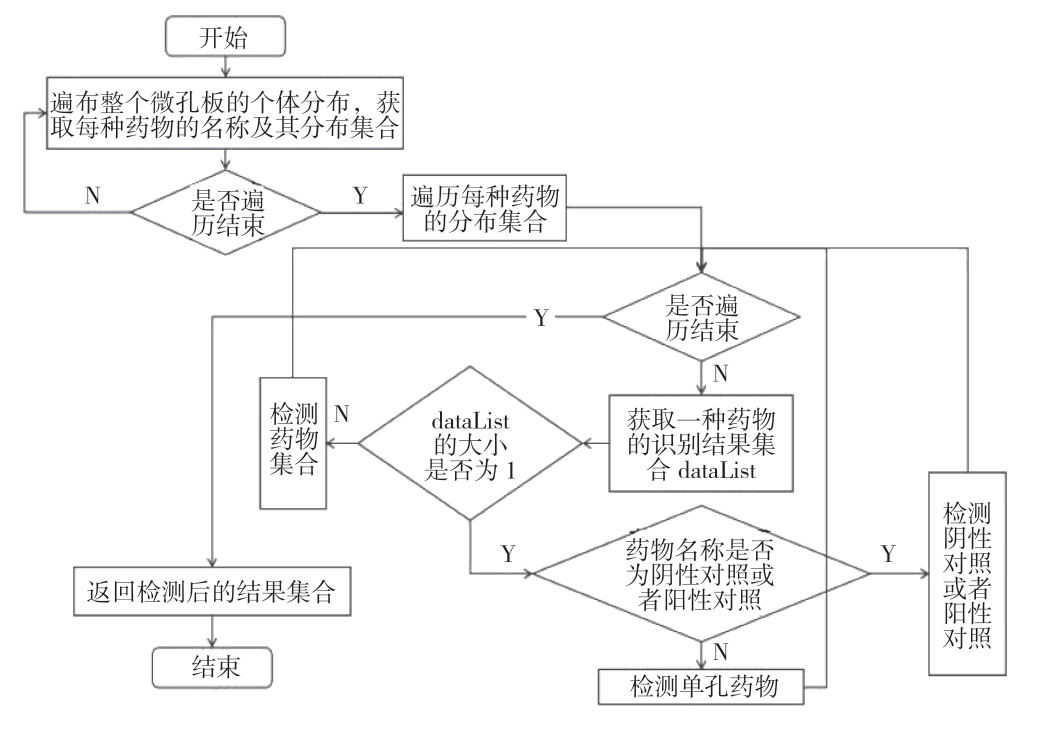
图6 MIC值计算流程
Fig.6 Flowchart of MIC calculation
计算流程中当一种药物的识别结果集合大小不为1时检测药物集合,该子流程具体情况如图7所示。识别结果集合大小为1时则分两种情况:一表明该“药物”是阴性对照或阳性对照,需进行阴性对照和阳性对照的检测;二表明微孔板中只为该药物设计了1个浓度值,故只需检测单孔。若识别结果为1,则该药物的MIC 值为该微孔所表示的浓度;若识别结果为0,则该药物无MIC值。
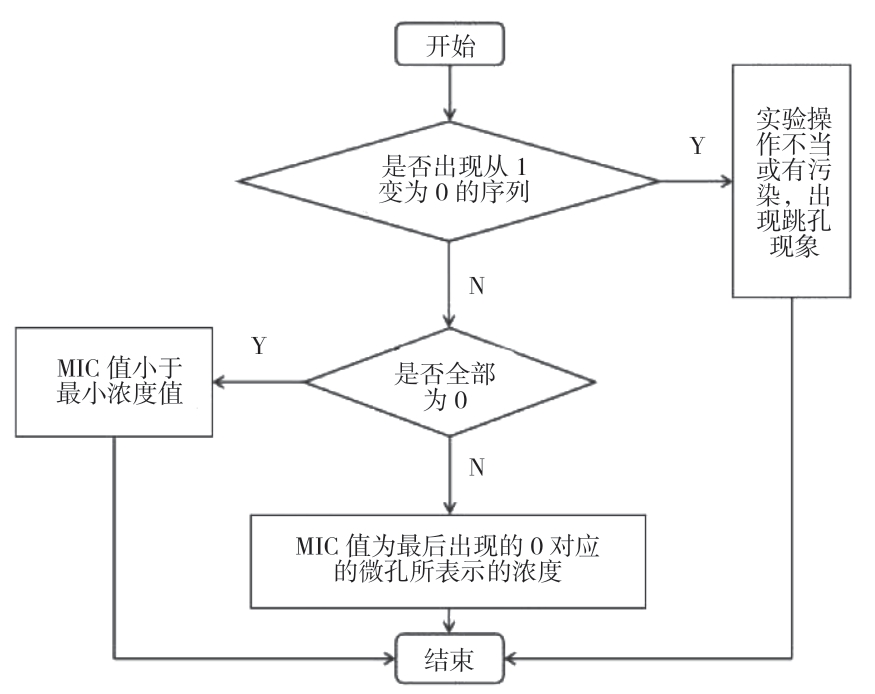
图7 药物集合检测流程
Fig.7 Detection flowchart of medicine set
3 结果与分析
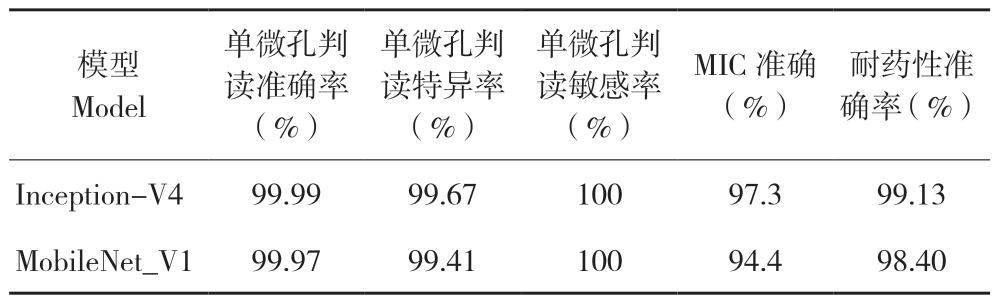
本研究测试组取80张试验结果96孔板图像,经过图像处理形成微孔图像,利用已训练好的卷积神经网络进行判读,得到8×12的01矩阵(0代表无菌,1代表有菌),再按图6、图7计算得到每种药物的MIC值,进而了解耐药情况,试验结果见表3。
表3 两种模型对药敏结果判读效果对比
Table 3 Comparison of the interpretation effects of two models on drug susceptibility results
模型Model单微孔判读准确率(%)单微孔判读特异率(%)单微孔判读敏感率(%)MIC准确(%)耐药性准确率(%)Inception-V4 99.99 99.67 100 97.3 99.13 MobileNet_V1 99.97 99.41 100 94.4 98.40
通过对Inception_V4和MobileNet_V1两种神经网络模型的训练,单微孔图像识别准确率分别达到99.99%、99.97%,特异性和敏感性比率也非常高。MIC准确率略低的原因在于每张96孔板由96单微孔组成,MIC值的计算依赖于不同浓度药物在微孔板上的排列。但两个模型的MIC准确率仍可达到97.30%、94.40%。因为耐药性计算依赖于判定标准的折点值,因此,其准确率高于MIC值准确率,两模型的耐药性准确率达到99.13%、98.40%。同时,本研究对Inception_V4和MobileNet_V1在相同验证集下进行了效率对比,对整张96孔板图像,两个模型的识别时间分别是55 s和234 s,Inception_V4模型的图像识别时间约是MobileNet_V1模型的4.25倍。由试验结果可得到MobileNet_V1网络图像识别准确率比Inception_V4较低,但是模型的识别效率比Inception_V4高,这是由于InceptionV4模型网络层数多,而Mobilenet_V1网络为轻量化网络模型,网络层数较少。
4 讨论
通过试验分析,Inception_V4和MobileNet_V1模型都可用于药敏结果判读。Inception_V4模型判读精度略高于MobileNet_V1模型,但其时间消耗高于MobileNet_V1模型。在精度要求高、时间要求不高的场合,可使用Inception_V4模型进行试验结果判读;而在时间要求高、精度要求不太高的场合,可使用MobileNet_V1模型进行试验结果判读。实时上,通过简单调整,MobileNet_V1模型达到可实用化的程度,即可构建应用系统,让实验人员通过网络上传试验结果图像,利用MobileNet_V1进行试验结果判读并返回结果。
与传统人工判读试验结果的方法相比,本研究提供的方法增加了试验结果拍照、相关资料电子等工作,将试验结果判读环节简化为拍照、上传、利用神经网络进行结果判读、读取试验结果。这一方面可提高实验人员工作效率,另一方面,可降低对实验人员的要求,不再需要经过专业的判读训练,只需按规程拍照上传即可获取试验结果。这些工作看似增加了实验人员的工作量,但此项工作可提高耐药性监测工作的信息化程度,为药敏监测工作积累原始数据,为后期耐药性监测、预测和指导临床用药等工作保存了原始证据。另外,也为利用深度学习、大数据技术与分析挖掘其中更具价值的规律性知识提供了基础。
目前药敏试验图像拍摄环节略有困难,使用手机、相机等设备拍照,易受视角、光照等因素影响,从而导致药敏板上个别微孔试验结果无法读取或判读错误。对此问题,可从两个方面考虑,一是开发专用拍照设备,固定角度与光照条件以提高图像质量;二是对MobileNet_V1模型进行改进,进一步提高单孔判读准确率。另外,针对药敏图像特点改进神经网络模型,进一步提高卷积神经网络模型训练和图像识别效率后续要开展的工作。
5 结论
本研究将卷积神经网络应用于药敏监测试验中MIC值判读和耐药性判定,利用Inception_V4和MobileNet_V1两种模型进行药敏试验单微孔图像识别,并给出MIC值计算、耐药性判定及试验结果异常判别的算法。单微孔图像识别试验结果表明,两种模型都可应用于药敏试验结果判读。药敏试验MIC值准确率分别达到97.30%、94.40%,耐药性判定的准确率更是达到99.13%、98.40%。以MobileNet_V1模型的判读算法为基础,可对此进行实用化推广,达到替代人工判读药敏试验结果的目标。与传统药敏试验结果的人工判读相比,本研究工作提高药敏试验结果判读准确率及效率,可提高药敏试验信息化程度。
[1] 孙坚,刘雅红,冯友军.动物源细菌耐药性研究现状与对策[J].生物工程学报,2018,34(8):1246-1258.doi:10.13345/j.cjb.180162.SUN J, LIU Y H.FENG Y J.Towards understanding antibiotic resistance in animals-borne bacterial pathogens[J].Chinese Journal of Biotechnology,2018,34(8):1246-1258.doi:10.13345/j.cjb.180162.
[2] 张可欣,李忠海,任佳丽.食源性细菌耐药性检测方法的研究进展[J].食 品 与机械,2018, 34(2):181-184.doi:CNKI:SUN:SPJX.0.2018-02-041.ZHANG K X, LI Z H, REN J L.Research progress on detection methods of food-borne bacteria resistant against drugs[J].Food & Machinery,2018, 34(2):181-184.doi:CNKI:SUN:SPJX.0.2018-02-041.
[3] SUN J,LIAO X P,LIU Y H.One Health:an approach to resolving antibiotic resistance[J].Infect Dis Trans Med, 2016, 2(3):122-124.doi:info:doi/10.11979/idtm.201603007.
[4] 胡明,李璐璐,赵敏,骆延波,张印,齐静,张庆,刘玉庆.96点阵琼脂稀释法与微量肉汤稀释法药敏试验结果的对比[J].中国抗生素杂志,2018,43(6):729-733.doi:10.13461/j.cnki.cja.006284.HU M, LI L L, ZHAO M, LUO Y B, ZHANG Y, QI J, ZHANG Q, LIU Y Q.Comparison of the results of 96 dot agar dilution method and broth microdilution method[J].Chinese Journal of Antibiotics, 2018, 43(6):729-733.doi:10.13461/j.cnki.cja.006284.
[5] 郭莉娜,于淑颖,王贺,徐英春.Sensititre YeastOne显色药敏板与微量肉汤稀释法检测阿萨希毛孢子菌体外药物敏感性比较研 究[J].中国真菌学杂志,2017,12(4):198-202.doi:10.3969/j.issn.1673-3827.2017.04.002.GUO L N, YU S Y, WANG H, XU Y C.Comparative evaluation of Sensititre Yeast One colorimetric panel with broth microdilution methods for antifungal susceptibility testing of Trichosporon asahii isolates[J].Chinese Journal of Mycology, 2017,12(4):198-202.doi:10.3969/j.issn.1673-3827.2017.04.002.
[6] 张筝,赵俊杰,李运喜,赵敏,刘焕奇,刘玉庆,吴润.微量肉汤稀释法药敏试验的误差分析[J].中国抗生素杂志,2016,41(11):858-864.ZHANG Z, ZHAO J J, LI Y X, ZHAO M, LIU H Q, LIU Y Q, WU R.The testing errors of broth microdilution methods for the antimicrobial susceptibility testing[J].Chinese Journal of Antibiotics,2016,41(11):858-864.
[7] 刘超英,汪霞,高洪,严玉霖,富国文,赵汝.FQs耐药基因aac(6’)-Ib-cr和qnrD及qepA三重PCR检测方法的建立及应用[J].中国兽医科学,2018,48(4):512-517.LIU C Y, WANG X, GAO H, YAN Y L, FU G W, ZHAO R.Establishment and application of detection methods for FQs-resistance genes aac(6′)-Ib-cr,qnrD and qepA in bacteria by multi-PCR[J].Chinese Veterinary Science,2018,48(4):512-517.
[8] BOGAERTS P, CUZON G, EVRAPRD S, HOEBEKE M.Evaluation of a DNA microarray for rapid detection of the most prevalent extendedspectrum β-lactamases, plasmid-mediated cephalosporinases and carbapenemases in Enterobcteriaceae, Pseudomonas and Acinetobacter[J].International Journal of Antimicrobial Agents, 2016, 48(2):189-193.
[9] 卢宏涛,张秦川.深度卷积神经网络在计算机视觉中的应用研究综述[J].数据采集与处理,2016,31(1):1-17.doi:CNKI:SUN:NC JI.0.2017-14-193.LU H T, ZHANG Q C.Applications of Deep Convolutional Neural Network in Computer Vision[J].Journal of Data Acquisition and Processing,2016,31(1):1-17.doi:CNKI:SUN:NCJI.0.2017-14-193.
[10] LU C, KRISHNA R, BERNSTEUN M.Visual Relationship Detection with Language Priors//European Conference on Computer Vision[C].Springer International Publishing, 2016.doi:10.1007/978-3-319-46448-0_51.
[11] 孙志军,薛磊,许阳明,王正.深度学习研究综述[J].计算机应用研究,2012,29(08):2806-2810.doi:CNKI:SUN:JSYJ.0.2012-08-003.SUN Z J, XUE L, XU Y M, WANG Z.Overview of deep learning[J].Application Research of Computers,2012,29(8):2806-2810.doi:CNKI:SUN:JSYJ.0.2012-08-003
[12] KRIZHEVSKY A,SUTSKEVER I,HINTON G.E.Imagenet classfication with deep convolutional neural networks//Advances in neural information processing systems[C].Cambridge:MIT Press.2012:1097-1105.doi:10.1145/3065386.
[13] ZHEN D, W YUWEI, PEI M T.Vehicle type classification using unsupervised convolutional neural network[J].IEEE Transactions on Intelligent Transportation Systems, 2015, 16(4):1-10.doi:10.1109/TITS.2015.2402438.
[14] HOWARD A G, ZHU M, CHEN B, KALENICHENKO D.MobileNets:Efficient convolutional neural networks for mobile vision applications[J/OL].(2017-4-17) https://arxiv.org/abs/1704.04861.
[15] ABADI M, BARHAM P, CHEN J, CHEN Z, DAVIS A.TensorFlow:a system for large-scale machine learning[J/OL].(2016-5-27)https://arxiv.org/abs/1605.08695.
[16] JIA Y, SHELHAMER E, DONAHUE J, KARAYEV S, LONG J.Caffe:Convolutional Architecture for Fast Feature Embedding//Proceedings of the 22nd ACM international conference on Multimedia[C].ACM,2014.doi:10.1145/2647868.2654889.
[17] KETKAR N.Introduction to PyTorch//Deep Learning with Python[C].2017.doi:10.1007/978-1-4842-2766-4_12.