【研究意义】苯胺(aniline)作为芳香族化合物的一种,是300多种化学物质的重要中间产物,广泛应用于染料、农药、塑料、橡胶添加剂、炸药等行业[1]。苯胺自身稳定性高、生物毒性大、难降解并具有“三致”作用,其在环境中的残留将会对动、植物造成严重危害,被列入我国“十四类环境优先污染物黑名单”及美国EPA“环境优先控制污染物黑名单”(EPA; www.epa.gov)。因此,针对环境中苯胺的有效处理、减少其对环境的污染是污染防治领域中面临的重要课题。由微生物介导的生物修复在此过程中展现出有效修复污染环境的巨大潜力,但因缺乏有关污染环境对微生物生长和代谢影响的信息,从而限制了微生物在环境修复中的应用[35]。而通过对苯胺胁迫下苯胺降解微生物响应机制的研究,将会丰富这一信息,并为进一步调控提高微生物降解苯胺能力提供良好的靶点、为其生物修复研究奠定基础。【前人研究进展】目前苯胺的处理方法很多,因生物法处理苯胺具有成本低、经济效益高、环境友好性等优点,近年来越来越多的苯胺降解菌被分离出来,如Klebsiella sp.ZL-1[2]、Pseudomonas sp.Z1[3]、Rhodococcus sp.Strain ATCC 49988[4]等,同时苯胺生物降解机理也得到了详细综述[5-6]。有研究指出,通过微生物胁迫响应机制对环境微生物准确调控将是一个强化微生物降解能力、提高修复效率的有效和通用解决途径[7]。如 Wang等[8]研究了 Rhodococcus ruber TH3在热激条件下的适应机制,并通过过表达显著响应蛋白Hsp16成功提高了菌株在热激、50%丙烯酰胺胁迫下的存活率。但在环境微生物苯胺胁迫响应机制方面仅在Rubrivivax benzoatilyticus JA2[9]菌株中被阐述,而作为生物修复最佳菌株之一的Rhodococcus sp.苯胺响应机制还不清楚。【本研究切入点】JA2菌株虽然对苯胺具有一定的耐受性,但其本身不具有苯胺的降解功能,也就是说目前对苯胺降解菌在适应苯胺胁迫方面的研究还未见有报道。Rhodococcus sp.AN-P1作为一株苯胺高效降解菌,可以降解和耐受30 ~ 40 mmol/L的苯胺,在2 000 mg/L苯胺条件下,可在32 h内就能使出水达到《污水综合排放标准》一级标准[10],且基于AN-P1的生物强化序批式反应器系统稳定运行后对苯胺及化学需氧量的去除率可分别超过96.3%和81.3%[11]。研究其在苯胺胁迫下的响应机制,不仅可以解释菌株AN-P1优良的苯胺降解和耐受能力的原因,还可以丰富微生物对环境污染物响应机制。【拟解决的关键问题】本研究采用双向电泳及质谱技术,分析AN-P1在苯胺胁迫下的蛋白质表达响应情况,揭示AN-P1苯胺降解途径,阐述苯胺降解菌在苯胺胁迫下的适应、降解机制,弥补苯胺降解菌对耐受、降解苯胺这一机制的空缺,并为提高环境微生物处理苯胺的能力奠定理论研究基础。
1 材料与方法
1.1 菌株及菌株培养
菌株AN-P1为实验室保藏菌株,根据16S rDNA基因序列分析显示该菌株属于Rhodococcus sp.(GenBank 登录号:FJ827255)[10]。将 AN-P1菌株活化后,分别接种于以1 500 mg/L苯胺和10 g/L柠檬酸钠为唯一碳源的无机盐培养基中,在30℃、200 r/min的恒温振荡器中培养。无机盐培养基组分:Na2HPO4 2 g/L、KH2PO4 0.5 g/L、MgSO4·7H2O 0.3 g/L、微量元素溶液5 mL/L,pH7.0,121℃灭菌 20 min。
1.2 蛋白质样品的制备
按上述培养方法将菌悬液培养至OD600达到0.8~1.0之间。10 000 g,4 ℃条件下离心15 min弃上清液,用pH 7.2的磷酸盐缓冲液洗涤菌体3次,再用双蒸水洗涤菌体2次后,收集菌体。收集的菌体采用超声破碎法提取蛋白质:取一定量菌体,用1 mL裂解液(8 mol/L尿素,4% CHAPS,2 % IPG缓冲液,40 mmol/L DTT,1 mmol/L PMSF,1 µL(500 U)核酸酶充分悬浮菌体;再用杯式超生细胞粉碎机(Scientz08-I,宁波新芝)进行超声处理直至菌悬液清澈,最后20 000 r/min、4 ℃离心20 min,取上清,-20 ℃保存备用。
1.3 双向电泳及图谱分析
用Bradford比色法[12]测定蛋白浓度,取2 mg蛋白质样品并将其固定在pH 4~7 IPG非线性梯度条(Bio-Rad, USA)上。第一维等电点聚焦(30 V,6 h;500 V,2 h;10 000 V,3 h;110 000 Vh)、胶平衡、第二维SDS-PAGE垂直电泳程序以及凝胶染色等步骤参照刘冰霜等实验过程[13]。显色后的凝胶用ImageScanner Ⅲ多功能激光扫描成像仪扫描获取图谱,再用凝胶分析软件ImageMaster 2D Platinum 6.0 进行双向电泳的图像分析。
1.4 蛋白质表达差异点鉴定
在图谱上标记差异的蛋白质点,并从胶上切取下来装入EP管中,加双蒸水保存。切取蛋白质点委托北京华大蛋白质研发中心有限公司进行基质辅助激光解析电离串联飞行时间质谱(MALDI-TOF-MS,UltrafleXtrem,Bruke)鉴定,并根据各肽段M/Z数据,利用Mascot(http://www.matrixscience.com)软件,在NCBInr蛋白数据库中搜索理论上能与酶解肽段相匹配的蛋白。
2 结果与分析
2.1 双向电泳图谱检测
蛋白质组学是分析细胞内所有蛋白质的高通量方法。而蛋白质又是细胞生命活动中的重要功能生物分子,因此通过分析生物体内蛋白质水平变化对于检测微生物在生物降解过程中的生理状态非常重要[14]。本研究以柠檬酸钠为唯一碳源培养的AN-P1蛋白组电泳图中共检测到579个蛋白质点(图1A);而以苯胺唯一碳源培养的AN-P1蛋白组电泳图中共检测到681个蛋白质点(图1B)。对这些蛋白质点进一步分析和鉴定将有助于阐述苯胺降解菌对苯胺胁迫的适应机制。
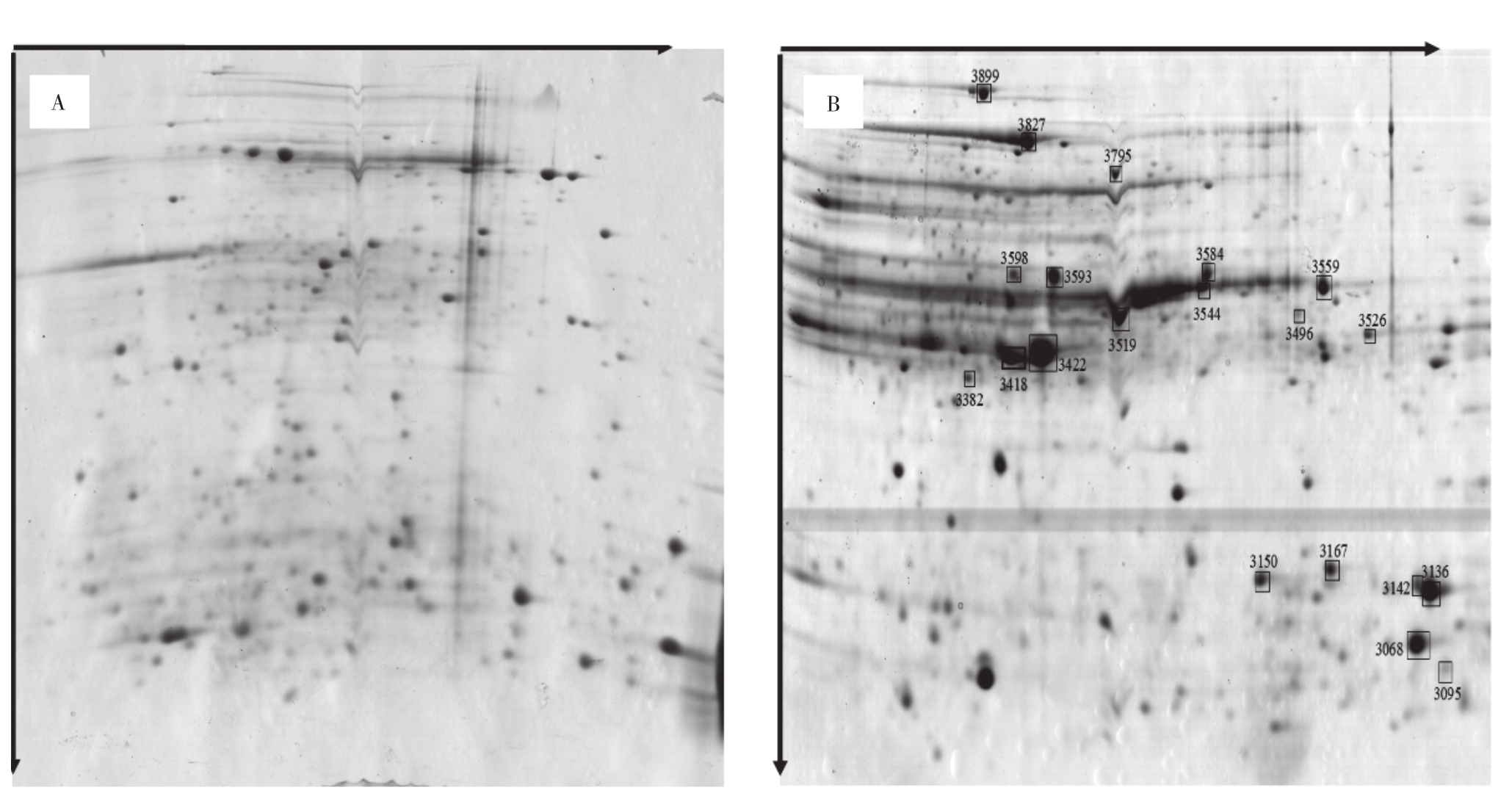
图1 Rhodococcus sp.AN-P1在不同条件下蛋白质组双向电泳图
Fig.1 Two-dimensional electrophoresis diagram of the proteome of Rhodococcus sp.AN-P1 under different conditions
A: 柠檬酸钠;B: 苯胺。B 图中标志方框处为被成功鉴定的蛋白质点
A: Sodium citrate; B: Aniline. The successfully identified protein spots are marked with squares in chart B
2.2 差异表达蛋白的鉴定
如图1B中黑色方框所示,从以苯胺为唯一碳源的电泳图谱中选取21个与柠檬酸钠为唯一碳源电泳图谱显著差异的蛋白质点,利用质谱进行分析,获得这些蛋白质点的肽质谱指纹图谱。根据得到的肽质谱指纹图谱及其分子量范围等数据,通过Mateix science网站提供的MASCOT软件查询与之相匹配的蛋白,如果蛋白质点的Mascot得分大于82(P<0.05)则认为该蛋白点被成功鉴定,并从给出的候选蛋白中选择与AN-P1最接近物种的蛋白质作为鉴定结果,否则视为未得到鉴定。如表1所示,蛋白质点Mascot得分大于82的共有17个,即成功鉴定17个蛋白质点,有4个蛋白质点未鉴定成功(未展示)。其中,成功鉴定的蛋白质点按功能可分为信号传导系统蛋白、氨基酸及能量代谢系统蛋白、细胞防御系统蛋白、苯胺降解酶等。另外,从蛋白质相对分子质量、等电点来看,大部分成功鉴定的蛋白质相对分子质量在31 000 ~ 58 000之间,等电点位于4~7之间,证明本研究中选择的IPG非线性梯度条非常适合该条件下物种蛋白质的二维电泳分离。从蛋白质匹配上的物种来看,17个蛋白质点均来自于非Rhodococcus sp.物种,这可能是由于AN-P1菌株属于Rhodococcus sp.新种或者是Rhodococcus sp.蛋白组信息不全面所导致。
表1 苯胺诱导条件下的AN-P1蛋白斑点的鉴定
Table 1 Identification of protein spots of AN-P1 induced under aniline conditions
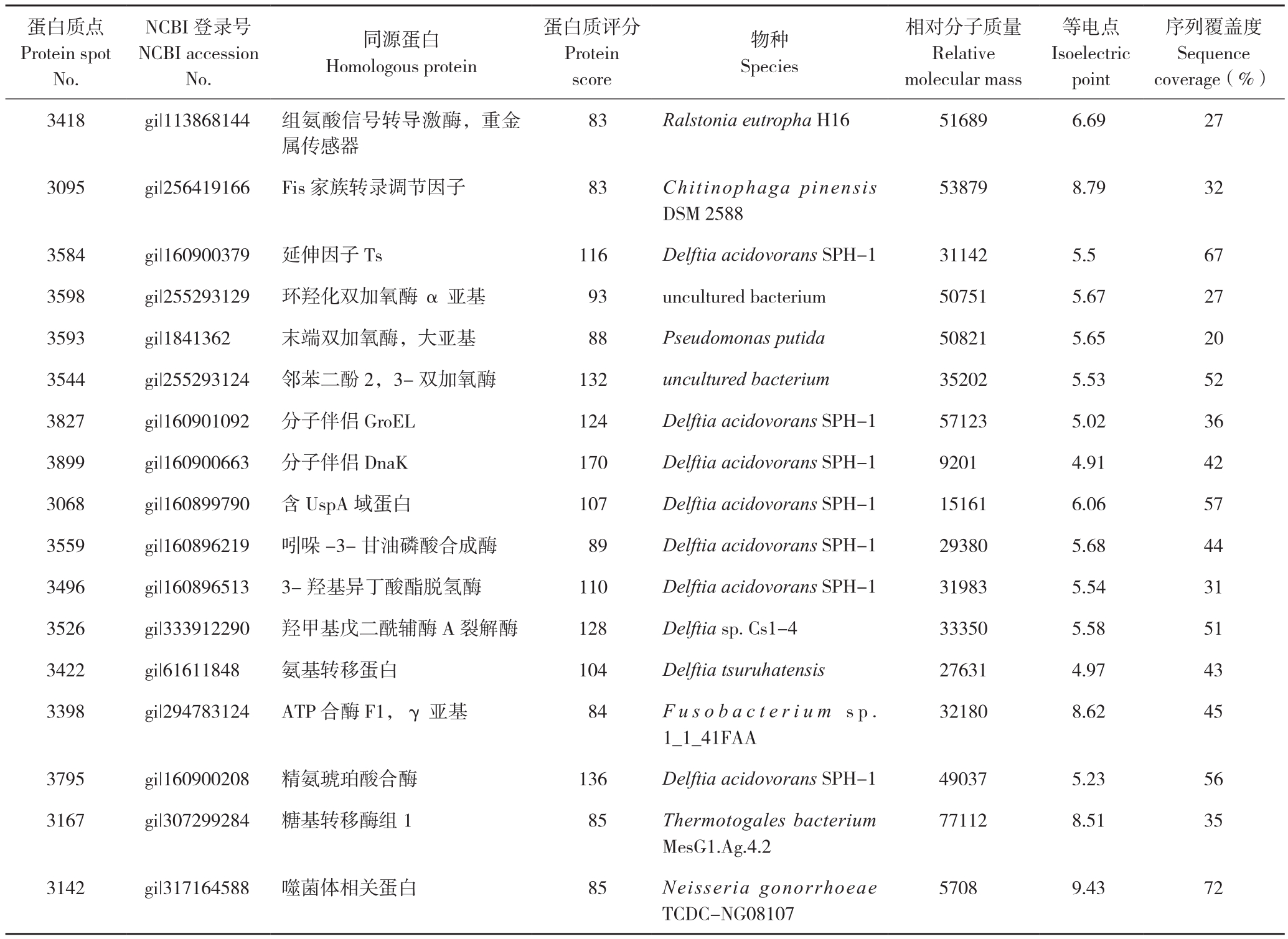
蛋白质点Protein spot No.NCBI登录号NCBI accession No.同源蛋白Homologous protein蛋白质评分Protein score物种Species相对分子质量Relative molecular mass等电点Isoelectric point序列覆盖度Sequence coverage(%)3418 gi|113868144 组氨酸信号转导激酶,重金属传感器83 Ralstonia eutropha H16 51689 6.69 27 3095 gi|256419166 Fis家族转录调节因子 83 Chitinophaga pinensis DSM 2588 53879 8.79 32 3584 gi|160900379 延伸因子Ts 116 Delftia acidovorans SPH-1 31142 5.5 67 3598 gi|255293129 环羟化双加氧酶α亚基 93 uncultured bacterium 50751 5.67 27 3593 gi|1841362 末端双加氧酶,大亚基 88 Pseudomonas putida 50821 5.65 20 3544 gi|255293124 邻苯二酚2,3-双加氧酶 132 uncultured bacterium 35202 5.53 52 3827 gi|160901092 分子伴侣GroEL 124 Delftia acidovorans SPH-1 57123 5.02 36 3899 gi|160900663 分子伴侣 DnaK 170 Delftia acidovorans SPH-1 9201 4.91 42 3068 gi|160899790 含UspA域蛋白 107 Delftia acidovorans SPH-1 15161 6.06 57 3559 gi|160896219 吲哚-3-甘油磷酸合成酶 89 Delftia acidovorans SPH-1 29380 5.68 44 3496 gi|160896513 3-羟基异丁酸酯脱氢酶 110 Delftia acidovorans SPH-1 31983 5.54 31 3526 gi|333912290 羟甲基戊二酰辅酶A裂解酶 128 Delftia sp.Cs1-4 33350 5.58 51 3422 gi|61611848 氨基转移蛋白 104 Delftia tsuruhatensis 27631 4.97 43 3398 gi|294783124 ATP合酶F1,γ亚基 84 F u s o b a c t e r i u m s p.1_1_41FAA 32180 8.62 45 3795 gi|160900208 精氨琥珀酸合酶 136 Delftia acidovorans SPH-1 49037 5.23 56 3167 gi|307299284 糖基转移酶组1 85 Thermotogales bacterium MesG1.Ag.4.2 77112 8.51 35 3142 gi|317164588 噬菌体相关蛋白 85 Neisseria gonorrhoeae TCDC-NG08107 5708 9.43 72
3 讨论
3.1 信号传导调节系统
虽然苯胺可以作为碳源、氮源及能源被微生物所利用,但同时作为有机溶剂也会对AN-P1产生毒害影响。当AN-P1暴露在苯胺环境中时,首先会通过信号传导系统感知外界环境的变化,如组氨酸激酶(#3418)。组氨酸激酶是一个双组分信号传导系统,其在感知胁迫存在及调节菌株适应环境方面发挥重要作用[15]。如Erythrobacter litoralis DSM8509在响应光照胁迫时可通过组氨酸激酶感知光照的改变进而调控其他基因表达[16]。而为了适应新环境,新蛋白质的合成又是必不可少的,这一点也从检测到与基因转录和翻译相关蛋白的表达得到了验证,如Fis族转录调节因子(#3095)、延伸因子Ts(#3584)的表达。
3.2 氨基酸及能量代谢
除上述参与调控转录和翻译相关蛋白外,参与氨基酸合成的酶类在细胞适应苯胺胁迫环境中也发挥着至关重要的作用。如吲哚-3-磷酸甘油合成酶(#3559),一种色氨酸生物合成酶,其参与合成的色氨酸在多种胁迫条件下发挥作用[17-18],可协助参与细胞壁的合成及脂质代谢[19]。同时其他氨基酸代谢相关的蛋白也被检测到,包括3-羟基异丁酸脱氢酶(#3496)(参与缬氨酸代谢)羟甲基戊二酰COA裂解酶(#3526)(参与亮氨酸代谢)、氨基转移蛋白(#3422)等。这些变化可维持胞内氨基酸平衡,满足胁迫条件下蛋白质合成增加的需求。另外,微生物在胁迫条件下为了抵抗胁迫环境往往对能量的需求也会增加[20],研究表明AN-P1在苯胺胁迫条件下ATP合酶F1(#3398)上调表达,有助于生产更多的能量用于抵抗苯胺胁迫环境。
3.3 细胞防御系统
通过进一步分析发现,AN-P1还可以调节GroEL(#3827)、DnaK(#3899)、UspA域蛋白(#3068)等细胞防御相关蛋白的表达,以适应苯胺胁迫。研究表明,热激蛋白(Heat shock protein,Hsp)GroEL和DnaK可在多种胁迫条件下响应表达,如有机溶剂/有毒化学物质胁迫[21]、高温胁迫[22]以及其他氧化应激胁迫[23]。热激蛋白除了在防止蛋白质变性、降解和聚集等方面发挥重要作用外,还可以提高细胞对胁迫的耐受力、维持细胞正常的代谢功能、提高细胞活力、减少环境胁迫造成的伤害[24]。通用应激蛋白(Universal stress protein, Usp)则可以提高长期暴露于胁迫条件下细胞的存活率,保护细胞免受有毒化学物质、渗透胁迫以及紫外线胁迫等的伤害[25]。因此,这些蛋白在AN-P1中的表达可能有助于提高细胞对苯胺胁迫的抵抗能力,从而达到适应苯胺环境的需求。
3.4 苯胺降解酶系统
在不同底物条件下,微生物有着不同的代谢途径,如Rhodococcus jostii RHA1是利用β-酮己二酸代谢途径代谢原儿茶酸、利用羟基喹啉代谢途径代谢4-羟基水杨酸等[26]。在本研究中也检测到一些与苯胺代谢相关的酶类,如环羟基化双加氧酶α亚基(#3598)、末端双加氧酶大亚基(#3593)以及邻苯二酚2,3-双加氧酶(#3544)。环羟基化是最常见的初始降解步骤之一,因此是环羟基化双加氧酶也是芳香环起始降解的重要催化酶之一[27-28]。典型的环羟基化双加氧酶由末端双加氧酶和电子转移酶组成,而末端加氧酶作为催化中心可以接收来自电子转移组分的电子[29]。苯胺降解途径表明,苯胺可在有氧条件下经过苯胺环羟基化双加氧酶被转化为邻苯二酚,然后邻苯二酚再在邻苯二酚1,2-双加氧酶的作用下降解为cis, cis-粘康酸(邻位代谢途径)或在邻苯二酚2,3-双加氧酶作用下降解为2-羟基粘康半醛(间位降解途径)[30]。由于本研究中检测到邻苯二酚2,3-双加氧酶的表达,证明了AN-P1是以间位降解途径来代谢苯胺的。AN-P1的这一苯胺代谢方式与Rhodococcus rhodochus strain CTM[31]、Rhodococcus sp.strain DK17[32]相同,但与 Rhodococcus erythropolis AN-13[33]、Rhodococcus sp.22[34]不同。
4 结论
苯胺作为重要的化工中间体,其在环境中的广泛分布及对水生态的影响引起众多学者的关注。就其生物降解途径至今已得到详细研究,但环境微生物对含苯胺环境的响应机制还鲜有研究。本文通过蛋白质组学技术,分析了苯胺降解菌Rhodococcus sp.AN-P1在苯胺胁迫下的蛋白质表达情况。研究结果表明,苯胺降解菌AN-P1在苯胺胁迫环境中会通过响应信号传导系统感知外界胁迫、调节氨基酸及能量代谢系统抵抗苯胺的毒害影响、利用细胞防御系统进一步提高其存活能力、表达苯胺降解酶系统降解苯胺获得碳源、氮源及能量来源,从而形成AN-P1菌株在苯胺胁迫条件下的适应及生存机制。另外,通过对AN-P1苯胺胁迫下蛋白组学的分析也揭示了AN-P1菌株是通过间位降解途径来降解苯胺的。本研究不仅补充了苯胺降解菌在苯胺胁迫条件适应机制的空白,本所鉴定的蛋白质点可为将来进一步调控提高微生物苯胺降解能力提供良好的靶点,为其生物修复研究奠定基础。
[1] ORGE C A,FARIA J L, PEREIRA M F.Removal of oxalic acid, oxamic acid and aniline by a combined photolysis and ozonation process[J].Environmental Technology, 2015, 36(9-12): 1075-1083.doi:10.108 0/09593330.2014.974682.
[2] 王亚娥,王萍,郑莹,柴志龙,李杰.1株具有铁氧化功能的苯胺降解菌的降解特性[J].环境科学与技术,2019,42(3):140-145.doi:10.19672/j.cnki.1003-6504.2019.03.020.WANG Y E,WANG P, ZHENG Y,CHAI Z L, LI J.Screening and degradation characteristics of a aniline degrading bacteria with iron oxidation function[J].Environmental Science and Technology, 2019,42(3): 140-145.doi: 10.19672/j.cnki.1003-6504.2019.03.020.
[3] 赵晶,刘玉香,呼婷婷,杨娅.复合菌同时去除苯胺与氨氮及其影响因素研究[J].环境科学与技术,2017,40(3): 133-137.ZHAO J, LIU Y X, HU T T, YANG Y.Degrading aniline and ammonianitrogen simultaneously by compound bacteria and studying the influence factors[J].Environmental Science and Technology, 2017,40(3): 133-137.
[4] GUPTA N, SKINNER K A, SUMMERS Z M, EDIRISINGHE J N,FARIA J P, MARSHALL C W, SHARMA A, GOTTEL N R, GILBERT J A, HENRY C S, O’LOUGHLIN E J.Draft genome sequence of Rhodococcus sp.strain ATCC 49988, a quinoline-degrading bacterium[J].Microbiology Resource Announcements, 2019, 8(25): 1-2.
[5] 马溪平,魏娜,徐成斌,王延刚.微生物降解高盐苯胺废水的研究进展[J].环境保护科学,2012,38(1):10-12, 18.doi: 10.16803/j.cnki.issn.1004-6216.2012.01.003.MA X P ,WEI N,XU C B, WAGN Y G.Advances in microbial degradation of aniline wastewater of high salt[J].Environmental Protection Science, 2012, 38(1): 10-12, 18.doi: 10.16803/j.cnki.issn.1004-6216.2012.01.003.
[6] 张逸飞,何卿,焦涛,程城,王贯中.微生物降解苯胺的研究进展[J].污染防治技术,2009, 22(1):35-39.ZHANG Y F, HE Q, JIAO T, CHENG C, WANG G Z.Research progress of microbiological degradation of aniline[J].Pollution Prevention Technology, 2009, 22(1): 35-39.
[7] HAZEN T C,STAHL D A.Using the stress response to monitor process control: pathways to more effective bioremediation[J].Current Opinion in Biotechnology,2006, 17(3): 285-290.doi:10.1016/j.copbio.2006.03.004.
[8] WANG M M, CHEN J, YU H M,SHEN Z Y.Improving stress tolerance and cell integrity of Rhodococcus ruber by overexpressing smallshock-protein Hsp16 of Rhodococcus[J].Journal of Industrial Microbiology & Biotechnology,2018,5(10):929-938.doi:10.1007/s10295-018-2066-9.
[9] MUJAHID M, PRASUNA M L, SASIKALA C, RAMANA C V.Integrated metabolomic and proteomic analysis reveals systemic responses of Rubrivivax benzoatilyticus JA2 to aniline stress[J].Journal of Proteome Research,2015,14(2):711-727.doi:10.1021/pr500725b.
[10] 武洪杰,谭周亮,刘庆华,李旭东.一株苯胺降解菌的筛选及处理突发污染的效果[J].中国环境科学,2009, 29(11): 1181-1185.WU H J, TAN Z L, LIU Q H, LI X D.Isolation of an efficient anilinedegrading bacterial strain and its application for accidents[J].China Environmental Science, 2009, 29(11): 1181-1185.
[11] 彭小明,谢翼飞,王桢,谭周亮,李旭东.生物强化技术应急处理苯胺泄漏事故[J].环境工程学报,2013,7(1): 14-18.PENG X M, XIE Y F, WANG Z, TAN Z L, LI X D.Bioaugmentation technology for emergency treatment of aniline leakage accidents[J].Chinese Journal of Environmental Engineering, 2013, 7(1): 14-18.
[12] BRADFORD M M.A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of proteindye binding[J].Analytical Biochemistry, 1976, 72, 248-254.doi:10.1006/abio.1976.9999.
[13] 刘冰霜,刘庆华,谭周亮,李旭东.苯胺降解菌蛋白质组双向电泳体系的建立与优化[J].西南大学学报(自然科学版),2012,34(8):73-78.doi:10.13718/j.cnki.xdzk.2012.08.014.LIU B S,LIU Q H,T Z L, LI X D.Establishment and optimization of two-dimensional gel electrophoresis for proteome of aniline-degrading bacterium[J].Journal of Southwest University, 2012, 34(8):73-78.doi:10.13718/j.cnki.xdzk.2012.08.014.
[14] CAO B, NAGARAJAN K, LOH K C.Biodegradation of aromatic compounds: current status and opportunities for biomolecular approaches[J].Applied Microbiology and Biotechnology, 2009, 85(2):207-228.doi:10.1007/s00253-009-2192-4.
[15] MASCHER T,HELMANN J D,UNDEN G.Stimulus perception in bacterial signal-transducing histidine kinases[J].Microbiology and Molecular Biology Reviews, 2006,70(4): 910-938.doi:10.1128/MMBR.00020-06.
[16] FIEBIG A, VARESIO L M, ALEJANDRO NAVARRETO X, CROSSON S.Regulation of the Erythrobacter litoralis DSM 8509 general stress response by visible light[J].Molecular Microbiology, 2019,112(2):442-460.doi:10.1111/mmi.14310.
[17] WANG C, CHEN Y, ZHOU H, LI X, TAN Z.Adaptation mechanisms of Rhodococcus sp.CNS16 under different temperature gradients:physiological and transcriptome[J].Chemosphere, 2020, 238,124571.doi:10.1016/j.chemosphere.2019.124571.
[18] HIRASAWA T, YOSHIKAWA K, NAKAKURA Y, NAGAHISA K,FURUSAWA C, KATAKURA Y, SHIOYA S.Identification of target genes conferring ethanol stress tolerance to Saccharomyces cerevisiae based on DNA microarray data analysis[J].Journal of Biotechnology,2007, 131(1): 34-44.doi:10.1016/j.jbiotec.2007.05.010.
[19] CZEKSTER C M, NETO B A, LAPIS A A,DUPONT J, SANTOS D S, BASSO L A.Steady-state kinetics of indole-3-glycerol phosphate synthase from Mycobacterium tuberculosis[J].Archives of Biochemistry and Biophysics,2009,486(1):19-26.doi:10.1016/j.abb.2009.04.001.
[20] JOUSSE C, DALLE C, CANET I, LAGREE M, TRAIKIA M, LYAN B,.DELORT A M.Metabolomic study of the response to cold shock in a strain of Pseudomonas syringae isolated from cloud water[J].Metabolomics,2017,14(1):11.doi:10.1007/s11306-017-1295-7.
[21] SEGURA A, MOLINA L, FILLET S, KRELL T, BERNAL P, MUNOZROJAS J, RAMOS J L.Solvent tolerance in Gram-negative bacteria[J].Current Opinion in Biotechnology, 2012, 23(3): 415-421.doi:10.1016/j.copbio.2011.11.015.
[22] MURATA M, FUJIMOTO H,NISHIMURA K,CHAROENSUK K,NAGAMITSU H, RAINA S, YAMADA M.Molecular strategy for survival at a critical high temperature in Eschierichia coli[J].Plos One, 2011, 6(6):1-9.doi:ARTN e2006310.1371/journal.pone.0020063.
[23] FREDRIKSSON A, BALLESTEROS M, DUKAN S, NYSTROM T.Induction of the heat shock regulon in response to increased mistranslation requires oxidative modification of the malformed proteins[J].Molecular Microbiology,2006,59(1):350-359.doi:10.1111/j.1365-2958.2005.04947.x.
[24] MOLIERE N, TURGAY K.Chaperone-protease systems in regulation and protein quality control in Bacillus subtilis[J].Research in Microbiology, 2009, 160(9): 637-644.doi:10.1016/j.resmic.2009.08.020.
[25] UDAWAT P, JHA R K, SINHA D, MISHRA A, JHA B.Overexpression of a cytosolic abiotic stress responsive universal stress protein(SbUSP)mitigates salt and osmotic stress in transgenic tobacco plants[J].Frontiers in Plant Science, 2016,7, 1-21.doi:ARTN 51810.3389/fpls.2016.00518.
[26] MCLEOD M P, WARREN R L, HSIAO W W L, ARAKI N, MYHRE M,FERNANDES C, ELTIS L D.The complete genome of Rhodococcus sp.RHA1 provides insights into a catabolic powerhouse[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(42): 15582-15587.doi:10.1073/pnas.0607048103.
[27] ULLRICH R, HOFRICHTER M.Enzymatic hydroxylation of aromatic compounds[J].Cellular and Molecular Life Sciences, 2007, 64(3):271-293.doi:10.1007/s00018-007-6362-1.
[28] GIBSON D T, PARALES R E.Aromatic hydrocarbon dioxygenases in environmental biotechnology[J].Current Opinion in Biotechnology,2000, 11(3): 236-243.doi: 10.1016/S0958-1669(00)00090-2.
[29] WOLFE M D, PARALES J V, GIBSON D T, LIPSCOMB J D.Single turnover chemistry and regulation of O2 activation by the oxygenase component of naphthalene 1,2-dioxygenase[J].Journal of Biological Chemistry,2001,276(3):1945-1953.doi: 10.1074/jbc.M007795200.
[30] PHALE P S, BASU A, MAJHI P D, DEVERYSHETTY J,VAMSEE-KRISHNA C, SHRIVASTAVA R.Metabolic diversity in bacterial degradation of aromatic compounds[J].Omics-a Journal of Integrative Biology,2007,11(3):252-279.doi:10.1089/omi.2007.0004.
[31] CANDIDUS S, VAN PEE K H, LINGENS F.The catechol 2,3-dioxygenase gene of Rhodococcus rhodochrous CTM: nucleotide sequence, comparison with isofunctional dioxygenases and evidence for an active-site histidine[J].Microbiology, 1994,140(Pt 2): 321-330.doi:10.1099/13500872-140-2-321.
[32] KIM D, CHAE J C,JANG J Y, ZYLSTRA G J, KIM Y M, KANG B S, KIM E.Functional characterization and molecular modeling of methylcatechol 2,3-dioxygenase from o-xylene-degrading Rhodococcus sp.strain DK17[J].Biochemical And Biophysical Research Communications, 2005, 326(4): 880-886.doi:10.1016/j.bbrc.2004.11.123.
[33] MURAKAMI S, KODAMA N, SHINKE R, AOKI K.Classification of catechol 1,2-dioxygenase family: sequence analysis of a gene for the catechol 1,2-dioxygenase showing high specificity for methylcatechols from Gram+ aniline-assimilating Rhodococcus erythropolis AN-13[J].Gene, 1997, 185(1): 49-54.doi:10.1016/s0378-1119(96)00629-4.
[34] MATSUMURA E, OOI S, MURAKAMI S, TAKENAKA S, AOKI K.Constitutive synthesis, purification, and characterization of catechol 1,2-dioxygenase from the aniline-assimilating bacterium Rhodococcus sp.AN-22[J].Journal of Bioscience and Bioengineering, 2004, 98(2):71-76.doi:10.1016/S1389-1723(04)70245-5.
[35] SINGH O V, NAGARAJ N S.Transcriptomics, proteomics and interactomics: unique approaches to track the insights of bioremediation[J].Briefings in Functional Genomics & Proteomics, 2006, 4(4):355-362.