【研究意义】目前,贵州把发展“三白”(即白菜、白萝卜、莲花白)作为脱贫攻坚的关键举措;而且蔬菜产业是贵州省委省政府确定大力发展的12大特色生态产业之一,因此,甘蓝产业的可持续发展尤为重要。近年来,对贵州甘蓝主产地区的病毒病调查发现,芜菁花叶病毒(Turnip mosaic virus,TuMV)是仅次于黄瓜花叶病毒的最重要的田间蔬菜病毒。受TuMV侵染的甘蓝植株表现出花叶、褪绿、叶片扭曲、斑驳等典型症状,严重影响甘蓝的品质和产量。TuMV是世界上最重要的植物病毒之一,能侵染43科156属318种植物,包括许多重要的大田作物[1]。TuMV可由至少89种蚜虫以非持续的方式传播[1],因此,TuMV的传播蔓延速度快,难以防控。而了解TuMV 在不同地区的发生分布和遗传变异是制定相应TuMV防控措施的科学依据。【前人研究进展】TuMV属于马铃薯Y病毒属,是一个正义单链RNA病毒,具有长700~750 nm的弯曲丝状颗粒,基因组约为10 000个核苷酸(nt)。基因组包含一个大的开放阅读框(ORF),它被翻译成一个大的多蛋白(自动催化水解成10种蛋白质),以及一个小的重叠ORF[2]。TuMV的CP基因是马铃薯Y病毒基因组的一个可变区,已被广泛应用于该属分离株的分类[3-5]。从世界各地收集的不同TuMV分离株的基因组序列进行系统发育关系研究表明,TuMV分离株可分为6个主要的TuMV系统发育类群:Orchis、basal-Brassica(basal-B)、Iranian、basal-Brassica/Raphanus(basal-BR),Asian-Brassica/Raphanus(Asian-BR)和 world- Brassica(world-B)[6-8]。TuMV分离株在生物学上可分为5种寄主侵染类型:侵染十字花科植物,但不侵染其他芸薹属植物;侵染寄主是潜在的,偶尔侵染芸苔属植物,不侵染萝卜属植物;系统地侵染大多数芸苔属植物(系统性花叶症状),但不侵染萝卜属植物;Basal-B(R)寄主侵染多种芸苔属植物,表现为花叶症状,但偶尔侵染萝卜属植物;Asian-BR寄主侵染芸苔属和萝卜属植物,表现为花叶症状[6]。basal-B是变异最大的组群,world-B是变化更大、分布更广的组群,basal-BR和Asian-BR的变异较小。大多数欧洲分离株不感染萝卜,而亚洲分离株同时感染芸苔和萝卜[9]。2002年在中国发现了Asian-BR和world-B组TuMV分离物,随后又发现了basal-BR组TuMV分离物[10-11]。【本研究切入点】TuMV在我国浙江、四川、重庆、山东、河北和北京等地的大白菜、萝卜、甘蓝、辣椒及中药材如太子参等作物上发生危害[12-15],对作物品质和产量造成严重影响。由于TuMV寄主范围广泛,推测可能与TuMV极易发生基因重组变异相关。目前,已有TuMV全基因组、CP基因、Hc-Pro基因、P3基因以及3’-UTR区段等核苷酸序列的分子变异报道[16-21]。研究TuMV的遗传和分子变异,有助于了解病毒生物学的重要特征,如毒力变化、宿主适应性、地理范围,以及它们作为新的流行病出现的可能性,这对于提出TuMV的防控措施至关重要。根据前期调查研究,TuMV是危害贵州甘蓝的主要病毒之一,但是对其病毒分子变异特性等尚未有研究报道。【拟解决的关键问题】本研究对贵州甘蓝主产地区的甘蓝病毒病样品进行ELISA检测,明确TuMV在甘蓝上的发生分布,用RT-PCR对甘蓝TuMV分离物的CP序列进行扩增,并用相关软件对CP序列进行遗传进化分析,以期为甘蓝抗TuMV的育种和防控提供科学基础。
1 材料与方法
1.1 试验材料
供试样品:采自贵州省安顺市西秀区、平坝区,威宁县草海镇、双龙镇和修文县龙场镇、久长镇等地花叶、黄化等疑似病毒病的甘蓝样品。每个区镇采集3块地,每块地按东西南北中5个点选取不同症状感染病毒病的甘蓝叶片。带回实验室后液氮速冻,置于-80℃超低温冰箱中保存备用。
试剂:芜菁花叶病毒(TuMV)多克隆抗体、碱性磷酸酶标记物、阳(阴)性质控物等试剂为美国阿格迪Agdia公司产品。植物总RNA提取试剂盒 TransZol Up Plus RNA Kit(ER501)、TransGen TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix(AT311-02)、EasyTaq®Mix buffer和EasyPure Quick Gel Extraction Kit(EG101)等生物试剂购自北京全式金生物技术有限公司。
1.2 试验方法
1.2.1 芜菁花叶病毒(TuMV)的血清学检测 称取0.5~1 g样品放入样品提取网眼袋,加入2 mL GEB通用样品提取缓冲液进行研磨。在酶标板加入包被抗体100 μL/孔,4 ℃过夜(或37 ℃放置3 h),PBST洗板4次,每次浸泡3 min。加入病叶粗汁液100 μL/孔,并设阳性、阴性和空白对照,37 ℃放置3 h。PBST洗板4次,每次浸泡5 min,然后加入含5%脱脂奶粉的PBST封闭30 min。加单克隆抗体 100 μL/孔,37℃放置 3 h。PBST洗板后加入羊抗鼠酶标抗体100 μL/孔,37 ℃放置3 h,PBST洗板。加对PNP substrate室温显色15~30 min。反应完全后用酶标仪检测各孔在405 nm波长处OD值,每份样品重复3次,以样品OD值比阴性对照OD405值≥2判为阳性。
1.2.2 TuMV CP基因的扩增,克隆和测序 植物总RNA提取试剂盒提取甘蓝叶片总RNA。以总RNA为模板,使用北京全式金生物技术有限公司的将总RNA反转录成cDNA。引物设计用已报道的TuMV CP基因序列(GU167976)为参考。引物序列为TuMV CP-F: GCAGATGAAA C G C T T G A C G C A G G C C;T u M V C P-R:TACAACTTCATAACCCCTGAACGCC。进行PCR扩增,PCR反应体系(25.0 μL):2 μL cDNA,正向引物、反向引物各 1 μL,12 μL 2×EasyTaq®,9 μL ddH2O。扩增程序:94℃预变性 5 min,94 ℃ 30 s,54 ℃ 40 s,72 ℃ 1 min,32个循环,72 ℃延伸5 min,4 ℃保存备用。取4 μL的PCR扩增产物于1%琼脂凝胶电泳检测扩增效果,将目的片段进行胶回收,用pEASY1–Blunt Simple Cloning Kit进行连接,并转化到DH5a,涂布于载体相应抗性的LB培养基平板上。阳性克隆长出后,挑取单菌落进行摇菌,提取质粒,送重庆擎科生物公司测序。
1.2.3 TuMV CP基因序列分析 测序序列用NCBI中 BLAST(https://www.ncbi.nlm.nih.gov/) 进行比对,利用Bioedit5对核苷酸序列数据进行组装,DNAMAN6.0软件对测序所得序列进行核苷酸和氨基酸一致性分析;MEGA7.0软件以邻接法构建系统发育进化树,各分支置信度(bootstrap)进行1 000次重复分析。以山药花叶病毒(JYMV)基因组的同源区作为分析的外群,由于BLAST搜索显示,它是国际序列数据库中与TuMV最为密切相关的序列。
2 结果与分析
2.1 TuMV 感染甘蓝的田间症状
甘蓝叶片感染病毒以后,叶片出现花叶、黄化、畸形或斑驳,甘蓝生长缓慢,比健康植株矮小(图1)。

图1 甘蓝感染TuMV的田间症状
Fig.1 Field symptoms of cabbage infected with TuMV
2.2 TuMV血清学检测
贵州威宁县、修文县和安顺市3个甘蓝主要种植区采集甘蓝病毒病样品152份,检测出TuMV阳性样品66份,检出率为43.42%。结果(表1)表明,TuMV在贵州甘蓝上普遍发生,而且不同地区发生分布存在差异。威宁县草海镇、双龙镇,修文县龙场镇、久长镇和安顺市平坝区甘蓝感染TuMV的检出率分别为46.88%、52.38%、58.33%、50.00%和61.54%,安顺市西秀区的甘蓝病毒病样品上未检出TuMV。
表1 贵州省甘蓝病毒病样品TuMV检测结果
Table 1 Test results of TuMV samples of cabbage from Guizhou Province

采样地点Sampling location样品数Number of samples阳性样品数Number of positive samples检出率Detection rate(%)威宁县草海镇Caohai Town,Weining County 32 15 46.88威宁县双龙镇Shuanglong Town,Weining County 21 11 52.38修文县龙场镇Longchang Town,Xiuwen County 24 14 58.33修文县久长镇Jiuchang Town,Xiuwen County 36 18 50.00安顺市平坝区Pingba District,Anshun City 13 8 61.54安顺市西秀区Xixiu District,Anshun City 26 0 0.00合计Total 152 66 43.42
2.3 TuMV CP基因的扩增
将所有的甘蓝病毒病样品用TuMV特异性引物进行RT-PCR扩增,获得目的片段进行回收、连接和转化,经测序获得864bp的TuMV CP基因。结果(图2)表明,威宁县草海镇、双龙镇,修文县龙场镇、久长镇和安顺市平坝区甘蓝上感染了TuMV,而安顺市西秀区未检出TuMV。
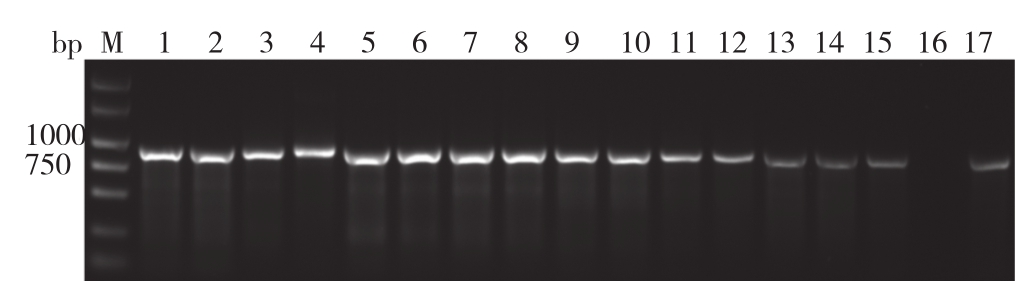
图2 TuMV CP基因RT-PCR扩增
Fig.2 RT-PCR amplification of TuMV CP genes
M:Maker; 1~15:不同地区感染TuMV甘蓝病样;16:阴性对照;17:阳性对照
M: Maker; 1-15: Cabbage samples infected with TuMV in different areas;16: Negative control; 17: Positive control
2.4 TuMV CP基因一致性分析
检测出TuMV的每个地区选择3个分离物进行CP基因核苷酸和氨基酸的比较,结果(表2)表明,15个分离物间的核苷酸同源性为88.70%~100%,与比对分离物的同源性要高,而组内和组间的氨基酸一致性均为100%。重组分析结果可知:15个TuMV分离物CP基因中仅检测到1个潜在事件,即wening-4可能为xiuwen-3和其他序列重组而成。RDP method、GENECONV、Bootscanning、MaxChi、Cimaera、3SEQ和SiScan 7种计算方法中只有MaxChi算出重组,为8.300×10-4,表明贵州侵染甘蓝的15个TuMV分离物CP基因无明显重组事件。
2.5 TuMV CP基因的系统进化分析
系统进化树表明,根据TuMV CP基因核苷酸序列的差异,15个分离物TuMV基因聚在两个不同的分支,Pingba-3与world-B分离物聚为一支,weining-1、2、3、4、5、6,xiuwei-1、2、3、4、5、6,pingba-1、2等14个分离物与basal-BR聚为一支(图3)。贵州甘蓝TuMV分离物没有分布在Orchis、basal-B、Iranian和Asian- BR组。结果表明威宁和修文TuMV分离物只有1个株系,而平坝TuMV分离物有2个株系。综上所述,15个TuMV甘蓝分离物存在2个株系,basal-BR是为害贵州甘蓝的优势株系。
3 讨论
目前,我国萝卜TuMV分离株可分为Asian-BR, basal-BR 和 world-B3个类群,其中半数以上(52.54%)为basal-BR类群[22]。随着对TuMV重组特性和病毒进化的研究,我国不同省份间优势株系存在差异。四川分离株聚在world-B群中[5],山东、吉林两省萝卜的TuMV分离株属于basal-BR[23]。山东、河南、辽宁、黑龙江及陕西等省份具有TuMV大白菜、萝卜分离物大致归属于world-B和basal -BR2个组[12]; 重庆萝卜TuMV分离物属于word-B组和basal-BR组[13]。陕西省32个油菜TuMV分离物与已知的138株来自世界其他地区TuMV分离株或序列进行遗传分类,获得了4个遗传簇:MB(主要是芸苔属分离株)、MR(主要是萝卜属分离株)、IBR(主要介于芸苔属和萝卜属簇之间)和OBR(主要是芸苔属和萝卜属簇之外)[1]。综上所述,在我国对感染TuMV十字花科作物的研究,多数集中在萝卜、白菜和油菜,而对侵染甘蓝的TuMV的株系研究报道较少。
表2 甘蓝病毒病样品TuMV CP基因核苷酸同源性分析
Table 2 Nucleotide homology analysis of TuMV CP genes in cabbage virus samples
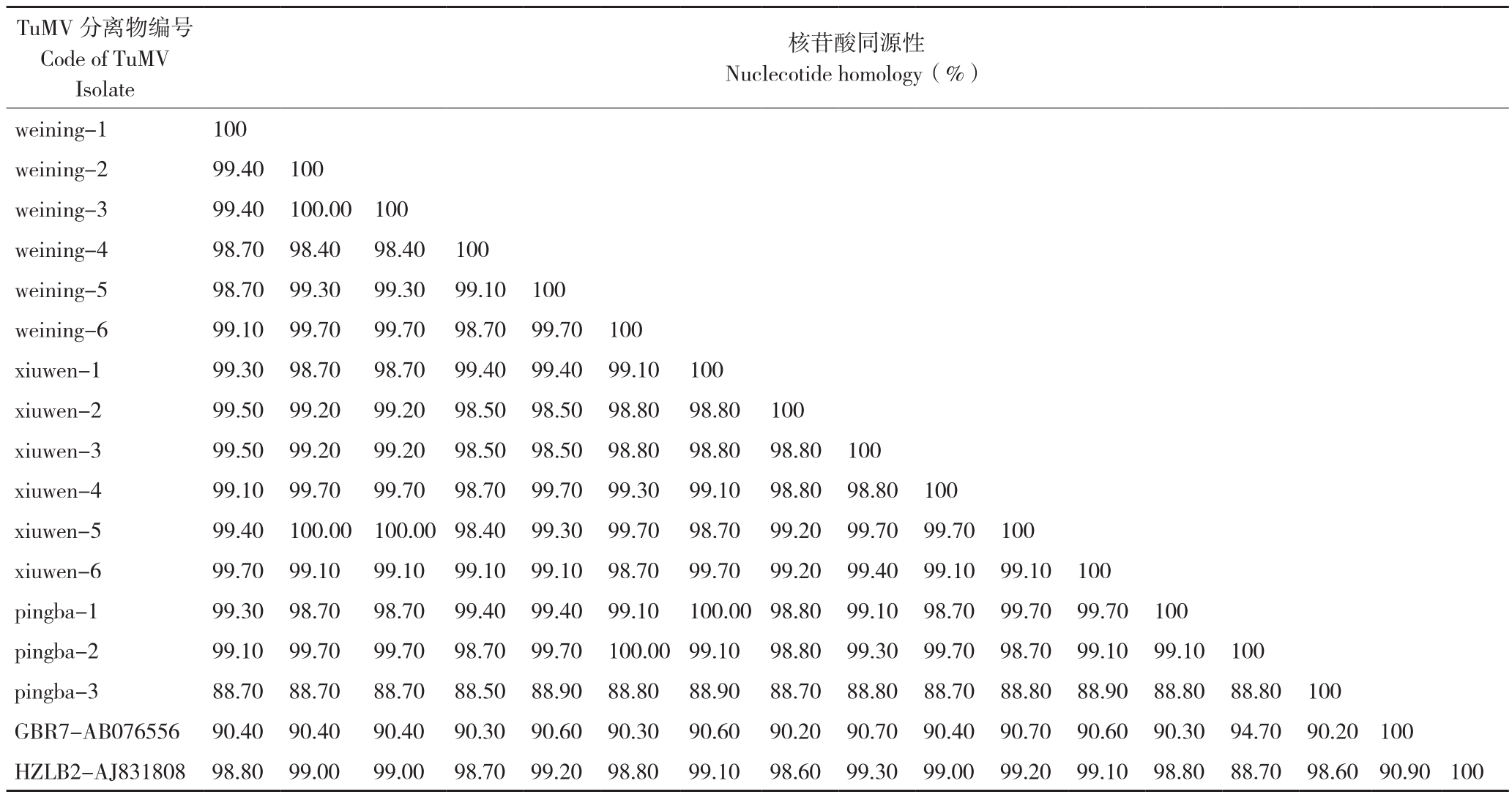
TuMV分离物编号Code of TuMV Isolate核苷酸同源性Nuclecotide homology(%)weining-1 100 weining-2 99.40 100 weining-3 99.40 100.00 100 weining-4 98.70 98.40 98.40 100 weining-5 98.70 99.30 99.30 99.10 100 weining-6 99.10 99.70 99.70 98.70 99.70 100 xiuwen-1 99.30 98.70 98.70 99.40 99.40 99.10 100 xiuwen-2 99.50 99.20 99.20 98.50 98.50 98.80 98.80 100 xiuwen-3 99.50 99.20 99.20 98.50 98.50 98.80 98.80 98.80 100 xiuwen-4 99.10 99.70 99.70 98.70 99.70 99.30 99.10 98.80 98.80 100 xiuwen-5 99.40 100.00 100.00 98.40 99.30 99.70 98.70 99.20 99.70 99.70 100 xiuwen-6 99.70 99.10 99.10 99.10 99.10 98.70 99.70 99.20 99.40 99.10 99.10 100 pingba-1 99.30 98.70 98.70 99.40 99.40 99.10 100.00 98.80 99.10 98.70 99.70 99.70 100 pingba-2 99.10 99.70 99.70 98.70 99.70 100.00 99.10 98.80 99.30 99.70 98.70 99.10 99.10 100 pingba-3 88.70 88.70 88.70 88.50 88.90 88.80 88.90 88.70 88.80 88.70 88.80 88.90 88.80 88.80 100 GBR7-AB076556 90.40 90.40 90.40 90.30 90.60 90.30 90.60 90.20 90.70 90.40 90.70 90.60 90.30 94.70 90.20 100 HZLB2-AJ831808 98.80 99.00 99.00 98.70 99.20 98.80 99.10 98.60 99.30 99.00 99.20 99.10 98.80 88.70 98.60 90.90 100
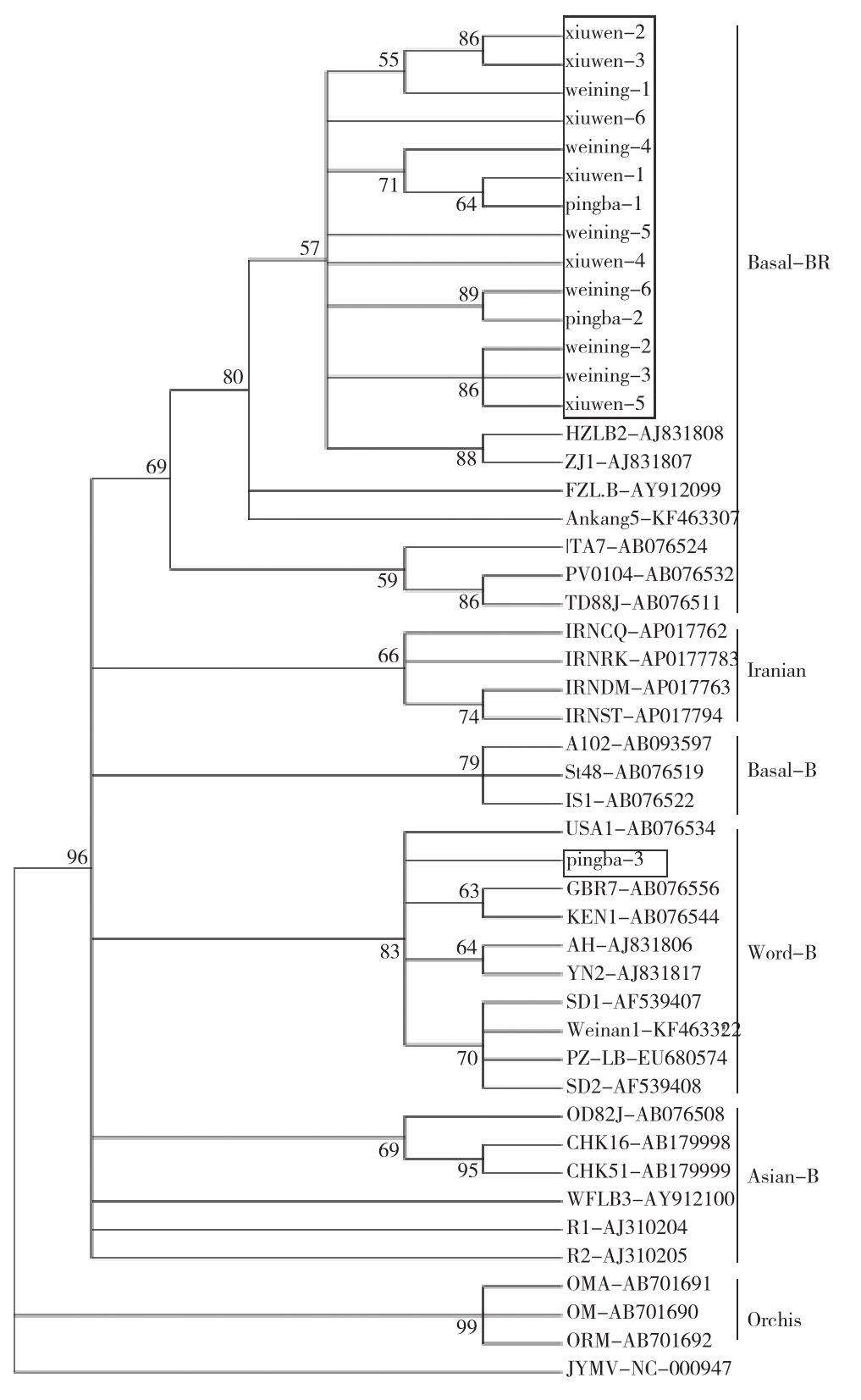
图3 TuMV CP基因分离物的系统进化树
Fig.3 Phylogenetic tree of TuMV CP gene isolates
本研究采用ELISA和RT-PCR方法对贵州甘蓝病毒病样品进行TuMV鉴定,结果表明两种检测方法的阳性检出结果一致,说明这两种方法均能保证TuMV检测的准确性。在贵州威宁、安顺和修文等地种植的甘蓝上均能检测出TuMV,表明该病毒在贵州的发生分布较广。
TuMV CP基因是该病毒分组的重要依据[24],而侵染贵州甘蓝的TuMV的种群结构特点未见报道。本研究通过TuMV CP基因进行扩增,测序比对,然后构建系统进化树发现贵州15个TuMV甘蓝分离物分布在basal-BR和 World-B组中,而北京感染TuMV甘蓝分离物用P3基因构建系统进化树发现,TuMV分离物属于 World-B组[25];表明贵州与北京甘蓝TuMV分离物的归属是一致的。许多文献报道重组在植物RNA和DNA病毒变异和适应性进化中起着重要作用。在马铃薯Y病毒中,74%的TuMV基因组是重组体[10]。贵州15个TuMV甘蓝分离物CP基因无重组事件,这与前人研究结果一致[12]。这可能与寄主植物、采样时间、地理分布或环境条件等因素密切相关。
贵州甘蓝TuMV是basal-BR为优势毒源,山东和吉林萝卜TuMV是basal-BR型,而world-B组为重庆市萝卜的优势毒源[23],这可能由于寄主植物和地理位置不同而产生的差异。江苏省徐州市感病的油菜、萝卜及菠菜上分离到 3个芜菁花叶病毒分离物位于 Basal-BR组[20]。表明basal-BR和 World-B组在我国的十字花科作物上普遍发生。对TuMV 序列进行x系统进化分析,能对其演变及致病性有深入的了解,可进行抗病育种鉴定。如在澳大利亚东部,一种新的芜菁花叶病毒(TuMV)突变株克服了TuMV抗性基因,抑制了该病毒在甘蓝型油菜作物中的传播[26]。因此,对感染TuMV的甘蓝分离物进行序列分析研究,有望进一步对我国甘蓝品种选育进行筛选鉴定,为TuMV的防控提供科学依据。
4 结论
在贵州甘蓝种植区进行调查发现,TuMV普遍发生危害,且引起甘蓝花叶、黄化等典型症状,用酶联免疫吸附法和分子生物学方法对贵州3个甘蓝种植区6个乡镇的152份甘蓝病毒病样品进行TuMV检测,结果表明有5个乡镇检测出该病毒,阳性检出率为43.42%。用TuMV CP基因进行序列分析表明,贵州TuMV甘蓝分离物有两个组群,分别为basal-BR和World-B组,除平坝有一个TuMV分离物聚在World-B组,多数TuMV分离物聚在basal-BR组。
[1] ZHAO L, FENG C H, HAO X G, WANG R R, HU L Y, WANG Q C,WU Y F.Detection and molecular variability of Turnip mosaic virus(TuMV)in Shaanxi, China[J].Journal of Phytopathology, 2013,162: 519-522.doi: 10.1111/jph.12207.
[2] GIBBS A J, OHSHIMA K.Potyviruses in the digital age[J].Annual review of Phytopathology, 2010, 48:205-223.doi.org/10.1146/annurev-phyto-073009-114404
[3] SHIRIN F, REZA P.Characterization of Turnip mosaic virus from the Asian-BR Population in Iran[J].Journal of Phytopathology, 2014,162: 824-828.doi: 10.1111/jph.12243.
[4] JIANG Y, WANG J H, YANG H, XU M Y, YUAN S, SUN W, XU W L, XI D H, LIN H H.Identification and sequence analysis of Turnip mosaic virus infection on cruciferous crops in southwest China[J].Journal of Plant Pathology, 2010, 92: 241-244.doi: 10.4454/jpp.v92i1.37.
[5] RAGHUVEER S, AMRITA B, SUSHEEL K S, ANUP C.Occurrence,Molecular Characterization, Physiological Study and Yield Loss Assessment of Broad-Leaved Mustard(Brassica juncea varrugosa)Infected with Turnip mosaic virus in Arunachal Pradesh[J].Environment and Ecology, 2018, 36(2): 489-494.https://www.researchgate.net/publication/325987813.
[6] NGUYEN H D, TOMITAKA Y, HO S Y W.Turnip mosaic potyvirus probably first spread to Eurasian brassica crops from wild orchids about 1000 years ago[J].PLoS ONE, 2013, 8: 1-13.doi:10.1371/journal.pone.0055336.
[7] YASAKA R, FUKAGAWA H, IKEMATSU M, HIROKO S, SAVAS K,ALIREZA G, NIKOLAOS K, SIMON Y W H , GIBBS A J, KAZUSATO O.The timescale of emergence and spread of turnip mosaic potyvirus[J].Nature Scientific Reports, 2017, 7: 4240.doi: 10.1038/s41598-017-01934-7
[8] SHEVCHENKO O, YASAKA R, TYMCHYSHYN O, SHEVCHEBKO T,OHSHIMA K.First evidence of the occurrence of Turnip mosaic virus in Ukraine and molecular characterization of its isolate[J].Journal of Phytopathology, 2018, 166(6): 429-437.doi: 10.1111/jph.12703.
[9] ZHU F X, SUN Y, WANG Y, PAN H Y, WANG F T, ZHANG X H,ZHANG Y H,LIU J L.Molecular characterization of the complete genome of three Basal-BR isolates of Turnip mosaic virus infecting Raphanus sativus in China[J].International Journal of Molecular Sciences.2016, 17: 888.doi:10.3390/ijms17060888.
[10] OHSHIMA K, YAMAGUCHI Y , HIROTA R, HAMAMOTO T,TOMIMURA K, TAN Z Y , SANO T, AZUHATA F, JOHN A W, JOHN F, CHEN J S, ABED G, GIBBS A J.Molecular evolution of Turnip mosaic virus: evidence of host adaptation, genetic recombination and geographical spread[J].Journal of General Virology, 2002, 83(6):1511-1521.doi: 10.1099/0022-1317-83-6-1511.
[11] OHSHIMA K, TOMITAKA Y, WOOD J T, MINEMATSU Y,KAJIYAMA H, TOMIMURA K, GIBBS A J.Patterns of recombination in Turnip mosaic virus genomic sequences indicate hotspots of recombination[J].Journal of General Virology, 2007, 88: 298–315.doi:10.1099/vir.0.82335-0.
[12] 吴斌,张眉,姜珊珊,张安盛,崔汉青,辛志梅,马立平,王升吉.大白菜、萝卜芜菁花叶病毒系统进化及CP序列分析[J].山东农业科学,2018, 50(8): 100-105.doi: 10.14083/j.issn.1001-4942.2018.08.021.WU B, ZHANG M, JIANG S S, ZHANG A S,CUI H Q,XIN Z M,MA L P,WANG S J.Phylogenetic evolution and CP nucleotide sequence analysis of TuMV in Chinese cabbage and radish[J].Shandong Agricultural Sciences, 2018, 50(8): 100-105.doi: 10.14083/j.issn.1001-4942.2018.08.021.
[13] 熊艳,孙淼,向华丰,王鹤冰,张洪成,青玲.重庆市萝卜芜菁花叶病毒的检测与序列分析[J].植物保护学报,2018, 45(3): 432-438.doi: 10.13802/j.cnki.zwbhxb.2018.2016188.XIONG Y, SUN M, XIANG H F, WANG H B,ZHANG H C,QING L.Detection and sequence analysis of CP gene of Turnip mosaic virus(TuMV)infecting radish plants in Chongqing City[J].Journal of Plant Protection, 2018, 45(3): 432-438.doi: 10.13802/j.cnki.zwbhxb.2018.2016188.
[14] CHEN J, CHEN J P , ADAMS M J.Variation between Turnip mosaic virus isolates in Zhejiang Province, China and evidence for recombination[J].Journal of Phytopathology, 2002,150(3): 142-145.doi:10.1046/j.1439-0434.2002.00731.x.
[15] 李向东,刘金亮,李文法,范在丰,李怀方,王洪刚.潍坊萝卜红心病病原鉴定[J].植物病理学报,2005, 35(1): 13-18.doi:10.13926/j.cnki.apps.2005.01.003.LI X D, LIU J L, LI W F, FAN Z F, LI H F, WANG H G.Identification of virus infecting radish in Weifang City[J].Acta Phytopathologica Sinica, 2005,35(1): 13-18.doi:10.13926/j.cnki.apps.2005.01.003
[16] TIAN Y P , ZHU X P , LIU J L, YU X Q, DU J, KREUZE J, LI X D.Molecular characterization of the 3’- terminal region of Turnip mosaic virus isolates from eastern China[J].Journal of Phytopathology,2007,155(6): 333-341.
[17] 蒋彧,席德慧,王建辉,袁澍,杨辉,许墨耘,林宏辉.芜菁花叶病毒四川分离物外壳蛋白基因的克隆及序列分析[J].四川大学学报(自然科学版),2010,47(3): 639-643.doi:103969/j.issn.0490-6756.2010.03.043.JIANG Y , XI D H, WANG J H, YUAN S, YANG H, XU M Y , LIN H H.Cloning and sequence analysis of coat protein genes of Turnip mosaic virus isolates obtained from Sichuan[J].Journal of Sichuan University(Natural Science Edition), 2010, 47(3): 639-643.doi:103969/j.issn.0490-6756.2010.03.043.
[18] 郭思瑶,童艳,黄娅,罗信福,青玲.重庆辣椒病毒病病原初步鉴定和 分 析[J].园 艺学报,2015,42(2): 263-270.doi:10.16420/j.issn.0513-353x.2014-0774.GUO SY , TONG Y , HUANG Y , LUO XF, QING L.Preliminary identification and analyses of viruses causing pepper virus disease in Chongqing[J].Acta Horticulturae Sinica, 2015,42(2): 263-270.doi:10.16420/j.issn.0513-353x.2014-0774.
[19] 匡云波,陈满足,陆伊荣,陈晶,叶祖云.太子参芜菁花叶病毒和蚕豆萎蔫病毒的双重RT-PCR检测[J].园艺学报,2017, 44(4):784-791.doi:10.16420/j.issn.0513-353x.2016-0940.KUANG Y B,CHEN M Z,LU Y R,CHEN J,YE Z Y.Detection of Turnip mosaic virus and Broad bean wilt virus in Pseudostellaria heterophylla by Duplex RT-PCR[J].Acta Horticulturae Sinica, 2017,44(4):784-791.doi:10.16420/j.issn.0513-353x.2016-0940.
[20] 张成玲,赵永强,杨冬静,孙厚俊,徐振,谢逸萍.不同寄主植物上芜菁花叶病毒外壳蛋白基因克隆及分析[J].江苏农业学报,2014,30(1): 37-41.doi:10.3969/j.issn.1000-4440.2014.01.007.ZHANG C L, ZHAO Y Q, YANG D J, SUN H J,XU Z,XIE Y P.Cloning and sequence analysis of coat protein genes of Turnip mosaic virus isolates from different hosts[J].Jiangsu Journal of Agricultural Sciences, 2014, 30(1): 37-41.doi:10.3969/j.issn.1000-4440.2014.01.007.
[21] 张雅琦,祝富祥,王凤婷,潘洪玉,李向东,刘金亮.芜菁花叶病毒长春分离物 p3 基因的克隆、序列分析及P3蛋白抗血清的制备[J].植物病理学报,2015, 45(6): 585-591.doi:10.13926/j.cnki.apps.2015.06.004.ZHANG Y Q,ZHU F X,WANG F T,PAN H Y,LI X D,LIU J L.Cloning,sequencing of p3 gene of Turnip m osaic virus isolates in Changchun and preparation of P3 protein antiserum[J].Acta phytopathologica sinica, 2015,45(6): 585-591.doi:10.13926/j.cnki.apps.2015.06.004.
[22] LI X D, ZHU T S, YIN X, ZHANG C L, CHEN J, TIAN Y P, LIU J L.The genetic structure of Turnip mosaic virus populationreveals the rapid expansion of a new emergent lineage in China[J].Virology Journal, 2017, 14: 165.doi: 10.1186/s12985-017-0832-3.
[23] ZHU F X, SUN Y, WANG Y, PAN H Y, WANG F T, ZHANG X H,ZHANG Y H, LIU J L.Molecular characterization of the complete genome of three Basal-BR isolates of Turnip mosaic virus infecting raphanus sativus in China[J].International Journal of Molecular Sciences, 2016, 17: 888.doi:10.3390/ijms17060888.
[24] PARMARL N,THAKUR A K, KUMARL P,THAKURL P D,BHARDWAJ S V.Molecular characterization of Turnip mosaic potyvirus(TuMV)infecting radish(Raphanus sativus L.)crop in India[J].Biotech ,2017,7:382.doi: 10.1007/s13205-017-1016-y.
[25] 叶艳英,曾钢,闫晓红,马荣才,吴才君,姚磊.芜菁花叶病毒萝卜与甘蓝分离物P3基因的克隆与序列分析[J].江西农业大学学报,2012,34(2): 264-269.doi:CNKI:SUN:JXND.0.2012-02-013.YE Y Y,ZENG G,YAN X H,MA R C,WU C Q,YAO L.Cloning and sequence analysis of P3 gene of Turnip mosaic virus isolates from Brassica and Raphanus[J].Acta Agriculturae Universitatis Jiangxiensis,2012,34(2): 264-269.doi:CNKI:SUN:JX ND.0.2012-02-013.
[26] GUERRET M G L, NYALUGWE E P, MAINA S, BARBETTI M J,VANLEUR J A G,JONES R A C.Biological and molecular properties of a Turnip mosaic virus(TuMV)strain that breaks TuMV 1 resistances in 2 Brassica napus[J].Plant Disease,2017,101:674-683.doi.org/10.1094/PDIS-08-16-1129-RE.