随着新一代测序技术的逐渐成熟,一些曾经被认为是“转录噪声”的基因越来越被关注,这种RNA具有潜在的调控功能,且不具有编码蛋白质的能力,我们称其为非编码RNAs(non-coding RNAs, ncRNAs),这些非编码RNA都由基因转录而来,并且占RNAs的大部分(98%),大量的研究结果表明ncRNAs在许多生命过程中起到了至关重要的作用[1]。ncRNA根据其作用机制可以分为两类,第一类为管家型ncRNA,即包括转运RNA(tRNA)、核内小RNA(snRNAs)、核仁小RNA(snoRNAs)和核糖体RNA(rRNA)等,管家型ncRNAs与细胞活动以及核糖体功能有关;第二类为调控型ncRNA,即包括短小干扰RNAs(siRNAs)、miRNA(microRNAs)、PIWI相互作用RNAs(piRNAs)和长链非编码RNA(lncRNAs)等[2]。其中lncRNAs因其二级结构发挥着重要的作用,越来越被关注,成为当前广泛研究的热点。
同动物lncRNAs的研究相比,植物lncRNAs的 研 究 起 步 较 晚,1993年 Yang等[3]在 大豆中鉴定出了最早表征的lncRNA基因之一GmENOD40,随后lncRNAs在多种植物中被鉴定和表征。1994年,在苜蓿中鉴定了与根瘤形成有关的 lncRNA-MtENOD40[4];1997年,在番茄中鉴定了与根中磷酸盐摄取有关的lncRNATPSI1[5];1999年,在水稻中鉴定了与根瘤形成有关的 lncRNA-ENOD40[6];2013年,在油菜中鉴定了与雄花不育有关的lncRNA-BcMF11[7]等。相关报道表明植物lncRNAs可以通过多种机制参与植物生长繁殖、开花周期、器官发育、幼苗光形态发生的生理调控[8],也在作物产量以及对生物和非生物胁迫的响应中起着关键作用。目前研究得最多的植物lncRNAs是调节开花的lncRNAs,其通过调节花位点C(FLC)来参与开花启动的调控,水稻中lncRNA-LDMAR被发现可以调节水稻的光周期敏感型雄性不育,lncRNA-CsM10在黄瓜品种的雄性分化中过程中可以发挥作用,lncRNA-Zm401在玉米中主要以调节发育的方式在花药中表达[9]。近年来,在许多植物物种中发现了大量响应生物和非生物胁迫的lncRNAs。通过高通量测序技术发现了大量响应氮、盐、冷和干旱胁迫等的lncRNAs[10],也有许多lncRNAs与植物对真菌、细菌和病毒病原体的防御有关。在拟南芥中,长的基因间非编码RNA LINC-AP2被上调,并且与芜菁皱纹病毒感染的AP2基因表达负相关。这些研究结果表明lncRNAs在植物生长发育中起到关键的作用[11]。
虽然在过去短暂的一段时间内植物lncRNAs被大量研究,但相关的研究报道仍然较少,除了在一些模式作物中的lncRNAs得到了表征和鉴定,在其他农作物中lncRNAs的探究仍具有局限性,许多lncRNAs仍然不知道确切的基因组注释和功能意义。本文主要概述了对植物lncRNAs的认识,包括长链非编码RNA的起源、主要特征、分类和作用机制,总结归纳了用于研究植物lncRNAs的数据库,同时对近几年主要农作物相关的lncRNAs中的研究进展进行了整理论述。
1 lncRNA来源和相关数据库
1.1 lncRNAs的来源
随着测序技术的进步和新技术的出现,lncRNAs第一阶段的发现在1980—2000年,通过传统的基因作图方法发现了单个的lncRNA,如H19和XIST,H19是被最早报道的长非编码RNA之一,XIST是X染色体失活的主调控子;lncRNAs发展的第二阶段即大规模cDNA测序的发展导致发现了数量惊人的lncRNAs转录本,特别是在植物中已经鉴定出数千种lncRNA,但同动物相关研究比较,植物中lncRNAs的鉴定和研究仍然较少;第三阶段随着微阵列、平铺阵列和下一代测序技术的发展,大量lncRNAs被鉴定在植物中充当重要的调节因子,参与植物的生长发育等过程[12]。
1.2 lncRNAs的相关数据库
目前长链非编码RNA已被证明在人类疾病相关领域发挥着重要的作用,有关植物长链非编码RNA的研究也越来越多,许多数据库为研究人员探索长链非编码RNA的功能提供了新的思路和方法,但这些数据库大多数都集中在人类和脊椎动物lncRNAs的研究上。以下为一些包含植物lncRNA转录本或者有助于植物lncRNA相关研究的数据库平台,可供研究人员更加便捷、有效的研究植物相关的 lncRNAs[13]。
PlncRNADB数据库(http://bis.zju.edu.cn/PlncRNADB/index.php)包含来自4个物种的5 000多个lncRNAs。该数据库可以快速区分非编码RNA及蛋白编码转录本,页面简介清晰,用户可以直接通过web界面查询lncRNA与各种RNA结合蛋白之间的关系。
PNRD数据库(http://structuralbiology.cau.edu.cn/PNRD/index.php)是于2010年建立的植物miRNA数据库,该数据库收录了来自166个物种的28 000余个非编码RNA,其中包含6 000多个lncRNAs,分别来自拟南芥、玉米、水稻、毛果杨、烟草、黄瓜等物种。除了基本的注释以外还提供了miRNAs与lncRNAs之间的联系,并且整合了一些新的研究技术及工具包,以加强数据库的能力,为科学用户提供一站式服务。
CANTATAdb数据库(http://yeti.amu.edu.pl/CANTATA/)是最大的植物lncRNAs资源数据库之一,该数据库收集了39种物种的239 631个lncRNAs,可以直接对lncRNA的ID进行检索,可得知lncRNA的序列、BLAST搜索结果、分析的RNA-Seq lncRNA表达谱条形图以及lncRNA预测的多肽信息。也可以对相关文献直接进行搜索。
NONCODE数据库(http://www.noncode.org)是一个系统数据库,致力于提供最完整的非编码RNA(ncRNA)集合和注释,特别是长非编码RNA(LncRNA)。该数据库可以得知lncRNAs的外显子数量,长度,序列和表达谱等信息。数据库中的数据通过GenBank和其他专业数据库收集得到。用户可以在数据库中对lncRNAs进行搜索,也可以直接对既有的ID或其他数据库中的名称进行搜索。
Starbase数据库(http://starbase.sysu.edu.cn/index.php)是研究RNAs之间调控相互作用网络的平台,其根据miRNA靶位点鉴定了10 000对ceRNA,提供了目前为止最全面的miRNA-lncRNA相互作用信息,并且提供大规模数据集的可视化、分析和下载。用户可以极其方便的使用数据库以扩大对ncRNA功能及其协调调控网络的理解。
PLNlncRbase(http://bioinformatics.ahau.edu.cn/PLNlncRbase)数据库人工收集了43个植物物种的1 187个植物lncRNA,包含了lncRNA的详细信息,即序列、分类、表达模式、表达的检测方法、参考文献以及从原始参考文献中提取的lncRNA的潜在靶基因等。该数据库定期更新,以极大地促进未来有关植物lncRNAs生物学意义的研究。
LncTar数据库(http://www.cuilab.cn/lnctar)通过寻找基于碱基配对的两个RNA分子的最小自由能连接结构来研究LncRNA-RNA之间的相互作用,其可以对lncRNA的RNA靶标进行大规模预测。LncTar对RNA大小没有限制,可以处理当前所有长度的RNA分子,并且提供了一个定量标准,可以自动判断两个RNA分子是否相互作用。
2 植物lncRNA的特征与作用机制
2.1 植物lncRNAs的特征
ncRNAs是长度大于200 nt的非编码RNA,其ORF小于100 nt,没有蛋白质编码能力,长非编码RNA大多数存在于细胞的细胞核和亚核室染色质中,其在不同的发育生长功能中具有特异性。lncRNAs在结构上与mRNA相似,具有polyA尾巴及带帽结构,但其转录物比mRNA更短,外显子更少,lncRNAs除了可以由RNA聚合酶Ⅱ、RNA聚合酶Ⅲ转录外,在植物中RNA聚合酶Pol Ⅳ和Ⅴ也可产生lncRNAs[14]。根据与蛋白质编码基因的关系进行分类,可将lncRNAs分为4类:(1)不与蛋白质编码基因重叠的长基因间非编码RNAs(lincRNAs,图1A);(2)内含子lncRNAs,即来自蛋白质编码基因内含子的lncRNAs。从具有相同启动子的蛋白编码基因重叠区域转录的lncRNAs被归类为正义lncRNAs(图1C);(3)反义RNA和天然反义转录本(NATs),由反义基因链转录而来(图1 B、D);(4)eRNAs是从增强子区域产生的外切体敏感增强子RNA,在哺乳动物细胞中占很大比例的非多聚腺苷酸lncRNAs,但在植物中尚未得到大量研究(图1 E)[15]。
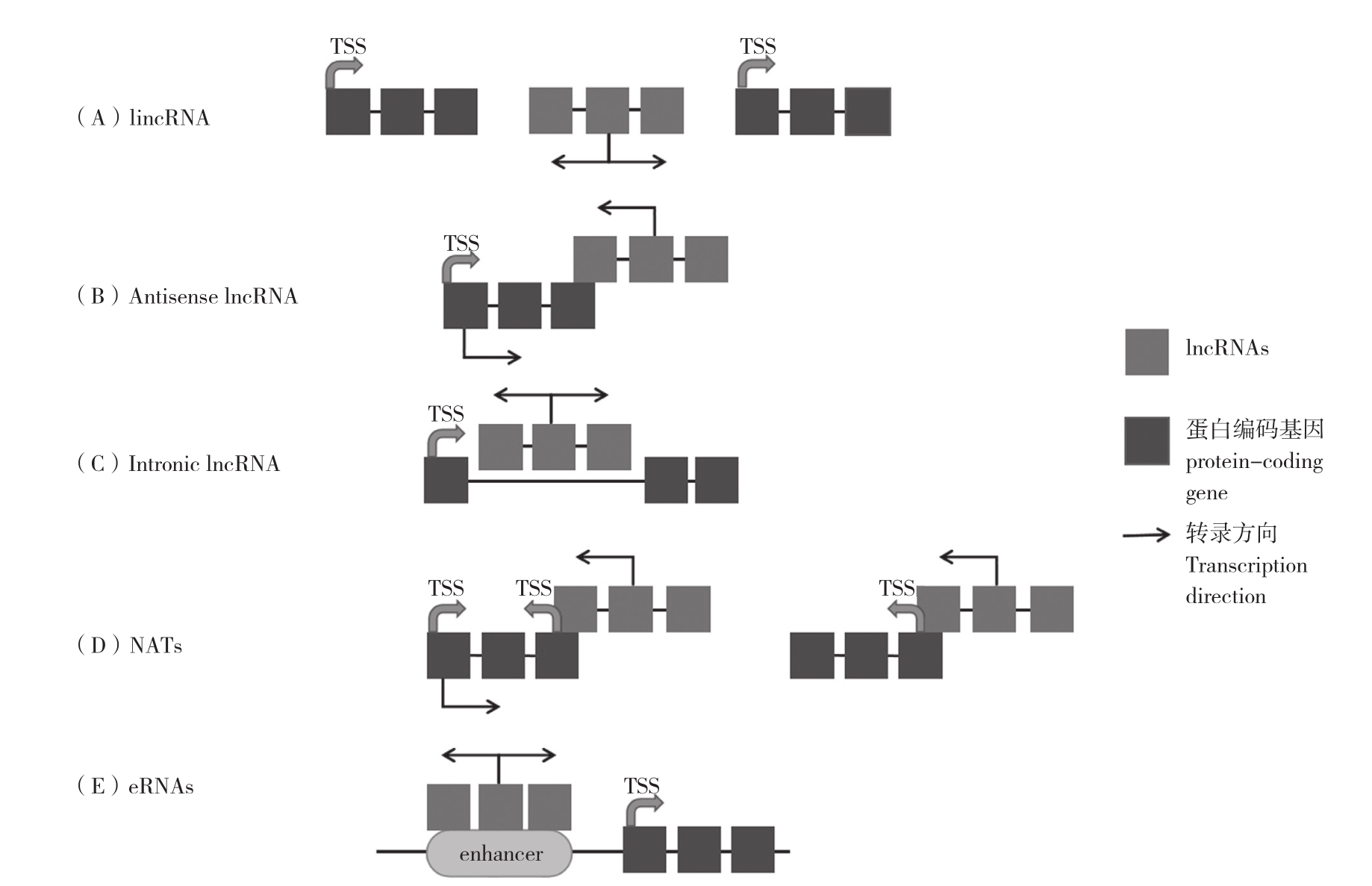
图1 基于lncRNAs与蛋白质编码基因关系的分类
Fig. 1 Classification of lncRNAs based on their relation with protein-coding genes
2.2 LncRNAs的作用机制
2.2.1 在染色质水平上,植物lncRNAs参与调节组蛋白修饰的作用机制 LncRNA主要在细胞核内调节表观遗传修饰,通过调节组蛋白或DNA修饰在转录水平上调节基因转录(图2A)。在拟南芥中发现的lncRNA能够调控开花位点C(FLC)染色质区域的组蛋白甲基化,从而编码一个重要的开花抑制蛋白,实现调节春化的功能。
2.2.2 在DNA水平上,lncRNAs参与调节DNA甲基化的作用机制 植物RdDM是一个表观遗传修饰过程,RNA Pol Ⅳ转录的lncRNAs被DCL3切割成siRNAs。这些siRNA被甲基化,并与Ago蛋白结合,从而形成Ago-siRNA复合物。RNA PolV转录的lncRNAs作为支架分子,通过序列互补招募AGO-siRNA复合物以及DNA甲基转移酶,介导从头甲基化,从而导致基因沉默(图2B)。
2.2.3 lncRNAs参与转录调控过程的作用机制 LncRNA可以直接通过与DNA序列结合来抑制基因转录或直接与蛋白质相互作用来调控下游基因的表达(图2C、D)。拟南芥中的ELENA1是一个与免疫应答相关的lncRNA,进一步的分析表明,ELENA1阻断了PR1免疫应答基因表达的负转录调节因子与介质亚单位19a(MED19a)的结合,促进了PR1启动子中免疫应答基因表达的负转录调节因子的释放,增强了PR1的表达。
2.2.4 lncRNAs参与转录后调控的作用机制 LncRNA通过选择性剪接或海绵机制参与转录后的调控。lncRNA可以直接与剪接因子或蛋白质相互作用,进而调控mRNA选择性剪接,剪接因子也可以直接调控lncRNA选择性剪接(图2E)。在拟南芥中lncRNA选择性剪接竞争对手RNA(ASCO-RNA)通过mRNA与AtNSRs竞争性结合,从而调节生长素信号中的选择性剪接(AS)。lncRNAs也作为miRNA靶标的模拟参与调控,在靶模拟过程中,miRNAs与其真实靶之间的相互作用被诱骗RNAs通过部分互补序列与miRNAs结合所阻断,这个过程也被称为内源性目标模仿(ETM,图2F)。在番茄中,lncRNAs slylnc0195和1077在番茄黄曲叶病毒(TYLCV)侵染后表达上调,lncRNAs-Slylnc0195和1077分别作为miR166和miR-399的目标模拟物参与调控[16]。除了作为miRNAs的模拟靶标外,miRNAs还可以直接或间接调控lncRNAs。lncRNAs可以直接作为miRNAs的靶标,并且可以受到miRNAs的负调控。以lncRNAs为靶点的miRNAs可以产生siRNA或阶段性小干扰RNA(PhasiRNAs),从而调节基因表达(图2G),桑树中新型lncRNA MuLnc1在成熟果实中以较高水平表达,并被mul-miR3954切割,产生次生siRNA,其中siRNA si16157可靶向并沉默钙调蛋白样蛋白基因CML27(MuCML27)的表达,在植物抗逆性中起着重要的作用。除此之外,植物lncRNAs也作为miRNA和其他siRNA的前体参与调控(图2H)。
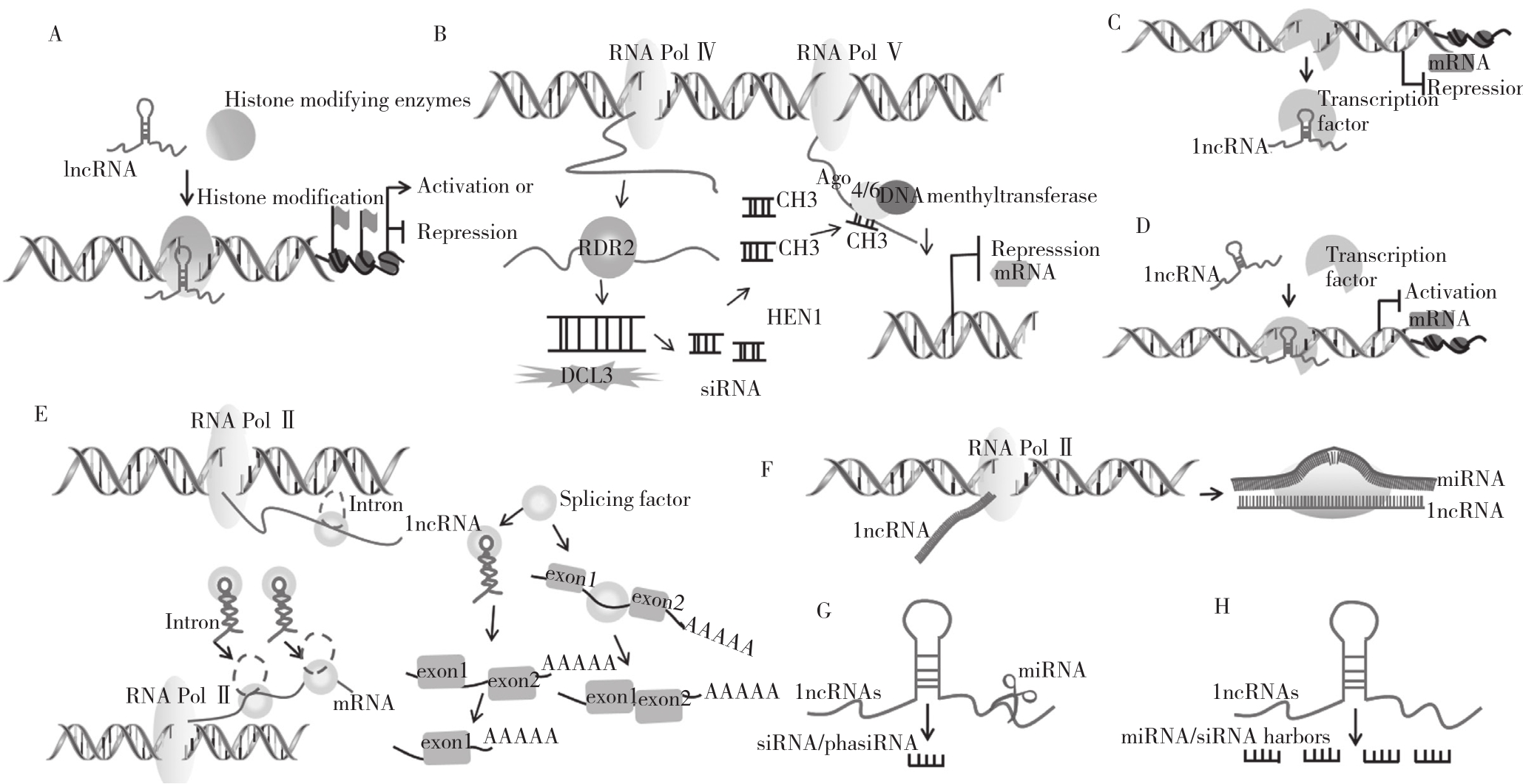
图2 植物lncRNA的分子作用机制(改自文献[17])
Fig. 2 Molecular mechanism of lncRNA in plant(changed from reference[17])
RNA Pol Ⅳ:RNA聚合酶 Ⅳ;RNA Pol Ⅴ:RNA聚合酶 Ⅴ;RDR2:依赖性RNA聚合酶;DCL3:Dicer-like3蛋白;Ago 4/6:Argonaute4/6蛋白;HEN1 Menthyltransferase:RNA甲基转移酶;RNA Pol Ⅱ:RNA聚合酶ⅡRNA Pol Ⅳ : RNA polymerase Ⅳ ; RNA Pol Ⅴ : RNA polymerase Ⅴ ; RDR2: RNA-dependent RNA polymerase2;DCL3: Dicer-like protein 3; Ago 4/6: Argonaute4/6 protein; HEN1 menthyltransferase: RNA enthyltransferase; RNA Pol Ⅱ : RNA polymerase Ⅱ
3 主要农作物相关lncRNA研究进展
3.1 玉米lncRNAs
研究发现lncRNA可以从生物胁迫及非生物胁迫、生长发育等多方面调控玉米的生长。在lncRNAs参与玉米非生物胁迫方面,lv等[18]在玉米中鉴定了1 077个差异表达的lncRNAs,其中包括509个TE-lncRNAs。通过共表达网络分析,发现39个lncRNAs作为响应非生物胁迫的共表达网络中的主要枢纽。Wang等[19]从玉米幼苗中鉴定出611个TE-lncRNAs,通过相关验证和分析表明转座元件相关的lincRNAs在植物非生物胁迫反应中起着重要作用。Pang等[20]对玉米4个发育阶段不同组织的RNA-SEQ数据进行全基因组分析,发现了一组3 488个高可信的lncRNA转录本,通过对它们在干旱胁迫和水分充足条件下表达水平的比较分析,共鉴定出1 535个对干旱有响应的lncRNAs;深入分析了lncRNA在干旱反应中的阶段依赖性和组织特异性表达,揭示了玉米在干旱胁迫响应过程中不同发育阶段不同组织中lncRNAs的表达模式。对适应极端条件的玉米地方品种中的lncRNAs进行鉴定,在盐和过量硼诱导的复合胁迫下,发现在玉米中的一组lncRNAs,在转录后调控水平上也可能在对高浓度盐分和硼的土壤的适应和胁迫反应中发挥重要作用[21]。Du等[22]鉴定了缺磷条件下诱导的lncRNA PILNCR1,结果表明PILNCR1通过与miRNA的相互作用参与玉米耐低磷的生命调控。lncRNAs也参与玉米生物胁迫及其他生长发育过程,lncRNAs在玉米胚乳发育过程中具有时空调节作用,Kim等[23]对玉米胚乳发育过程中长的非编码转录本进行时空分析,从3种胚乳细胞发育不同阶段的编码和非编码转录本中,鉴定了1 540个新的长非编码转录本,发现1 312个具有时空调节的转录本,并通过逆转录、定量PCR(RT-PCR)和原位杂交等方法验证了已鉴定的lncRNAs。玉米在赤霉素(gibberellin, GA)条件下的处理中,lncRNA也参与调控,研究发现34个显著差异表达的lncRNAs分别对GA在正常株高和矮秆植株中有响应[24]。在探究玉米中从枝菌根(arbuscular mycorrhizal, AM)响应的lncRNAs的实验中,有5 941个lncRNAs被鉴定出来,其中63个lncRNAs差异表达,证明了lncRNA在AM真菌与植物根部共生中起到了关键作用[25]。
3.2 水稻lncRNAs
水稻作为主要的粮食作物,其遗传改良具有重要意义。lncRNAs在水稻花粉发育、生殖生长方面发挥重要作用,Kiegle等[26]在种子、胚乳中共鉴定出15个lncRNAs,其中lncRNA LOC9270896可能通过选择性保留内含子和外显子来调节肽的表达,相关研究结果进一步证明lncRNAs在水稻种子的发育过程中发生了选择性剪接。Liu等[27]对高频雌性不育系和野生水稻品系的3个阶段进行全转录组测序,在发育阶段分别鉴定了152 233和197个差异表达的lncRNAs,通过对其靶基因进行分析,发现lncRNAs可以通过和mRNAs竞争与miRNAs的结合来维持胚珠和雌配子体发育相关基因的正常表达。Li等[28]进一步研究了同源四倍体水稻lncRNAs参与育性的作用机制,发现了444个差异表达基因,其中过表达lncRNA57811水稻植株会导致育性低和结实率差,说明lncRNA在多倍体水稻花粉发育过程中起重要作用。LncRNA LAIR是从富含亮氨酸重复序列受体激酶基因簇的反义链上转录而来的,对其进行相关的功能分析,发现其可以增强水稻的生长和产量的积累[29]。lncRNAs在水稻生物胁迫及非生物胁迫方面也有相关的研究,在对水稻4种非生物胁迫(冷、热、干旱和盐胁迫)的研究发现多聚腺苷酸化的lncRNAs参与转录后调控,这些lncRNAs在一些应激反应调控中与蛋白质编码基因共同表达,从而在水稻生长过程中发挥重要的作用[30]。Chen等[31]鉴定了镉(Cd)胁迫和正常水稻植株存在差异表达的lncRNAs,发现lncRNAs参与到水稻镉胁迫的调控中。在茉莉酸途径下lncRNAs参与到植物抗病性的调控中,Yu等[32]对不同时期感病水稻的lncRNAs进行鉴定,有567个响应的lncRNAs被鉴定出来,其中73个lncRNAs与39个茉莉酸途径相关蛋白编码基因相互作用,lncRNA ALEX1对水稻白叶枯病表现出显着的抗性。
3.3 小麦lncRNAs
lncRNA在小麦中的研究主要集中在生物胁迫及非生物胁迫调控中,Zhang等[33]探究了小麦lncRNAs在生物胁迫的调控机制,在小麦患条锈病(Puccinia striiformis West f. sp. Tritici)和小麦患白粉病(Blumeria graminis f. sp. Tritici; Bgt)品系中鉴定出283个差异表达的lincRNAs,通过相关分析表明小麦lincRNA响应BGT和PST胁迫。小麦lncRNAs也响应非生物胁迫,Cagirici等[34]等探究了干旱胁迫下小麦中lncRNAs的表征,选用对耐旱程度不同的3个品系的四倍体小麦进行测序,鉴定了5 000个差异表达的转录本,并对miRNA及其靶标相互作用进行分析,进一步揭示了干旱响应下lncRNA的作用机制。Shumayla等[35]对干旱胁迫、高温胁迫、盐胁迫下的小麦组织进行测序和分析,对得到的7 743个lncRNAs进行分析,预测它们对光合作用、生物和非生物胁迫的响应以及在其他过程中的作用。Díaz等[36]在小麦抗寒研究中,通过对繁殖期硬粒小麦和低温处理小麦的样品进行转录组测序,发现了31个差异表达的lncRNAs,并且其中的两个lncRNAs可以通过miRNAs靶模拟来发挥作用。此外,小麦lncRNAs还参与植物对钙通道阻断反应中细胞周期调控的机制,已经发现23个lncRNAs可能作为转录因子(TF)发挥作用,8个lncRNAs参与细胞周期调控[37]。
3.4 棉花lncRNAs
棉花是主要作物之一,棉纤维具有多种用途,lncRNAs参与对棉纤维生长发育的调控。为了探究长非编码RNA参与皮棉和绒毛纤维生长发育的机制,Hu等[38]鉴定了35 802个lncRNAs,其中在无纤维品系和有纤维品系中优先表达的分别有645和651个lncRNAs,通过相关分析表明lncRNAs在棉花纤维发育中的重要作用。Salih等[39]从新的角度对lncRNAs在调控棉花纤维发育的途径进行研究,对陆地棉Ligon-less-1突变体和野生型中差异表达的lncRNAs进行系统鉴定和分析,共鉴定出18 333个lncRNAs,其中lnc001237、lnc017085是棉纤维发育过程中可能参与多种发育途径的完整分子元件。在棉花胁迫过程中,lncRNAs也充当重要的角色,Deng等[40]从陆地棉(Gossypium hirsutum)中鉴定出1 117个lncRNAs,其中44个为盐胁迫下差异表达的基因间lncRNAs(LincRNAs),通过验证表明其均参与到棉花盐胁迫的响应中。Zhang等[41]在探究棉花盐胁迫的研究中,发现过表达lncRNA973可以增强棉花对盐胁迫的耐性,下调lncRNA973表达则降低棉花的耐盐性,并影响一些与盐胁迫相关的基因的表达。在棉花真菌黄萎病防御反应的研究中,首次鉴定了参与抗病的lncRNAs,对感病海岛棉和抗病海岛棉两个绵种的种间抗病过程的保守性和特异性进行研究,描述了参与植物对黄萎病侵染反应的lncRNAs的表达谱,为阐明棉花病害的反应机制提供了新的线索[42]。lncRNAs还参与棉花的花药发育,lncRNAs在棉花可育系和不育系的花药发育过程中均有差异表达,研究表明差异表达的lncRNAs与棉花四分体期、花粉母细胞期和小孢子期的花药发育有关,其表达异常会出现花药败育、棉花雄性不育等现象[43]。
3.5 主要豆科植物lncRNAs
豆类种子是人类和动物食物的第二大来源,仅次于谷类,主要包括大豆、花生、苜蓿等。lncRNAs参与大豆生物胁迫、非生物胁迫的调控过程,并在大豆种质、产量方面起到关键作用。根据链特异RNA测序技术得到的13个大豆样品的种子转录本中,鉴定了与种子重量相关的差异表达基因和1 251个长基因间非编码RNAs(LincRNAs),基因转录本和lincRNAs共表达网络分析表明,lncRNAs是共表达网络的枢纽,因而可能在大豆籽粒重中发挥重要的作用[44]。Kang等[45]测定了10份大豆材料的裂果力和裂荚率,并将其分为破碎敏感型(SS)和抗裂型(SR)两类进行分析,共鉴定出225个SS大豆和SR大豆的差异表达基因(DEG),并从这些转录本中鉴定了246个大豆荚长基因间ncRNAs(LincRNAs)此外还构建了lincRNAs共表达网络,通过分析表明lncRNAs在大豆荚的发育中起着潜在的重要调控作用。LincRNAs参与了大豆胁迫反应、信号转导和发育等过程,有6 018个lincRNAs在37个大豆样本的超过10亿RNA-seq读取中被发现。通过对lincRNA和蛋白质编码基因的共表达分析,证明大豆lncRNAs参与了多种调控,并提供了大豆基因组中lincRNAs的综合图谱[46]。Chen等[47]在持续盐胁迫和对照条件下,通过全转录组测序和生物信息学分析,在大豆根系中鉴定出3 030个lincRNAs和275个lncNATs,并进行了相关鉴定和功能分析,证明lncRNAs在连续盐胁迫诱导下大豆根中的潜在功能。Lin等[48]对不同品种的大豆进行了lncRNAs的综合鉴定,鉴定了约69 000个lncRNAs基因位点,发现了以前未报告的小肽编码转录物子集,避免了具有小的开放阅读框(ORF)的lncRNAs基因位点被视为蛋白质编码基因的可能。
目前在花生中lncRNAs的相关报道较少,Zhao等[49]在包含来自15个不同组织的124个样本的测序数据中鉴定了50 873个花生lncRNAs,利用4 713个共表达的lncRNAs构建共表达网络,获得了与生长发育相关的花生lncRNAs,并预测了共表达的386个关键 lncRNAs的靶基因,为花生提供了丰富的全基因组lncRNAs资源,为培育高产高抗花生品种提供了有价值的理论依据。
随着生物技术的发展,在苜蓿中的测序研究也逐渐展开。Wang等[50]在豆科模式物种紫花苜蓿中鉴定了10 785个lncRNAs,其中有358个和224个分别对叶片和根的磷缺乏有反应,通过进一步分析和研究,证明lncRNAs参与苜蓿对缺磷反应的调控。Chao等[51]对紫花苜蓿转录组进行了单分子长阅读测序,共获得113 321个苜蓿幼叶、成熟叶和衰老叶片的转录本,鉴定了17 740个长的非编码RNA。
4 展望
虽然目前对lncRNAs在农作物中的功能研究越来越多,有关一些主要农作物的研究仍处于起步阶段,已经被大量鉴定的lncRNAs大部分来源于模式作物,在其他农作物的鉴定仍然非常有限,虽然已有大量研究发现lncRNAs参与农作物生物胁迫、非生物胁迫等过程,但针对其响应的全面调查仍然有限,许多lncRNAs仍然不知道确切的基因组注释和功能意义,lncRNA新的来源仍然不断被发现,其分类的方法仍然在不断更新。同时,lncRNA并不是以单一的方式发挥作用,通常与其他非编码基因和蛋白编码基因相互作用而发挥功能,目前很多研究都表明lncRNA可与miRNA相互作用发挥调控机制,还不清楚lncRNA在植物中和circRNA等其他非编码RNA的相互作用机制。如果想进一步探究lncRNA在农作物中的调控机制,还需要结合多个学科,如生物信息学、遗传学等,同时随着相关技术的进一步发展,我们将对lncRNA功能和机制有更加完整的理解和研究,lncRNAs也将在作物遗传育种、生物资源开发、植物细胞工程等领域发挥更加重要的作用。
[1] GAO R, LIU P, IRWANTO N, LOH R, WONG S M. Upregulation of LINC-AP2 is negatively correlated with AP2 gene expression with Turnip crinkle virus infection in Arabidopsis thaliana[J]. Plant Cell Reports, 2016,35(11):2257-2267. doi :10.1007/s00299-016-2032-9.
[2] RAI M I, ALAM M, LIGHTFOOT D A, GURHA P, AFZAL A J.Classification and experimental identification of plant long noncoding RNAs[J]. Genomics,2019,111(5):997-1005. doi:10.1016/j.ygeno.2018.04.014.
[3] YANG W C, KATINAKIS P, HENDRIKS P, SMOLDERS A,VRIES F, SPEE J, KAMMEN A, BISSELING T, FRANSSEN H.Characterization of GmENOD40, a gene showing novel patterns of cell-specific expression during soybean nodule development.[J]. The Plant Journal, 1993, 3(4):573-85. doi:10.1046/j.1365-313x.1993.03040573.x.
[4] CRESPI M D,JURKEVITCH E,POIRET M,D'AUBENTON-CARAFA Y,PETROVICES G,KONDOROSI E, KONDOROSI A. ENOD40, a gene expressed during nodule organogenesis, codes for a non-translatable RNA involved in plant growth[J]. The EMBO Journal, 1994,13(21):5099-5112. doi:10.1002/j.1460-2075.1994.tb06839.x.
[5] LIU C, MUCHHAL U S, RAGHOTHAMA K G. Differential expression of TPS11, a phosphate starvation-induced sene in tomato[J]. Plant Molecular Biology, 1997, 33(5):867-74. doi:10.1023/a:1005729309569.
[6] KOUCHI H, TAKANE K, SO R B, LADHA J K, REDDY P M. Rice ENOD40: isolation and expression analysis in rice and transgenic soybean root nodules[J]. The Plant Journal, 1999,18(2):121-129.doi:10.1046/j.1365-313x.1999.00432.x.
[7] SONG J H, CAO J S, WANG C G. BcMF11, a novel non-coding RNA gene from Brassica campestris, is required for pollen development and male fertility[J]. Plant Cell Reports, 2013,32(1):21-30.doi:10.1007/s00299-012-1337-6.
[8] WU L, LIU S, QI H, CAI H, XU M. Research progress on plant long non-coding RNA[J]. Plants(Basel), 2020,9(4):408-417.doi:10.3390/plants9040408.
[9] DATTA R, PAUL S. Long non-coding RNAs:Fine-tuning the developmental responses in plants[J]. Journal of Biosciences,2019,44(4):77-87. doi:10.1007/s12038-019-9910-6.
[10] NEJAT N, MANTRI N. Emerging roles of long non-coding RNAs in plant response to biotic and abiotic stresses[J].Critical Reviews in Biotechnology, 2018,38(1):93-105. doi:10.1080/07388551.2017.1312270.
[11] 马博涵,姚丹,焦苏淇,鲁中爽,李泽远,张爱晶,何浩博,张家野.非编码RNA在植物中的研究进展[J].广东农业科学,2019,46(7):8-16.doi: 10.16768/j.issn.1004-874X.2019.07.002.MA B H, YAO D, JIAO S Q, LU Z S, LI Z Y, ZHANG A J, HE H B,ZHANG J Y. Research progress of non-coding RNA in plants[J].Guangdong Agricultural Sciences, 2019,46(7):8-16. doi:10.16768/j.issn.1004-874X.2019.07.002
[12] JARROUX J, MORILLON A, PINSKAYA M. History, discovery, and classification of lncRNAs[J]. Advances in Experimental Medicine and Biology, 2017, 1008:1-46. doi:10.1007/978-981-10-5203-3_1.
[13] CHARLES R J L, EICHHORN P J A. Platforms for investigating lncRNA functions[J]. SLAS Technology, 2018,23(6):493-506. doi:10.1177/2472630318780639.
[14] BUDAK H, KAYA S B, CAGIRICI H B. Long non-coding RNA in plants in the Era of reference sequences[J]. Frontiers in Plant Science, 2020,11:276-285. doi: 10.3389/fpls.2020.00276.
[15] WANG H V, CHEKANOVA J A. Long Noncoding RNAs in Plants[J].Advances in Experimental Medicine and Biology, 2017,1008:133-154. doi: 10.1007/978-981-10-5203-3_5.
[16] WANG J, YANG Y, JIN L, LING X, Liu T, CHEN T, JI Y, YU W,ZHANG B. Re-analysis of long non-coding RNAs and prediction of circRNAs reveal their novel roles in susceptible tomato following TYLCV infection[J]. BMC Plant Biology, 2018,18(1):104-119.doi:10.1186/s12870-018-1332-3.
[17] ZHANG X, WANG W, ZHU W, DONG J, CHENG Y, YIN Z, SHEN F. Mechanisms and functions of long non-coding RNAs at multiple regulatory levels[J]. International Journal of Molecular Sciences,2019,20(22):5573-5601. doi: 10.3390/ijms20225573.
[18] LV Y, HU F, ZHOU Y, WU F, GAUT B S. Maize transposable elements contribute to long non-coding RNAs that are regulatory hubs for abiotic stress response[J]. BMC Genomics, 2019,20(1):864-880.doi:10.1186/s12864-019-6245-5.
[19] WANG D, QU Z, YANG L, ZHANG Q, LIU Z H, DO T, ADELSON D L, WANG Z Y, SEARLE I, ZHU J K. Transposable elements(TEs)contribute to stress-related long intergenic noncoding RNAs in Plants[J]. The Plant Journal, 2017,90(1):133-146. doi: 10.1111/tpj.13481.
[20] PANG J, ZHANG X, MA X H, ZHAO J. Spatio-temporal transcriptional dynamics of maize long non-coding RNAs responsive to drought stress[J]. Genes(Basel), 2019,10(2):138-152. doi:10.3390/genes10020138.
[21] HUANCA-MAMANI W, ARIAS-CARRASCO R, CARDENASNINASIVINCHA S, ROJAS-HERRERA M, SEPULVEDAHERMOSILLA G, CARIS-MALDONADO J C, BASTIAS E,MARACAJA-COUTINHO V. Long non-coding RNAs responsive to salt and boron stress in the Hyper-Arid Lluteño maize from Atacama Desert[J]. Genes(Basel), 2018,9(3):170-191. doi:10.3390/genes9030170.
[22] DU Q G, WANG K, ZOU C, XU C, LI W X. The PILNCR1-miR399 regulatory module is important for low-phosphate tolerance in maize[J]. Plant Physiology, 2018,177(4):1743-1753. doi:10.1104/pp.18.00034.
[23] KIM E D, XIONG Y, PYO Y, KIM D H, KANG B H, SUNG S. Spatiotemporal analysis of coding and long noncoding transcripts during maize endosperm development[J]. Scientific Reports, 2017,7(1):3838-3851. doi:10.1038/s41598-017-03878-4.
[24] WANG Y J, WANG Y L, ZHAO J, HUANG J Y, SHI Y N, DENG D X.Unveiling gibberellin-responsive coding and long noncoding RNAs in maize[J]. Plant Molecular Biology, 2018,98(4-5):427-438.doi:10.1007/s11103-018-0788-8.
[25] HAN G, CHENG C, ZHENG Y, WANG X, XU Y, WANG W, ZHU S,CHENG B. Identification of long non-coding RNAs and the regulatory network responsive to Arbuscular mycorrhizal fungi colonization in maize roots[J]. International Journal of Molecular Sciences, 2019,20(18):4491-4505. doi:10.3390/ijms20184491.
[26] KIEGLE E A, GARDEN A, LACCHINI E, KATER M M. A genomic view of alternative splicing of long non-coding RNAs during rice seed development reveals extensive splicing and lncRNA gene families[J]. Frontiers in Plant Science,2018,9:115-126. doi:10.3389/fpls.2018.00115.
[27] LIU H, WANG R, MAO B, ZHAO B, WANG J. Identification of lncRNAs involved in rice ovule development and female gametophyte abortion by genome-wide screening and functional analysis[J]. BMC Genomics, 2019,20(1):90-105. doi:10.1186/s12864-019-5442-6.
[28] LI X, SHAHID M Q, WEN M, CHEN S, YU H, JIAO Y, LU Z, LI Y, LIU X. Global identification and analysis revealed differentially expressed lncRNAs associated with meiosis and low fertility in autotetraploid rice[J]. BMC Plant Biology, 2020,20(1):82-100. doi:10.1186/s12870-020-2290-0.
[29] WANG Y, LUO X J, SUN F, HU J H, ZHA X J, SU W, YANG J S. Overexpressing lncRNA LAIR increases grain yield and regulates neighbouring gene cluster expression in rice[J]. Nature Communications, 2018,9(1):3516-3524. doi:10.1038/s41467-018-05829-7.
[30] YUAN J P, LI J, YANG Y, TAN C, ZHU Y, HU L, QI Y, LU Z J. Stressresponsive regulation of long non-coding RNA polyadenylation in Oryza sativa[J]. The Plant Journal, 2018,93(5): 814-827. doi:10.1111/tpj.13804.
[31] CHEN L, SHI S, JIANG N, KHANZADA H, WASSAN G M, ZHU C, PENG X, XU J, CHEN Y, YU Q, HE X, FU J, CHEN X, HU L,OUYANG L, SUN X, HE H, BIAN J. Genome-wide analysis of long non-coding RNAs affecting roots development at an early stage in the rice response to cadmium stress[J]. BMC Genomics, 2018,19(1):460-469. doi: 10.1186/s12864-018-4807-6.
[32] YU Y, ZHOU Y F, FENG Y Z, HE H, LIAN J P, YANG Y W, LEI M Q, ZHANG Y C, CHEN Y Q. Transcriptional landscape of pathogenresponsive lncRNAs in rice unveils the role of ALEX1 in jasmonate pathway and disease resistance[J]. Plant Biotechnology Journal,2020,18(3):679-690. doi:10.1111/pbi.13234.
[33] ZHANG H, HU W, HAO J, LV S, WANG C, TONG W, WANG Y,WANG Y, LIU X, JI W. Genome-wide identification and functional prediction of novel and fungi-responsive lincRNAs in Triticum aestivum[J]. BMC Genomics, 2016,17(1):238-248. doi:10.1186/s12864-016-2570-0.
[34] CAGIRICI H B, ALPTEKIN B, BUDAK H. RNA sequencing and coexpressedl ong non-coding RNA in modern and wild wheats[J].Scientific Reports, 2017,7(1): 10670-10685. doi:10.1038/s41598-017-11170-8.
[35] SHUMAYLA, SHARMA S, TANEJA M, TYAGI S, SINGH K,UPADHYAY S K. Survey of high throughput RNA-Seq data reveals potential roles for lncRNAs during development and stress response in bread wheat[J]. Frontiers in Plant Science, 2017,8:1019-1036.doi:10.3389/fpls.2017.01019.
[36] DIAZ M L, SORESI D S, BASUALDO J, CUPPARI S J, CARRERA A.Transcriptomic response of durum wheat to cold stress at reproductive stage[J]. Molecular Biology Reports, 2018,46(2):2427-2445.doi:10.1007/s11033-019-04704-y.
[37] MA K, SHI W, XU M, LIU J, ZHANG F. Genome-wide identification and characterization of long non-coding RNA in wheat roots in response to Ca2+ channel blocker[J]. Frontiers in Plant Science, 2018,9 :244-259. doi:10.3389/fpls.2018.00244.
[38] HU H, WANG M, DING Y, ZHU S, ZHAO G, TU L, ZHANG X.Transcriptomic repertoires depict the initiation of lint and fuzz fibres in cotton(Gossypium hirsutum L.)[J]. Plant Biotechnology Journal,2018,16(5):1002-1012. doi: 10.1111/pbi.1284.
[39] SALIH H, GONG W, HE S, XIA W, ODONGO M R, DU X. Long non-coding RNAs and their potential functions in Ligon-lintless-1 mutant cotton during fiber development[J]. BMC Genomics, 2019,20(1):661-676. doi:10.1186/s12864-019-5978-5.
[40] DENG F, ZHANG X, WANG W, YUAN R, SHEN F. Identification of gossypium hirsutum long non-coding RNAs(lncRNAs)under salt stress[J]. BMC Plant Biology, 2018,18(1):23-36. doi:10.1186/s12870-018-1238-0.
[41] ZHANG X, DONG J, DENG F, WANG W, CHENG Y, SONG L, HU M, SHEN J, XU Q, SHEN F. The long non-coding RNA lncRNA973 is involved in cotton response to salt stress[J]. BMC Plant Biology,2019,19(1):459-474. doi:10.1186/s12870-019-2088-0.
[42] ZHANG L, WANG M, LI N, WANG H, QIU P, PEI L, XU Z, WANG T, GAO E, LIU J, LIU S, HU Q, MIAO Y, LINDEEY K, TU L, ZHU L,ZHANG X. Long noncoding RNAs involve in resistance to Verticillium dahliae, a fungal disease in cotton[J]. Plant Biotechnology Journal,2018,16(6):1172-1185. doi:10.1111/pbi.12861.
[43] LI Y, QIN T, DONG N, WEI C, ZHANG Y, SUN R, DONG T, CHEN Q, ZHOU R, WANG Q. Integrative analysis of the lncRNA and mRNA transcriptome revealed genes and pathways potentially involved in the anther abortion of cotton[J]. Genes(Basel),2019,10(12):947-964.doi: 10.3390/genes10120947.
[44] YU C, QU Z, ZHANG Y, ZHANG X, LAN T, ADELSON D L, WANG D, ZHU Y. Seed weight differences between wild and domesticated soybeans are associated with specific changes in gene expression[J].Plant Cell Reports, 2017,36(9):1417-1426. doi:10.1007/s00299-017-2165-5.
[45] KANG X, CAI J, CHEN Y, YAN Y, YANG S, HE R, WANG D, ZHU Y. Pod-shattering characteristics differences between two groups of soybeans are associated with specific changes in gene expression[J].Functional and Integrative Genomics, 2019,20(2):201-210. doi:10.1007/s10142-019-00702-2.
[46] GOLICZ A A, SINGH M B, BHALA P L. The long intergenic noncoding RNA(LincRNA)landscape of the soybean genome[J]. Plant Physiology, 2018,176(3):2133-2147. doi:10.1104/pp.17.01657.
[47] CHEN R, LI M, ZHANG H, DUAN L, SUN X, JIANG Q, ZHANG H,HU Z. Continuous salt stress-induced long non-coding RNAs and DNA methylation patterns in soybean roots[J]. BMC Genomics, 2019, 20(1):730-141. doi:10.1186/s12864-019-6101-7.
[48] LIN X, LIN W, KU Y S, WONG F L, LI M W, LAM H M, NGAI S M,CHAN T F. Analysis of soybean long non-coding RNAs reveals a subset of small peptide-coding transcripts[J]. Plant Physiology, 2020,182(3):1359-1374. doi:10.1104/pp.19.01324.
[49] ZHAO X, GAN L, YAN C, LI C, SUN Q, WANG J, YUAN C, ZHANG H, SHAN S, LIU J N. Genome-wide identification and characterization of long non-coding RNAs in peanut[J]. Genes(Basel), 2019,10(7):536-548. doi:10.3390/genes10070536.
[50] WANG T, ZHAO M, ZHANG X, LIU M, YANG C, CHEN Y, CHEN R,WEN J, MYSORE K S, ZHANG W H. Novel phosphate deficiencyresponsive long non-coding RNAs in the legume model plant Medicago truncatula[J]. Journal of Experimental Botany, 2017,68(21-22):5937-5948. doi:10.1093/jxb/erx384.
[51] CHAO Y, YUAN J, GUO T, XU L, MU Z, HAN L. Analysis of transcripts and splice isoforms in Medicago sativa L. by singlemolecule long-read sequencing[J]. Plant Molecular Biology, 2019,99(3): 219-235. doi:10.1007/s11103-018-0813-y.