【研究意义】药用植物遗传资源是中药新药研发、优良品种选育的基础,但近年来国内外对中药材的需求量急剧增长,导致中药材面临种质退化、资源耗竭、品质不稳定等问题。因此,利用现代生物学技术解析药用活性成分的合成途径和调控机制来推动合成生物学的发展,是药用植物资源的保护与利用的重要课题。【前人研究进展】龙脷叶(Sauropus spatulifolius Beille),又名龙利叶、龙肝叶、龙舌叶、龙味叶和牛耳叶等,为大戟科守宫木属常绿小灌木,广泛分布于我国福建、广东、广西等地。龙脷叶具有重要的药用价值和观赏价值,其性甘、淡、平,具有清热解毒、祛痰止咳的功效,常用于治疗肺热咳嗽以及上呼吸道炎症引起的咽痛、急性支气管炎等症[1]。龙脷叶在我国南方也常作为食疗药材,其干燥的叶也是凉茶的主要原料之一。现代药理学研究表明龙脷叶具有抗炎镇痛、抗过敏、抗氧化、止咳祛痰等活性[2-4]。化学成分研究表明龙脷叶中富含氨基酸、多糖、多酚类、有机酸类、甾醇、生物碱类、萜类、苯丙素类和黄酮类等物质[5-6]。黄酮及其糖苷类化合物是龙脷叶主要的活性成分之一,2015年版《中华人民共和国药典》中将山奈酚3-O-龙胆二糖苷的含量作为评价龙脷叶质量的标准。基于高通量测序技术的转录组和生物信息学分析不受基因组信息的限制,在阐明药用活性成分合成途径、挖掘相关功能基因、揭示其生长发育规律、开发中药鉴别的DNA分子标记等方面奠定了重要基础[7-8]。目前,许多药用植物的转录组学研究已经开展,例如半枝莲、白木香、茜草等,并鉴定到大量涉及黄酮类、萜类、蒽醌类等活性物质生物合成途径相关的基因[9-11]。【本研究切入点】目前,龙脷叶的研究多集中于药理作用和化学成分鉴定等方面,关于龙脷叶的转录组学分析和次生代谢产物的合成途径解析,尤其是黄酮类化合物生物合成关键基因的鉴定与研究还未见相关报道。【拟解决的关键问题】由于缺乏龙脷叶基因组信息,本研究采用Illumina HiSeqTM 4000测序技术对龙脷叶不同组织器官(叶、花和根)进行混池建库和合并组装构建龙脷叶的转录组unigene库,并基于公共数据库进行功能分析,解析龙脷叶黄酮类化合物生物合成途径及相关的功能基因,为下一步克隆黄酮类生物合成关键基因、功能验证以及开展合成调控机制研究提供基础数据。
1 材料与方法
1.1 试验材料
供试材料种植于广东省中山市永正中草药研究所的温室,经广东省农业科学院作物研究所蔡时可高级农艺师鉴定为龙脷叶(S.spatulifolius)。选取健康的3年生龙脷叶5株,分别采集叶、花和根3个不同器官,立即在液氮中冷冻,存储于-80 ℃冰箱保留备用。
1.2 试验方法
1.2.1 RNA提取 采用植物RNA提取试剂盒(Omega)分别提取龙脷叶3个器官的总RNA,用1%琼脂糖凝胶电泳检测RNA的完整性。使用Agilent 2100 Bioanalyzer评估RNA的质量和完整性。将检测合格的总RNA等量混合,用于Illumina平台进行建库与测序。
1.2.2 建库测序与拼接组装 采用带有Oligo(dT)的磁珠富集mRNA并加入fragmentation buffer片段化,通过六碱基随机引物反转录合成第一条cDNA链,然后通过DNA polymerase Ⅰ合成cDNA第二链,经过QiaQuick PCR试剂盒纯化并做末端修复、加poly(A)并连接测序接头,通过片段大小选择后进行PCR扩增,构建的龙脷叶测序文库采用Illumina HiSeqTM 4000进行测序。得到原始数据后,利用Fast QC进行评估,并使用NGS QC工具包进行过滤获得高质量的clean reads,然后采用Trinily行转录本的拼接组装获得unigene[12-13]。
1.2.3 Unigene的功能注释 采用blastx程序(E-value<0.00001)将组装的unigene在Nr、SwissProt、KEGG和KOG数据库进行比对分析。用Blast2GO软件分析GO的功能注释,并采用WEGO软件进行功能分类。根据文献报道的高等植物黄酮类合成途径,结合转录组注释的结果,采用本地blast进行比对分析,筛选参与龙脷叶黄酮类化合物合成的相关基因[14-15]。
1.2.4 TFs、R基因和SSRs鉴定分析 采用blastp将unigene编码的蛋白序列与植物转录因子数据库v4.0(http://planttfdb.cbi.pku.edu.cn/)进行比对,预测龙脷叶TFs家族;通过与植物R基因数据库PRGdb(http://PRGdb.crg.eu/wiki/Main_Page) 进行比对分析,预测龙脷叶中的R基因。采用软件MISA(http://pgrc.ipk-gatersleben.de/misa/)对龙脷叶转录组所有Unigene 存在SSRs进行鉴定分析。
2 结果与分析
2.1 龙脷叶转录组测序与de novo组装
采用Illumina HiSeqTM 4000测序平台进行龙脷叶转录组的高通量测序,共获得90 679 574条raw reads,经过数据过滤后共得到88 396 692条 clean reads,Q20达97.02%,Q30达92.49%,GC量为44.61%,表明测序质量较高,能够满足后续的生物信息学分析。利用Trinity软件,共组装46 600个unigene,平均长度877 bp,N50为1 441 bp,GC含量为41.66%。组装的unigene长度分布表明,47.77%的unigene长度在200~500 bp之间,长度大于1 000 bp和大于2 000 bp的unigene分别占29.76%和9.82%(图1A)。
2.2 龙脷叶转录组功能注释
使用BLAST软件将组装的unigene与NR、SwissProt、KOG和KEGG数据库进行比对分析获得龙脷叶转录组unigene的注释信息。由图1 B可知,共有34 188个(73.36%)unigene获得功能注释,其中34 092个(73.16%)和24 336个(52.22%)unigene分别在NR和SwissProt数据库中得到注释,9 799个(28.66%)unigene在各数据库中均得到注释,而12 412个(26.64%)unigene没有获得功能注释。由图1 C可知,龙脷叶unigene 在NR数据库相似序列匹配的近缘物种中,麻风树(Jatropha curcas)的匹配率最高,共有5 628个(16.51%)unigene,其后依次是蓖麻(Ricinus communis, 4 833个、14.18%)、胡杨(Populus euphratica, 3 819个、11.20%)、可可(Theobroma cacao, 2 086个、6.12%)、毛果杨(Populus trichocarpa, 1 924个、5.64%)。
2.3 KOG功能分类
共有(73.53%)34 267个unigene被注释到25个KOG功能分类。其中涉及一般功能预测的基因最多,共有8 501个、占24.81%,其后依次是翻译后修饰、蛋白质转换、伴侣(3 662个、10.69%)、信号转导机制(3 556个、10.38%)和转录(1 934个、5.64%),只有少数unigene被注释到细胞运动和细胞外结构(分别为15、64个),还有1 367个unigene涉及功能未知;共有1 151个unigene分配到次生代谢物生物合成、运输和分解代谢,占总数的3.36%,这些基因可能涉及龙脷叶中次生代谢物的生物合成、运输和积累。
2.4 GO功能注释分类
共有41 181个(88.37%)unigene被注释为42个GO功能分类。其中45.39%的unigene注释到生物过程中,依次为代谢过程(6 114个、14.85%)、细胞过程(4 889个、11.87%)、单有机体过程(3 720个、9.03%)和定位过程(1 043个、2.53%)等;28.94%的unigene注释到分子功能中,依次为催化活性(6 564个、15.94%)、代谢过程(6 114个、14.85%)、细胞过程(4 889个、11.87%)和结合过程(4 319个、10.62%)等;25.66%的unigene分配到细胞成分,其中细胞和细胞部分注释到的unigene数量最多(2 256个、5.48%),其后依次是细胞器(1 597个、3.83%)、膜(1 520个、3.69%)和膜部分(1 225个、2.98%)。
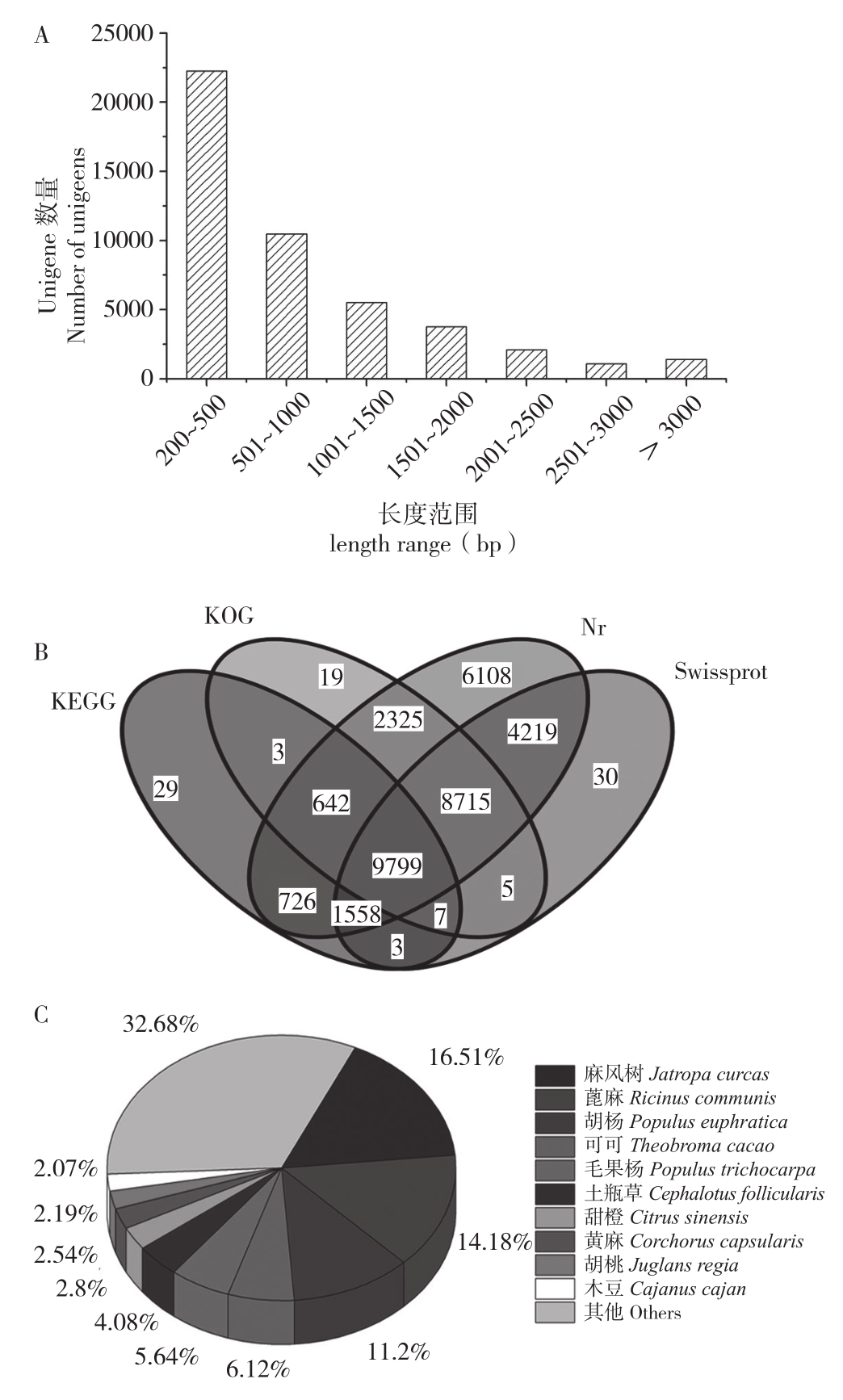
图1 龙脷叶转录组组装的unigene长度分布和功能注释
Fig. 1 Length distribution and functional annotation of unigenes assembled by S. spatulifolius Beille transcriptome
A: 龙脷叶unigene的长度分布;B: 公共数据库中注释的unigene数量;C: NR库注释到的近缘物种分布(E值≤1.0E-5)A: Length distribution of the assembled unigenes;B: The number of unigenes annotated in public databases;C: Distribution of closely related species annotated in NR database(E-value≤1.0E-5)
2.5 KEGG代谢通路分析
由图2可知,共有6 902个unigene涉及参与萜类、黄酮类、脂质等132条代谢途径。其中185个unigene涉及苯丙素生物合成(ko00940),56个unigene涉及类黄酮生物合成(ko00941),9个unigene参与黄酮和黄酮醇生物合成(ko00944),2个unigene参与异黄酮生物合成(ko00943)(图2 A);72个unigene涉及萜类骨架生物合成(ko00900),46个unigene涉及类胡萝卜素生物合成(ko00906),39个unigene涉及二萜类生物合成(ko00904),35个unigene涉及倍半萜和三萜生物合成(ko00909)和12个unigene涉及单萜生物合成(ko00902)(图2 B)。
2.6 黄酮类生物合成基因的鉴定
黄酮类通常以糖苷或其他结合物的形式积聚,合成的底物来源于苯丙酸途径和三羧酸循环。黄酮类物质是龙脷叶的主要活性成分,其中山奈酚-3-O-龙胆二糖苷的含量是评价龙脷叶质量的标准。根据龙脷叶的转录组数据绘制龙脷叶黄酮类生物合成代谢途径(图3)。在苯丙酸生物合成途径中,苯丙氨酸由苯丙氨酸解氨酶(PAL)、4-香豆酸-CoA连接酶(4CL)和肉桂酸4-羟化酶(CYP73A/C4H)催化合成肉桂酰CoA和对香豆酰CoA,作为黄酮类化合物合成的起始底物。在黄酮类化合物的生物合成中,对-香豆酰CoA和丙二酰CoA(来源于三羧酸循环)在查尔酮合酶(CHS,黄酮类化合物生物合成的第一个关键酶)的催化下产生柚皮素查尔酮或异甘草素,然后在黄酮代谢途径上游的关键酶查尔酮异构酶(CHI)的作用下,柚皮素查尔酮或异甘草素被迅速异构化形成柚皮素或甘草素。随后,甘草素作为底物主要进入异黄酮生物合成途径,柚皮素作为主要的中间产物进入多种代谢途径。黄烷酮3-羟化酶(F3H)催化柚皮素合成二氢山奈酚,直接影响黄酮类化合物的合成。黄酮醇合酶(FLS)催化二氢山奈酚形成山奈酚,作为黄酮合成途径的底物。龙脷叶中类黄酮生物合成过程中的产物在不同酶的催化下参与不同的代谢途径,产生多种次生代谢产物。在龙脷叶的转录组数据中共发现38个主要参与黄酮类的生物合成的关键基因,其中5个编码CHS,2个编码CHI,3个编码FLS(表1)。
2.7 转录组中TFs和R基因分析
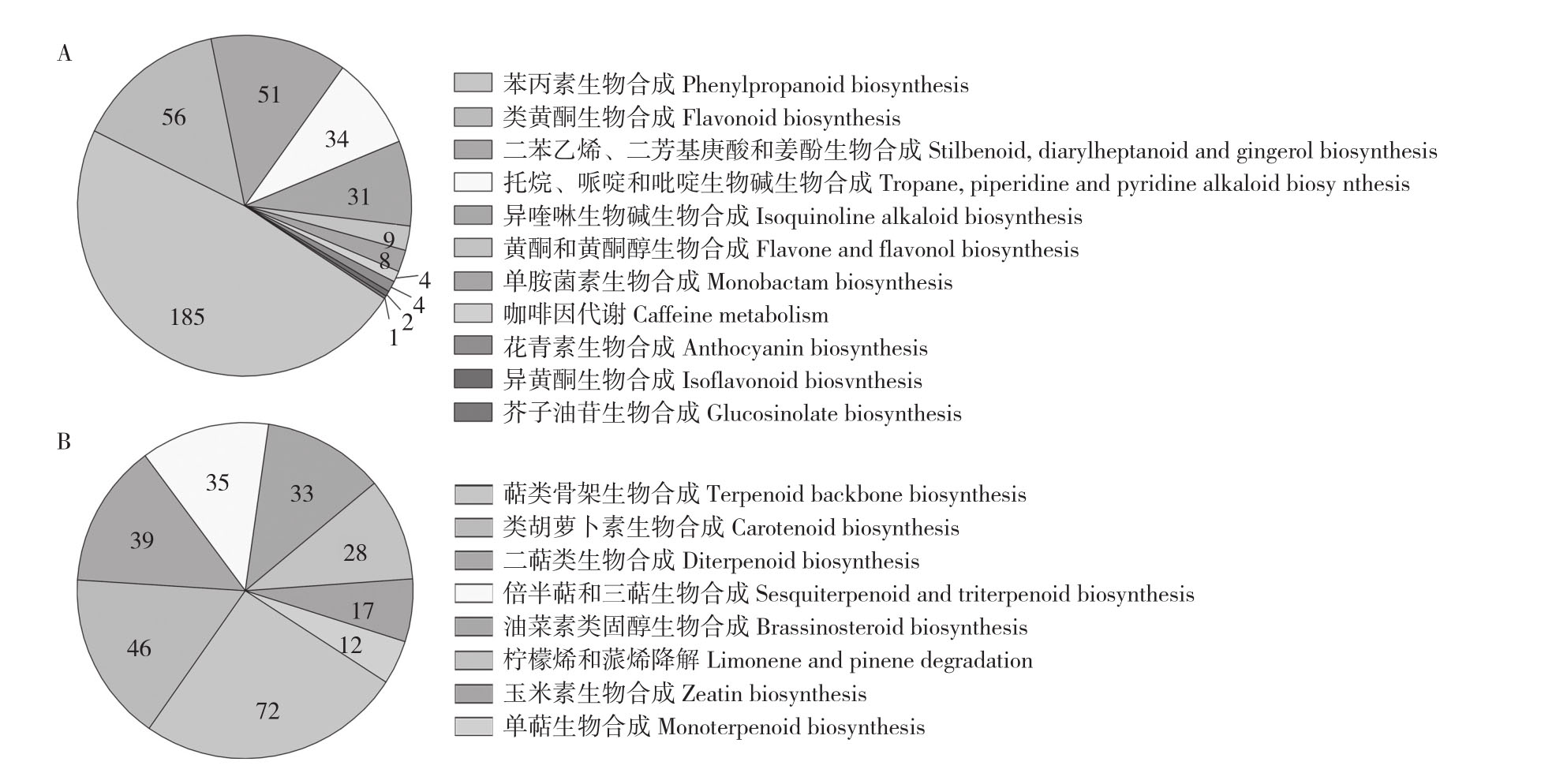
图2 基于KEGG数据库的代谢途径分析
Fig. 2 Analysis of metabolic pathways based on KEGG database
A: 其他次生代谢产物的生物合成;B: 萜类化合物和聚酮类化合物的代谢
A: Biosynthesis of other secondary metabolites; B: Metabolism of terpenoids and polyketides
TFs主要调节各种生物过程的基因表达模式,如黄酮类化合物的生物合成途径[16]。根据比对结果,1 256个unigene被分为56个不同的转录因子家族,其中Basic/Helix-Loop-Helix(bHLH)转录因子类的unigene数量最多(92个),其次是ERF、MYB、WRKY、NAC等,这些转录因子信息为进一步用于提高龙脷叶类黄酮生物合成和抗逆性提供了理论依据。植物R基因在识别病原菌的特异无毒性(Avr)基因和刺激诱导抗病的信号转导级联中起着关键作用[17]。共有3 942个unigene被分为15个不同的R基因类别,其中受体样蛋白(RLP)的种类最多(1 047个、占26.56%),其后依次是TNL(660个、占16.74%)、N(603个、占15.30%)和NL(530个、占13.44%),只有少数unigene被分配到RLPMalectin和L(分别为3、8个unigene)。
2.8 转录组中SSRs标记位点分布
SSRs是检测遗传多样性和构建遗传图谱的有效分子标记之一[18]。共从46 600个unigene中鉴定出3 356个SSRs,其中348个序列含有1个以上的SSRs,201个SSRs以复合形式存在,SSRs的平均分布密度为1/12.2kb。3 356个SSRs位点共检测到333种核苷酸基序重复序列,其中以二核苷酸(1 785个、占53.19%)和三核苷酸(1 198个、占35.70%)最多,其次是四核苷酸(147个、占4.38%)、五核苷酸(129个、占3.84%)和六核苷酸(97个、占2.89%)。龙脷叶中SSRs最丰富的重复类型是AG/CT(1 496个、占44.58%),其次是AAG/CTT(396个、占11.80%),ATC/ATG(175个、占5.21%)和ACC/GGT(172个、占5.13%)。基于3 356个SSRs,使用primer 1.1.4设计引物,为进一步研究龙脷叶的遗传多样性和构建遗传图谱提供了有价值的数据资源。
3 讨论
近年来,本草基因组学的发展加速了药用植物基因资源的保护与利用[19]。转录组学研究是本草基因组学研究的重要手段之一,有助于揭示其生长发育、响应逆境胁迫和次生代谢产物富集的调控机制,特别适用于没有完整基因组序列的药用植物的基因结构和功能预测等研究。中药龙脷叶是大戟科植物龙脷叶的干燥叶,常与其他药物配伍治疗间质性肺炎、急性支气管炎。目前关于龙脷叶化学成分和药理作用的研究不断深入,而基因组、转录组学的研究尚未见报道,制约了龙脷叶次生代谢合成途径的解析。本研究采用Illumina HiSeqTM 4000测序平台,对龙脷叶不同组织进行转录组测序并构建龙脷叶的unigene库。通过de novo组装共获得46 600个unigene,N50为1 441 bp,长度大于1 000 bp的unigene占39.58%。N50的长度是评价转录组组装质量的重要指标,龙脷叶转录组组装的N50长度与茶树(1 081 bp)的长度相近,但比新塔花的N50长(704 bp),表明龙脷叶转录组测序数据组装效果较好,能够满足后续数据分析的要求[20-21]。
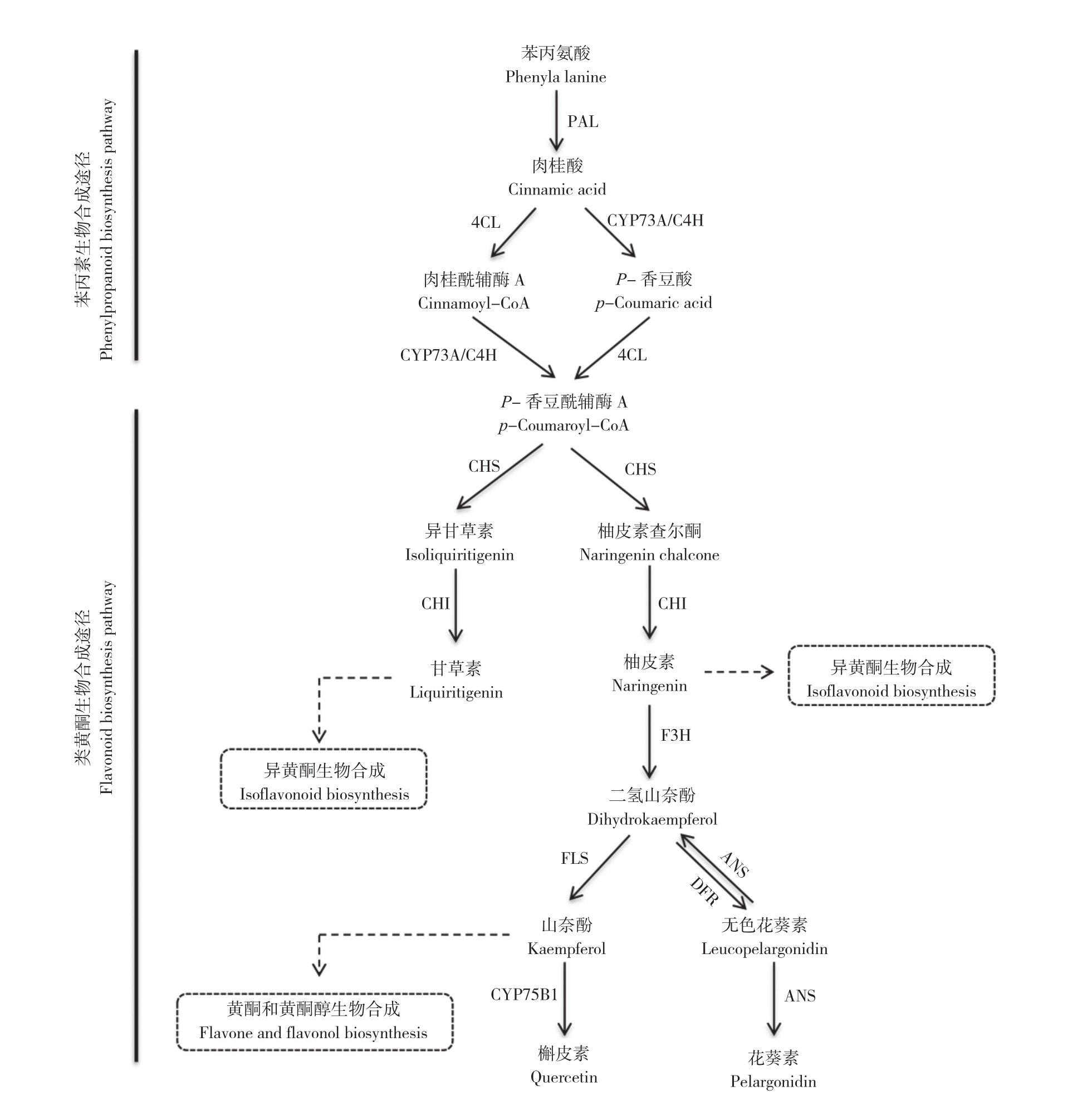
图3 龙脷叶转录组中类黄酮生物合成途径
Fig. 3 Pathway of flavonoids biosynthesis in the S. spatulifolius Beille transcriptome
PAL:苯丙氨酸解氨酶;4CL:4-香豆酰辅酶A连接酶;CYP73A/C4H: 肉桂酸4-羟化酶;CHS:查尔酮合酶;CHI:查尔酮异构酶;F3H: 黄烷酮3-羟化酶;CYB75B1:黄酮3'-单加氧酶;FLS:黄酮醇合成酶;DFR:二氢黄酮醇4-还原酶;ANS:花青素合成酶PAL: phenylalanine ammonia-lyase; 4CL: 4-coumarate-CoA ligase; CYP73A/C4H: cinnamate 4-hydroxylase; CHS: chalcone synthase;CHI: chalcone isomerase; F3H: flavanone 3-hydroxylase; CYB75B1: flavonoid 3'-monooxygenase; FLS: flavonol synthase;DFR: bifunctional dihydroflavonol 4-reductase; ANS: anthocyanidin synthase
通过与公共数据库进行比对分析获得unigene的功能注释,结果显示共有73.36%的unigene获得功能注释,其中在NR数据库中比对到最多的物种为大戟科近缘物种麻风树和蓖麻,表明龙脷叶转录组组装的序列结果可靠。但还有26.64%的unigene没有获得功能注释,可能是由于组装的unigene序列太短而缺乏保守结构域或者由于数据库中龙脷叶基因组、转录组信息匮乏导致无法获得功能注释。在KEGG数据库中共注释到6 902个unigene参与132个代谢途径,包括萜类、类胡萝卜素、生物碱类、黄酮类等次级代谢的生物合成途径。黄酮类化合物山奈酚及其衍生物具有抗氧化、抗炎、抗菌、抗癌、心脏保护、神经保护、降糖和抗骨质疏松等药理活性[22]。山奈酚3-O-龙胆苷是龙脷叶的主要活性物质之一,其含量常作为龙脷叶质量的检测标准。目前黄酮类化合物的生物合成途径在许多药用植物中已经明确,但关于龙脷叶中黄酮类的生物合成途径和相关的功能基因还未见报道。本研究通过转录组测序和功能注释共鉴定到38个unigene涉及黄酮类化合物生物合成途径的关键基因,其中5个编码CHS,2个编码CHI,3个编码FLS。转录因子能够结合到基因上游的DNA结合域,从而对基因的表达起抑制或增强的作用。Huang等[23]在紫皮柚中鉴定到MYB类转录因子CgRuby1可以激活花青苷合成的结构基因CgF3'H和CgDFR的转录表达促进花青苷的合成。本研究在龙脷叶中还鉴定到56个unigene编码 MYB转录因子。这些代谢通路和转录因子的发现为揭示龙脷叶次级代谢物的生物合成途径和调控机制提供了基础数据。
表1 类黄酮生物合成相关的候选基因数量
Table 1 Summary of candidate genes related to flavonoids biosynthesis

基因功能Gene function 标志Symbol 基因数量Gene number苯丙氨酸解氨酶 Phenylalanine ammonia-lyase PAL 8 4-香豆酸-CoA连接酶 4-coumarate-CoA ligase 4CL 10肉桂酸4-羟化酶 Cinnamate 4-hydroxylase CYP73A/C4H 1查尔酮合酶 Chalcone synthase CHS 5查尔酮异构酶 Chalcone isomerase CHI 2黄酮3'-单加氧酶 Flavonoid 3'-monooxygenase CYP75B1 5黄烷酮3-羟化酶 Flavanone 3-hydroxylase F3H 1黄酮醇合成酶 Flavonol synthase FLS 3二氢黄酮醇4-还原酶 Bifunctional dihydroflavonol 4-reductase DFR 2花青素合成酶 Anthocyanidin synthase ANS 1
4 结论
本研究利用高通量测序技术获得了龙脷叶的转录组特征,通过不同组织合并de novo组装共获得46 600个unigene,其中34 188(73.36%)个unigene在公共数据库中获得功能注释。同时,在龙脷叶中还鉴定到1 256个转录因子、3 942个R基因和3 356个SSRs。KEGG代谢通路分析表明共有6 902个unigene涉及参与萜类、黄酮类、脂质等132条代谢途径,通过功能注释筛选到56个unigene涉及类黄酮生物合成、9个unigene参与类黄酮和类黄酮生物合成,2个unigene参与异黄酮生物合成。
[1] WEI X C, LI Y M, LI M, MIN C Y, LU H, LI Q R, YUAN J, CHEN C,LI H R, ZHANG J. The protective effects of Sauropus spatulifolius on acute lung injury induced by lipopolysaccharide[J]. Journal of the Science of Food and Agriculture, 2018, 98(12): 4420-4426. doi:10.1002/jsfa.8964.
[2] 丘琴,刘玉雯,甄汉深,范秀春,甄丹丹. 龙利叶抗炎活性部位筛选及其作用机制的研究[J]. 中华中医药学刊,2020, 38(4): 37-42.doi: 10.13193/j.issn.1673-7717.2020.04.009.QIU Q, LIU Y W, ZHEN H S, FAN X C, ZHEN D D. Studies on antiinflammatory effect and mechanismof different extraction parts from Sauropus rostratus Miq[J]. Chinese Archives of Traditional Chinese Medicine, 2020, 38(4): 37-42. doi: 10.13193/j.issn.1673-7 717.2020.04.009.
[3] 陆秋娜,李兆叠,郑鸿娟,黄春阳,齐梁煜,黄月维,黄锁义. 龙脷叶提取物的抗氧化活性研究[J]. 湖北农业科学,2017, 56(1):89-90,94. doi: 10.14088/j.cnki.issn0439-8114.2017.01.022.LU Q N, LI Z D, ZHENG H J, HUANG C Y, QI L Y, HUANG Y W, HUANG S Y. Investigation of antioxidant activity of Sauropus spatulifolius Beille extract[J]. Hubei Agricultural Sciences, 2017, 56(1):89-90,94. doi: 10.14088/j.cnki.issn0439-8114.2017.01.022.
[4] 丘琴,赵应学,范秀春,甄丹丹,周颖,黄敏琪,甄汉深,庾丽峰. 龙利叶止咳活性部位筛选及其作用机制研究[J]. 中草药,2019, 50(6):1430-1434. doi: 10.7501/j.issn.0253-2670.2019.06.026.QIU Q, ZHAO Y X, FAN X C, ZHEN D D, ZHOU Y, HUANG M Q, ZHEN H S, YU L F. Antitussive effect and mechanism study of effective fractions from Sauropus rostratus[J]. Chinese Traditional and Herbal Drugs, 2019, 50(6):1430-1434. doi: 10.7501/j.issn.0253-2670.2019.06.026.
[5] 张素斌,张欣,李敏. 龙利叶多酚提取条件优化及其抗氧化活性研究[J]. 河南农业科学,2017, 46(2): 148-152. doi: 10.15933/j.cnki.1004-3268.2017.02.031.ZHANG S B, ZHANG X, LI M. Optimization of extraction conditions and antioxidant activities of polyphenols from Sauropus rostratus Miq[J]. Journal of Henan Agricultural Sciences, 2017, 46(2): 148-152. doi: 10.15933/j. cnki.1004-3268.2017.02.031.
[6] 韦建华,莫惠雯,蒙秋艳,赵惠玲,卢汝梅. 壮药龙脷叶化学成分研 究(Ⅱ)[J]. 中草药,2016, 47(20): 3560-3564. doi: 10.7501/j.issn.0253-2670.2016.20.002.WEI J H, MO H W, MENG Q Y, ZHAO H L, LU R M. Chemical constituents from Zhuang medicine Sauropus rostratus(Ⅱ)[J].Chinese Traditional and Herbal Drugs, 2016, 47(20): 3560-3564.doi: 10.7501/j.issn.0253-2670.2016.20.002.
[7] 黑小斌,李欢,李依民,王光辉,徐进,彭亮,邓翀,颜永刚,郭顺星,张岗. 药用大黄幼苗转录组高通量测序及蒽醌类生物合成基因筛选[J]. 中国药学杂志,2019, 54(7): 526-535. doi: 10.11669/cpj.2019.07.003.HEI X B, LI H, LI Y M, WANG G H, XU J, PENG L, DENG C, YAN Y G, GUO S X, ZHANG G. High-throughput transcriptomic sequencing of Rheum officinale Baill. Seedlings and screening of genes in anthraquinone biosynthesis[J]. China Journal of Chinese Materia Medica, 2019, 54(7): 526-535. doi: 10.11669/cpj.2019.07.003.
[8] 时圣明,潘明佳,王洁,陈常青. 分子鉴定技术在中药中的应用[J]. 中草药,2016, 47(17): 3121-3126. doi: 10.7501/j.issn.0253-2670.2016.17.027.SHI S M, PAN M J, WANG J, CHEN C Q. Application of molecular identification techniques in Chinese materia medica[J]. Chinese Traditional and Herbal Drugs, 2016, 47(17): 3121-3126. doi:10.7501/j.issn.0253-2670.2016.17.027.
[9] 邹毅辉,刘昌勇,林泽燕,李珍. 半枝莲转录组高通量测序及黄酮类相关基因解析[J]. 福建农业学报,2018, 33(12): 1242-1250.doi: 10.19303/j.issn.1008-0384.2018.12.003.ZOU Y H, LIU C Y, LIN Z Y, LI Z. Transcriptome sequencing and flavonoid biosynthesis-related genes of Scutellaria barbata D. Don [J].Fujian Journal of Agricultural Sciences, 2018, 33(12): 1242-1250.doi: 10.19303/j.issn.1008-0384.2018.12.003.
[10] 高志晖,赵文婷,金钺,熊换英,杨云,黄俊卿. 濒危南药白木香萜类合成酶的逆境表达模式分析[J]. 中草药,2013, 44(6): 749-754.doi: 10.7501/j.issn.0253-2670.2013.06.022.GAO Z H, ZHAO W T, JIN Y, XIONG H Y, YANG Y, HUANG J Q. Expression pattern analysis of terpene synthases of endangered Aquilaria sinensis under different stresses[J]. Chinese Traditional and Herbal Drugs, 2013, 44(6): 749-754. doi: 10.7501/j.issn.0253-2670.2013.06.022.
[11] 彭亮,张岗,颜永刚,孙涛,王媚,李洁,罗露,曹福麟,胡本祥,杨冰月. 茜草转录组分析及其蒽醌类化合物合成相关基因的挖掘[J]. 中草药,2018, 49(8):1890-1896. doi: 10.7501/j.issn.0253-2670.2018.08.024.PENG L, ZHANG G, YAN Y G, SUN T, WANG M, LI J, LUO L, CAO F L, HU B X, YANG B Y. Analysis of transcriptomes and exploring of anthraquinones biosynthetic pathway genes in Rubia cordifolia L.[J].Chinese Traditional and Herbal Drugs, 2018, 49(8):1890-1896. doi:10.7501/j.issn.0253-2670.2018.08.024.
[12] PATEL R K, JAIN M. NGS QC Toolkit: a toolkit for quality control of next generation sequencing data[J]. PloS One, 2012, 7(2): e30619.doi:10.1371/journal.pone.0030619.
[13] GRABHERR M G, HAAS B J, YASSOUR M, LEVIN J Z, THOMPSON D A, AMIT I, ADICONIS X, FAN L, RAYCHOWDHURY R, ZENG Q D, CHEN Z H, MAUCELI E, HACOHEN N, GNIRKE A, RHIND N, PALMA F D, BIRREN B W, NUSBAUM C, LINDBLAD-TOH K, FRIEDMAN N, REGEV A. Trinity: reconstructing a full-length transcriptome without a genome from RNA-Seq data[J]. Nature Biotechnology, 2011, 29(7): 644. doi: 10.1038/nbt.1883.
[14] CONESA A, GÖTZ S, GARCÍA-GÓMEZ J M, TEROL J, TALÓN M,ROBLES M. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics,2005, 21(18): 3674-3676. doi: 10.1093/bioinformatics/bti610.
[15] YE J, FANG L, ZHENG H K, ZHANG Y, CHEN J, ZHANG Z J,WANG J, LI S T, LI R Q, BOLUND L, WANG J. WEGO: a web tool for plotting GO annotations[J]. Nucleic Acids Research, 2006, 34(S2):293-297. doi: 10.1093/nar/gkl031.
[16] XU W, DUBOS C, LEPINIEC L. Transcriptional control of flavonoid biosynthesis by MYB-bHLH-WDR complexes[J]. Trends in Plant Science, 2015, 20(3): 176-185. doi: 10.1016/j.tplants.2014.12.001.
[17] SHIRANO Y, KACHROO P, SHAH J, KLESSIG D F. A gain-offunction mutation in an Arabidopsis toll interleukin1 receptornucleotide binding site-leucine-rich repeat type R gene triggers defense responses and results in enhanced disease resistance[J]. The Plant Cell, 2002, 14(12): 3149-3162. doi: 10.1105/tpc.005348.
[18] 李桂花,陈汉才,张艳,黎庭耀. 小白菜种质遗传多样性与亲缘关系的SRAP和SSR分析[J]. 广东农业科学,2017, 44(5): 37-45.doi: 10.16768/j.issn.1004-874X.2017.05.007.LI G H, CHEN H C, ZHANG Y, LI T Y. Genetic diversity and phylogenetic relationships analysis of Chinese cabbage germplasm resources by SRAP and SSR[J]. Guangdong Agricultural Sciences,2017, 44(5): 37-45. doi: 10.16768/j.issn.1004-874X.2017.05. 007.
[19] 陈士林,宋经元 . 本草基因组学[J]. 中国中药杂志,2016, 41(21):3881-3889. doi: 10.4268/cjcmm20162101.CHEN S L, SONG J Y. Herb genomics[J]. China Journal of Chinese Materia Medica, 2016, 41(21):3881-3889. doi: 10.4268/cjcmm 20162101.
[20] 庞丹丹,张亚真,徐礼羿,王丽鸳,成浩,韦康. 基于转录组测序的茶树儿茶素合成调控相关基因的挖掘[J]. 分子植物育种,2019, 17(17): 5604-5612. doi: 10.13271/j.mpb.017.005604.PANG D D, ZHANG Y Z, XU L Y, WANG L Y, CHENG H, WEI K.Excavation of key genes involved in the regulation of tea tree catechin synthesis based on transcriptome sequencing[J]. Molecular Plant Breeding, 2019, 17(17): 5604-5612. doi: 10.13271/ j.mpb.017.005604.
[21] 何江,马艺沔,杨伟俊,程波,地力努尔,马丽娜,李冠. 基于转录组测序挖掘新塔花黄酮类物质生物合成相关基因[J]. 中国中药杂志,2019, 44(15): 3178-3186. doi: 10.19540/j.cnki.cjcmm.20190628.202.HE J, MA Y M, YANG W J , CHENG B, DI L N E, MA L N, LI G.Transcriptome analysis reveals candidate genes involved in flavonoid biosynthesis in Ziziphora bunge[J]. China Journal of Chinese Materia Medica, 2019, 44(15): 3178-3186. doi: 10.19540/j.cnki.cjcmm.20190628.202.
[22] NAGULA R L, WAIRKAR S. Recent advances in topical delivery of flavonoids: A review[J]. Journal of Controlled Release, 2019, 296:190-201. doi: 10.1016/j.jconrel.2019.01.029.
[23] HUANG D, WANG X, TANG Z Z, YUAN Y, XU Y T, HE J X,JIANG X L, PENG S A, LI L, EUGENIO B, DENG X X, XU Q.Subfunctionalization of the Ruby2-Ruby1 gene cluster during the domestication of citrus[J]. Nature Plants, 2018, 4(11): 930-941.doi: 10.1038/s41477-018-0287-6.