【研究意义】红树林是重要的海洋资源,红树植物具有较强的耐潮汐能力,多分布在热带、亚热带海岸。红树林在沿海生态系统修复中发挥重要作用,但红树植物与陆生植物相比种类极少[1]。随着全球经济和环境的影响[2-3],红树植物濒危程度增加。引起红树物种濒危的原因有植物长期进化的内在因素以及一些外部因素。其中,有性生殖障碍是影响物种濒危的重要原因之一。红榄李〔Lumnitzera littorea(Jack.)Voigt〕为使君子科(Combretaceae)非胎生红树植物,分布于印度、斯里兰卡、缅甸、马来西亚、泰国和我国的海南[1,4]。由于自然界和人类活动的影响,该物种的种群出现高度破碎化并持续受到高度干扰[5]。在我国,红榄李仅自然分布于海南的三亚和陵水,由于分布区域狭小、种子败育和个体数量稀少而濒临灭绝[4-7]。目前红榄李已被列为国家二级保护植物,在世界自然保护联盟(IUCN)红色名录中被列为受威胁物种[8]。2006—2018年该物种的生物学和生态学特征被相继报道[4-7,9],但相关研究中对红榄李种子败育的分子机制仍未知。
【前人研究进展】质谱定量分析方法已经成为植物蛋白质组研究的一个重要工具。同位素标记相对和绝对定量(isobaric tags for relative and absolute quantification,iTRAQ)技术在植物蛋白组学中的应用越来越广泛[10]。iTRAQ技术已应用在植物根、叶、花药、壳和芽等各组织蛋白质组的鉴定中[10-12]。此外,植物对盐胁迫、干旱胁迫、渗透压、热、铝、汞和烟草花叶病毒(TMV)[12-15]等环境胁迫的响应也已使用iTRAQ技术进行分析。应用iTRAQ技术已鉴定出陆地棉[13]、小麦[16]、番茄[17]和葡萄[18]等胚胎发育过程中的相关蛋白。【本研究切入点】濒危植物红榄李是国家二级保护植物,种子败育是影响其濒危的一个重要因素。本研究通过iTRAQ技术初步分析其种子败育的分子机制。【拟解决的关键问题】借助iTRAQ定量蛋白质组学技术分析红榄李不同育性种子蛋白质组的表达差异。对质检合格的两个蛋白质组进行酶解,iTRAQ标记后进行高效液相色谱、质谱检测;利用Mascot软件进行蛋白质鉴定,通过iTRAQ定量寻找差异表达蛋白,并进行GO功能注释、KEGG代谢通路和蛋白互作分析,并通过实时荧光定量PCR对候选差异表达基因进行验证,以期探讨红榄李种子败育过程的分子机制。
1 材料与方法
1.1 样品收集
收集红榄李花瓣完全脱落后的种子。可育种子采集于泰国东海岸(12°23′N,102°16′E)的Lam Ri-ngun和Chanthaburi,败育种子采集于海南省三亚市铁炉港红树林保护区(18°15′N,109°42′E)。刀剖种子检测胚是否发育。
1.2 蛋白质提取与定量
采用Kambiranda等[18]的方法提取红榄李幼嫩种子的蛋白质,采用Bradford蛋白浓度测定试剂盒(Thermoffisher Scientific, UN, China)对蛋白质进行定量分析(具体操作见说明书),最后将定量好的蛋白质保存在-80℃冰箱中备用。
1.3 iTRAQ标记与SCX分馏
取100 μg总蛋白,加入Trypisn Gold溶液(样品∶试剂=30∶1),37 ℃消化16 h,真空离心干燥获得肽段干粉。用0.5 mol/L的TEAB复溶肽段,然后按照iTRAQ Reagen t8-plex试剂盒(SCIEX,Madison, WI, USA)进行标记(具体操作见说明书)。使用LC-20AB HPLC(Shimadzu, Kyoto, Japan)高效液相色谱系统进行SCX色谱分析(具体操作见说明书)。
1.4 基于QEXACTIVE的LC-ESI-MS/MS分析
采用LC-20AD纳米高效HPLC液相系统(Shimadzu, Kyoto, Japan),根据说明书用缓冲液A悬浮肽段,终浓度约为0.5 μg/μL。用自动进样系统吸入10 μL上清液,置于2 cm的C18捕集柱上,然后洗脱入10 cm 的C18分析柱(内径75 μm)上。将样品以8 μL/min的速度加载4 min,然后使用浓度梯度2%~35%的缓冲液B(98% ACN,0.1% FA)以300 nL/min的流速运行44 min;接着在2 min内使缓冲液B浓度上升至80%,保持4 min;最后在1 min内使缓冲液B浓度降到5%。将肽段通过纳米液相色谱系统(HPLC)分离,该系统直接与Q-EXACTIVE质 谱 仪(MS,ThermoFisher Scientific, San Jose,CA)连接,分离的样品进入Q-EXACTIVE质谱仪分析。MS扫描,一级MS全扫描的m/z范围为350~2 000 u,二级MS全扫描的m/z范围为100~1 800 u。
1.5 数据分析
用 Proteome Discoverer 1.2(PD 1.2,Thermo)软件和5600 MS转换器将从Orbitrap获得的原始质谱数据转换成MGF文件。蛋白质的鉴定使用Mascot软 件(Version 2.3.02, Matrix Science Inc.,MA, USA),选择已经建立好的红榄李转录组数据库(NCBI登录号:SRP127706)进行数据库检索。蛋白质鉴定,完整肽段匹配误差控制在20 mg/L以内,片段匹配误差控制在0.02 u以内,胰蛋白酶消化最多允许1个发生错位。为了减少肽段鉴定的假阳性,待鉴定蛋白须符合Mascot概率分析软件鉴定多肽的置信区间为99%以上,且在置信区间内显著性得分≥20。每个可信的鉴定蛋白都至少含有1个unique肽段。
1.6 生物信息学分析
在蛋白质定量过程中,要求每个蛋白至少包含2个unique肽段。使用Mascot软件中位数比值对蛋白质的量进行测定和标准化。当P<0.05且差异倍数>1.5时认为该蛋白差异显著。
利用Gene Ontology(GO)软件将差异表达蛋白与GO-Annotation@EBI网站下载的数据进行比对分析。为明确差异表达蛋白的生物学功能特性,采 用 COG(http://www.ncbi.nlm.nih.gov/COG/) 和KEGG(http://www.genome.jp/kegg/pathway.html)数据库进行差异蛋白分析。蛋白质的互作网络分析使用STRING软件[19]。
1.7 实时荧光定量PCR
为验证iTRAQ结果,根据姚嘉龙等[20]的方法选择8个候选基因对蛋白质组鉴定结果进行验证。使用Primer Express软件(Applied Biosystems)引物设计,引物序列见表1。以基因ubiquitin为内参,基因相对表达量的计算公式为F=2-ΔΔCt。
2 结果与分析
2.1 质谱鉴定结果
以红榄李可育种子和败育种子建立蛋白质组,通过iTRAQ技术检测共得到206 580个质谱,通过Mascot 软件分析,33 481个质谱匹配到已知质谱,30 435个质谱与独特的肽相匹配。另鉴定出6 138个已知肽段、5 769个unique肽和2 380个蛋白。肽段的数量分布如图1所示,其中至少包含2个unique肽段的蛋白质占比47%以上。而大于4肽的蛋白数量占比12.52%。
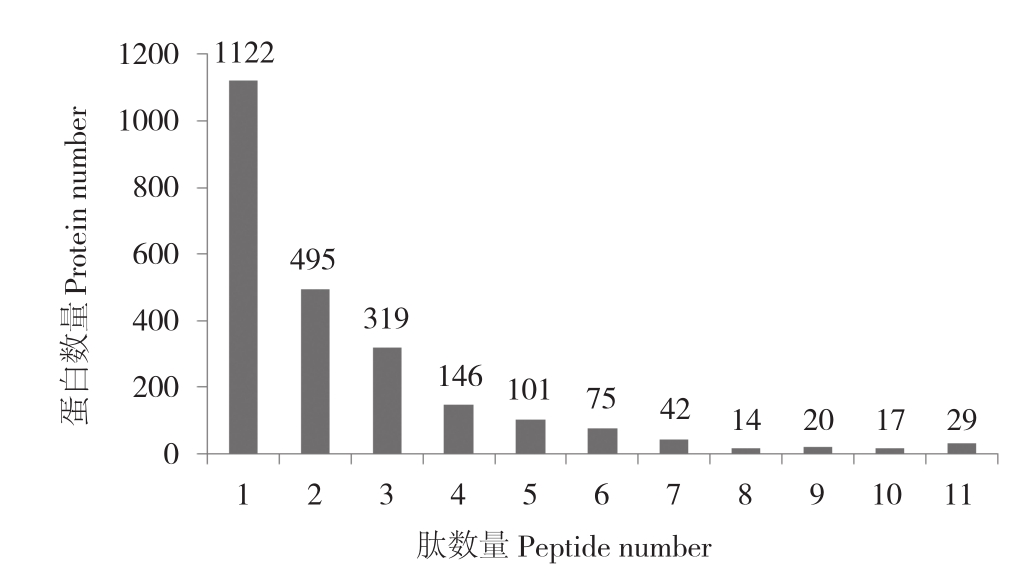
图1 蛋白数量分布
Fig. 1 Distribution of protein number
2.2 不同育性种子差异表达蛋白的生物学过程及生物学功能特性分析
在败育种子中共鉴定出448个差异表达蛋白,其中上调蛋白238个(53%)、下调蛋白210个(47%)。根据生物学过程进行分类,其中1.86%的蛋白与生殖相关,16.45%的蛋白参与代谢过程,15.78%的蛋白参与细胞过程,还有10.74%的蛋白被分类为单一生物过程蛋白(表2)。生物学功能特性分析结果表明,大多数蛋白都参与RNA加工和转运、细胞运动、无机离子转运和代谢、翻译后修饰以及蛋白质更新和蛋白伴侣等过程(图2)。
表1 候选基因qRT-PCR引物序列
Table 1 qRT-PCR primer sequences for the candidate proteins
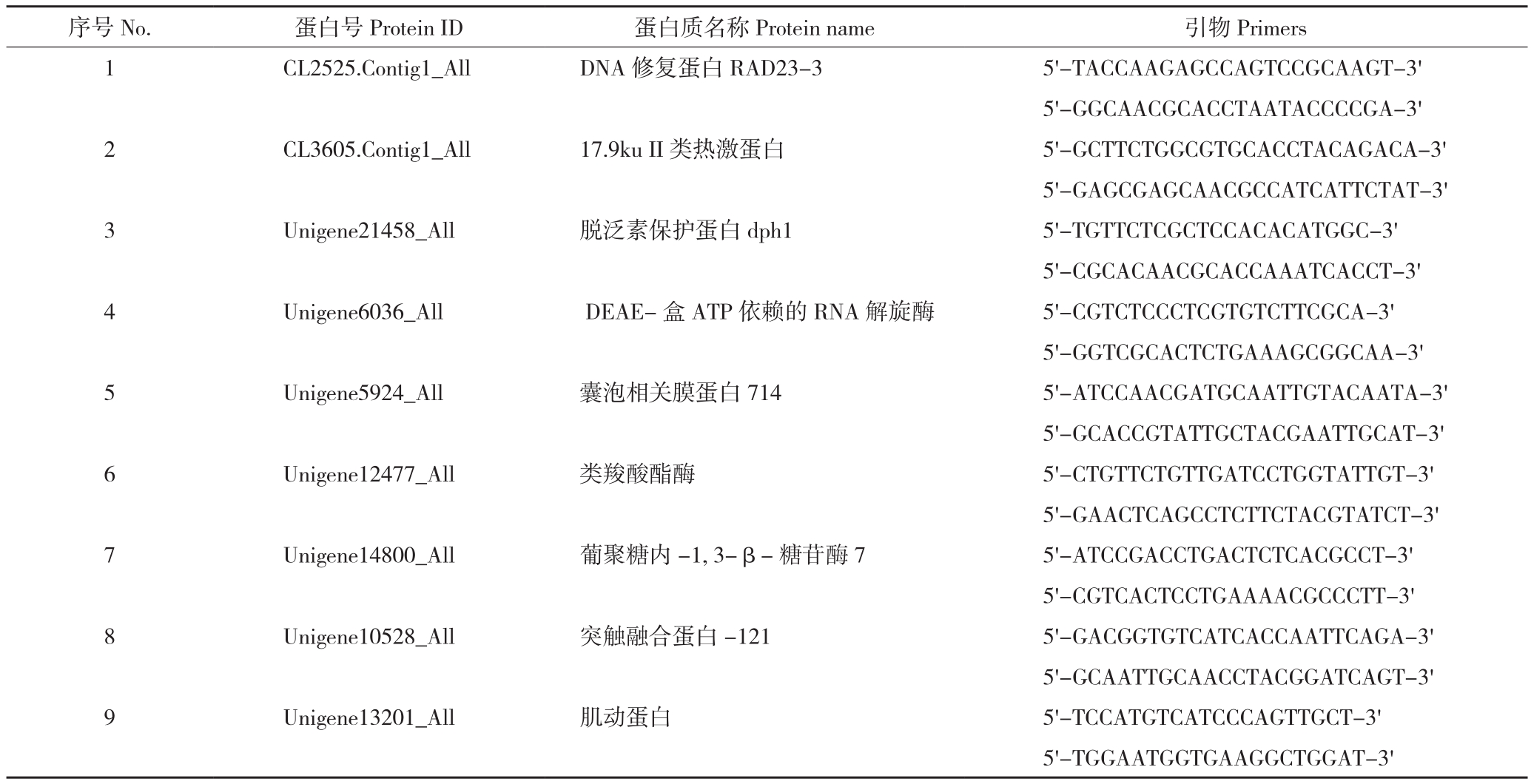
序号No. 蛋白号Protein ID 蛋白质名称Protein name 引物Primers 1 CL2525.Contig1_All DNA 修复蛋白RAD23-3 5'-TACCAAGAGCCAGTCCGCAAGT-3'5'-GGCAACGCACCTAATACCCCGA-3'2 CL3605.Contig1_All 17.9ku II类热激蛋白 5'-GCTTCTGGCGTGCACCTACAGACA-3'5'-GAGCGAGCAACGCCATCATTCTAT-3'3 Unigene21458_All 脱泛素保护蛋白 dph1 5'-TGTTCTCGCTCCACACATGGC-3'5'-CGCACAACGCACCAAATCACCT-3'4 Unigene6036_All DEAE-盒 ATP依赖的RNA解旋酶 5'-CGTCTCCCTCGTGTCTTCGCA-3'5'-GGTCGCACTCTGAAAGCGGCAA-3'5 Unigene5924_All 囊泡相关膜蛋白714 5'-ATCCAACGATGCAATTGTACAATA-3'5'-GCACCGTATTGCTACGAATTGCAT-3'6 Unigene12477_All 类羧酸酯酶 5'-CTGTTCTGTTGATCCTGGTATTGT-3'5'-GAACTCAGCCTCTTCTACGTATCT-3'7 Unigene14800_All 葡聚糖内-1, 3-β-糖苷酶7 5'-ATCCGACCTGACTCTCACGCCT-3'5'-CGTCACTCCTGAAAACGCCCTT-3'8 Unigene10528_All 突触融合蛋白-121 5'-GACGGTGTCATCACCAATTCAGA-3'5'-GCAATTGCAACCTACGGATCAGT-3'9 Unigene13201_All 肌动蛋白 5'-TCCATGTCATCCCAGTTGCT-3'5'-TGGAATGGTGAAGGCTGGAT-3'
2.3 不同育性种子差异表达蛋白GO分析
GO分析结果表明,大多数差异表达蛋白主要参与了内质网蛋白的加工、RNA转运、RNA降解、囊泡转运的SNARE相互作用、植物信号转导以及mRNA的监测等生物学途径(表3)。图3所示蛋白相互关系,参照表3中蛋白号得出,与内质网蛋白加工相关差异蛋白中的sec13同源蛋白、热休克蛋白(Hsp70)、DNA修复蛋白(RAD23-3)、核转录因子、转运相关蛋白和去泛素保护蛋白(dph1)下调,而UDP-葡萄糖转移酶和内质网素上调;与RNA转运相关的差异表达蛋白伸长因子-1-α、真核生物翻译起始因子3亚基B和C、小泛素相关修饰因子2、聚腺苷酸结合蛋白2和4、依赖ATP的DEAD-盒子RNA解旋酶38以及程序性细胞死亡蛋白4均为上调表达,而真核翻译起始因子3亚基f(elF3f)和RAN GTPase激活蛋白2在红榄李败育种子中出现下调表达;与RNA降解相关差异表达蛋白中,只有Hsp70和烯醇化酶2表现为下调,其余蛋白如热休克结合蛋白1、U6 snRNA相关Sm类蛋白LSm5和多聚腺苷酸结合蛋白2和4均为上调;与SNARE相互作用相关差异表达蛋白Syntaxin-132结合121和52上调,而Syntaxin-23和24下调;葡聚糖内切-1,3-β-葡萄糖苷酶3是唯一的植物激素信号转导相关的上调蛋白;红榄李败育种子中与mRNA监测相关的差异表达蛋白多聚腺苷酸结合蛋白表达量上调。
表2 不同育性种子差异表达蛋白的生物学过程分析
Table 2 Biological process of the differentially expressed proteins of different fertility seeds
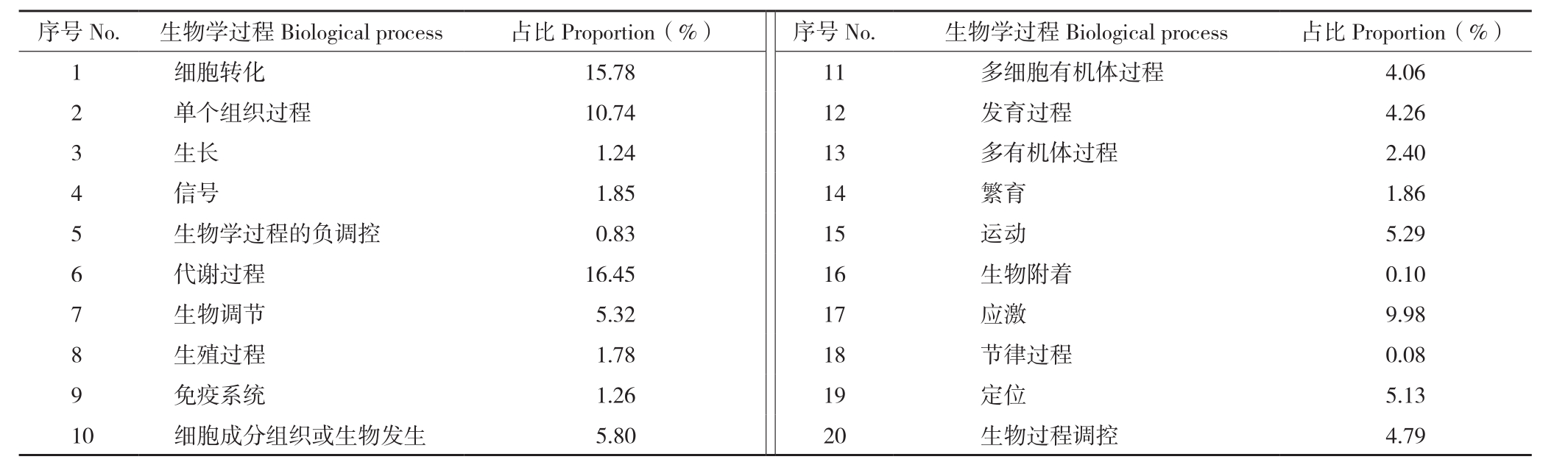
序号No. 生物学过程Biological process 占比Proportion(%) 序号No. 生物学过程 Biological process 占比Proportion(%)1 细胞转化 15.78 11 多细胞有机体过程 4.06 2 单个组织过程 10.74 12 发育过程 4.26 3 生长 1.24 13 多有机体过程 2.40 4 信号 1.85 14 繁育 1.86 5 生物学过程的负调控 0.83 15 运动 5.29 6 代谢过程 16.45 16 生物附着 0.10 7 生物调节 5.32 17 应激 9.98 8 生殖过程 1.78 18 节律过程 0.08 9 免疫系统 1.26 19 定位 5.13 10 细胞成分组织或生物发生 5.80 20 生物过程调控 4.79
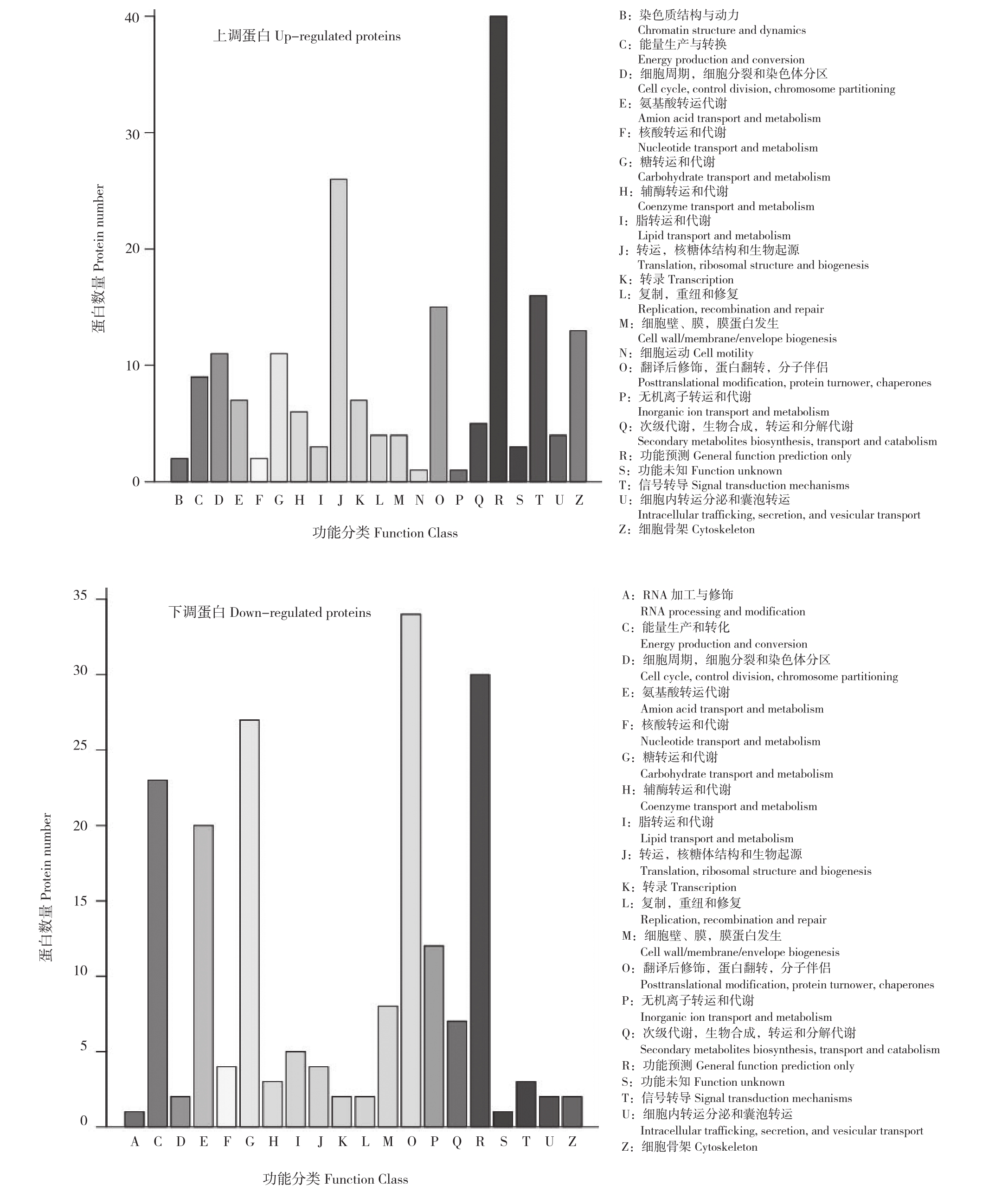
图2 不同育性种子差异表达蛋白的功能分类
Fig. 2 Functional classification of the differentially expressed proteins in different fertility seeds
表3 败育红榄李种子中差异表达蛋白的GO分析
Table 3 Differentially expressed proteins in abortive L. littorea seeds by GO analysis
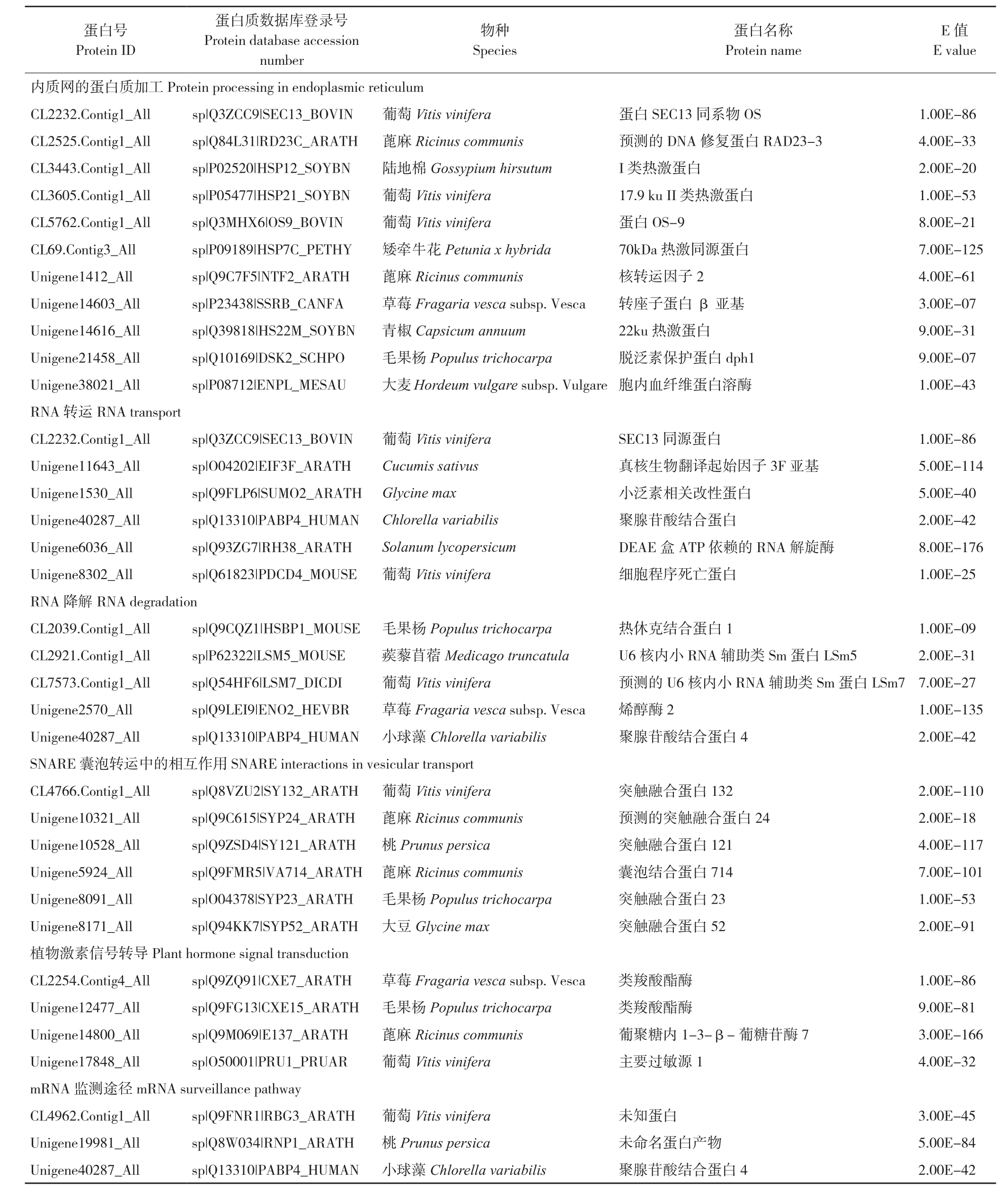
E值E value内质网的蛋白质加工Protein processing in endoplasmic reticulum CL2232.Contig1_All sp|Q3ZCC9|SEC13_BOVIN 葡萄Vitis vinifera 蛋白SEC13同系物OS 1.00E-86 CL2525.Contig1_All sp|Q84L31|RD23C_ARATH 蓖麻Ricinus communis 预测的DNA修复蛋白RAD23-3 4.00E-33 CL3443.Contig1_All sp|P02520|HSP12_SOYBN 陆地棉Gossypium hirsutum I类热激蛋白 2.00E-20 CL3605.Contig1_All sp|P05477|HSP21_SOYBN 葡萄Vitis vinifera 17.9 ku II类热激蛋白 1.00E-53 CL5762.Contig1_All sp|Q3MHX6|OS9_BOVIN 葡萄Vitis vinifera 蛋白 OS-9 8.00E-21 CL69.Contig3_All sp|P09189|HSP7C_PETHY 矮牵牛花Petunia x hybrida 70kDa 热激同源蛋白 7.00E-125 Unigene1412_All sp|Q9C7F5|NTF2_ARATH 蓖麻Ricinus communis 核转运因子2 4.00E-61 Unigene14603_All sp|P23438|SSRB_CANFA 草莓Fragaria vesca subsp. Vesca 转座子蛋白β亚基 3.00E-07 Unigene14616_All sp|Q39818|HS22M_SOYBN 青椒Capsicum annuum 22ku热激蛋白 9.00E-31 Unigene21458_All sp|Q10169|DSK2_SCHPO 毛果杨Populus trichocarpa 脱泛素保护蛋白 dph1 9.00E-07 Unigene38021_All sp|P08712|ENPL_MESAU 大麦Hordeum vulgare subsp. Vulgare胞内血纤维蛋白溶酶 1.00E-43 RNA转运RNA transport CL2232.Contig1_All sp|Q3ZCC9|SEC13_BOVIN 葡萄Vitis vinifera SEC13同源蛋白 1.00E-86 Unigene11643_All sp|O04202|EIF3F_ARATH Cucumis sativus 真核生物翻译起始因子3F亚基 5.00E-114 Unigene1530_All sp|Q9FLP6|SUMO2_ARATH Glycine max 小泛素相关改性蛋白 5.00E-40 Unigene40287_All sp|Q13310|PABP4_HUMAN Chlorella variabilis 聚腺苷酸结合蛋白 2.00E-42 Unigene6036_All sp|Q93ZG7|RH38_ARATH Solanum lycopersicum DEAE盒ATP依赖的RNA解旋酶 8.00E-176 Unigene8302_All sp|Q61823|PDCD4_MOUSE 葡萄Vitis vinifera 细胞程序死亡蛋白 1.00E-25 RNA降解RNA degradation CL2039.Contig1_All sp|Q9CQZ1|HSBP1_MOUSE 毛果杨Populus trichocarpa 热休克结合蛋白1 1.00E-09 CL2921.Contig1_All sp|P62322|LSM5_MOUSE 蒺藜苜蓿Medicago truncatula U6核内小RNA辅助类Sm蛋白 LSm5 2.00E-31 CL7573.Contig1_All sp|Q54HF6|LSM7_DICDI 葡萄Vitis vinifera 预测的U6核内小RNA辅助类Sm蛋白 LSm7 7.00E-27 Unigene2570_All sp|Q9LEI9|ENO2_HEVBR 草莓Fragaria vesca subsp. Vesca 烯醇酶2 1.00E-135 Unigene40287_All sp|Q13310|PABP4_HUMAN 小球藻Chlorella variabilis 聚腺苷酸结合蛋白4 2.00E-42 SNARE囊泡转运中的相互作用SNARE interactions in vesicular transport CL4766.Contig1_All sp|Q8VZU2|SY132_ARATH 葡萄Vitis vinifera 突触融合蛋白132 2.00E-110 Unigene10321_All sp|Q9C615|SYP24_ARATH 蓖麻Ricinus communis 预测的突触融合蛋白24 2.00E-18 Unigene10528_All sp|Q9ZSD4|SY121_ARATH 桃Prunus persica 突触融合蛋白121 4.00E-117 Unigene5924_All sp|Q9FMR5|VA714_ARATH 蓖麻Ricinus communis 囊泡结合蛋白714 7.00E-101 Unigene8091_All sp|O04378|SYP23_ARATH 毛果杨Populus trichocarpa 突触融合蛋白23 1.00E-53 Unigene8171_All sp|Q94KK7|SYP52_ARATH 大豆Glycine max 突触融合蛋白52 2.00E-91植物激素信号转导Plant hormone signal transduction CL2254.Contig4_All sp|Q9ZQ91|CXE7_ARATH 草莓Fragaria vesca subsp. Vesca 类羧酸酯酶 1.00E-86 Unigene12477_All sp|Q9FG13|CXE15_ARATH 毛果杨Populus trichocarpa 类羧酸酯酶 9.00E-81 Unigene14800_All sp|Q9M069|E137_ARATH 蓖麻Ricinus communis 葡聚糖内1-3-β-葡糖苷酶 7 3.00E-166 Unigene17848_All sp|O50001|PRU1_PRUAR 葡萄Vitis vinifera 主要过敏源1 4.00E-32 mRNA监测途径mRNA surveillance pathway CL4962.Contig1_All sp|Q9FNR1|RBG3_ARATH 葡萄Vitis vinifera 未知蛋白 3.00E-45 Unigene19981_All sp|Q8W034|RNP1_ARATH 桃Prunus persica 未命名蛋白产物 5.00E-84 Unigene40287_All sp|Q13310|PABP4_HUMAN 小球藻Chlorella variabilis 聚腺苷酸结合蛋白4 2.00E-42蛋白号Protein ID蛋白质数据库登录号Protein database accession number物种Species蛋白名称Protein name
2.4 不同育性种子差异表达基因的实时荧光定量PCR验证
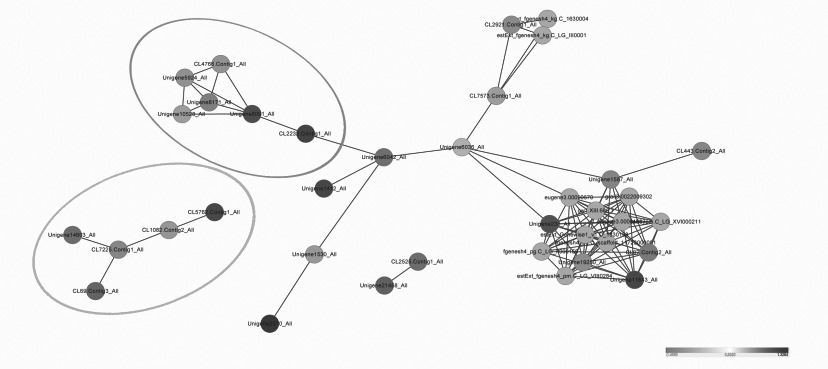
图3 败育种子中差异表达蛋白的互作网络
Fig. 3 Protein interaction network of the differentially expressed proteins in abortive seeds
蛋白质及其相互作用表现为节点和覆盖边,没有相关性的蛋白质不包括在内;深蓝色表示上调,浅蓝色表示下调。Proteins and their interactions are shown as nodes and edges. Proteins without connections are not included.Dark blue means up-regulation and light blue means down-regulation.
为了验证iTRAQ分析的结果,在GO分析的基础上从差异表达蛋白中筛选出8个候选基因进行qRT-PCR检测,从转录表达层面上对蛋白质组鉴定结果进行验证。其中4个基因RAD23、protein714、Syntaxin-121、依赖ATP的DEAD-盒子RNA解旋酶基因在败育种子中上调表达,而另外4个基因17.9 ku的Ⅱ类热休克蛋白基因、去泛素保护蛋白dph1基因、葡聚糖内切-1,3-β-葡萄糖苷酶基因、羧酸酯酶基因则下调表达(图4)。在这8个差异基因中除了RAD23外其他7个基因的表达量与蛋白质组检测结果一致。
3 讨论
在我国,濒危红树植物红榄李存在严重的有性生殖障碍,表现为种子败育率高,在没有人工辅助育种的情况下种子萌发率几乎为零[6,8],存在大量空胚现象[5,8]。但通过人工辅助育种等技术手段可实现红榄李种子的萌发和幼苗抚育[4]。本研究利用iTRAQ的方法分析了红榄李败育种子和可育种子的蛋白组学差异情况,旨在揭示我国红榄李种子发育过程中种子败育的分子机制。在448个差异表达蛋白中,内质网加工、RNA转运、RNA降解、SNARE在囊泡运输中的相互作用、植物激素信号转导和mRNA监测等7类植物生理生化过程的蛋白都表现出不同的表达量。前人研究发现,这些相关蛋白均不同程度参与陆地棉[13]、小麦[14] 、金鱼草[21]、番茄[22]、大豆[23]、拟南芥[24]、玉米[25]等众多植物的胚胎发育过程,且不同程度调控植物的育性。这些差异蛋白的揭示为研究红榄李种子败育机制和采用基因工程手段进行深入研究提供了研究基础。
3.1 内质网加工蛋白
在败育红榄李种子中有11种内质网加工相关蛋白差异表达,其中sec13同源蛋白、DNA修复蛋白(RAD23-3)表达下调。在拟南芥中,sec13同源蛋白1通过控制AtDRP2A的膜结合在液泡运输中发挥作用[26]。在金鱼草花发育过程中,RAD蛋白通过抑制DIV蛋白的表达抑制花的发育[21],而花的发育状态直接影响种子的发育进程。在番茄果实发育过程中,类RAD蛋白的功能非常保守,但其表达却由环境和分子结合伴侣共同决定[22]。前期研究表明,参与淀粉代谢相关蛋白的表达会调控大豆雄性不育性状[23]。本研究中红榄李败育种子的UDP-葡萄糖转移酶出现了上调表达。植物的内质网素对植物蛋白分泌代谢起重要作用,对植物组织的生长起促进作用[27],该类蛋白在红榄李败育种子的蛋白质组中表现为上调。
3.2 RNA转运
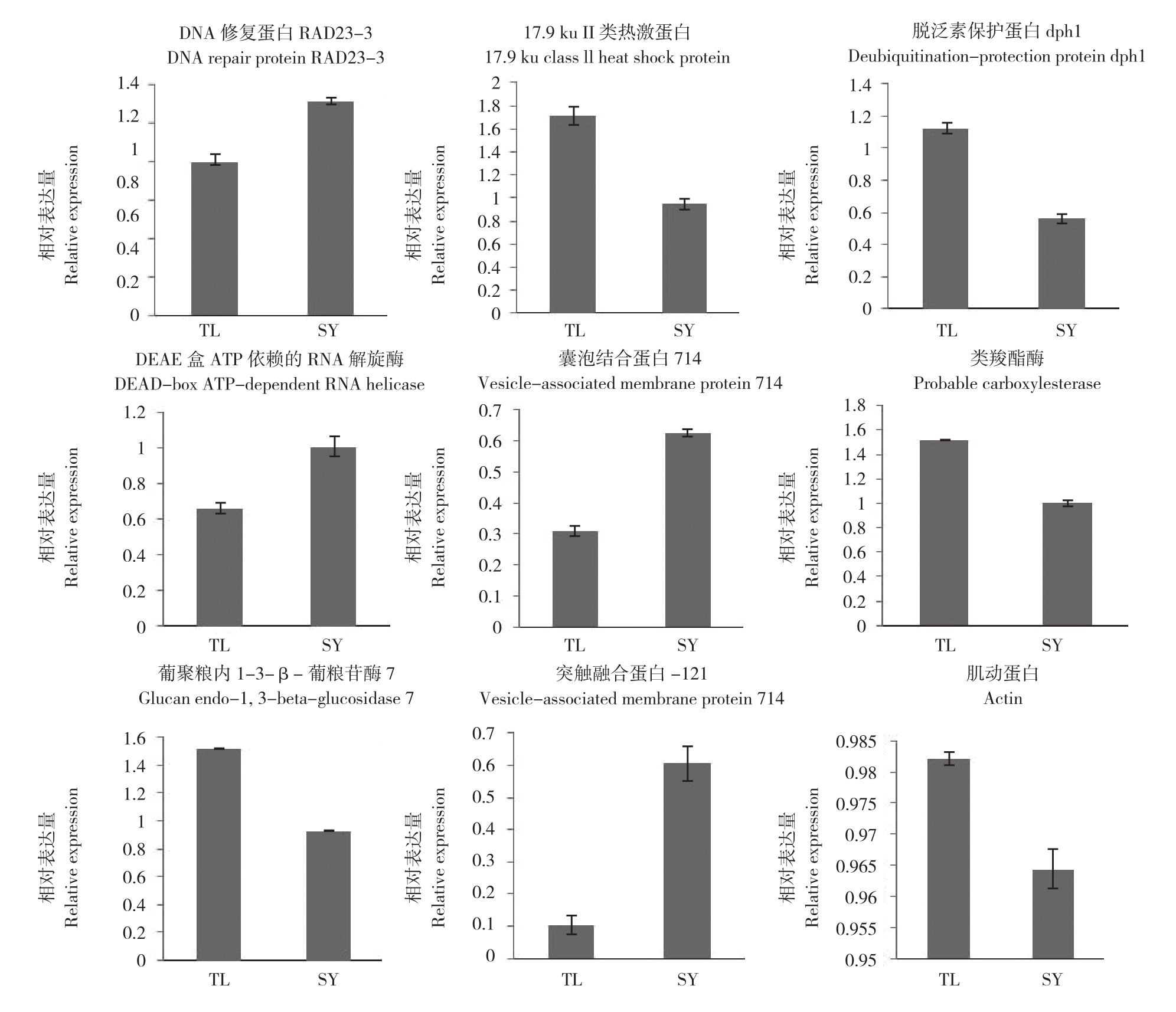
图4 差异表达基因的qRT-PCR验证
Fig. 4 qRT-PCR validation of differentially expressed genes
TL:采自泰国红榄李的种子;SY:采自我国三亚红榄李的种子TL: L. littorea seeds from Thailand; SY: L. littorea seeds from Sanya, China
植物延长因子参与细胞周期进程和部分细胞延长过程中蛋白质的合成。本研究在败育种子差异表达蛋白中发现了一种参与RNA翻译过程的因子即延长因子-1-α,以及其他7种蛋白,包括真核生物翻译起始因子3亚基B和C、小泛素相关修饰因子2、聚腺苷酸结合蛋白2和4、依赖ATP的DEAD-盒子RNA解旋酶38以及程序性细胞死亡蛋白4,均为上调表达。已有研究表明,翻译起始因子可控制拟南芥热响应性生长的翻译终止和终止密码子的通读[24],同时也会影响植物幼苗mRNA的选择性翻译[28]。RNA解旋酶是一种依赖NTP水解释放能量来解旋双链RNA分子的酶。RNA解旋酶与RNA分子所有活动过程相关,包括核转录、剪切、核糖体生物合成、RNA输出和细胞器的基因表达,广泛参与植物的生长发育和胁迫反应[25]。另外3种RNA转运相关蛋白SEC13同源蛋白OS、真核翻译起始因子3亚基f(elF3f)和RAN GTPase激活蛋白2在红榄李败育种子中出现下调表达。elF3f蛋白表达与植物的小配子发生有关,从而影响花粉育性和种子发育情况[29]。RAN类蛋白是ras超家族中一种保守的小信号GTPase,参与RNAs和蛋白质的核质运输[30]。同时也参与有丝分裂过程,包括DNA的合成调控,纺锤体组装,中心体复制,染色体排列、分离和解聚和核膜形成等[31]。在马铃薯中,RAN GTPase激活蛋白2在其免疫防护中发挥重要作用[32]。
3.3 RNA降解
在RNA降解相关蛋白中,热休克蛋白(HSP)、谷胱甘肽S-转移酶、抗坏血酸过氧化物酶和铜/锌超氧化物歧化酶均积极参与植物胚胎发育过程且发挥重要作用[26,31-32]。本研究鉴定出了一些小分子量的热休克蛋白,如22 ku的Ⅱ类和17.9 ku的I类热休克蛋白,这些小分子量的热休克蛋白参与大豆的胚胎发育过程[33]。由蛋白互作网络图可见,HSP蛋白表现上调,而UGGT蛋白表现下调,这两种蛋白质协同作用,可能在红榄李败育种子的发育过程中发挥作用。
3.4 囊泡运输中的SNARE相互作用
在植物细胞中SNAER介导的囊泡运输将细胞壁和膜上的物质传递到细胞表面,在细胞的稳态、扩张和生长中发挥重要作用[34]。SNAER蛋白参与植物对细菌的抗性和病理相关蛋白的分泌[35],能够增强植物对干旱和病原体的抗性[36]。前期研究表明,红榄李败育种子受病害和虫害的比例极高[4-6]。本研究参与到囊泡运输中的SNARE相互作用的差异表达是否为红榄李种子发育过程中对病害和虫害的应急响应仍需要持续验证。
3.5 植物激素信号转导
在属于植物激素信号转导的蛋白中,内切-1,3-β-葡聚糖酶广泛分布在细菌、真菌和高等植物中,可以水解多糖的特定糖苷键(β-1,3-葡聚糖残基键)。在红榄李败育种子中葡聚糖内切-1,3-β-葡萄糖苷酶3是唯一与植物激素信号转导相关的上调蛋白。内切-1,3-β-葡聚糖酶常在整个植物体内表达,在小花、叶鞘和叶片中高度表达,它也是水稻花粉发育过程中胼胝质降解所必需的[37]。在败育红榄李种子中,两个与病理相关蛋白表现下调,这两个蛋白是植物激素信号转导蛋白中较为重要的蛋白,受到生物和非生物胁迫诱导[38]。
3.6 mRNA监测途径
本研究发现在mRNA监测途径中的差异蛋白有3个,其中2个功能未知,1 个为聚腺苷酸结合蛋白。通过蛋白质组表达谱比较分析发现,多聚腺苷酸结合蛋白与水稻温敏性雄性不育相关[39]。酵母多聚腺苷酸结合蛋白基因(PAB1)在植物中表达时可作为一种致病模拟基因[40]。在红榄李败育种子中多聚腺苷酸结合蛋白表达量上调。葡萄糖和植物激素(如ABA、GA和乙烯)协同调控植物种子的萌发和幼苗早期发育。真核生物释放因子1-2(在半不育种子中表达量下调)影响拟南芥种子萌发和幼苗早期发育过程中对葡萄糖和植物激素的响应[41]。
4 结论
本研究借助iTRAQ技术对红榄李不同育性种子的蛋白质组表达差异进行分析,经GO分析发现大多数差异表达转录因子参与了内质网蛋白加工、RNA转运、RNA降解、囊泡转运的SNARE相互作用、植物信号转导和mRNA监测途径。而许多研究都证明这些差异表达转录因子大多与花粉和种子发育、种子萌发、幼苗早期发育、环境胁迫和抗病性有关。在GO分析的基础上筛选出8个候选基因进行qRT-PCR分析,结果显示有7个基因的表达量与蛋白质检测结果一致,这从转录表达层面上对蛋白质鉴定的结果进行了验证,从而肯定了蛋白质组检测结果的正确性,为红榄李败育种子的分子机制深入研究奠定了基础。
[1] POLIDORO B A, CARPENTER K E, COLLINS L, DUKE N C, ELLISON A M, FARNSWORTH E J, FERMANDO E S,KATHIRESAN K, KOEDAM N E, LIVINGSTONE S R, MIYAGI T,MOORE G E, NAM V N, ONG J E, PRIMACERA J H, SALMO S G,SANCIANGCO J C, SUKARDJO S, WANG Y M, YONG H J W. The loss of species: mangrove extinction risk and geographic areas of global concern[J]. PLOS ONE, 2010, 5(4): e10095. doi:10.1371/journal.pone.0010095.
[2] DUKE N C, MEYNECKE J O, DITTMANN S, ELLISON A M, ANGER K, BERGER U, CANNICCI S, DIELE K, EWEL K C, FIELD CD,KOEDAM N, LEE S Y, MARCHAND C, NORDHAUS I, DAHDOUHGUEBAS F. A world without mangroves[J]. Science, 2007, 317(5834):41-42. doi:10.1126/science.317.5834.41b.
[3] DARU B H, YESSOUFOU K, MANKGA L T, DAVIES T J. A global trend towards the loss of evolutionarily unique species in mangrove ecosystems[J]. PLOS ONE, 2013, 8(6): e66686. doi:10.1371/journal.pone.0066686.
[4] 张颖,钟才荣,杨勇,钟红兵,曾志平,张晶,张世杰. 濒危红树植物红榄李种质资源挽救[J]. 分子植物育种,2018 16(12):4112-4118.doi:10.13271/j.mpb.016.004112.ZHANG Y, ZHONG C R, YANG Y, ZHONG H B, ZENG Z P, ZHANG J, ZHANG S J. Rescue of germplasm resources of endangered mangrove plant Lumnitzera littorea[J]. Molecular Plant Breeding, 2018 16(12):4112-4118. doi:10.13271/j.mpb.016.004112.
[5] 张颖,李燕华,张晓楠,杨勇. 濒危红树植物红榄李开花生物学特征及繁育系统[J]. 应用与环境生物学报,2017, 23(1):77-81.ZHANG Y, LI Y H, ZHANG X N, YANG Y. Flower phenology and breeding system of endangered mangrove Lumnitzera littorea(Jack.)Voigt[J]. Applied and Environmental Biology, 2017, 23(1):77-81.
[6] 张颖,钟才荣,李诗川,严廷良,管伟. 濒危红树植物红榄李[J]. 林 业 资 源 管 理,2013, 5:103-107,151. doi:10.13466/j.cnki.lyzygl.2013.05.026.ZHANG Y, ZHONG C R, LI S C, YAN T L, GUAN W. Endangered mangrove Lumnitzera littorea(Jack.)Voigt[J]. Forest Resources Management, 2013, 5:103-107,151. doi:10.13466/j.cnki.lyzygl.2013.05.026.
[7] 杨勇,钟才荣,李燕华,张颖. 濒危红树植物红榄李种子形态及萌发特性[J]. 分子植物育种,2016, 14(10):2851-2858. doi:10.13271/j.mpb.014.002851.YANG Y, ZHONG C R, LI Y H, ZHANG Y. The morphological structure and germination characters of seed of endangered mangrove Lumnitzera littorea(Jack.)Voigt[J]. Molecular Plant Breeding,2016, 14(10):2851-2858. doi:10.13271/j.mpb.014.002851.
[8] 范航清,陈利洪. 中国濒危红树植物红榄李的种群数量及其分布[J].广西科学,2006, 13(3):226-227.FAN H Q, CHEN L H. Current distribution of endangered mangrove lumnitzera littorea(Jack.)voigt in China[J]. Guangxi Sciences,2006, 13(3):226-227.
[9] 张晓楠,钟才荣,严廷良,张颖. 人工辅助授粉探寻濒危红树植物红榄李种质资源挽救途径[J]. 生态科学,2016, 35(5):38-42.doi:10.14108/j.cnki.1008-8873.2016.05.006.ZHANG X N, ZHONG C R, YAN T L, ZHANG Y. The germplasm resource rescue of endangered mangrove(Lumnitzera littorea(Jack.)Voigt)by artificial pollination[J]. Ecological Science, 2016, 35(5):38-42. doi:10.14108/j.cnki.1008-8873.2016.05.006.
[10] 唐秀英,王会民,龙起樟,黄永兰,芦明,曹志斌,万建林. 基于iTRAQ技术筛选东乡野生稻根系低温响应蛋白[J]. 分子植物育种,2019, 17(11): 3624-3631. doi:10.13271/j.mpb.017.003624.TANG X Y, WANG H M, LONG, Q Z, HUANG Y L, LU M, CAO Z B,WAN J L. Identification of cold responsive proteins in Dongxiang wild rice root based on iTRAQ technique[J]. Molecular Plant Breeding,2019, 17(11): 3624-3631. doi: 10.13271/j.mpb.017.003624.
[11] LI W, ZHAO F A, FANG W P, XIE D Y, HOU J A, YANG X J, ZHAO Y M, TANG Z J, NIE L H, LV S P. Identification of early salt stress responsive proteins in seedling roots of upland cotton(Gossypium hirsutum L.)employing iTRAQ-based proteomic technique[J].Frontiers in Plant Science, 2015(6): 732-732. doi:10.3389/fpls.2015.00732.
[12] WANG J, WANG X R, ZHOU Q, YANG J M, GUO H X, YANG L J,LIU W Q. iTRAQ protein profile analysis provides integrated insight into mechanisms of tolerance to TMV in tobacco(Nicotiana tabacum)[J]. Journal of Proteomics, 2016 28(132): 21-30. doi: 10.1016/j.jprot.2015.11.009
[13] GE X, ZHANG C J, WANG Q H, YANG Z R, WANG Y, ZHANG X Y,WU Z X, HOU Y X, WU J H, LI F G. iTRAQ protein profile differential analysis between somatic globular and cotyledonary embryos reveals stress, hormone, and respiration involved in increasing plantlet regeneration of gossypium hirsutum L[J]. Journal of Proteome Research, 2015, 14(1):268-278. doi: /10.1021/pr500688g.
[14] KANG G Z, LI G Z, WANG L N, WEI L T, YANG Y, WANG P F,YANG Y Y, WANG Y H, FENG W, WANG C Y, GUO T C. Hgresponsive proteins identified in wheat seedlings using iTRAQ analysis and the role of ABA in Hg stress[J]. Journal of Proteome Research,2015, 14(1): 249-267. doi:10.1021/pr5006873.
[15] 王晓丹,肖钢,张振乾,肖楠,陈浩,官春云. 转录组学和蛋白质组学关联分析在植物研究中的应用[J]. 基因组学与应用生物学,2018,37(1):432-439. doi:10.13417/j.gab.037.000432.WANG X D, XIAO G, ZHANG Z Q, XIAO N, CHEN H, GUAN C Y,Application of transcriptomics and proteomics correlation abalysis in plant research[J]. Genomics and Applied Biology, 2018, 37(1):432-439. doi:10.13417/j.gab.037.000432.
[16] MA C Y, ZHOU J W, CHEN G X, BIAN Y W, LV D W, LI X H,WANG Z M, YAN Y M. iTRAQ-based quantitative proteome and phosphoprotein characterization reveals the central metabolism changes involved in wheat grain development[J]. BMC Genomics, 2014, 15(1):1029-1029. doi:10.1186/1471-2164-15-1029.
[17] 叶必顺,林敏梨,陈萱,王小红,周婷,赖童飞. 利用iTRAQ分析番茄果实从绿熟向破色转化过程中蛋白质组的变化[J]. 杭州师范大学学报(自然科学版),2019, 18(6): 592-598,650. doi:10.3969/j.issn.1674-232X.2019.06.006.YE B S, LIN M L, CHEN X, WANG X H, ZHOU T, LAI T F. Proteomic variation of tomato fruit in the transition process from mature green to breaker by iTRAQ[J]. Journal of Hangzhou Normal University(Natural Science Edition), 2019, 18(6): 592-598,650. doi:10.3969/j.issn.1674-232X.2019.06.006.
[18] KAMBRIANDA D M, KATAM R, BASHA S M, SIEBERT S. iTRAQ-based quantitative proteomics of developing and ripening muscadine grape berry[J]. Journal of Proteome Research, 2014, 13(2): 555-569. doi:10.1021/pr400731p.
[19] FRANCESCHINI A, SZKLARCZYK D, FRANKILD S, KUHN M,SIMONOVIC M, ROTH a, LIN J, MINGUEZ P, BORK P, MERING C V,JENSEN L J. STRING v9.1: protein-protein interaction networks, with increased coverage and integration[J]. Nucleic Acids Res, 2013, 41:D808-D815. doi:10.1093/nar/gks1094.
[20] 姚嘉龙,郭蓓,谢皓.叶绿素合成关键酶基因表达的半定量RTPCR分析[J]. 基因组学与应用生物学,2015,34(3):593-598.YAO J L, GUO B, XIE H. Semi-quantitative RT-PCR analysis of gene expression of key enzymes for chlorophyll synthesis[J]. Genomics and Applied Biology, 2015,34(3):593-598.
[21] CORLEY S B, CARPENTER R, COPSEY L, COEN E. Floral asymmetry involves an interplay between TCP and MYB transcription factors in Antirrhinum[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(14): 5068-5073. doi:10.1073/pnas.0501340102.
[22] MACHEMER K, SHAIMAN O, SALTS Y, SHABTAI S, SOBOLEV I,BELAUSOVE, GROTEWOLD E, BARG R. Interplay of MYB factors in differential cell expansion, and consequences for tomato fruit development[J]. Plant Journal, 2011, 68(2): 337-350. doi:10.1111/j.1365-313X.2011.04690.x.
[23] LI J J, HAN S H, DING X L, HE T T, DAI J Y, YANG S P,GAI J Y.Comparative transcriptome analysis between the cytoplasmic male sterile line NJCMS1A and its maintainer NJCMS1B in Soybean(Glycine max(L.)Merr.[J]. PLOS ONE, 2015, 10(5): e0126771. doi:10.1371/journal.pone.0126771.
[24] BOX M S, HUANG BE, DOMIJAN M, JAEGER K E, KHATTAK A K, YOO S J, SEDIVY E L, JONES D M, HEARN T J, WEBB A A R, GRANT A, LOCKE J C W, WIGGE P A. ELF3 controls thermoresponsive growth in Arabidopsis[J]. Current Biology, 2015,25(2): 194-199. doi:10.1016/j.cub.2014.10.076.
[25] XU R R, ZHANG S Z, HUANG J G, ZHENG C C. Genome-wide comparative in silico analysis of the RNA helicase gene family in Zea mays and Glycine max: a comparison with Arabidopsis and Oryza sativa[J]. PLOS ONE, 2013, 8(11):e78982. doi:10.1371/journal.pone.0078982.
[26] LEE M, LEE S, KIM H, Jin JB, Kim D, Hwang I. A WD40 repeat protein, Arabidopsis Sec13 homolog 1, may play a role in vacuolar trafficking by controlling the membrane association of AtDRP2A[J].Molecules and Cells, 2006, 22(2): 210-219.
[27] KLEIN E M, MASCHERONI L, POMPA A, RAGNI L, WEIMAR T,LILLEY K S, DUPREE P, VITALE A. Plant endoplasmin supports the protein secretory pathway and has a role in proliferating tissues[J]. Plant Journal, 2006, 48(5): 657-673. doi:10.1111/j.1365-313X.2006.02904.x.
[28] LU S X, WEBB C J, KNOWLES S M, KIM S H, WANG Z Y, TOBIN E M. CCA1 and ELF3 interact in the control of hypocotyl length and flowering time in Arabidopsis[J]. Plant Physiology, 2012, 158(2):1079-1088. doi:10.1104/pp.111.189670.
[29] LI Q, DENG Z Y, GONG C Y, WANG T. The rice eukaryotic translation initiation factor 3 subunit f(OseIF3f)is involved in microgametogenesis[J]. Frontiers in Plant Science, 2016(7): 532-532. doi:10.3389/fpls.2016.00532.
[30] AVIS J M, CLARKE P R. Ran, a GTPase involved in nuclear processes:its regulators and effectors[J]. Journal of Cell Science, 1996, 109(10):2423-2427.
[31] BORUC J, GRIFFIS A H N, RODRIGOPEIRIS T, ZHOU, X, TIFLORD B, DAMME D V, MEIER I. GAP activity, but not subcellular targeting,is required for Arabidopsis RanGAP cellular and developmental functions[J]. The Plant Cell, 2015, 27(7): 1985-1998. doi:10.1105/tpc.114.135780.
[32] SOBHANIAN S, SACCO M A. Divergent evolution of potato immune receptor CC domain interactions with the Ran GTPase-activating protein 2[J]. Plant Signaling & Behavior, 2014, 9(9): e29772.doi:10.4161/psb.29772.
[33] ZHU B G, YE C J, LU H Y, CHEN X J, CHAI G H, CHEN J N, WANG C.Identification and characterization of a novel heat shock transcription factor gene, GmHsfA1, in soybeans(Glycine max)[J]. Journal of Plant Research, 2006, 119(3): 247-256. doi:10.1007/s10265-006-0267-1.
[34] KARNIK R, ZHANG B, WAGHMARE S, ADERHOLD C, GREFEN C,BLATT M R. Binding of SEC11 indicates its role in SNARE recycling after vesicle fusion and identifies two pathways for vesicular traffic to the plasma membrane[J]. The Plant Cell, 2015, 27(3): 675-694.doi:10.1105/tpc.114.134429.
[35] KALDE M, NUHSE T S, FINDLAY K, PECK S C. The syntaxin SYP132 contributes to plant resistance against bacteria and secretion of pathogenesis-related protein 1[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(28):11850-11855. doi:10.1073/pnas.0701083104.
[36] 薛媛. 拟南芥SNARE蛋白VAMP711通过调控质膜H+-ATPase响应干旱胁迫的机制研究[D]. 北京:中国农业大学,2019.XUE Y. Regulation of plasma membrane H+-ATPase activity by SNARE protein VAMP711 under drought stress in Arabidopsis[D].Beijing: China Agriculture University, 2019.
[37] WAN L, ZHA W, CHENG X, LIU C, LV L, LIU C X, WANG Z Q, DU B, CHEN R Z, ZHU L L, HE G C. A rice β-1,3-glucanase gene Osg1 is required for callose degradation in pollen development[J]. Planta,2011, 233(2): 309-323. doi:10.1007/s00425-010-1301-z.
[38] VAN LOON L C, REP M, PIETERSE C M. Significance of inducible defense-related proteins in infected plants[J]. Annual Review of Phytopathology, 2006, 44(1): 135-162. doi:10.1146/annurev.phyto.44.070505.143425.
[39] PAN Y, LI Q, WANG Z, WANG Y, MA R, ZHU L L, HE G C, CHEN R Z. Genes associated with thermosensitive genic male sterility in rice identified by comparative expression profiling[J]. BMC Genomics,2014, 15(1): 1114-1114. doi:10.1186/1471-2164-15-1114.
[40] LI Q S, VON LANKEN C, YANG J J, LAWERNCE C B, HUNT A G.The yeast polyadenylate-binding protein(PAB1)gene acts as a disease lesion mimic gene when expressed in plants[J]. Plant Molecular Biology, 2000, 42(2): 335-344. doi:10.1023/A:1006354010997.
[41] ZHOU X, COOKE P, LI L. Eukaryotic release factor 1-2 affects Arabidopsis responses to glucose and phytohormones during germination and early seedling development[J]. Journal of Experimental Botany, 2010, 61(2): 357-367. doi:10.1093/jxb/erp308.