【研究意义】非洲猪瘟病(African swine fever,ASF)是一种由非洲猪瘟病毒(African swine fever virus,ASFV)导致的,能引起猪高度致死(死亡率可达100%)的急性传染病[1-2]。我国是全球猪肉消费大国,养殖生猪总量占世界的50%,但我国养猪条件存在许多生物安全问题[3]。ASF由俄罗斯蔓延到我国,2018年8月我国首次报告ASF疫情[4-6],之后ASF在我国多地迅速传播,疫情形势严峻复杂,对我国养猪业造成了严重打击。由于ASFV结构复杂且容易变异[7-10],至今还未有有效的ASF疫苗,因此掌握精确的ASF诊断方法显得尤为重要。
【前人研究进展】ASFV是一种有囊膜的双链DNA病毒,也是唯一的虫媒DNA病毒[11]。研究证实,ASFV拥有庞大的基因组,基因组长约160~190 kb,含有约150个开放阅读框,编码160多种蛋白,其中结构蛋白50多种,如编码病毒抗原表位的常见结构蛋白P72(B646L)、P30(CP204L)、P54(E183L) 等[11-13]。ASFV 是通过细胞内化作用进入自然感染的细胞中的,P30抗原蛋白也可能参与了病毒的内化作用而进入细胞中。P30蛋白是由CP204L调控基因编码的磷酸蛋白[13],该蛋白在病毒感染早期,氨基末端丝氨酸残基发生磷酸化后被包进病毒粒子中。由于P30蛋白有良好的免疫原性,在动物体中容易产生较强的免疫应答,产生含量较高的抗体[14-16],因此P30蛋白常被用做血清学诊断。
【本研究切入点】目前诊断和监测ASFV感染猪群的重要方法是血清学检测法。由于ASFV基因组较庞大,通过针对保守性、特异性好的蛋白进行血清学检测,才能更快、更有效、更准确地实现诊断和监测。【拟解决的关键问题】P30蛋白为稳定蛋白,前人研究表明该蛋白是膜相关抗原蛋白,参与病毒内化,具有较好的诊断抗原性。本研究以P30抗原蛋白作为血清学诊断试剂的主要研究蛋白,为后期ASFV感染后提供血清学检验基础。以编码P30蛋白的CP204L保守基因作为研究对象,并通过生物信息学分析,构建表达重组载体及诱导蛋白表达,为P30蛋白的深入研究奠定基础。
1 材料与方法
1.1 试验材料
ASFV基因组由军事医学科学院军事兽医研究所扈荣良实验室惠赠。pET-32a(+)、pEASY-T5 Zero Cloing Kit、2×Rapid Taq Master Mix购于南京诺唯赞生物科技有限公司;E. coli DH5α、Rosetta(DE3)、xHoⅠ、EcoRⅠ、T4 DNA Ligase、Maker购于宝日医生物技术有限公司;基因组提取试剂盒、质粒试剂盒、胶回收试剂盒购于康宁生命科学有限公司。
1.2 基因克隆
根据NCBI中CP204L基因序列(登录号:MN172368.1)设计特异性引物(CP204L-F:CCGGAATTC ATGGATTTTATTTTAAATATATCC,CP204L-R:CCGCTCGAG TTATTTTTTTTTTAAAA GTTTAAT),引物由生工生物工程(上海)股份有限公司长春分公司合成。以ASFV基因组作为模板进行PCR扩增,PCR反应体系(20 μL):10 μL Mix,上下游引物(10 μmol/L)各 1 μL,6 μL ddH2O,2 μL模板;PCR反应程序:95 ℃预变性 3 min;95 ℃变性 15 s、58 ℃退火 15 s、72 ℃延伸15 s,35个循环;72℃后延伸5 min。
1.3 克隆载体构建与鉴定
对PCR扩增出来的目的条带进行切胶,并用胶回收试剂盒进行回收,将回收的PCR产物片段连接pEASY-T5克隆载体,将连接产物转化到E.coli DH5α感受态中并涂布在含有卡那抗性的LB培养基中,37 ℃培养8~10 h后对挑取单菌落摇菌,并进行菌液PCR验证,将验证成功的菌液进行测序比对。
1.4 生物信息学分析
采用TExpasy方法对蛋白质进行理化性质预测;分别采用Tmpred、HMM方法对蛋白质的跨膜区进行预测;采用SignalP方法进行信号肽预测;采用PSIPRED方法进行蛋白质二级结构预测;采用SWISS-MODEL方法进行蛋白质三级结构预测。
1.5 重组表达载体构建与鉴定
用xHoⅠ和EcoRⅠ将pET-32a(+)表达载体和pEASY-T5-CP204L克隆载体分别进行双酶切,经琼脂糖凝胶电泳后切胶,pET-32a(+)载体大片段和目的基因片段用凝胶回收试剂盒回收,回收产物用T4 DNA Ligase在16 ℃下过夜连接。将连接产物转入E.coli DH5α中,在含卡那抗性的LB培养基上37 ℃培养,进行抗性筛选,挑取单菌落进行摇菌提质粒,进行质粒PCR验证、双酶切验证及测序比对验证。
1.6 重组蛋白的诱导表达
将鉴定正确的重组载体pET-32a(+)-CP204L质粒转化至Rosetta(DE3)感受态细胞中,次日挑取单个菌落接种于LB液体培养基中37 ℃过夜培养,次日取200 μL菌液接种于20 mL含氨苄青霉素抗性的液体LB培养基中,加入1.0 mmol/L IPTG诱导8 h,收集菌液。
1.7 重组蛋白的SDS-PAGE电泳鉴定
将收集的菌液在12 000 r/min条件下离心1 min,收集菌体,用PBS冲洗2次后进行超声破碎10 min,期间超声破碎10 s、间歇10 s,重复进行,电压220 V,10 000 r/min离心后分别取上清和沉淀,将沉淀用PBS重悬后重新稀释,上清和沉淀分别进行SDS-PAGE电泳分析。
2 结果与分析
2.1 基因扩增
利用特异性引物进行CP204L基因扩增,经琼脂糖凝胶电泳检测PCR产物,在585 bp处有明亮的条带(图1),结果与预期相符。
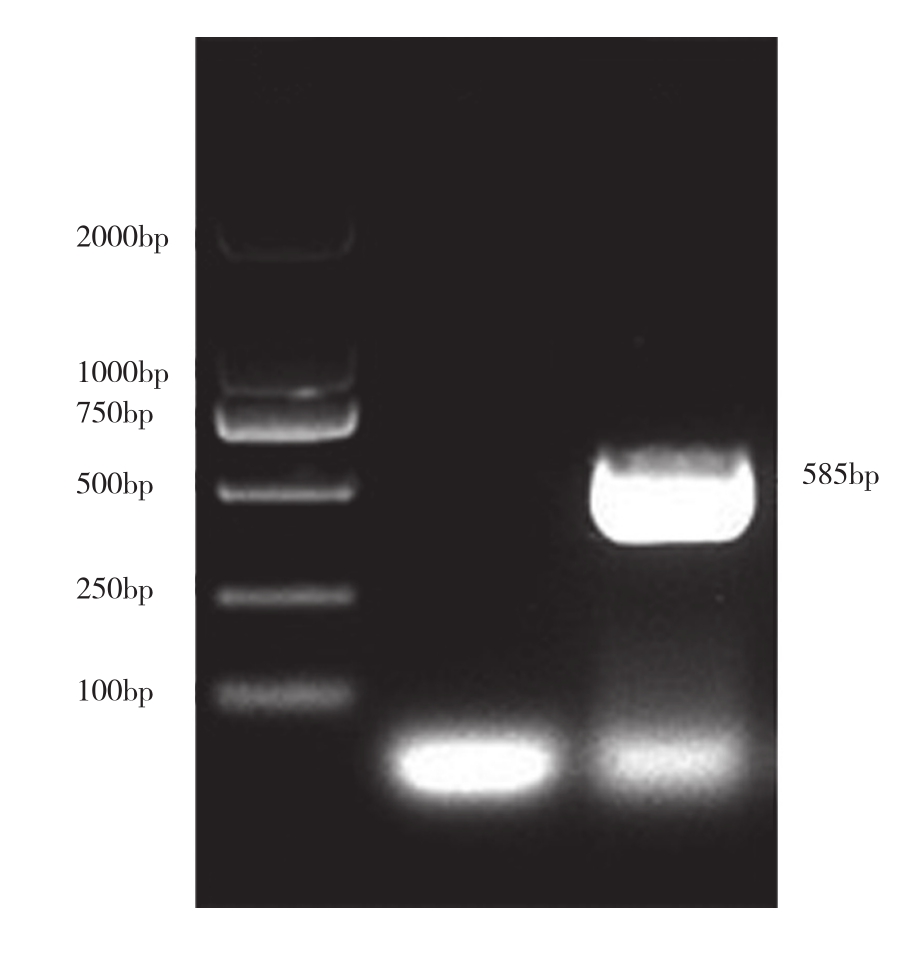
图1 CP204L基因克隆结果
Fig. 1 Cloning result of CP204L gene
M:DL2000;1:阴性对照;2:PCR产物M: DL2000; 1: Negative control; 2: PCR production
2.2 克隆载体构建及鉴定

图2 菌液PCR验证
Fig. 2 PCR verification of bacterial solution
M:DL2000 ;1:阴性对照;2:PCR产物M:DL2000 ; 1:Negative control; 2: PCR production
将目的基因PCR产物用DNA产物凝胶回收试剂盒回收,将目的基因片段与pEASY-T5载体连接并转化,挑取单菌落扩大培养,进行菌液PCR验证,将图2目的条带的菌液送往上海生工有限公司测序。使用DNAMAN软件对测序结果与CP204L基因序列进行比对分析,结果显示测序结果与CP204L基因序列同源性为100%,证明CP204L基因与克隆载体连接成功。
2.3 生物信息学分析
2.3.1 蛋白理化性质 TExpasy在线预测分析结果显示,CP204L基因编码195个氨基酸,序列如下:
MDFILNISMK MEVIFKTDLR
SSSQVVFHAG SLYNWFSVEI
INSGRIVTTA IKTLLSTVKY
DIVKSARIYA GQGYTEHQAQ
EEWNMILHVL FEEETESSAS
SENIHEKNDN ETNECTSSFE
TLFEQEPSSE VPKDSKLYML
AQKTVQHIEQ YGKAPDFNKV
IRAHNFIQTI YGTPLKEEEK
EVVRLMVIKL LKKK*
P30蛋白不稳定指数为34.12,为稳定蛋白;总亲水性平均值为0.739,为亲水蛋白。
2.3.2 蛋白质跨膜区预测 基于对Tmbase数据库的统计分析预测P30蛋白的跨膜区和跨膜方向,用Tmpred法分析蛋白质跨膜区,结果(图3)显示,P30蛋白TM-螺旋长度都在17~33之间;基于HMM方法预测蛋白质跨膜区,图4显示TMH数量为0,P30蛋白为非跨膜蛋白。
2.3.3 蛋白质信号肽预测 采用SignalP在线软件对P30蛋白进行信号肽预测,结果(图5)显示,基因编码的P30蛋白在编码区内均无有信号肽信号,说明该蛋白不是分泌性蛋白。

图3 Tmpred法蛋白跨膜域分析
Fig. 3 Transmembrane domain analysis of protein by Tmpred method
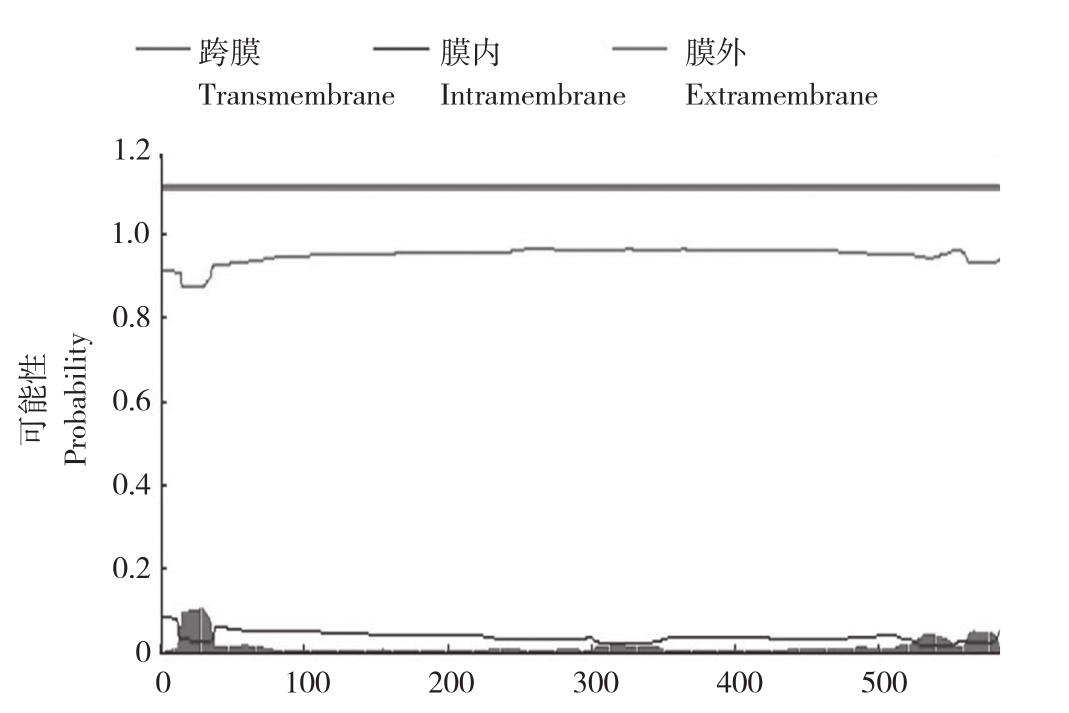
图4 HMM法蛋白跨膜域分析
Fig. 4 Transmembrane domain analysis of protein by TMM method
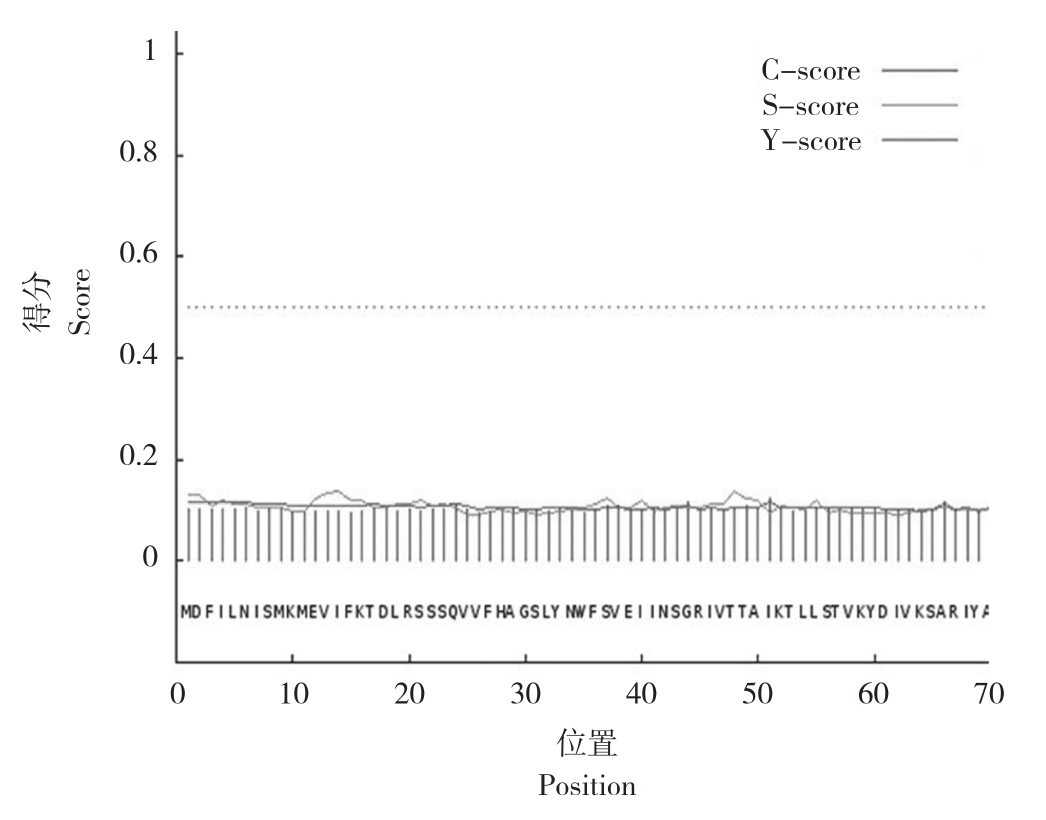
图5 P30蛋白信号肽分析
Fig. 5 Analysis of protein signal peptide
2.3.4 蛋白质二级结构预测 PSIPRED法在线预测分析P30蛋白的二级结构,结果(图6)显示,该蛋白主要结构由无规卷曲、α螺旋和β折叠组成。
2.3.5 蛋白质三级结构预测 通过SWISSMODEL在线预测P30蛋白质同源建模结构(图7),无规则卷曲、α螺旋和β折叠是三级结构的主要成分,与二级结构预测结果相符。
2.4 重组表达载体的构建及鉴定
用EcoRⅠ、xHoⅠ酶同时将构建好的克隆载体和表达载体pET-32a(+)质粒进行双酶切验证,经1%琼脂糖凝胶电泳,结果(图8、图9)显示,酶切产物可获得目的基因片段和载体片段,将基因片段和线性载体大片段用琼脂糖凝胶回收试剂盒进行切胶回收。

图6 蛋白质二级结构分析
Fig. 6 Analysis of the secondary structure of protein
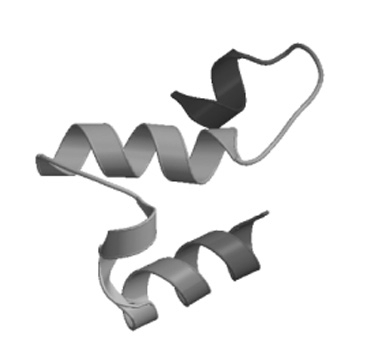
图7 蛋白质三级结构预测结果
Fig. 7 Prediction result of the tertiary structure of protein
用T4连接酶连接目的基因CP204L与pET-32a(+)载体大片段构建重组表达载体pET-32a(+)-CP204L并转化到大肠杆菌,对转化后的单菌落进行菌液PCR验证,对验证成功的提取pET-32a(+)-CP204L质粒进行测序,测序结果与目的基因序列一致,证明重组表达载体pET-32a(+)-CP204L构建成功(图10、图11)。
2.5 P30蛋白的诱导表达及SDS-PAGE检测
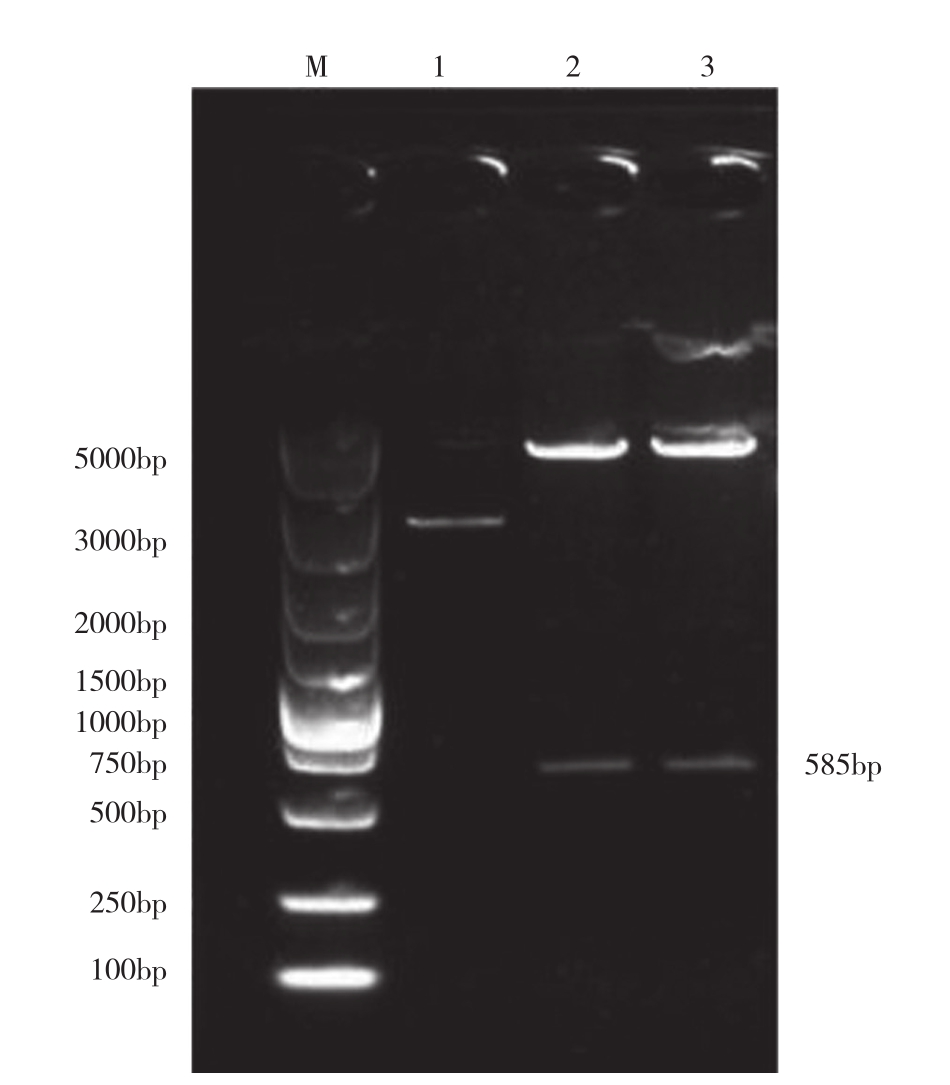
图8 CP204L基因克隆载体质粒双酶切结果
Fig. 8 Cloning vector plasmid of CP204L gene verified by double digestion
M:DL2000;1:克隆载体质粒;2~3:双酶切产物
M: DL2000; 1: Cloning vector plasmid; 2-3: Production of double digestion
重组原核表达质粒pET-32a(+)-CP204L转化大肠杆菌Rosetta(DE3)感受态细胞,37℃经IPTG诱导后菌体超声破碎,上清和沉淀进行SDS-PAGE电泳。由图12可知,pET-32a(+)-CP204L以可溶形式在宿主菌中未表达,以质溶形式在沉淀中有表达。蛋白分子质量约为23.4 ku,与理论值相符。
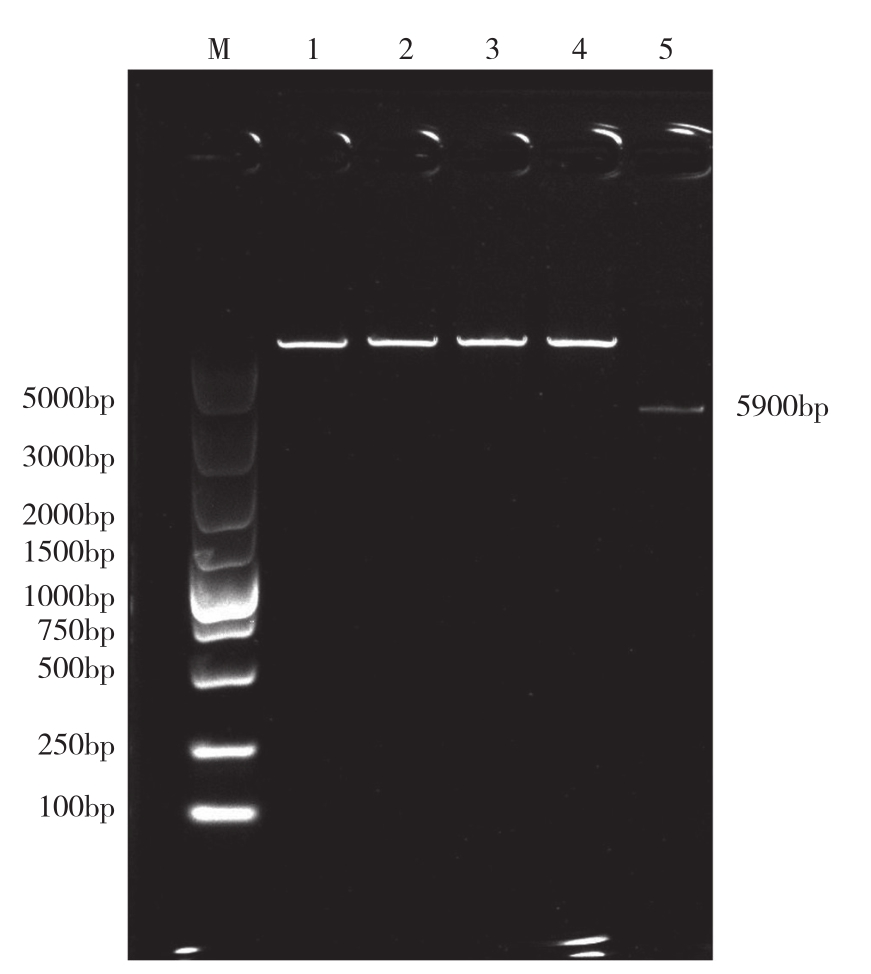
图9 pET-32a(+)表达载体质粒双酶切结果
Fig. 9 Identification of expression vector plasmid pET-32a(+)by double digestion
M:DL2000;1:质粒;2~5:双酶切产物
M:DL2000;1: Expression vector plasmid; 2-5: Production of double digestion
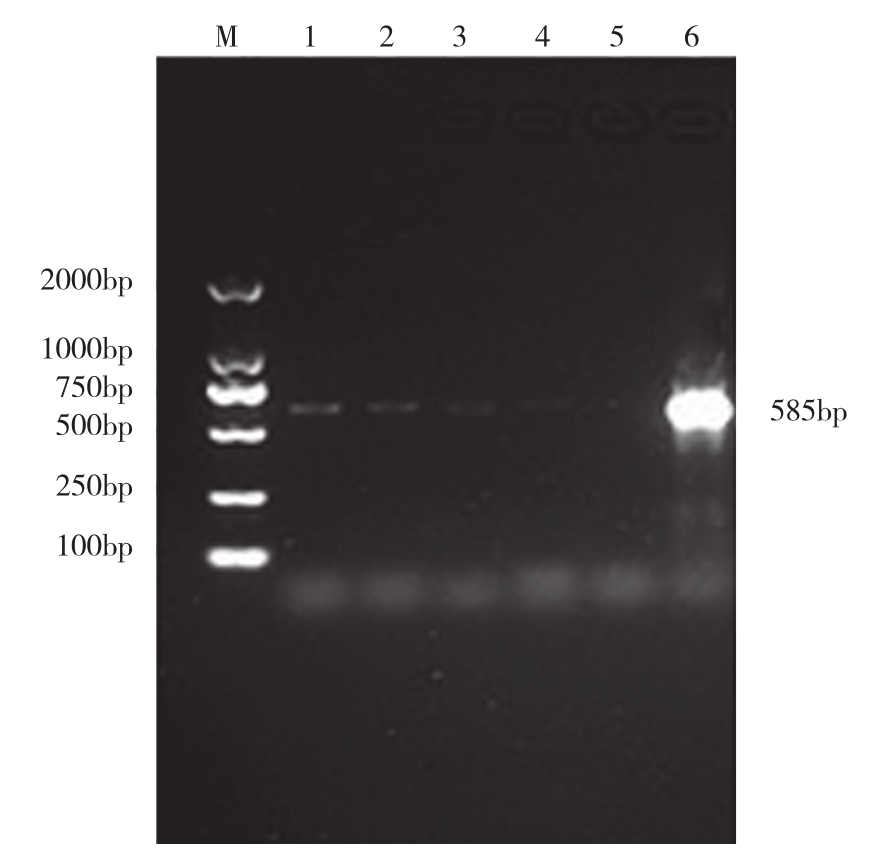
图10 菌液PCR验证
Fig. 10 PCR verification of bacterial solution
M:DL2000;1~5:PCR产物;6:阳性对照M: DL2000; 1-5: PCR production; 6: Positive control
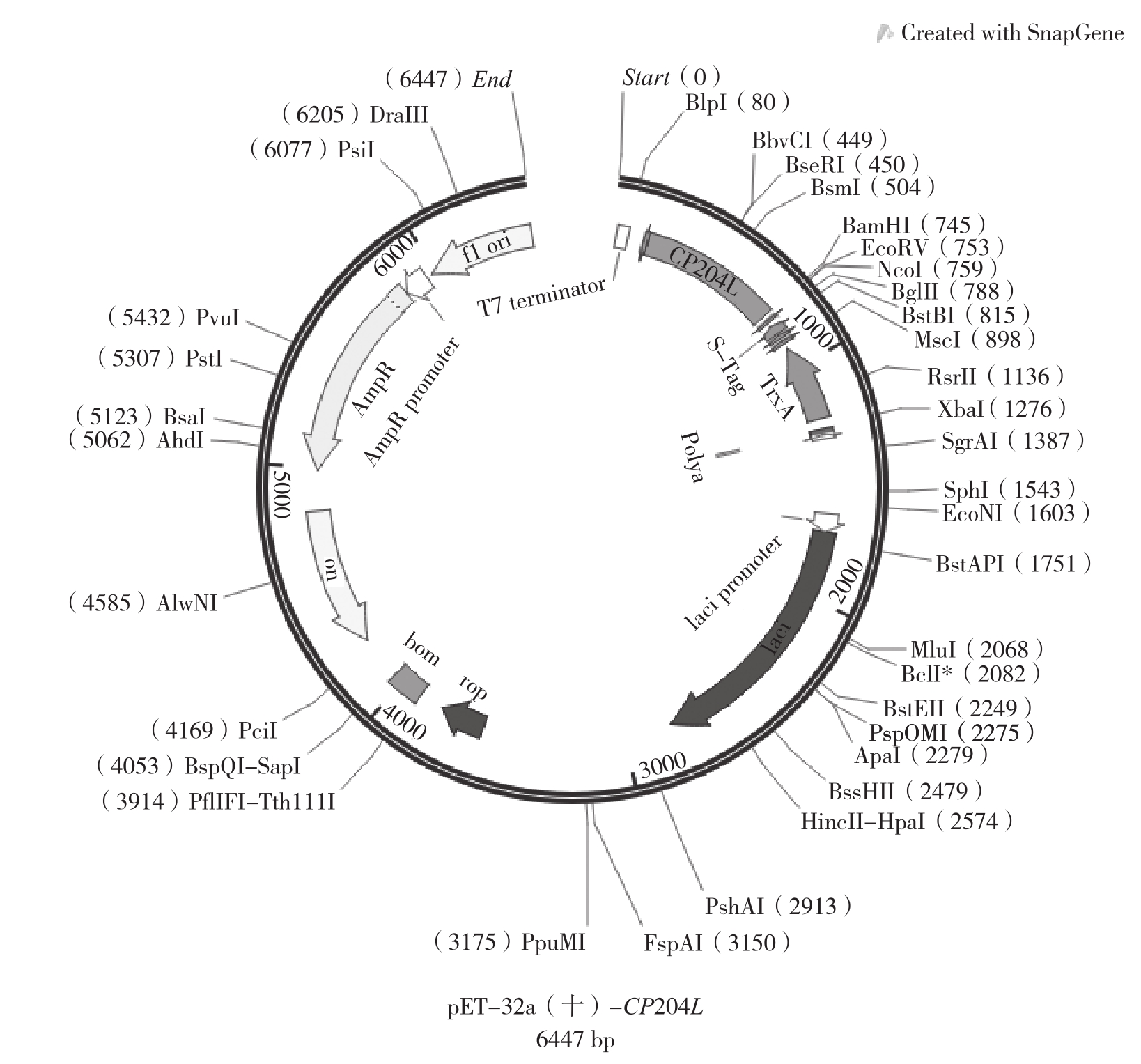
图11 pET-32a(+)-CP204L重组表达载体示意图
Fig. 11 Schematic diagram of pET-32a(+)-CP204L recombinant expression vector
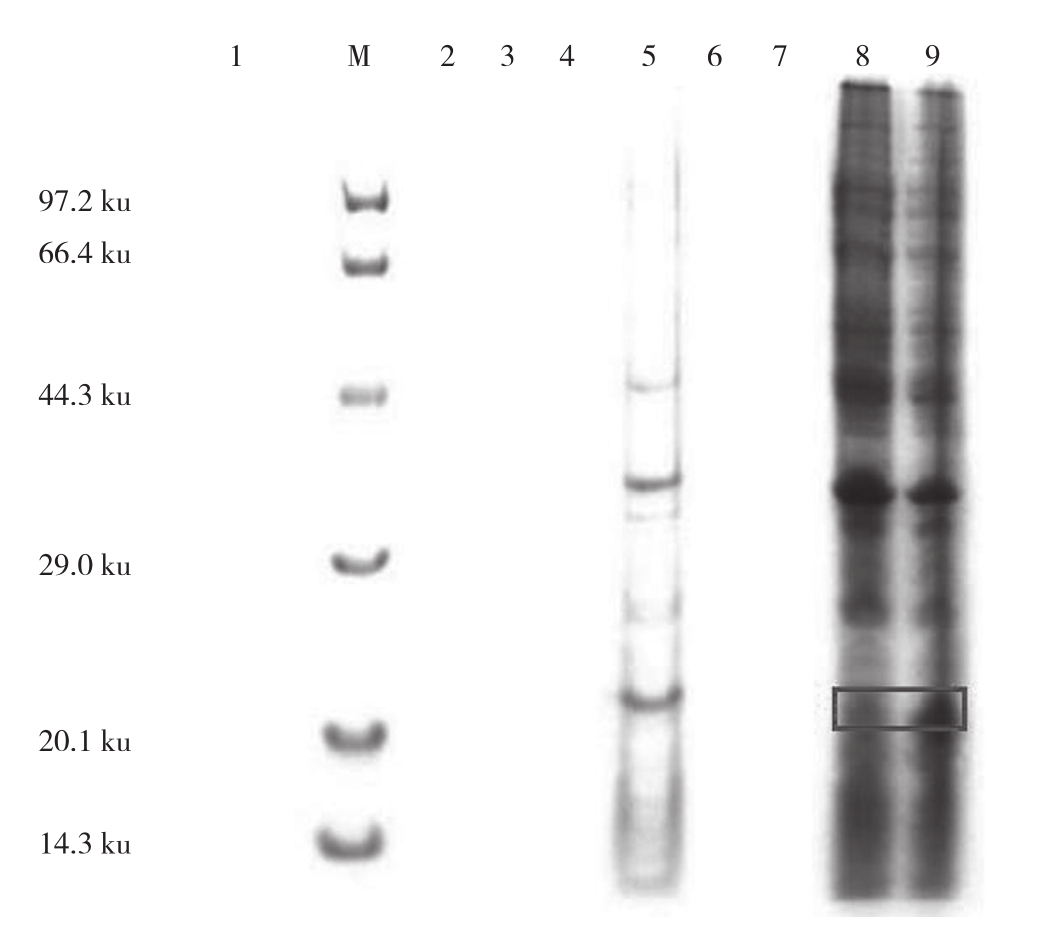
图12 P30蛋白SDS-PAGE检测结果
Fig. 12 SDS-PAGE detection results of P30 protein
M: DL2000;1: pET-32a(+)诱导后上清;2: pET-32a(+)上清;3: pET-32a(+)沉淀;4: pET-32a(+)诱导后沉淀;5: pET-32a(+)-CP204L诱导后上清;6: pET-32a(+)-CP204L诱导前上清;7: pET-32a(+)-CP204L诱导前沉淀;8~9: pET-32a(+)-CP204L诱导后沉淀
M: DL2000; 1: pET-32a(+)supernatant after induction; 2: pET-32a(+)supernatant; 3: pET-32a(+)precipitation; 4: pET -32a(+)precipitation after induction; 5: pET-32a(+)-CP204L supernatant after induction; 6: pET-32a(+)-CP204L supernatant before induction;7: pET-32a(+)-CP204L precipitation before induction; 8-9: pET-32a(+)-CP204L precipitation after induction
3 讨论
ASF是一类发病机理和临床症状极为复杂的传染病,如不能及早发现和实施严格控制措施会迅速蔓延和持续传播,将对社会造成严重经济损失[17-18,22]。ASF可根据流行病学、临床症状和病理特点作出初步判断,但最终需根据实验室诊断结果进行确诊。虽然目前市场上没有有效的疫苗,国内外的研究人员也从未停止对非洲猪瘟的研究。吴竞等[19]通过P30蛋白的原核表达建立了间接ELISA检测方法;Alexandra等[20]对P30基因进行CRISPR/Cas9靶向编辑后,有效抑制了ASFV病毒的复制;李杰等[5]将ASFV的P30-54融合蛋白在杆状病毒表达系统中表达,该重组蛋白可以与多克隆抗体发生免疫学反应;杨莎莎等[22]制备了非洲猪瘟pET-28a-P30单克隆抗体,为ASFV的诊断和致病机制研究奠定基础。
前人诸多研究表明,P30蛋白是ASFV中的关键蛋白,具有良好的反应原性,吴竞等[19]建立的ELISA检测方法表明,克隆和表达P30蛋白可以用来监测ASF疫情;Alexandra等[20]对P30蛋白重新编辑之后,抑制了ASFV蛋白的表达,因此可以通过调控P30蛋白来控制ASFV病毒的致病性;杨莎莎等[21]选用Georgia2007/1毒株基因组也成功构建了单克隆抗体,更能判定P30蛋白的表达在建立间接ELISA方法应用于监测疫情是可行的,而且非常重要。前人的研究缺少在生物信息学方面进行蛋白的分析,结合蛋白层面的分析能够更好地了解蛋白及分析蛋白功能,这些对后期研究具有重要意义。本试验对ASFV调控蛋白P30进行了研究,在生物信息学层面对CAS19-01株病毒中的CP204L基因组进行剖析,在此基础上构建表达载体并诱导其成功表达P30蛋白,为制备检测试剂盒做好准备。
4 结论
ASFV基因组可编码的蛋白非常多,而且ASFV的免疫逃避机制复杂,使得非洲猪瘟疫苗的研发工作受阻。未来需要继续深入研究ASFV的病原学、致病机理和免疫机制,才能更好地应对疫情发生。目前非洲猪瘟疫苗开始进入临床阶段,对疫病的抗体检测迫在眉睫,为了加快自主研发的步伐,尽快研制出高效且灵敏的抗体检测试剂盒尤为关键。本研究根据CAS19-01株病毒基因组成功克隆了在ASFV中稳定存在、保守的CP204L基因,并成功构建其表达载体pET-32a(+)-CP204L,通过用IPTG诱导8 h后成功表达蛋白,为制备ASF的抗体检测试剂提供技术基础。
[1] 罗忠永 . CSFV、HP-PRRSV、PEDV和 ASFV等病原GeXP多重检测方法的建立和试剂盒的初步组装[D].成都:四川农业大学,2017.doi:cnki:cdmd:2.1018.071351.LUO Z Y. CSFV, HP-PRRSV, PEDV, ASFV and other pathogen GeGe multiple detection method establishment and preliminary assembly of the kit[D]. Chengdu: Sichuan Agricultural University, 2017..doi:cnki:cdmd:2.1018.071351.
[2] 唐迪,刘迪,刘林,王众,郑宁,郑业鲁.非洲猪瘟检测技术研究进展[J].广 东 农 业 科 学,2019,46(11):104-111.doi:10.16768/j.issn.1004-874X.2019.11.015.TANG D, LIU D, LIU L, WANG Z, ZHENG N, ZENG Y L. Research progress of African swine fever detection technology[J]. Guangdong Agricultural Sciences, 2019,46(11):104-111.doi:10.16768/j.issn.1004-874X.2019.11.015.
[3] 张艳艳,陈腾,张静远,齐宇,缪发明,薄宗义,王立冬,郭晓宇,周鑫韬,杨金梅,王晓虎,高玉龙,汪春雨,鲍晨沂,米立娟,孙雪霏,冯娜,杨金金,王承宇,万忠海,李记平,孟轲音,田多,李楠,王述超,王颖,张菲,张锦霞,蒋依倩,刘晔,张守峰,张中洋,钱莺娟,朱鸿飞,高玉伟,陈鸿军,李金祥,扈荣良. 非洲猪瘟病毒基因缺失疫苗株的构建和免疫保护特性[J]. 中国兽医科学,2019,39(8):1421-1427.doi:10.16303/j.cnki.1005-4545.2019.08.01.ZHANG Y Y, CHEN T, ZHANG J Y, QI Y, MIAO F M, BO Z Y, WANG L D, GUO X Y, ZHOU X T, YANG J M, WANG X H, GAO Y L, WANG C Y, BAO C Y, MI L J, SUN X F, FENG N, YANG J J, WANG C Y,WAN Z H, LI J P, MENG K Y, TIAN D, LI N, WANG S C, WANG Y,ZHANG F, ZHANG J X, JIANG Y Q, LIU Y, ZHANG S F, ZHANG Z Y,QIAN Y J, ZHU H F, GAO Y W, CHEN H J, LI J X, HU R L. African swine fever virus gene deletion construction of vaccine strains and immune protection characteristics[J]. Chinese Veterinary Science,2019, 39(8): 1421-1427.doi:10.16303/j.cnki.1005-4545.2019.08.01.
[4] 赵亚茹,韩蓉,杨吉飞,牛庆丽,关贵全,罗建勋,刘光远,殷宏,GREGORZ Wo niakowski,刘志杰. 非洲猪瘟病毒K205R 蛋白单克隆抗体的制备与鉴定[J]. 中国兽医科学,2019,49(7):805-811.doi:10.16656/j.issn.1673-4696.2019.0092.ZHAO Y R, HAN R, YANG J F, NIU Q L, GUAN G Q, LUO J X, LIU G Y, YIN H, GREGORZ W N, LIU Z J. Preparation and identification of monoclonal antibody against African swine fever virus K205R protein[J]. Chinese Veterinary Science, 2019, 49(7):805-811. doi:10.16656/j.issn.1673-4696.2019.0092.
[5] 李杰,张星星,郭晶,孟庆玲,乔军,张国武,王晓婷,李妍,才学鹏. 非洲猪瘟病毒p30-54融合蛋白基因的构建、表达及其多克隆抗体制备[J]. 河南农业科学,2018,7(9): 126-130. doi:10.15933/j.cnki.1004-3268.2018.09.021.LI J, ZHANG X X, GUO J, MENG Q L, QIAO J, ZHANG G W, WANG X T, LI Y, CAI X P. Construction, expression and polyclonal antibody preparation of the African swine fever virus p30-54 fusion protein gene[J]. Henan Agricultural Sciences, 2018, 7(9): 126-130. doi:10.15933 / j.cnki.1004-3268.2018.09.021.
[6] ZHOU X T, LI N, LUO Y Z, LIU Y, MIAO F M, CHEN T, ZHANG S F,CAO P L, LI X D, TIAN K G, QIU H J, HU R L. Emergence of African swine fever in China[J]. Transboundary & Emerging Diseases,2018,65(6):1482-1484. doi: 10.1111/tbed.12989.
[7] 余杞强.非洲猪瘟的疫情分布及其防制[J]. 今日畜牧兽医,2019(6):30.YU Q Q. The distribution and control of African swine fever epidemic[J]. Today Animal Husbandry and Veterinary Medicine, 2019(6):30.
[8] TARIQ H, ANETTE B, STEN M, HANNE C, NILS T. AnetteBoklund.Simulating the epidemiological and economic effects of an African swine fever epidemic in industrialized swine populations[J]. Veterinary Microbiology, 2016, 193:7-16.doi:10.1016/j.vetmic.2016.08.004.
[9] WANG T, SUN Y, QIU H J. African swine fever: an unprecedented disaster and challenge to China[J]. Infectious Diseases of Poverty Volume, 2018,111(7):1-5. doi:10.1186/s40249-018-0495-3.
[10] LI X D, TIAN K G. African swine fever in China[J]. Veterinary Record, 2018,183(9):300-301. doi:10.1136/vr.k3774.
[11] 王彩霞,冯春燕,杜方原,刘丹丹,张永宁,林祥梅,吴绍强. 非洲猪瘟病毒主要抗原p54-1的原核表达及多克隆抗体的制备与鉴 定[J]. 中国 畜 牧兽 医,2018, 45(10):179-186. doi:10.16431/j.cnki.1671-7236.2018.10.019.WANG C X, FENG C Y, DU F Y, LIU D D, ZHANG Y N, LIN X M, WU S Q. Prokaryotic expression of the major antigen of African swine fever virus p54-1 and preparation and identification of polyclonal antibodies[J]. Chinese Journal of Animal Husbandry and Veterinary Medicine, 2018, 45(10):179-186. doi:10.16431/j.cnki.1671-7236.2018.10.019.
[12] KOLLNBERGER S G B,OSTER-CUEVAS M,ME C A P.Identificationof the principal serological immunodeterminants of African swinefever virus by screening a virus cDNA library with antibody[J]. Journal of General Virology, 2002, 3(6):1331 -1342.doi:10.1099/0022-1317-83-6-1331.
[13] 曹琛福. 基于非洲猪瘟病毒P54蛋白抗原表位的ELISA检测方法建立及应用[D]. 广州:华南农业大学,2016.CAO C F. Establishment and application of ELISA detection method based on the epitope of African swine fever virus P54 protein[D].Guangzhou: South China Agricultural University, 2016.
[14] 张会雷,刘任强,张敏敏,王喜军,葛金英,温志远,步志高. 非洲猪瘟病毒结构蛋白基因重组痘苗病毒的构建及鉴定[J]. 中国预防兽医学报,2017,10(9):789-793. doi: 10.3969/j.issn.1008-0425.201703042.ZHANG H L, LIU R Q, ZHANG M M, WANG X J, GE J Y, WEN Z Y, BU Z G. Construction and identification of recombinant vaccinia virus of African swine fever virus structural protein gene[J]. Chinese Journal of Preventive Veterinary Medicine, 2017, 10(9): 789-793.doi: 10.3969/j.issn.1008-0425.201703042.
[15] COBBOLD C, WINDSOR M, WILEMAN T. A virally encoded chaperone specialized for folding of the major capsid protein of African swine fever virus[J]. Journal of Virology, 2001, 75(16):7221-7229.doi: 10.1128/JVI.75.16.7221-7229.2001.
[16] 张鑫宇.非洲猪癌病毒实验室诊断技术研究及试剂储备[D].扬州:扬州大学,2015.ZHANG X Y. Research on diagnostic technology and reagent reserve of African swine carcinoma virus laboratory[D]. Yangzhou: Yangzhou University, 2015.
[17] 温肖会,魏文康,黄忠,吕殿红.非洲猪瘟的研究进展[J]. 广东农业科学,2010,37(10):137-140.doi:10.16768/j.issn.1004-874x.2010.10.055.WEN X H,WEI W K, HUANG Z, LYU D H. Research progress of African swine fever[J]. Guangdong Agricultural Sciences, 2010,37(10):137-140..doi:10.16768/j.issn.1004-874x.2010.10.055.
[18] 康桦华,移兰丽,王片片,陈兴勃,彭新宇,向蓉,梅盈洁,徐志宏.广东屠宰行业现状及非洲猪瘟后的发展对策[J]. 广东农业科学,2019,46(7):130-137.doi:10.16768/j.issn.1004-874X.2019.07.019.KANG H H, YI L L, WANG P P, CHEN X B, PENG X Y, XIANG R, MEI Y J, XU Z H. Current status of the slaughter industry in Guangdong and development strategies after African swine fever[J]. Guangdong Agricultural Sciences, 2019,46(7):130-137. doi:10.16768/j.issn.1004-874X.2019.07.019.
[19] 吴竞,王西西,吴映彤,任肖,郭晓宇. 非洲猪瘟病毒p30基因的原核表达及间接ELISA抗体检测方法的建立[J]. 中国畜牧兽医,2018, 45(12):3555-3562.doi:10.16431/j.cnki.1671-7236.2018.12.029.WU J, WANG X X, WU Y T, REN X, GUO X Y. Prokaryotic expression of p30 gene of African swine fever virus and establishment of indirect ELISA antibody detection method[J]. Chinese Journal of Animal Husbandry and Veterinary Medicine, 2018, 45(12):3555-3562.doi:10.16431/j.cnki.1671-7236.2018.12.029.
[20] ALEXANDRA H, BJOERN P, GÜNTHER M K, HEINER N,THOMAS C,METTENL E, WALTER F. Efficient inhibition of African swine fever virus replication by CRISPR/Cas9 targeting of the viral p30 gene(CP204L)[J]. Scientific Reports, 2018, 8(1):1449-1456.doi:10.1038/s41598-018-19626-1.
[21] 杨莎莎,苗雨润,薄宗义,王振忠,谭凯,鲍晨沂,吴晓东,陈鸿军,郑龙三,钱莺娟,戴建君.非洲猪瘟病毒p30单克隆抗体的制备和鉴定[J]. 中国兽医科学,2020(12):1-7. doi:10.16656/j.issn.1673-4696.2020.0117.YANG S S, MIAO Y R, BO Z Y, WANG Z Z, TAN K, BAO C Y, WU X D, CHEN H J, ZHENG L S, QIAN Y J, DAI J J. Preparation and identification of p30 monoclonal antibody against African swine fever virus[J]. China Veterinary Science, 2020(12): 1-7. doi:10.16656/j.issn.1673-4696.2020.0117.
[22] 张险朋.非洲猪瘟实验室检测技术研究进展[J]. 畜牧兽医科技信息,2019(7):18-20.ZHANG X P. Research progress of African swine fever laboratory detection technology[J]. Animal Husbandry and Veterinary Science and Technology Information, 2019(7):18-20.