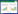文章信息
基金项目
- 中央财政林业科技推广示范资金项目(2015GDTK-02)
作者简介
- 徐放(1987—),男,博士,助理研究员,研究方向为林木群体与数量遗传学,E-mail:xufang@sinogaf.cn.
文章历史
- 收稿日期:2020-11-09
2. 广州市增城区林业和园林科学研究所,广东 广州 511300;
3. 棕榈生态城镇发展股份有限公司,广东 广州 510627
2. Guangzhou Zengcheng Forestry and Landscape Science Research Institute, Guangzhou 511300, China;
3. Zonglv Eco-town Development Co., Ltd. Guangzhou 510627, China
【研究意义】木兰科(Magnoliaceae)含笑属(Michelia L.)有80余种植物,主要分布于亚洲热带和亚热带地区。该属树种树型优美、香气怡人,被广泛应用于园林绿化中,且兼具材用、药用和油脂用等用途,具有广阔的应用前景[1-5]。含笑属种间生殖隔离较弱,表型变异丰富,部分植物的分类和亲缘关系在学术界仍有争议[6],急需在含笑属中开展亲缘关系研究,明晰各种间遗传关系,为后续开展种间杂交和优良新品种选育提供有力的指导。【前人研究进展】随着分子标记技术的发展,分子标记已经在含笑属植物遗传多样性和亲缘关系研究中得到应用。王艇等[7]首次利用RAPD标记开展了含笑属中4个含笑亚属的系统发育研究。王亚玲等[8]利用trnL内含子及trnL-trnF间隔区序列对多个含笑属植物进行了亲缘关系分析,发现其中一个简单重复序列的插入可以作为系统发育分析的依据之一。滕花景[9]筛选了19个RAPD标记对乐昌含笑(Michelia chapensis)6个群体进行聚类分析。柴弋霞[10]开发ISSR标记对紫花含笑(M. crassipes)及其与近缘种的杂交子代进行了亲缘关系分析,发现子代分子标记分型结果与形态学标记一致。李剑等[11]将AFLP和ISSR标记共同用于含笑属植物的亲缘关系分析,初步得出7种进化历程。王猛等[12]利用10个ISSR分子标记对云南省内的48种野生木兰科植物进行亲缘关系初步分析,并划分为7个类群,其中含笑属17个种被划分到6个不同类群中,该研究发现基于分子标记的聚类结果与传统分类学存在一定的差异。温强等[13]利用ISSR标记对3个墨紫含笑新品种和4个近缘种进行了亲缘关系的评价。【本研究切入点】目前本研究供试的广东含笑等11个含笑属物种表型多样,亲缘关系尚不清楚,急需通过稳定的分子标记对不同物种进行基因分型和亲缘关系分析。【拟解决的关键问题】本研究在收集含笑属资源的基础上,利用基于M13荧光标记的12对SSR引物获取11个含笑属物种的基因型,并对不同物种的亲缘关系进行分析,计算不同物种间的遗传距离,旨在建立一个高效的含笑属植物的基因分型体系,为含笑属的保护和开发利用提供技术支持。
1 材料与方法 1.1 试验材料与DNA提取供试的11个含笑属物种采自广东省林业科学研究院种质资源圃(表 1),分别编号M01~M11,采集生长良好的叶片,液氮速冻后放入-80℃超低温冰箱保存。选用0.2~0.3 g叶片利用震荡研磨仪破碎,采用改良CTAB法[14]进行DNA提取,利用Nanodrop2000(Thermo,美国)核酸检测仪检测DNA浓度,稀释至50 ng/μL备用。
1.2 引物筛选
参照Sun等[15]和李红英等[16]论文中的引物信息合成40对引物,稀释至10 μmol/L备用,选择11个物种的DNA进行PCR扩增,PCR反应体系为20 μL,包含DNA模板2 μL、正反引物各0.4 μL、PCR Mix(上海生工,上海)10 μL和ddH2O 7.2 μL。扩增反应程序为94℃预变性5 min;94℃变性40 s、56℃退火45 s、72℃延伸45 s,35个循环;72℃延伸5 min。产物利用2% 琼脂糖凝胶电泳进行检测。选取扩增成功率90% 以上的引物共计12对,用于后续试验以获取各物种的基因型。
1.3 基因分型体系建立选用基于M13序列的荧光标记方法获取基因型[17],在M13序列TGTAAAACGACGGCCAGT的3′端分别添加6-FAM、HEX、TAMRA和ROX作为荧光标记引物,12个标记的正引物进行重新合成,在正引物3′端添加M13序列,其序列见表 2。基因分型PCR反应体系为20 μL,包含DNA模板2 μL、M13标记0.4 μL、正引物0.15 μL、反引物0.4 μL、PCR Mix 10 μL和ddH2O 7.05 μL。扩增反应程序为94 ℃预变性5 min;94℃变性40 s、56℃退火45 s、72℃延伸45 s,30个循环;94℃变性40 s、53℃退火45 s、72℃延伸45 s,8个循环;72℃延伸5 min。产物利用ABI3730测序仪进行毛细管电泳检测,使用LIZ-500作为内标。
1.4 数据分析
利用GeneMarker v 2.05软件[18]对基因分型的结果进行判读,获取原始片段长度数据后,利用Excel的宏文件Flexibin进行数据整理。利用GenAlex6.5[19]计算等位基因数(Na)、有效等位基因数(Ne)、Shannon多样性指数(I)和表观杂合度(Ho)。利用Cervus[20]计算多态性信息含量(PIC)。获取各物种的基因型数据后,利用DARwin软件[21]进行不同物种间的亲缘关系分析,构建基于非加权组平均法(Unweighted pair-group method with arithmetic means,UPGMA)的Neighbor-Joining(NJ)聚类图。
2 结果与分析 2.1 引物的种间通用性利用40对引物对11个物种进行扩增的结果显示,不同引物扩增成功率在40%~100% 之间,总体扩增成功率为66.5%。2% 琼脂糖电泳显示无非特异产物的引物共计12对,PCR产物片段长度均在350 bp以下(图 1),其中5对引物可以在全部11个物种中高效扩增(表 3),12对引物扩增成功率从72.7%~100% 不等、平均为90.9%,以上12对引物用于本研究的后续试验。
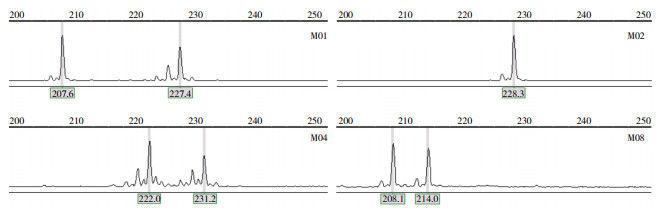
|
| 图 1 标记SGA15在4个含笑属物种中的扩增产物长度 Fig. 1 PCR amplification product length of marker SGA15 in four Michelia species |
2.2 遗传多样性分析
获取11个物种的基因型后开展遗传多样性分析,结果见表 4。位点LT106扩增获得的等位基因数最多、为12个,位点LT47扩增获得的等位基因数最少、仅4个。12个标记的有效等位基因数的变化范围为2.909~10.083;Shannon多样性指数(I)为1.303~2.437;观测杂合度(Ho)最小为0、最大为0.818;期望杂合度(He)变化区间为0. 656~0.901,多态信息含量(PIC)为0.671~0.880。
2.3 11个含笑属物种的亲缘关系分析
DARwin软件获得的NJ聚类图如图 2所示,由图 2可知,11个物种被划分成3个不同的重要分支,其中第一分支的物种为广东含笑(M. guangdongensis)、金叶含笑(M. foveolata)、醉香含笑(M. macclurei)、石碌含笑(M. shiluensis)和苦梓含笑(M. balansae);第二分支包括2个物种,分别为深山含笑(M. Maudiae)和含笑(M. figo);第三个分支的4个物种为白兰(M. alba)、黄兰(M. champaca)、紫花含笑(M. crassipes)和乐昌含笑(M. chapensis)。第一分支和第二分支上的7个物种均属于后生含笑亚属,而第三个分支上的4个物种中,乐昌含笑属于后生含笑亚属,另外3个物种属于含笑亚属。

|
| 图 2 11个含笑属物种的Neighbor-Joining聚类结果 Fig. 2 Neighbor-Joining tree of 11 Michelia species |
3 讨论
SSR标记广泛分布于真核生物的基因组中,作为共显性标记,相比于传统的RADP、ISSR和AFLP等显性标记类型,可以更准确地获得生物遗传信息。由于SSR标记具有高度变异、操作简单、重复性好、准确率高等优点,因此可以作为物种分类、种质资源评价、杂交亲本选择等遗传分析的重要工具。李清莹[22]在醉香含笑中开发了SSR标记,并对醉香含笑的种质资源进行了遗传多样性研究,结合生长表型评选出优良种源和家系。郑纪伟等[6]利用13个SSR标记对8种含笑进行了种质指纹图谱的构建。另外,在木兰科鹅掌楸(Liriodendron chinense)[23-25]中分别进行了SSR引物开发和杂交种判别和自然条件下物种渐渗等研究,为鹅掌楸的新品种选育提供了重要指导。以上研究均表明SSR标记可在遗传分析中获得良好效果。本研究中选用的SSR标记多态性好,平均PIC值达到0.769,扩增成功率较高、达90.9%,且100% 位点具有多态性,相比于近年来前人在十字花科(Brassicaceae)[26]、榛属(Corylus)[27]和玉叶金花属(Mussaenda)[28]中的亲缘关系分析研究,本研究中引物扩增成功率和多态性均有显著提升,以上数据表明应用该12组引物可以较为准确地反映试验材料的遗传信息,可以作为后续杂交亲本筛选和杂交子代判定的重要工具。本研究中选用了经济性较好的基于M13序列的荧光标记方法,相比于传统的聚丙烯酰胺(PAGE)检测,可显著提升实验效率,并实现了试验结果的数字化读取,避免了人工读取造成的较大误差。另外,与在正引物的3′端直接进行荧光标记的试验方法相比,本试验中采用的方法可以使总体试验成本降低40% 以上。
木兰科中属种的划分,因不同物种的性状相似性较高,一直是分类学家争论的热点,而含笑属的系统分类更是争论的焦点[3]。含笑属植物在属下等级的划分经历了较长的发展历史,但仍存在较大分歧,目前主要有刘玉壶[29]及Chen等[30]提出的两套不同分类系统。本研究将11个含笑属物种进行了聚类,其中广东含笑、金叶含笑和醉香含笑的亲缘关系较近,Yan等[31]于2004年首次报道该物种时,根据其叶片、花瓣和花蕊形态和发现地的地理环境等,认为广东含笑与金叶含笑和醉香含笑的亲缘关系最为接近,有可能为自然条件下二者的变种或杂交种,本研究支持了相关结论。另外,本研究可以将11个物种分属两个亚属的差异体现出来,证明本研究开发的基因分型体系准确性较高。目前,含笑属新品种的杂交选育研究逐步兴起,如中南林业科技大学通过种间杂交获得多个含笑的新品种,并获得国家林业局的新品种授权[32]。王晶等[33]利用金叶含笑和广东含笑进行杂交,选育出矮化、多花且花色特异的新品种。随着含笑属物种和新品种的逐渐增加,为了更加准确地进行含笑属物种的分类和亲本溯源,可以使用本研究开发的分子标记,结合形态学、细胞生物学等多种方法进行分析,从而进行更加准确的亲缘关系划分,为后续的含笑属植物保护和利用提供有力指导。
4 结论利用SSR标记进行含笑属亲缘关系的研究,有助于开展后续的物种利用和保护工作。本研究利用12对高多态性引物构建了11个含笑属物种的基因分型体系,分析了不同物种间的亲缘关系,构建了Neighbor-Joining进化树,结果表明广东含笑、金叶含笑、醉香含笑、石碌含笑和苦梓含笑的亲缘关系较近,深山含笑和含笑的遗传距离较近,乐昌含笑与属于含笑亚属的白兰、黄兰和紫花含笑遗传关系较为紧密。
| [1] |
中国科学院中国植物志编辑委员会. 中国植物志[M]. 北京: 科学出版社, 1996. Flora of China Editorial Committee. Flora of China[M]. Beijing: Science Press, 1996. |
| [2] |
中国树木志编辑委员会. 中国树木志[M]. 北京: 中国林业出版社, 1983. Sylva Sinica Editorial Committee. Sylva Sinica[M]. Beijing: China Forestry Press, 1983. |
| [3] |
刘玉壶. 中国木兰[M]. 北京: 科学技术出版社, 2004. LIU Y H. Magnolia of China[M]. Beijing: Science and Technology Press, 2004. |
| [4] |
张新华. 含笑属植物研究进展[J]. 广西植物, 2007, 5(5): 712-719. DOI:10.3969/j.issn.1000-3142.2007.05.009 ZHANG X H. Advances in research of Michelia Linn[J]. Guihaia, 2007, 5(27): 712-719. DOI:10.3969/j.issn.1000-3142.2007.05.009 |
| [5] |
叶小玲, 胡晓敏, 朱开甫. 含笑属植物繁殖技术及其应用综述[J]. 安徽农学通报, 2013, 19(13): 35-36, 60. DOI:10.16377/j.cnki.issn1007-7731.2013.13.056 YE X L, HU X M, ZHU K P. Reproductive technology study and application of Michelia Linn[J]. Anhui Agricultural Science Bulletin, 2013, 19(13): 35-36, 60. DOI:10.16377/j.cnki.issn1007-7731.2013.13.056 |
| [6] |
郑纪伟, 教忠意, 窦全琴, 黄利斌, 何旭东. 利用荧光SSR标记构建含笑种质指纹图谱[J]. 分子植物育种, 2018, 16(14): 243-252. DOI:10.13271/j.mpb.016.004705.j.mpb.016.004705 ZHENG J W, JIAO Z Y, DOU Q Q, HUANG L B, HE X D. Construction of fingerprint of Michelia germplasm by fluorescent SSR markers[J]. Molecular Plant Breeding, 2018, 16(14): 243-252. DOI:10.13271/j.mpb.016.004705.j.mpb.016.004705 |
| [7] |
王艇, 朱建明, 苏应娟, 曾庆文, 黄超. 含笑亚族植物的RAPD分析及其系统学意义[J]. 中山大学学报(自然科学版), 1999, 38(6): 67-71. DOI:10.3321/j.issn:0529-6579.1999.06.005 WANG T, ZHU J M, SU Y J, ZENG Q W, HUANG C. Systematic relationship of subtrib micheliinae based on random amplified polymorphic DNA[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni, 1999, 38(6): 67-71. DOI:10.3321/j.issn:0529-6579.1999.06.005 |
| [8] |
王亚玲, 张寿洲, 崔铁成. trnL内含子及trnL-trnF间隔区序列在木兰科系统发育研究中的应用[J]. 西北植物学报, 2003, 23(2): 247-252. DOI:10.3321/j.issn:1000-4025.2003.02.010 WANG Y L, ZHANG S Z, CUI T C. The utility of trnL intron and trnLtrnF IGS in phylogenetic analysis of Magnoliaceae[J]. Acta Botanica Boreali-occidentalia Sinica, 2003, 23(2): 247-252. DOI:10.3321/j.issn:1000-4025.2003.02.010 |
| [9] |
滕花景. 乐昌含笑遗传变异的评价及繁育技术的研究[D]. 北京: 中国林业科学研究院, 2004. TENG H J. Evaluation of genetic variation of Michelia chapensis Dandy and research on the breeding techniques[D]. Beijing: Chinese Academy of Forestry, 2004. |
| [10] |
柴弋霞. 紫花含笑传粉生物学与杂交育种研究[D]. 长沙: 中南林业科技大学, 2018. CHAI Y X. Study on pollination biology and hybridization breeding of Michelia crassipes[D]. Changsha: Central South University of Forestry and Technology, 2018. |
| [11] |
李剑, 靳晓东, 杨秋生, 裴海潮, 刘增喜. 7种木兰科常绿植物亲缘关系的ISSR和AFLP分析[J]. 河南农业大学学报, 2013, 47(3): 289-295. DOI:10.3969/j.issn.1000-2340.2013.03.011 LI J, JIN X, YANG Q, PEI H C, LIU Z X. AFLP and ISSR markers analysis of the genetic relationship of 7 kinds of Magnoliaceae evergreen plants[J]. Journal of Henan Agricultural University, 2013, 47(3): 289-295. DOI:10.3969/j.issn.1000-2340.2013.03.011 |
| [12] |
王猛, 彭继庆, 曹基武, 曹福祥, 李娇婕, 薛超, 吴毅. 云南木兰科48种野生植物资源的遗传多样性研究[J]. 热带亚热带植物学报, 2020, 28(3): 277-284. WANG M, PENG J Q, CAO J W, CAO F X, LI J J, XUE C, WU Y. Studies on genetic diversity of 48 wild species of Magnoliaceae in Yunnan[J]. Journal of Tropical and Subtropical Botany, 2020, 28(3): 277-284. |
| [13] |
温强, 宋晓琛, 田径, 肖复明, 江香梅. 紫花含笑新品种鉴别及与4种含笑属植物亲缘关系分析[J]. 分子植物育种, 2014, 12(3): 509-516. DOI:10.13271/j.mpb.012.000509 WEN Q, SONG X C, TIAN J, XIAO F M, JIANG X M. Molecular identification of new cultivated varieties in Michelia crassipes and the relationship analysis with four relative Michelia species[J]. Molecular Plant Breeding, 2014, 12(3): 509-516. DOI:10.13271/j.mpb.012.000509 |
| [14] |
XU F, FENG S, WU R, DU F. Two highly validated SSR multiplexes (8‐plex) for Euphrates' poplar, Populus euphratica(Salicaceae)[J]. Molecular Ecology Resources, 2013, 13(1): 144-153. DOI:10.1111/1755-0998.12030 |
| [15] |
SUN Y, LIU Y F, WAN G J, GUO Y, HUANG H W. Ten polymorphic microsatellite markers in Michelia maudiae(Magnoliaceae)[J]. American Journal of Botany, 2010, 97(12): 157-158. DOI:10.3732/ajb.1000332 |
| [16] |
李红英. 鹅掌楸EST-SSR在木兰科属间通用性及系统学探讨[D]. 南京: 南京林业大学, 2008. LI H Y. The cross-genus transferability of EST-SSR from Liriodendron tulipifera Linn. and the implications for the phylogeny of Magnoliaceae [D]. Nanjing: Nanjing Forestry University, 2008. |
| [17] |
SCHUELKE M. An economic method for the fluorescent labeling of PCR fragments[J]. Nature Biotechnology, 2000, 18(2): 233-234. DOI:10.1038/72708 |
| [18] |
HOLLAND M M, PARSON W. GeneMarker HID: A reliable software tool for the analysis of forensic STR data[J]. Journal of Forensic Sciences, 2011, 56(1): 29-35. DOI:10.1111/j.1556-4029.2010.01565.x |
| [19] |
PEAKALL R, SMOUSE P E. GENALEX 6: Genetic analysis in Excel[J]. Molecular Ecology, 2012, 6(1): 288-295. |
| [20] |
WALLING C A, PEMBERTON J M, HADFIELD J D, LOESKE K. Comparing parentage inference software: reanalysis of a red deer pedigree[J]. Molecular Ecology, 2010, 19(9): 1914-1928. DOI:10.1111/j.1365-294X.2010.04604.x |
| [21] |
PERRIER X, FLORI A, BONNOT F, HAMON P, SEGUIN M, PERRIER X, GLASZMANN J C. Data analysis methods//Genetic diversity of cultivated tropical plants. Enfield[M]. Montpellier: Science Publishers, 2003.
|
| [22] |
李清莹. 火力楠种质资源遗传多样性研究[D]. 北京: 中国林业科学研究院, 2018. LI Q Y. Genetic diversity of germplasm resources of Michelia macclurei Dandy[D]. Beijing: Chinese Academy of Forestry, 2018. |
| [23] |
胥猛, 李火根. 鹅掌楸EST-SSR引物开发及通用性分析[J]. 分子植物育种, 2008, 6(3): 615-618. DOI:10.3969/j.issn.1672-416X.2008.03.035 XU M, LI H G. Development and characterization of microsatellite markers from expressed sequence tags for Liriodendron[J]. Molecular Plant Breeding, 2008, 6(3): 615-618. DOI:10.3969/j.issn.1672-416X.2008.03.035 |
| [24] |
张红莲, 李火根. 利用EST-SSR分子标记检测鹅掌楸种间渐渗杂交[J]. 生物多样性, 2010, 18(2): 120-128. ZHANG H L, LI H G. Detection of interspecific introgressive hybridization in Liriodendron with EST-SSR markers[J]. Biodiversity Science, 2010, 18(2): 120-128. |
| [25] |
张红莲, 李火根, 胥猛, 冯源恒. 鹅掌楸属种及杂种的SSR分子鉴定[J]. 林业科学, 2010, 46(1): 36-39. ZHANG H L, L I H G, XU M, FENG Y H. Identification of Liriodendron tulipifera, Liriodendron chinense and hybrid Liriodendron using species-specific SSR markers[J]. Scientia Silvae Sinicae, 2010, 46(1): 36-39. |
| [26] |
杨帅, 李慧, 侯欣, 张丽. 大规模开发及特性分析十字花科SSR分子标记及其数据库的构建[J]. 植物学研究, 2017, 6(3): 86-95. DOI:10.12677/BR.2017.63013 ZHANG S, LI H, HOU X, ZHANG L. Large-scale development and character analysis of SSR markers and database build in Brassicaceae[J]. Botanical Research, 2017, 6(3): 86-95. DOI:10.12677/BR.2017.63013 |
| [27] |
陈豫静. 基于SSR、SRAP标记的榛属种质资源遗传多样性及亲缘关系分析[D]. 沈阳: 沈阳农业大学, 2020. CHEN Y J. Genetic diversity and relationship analysis of Hazelnut germplasm resources based on SSR, SRAP marker[D]. Shenyang: Shenyang Agriculture University, 2020. |
| [28] |
郑艳. 玉叶金花属植物SSR分子标记全面开发及种间亲缘关系初步研究[D]. 深圳: 深圳大学, 2018. ZHENG Y. Development of SSR molecular markers in Mussaenda and preliminary studies on the genetic relationship of species[D]. Shenzhen: Shenzhen University, 2018. |
| [29] |
刘玉壶. 木兰科分类系统的初步研究[J]. 中国科学院大学学报, 1984, 22(2): 89-109. LIU Y H. A preliminary study on the taxonomy of the family Magnoliaceae[J]. Journal of University of Chinese Academy of Sciences, 1984, 22(2): 89-109. |
| [30] |
CHEN B L, NOOTEBOOM H P. Notes on MagnoliaceaeⅢ: the Magnoliaceae of China[J]. Annals of the Missouri Botanical Garden, 1993, 80(4): 999-1104. DOI:10.2307/2399942 |
| [31] |
YAN Y H, ZENG Q W, XING F W. Michelia guangdongensis (Magnoliaceae), a new species from China[J]. Annales Botanici Fennici, 2004, 41(6): 491-493. |
| [32] |
谭殷殷, 金晓玲, 余秋岫, 孙凌霄. '玉霞'含笑与其亲本抗寒生理特性比较[J]. 植物生理学报, 2019, 55(11): 1721-1728. DOI:10.13592/j.cnki.ppj.2019.0167 TAN Y Y, JIN X L, YU Q X, SUN L X. Comparison of physiological characteristics of cold resistance between Michelia 'Yuxia' and its parents[J]. Plant Physiology Journal, 2019, 55(11): 1721-1728. DOI:10.13592/j.cnki.ppj.2019.0167 |
| [33] |
王晶, 赵强民, 高泽正. 含笑属Michelia新品种'香绯'和'香雪' 的选育[J]. 广东园林, 2019, 41(6): 53-55. WANG J, ZHAO Q M, GAO Z Z. Breeding of two new Michelia hybrid varieties[J]. Guangdong Landscape Architecture, 2019, 41(6): 53-55. |
 2021, Vol. 48
2021, Vol. 48