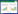文章信息
基金项目
- 浙江省基础公益研究项目(GN18C140001)
作者简介
- 单长林(1987—),男,硕士,农艺师,研究方向为植物检疫,E-mail:changlin_shan@163.com.
文章历史
- 收稿日期:2020-10-24
【研究意义】玉米细菌性枯萎病菌(Pantoea stewartii subsp. stewarii,Pss)是我国进境植物检疫性病原物之一,主要分布在美国、加拿大、墨西哥、巴西、秘鲁、意大利、波兰、罗马尼亚、马来西亚、泰国、越南以及俄罗斯、乌克兰等原苏联国家和地区[1]。尽管我国是仅次于美国的第二大玉米生产国,但随着国内玉米需求量的激增,未来中国仍需要通过进口来填补玉米产需缺口,玉米净进口将成为常态[2]。目前玉米进口来源主要集中在乌克兰、美国等玉米细菌性枯萎病菌发生国家[3]。随着玉米进口贸易的发展,玉米细菌性枯萎病菌侵入我国风险剧增,该病原物在我国主要玉米产区均适合定殖,具有广泛的适生区域,一旦入侵将严重威胁我国玉米生产安全[4]。因此,建立并掌握针对玉米细菌性枯萎病菌快速检测技术至关重要。
【前人研究进展】目前,针对玉米细菌性枯萎病菌的检测主要有传统的分离培养[5]、血清学检测[6]、分子生物学检测[7-8]和红外光谱检测[9],这些方法或操作复杂、耗时较长,或需大型仪器的支持,无法实现体外恒温条件的快速检测。与传统PCR的变性-退火-延伸等热循环解链原理不同,环介导基因恒温扩增技术(loop-mediated isothermal amplification, LAMP)和重组酶介导等温扩增技术(recombinase-aided amplification, RAA)均可在恒温条件下实现目标序列的扩增。LAMP技术[10]在65℃条件下,使用链置换型Bst DNA聚合酶,利用4~6个引物,通过类似于滚环复制的链替换反应产生互补序列,形成大小不一的一系列条带。LAMP技术是目前体外恒温扩增检测病原物主要方法之一,广泛用于细菌、真菌、病毒等病原物检测[11-15]。封立平等[11]利用LAMP技术成功建立了针对Pss的快速检测方法,该方法特异、灵敏且结果可视化。但利用LAMP技术检测Pss所得产物为大小不等的一系列条带,无法进行克隆、测序等进一步验证。而RAA技术[16]则利用重组酶,在单链DNA结合蛋白(singlestranded DNA binding, SSB)的帮助下, 使模板DNA解链,经DNA聚合酶的作用,恒温条件下实现目标序列的快速扩增,且产物为单一条带。
【本研究切入点】参与磷酸代谢的pst操纵子和相邻的glmS基因区间序列(pstS-glmS序列)在不同菌种间存在明显差异,可用于Pss菌株鉴定[17-18]。RAA方法具有耗时短、特异性强、灵敏度高等特点,是目前应用于细菌、病毒和支原体等病原微生物恒温检测[19-21]研究的热门方法之一。【拟解决的关键问题】本试验使用荧光重组酶介导等温扩增技术,以pstS-glmS序列为模板设计引物和探针,以建立在恒温条件下快速检测玉米细菌性枯萎病菌的荧光RAA检测方法。
1 材料与方法 1.1 试验材料供试玉米样本为乌克兰进境饲用玉米,进境口岸为舟山,进境时间为2018—2019年,共计24个样品,每个样品2 kg。
供试菌株共计12株,其中玉米细菌性枯萎病菌菌株(Pantoea stewartii subsp. stewarii, Pss)为ATCC标准菌株(编号ATCC 29229),玉米内州萎蔫病菌(Clavibacter michiganensis subsp. nebraskensis, Cmn)为ATCC标准菌株(编号ATCC 27794),由杭州海关综合技术服务中心赠送;丁香假单胞杆菌番茄致病变种(Pseudomonas syringae pv. syringae, Pssy)、砖红色微杆菌(Microbacterium testaceum, Mt)、密执安棍状杆菌诡谲亚种(Clavibacter michiganensis subsp. insidiosus, Cmi)、密执安棍状杆菌密执安(Clavibacter michiganensis subsp. michiganensis, Cmm)为本实验室保存;成团泛菌(Pantoea agglomeran, Pag)、菠萝泛菌(Pantoea ananatis, Pan)、分散泛菌(Pantoea dispersa, Pd)、伯克霍尔德氏菌(Burkholderia andropogonis, Ba)、玉米迪克氏菌(Dickeya zeae, Dz)、欧氏杆菌(Erwinia chrysanthemi var. zea, Ecz),购自北京百欧博伟生物技术有限公司。
主要试剂:荧光RAA试剂盒(S002ZC)购自杭州众测生物技术有限公司,细菌基因组DNA提取试剂盒(9763)购自宝生物工程(大连)有限公司,新型植物基因组DNA提取试剂盒(DP320)购自天根生化科技(北京)有限公司,2× Taq PCR StarMix with Loading Dye(A012)购自GenStar-康润生物。主要仪器:荧光定量PCR仪(ABI 7500)。
1.2 试验方法1.2.1 细菌菌株准备、基因组提取 在NA平板划线,28 ℃培养48 h,接种环刮板收集菌株。采用细菌基因组DNA提取试剂盒提取细菌基因组,用微量分光光度计(Nanodrop 2000)验证提取效果。-20 ℃保存备用。
1.2.2 目标片段获取、引物探针设计 以Pss全基因组序列(Gen Bank登录号:CP 017581)为靶标,设计扩增pstS-glmS靶标序列引物pss3/pss4,分别对应基因组444415 nt和444914 nt,扩增靶标序列。PCR反应体系(50 μL):2×Taq PCR StarMix with Loading Dye 25 μL,pss3/pss4(10 μmol/L)各2 μL,DNA模板2 μL,ddH2O 19 μL。PCR扩增程序:94 ℃ 3 min;94 ℃ 30 s、55 ℃ 30 s、72 ℃ 45 s,35个循环;72 ℃延伸7 min。PCR产物经1.5% 琼脂糖凝胶电泳割胶回收,进行克隆测序。
以测序获取序列片段(图 1)为模板,按照RAA引物、探针设计原则,设计正向引物F1、F2、F3、F4、反向引物R1、R2、R3和探针P,BLAST分析验证引物、探针特异性。以上引物、探针序列信息见表 1、图 1。本试验目的序列克隆测序,引物、探针的合成均由生物工程技术(上海)有限公司完成。

|
| 图 1 荧光RAA引物与探针在靶标序列上的位置 Fig. 1 Location of primers and probes designed for fluorescent RAA on target DNA sequence |
1.2.3 引物筛选与体系建立 采用商品化荧光RAA试剂盒,并利用荧光定量PCR仪进行荧光RAA实验。按照方法1.2.1提取Pss DNA(280 ng/μL),以无菌ddH2O进行10倍等梯度稀释,梯度浓度分别为280、28、2.8、2.8× 10-1、2.8×10-2、2.8×10-3、2.8×10-4、2.8× 10-5 ng/μL。以不同浓度的Pss DNA为模板,选取F1/R1引物组合,初步探索荧光RAA最低检测浓度。
以Pss最低检测浓度DNA为模板,分别探索不同引物组合F1/R1、F1/R2、F1/R3、F2/R1、F2/R2、F2/R3、F3/R1、F3/R2、F3/R3、F4/R1、F4/R2、F4/R3对Pss的荧光RAA检测效果,筛选出扩增效率最高的引物组合。反应体系采用试剂盒说明书推荐体系(50 μL):A Buffer 40.9 μL,B Buffer 2.5 μL,上游引物和下游引物(10 μmol/L)各2.0 μL,荧光探针(10 μmol/L)0.6 μL,DNA模板2.0 μL;扩增程序为39 ℃ 30 s,共40个循环。1.2.4 灵敏度测定及特异性验证 参照1.2.3试验结果,合理选取不同浓度的Pss DNA样本,并利用筛选出的最佳引物组合,测定建立荧光RAA检测方法的灵敏度。
荧光RAA特异性检测分别以3株泛菌属其他菌株(Pan、Pag、Pd)及8株病原细菌(Cmn、Pssy、Mt、Cmi、Cmm、Ba、Dz、Ecz)DNA为参考模板进行验证。1.2.5 荧光RAA检测玉米样本的应用 将1.2.1收集的菌体,用无菌ddH2O制备菌悬液,采用涂板法测定菌悬液浓度(3.5×103 CFU/mL)。选取鉴定结果为阴性的进境饲用玉米(实验室编号1-3103304),磨成粉末状,每份10 g,共6份,分别加入浓度为3.5×103 CFU/mL的Pss菌液,搅拌混匀,制备阳性模拟玉米样本。利用新型植物基因组DNA提取试剂盒提取模拟样本与真实玉米样本DNA,进行荧光RAA检测。同时以标准SN/T 1375-2004:玉米细菌性枯萎病菌检疫鉴定方法复核验证。
2 结果与分析 2.1 引物和探针序列分析利用BLAST与全数据库序列比对,分析实验中设计的引物,结果显示:实验设计所有引物组合,包括pss3/pss4、F1/R1、F1/R2、F1/R3、F2/R1、F2/R2、F2/R3、F3/R1、F3/R2、F3/R3、F4/R1、F4/R2、F4/R3对Pss菌株DC283(登录号:CP017581)、ZJ-FGZX1(登录号:CP049115)的识别率和覆盖率均为100%,对数据库其他序列没有明显识别现象;探针P对Pss菌株DC283(登录号:CP017581)、菌株ICMP275 pstS-glmS序列(登录号:AB894430)的识别率为100%,菌株ZJFGZX1(登录号:CP049115)识别率为97.87%,对数据库其他序列无识别现象。
2.2 引物筛选与体系建立以Pss梯度稀释DNA为模板,探索引物组合F1/R1的荧光RAA最低检测浓度,结果见图 2A。浓度为2.8、2.8×10-1、2.8×10-2、2.8×10-3 ng/μL的DNA模板进行荧光RAA检测,均具有明显的扩增曲线,浓度为2.8×10-4 ng/μL的DNA则没有典型的扩增曲线。故引物组合F1/R1的荧光RAA最低检测浓度为2.8×10-3 ng/μL。本实验以2.8×10-3 ng/μL DNA为模板进行不同引物组合扩增效果实验。
|
|
图 2 荧光RAA灵敏性检测结果 Fig. 2 Sensitivity detection result of fluorescent RAA A: 引物组合F1/R1荧光RAA最低检测浓度初步测定结果;B:引物F1/R2荧光RAA灵敏性检测结果 E3~E8 DNA浓度分别为2.8、2.8×10-1、2.8×10-2、2.8×10-3、2.8×10-4、2.8×10-5 ng/μL; N: 阴性对照 A: The minimum detectable concentration of fluorescent RAA with F1/R1 primers; B Sensitivity result of fluorescent RAA with F1/R2M E3-E8: the concentrations of DNA were 2.8, 2.8×10-1, 2.8×10-2, 2.8×10-3, 2.8×10-4, and 2.8×10-5 ng/μL, respectively; N: Negative control |
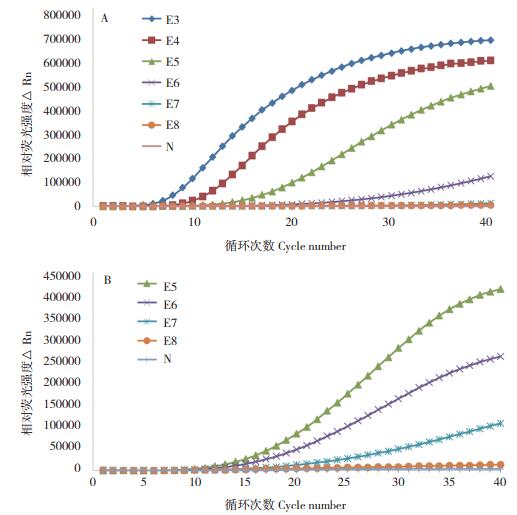
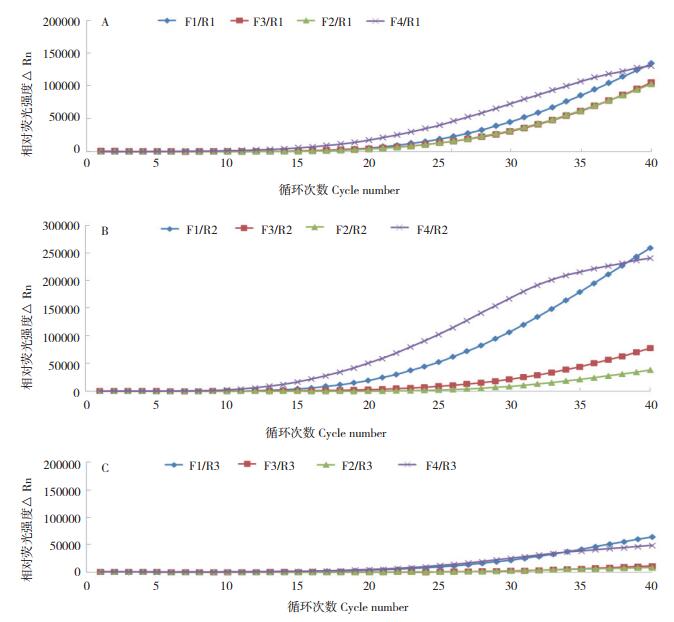
图 3A、B、C显示的是以2.8×10-3 ng/μL DNA为模板,反向引物R1、R2、R3分别与正向引物F1、F2、F3、F4组合的荧光RAA扩增曲线。结果显示,每种组合均能收集到扩增曲线,但引物组合F1/R2与F4/R2在相同反应循环数下,相对荧光强度均高于其他引物组合,扩增效率明显高于其他引物组合。不同引物组合荧光RAA经40个循环收集到的相对荧光强度(∆Rn)大小依次为F1/R2 > F4/R2 > 250 000 > F1R1/> F4/R1 > 150 000 > F3/R1 > F2/R1 > F3/R2 > 100 000 > F2/R2 > F1/R3 > F4/R3 > 50 000 > F3/R3 > F2/R3。比较引物组合F1/R2和F4/R2,在38个循环前引物组合扩增效率F4/R2 > F1/R2,但在38个循环后F1/R2扩增效率明显高于F4/R2。经多次重复,最终选定引物组合F1/R2为玉米细菌性枯萎病菌荧光RAA检测最佳引物组合。
|
| 图 3 引物筛选荧光RAA结果 Fig. 3 Fluorescent RAA results of primer screening A:分别以引物F1/R1、F2/R1、F3/R1、F4/R1组合的荧光RAA反应结果;B:分别以引物F1/R2、F2/R2、F3/R2、F4/R2组合的荧光RAA反应结果;C:分别以引物F1/R3、F2/R3、F3/R3、F4/R3组合的荧光RAA反应结果 A: Results of fluorescent RAA with primers F1/R1, F2/R1, F3/R1, F4/R1 respectively; B: Results of fluorescent RAA with primers F1/R2, F2/R2, F3/R2, F4/R2 respectively; C: Results of fluorescent RAA with primers F1/R3, F2/R3, F3/R3, F4/R3 respectively |
2.3 灵敏性及特异性测定
参照2.2中引物组合F1/R1的荧光RAA最低检测浓度为2.8×10-3 ng/μL(图 2A),本实验选取浓度为2.8×10-2、2.8×10-3、2.8×10-4及2.8×10-5 ng/μL的玉米细菌性枯萎病菌DNA样本,进行引物组合F1/R2荧光RAA灵敏度测定,结果见图 2B。浓度为2.8×10-2、2.8×10-3和2.8×10-4 ng/μL的样本均呈现明显扩增曲线,浓度为2.8×10-5 ng/μL的样本无明显扩增现象。因此,以引物组合F1/R2建立的荧光RAA检测方法的灵敏度为2.8×10-4 ng/μL(280 fg/μL),比引物F1/R1组合的荧光RAA灵敏度提高10倍。
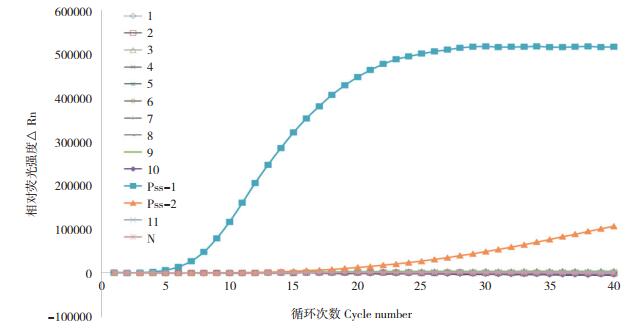
特异性验证实验选取3株泛菌属其他菌株及8株病原细菌DNA为参考模板,以不同浓度的Pss DNA(编号Pss-1:280 ng/μL;Pss-2:2.8×10-4 ng/μL)为阳性对照,无菌ddH2O为阴性对照,验证建立的荧光RAA检测体系的特异性,结果见图 4,玉米细菌性枯萎病菌DNA样品Pss-1、Pss-2均具有明显扩增曲线,包括3株泛菌属Pag、Pan、Pd基因组在内的其他11株参照DNA模板均没有扩增现象,表明以引物F1/R2和探针P组合的荧光RAA检测体系可以特异检出Pss。
|
| 图 4 荧光RAA特异性实验 Fig. 4 Specificity result of fluorescent RAA 1~11:分别为玉米内州萎蔫病菌、丁香假单胞杆菌番茄致病变种、砖红色微杆菌、密执安棍状杆菌诡谲亚种、密执安棍状杆菌密执安亚种、成团泛菌、菠萝泛菌、分散泛菌、伯克霍尔德氏菌、玉米迪克氏菌、欧氏杆菌;N:无菌水;Pss-1:浓度为280 ng/μL Pss DNA;Pss-2:浓度为2.8×10-4 ng/μL Pss DNA 1-11:Clavibacter michiganensis subsp. nebraskensis, Pseudomonas syringae pv. syringae, Microbacterium testaceum, Clavibacter michiganensis subsp. insidiosus, Clavibacter michiganensis subsp. michiganensis, Pantoea agglomerans, Pantoea ananatis, Pantoea dispersa, Burkholderia andropogonis, Dickeya zeae, Erwinia chrysanthemi var. zea; N: Negative control; Pss-1: 280 ng/μL Pss DNA; Pss-2: 2.8×10-4 ng/μL Pss DNA |
2.4 模拟样本及真实样品检测效果
利用新型植物DNA提取试剂盒,提取进境玉米样本和模拟玉米样本DNA。使用建立的荧光RAA检测方法和SN/T 1375-2004标准方法同时进行Pss检测,结果利用荧光RAA检测方法检出6份模拟玉米样本,未检出24份真实玉米样本,检测结果与SN/T 1375-2004方法一致,准确率100%。证实新建立的荧光RAA检测方法可针对玉米种子样本进行玉米细菌性枯萎病菌的检测。
3 讨论RAA技术具有灵敏、特异、简便和快速的优点,可实现常温下对目的序列的指数级扩增。目前针对RAA引物、探针设计,主要要求引物长度在30~35 nt之间,序列中无回文结构、连续单碱基重复序列和二级结构,暂无针对引物与重组酶蛋白结合方面的设计要求。本实验中以玉米细菌性枯萎病菌pstS-glmS特异性序列为模板,设计4条正向引物和3条反向引物,尽管12组引物对都能实现扩增,但分析荧光RAA经40个循环收集的相对荧光强度(∆Rn),扩增效率差异很大。同一反向引物中,与正向引物F1、F4组合扩增效率明显高于与引物F2、F3组合扩增效率;与F1、F4组合的反向引物R1、R2、R3中,R2组合扩增效率明显高于R1、R3。究其原因,应是引物与重组酶蛋白结合效率存在差异,正向引物F1、F4与重组酶蛋白结合效率比F2、F3高,反向引物R2与重组酶蛋白结合效率比R1、R3高。因此,有必要进一步研究RAA中引物与重组酶蛋白之间的结合机制,以期指导引物的设计,提高效率。
分子生物学检测是目前实验室进行植物病原菌检测的重要手段,而靶标序列的选择则是实现特异性检测的关键所在。目前针对玉米细菌性枯萎病菌进行的分子生物学检测,选取的靶标序列主要有16S-23S序列、16S序列[7]、cpsD基因序列[8]和pstS-glmS序列[18]。依据16S-23S序列筛选特异性引物制定的标准SN/T 3756-2013:玉米细菌性枯萎病菌检疫鉴定方法PCR方法,因已被证实引物序列无法区分Pss和Pantoea stewartii subsp. indologenes(Psi),于2019年12月31日废止(海关总署公告2019年第231号)。王赢等[7]利用16S序列成功设计了玉米细菌性枯萎病菌特异性引物Ps2r/Ps3r,并对部分文献中设计的引物序列进行特异性验证,发现包括DEP1/DEP2、CPSL1/CPSL2c在内的多对依靠16S序列和cps基因序列设计的引物均无法区分Pss和Psi。Gehring等[17]和Wensing等[18]通过序列分析证实pstS-glmS序列在不同菌种间存在明显差异,可以区分Pss和Psi,用于Pss菌株鉴定。本实验利用pstS-glmS序列为靶标序列,设计引物和探针,通过BLAST与全库数据序列分析和参考菌株特异性检测实验,证实引物探针的特异性,成功建立了针对玉米细菌性枯萎病菌的荧光RAA检测方法。
新建立的以引物F1/R2和荧光探针P为组合的荧光RAA检测方法,实现了针对玉米细菌性枯萎病菌的特异性扩增,灵敏度高达280 fg/μL,与实时荧光PCR方法[8]相比,荧光RAA灵敏度有所提升(Tambong等建立的TaqMan实时荧光法灵敏度为1 pg),且检测可利用小型仪器—荧光测试仪在恒温条件下完成反应,不需要大型仪器支持;与酶联免疫试纸条法检测玉米细菌性枯萎病菌[6]相比,仅需20 min就可完成反应,不需要在4 ℃条件下0.9% NaCl过夜孵育,节省检测时间;与LAMP技术用于检测玉米细菌性枯萎病菌[11]相比,因为其扩增产物为单一条带,而不是大小不等的一系列片段,可用于后续的克隆、测序等进一步验证,也可实时监控扩增情况,为后续定量研究奠定基础。
4 结论本研究针对玉米细菌性枯萎病菌特异性pstS-glmS序列片段,参照RAA原理设计引物和探针,并通过引物筛选、特异性验证和灵敏性测定等实验,成功建立了玉米细菌性枯萎病菌荧光RAA检测方法。该方法可在20 min内,特异性地完成玉米细菌性枯萎病菌的检测,灵敏度高达280 fg/μL,且通过了玉米真实样本与玉米模拟样本检测验证,为玉米细菌性枯萎病菌的快速检测提供了新的方法选择。
| [1] |
DIRK J G, MARTIJIN S, ALICE D, SYBREN V. Pest survey card on Pantoea stewartii subsp. stewartii[J]. EFSA Supporting Publications, 2020, 17(6). DOI:10.2903/sp.efsa.2020.EN-1878 |
| [2] |
毛爽, 高庆鹏, 朱丰超. 未来国际市场对中国玉米进口的适应性分析[J]. 世界农业, 2012(5): 57-62. DOI:10.3969/j.issn.1002-4433.2012.05.013 MAO S, GAO Q P, ZHU F C. Analysis on the adapt abilit y of International market to Chinese corn import in the future[J]. World Agriculture, 2012(5): 57-62. DOI:10.3969/j.issn.1002-4433.2012.05.013 |
| [3] |
李慧燕, 岳云娇, 陆丽萍. 中国玉米进口来源地可依赖度比较分析[J]. 世界农业, 2017(8): 140-146. DOI:10.13856/j.cn11-1097/s.2017.08.023 LI H Y, YUE Y J, LU L P. Comparative analysis of source dependence of Corn import in China[J]. World Agriculture, 2017(8): 140-146. DOI:10.13856/j.cn11-1097/s.2017.08.023 |
| [4] |
刘翔, 刘凤权. 基于地理信息系统的玉米细菌性枯萎病在中国适宜流行区的初步研究[J]. 植物检疫, 2008, 22(1): 9-11. DOI:10.3969/j.issn.1005-2755.2008.01.004 LIU X, LIUF Q. A preliminary study on the potential area for establishment of Stewart's wilt in China assessed by geographical information system[J]. Plant Quarantine, 2008, 22(1): 9-11. DOI:10.3969/j.issn.1005-2755.2008.01.004 |
| [5] |
郭翼奋, 梁再群, 俞大绂. 玉米细菌性枯萎病选择性培养基——黑色素培养基[J]. 微生物学报, 1982, 22(4): 339-344. GUO Y F, LIANG Z Q, YU D F. Nigrosine medium—selective medium for isolation of Erwinia stewarii from imported corn[J]. Acta Microbiologica Sinica, 1982, 22(4): 339-344. |
| [6] |
FENG M, KONG D Z, WANG W B, LIU L Q, SONG S S, XU C L. Development of an immunochromatographic strip for rapid detection of Pantoea stewartii subsp. stewartii[J]. Sensors, 2015, 15(2): 4291-4301. DOI:10.3390/s150204291 |
| [7] |
王赢, 周国梁, 印丽萍, 袁平, 王文兵, 易建平. 玉米细菌性枯萎病菌PCR检测[J]. 植物病理学报, 2009, 39(4): 368-376. WAN Y, ZHOU G L, YIN L P, YUAN P, WANG W B, YI J P. PCR detection of Pantoea stewartii subsp.stewartii[J]. Acta Phytopathologica Sinica, 2009, 39(4): 368-376. |
| [8] |
TAMBONG J T, MWANGE K N, BERGERON M, DING T, MANDY F, REID L M, ZHU X. Rapid detection and identification of the bacterium Pantoea stewartii in maize by TaqMan® real-time PCR assay targeting the cpsD gene[J]. Journal of Applied Microbiology, 2008, 104(5): 1525-1537. DOI:10.1111/j.1365-2672.2007.03674.x |
| [9] |
夏拯, 陈曦, 张明哲, 吴志毅, 林晓佳, 陈吴健. 玉米细菌性枯萎病和玉米内州萎焉病的红外光谱检测技术研究[J]. 西南农业学报, 2015, 28(3): 1083-1087. DOI:10.16213/j.cnki.scjas.2015.03.030 XIA Z, CHEN X, ZHANG M Z, WU Z Y, LIN X J, CHEN W J. IR detection technology research of bacterial wilt of corn or stewart's wilt and goss's bacterial wilt and leaf blight[J]. Southwest China Journal of Agriculture Sciences, 2015, 28(3): 1083-1087. DOI:10.16213/j.cnki.scjas.2015.03.030 |
| [10] |
NOTOMI T, OKAYAMA H, MASUBUCHI H, YONEKAWA T, WATANABE K, AMINO N, HASE T. Loop-mediated isothermal amplification of DNA[J]. Nucleic acids research, 2000, 28(12): 63-63. DOI:10.1093/nar/28.12.e63 |
| [11] |
封立平, 倪新, 吴兴海, 伦才智, 吴翠萍, 栾晶. 玉米细菌性枯萎病菌的环介导恒温扩增(LAMP)检测方法[J]. 植物保护学报, 2015, 42(3): 347-352. DOI:10.13802/j.cnki.zwbhxb.2015.03.010 FENG L P, NI X, WU X H, LUN C Z, WU C P, LUAN J. Loop-mediated isothermal amplification(LAMP)for detection of Pantoea stewartii subsp. stewartii[J]. Journal of Plant protection, 2015, 42(3): 347-352. DOI:10.13802/j.cnki.zwbhxb.2015.03.010 |
| [12] |
单长林, 李雪松, 李孝军, 周圆, 杨赛军, 王健, 张建成. 玉米内州萎蔫病菌LAMP快速检测方法的建立[J]. 安徽农业科学, 2015, 43(22): 17-20, 22. DOI:10.3969/j.issn.0517-6611.2015.22.008 SHAN C L, LI X S, LI X J, ZHOU Y, YANG S J, WANG J, ZHANG J C. The establishment of a rapid detection method of Clavibacter michiganensis subsp. nebraskensis LAMP[J]. Journal of Anhui Agriculture Science, 2015, 43(22): 17-20, 22. DOI:10.3969/j.issn.0517-6611.2015.22.008 |
| [13] |
单长林, 周圆, 李孝军, 杨赛军, 邵炜冬. RT-LAMP及RT-PCR方法快速检测玉米褪绿斑驳病毒的比较与应用[J]. 广东农业科学, 2017, 44(7): 83-90. DOI:10.16768/j.issn.1004-874X.2017.07.014 SHAN C L, ZHOU Y, LI X J, YANG S J, SHAO W D. Comparison and application of RT-LAMP and RT-PCR methods for MCMV detection[J]. Guangdong Agricultural Sciences, 2017, 44(7): 83-90. DOI:10.16768/j.issn.1004-874X.2017.07.014 |
| [14] |
杨雷亮, 管维, 孙丽霞, 吴颖儿, 单振菊, 王章根, 陈定虎. 香蕉细菌性枯萎病菌荧光LAMP检测体系的建立[J]. 广东农业科学, 2020, 47(5): 66-73. DOI:10.16768/j.issn.1004-874X.2020.05.009 YANG L L, GUAN W, SUN L X, WU Y E, SHAN C J, WANG Z G, CHEN D H. Establishment of fluorescence LAMP detection system for pathogen of moko disease[J]. Guangdong Agricultural Sciences, 2020, 47(5): 66-73. DOI:10.16768/j.issn.1004-874X.2020.05.009 |
| [15] |
龙阳, 马新华, 袁俊杰, 卢乃会, 杨卓瑜, 魏霜, 王卫芳. 油菜茎基溃疡病菌LAMP-HNB检测方法的建立[J]. 广东农业科学, 2017, 44(10): 66-69. DOI:10.16768/j.issn.1004-874X.2017.10.011 LONG Y, MA X H, YUAN J J, LU N H, YANG Z Y, WEI S, WANG W F. Establishment of LAMP-HNB method for detection of Leptosphaeria maculans in rape[J]. Guangdong Agricultural Sciences, 2017, 44(10): 66-69. DOI:10.16768/j.issn.1004-874X.2017.10.011 |
| [16] |
LU B, CHENG H R, YAN Q F. Recombinase-aid amplification: a novel technology of in vitro rapid nucleic acid amplification[J]. Scientla Sinica Vitae, 2010, 40: 983-988. DOI:10.1360/052010-508 |
| [17] |
GEHRING I, WENSING A, GERNOLD M, COPLIN D L, GEIDER K. Molecular differentiation of Pantoea stewartii subsp. indologenes from subspecies stewartii and identification of new isolates from maize seeds[J]. Journal of Applied Microbiology, 2014, 116(6): 1553-1562. DOI:10.1111/jam.12467 |
| [18] |
WENSING A, ZIMMERMANN S, GEIDER K. Identification of the corn pathogen Pantoea stewartii by mass spectrometry of whole-cell extracts and its detection with novel PCR primers[J]. Applied and Environmental Microbiology, 2010, 76(18). DOI:10.1128/AEM.01032-10 |
| [19] |
周冬根, 罗洁. 沙门菌荧光重组酶介导等温扩增检测方法的应用研究[J]. 中国热带医学, 2019, 19(2): 107-110, 119. DOI:10.13604/j.cnki.46-1064/r.2019.02.02 ZHOU D G, LUO J. Establishment and application of a fluorescent recombinase-aided amplification assay to detect Salmonella[J]. China Tropical Medicine, 2019, 19(2): 107-110, 119. DOI:10.13604/j.cnki.46-1064/r.2019.02.02 |
| [20] |
SHEN X X, QIU F Z, SHEN L P, YAN T F, ZHAO M C, QI J J, CHEN C, ZHAO L, WANG L, FENG Z S, MA X J. A rapid and sensitive recombinase aided amplification assay to detect hepatitis B virus without DNA extraction[J]. BMC Infectious Diseases, 2019, 19(1): 229-229. DOI:10.1186/s12879-019-3814-9 |
| [21] |
XUE G H, LI S L, ZHAO H Q, YAN C, FENG Y L, CUI J H, JIANG T T, YUAN J. Use of a rapid recombinase-aided amplification assay for Mycoplasma pneumonia detection[J]. BMC Infectious Diseases, 2020, 20(1): 79-79. DOI:10.1186/s12879-019-4750-4 |
 2021, Vol. 48
2021, Vol. 48