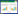文章信息
基金项目
- 广东省自然科学基金面上项目(2021A1515011112);广东省重点领域研发计划项目(2020B0202090003);广东省重点实验室运行费专项(2020B1212060047)
作者简介
- 陈可,博士,助理研究员,2012年7月毕业于华南农业大学生物技术专业,获学士学位。2019年博士毕业于中国科学院大学上海植物科学卓越创新中心,师从林鸿宣院士。2019年8月-2021年3月在沙特阿拉伯阿卜杜拉国王科技大学进行博士后工作。2021年3月入职广东省农业科学院水稻研究所功能性水稻选育研究室。目前主要从事水稻抗逆分子机理研究及水稻产量性状改良,主持国家自然科学基金青年项目1项,在国内外学术期刊发表科技论文近10篇.
通讯作者
- 刘传光(1968—),男,博士,研究员,研究方向为功能性水稻选育,E-mail:guyliu@126.com.
文章历史
- 收稿日期:2021-08-24
2. 广东海洋大学滨海农业学院,广东 湛江 524088
2. Binhai Agricultural College, Guangdong Ocean University, Zhanjiang 524088, China
作为全球范围内最重要的粮食作物之一,水稻提供了约50% 世界人口的每日主粮[1]。因此,研究高产、优质的水稻及其分子机理有着重要的生物学和生产实践意义。20世纪50年代开始的第一次“绿色革命”,包括水稻育种的半矮秆基因(semi-dwarf 1)sd1的应用、70年代三系杂交水稻的应用,极大地促进了谷物产量增长[2]。这一阶段主要关注水稻和小麦半显性基因的应用及以胞质雄性不育(CMS)在杂交水稻的应用[3]。进入21世纪,“超级稻”概念、“理想株型”模型及基因编辑技术的发展为我国水稻产量持续增长奠定了理论基础[4]。特别是在新冠病毒肆虐的2020年以后,我国水稻生产又面临新的严峻挑战,“三胎”政策的开放促进人口增长、经济发展导致耕地过快减少的态势与“18亿亩耕地红线”严防死守的矛盾等。2021年中央一号文件把提升粮食和重要农产品供给保障能力作为加快推进农业现代化的首要任务。因此,如何突破传统育种瓶颈,将水稻分子育种和功能基因组学系统结合应用于水稻育种改良,已经成为新时代我国粮食安全和农业现代化面临的亟待解决的重大科学问题[5]。
水稻单株产量主要由有效穗数、每穗粒数、粒重3个因素决定。多年来的水稻基础研究不断丰富水稻基因组序列信息和注释,并且超过120个主效及微效(数量性状遗传位点)QTLs位点已经成功克隆、鉴定及功能分析[6]。在超过1万年的水稻驯化过程中,人类通过人工选择产生了许多水稻亚种和品种,并且使得水稻的分布从热带地区扩展到温带地区的五大洲。近年来,基于图位克隆的遗传分析、规律成簇的间隔短回文重复(Clustered Regularly Interspaced Short Palindromic Repeats,CRISPR)及其衍生的基因编辑技术快速发展,在多种水稻农艺性状的研究中发挥了重要作用[7]。本文综述总结了近年来水稻粒重相关基因的研究进展,包括QTL和质量性状相关基因,并且深入讨论其在现代农业育种上的应用,结合5G时代数字化智能化生物现代育种,对我国目前生物育种4.0时代进行展望。
1 水稻粒重影响因素及相关基因水稻粒重(千粒重、鲜重)受到水稻粒长、粒宽、粒厚和灌浆等因素影响[8],这些基因协同作用,共同调控水稻粒重这一数量性状,维持水稻产量的动态平衡。水稻的拔节孕穗期是营养生长与生殖生长的并进时期,这期间水稻生物量迅速增大,以幼穗发育为中心的生殖发育迅速起始,特别是水稻幼穗发育8个时期中的第3期- 第2次枝梗原基及颖花原基分化期决定水稻穗型、第5期到第8期颖壳发育期决定水稻粒型。从细胞生物学角度来评估水稻粒重的变化,可以追溯到原基发育时分生组织的细胞数量、细胞分裂能力和细胞伸长范围等。而水稻幼穗分化完成期及水稻灌浆期是对已定型的完成授粉的水稻籽粒进行营养运输的二次分配(源- 库- 流理论)的时期,灌浆效率及淀粉等营养物质的组成均对水稻粒重产生明显影响。迄今为止,几乎大部分的主效粒重相关QTLs已经被报道,多个综述也从不同角度总结这些基因及其作用[8-9]。除了上述因素外,颖壳的发育对水稻粒型的长度和宽度也有重要影响。水稻灌浆过程和胚乳发育过程影响光合产物及其积累。表 1为近年来克隆鉴定的影响水稻粒宽、粒长、粒厚及灌浆速率的QTL和功能基因。
2 不同信号调控途径的水稻粒重相关基因
过去20年的研究揭示了水稻粒重受到多种信号途径调控,包括泛素化修饰途径、植物激素信号途径、G蛋白信号途径、光合产物积累运输相关途径、胚乳发育途径、表观修饰途径及小微RNA途径等[27]。本文对上述途径的水稻粒重相关基因进行总结与梳理。
2.1 泛素化调控途径泛素介导的蛋白酶体降解途径是细胞体内代谢和蛋白动态变化的主要途径。泛素化调控途径包括3种泛素酶的协同作用:由泛素激活酶E1、泛素结合酶E2和泛素连接酶E3特异性识别靶蛋白并且标记泛素,进而受到蛋白酶体识别降解[28]。Grain Width 2(GW2)是粳稻主效调控粒宽的QTL, 编码一个细胞核定位RING-type的E3泛素连接酶。失活的GW2等位基因能增大细胞面积,过表达GW2造成粒型减少,表明GW2是一个水稻粒宽的负调控因子。GW2能与几丁质酶(CHT14)和磷酸甘油酸酯激酶(PGK)相互作用,并通过改变这两个蛋白的活性和稳定性影响水稻种子碳代谢途径[18]。另外,水稻去泛素化酶15(OsUBP15)能够与水稻的泛素受体(OsDA1)相互作用,遗传证据显示OsUBP15可能在功能上部分依赖GW2通过泛素化和去泛素化的动态变化调控下游底物蛋白量的变化[19]。另一个去泛素化酶WIDE AND THICK GRAIN 1(WTG1,同OsOTUB1和GWC1)能够正向调控颖壳细胞延伸和稻米品质。Heading and Grain Weight(HGW)编码一个能够与多种水稻种子发育和开花期相关蛋白共表达的新的核质共定位多聚泛素结合酶(UBA)[20]。拟南芥中发现将泛素连接酶E3和UBA突变后明显影响种子护颖及表皮的发育,表明这条泛素介导的蛋白酶体降解途径在植物种子发育过程非常保守。
2.2 植物激素调控途径植物激素参与了多种植物生长发育过程。到目前为止,几乎所有的植物激素(生长素、乙烯、赤霉素、细胞分裂素、油菜素内酯和茉莉酸等)均能调控水稻粒重,这些激素可能通过协同作用或拮抗作用调控水稻粒重。多种激素途径相关基因能够改变并产生多种水稻表型。
2.2.1 生长素 作为第一个被发现的植物激素,生长素(Auxin)的作用是促进细胞分化与分裂。生长素在包括种子生长、胚胎发育及配子体形成等植物多个发育阶段发挥重要作用。生长素极性运输(PAT)和生长素信号传导同样影响籽粒发育成熟过程[29]。Tillering and Small Grain 1(TSG1)是FISH BONE(FIB)基因的等位基因,编码一个色氨酸氨基转移酶[30]。tsg1突变体对外源吲哚乙酸敏感,在水稻的4个色氨酸氨基转移酶同源基因中,TSG1对生长素合成起主要作用。2018年3个课题组同时在Molecular Plant发表qTGW3/GL3.3/TGW3作为水稻新的粒重QTL负调控粒型的研究成果[11, 14-15]。这个QTL位点编码一个糖原合成酶激酶3,能够分别与生长素响应因子OsARF4和水稻主效粒型QTL GS3发生相互作用。OsARF多个基因参与水稻粒型调控作用。另一个水稻粒重的微效QTL GSA1编码一个UDP- 葡萄糖转移酶,通过影响类黄酮介导的生长素含量变化及相关基因表达量形成抗逆和产量的动态平衡关系[31]。除了上文提到的OsARF4影响水稻粒型外,Qiao等揭示一条全新的miR167a-OsARF6-OsAUX3调控水稻粒长和粒重的分子调控模块[32];OsARF25的T-DNA插入突变体表现出粒型变短的表型;稀穗基因Gnp4/LAX2能与OsIAA3互作干扰OsIAA3-OsARF25调控模块,进而影响下游OsERF142/SMOS1(Small Organ Size 1)基因表达[13]。OsARF11正调控水稻粒型及植物发育过程[33]。SMOS1和SMOS2分别编码一个生长素调控的APETALA2转录因子和与赤霉素相关的GRAS转录因子〔与DWARF AND LOW-TILLERING(DLT)基因相同〕。这其中是否存在更广义的互作及调控机制,有待进一步研究。
2.2.2 乙烯 作为唯一的气体植物激素,乙烯(Ethylene)在促进叶片衰老、水果成熟、种子休眠及种子的三重反应中发挥重要作用[34]。以“猫胡子”(MHZs)表型的乙烯相关的突变体揭示乙烯调控水稻发育过程的重要调控网络。MHZ3编码一个内质网(ER)定位蛋白。其蛋白N端C端能够与OsEIN2发生相互作用。过表达MHZ3可显著增加OsEIN2的数量和水稻种子大小[35]。EIN3水稻同源基因OsEIL/MHZ6也通过实验证明与水稻种子大小呈正相关[36]。过量表达乙烯受体ETR2导致水稻粒重减少,类似结果在百脉根(Medicago sativa L.)也得到验证[37]。这些结果表明水稻粒型与乙烯信号传导呈正相关关系。与其相关的一个水稻基因OsSPMS1编码一个热精胺合成酶,通过负调控乙烯前体1- 氨基环丙烷羧酸(ACC)含量调控水稻粒型[38]。Ahmadizadeh等总结和预测了拟南芥和水稻乙烯合成途径具有功能的编码基因,并且这些基因的启动子区顺式元件与光反应、缺水、细胞周期及水杨酸诱导相关。可以预期未来会有更多的乙烯相关基因与水稻粒重的调控关系被揭示[39]。
2.2.3 赤霉素 赤霉素(Gibberellins,GAs)由一系列具有共同结构的植物激素家族组成,对种子萌发、株高增加、叶形态建成、开花诱导等有重要促进作用[40]。Grain Number per Panicle 1(GNP1)编码GA 20氧化酶1(OsGAox1),参与水稻品种特青的穗粒数和产量的调控[41]。值得注意的是,其同源基因OsGAox2正是“第一次绿色革命”广泛应用的sd1基因[2]。Small and dwarf 2(sgd2)突变体表现出植株株高变矮及粒型减少等表型,通过正向定位发现该基因编码一个亮氨酸拉链Ⅱ(HD-Zip)家族转录因子,其通过转录水平调控多个GA合成相关基因的表达量,从而影响水稻粒型[42]。GAST家族基因编码具有一个保守的富半胱氨酸多肽,其表达量受到GA的诱导[43],迄今为止,3个GAST家族基因都被报道与水稻粒型相关,包括OsGASR9、GW6和OsGASR1[21, 44-45]。这些结果表明,GAST家族基因在调控GA通路和水稻穗部发育功能方面高度保守和一致。细叶基因Narrow Leaf 2和Narrow Leaf 3编码WUSCHEL同源盒3A(WOX3A)转录因子,能够特异性负调控GA合成和GA前体代谢物的动态平衡。过表达OsWOX3A基因能够模拟GA缺陷表型,包括株高变矮、叶片变细、穗粒数减少及粒型变小[46]。综上,GA信号调控途径能够显著影响水稻粒重相关表型。
2.2.4 细胞分裂素 植物激素细胞分裂素(Cytokinin,CK)能够促进细胞分裂和细胞分化,主要在植物根部、茎部、顶端优势、细胞衰老、果实成熟、生物和非生物胁迫等生长发育过程发挥重要作用。在CK合成过程中,异戊二酰转移酶(IPT)是重要的合成限速步骤,而细胞分裂素氧化酶(CKXs)调控CK降解。外源施加CK能够抑制CKXs表达,增加种子大小[47]。林鸿宣课题组报道一个水稻的盐旱耐受突变体dst能够增加水稻非生物胁迫抗性同时影响叶片宽度和水稻粒型。作为一个锌指转录因子,DST能够结合H2O2代谢相关基因启动子区调控水稻对于非生物胁迫响应。进一步研究发现,DST能够结合OsCKX2启动子调控OsCKX2在穗部表达。Glo等发现MAPK6能够直接结合DST并且对其磷酸化以激活OsCKX2的表达[48]。水稻中过表达IPT基因能够增加水稻产量[49]。OsSGL编码一个未知功能结构域(DUF1645)蛋白正向调控CK信号途径和水稻非生物胁迫。除此之外,对嘌呤渗透酶家族(OsPUPs)的研究表明其可以调控CK运输和改变种子大小和重量。OsPUP4(BG3)直接对远距离CK运输和本地分布产生影响[50]。以上结果表明,CK参与水稻的非生物胁迫响应和幼穗发育过程。
2.2.5 油菜素内酯 早在1979年,科学家就从油菜分离出一类与人类类固醇结构相近的化合物并且命名为油菜素内酯(Brassinosteroids,BRs)[51]。BR能够影响种子萌发、气孔形成、维管束分化、花粉育性及衰老等重要生理生化过程,特别是对株高、叶倾角、粒型及分蘖数等重要农艺性状产生显著影响。BR合成途径或者信号传导缺陷突变体,包括D11,OsBR6ox/BRD1,D2,BRD2等,均表现出株高变矮、穗长变短、叶片颜色变深、水稻粒型变小等表型[52]。突变体Slender grain Dominant(slg-D)表现出BR过量表型,包括植株松散、较大的叶倾角和更长水稻粒长[53]。GW5与一个糖原合成酶激酶2(GSK2)相互作用,通过对BR信号传导通路BZR1和DLT磷酸化调控BR介导的水稻粒型建成[22]。值得注意的是,GW5及其同源基因能够与BR信号通路关键成分GSK2和BIN2互作导致DLT和OsBZR1的磷酸化程度降低[54]。一个含有OVATE结构的家族蛋白成员(OFPs)被证明受到GSK2的磷酸化过程参与BR信号途径介导的水稻粒型调控。包括OFP1、OFP3、OFP8及OFP14等[55]。Yu等在对水稻OFP家族蛋白表达模式和亚细胞定位分析发现OFP15也是具有BR信号诱导表达的特征,可能也参与BR信号调控水稻株型和粒型性状[56]。
除了上述一直报道的BR信号成员调控水稻粒型外,仍有许多新的未知的分子机制参与BR信号介导的水稻粒型调控。G- 蛋白α亚基D1/RGA1对BR处理呈正相关,其功能失活突变体d1表现出株高变矮及小粒[57]。进一步研究发现D1/RGA1能够与一个U-BOX结构域的E3泛素连接酶(TUD1)发生相互作用,d1tud1-5双突变体表现出节间缩短、叶倾角减少、粒型变小等表型[58]。另一个大的转录因子家族GATA包括锌指结构域和DNA结合区域,对OsGATA7进行RNAi和基因敲除发现,转基因水稻对BR处理不敏感,而过量表达则表现出相反表型[59]。除了上述这些基因外,丝裂原活化蛋白激酶(MAPKs)与BR信号通路有很强的交互作用。SMALL GRAIN 1(SMG1)编码水稻OsMKK4基因,其突变体smg1表现出与D61/OsBRI1,OsBZR1,OsGSK2和DLT缺陷突变体类似的BR缺乏表型[60]。Dwarf and Small Grain 1(DSG1)编码OsMAPK6,其作为OsMKK4下游靶基因共同调控水稻粒型。MAPK上游OsMKKK10结合并且磷酸化OsMKK4调控水稻粒型大小。其功能失活突变体osmkkk10突变体表现出更小的水稻粒型和粒重。组成性过表达(CA-OsMKKK10)表现出穗型和粒型增大。GRAIN SIZE AND NUMBER 1(GSN1)能够对MAPK6进行去磷酸化修饰,参与GSN1-MAPK分子调控模块[61]。在MAPK调控途径最上游,类受体激酶(RLK)OsER1能够对OsMPKKK10进行磷酸化,通过MAPK信号调控途径影响DST[48]。除此之外,小G蛋白OsRac1也可以与OsMAPK6相互作用正向调控水稻粒型[62]。作为主要的调控水稻粒长QTL,qGL3/GL3.1编码一个Kelch重复丝/ 苏氨酸蛋白磷酸激酶OsPPKL1,其底物包括细胞周期蛋白Cyclin-T1;3和GSK3[12]。综上所述,BR信号途径、MAPK信号途径通过磷酸化修饰精确调控水稻粒型和穗型动态平衡。
2.3 G蛋白调控途径异源三聚体G蛋白(G-proteins)在植物和动物信号传导通路差异较大,由Gα、Gβ和Gγ组成,水稻中调控多种激素、干旱、抗病、侧根形成等发育过程[63]。水稻中G蛋白均已报道与穗部发育、粒型、灌浆速率及每穗粒数等直接相关。上文提到Gα(RGA1)蛋白通过调控BR信号途径影响水稻粒型。Gβ(RGB1)正调控水稻粒型和株型[64]。Gγ则包括RGG1、RGG2、GS3、GGC2及DEP1[65]。然而,虽然DEP1蛋白结构上类似GS3,但是DEP1的表型却与GS3相反,DEP1是正调控水稻粒型基因[66]。G- 蛋白下游靶基因包括编码OsMADS1基因等的转录活性影响水稻粒型和胚乳淀粉含量[67]。综上所述,G蛋白信号途径对水稻粒型影响起重要调控作用。
2.4 光合作用及胚乳发育调控途径颖壳大小及灌浆程度是影响水稻粒重和品质的重要因素。对于水稻粒型的有关综述已多次指出调控颖壳细胞伸长和分裂和胚乳发育相关基因对水稻粒重的影响[68]。QUASIMODO2(OsQUA2)编码一个果胶甲基转移酶,对胚乳细胞壁乳糖醛酸的甲基酯化有重要作用,支持了光合产物远距离运输的“源- 库”理论[69]。液泡转换酶OsINV2和OsINV3正向调控水稻淀粉组成和糖代谢过程,特别是OsINV3对水稻粒型正向调控[70]。OsSWEET4编码一个糖转运蛋白促进六碳糖从韧皮部向胚乳细胞的转运影响水稻粒重[26]。另外一个膜蛋白GRAIN-FILLING RATE 1(GFR1),通过与Rubisco小亚基相互作用调控水稻种子灌浆速率[25]。
胚乳颗粒的形成是一个复杂过程,胚乳主要由两种多聚物组成,直链淀粉和支链淀粉的比例影响稻米品质。多年来,大量稻米品质相关突变体如floury endosperm(FLOs)已经被揭示与胚乳正常形成、水稻粒重密切相关。包括FLO2、FLO7、FLO10、FLO11-2、FLO13及FLO19等,他们编码不同的基因在不同调控途径发挥作用,影响胚乳及正常灌浆[27]。此外,水稻细胞核Y家族对于水稻灌浆期调控非常重要。如OsNFYB1的RNAi转基因植株表现出垩白率增加,通过结合GCC盒ERF转录因子和OsYUC11等调控水稻灌浆期表型[10, 71-72];OsNF-YB1、OsNFYC12和OsbHLH144三者形成复合体通过减少OsYNF-YB1被26S蛋白酶体降解的数量维持OsNF-YB1的稳定性,OsNF-YC12也可以通过结合FLO6和OsGS1;3启动子区调控稻米品质[27]。除了上述这些基因外,OsNF-YC10、OsNF-YB9、OsNF-YC2、及OsNF-YC4参与水稻穗形态建成和粒型发育调控[73-75]。上述这些结果表明,细胞核Y家族蛋白调控水稻粒型和品质。
2.5 表观修饰调控途径表观修饰主要指不改变DNA序列的可遗传表型变化[76]。对组蛋白的包括H3和H4的甲基化、乙酰化修饰在水稻的功能研究最为透彻。作为第一个参与表观修饰调控的水稻粒型QTL,GW6a编码一个含有GNAT结构域组蛋白乙酰转移酶Osg1HAT1,过表达GW6a能增加颖壳细胞数量、籽粒灌浆速率、籽粒大小及千粒重;此外,Osg1HAT1也促进参与组蛋白H4乙酰化修饰[23]。通过酵母双杂交实验筛选,水稻DA1的同源蛋白HDR3能够与GW6a发生相互作用。HDR3是一个能够正向调控水稻籽粒大小的具有泛素结合活性的泛素受体蛋白,有趣的是,HDR3是通过增强GW6a泛素化水平来延缓而不是促进GW6a依赖26S蛋白酶体的降解,从而提高GW6a蛋白数量和酶活增强后者对下游靶基因的表达调控[77]。SDG725编码一个H3K36甲基转移酶,其功能缺失突变体展现出类似BR缺陷表型,包括植株矮化、叶倾角减少基小圆粒表型[78]。另一类PcG多梳蛋白也被验证通过H3甲基化调控影响胚乳发育状态。两个同源基因OsFIE1和OsFIE2对水稻粒宽、稻米品质等产生多种影响,特别是OsFIE2能够通过影响OsMADS6的H3K27me3甲基化程度影响水稻花器官发育过程[24, 79]。这些基因表明各类表观修过调控与水稻粒重有密切的调控关系。
2.6 microRNA调控途径microRNAs(miRNAs)是一类存在植物体内非编码RNA。经过多种miRNA加工酶加工后,其最终产物能够靶向结合某些特定基因序列调控他们的翻译起始或者促进这些基因降解。miRNA参与许多植物生长发育过程调控,包括生物胁迫及非生物胁迫反应、环境适应性及衰老等[80]。OsSPL14(IPA1)编码一个SPL家族转录因子,是重要的调控水稻茎秆和粒型的QTL。其受到水稻miR156调控[81-82]。OsSPL14基因通过结合水稻株型基因OsTB1和OsDEP1启动子区分别调控植物的株型和穗型。过表达miR156能显著减少OsSPL14的表达量而表现出矮化、茎杆变细及分蘖变少等表型。另外一个SHORT INTERNODES 1(OsSHI1)编码一个含有IGGH结构域的转录因子也可以与IPA1结合,相互拮抗调控水稻株型[83]。上述这些基因构建了以IPA1为中心的水稻株型和粒型的调控网络。SPL家族的另外一个成员OsSPL16编码一个正向调控水稻粒宽的QTL,同样受到miR156的调控,同时其作为转录因子调控结合GL7/GW7启动子区促进颖壳细胞伸长[16, 84-85]。OsSPL13是第一个通过全基因组关联分析(GWAS)找到的调控水稻粒重和产量的QTL[86]。其3’-UTR同样包含一个miR156的结合区域。此外,OsSPL13结合在Small and Round Seed 5(SRS5)基因的启动子区调控颖壳细胞大小。上述结果构建miR156-OsSPL13/GLW7-SRS5水稻粒型调控网络[17]。OsSPL18可以结合在OsDEP1启动子区,也受到OsmiR156k的靶向降解,表明一个新的OsmiR156k-OsSPL18-OsDEP1的调控网络参与水稻粒型建成[87]。OsSPL9通过直接激活RCN1调控水稻早期穗粒数原基发生[88]。Jiang等对水稻的SPL家族蛋白进行总结和分类,也揭示了另外8个SPL家族蛋白存在穗部表型[89]。
除了miR156调控水稻粒型穗型外,其他miRNA也参与了水稻粒型调控过程。OsmiR535是OsSPL7/12/16的靶向miRNA,下游对OsPIN1B、OsDEP1、OsLOG及OsSLR1的表达量调控[90]。OsmiR397调控OsLAC基因影响穗部的粒长、粒宽和千粒重[91]。OsmiR396通过影响Growth Regulating Factor(GRF)转录因子的表达调控水稻穗粒数和粒型关系,建立了OsmiR396-GRF-GIF调控网络[92]。Zhao等通过应用Short Tandem Target Mimic(STTM)方法阻断OsmiR159与靶基因的相互作用,发现多个MYB结构域基因,如OsGAMYB和OsGAMYBL1,表达量明显上调,进而对水稻株高、叶长和粒型进行调控[93]。OsmiR530分别靶向OsPIL15和OsPL3正调控水稻粒型[94]。上述结果说明,miRNA通过靶向与水稻粒重相关基因来影响水稻粒型。
3 结语与展望水稻粒型发育及调控是一个复杂过程,除了受到许多基因参与调控,环境因素包括营养、田间管理、种植区域等也有显著影响。本文综述总结了水稻粒重相关的表观调控途径、转录水平、蛋白质互作等分子调控过程,与光合作用、蔗糖运输等代谢过程及细胞分裂与分化细胞学层面调控过程。这些基因相互作用,有的形成调控网络发挥作用,有的仍然不清楚其具体功能。目前多个证据表明,水稻不同性状之间存在微妙的动态平衡[31, 48, 61]。本文从泛素化途径、G- 蛋白偶联途径、激素调控途径、光合作用调控途径、表观调控途径和micro-RNA调控途径列举和整理与水稻粒重相关的重要QTL和功能基因。由于合子(胚和胚乳)决定种子大小,在作物驯化过程受到强烈人工选择以优化种子大小与品种有益等位基因组合。不同物种在进化过程可能存在相似的调控机制[9]。比如拟南芥的DA1基因调控种子大小,在水稻中也有相似机制[19];拟南芥MAPK信号途径调控植物广谱抗病性分子机制,然而在水稻中主要在调控水稻粒型和穗粒数平衡[61]。此外,对于多基因调控网络的研究也大大加深我们对于水稻粒重这一复杂性状的分子机理了解。如OsmiR156k-OsSPL18-OsDEP1、OsmiR396-GRFGIF调控网络等[87, 92]。这些信号调控网络之间的相互影响和调控关系需要在未来投入更多的研究,包括多组学联合分析等以便能更加全面构筑水稻粒重调控网络,为未来实现真正意义上分子模块设计育种打下坚实理论基础。
基因编辑技术经过过去十多年的蓬勃发展,目前已经开发出TALEN系统、Cas9系统、Cas12系统以及单碱基编辑器ABE等[95]。值得注意的是,目前利用启动子编辑微调基因表达产生多种不同性状已有课题组出现相关报道。早在2017年,美国冷泉港Zachary B. Lippman课题组就提出利用多个不同基因家族成员的自然或者人工改良等位基因进行花序的改良,达到从野生番茄向人工番茄快速驯化的目的[96]。扬州大学刘巧泉课题组和华南农业大学刘耀光课题组分别利用CRISPRCas9技术对稻米品质基因Waxy基因进行启动子、5’-UTR及内含子区进行基因编辑,获得不同的新等位Waxy基因,改良水稻稻米品质[97-98]。最近,李钱锋课题组等对水稻Glucan, Water-Dikinase 1(GWD1)基因进行启动子编辑获得其弱突变的遗传改良材料,能够实现在不改变水稻主要农艺性状的同时对水稻稻米透明度和种子萌发状态的改良[99]。除了水稻稻米品质改良,对水稻细菌性条纹病抗病基因Xa13启动子编辑获得提高水稻细条病抗性但是并没有降低花粉的育种改良材料在以后水稻高产抗病育种过程的应用也是一个很好的例子[100]。这些都预示着对水稻粒重相关基因的启动子编辑具有重要的生物学和生产实践意义。
随着水稻基因组变异组学的发展,更多的不同水稻品种材料在单碱基SNP差异、序列变异(SV)差异为水稻育种和分子设计育种聚合不同优良性状改良提供新的思路[101]。这些在不同品种的水稻序列变异不仅挖掘更多新的有效等位基因,在育种生产上应用;另外也可能解决过去QTL定位出现的问题,新的变异可能隐藏着的新的功能性基因在水稻“四性”的作用值得重视。除了经典遗传学在水稻育种方面的应用,发展智慧农业,物联网和农村5G建设也是推动我国农业高质量发展,乡村振兴的重要举措。目前,华南地区以广东省农业科学院和地市政府联手打造的产学研结合基地,农村产业园、大田规模化种植基地、设施园艺标准示范基地等也在逐步建设,这将引领农业农村现代化在2025年、2035年不同时间取得阶段性进展。与之相配套的技术也在不断开发。比如,中国农业科学院都市农业研究所最近利用6种矮秆品种结合定制光谱LED光源为水稻不同生育期提供最佳光环境同时精确调控各种环境因素,实现对大田环境120 d生育期水稻压缩至60 d,并且实现相当于单产652 kg/667m2巨大应用潜力。2007年研发出水稻“三控”施肥技术,该技术是以控肥、控苗、控病虫为主要内容的高效施肥及配套技术体系,与传统施肥方法相比,一般减少氮肥20%,水稻增产10% 左右,氮肥利用率提高10% 以上,有效分蘖增多,无效分蘖减少,病虫害和倒伏大幅减轻,多年来的成功应用使得该技术已成为广东省和农业部主推技术[102-103]。除此之外,利用农机自动导航作业与无人旋耕机、播种机、插秧机、喷雾机和收获机的有机结合,华南农业大学罗锡文院士打造的“无人农场”是国家“智慧农业”示范的典型例子,争取“无人农场”5年后进入推广阶段、10年后加快推广速度,完成技术转化。这些技术的配套实施将极大有助于我们国家水稻生物育种、打造智慧农业,打赢种业翻身仗。
| [1] |
KUROHA T, ASHIKARI M. Molecular mechanisms and future improvement of submergence tolerance in rice[J]. Molecular Breeding, 2020, 40: 41. DOI:10.1007/s11032-020-01122-y |
| [2] |
QIAN Q, GUO L, SMITH S M, LI J. Breeding high-yield superior quality hybrid super rice by rational design[J]. National Science Review, 2016, 3(3): 283-294. DOI:10.1093/nsr/nww006 |
| [3] |
LI S, TIAN Y, WU K, YE Y, YU J, ZHANG J, LIU Q, HU M, LI H, TONG Y, HARBERD P N, FU X. Modulating plant growth-metabolism coordination for sustainable agriculture[J]. Nature, 2018, 560(7720): 595-600. DOI:10.1038/s41586-018-0415-5 |
| [4] |
FIAZ S, AHMAD S, NOOR M A, WANG X, YOUNAS A, RIAZ A, ALI F. Applications of the CRISPR/Cas9 system for rice grain quality improvement: perspectives and opportunities[J]. International Journal of Molecular Sciences, 2019, 20: 888. DOI:10.3390/ijms20040888 |
| [5] |
郭韬, 余泓, 邱杰, 李家洋, 韩斌, 林鸿宣. 中国水稻遗传学研究进展与分子设计育种[J]. 中国科学: 生命科学, 2019, 49: 1185-1212. DOI:10.1360/SSV-2019-0209 GUO T, YU H, QIU J, LI J Y, HAN B, LIN H X. Advances in rice genetics and breeding by molecular design in China[J]. Scientia Sinica Vitae, 2019, 49: 1185-1212. DOI:10.1360/SSV-2019-0209 |
| [6] |
张静, 李晨, 潘大建, 陈文丰, 孙炳蕊, 刘清, 吕树伟, 江立群, 毛兴学, 范芝兰. 水稻粒长遗传及其功能基因研究进展[J]. 广东农业科学, 2021, 48(3): 1-10. DOI:10.16768/j.issn.1004-874X.2021.03.001 ZHANG J, LI C, PAN D J, CHEN W F, SUN R B, LIU Q, LYU S W, JIANG L Q, MAO X X, FAN Z L. Advances in inheitance of grain length and its functional genes in rice[J]. Guangdong Agricultural Sciences, 2021, 48(3): 1-10. DOI:10.16768/j.issn.1004-874X.2021.03.001 |
| [7] |
HU X, WANG C, FU Y, LIU Q, JIAO X, WANG K. Expanding the range of CRISPR/Cas9 genome editing in rice[J]. Molecular Plant, 2016, 9(6): 943-945. DOI:10.1016/j.molp.2016.03.003 |
| [8] |
LI G, TANG J, ZHENG J, CHU C. Exploration of rice yield potential: Decoding agronomic and physiological traits[J]. The Crop Journal, 2021, 9(3): 577-589. DOI:10.1016/j.cj.2021.03.014 |
| [9] |
LI N, XU R, LI Y. Molecular networks of seed size control in plants[J]. Annual Review of Plant Biology, 2019, 70: 435-463. DOI:10.1146/annurev-arplant-050718-095851 |
| [10] |
SUN P, ZHANG W, WANG Y, HE Q, SHU F, LIU H, WANG J, WANG J, YUAN L, DENG H. OsGRF4 controls grain shape, panicle length and seed shattering in rice[J]. Journal of Integrative Plant Biology, 2016, 58(10): 836-847. DOI:10.1111/jipb.12473 |
| [11] |
XIA D, ZHOU H, LIU R, DAN W, LI P, WU B, CHEN J, WANG L, GAO G, ZHANG Q, HE Y. GL3.3, a novel QTL encoding a GSK3/ SHAGGY-like kinase, epistatically interacts with GS3 to produce extra-long grains in rice[J]. Molecular Plant, 2018, 11(5): 754-756. DOI:10.1016/j.molp.2018.03.006 |
| [12] |
GAO X, ZHANG J Q, ZHANG X, ZHOU J, JIANG Z, HUANG P, TANG Z, BAO Y, CHENG J, TANG H, ZHANG W, ZHANG H, HUANG J. Rice qGL3/OsPPKL1 functions with the GSK3/SHAGGY-Like kinase OsGSK3 to modulate brassinosteroid signaling[J]. Plant Cell, 2019, 31(5): 1077-1093. DOI:10.1105/tpc.18.00836 |
| [13] |
ZHANG Z, LI J, TANG Z, SUN X, ZHANG H, YU J, YAO G, LI G, GUO H, LI J, WU H, HUANG H, XU Y, YIN Z, QI Y, HUANG R, YANG W, LI Z. Gnp4/LAX2, a RAWUL protein, interferes with the OsIAA3-OsARF25 interaction to regulate grain length via the auxin signaling pathway in rice[J]. Journal of Experimental Botany, 2018, 69(20): 4723-4737. DOI:10.1093/jxb/ery256 |
| [14] |
HU Z, LU S J, WANG M J, HE H, SUN L, WANG H, LIU X H, JIANG L, SUN J L, XIN X, KONG W, CHU C, XUE H, YANG J, LUO X, LIU J. A novel QTL qTGW3 encodes the GSK3/SHAGGY-Like Kinase OsGSK5/OsSK41 that interacts with OsARF4 to negatively regulate grain size and weight in rice[J]. Molecular Plant, 2018, 11(5): 736-749. DOI:10.1016/j.molp.2018.03.005 |
| [15] |
YING J Z, MA M, BAI C, HUANG X H, LIU J L, FAN Y Y, SONG X J. TGW3, a major QTL that negatively modulates grain length and weight in rice[J]. Molecular Plant, 2018, 11(5): 750-753. DOI:10.1016/j.molp.2018.03.007 |
| [16] |
WANG S, LI S, LIU Q, WU K, ZHANG J, WANG S, WANG Y, CHEN X, ZHANG Y, GAO C, WANG F, HUANG H, FU X. The OsSPL16-GW7 regulatory module determines grain shape and simultaneously improves rice yield and grain quality[J]. Nature Genetics, 2015, 47(8): 949-954. DOI:10.1038/ng.3352 |
| [17] |
SEGAMI S, TAKEHARA K, YAMAMOTO T, KIDO S, KONDO S, IWASAKI Y, MIURA K. Overexpression of SRS5 improves grain size of brassinosteroid-related dwarf mutants in rice(Oryza sativa L.)[J]. Breeding Science, 2017, 67(4): 393-397. DOI:10.1270/jsbbs.16198 |
| [18] |
LEE K H, PARK S W, KIM Y J, KOO Y J, SONG J T, SEO H S. Grain width 2(GW2)and its interacting proteins regulate seed development in rice(Oryza sativa L.)[J]. Botanical Studies, 2018, 59(1): 23. DOI:10.1186/s40529-018-0240-z |
| [19] |
SHI C, REN Y, LIU L, WANG F, ZHANG H, TIAN P, PAN T, WANG Y, JING R, LIU T, WU F, LIN Q, LEI C, ZHANG X, ZHU S, GUO X, WANG J, ZHAO Z, WANG J, ZHAI H, CHENG Z, WAN J. Ubiquitin specific protease 15 has an important role in regulating grain width and size in rice[J]. Plant Physiology, 2019, 180(1): 381-391. DOI:10.1104/pp.19.00065 |
| [20] |
HUANG K, WANG D, DUAN P, ZHANG B, XU R, LI N, LI Y. WIDE AND THICK GRAIN 1, which encodes an otubain-like protease with deubiquitination activity, influences grain size and shape in rice[J]. Plant Journal, 2017, 91(5): 849-860. DOI:10.1111/tpj.13613 |
| [21] |
SHI C L, DONG N Q, GUO T, YE W W, SHAN J X, LIN H X. A quantitative trait locus GW6 controls rice grain size and yield through the gibberellin pathway[J]. Plant Journal, 2020, 103(3): 1174-1188. DOI:10.1111/tpj.14793 |
| [22] |
LIU J, CHEN J, ZHENG X, WU F, LIN Q, HENG Y, TIAN P, CHENG Z, YU X, ZHOU K, ZHANG X, GUO X, WANG J, WANG H, WAN J. GW5 acts in the brassinosteroid signalling pathway to regulate grain width and weight in rice[J]. Nature Plants, 2017, 3: 17043. DOI:10.1038/nplants.2017.43 |
| [23] |
SONG X J, KUROHA T, AYANO M, FURUTA T, NAGAI K, KOMEDA N, SEGAMI S, MIURA K, OGAWA D, KAMURA T, SUZUKI T, HIGASHIYAMA T, YAMASAKI M, MORI H, INUKAI Y, WU J, KITANO H, SAKAKIBARA H, JACOBSEN S E, ASHIKARI M. Rare allele of a previously unidentified histone H4 acetyltransferase enhances grain weight, yield, and plant biomass in rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(1): 76-81. DOI:10.1073/pnas.1421127112 |
| [24] |
CHENG X, PAN M, E Z, ZHOU Y, NIU B, CHEN C. Functional divergence of two duplicated fertilization independent endosperm genes in rice with respect to seed development[J]. Plant Journal, 2020, 104(1): 124-137. DOI:10.1111/tpj.14911 |
| [25] |
LIU E, ZENG S, ZHU S, LIU Y, WU G, ZHAO K, LIU X, LIU Q, DONG Z, DANG X, XIE H, LI D, HU X, HONG D. Favorable alleles of GRAIN-FILLING RATE1 increase the grain-filling rate and yield of rice[J]. Plant Physiology, 2019, 181(3): 1207-1222. DOI:10.1104/pp.19.00413 |
| [26] |
SOSSO D, LUO D, LI Q B, SASSE J, YANG J, GENDROT G, SUZUKI M, KOCH K E, MCCARTY D R, CHOUREY P S, ROGOWSKY P M, ROSS-IBARRA J, YANG B, FROMMER W B. Seed filling in domesticated maize and rice depends on SWEET-mediated hexose transport[J]. Nature Genetics, 2015, 47(12): 1489-1493. DOI:10.1038/ng.3422 |
| [27] |
CHEN K, LYSKOWSKI A, JAREMKO L, JAREMKO M. Genetic and molecular factors determining grain weight in rice[J]. Frontiers in Plant Science, 2021, 12: 605799. DOI:10.3389/fpls.2021.605799 |
| [28] |
胡婷婷, 王健康, 丁成伟, 郭荣良, 吴玉玲, 徐家安, 王友霜, 赵轶鹏, 何弯弯. 植物种子的发育调控研究进展[J]. 中国农业科技导报, 2021, 23(5): 27-34. DOI:10.13304/j.nykjdb.2020.0048 HU T T, WANG J K, DING C W, GUO R L, WU Y L, XU J A, WANG Y S, ZHAO Y P, HE W W. Progress on regulation mechanism of plant seed development[J]. Journal of Agricultural Science and Technology, 2021, 23(5): 27-34. DOI:10.13304/j.nykjdb.2020.0048 |
| [29] |
WANG Y, ZHANG T, WANG R, ZHAO Y. Recent advances in auxin research in rice and their implications for crop improvement[J]. Journal of Experimental Botany, 2018, 69(2): 255-263. DOI:10.1093/jxb/erx228 |
| [30] |
GUO T, CHEN K, DONG N Q, YE W W, SHAN J X, LIN H X. Tillering and small grain 1 dominates the tryptophan aminotransferase family required for local auxin biosynthesis in rice[J]. Journal of Integrative Plant Biology, 2020, 62(5): 581-600. DOI:10.1111/jipb.12820 |
| [31] |
DONG N Q, SUN Y, GUO T, SHI C L, ZHANG YM, KAN Y, XIANG Y H, ZHANG H, YANG Y B, LI Y C, ZHAO H Y, YU H X, LU Z Q, WANG Y, YE W W, SHAN J X, LIN H X. UDP-glucosyltransferase regulates grain size and abiotic stress tolerance associated with metabolic flux redirection in rice[J]. Nature Communications, 2020, 11(1): 2629. DOI:10.1038/s41467-020-16403-5 |
| [32] |
QIAO J, JIANG H, LIN Y, SHANG L, WANG M, LI D, FU X, GEISLER M, QI Y, GAO Z, QIAN Q. A novel miR167a-OsARF6- OsAUX3 module regulates grain length and weight in rice[J]. Molecular Plant, 2021, S1674-2052(21): 00235-5. DOI:10.1016/j.molp.2021.06.023 |
| [33] |
SIMS K, ABEDI-SAMAKUSH F, SZULC N, MACIAS H M G, MATTSSON J. OsARF11 promotes growth, meristem, seed, and vein formation during rice plant development[J]. International Journal of Molecular Sciences, 2021, 22(8). DOI:10.3390/ijms22084089 |
| [34] |
YIN C C, ZHAO H, MA B, CHEN S Y, ZHANG J S. Diverse roles of ethylene in regulating agronomic traits in rice[J]. Frontiers in Plant Science, 2017, 8: 1676. DOI:10.3389/fpls.2017.01676 |
| [35] |
MA B, ZHOU Y, CHEN H, HE S J, HUANG Y H, ZHAO H, LU X, ZHANG W K, PANG J H, CHEN S Y, ZHANG J S. Membrane protein MHZ3 stabilizes OsEIN2 in rice by interacting with its Nramp-like domain[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(10): 2520-2525. DOI:10.1073/pnas.1718377115 |
| [36] |
YANG C, MA B, HE S J, XIONG Q, DUAN K X, YIN C C, CHEN H, LU X, CHEN S Y, ZHANG J S. MAOHUZI6/ ETHYLENE INSENSITIVE3-LIKE1 and ETHYLENE INSENSITIVE3-LIKE2 regulate ethylene response of roots and coleoptiles and negatively affect salt tolerance in rice[J]. Plant Physiology, 2015, 169(1): 148-165. DOI:10.1104/pp.15.00353 |
| [37] |
WANG Y, DIAO P, KONG L, YU R, ZHANG M, ZUO T, FAN Y, NIU Y, YAN F, WURIYANGHAN H. Ethylene enhances seed germination and deedling growth under salinity by reducing oxidative stress and promoting chlorophyll content via ETR2 pathway[J]. Frontiers in Plant Science, 2020, 11: 1066. DOI:10.3389/fpls.2020.01066 |
| [38] |
TAO Y, WANG J, MIAO J, CHEN J, WU S, ZHU J, ZHANG, D, GU H, CUI H, SHI S, XU M, YAO Y, GONG Z, YANG Z, GU M, ZHOU Y, LIANG G. The spermine synthase OsSPMS1 regulates seed germination, grain size, and yield[J]. Plant Physiology, 2018, 178(4): 1522-1536. DOI:10.1104/pp.18.00877 |
| [39] |
AHMADIZADEH M, CHEN J T, HASANZADEH S, AHMAR S, HEIDARI P. Insights into the genes involved in the ethylene biosynthesis pathway in Arabidopsis thaliana and Oryza sativa[J]. Journal of Genetic Engineering and Biotechnology, 2020, 18(1): 62. DOI:10.1186/s43141-020-00083-1 |
| [40] |
KWON C T, PAEK N C. Gibberellic acid: a key phytohormone for spikelet fertility in rice grain production[J]. International Journal of Molecular Sciences, 2016, 17(5). DOI:10.3390/ijms17050794 |
| [41] |
WU Y, WANG Y, MI X F, SHAN J X, LI X M, XU J L, LIN H X. The QTL GNP1 encodes GA20ox1, which increases grain number and yield by increasing cytokinin activity in rice panicle meristems[J]. PLoS Genetics, 2016, 12(10): e1006386. DOI:10.1371/journal.pgen.1006386 |
| [42] |
CHEN W, CHENG Z, LIU L, WANG M, YOU X, WANG J, ZHANG F, ZHOU C, ZHANG Z, ZHANG H, YOU S, WANG Y, LUO S, ZHANG J, WANG J, WANG J, ZHAO Z, GUO X, LEI C, ZHANG X, LIN Q, REN Y, ZHU S, WAN J. Small Grain and Dwarf 2, encoding an HD-Zip Ⅱ family transcription factor, regulates plant development by modulating gibberellin biosynthesis in rice[J]. Plant Science, 2019, 288: 110208. DOI:10.1016/j.plantsci.2019.110208 |
| [43] |
KUMAR A, SINGH A, KUMAR P, SARKAR A K. Giberellic acidstimulated transcript proteins evolved through successive conjugation of novel motifs and their subfunctionalization[J]. Plant Physiology, 2019, 180(2): 998-1012. DOI:10.1104/pp.19.00305 |
| [44] |
LEE S C, KIM S J, HAN S K, AN G, KIM S R. A gibberellinstimulated transcript, OsGASR1, controls seedling growth and alphaamylase expression in rice[J]. Journal of Plant Physiology, 2017, 214: 116-122. DOI:10.1016/j.jplph.2017.04.010 |
| [45] |
LI X, SHI S, TAO Q, TAO Y, MIAO J, PENG X, LI C, YANG Z, ZHOU Y, LIANG G. OsGASR9 positively regulates grain size and yield in rice (Oryza sativa)[J]. Plant Science, 2019, 286: 17-27. DOI:10.1016/j.plantsci.2019.03.008 |
| [46] |
CHO S H, KANG K, LEE S H, LEE I J, PAEK N C. OsWOX3A is involved in negative feedback regulation of the gibberellic acid biosynthetic pathway in rice(Oryza sativa)[J]. Journal of Experimental Botany, 2016, 67(6): 1677-1687. DOI:10.1093/jxb/erv559 |
| [47] |
JAMESON P E, SONG J. Cytokinin: a key driver of seed yield[J]. Journal of Experimental Botany, 2016, 67(3): 593-606. DOI:10.1093/jxb/erv461 |
| [48] |
GUO T, LU Z Q, SHAN J X, Y E, W W, DONG NQ, LIN H X. ERECTA1 acts upstream of the OsMKKK10-OsMKK4-OsMPK6 cascade to control spikelet number by regulating cytokinin metabolism in rice[J]. Plant Cell, 2020, 32(9): 2763-2779. DOI:10.1105/tpc.20.00351 |
| [49] |
PELEG Z, REGUERA M, TUMIMBANG E, WALIA H, BLUMWALD E. Cytokinin-mediated source/sink modifications improve drought tolerance and increase grain yield in rice under water-stress[J]. Plant Biotechnology Journal, 2011, 9(7): 747-758. DOI:10.1111/j.1467-7652.2010.00584.x |
| [50] |
XIAO Y, LIU D, ZHANG G, GAO S, LIU L, XU F, CHE R, WANG Y, TONG H, CHU C. Big Grain3, encoding a purine permease, regulates grain size via modulating cytokinin transport in rice[J]. Journal of Integrative Plant Biology, 2019, 61(5): 581-597. DOI:10.1111/jipb.12727 |
| [51] |
SALVI P, MANNA M, KAUR H, THAKUR T, GANDASS N, BHATT D, MUTHAMILARASAN M. Phytohormone signaling and crosstalk in regulating drought stress response in plants[J]. Plant Cell Reports, 2021, 40(8): 1305-1329. DOI:10.1007/s00299-021-02683-8 |
| [52] |
ZHANG C, BAI M Y, CHONG K. Brassinosteroid-mediated regulation of agronomic traits in rice[J]. Plant Cell Reports, 2014, 33(5): 683-696. DOI:10.1007/s00299-014-1578-7 |
| [53] |
FENG Z, WU C, WANG C, ROH J, ZHANG L, CHEN J, ZHANG S, ZHANG H, YANG C, HU J, YOU X, LIU X, YANG X, GUO X, ZHANG X, WU F, TERZAGHI W, KIM S, JIANG L, WAN J. SLG controls grain size and leaf angle by modulating brassinosteroid homeostasis in rice[J]. Journal of Experimental Botany, 2016, 67(14): 4241-4253. DOI:10.1093/jxb/erw204 |
| [54] |
TIAN P, LIU J, MOU C, SHI C, ZHANG H, ZHAO Z, LIN Q, WANG J, WANG J, ZHANG X, GUO X, CHENG Z, ZHU S, REN Y, LEI C, WANG H, WAN J. GW5-Like, a homolog of GW5, negatively regulates grain width, weight and salt resistance in rice[J]. Journal of Integrative Plant Biology, 2019, 61(11): 1171-1185. DOI:10.1111/jipb.12745 |
| [55] |
YANG C, SHEN W, HE Y, TIAN Z, LI J. OVATE family protein 8 positively mediates brassinosteroid signaling through interacting with the GSK3-like kinase in rice[J]. PLoS Genetics, 2016, 12(6): e1006118. DOI:10.1371/journal.pgen.1006118 |
| [56] |
YU H, JIANG W, LIU Q, ZHANG H, PIAO M, CHEN Z, BIAN M. Expression pattern and subcellular localization of the ovate protein family in rice[J]. PLoS One, 2015, 10(3): e0118966. DOI:10.1371/journal.pone.0118966 |
| [57] |
WANG L, XU Y Y, MA Q B, LI D, XU Z H, CHONG K. Heterotrimeric G protein α subunit is involved in rice brassinosteroid response[J]. Cell Research, 2006, 16(12): 916-922. DOI:10.1038/sj.cr.7310111 |
| [58] |
HU X, QIAN Q, XU T, ZHANG Y, DONG G, GAO T, XIE Q, XUE Y. The U-box E3 ubiquitin ligase TUD1 functions with a heterotrimeric G alpha subunit to regulate Brassinosteroid-mediated growth in rice[J]. PLoS Genetics, 2013, 9(3): e1003391. DOI:10.1371/journal.pgen.1003391 |
| [59] |
ZHANG Y J, ZHANG Y, ZHANG L L, HUANG H Y, YANG B J, LUAN S, XUE H W, LIN W H. OsGATA7 modulates brassinosteroidsmediated growth regulation and influences architecture and grain shape[J]. Plant Biotechnol Journal, 2018, 16(7): 1261-1264. DOI:10.1111/pbi.12887 |
| [60] |
DUAN P, RAO Y, ZENG D, YANG Y, XU R, ZHANG B, DONG G, QIAN Q, LI Y. SMALL GRAIN 1, which encodes a mitogen-activated protein kinase kinase 4, influences grain size in rice[J]. Plant Journal, 2014, 77(4): 547-557. DOI:10.1111/tpj.12405 |
| [61] |
GUO T, CHEN K, DONG N Q, SHI C L, YE W W, GAO J P, SHAN J X, LIN H X. GRAIN SIZE AND NUMBER1 negatively regulates the OsMKKK10-OsMKK4-OsMPK6 cascade to coordinate the tradeoff between grain number per panicle and grain size in rice[J]. Plant Cell, 2018, 30(4): 871-888. DOI:10.1105/tpc.17.00959 |
| [62] |
ZHANG Y, XIONG Y, LIU R, XUE H W, YANG Z. The Rho-family GTPase OsRac1 controls rice grain size and yield by regulating cell division[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(32): 16121-16126. DOI:10.1073/pnas.1902321116 |
| [63] |
CUI Y, JIANG N, XU Z, XU Q. Heterotrimeric G protein are involved in the regulation of multiple agronomic traits and stress tolerance in rice[J]. BMC Plant Biology, 2020, 20(1): 90. DOI:10.1186/s12870-020-2289-6 |
| [64] |
X U, Q, ZHAO M, WU K, FU X, LIU Q. Emerging insights into heterotrimeric G protein signaling in plants[J]. Journal of Genetics and Genomics, 2016, 43(8): 495-502. DOI:10.1016/j.jgg.2016.06.004 |
| [65] |
MIAO J, YANG Z, ZHANG D, WANG Y, XU M, ZHOU L, WANG J, WU S, YAO Y, DU X, GU F, GONG Z, GU M, LIANG G, ZHOU Y. Mutation of RGG2, which encodes a type B heterotrimeric G protein γ subunit, increases grain size and yield production in rice[J]. Plant Biotechnology Journal, 2019, 17(3): 650-664. DOI:10.1111/pbi.13005 |
| [66] |
SUN S, WANG L, MAO H, SHAO L, LI X, XIAO J, OUYANG Y, ZH AN, Q. A G-protein pathway determines grain size in rice[J]. Nature Communications, 2018, 9(1): 851. DOI:10.1038/s41467-018-03141-y |
| [67] |
LIU Q, HAN R, WU K, ZHANG J, YE Y, WANG S, CHEN J, PAN Y, LI Q, XU X, ZHOU J, TAO D, WU Y, FU X. G-protein betagamma subunits determine grain size through interaction with MADS-domain transcription factors in rice[J]. Nature Communications, 2018, 9(1): 852. DOI:10.1038/s41467-018-03047-9 |
| [68] |
LI N, XU R, DUAN P, LI Y. Control of grain size in rice[J]. Plant Reproduction, 2018, 31(3): 237-251. DOI:10.1007/s00497-018-0333-6 |
| [69] |
XU Y, SECHET J, WU Y, FU Y, ZHU L, LI J, ZHANG Y, GINEAU E, GAERTNER C, ZHOU J, FAN X, LIU Y, ZHOU L, MOUILLE G, LIN X. Rice sucrose partitioning mediated by a putative pectin methyltransferase and homogalacturonan methylesterification[J]. Plant Physiology, 2017, 174(3): 1595-1608. DOI:10.1104/pp.16.01555 |
| [70] |
DENG X, HAN X, YU S, LIU Z, GUO D, HE Y, LI W, TAO Y, SUN C, XU P, LIAO Y, CHEN X, ZHANG H, WU X. OsINV3 and its homolog, OsINV2, control grain size in rice[J]. International Journal of Molecular Sciences, 2020, 21(6): 2199. DOI:10.3390/ijms21062199 |
| [71] |
BAI A N, LU X D, LI D Q, LIU J X, LIU C M. NF-YB1-regulated expression of sucrose transporters in aleurone facilitates sugar loading to rice endosperm[J]. Cell Research, 2016, 26(3): 384-388. DOI:10.1038/cr.2015.116 |
| [72] |
XU J J, ZHANG X F, XUE H W. Rice aleurone layer specific OsNFYB1 regulates grain filling and endosperm development by interacting with an ERF transcription factor[J]. Journal of Experimental Botany, 2016, 67(22): 6399-6411. DOI:10.1093/jxb/erw409 |
| [73] |
KIM S K, PARK H Y, JA NG, Y H, LEE K C, CHUNG Y S, LEE J H, KIM J K. OsNF-YC2 and OsNF-YC4 proteins inhibit flowering under long-day conditions in rice[J]. Planta, 2016, 243(3): 563-576. DOI:10.1007/s00425-015-2426-x |
| [74] |
DAS S, PARIDA S K, AGARWAL P, TYAGI A K. Transcription factor OsNF-YB9 regulates reproductive growth and development in rice[J]. Planta, 2019, 250(6): 1849-1865. DOI:10.1007/s00425-019-03268-2 |
| [75] |
JIA S, XIONG Y, XIAO P, WANG X, YAO J. OsNF-YC10, a seed preferentially expressed gene regulates grain width by affecting cell proliferation in rice[J]. Plant Science, 2019, 280: 219-227. DOI:10.1016/j.plantsci.2018.09.021 |
| [76] |
DUPONT C, ARMANT D R, BRENNER C A. Epigenetics: definition, mechanisms and clinical perspective[J]. Seminars in Reproductive Medicine, 2009, 27(5): 351-357. DOI:10.1055/s-0029-1237423 |
| [77] |
GAO Q, ZHANG N, WANG W Q, SHEN S Y, BAI C, SONG X J. The ubiquitin-interacting motif-type ubiquitin receptor HDR3 interacts with and stabilizes the histone acetyltransferase GW6a to control grain size in rice[J]. Plant Cell, 2021. DOI:10.1093/plcell/koab194 |
| [78] |
SUI P, JIN J, YE S, MU C, GAO J, FENG H, SHEN W H, YU Y, DONG A. H3K36 methylation is critical for brassinosteroid-regulated plant growth and development in rice[J]. Plant Journal, 2012, 70(2): 340-347. DOI:10.1111/j.1365-313X.2011.04873.x |
| [79] |
DHATT B K, PAUL P, SANDHU J, HUSSAIN W, IRVIN L, ZHU F, ADVIENTO-BORBE M A, LORENCE A, STASWICK P, YU H, MOROTA G, WALIA H. Allelic variation in rice Fertilization Independent Endosperm 1 contributes to grain width under high night temperature stress[J]. New Phytologist, 2021, 229(1): 335-350. DOI:10.1111/nph.16897 |
| [80] |
TANG J, CHU C. MicroRNAs in crop improvement: fine-tuners for complex traits[J]. Nature Plants, 2017, 3: 17077. DOI:10.1038/nplants.2017.77 |
| [81] |
JIAO Y, WANG Y, XUE D, WANG J, YAN M, LIU G, DONG G, ZENG D, LU Z, ZHU X, QIAN Q, LI J. Regulation of OsSPL14 by OsmiR156 defines ideal plant architecture in rice[J]. Nature Genetics, 2010, 42(6): 541-544. DOI:10.1038/ng.591 |
| [82] |
MIURA K, IKEDA M, MATSUBARA A, SONG X J, ITO M, ASANO K, MATSUOKA M, KITANO H, ASHIKARI M. OsSPL14 promotes panicle branching and higher grain productivity in rice[J]. Nature Genetics, 2010, 42(6): 545-549. DOI:10.1038/ng.592 |
| [83] |
DUAN E, WANG Y, LI X, LIN Q, ZHANG T, WANG Y, ZHOU C, ZHANG H, JIANG L, WANG J, LEI C, ZHANG X, GUO X, WANG H, WAN J. OsSHI1 regulates plant architecture through modulating the transcriptional activity of IPA1 in rice[J]. Plant Cell, 2019, 31(5): 1026-1042. DOI:10.1105/tpc.19.00023 |
| [84] |
WANG S, WU K, YUAN Q, LIU X, LIU Z, LIN X, ZENG R, ZHU H, DONG G, QIAN Q, ZHANG G, FU X. Control of grain size, shape and quality by OsSPL16 in rice[J]. Nature Genetics, 2012, 44(8): 950-954. DOI:10.1038/ng.2327 |
| [85] |
WANG Y, XIONG G, HU J, JIANG L, YU H, XU J, FANG Y, ZENG L, XU E, XU J, YE W, MENG X, LIU R, CHEN H, JING Y, WANG Y, ZHU X, LI J, QIAN Q. Copy number variation at the GL7 locus contributes to grain size diversity in rice[J]. Nature Genetics, 2015, 47(8): 944-948. DOI:10.1038/ng.3346 |
| [86] |
SI L, CHEN J, HUANG X, GONG H, LUO J, HOU Q, ZHOU T, LU T, ZHU J, SHANGGUAN Y, CHEN E, GONG C, ZHAO Q, JING Y, ZHAO Y, LI Y, CUI L, FAN D, LU Y, WENG Q, WANG Y, ZHAN Q, LIU K, WEI X, AN K, AN G, BIN H. OsSPL13 controls grain size in cultivated rice[J]. Nature Genetics, 2016, 48(4): 447-456. DOI:10.1038/ng.3518 |
| [87] |
YUAN H, QIN P, HU L, ZHAN S, WANG S, GAO P, LI J, JIN M, XU Z, GAO Q, DU A, TU B, CHEN W, MA B, WANG Y, LI S. OsSPL18 controls grain weight and grain number in rice[J]. Journal of Genetics and Genomics, 2019, 46(1): 41-51. DOI:10.1016/j.jgg.2019.01.003 |
| [88] |
HU L, CHEN W, YANG W, LI X, ZHANG C, ZHANG X, ZHENG L, ZHU X, YIN J, QIN P, WANG Y, MA B, LI S, YUAN H, TU B. OsSPL9 regulates grain number and grain yield in rice[J]. Frontiers in Plant Science, 2021, 12: 682018. DOI:10.3389/fpls.2021.682018 |
| [89] |
JIANG M, HE Y, CHEN X, ZHANG X, GUO Y, YANG S, HUANG J, TRAW M B. CRISPR-based assessment of genomic structure in the conserved SQUAMOSA promoter-binding-like gene clusters in rice[J]. Plant Journal, 2020, 104(5): 1301-1314. DOI:10.1111/tpj.15001 |
| [90] |
SUN M, SHEN Y, LI H, YANG J, CAI X, ZHENG G, ZHU Y, JIA B, SUN X. The multiple roles of OsmiR535 in modulating plant height, panicle branching and grain shape[J]. Plant Science, 2019, 283: 60-69. DOI:10.1016/j.plantsci.2019.02.002 |
| [91] |
ZHANG Y C, YU Y, WANG C Y, LI Z Y, LIU Q, XU J, LIAO J Y, WANG X J, QU L H, CHEN F, XIN P, YAN C, CHU J, LI H Q, CHEN Y Q. Overexpression of microRNA OsmiR397 improves rice yield by increasing grain size and promoting panicle branching[J]. Nature Biotechnology, 2013, 31(9): 848-852. DOI:10.1038/nbt.2646 |
| [92] |
TSUKAYA H. Yield increase: GRFs provide the key[J]. Nature Plants, 2015, 2: 15210. DOI:10.1038/nplants.2015.210 |
| [93] |
ZHAO Y, WEN H, TEOTIA S, DU Y, ZHANG J, LI J, SUN H, TANG G, PENG T, ZHAO Q. Suppression of microRNA159 impacts multiple agronomic traits in rice(Oryza sativa L.)[J]. BMC Plant Biology, 2017, 17(1): 215. DOI:10.1186/s12870-017-1171-7 |
| [94] |
SUN W, XU X H, LI Y, XIE L, HE Y, LI W, LU X, SUN H, XIE X. OsmiR530 acts downstream of OsPIL15 to regulate grain yield in rice[J]. New Phytologist, 2020, 226(3): 823-837. DOI:10.1111/nph.16399 |
| [95] |
CHEN K, WANG Y, ZHANG R, ZHANG H, GAO C. CRISPR/Cas genome editing and precision plant breeding in agriculture[J]. Annual Review of Plant Biology, 2019, 70: 667-697. DOI:10.1146/annurevarplant-050718-100049 |
| [96] |
SOYK S, LEMMON Z H, OVED M, FISHER J, LIBERATORE K L, PARK S J, GOREN A, JIANG K, RAMOS A, VAN DER KNAAP E, ECK J V, ZAMIR D, ESHED Y, LIPPMAN Z B. Bypassing negative epistasis on yield in tomato imposed by a domestication gene[J]. Cell, 2017, 169(6): 1142-1155. DOI:10.1016/j.cell.2017.04.032 |
| [97] |
HUANG L, LI Q, ZHANG C, CHU R, GU Z, TAN H, ZHAO D, FAN X, LIU Q. Creating novel Wx alleles with fine-tuned amylose levels and improved grain quality in rice by promoter editing using CRISPR/Cas9 system[J]. Plant Biotechnology Journal, 2020, 18(11): 2164-2166. DOI:10.1111/pbi.13391 |
| [98] |
ZENG D, LIU T, MA X, WANG B, ZHENG Z, ZHANG Y, XIE X, YANG B, ZHAO Z, ZHU Q, LIU Y. Quantitative regulation of Waxy expression by CRISPR/Cas9-based promoter and 5'UTR-intron editing improves grain quality in rice[J]. Plant Biotechnology Journal, 2020, 18(12): 2385-2387. DOI:10.1111/pbi.13427 |
| [99] |
WANG Z, WEI K, XIONG M, WANG J D, ZHANG C Q, FAN X L, HUANG L C, ZHAO D S, LIU Q Q, LI Q F. Glucan, Water-Dikinase 1 (GWD1), an ideal biotechnological target for potential improving yield and quality in rice[J]. Plant Biotechnology Journal, 2021. DOI:10.1111/pbi.13686 |
| [100] |
LI C, LI W, ZHOU Z, CHEN H, XIE C, LIN Y. A new rice breeding method: CRISPR/Cas9 system editing of the Xa13 promoter to cultivate transgene-free bacterial blight-resistant rice[J]. Plant Biotechnology Journal, 2020, 18(2): 313-315. DOI:10.1111/pbi.13217 |
| [101] |
QIN P, LU H, DU H, WANG H, CHEN W, CHEN Z, HE Q, OU S, ZHANG H, LI X, LI X, LI Y, LIAO Y, GAO Q, TU B, YUAN H, MA B, WANG Y, QIAN Y, FAN S, LI W, WANG J, HE M, YIN J, LI T, JIANG N, CHEN X, LIANG C, LI S. Pan-genome analysis of 33 genetically diverse rice accessions reveals hidden genomic variations[J]. Cell, 2021, 184(13): 3542-3558. DOI:10.1016/j.cell.2021.04.046 |
| [102] |
钟旭华, 黄农荣, 郑海波, 彭少兵, ROLAND J B. 水稻"三控"施肥技术规程[J]. 广东农业科学, 2007(5): 13-15. DOI:10.16768/j.issn.1004-874X.2007.05.003 ZHONG X H, HUANG N R, ZHENG H B, PENG S B, ROLAND J B. Specification for the"three controls"nutrient management technology for irrigated rice[J]. Guangdong Agricultural Sciences, 2007(5): 13-15. DOI:10.16768/j.issn.1004-874X.2007.05.003 |
| [103] |
彭碧琳, 李妹娟, 胡香玉, 钟旭华, 唐湘如, 刘彦卓, 梁开明, 潘俊峰, 黄农荣, 傅友强, 胡锐. 轻简氮肥管理对华南双季稻产量和氮肥利用率的影响[J]. 中国农业科学, 2021, 54(7): 1424-1438. DOI:10.3864/j.issn.0578-1752.2021.07.009.j.issn.0578-1752.2021.07.009 PENG B L, LI M J, HU X Y, ZHONG X H, TANG X R, LIU Y Z, LIANG K M, PAN J F, HUANG N R, FU Y Q, HU R. Effects of simplified nitrogen managements on grain yield and nitrogen use efficiency of double-cropping rice in South China[J]. Scientia Agricultura Sinica, 2021, 54(7): 1424-1438. DOI:10.3864/j.issn.0578-1752.2021.07.009.j.issn.0578-1752.2021.07.009 |
(责任编辑 杨贤智)
 2021, Vol. 48
2021, Vol. 48