文章信息
基金项目
- 安徽省重点研究与开发计划面上攻关-农业领域项目(202104a06020039)
作者简介
- 任代胜(1989—),男,硕士,农艺师,研究方向为水稻遗传育种,E-mail:rendaisheng@163.com.
通讯作者
- 乔保建(1972—),男,博士,副研究员,研究方向为高产优质杂交稻育种,E-mail:qiaobaojian@ylrice.com.
文章历史
- 收稿日期:2021-07-31
2. 广东省农业科学院作物研究所/广东省农作物遗传改良重点实验室,广东 广州 510640
2. Crops Research Institute, Guangdong Academy of Agricultural Sciences/Guangdong Key Laboratory of Crop Genetic Improvement, Guangzhou 510640, China
【研究意义】水稻是我国重要的粮食作物,在日常饮食中作为主食为人体能量代谢提供碳水化合物。由于我国人口基数大,过去粮食供应紧缺,传统的育种与栽培观念更倾向于提高水稻的总产量[1]。但随着时代的变化,改变稻米及其副产品的营养品质成为新的目标。稻谷在加工过程中被去除的碎屑组织称为米糠或米粕,富含脂肪酸、氨基酸、花青素、多酚等物质[2],所提取的米糠油含有脂肪酸组分、维生素E、甾醇、谷维素等有利于人体吸收的营养成分,吸收率高达90%,并具有清除血液中的胆固醇、降低血脂等保健作用[3]。因此,提高并改良稻米油的营养组分对产业发展具有重要意义。
【前人研究】米糠油中棕榈酸约占总油脂的15%,硬脂酸含量小于3%,油酸约为45%,亚油酸变化幅度较大约为29%~42%,亚麻酸含量小于1.8%。油酸分子为18碳烯酸,因第9位与第10位碳原子间存在双键而形成单不饱和脂肪酸。油酸含量较高的植物油对人体健康较为有益,长期食用可降低心脑血管发病率,增强人体免疫力[4]。此外,油酸分子的18碳长链中仅含有单个碳碳不饱和双键(C=C),其化学分子式结构稳定可提高油品的抗氧化能力、延长油品存储期,因此植物高油酸育种是产业化发展的主攻方向。目前油酸合成的分子机制已经明确,脂肪酸脱饱和基因FAD2(Fatty Acid Desaturase 2)位于油酸合成通路的下游,该基因突变后阻断油酸向亚油酸转化,进而提高油酸含量[5]。基于此原理,通过选育含有fad2的品种是植物高油酸育种的主要策略。木本油料作物中橄榄油与山茶油的油酸含量可占总油脂的90% 以上,豆科植物中大豆与花生的高油酸品种可达70%,并且高油酸育种已经在油菜、向日葵等大宗油料作物中广泛开展。以水稻为代表的禾本科作物仅高油酸胚芽油有少量报道,但也只限于基础研究层面,鉴于缺少可利用的高油酸种质资源,产业化应用仍然相当落后。
【本研究切入点】传统育种获得突变体材料主要依靠诱变育种,而目标个体筛选却费时费力。CRISPR/Cas9基因编辑技术是通过设计靶向位点引导sgRNA与CRISPR/Cas9蛋白相融合,对目的基因DNA序列进行特异性剪切,然后机体通过非末端同源重组修复受损的序列,导致核酸序列错配、插入、缺失以实现目标基因突变[6]。有关利用CRISPR/Cas9技术靶向OsFAD2及其旁系同源基因的研究鲜有报道,这限制了对油酸调控基因功能的了解。【拟解决的关键问题】本研究以水稻OsFAD2为目的基因,通过CRISPR/Cas9基因编辑与转基因技术相结合,获得水稻高油酸突变体材料。研究结果可为后续开展水稻高油酸育种提供新的种质资源,并为稻米油产业发展奠定理论基础。
1 材料与方法 1.1 试验材料供试水稻材料为日本晴种子,由安徽袁粮水稻产业有限公司提供。供载体构建转化的大肠杆菌菌株为DH5α,供水稻遗传转化的土壤脓杆菌菌株为EHA105,由广东省农业科学院作物研究所提供。OsU6a-sgRNA表达盒载体与pYLCRISPR/Cas9由华南农业大学刘耀光院士课题组馈赠。BsaⅠ限制性内切酶与T4 DNA连接酶购于NEB(New England Biolabs)有限公司,琼脂糖凝胶核酸纯化回收试剂盒为OMEGA凝胶回收试剂盒D2500-01,反转录cDNA试剂盒为TOYOBO FSK-100,荧光定量PCR试剂为ThermoFisher公司的Fast SYBR Green Master Mix,脂肪酸检测试剂购于Sigma-Aldrich公司。荧光定量PCR仪由华南农业大学国家植物航天育种工程技术中心提供,GC-MS连用仪由华南农业创新中心提供。
1.2 试验方法1.2.1 OsFAD2-sgRNA表达盒载体构建 OsFAD2基因组序列参考MSU Rice Genome Annotation Project数据库公布的基因登录号LOC_Os02g48560。sgRNA表达盒以Amp抗性的pUC18载体为骨架经改造而来,大小约为3 kb。利用CRISPRGE[7]网站设计OsFAD2的敲除靶点,该靶点为20 bp序列,位于第一外显子靠近起始密码子的位置,由Intervitrogene公司以引物的形式进行合成。OsFAD2-sgRNA表达盒构建,利用PCR仪设置95 ℃处理靶点接头引物(前引物:GCCGGAGGAGCAGAAGCTGCT,后引物:AAACAGCAGCTTCTGCTCCTC)30 s,用T4 DNA连接酶将靶点接头引物与BsaⅠ酶切后的pYLCRISPR/OsU6a-sgRNA载体相连,利用特定载体引物(前引物:CTCCGTTTTACCTGTGGA ATCG,后引物:CGGAGGAAAATTCCATCCAC)扩增OsU6a-FAD2-sgRNA序列,PCR产物切胶纯化后备用。
1.2.2 单靶点pYLCRISPR/Cas9-FAD2-sgRNA载体构建 利用BsaⅠ酶切载体pYLCRISPR/Cas9-FAD2-sgRNA,琼脂糖凝胶电泳检测酶切载体片段,切胶纯化回收。再利用BsaⅠ单酶切OsU6a-FAD2-sgRNA的PCR产物,充分酶切后直接利用Omega DNA纯化试剂盒回收酶切产物,全部线性化的纯化片段利用T4连接酶于20 ℃连接2 h后,将10 μL连接产物加入到100 μL的DH5α大肠杆菌感受态细胞于42 ℃进行热激法转化。阳性克隆子鉴定采用天根质粒小提试剂盒提取大肠杆菌质粒DNA,利用AscⅠ酶切进行验证,阳性克隆经Sanger测序后确定Cas9-FAD2sgRNA载体构建成功[8]。
1.2.3 OsFAD2敲除植株的转基因构建 将构建的Cas9-FAD2-sgRNA载体转化土壤脓杆菌,含有阳性载体的农杆菌菌株侵染日本晴的愈伤组织,共培养3 d后,转移侵染后的愈伤于潮霉素抗性培养基筛选2次共40 d,愈伤再经过分化、生根获得转基因再生植株。采用CTAB法提取转基因水稻叶片基因组DNA,在距离靶点上下游约200 bp的位置设计引物,通过PCR反应与Sanger测序鉴定靶点序列突变情况,并将阳性植株(T0代)练苗后栽至温室,所产生的种子继续栽种至T1代[9]。
1.2.4 OsFAD2表达量检测 取0.1 g叶片经液氮研磨,加入1 mL Invitrogene的TRIZOL试剂静置;加入300 μL氯仿,13 000 r/min离心5 min,取上清液400 μL并加入等体积异丙醇,13 000 r/min离心5 min,70% 无水乙醇洗涤,DEPC水溶解总RNA。取1 μg总RNA,利用反转录试剂盒FSK-100获得全长cDNA,以此作为模版,采用ABI StepOne Plus系统,使用SYBR Premix ExTaq进行荧光定量PCR检测,反应体系为20 μL,用2-ΔΔct法计算目标基因各组织的相对表达量,以野生型日本晴植株作为对照[10]。
1.2.5 OsFAD2敲除植株籽粒脂肪酸组分检测 脂肪酸检测所用试剂,包括脂肪酸标品(SigmaAldrich)、三氯甲烷(Sigma-Aldrich)、甲醇(色谱纯)、石油醚、乙醚、氢氧化钾,所用仪器为安捷伦GC-MS气质联用仪7890A+5975C,色谱柱为DB-225MS(30 m×0.25 mm×0.25 μm),进样量1 μL,气化温度280 ℃,分流比10 ︰ 1,载气为氦气(99.999%),流量1 mL/min,柱温初始温度50 ℃,5 ℃ /min升温至200 ℃,2 ℃ /min升至230 ℃,保持10 min。色谱条件依次检测各标准溶液的峰面积,以峰面积为纵坐标,浓度为横坐标,计算得到各种标准曲线。称取水稻籽粒研磨后0.2 g样品,加入0.5 mL石油醚一乙醚混合液,涡旋振荡、静置1 h,4 000 r/min离心1 min,加入0.25 mL氢氧化钾一甲醇溶液为甲酯化试剂,涡旋振荡、静置1 h,再次涡旋振荡,加入0.5 mL去离子水,静置30 min分层、以4 500 r/min离心2 min、取上清液,供气相色谱质谱联用仪测定。对样品质谱峰图与标准品峰图进行比对,计算各组分相对含量[11]。
2 结果与分析 2.1 水稻OsFAD2基因与蛋白序列特性分析水稻OsFAD2(LOC_Os02g48560)位于第2条染色体上,基因序列5 244 bp,仅含有1个外显子区段,该基因的信使RNA同时包含3种可变剪切形式,大小分别为1 359、1 166、1 148 bp(图 1A)。其中可变剪切转录本OsFAD2-1的蛋白序列长度452个氨基酸,在72~137 aa处含有保守的DUF3474结构域,在155~416 aa处含有脂肪酸脱饱和酶结构域;OsFAD2-2与OsFAD2-3蛋白序列长度相似,仅有少量氨基酸缺失差异,两者分别在18~73 aa与85~353 aa处含有保守的DUF3474与脂肪酸脱饱和酶结构域(图 1B)。由此推测,虽然OsFAD2具有3种不同的可变剪切转录本,但是所编码的蛋白均含有保守的脂肪酸脱饱和酶结构域,该保守区段可能是其促进油酸脱氢后产生亚油酸的主要结构。

|
| A:OsFAD2不同可变剪切转录本的基因组分布;B:OsFAD2不同可变剪切体对应的蛋白质保守结构 A: Genome distribution of different alternative splice transcripts of OsFAD2; B: Conservative domain in OsFAD2 alternative splice encoded protein sequences 图 1 OsFAD2基因与蛋白结构示意图 Fig. 1 Schematic gene and protein structures of OsFAD2 |
2.2 OsFAD2组织表达量分析
为明确OsFAD2能否在种子内具有表达活性,利用RiceXPro数据库[12]检索OsFAD2基因表达量,数据经过整合重新分析后发现,OsFAD2在营养生长阶段的表达量以在叶片中最高,其次是叶鞘,在茎与根中表达量较低,表明该基因在地上部分组织的表达水平整体高于地下部分。而进入生殖生长阶段,该基因在种子内稃中的表达量最高,而在受精后胚以及胚乳中的表达量略高于花序、花药、雌蕊和子房,总体而言在种子发育过程中的表达量要高于营养生长阶段叶片的表达量(图 2),表明该基因对于籽粒积累脂肪酸的调控有重要的生物学功能。组织表达量分析也明确了该基因的潜在功能,若该基因不能在水稻籽粒发育阶段表达,则后续开展CRISPR/Cas9基因敲除将失去实际意义。

|
| 图 2 OsFAD2在不同组织内的基因表达模式分析 Fig. 2 Analysis of gene expression pattern of OsFAD2 in different tissues |
2.3 CRISPR/Cas9诱导的OsFAD2敲除植株检测
针对OsFAD2基因3个可变剪切的共有序列设计CRISPR/Cas9敲除的单个靶点,该引导RNA的位置位于编码区距离起始密码子(ATG)250 bp处。最终构建好的Cas9-FAD2-sgRNA经酶切与测序验证表明,FAD2-sgRNA表达盒与玉米泛素化启动子Ubi驱动的Cas9蛋白连接成功(图 3A)。
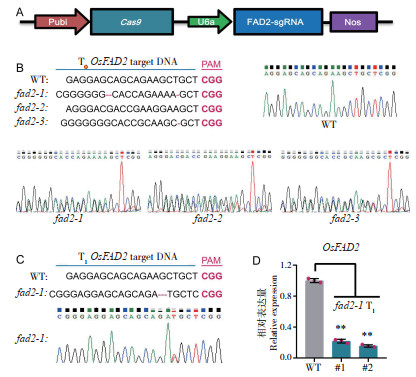
|
| A:Cas9-FAD2-sgRNA载体构建;B:fad2突变体T0代植株靶点检测;C:fad2-1转基因T1代植株靶点检测;D:荧光定量PCR检测OsFAD2在fad2-1植株中的相对表达量 A: Construction of Cas9-FAD2-sgRNA vector; B: Target detection in fad2 mutant lines(T0 generation); C: Target detection in fad2-1 transgenic plant(T1 generation); D: Relative expression level of OsFAD2 in fad2-1 plants by using qPCR detection 图 3 OsFAD2的CRISPR/Cas9敲除植株验证 Fig. 3 Validation of OsFAD2 knock-outed plants via CRISPR/Cas9 |
利用农杆菌介导法将表达载体转入日本晴的愈伤组织,以30个长势良好的愈伤组织团聚体作为外植体进行转染,经潮霉素抗性标记筛选后获得17株转基因再生苗。其中4株为白化苗且未能存活,5株未能产生突变,剩余8株的OsFAD2序列均有碱基突变,总体转化效率接近50%,可见该类载体的编辑效率非常高。经过序列比对后,最终确定了3个独立的转基因T0代植株,命名为fad2-1、fad2-2、fad2-3。T0代转基因植株测序结果表明,fad2-1同时存在缺失与碱基替换,fad2-2在靶点位置均为碱基替换,fad2-3有1个位点发生碱基缺失,其余均为碱基替换类型。由于T0代植株属于嵌合体,因而与野生型植株相比在进行测序时,峰图均存在双峰现象,该结果属于正常现象(图 3B)。同时,利用CRISPR-GE网站预测所设计靶点的潜在脱靶基因,筛选发现有10个基因的编码区序列可能是潜在的敲除位点,经序列检测后发现,fad2-1不存在脱靶现象,而fad2-2与fad2-3均有脱靶现象(表 1),表明fad2-1可作为阳性植株开展后续脂肪酸检测试验。
本试验收获3个独立T0代株系的种子,进行扩繁得到T1代植株,但检测T1代植株靶点时发现,fad2-2与fad2-3产生的种子后代在敲除靶点全部变为正常序列,原本T0代的变异序列被同源重组修复后变成了与野生型一致的序列,加之该两个株系存在脱靶现象,因此不可作为后续试验材料。而fad2-1在敲除靶点存在3个碱基缺失与碱基替换现象,表明fad2-1为纯合株系,并且测序峰图并未出现严重的双峰现象(图 3C)。荧光定量PCR检测fad2-1两个独立株系的OsFAD2基因表达量,结果显示该基因的表达量显著低于野生型内的表达水平(图 3D),可见本研究获得了稳定性遗传的CRISPR/Cas9敲除OsFAD2的转基因植株。
2.4 OsFAD2转基因敲除植株籽粒的脂肪酸组分检测经验证明确fad2-1为阳性敲除植株后,我们收获了两个独立株系的转基因种子,利用GC-MS气质联用仪检测脱壳后种子的脂肪酸含量,结果表明,棕榈酸(C16∶0)含量在fad2-1中略有减少,但是单不饱和棕榈酸(C16∶1)含量有所增加;18碳长链脂肪酸中的硬脂酸(C18∶0)含量虽在敲除植株中有所减少,但该类脂肪酸总体含量较少,未达到显著差异;油酸(C18∶1)含量则明显发生变化,其含量显著高于野生型中的含量,而相应的亚油酸(C18∶2)含量则出现显著下降,亚麻酸(C18∶3)含量总体变化不大(图 4)。通过种子脂肪酸组分含量检测可以确定,OsFAD2突变后对油酸含量的增加有显著促进作用。本研究通过利用CRISPR/Cas9敲除OsFAD2获得了稳定遗传的高油酸稻突变体材料。
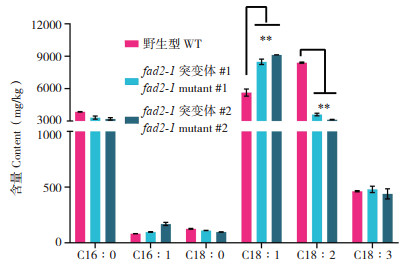
|
| 图 4 OsFAD2敲除突变体植株种子的脂肪酸组分检测 Fig. 4 Detection of fatty acid composition in OsFAD2 knock-outed mutants |
3 讨论
CRISPR/Cas9基因定点编辑技术具有突变效率高、载体构建简便的优势,是水稻种质资源创新的前沿技术,与传统获得突变体的方式不同,通过靶向引导RNA的介入,CRISPR/Cas9蛋白酶可对DNA序列进行高效剪切,机体的非同源重组修复再产生多种类型的碱基变异模式[13]。而后续通过杂交或轮回选择的方式可剔除内源性插入的CRISPR/Cas9蛋白的编码序列与筛选标记,该技术与传统育种方式的有效结合可构建水稻基因突变体库[14]。本研究利用CRISPR/Cas9基因编辑技术对水稻脂肪酸脱饱和酶基因OsFAD2编码区序列进行定点编辑,获得了能够稳定遗传的高油酸种质材料,为后续开展高油酸水稻育种奠定了基础。
OsFAD2在水稻体存在4个同源基因,本研究主要针对OsFAD2-1(LOC_Os02g48560)进行研究,其他3个同源基因如OsFAD2-2(LOC_Os07g23430)、OsFAD2-3(LOC_Os07g23410)、OsFAD2-4(LOC_Os07g23390)均被预测定位于第7条染色体上,三者是否具有与OsFAD2-1类似的功能仍有待深入研究,若采用CRISPR/Cas9基因编辑创制三者的突变体,则能够有效解释水稻OsFAD2及其同源基因之间的功能冗余性问题[15]。此外,OsFAD2突变体不仅能够被应用于实际育种工作,还可以作为基础研究材料用于研究水稻脂肪酸合成代谢的分子机制。此前有研究报道OsFAD2-1的RNAi干扰植株可表达生成高油酸米糠油,米糠油具有较高的氧化稳定性,无需加氢过程而直接被用于食品工业[16]。敲减OsFAD2-1基因表达会伴随脂质合成通路中多个关键基因表达下调,表明该基因能够在整体水平上影响种子脂肪酸的合成[17-18]。本研究鉴定到敲除OsFAD2导致该基因丧失表达能力,可显著提高油酸含量,并且发现单不饱和棕榈酸(C16∶1)含量有所增加,该结果与花生中高油酸突变体结果类似,花生FAD2突变后也可以提高C16∶1含量[19],但具体机制仍有待深入研究。因此,利用OsFAD2突变体可为水稻脂肪酸合成代谢研究提供新的切入点。
我国是水稻生产大国,每年总产量约2亿t,米糠等副产品的产量约占6.5%,粗略估计如充分利用可产稻米油180万t,相当于1 000多万t大豆的含油量,可缓解食用油供给压力[20]。随着我国稻米精深加工业整体水平的提高,以及人们对油类产品营养保健作用的重视,米糠油的产业链正在逐步发展壮大。油酸含量较高的植物油营养价值、货架存储期均优于普通植物油,现已成为衡量植物油价格高低的评价标准,例如橄榄油、山茶油等价格是普通植物油的5~10倍[21]。因此,大力推广高产、高油酸水稻品种能够促进稻米油产业全面发展,本研究所创制的高油酸水稻种质资源则具有潜在的产业应用价值。
4 结论本研究利用CRISPR/Cas9基因编辑技术获得OsFAD2的突变体材料,为水稻高油酸育种提供了新的种质资源。通过水稻全生育期基因转录表达量分析发现,OsFAD2的表达量在生殖生长阶段显著高于营养生长阶段,表明该基因可调控籽粒中的油酸合成。鉴于CRISPR/Cas9基因编辑可特异性敲除目标DNA序列,本研究所设计的敲除靶点位于OsFAD2起始密码子(ATG)下游约250 bp处,因此创制出新OsFAD2突变类型。其遗传特性稳定的T1代转基因植株油酸含量比野生型植株提高了25%~30%,而种子亚油酸含量相应减少约35%,该结果表明通过靶向敲除OsFAD2的编码序列,可有效改良水稻油酸及其他脂肪酸的组成。
| [1] |
XU L, YUAN S, MAN J G. Changes in rice yield and yield stability in China during the past six decades[J]. Journal of the Science of Food Agriculture, 2020, 100(8): 3560-3569. DOI:10.1002/jsfa.10385 |
| [2] |
左英秀, 沈军. 稻米加工副产物米糠综合利用的研究[J]. 粮食与食品工业, 2020, 27(6): 11-14. ZUO Y X, SHEN J. Studies on comprehensive utilization of rice bran, a byproduct of rice processing[J]. Cereal and Food Industry, 2020, 27(6): 11-14. |
| [3] |
ZHOU H, XIA D, LI P B, AO Y T, XU X D, WAN S S, LI Y H, WU B, SHI H, WANG K Y, GAO G J, ZHANG Q L, WANG G W, XIAO J H, LI X H, YU S B, LIAN X M, HE Y Q. Genetic architecture and key genes controlling the diversity of oil composition in rice grains[J]. Molecular Plant, 2021, 14(3): 456-469. DOI:10.1016/j.molp.2020.12.001 |
| [4] |
ZHAO Z H, SHI A M, WANG Q, ZHOU J R. High Oleic acid peanut oil and extra virgin olive oil supplementation attenuate metabolic syndrome in rats by modulating the gut microbiota[J]. Nutrients, 2019, 11(12): 3005. DOI:10.3390/nu11123005 |
| [5] |
ZAPLIN E S, LIU Q, LI Z Y, BUTARDO V M, BLANCHARD C L, RAHMAN S. Production of high oleic rice grains by suppressing the expression of theOsFAD2-1 gene[J]. Functional Plant Biology, 2013, 40(10): 996-1004. DOI:10.1071/FP12301 |
| [6] |
CHEN K L, WANG Y P, ZHANG R, ZHANG H W, GAO C X. CRISPR/ Cas genome editing and precision plant breeding in agriculture[J]. Annual Review of Plant Biology, 2019, 70: 667-697. DOI:10.1146/annurev-arplant-050718-100049 |
| [7] |
XIE X R, MA X L, ZHU Q L, ZENG D C, LI G S, LIU Y G. CRISPRGE: a convenient software toolkit for CRISPR-based genome editing[J]. Molecular Plant, 2017, 10(9): 1246-1249. DOI:10.1016/j.molp.2017.06.004 |
| [8] |
MA X L, ZHANG Q Y, ZHU Q L, LIU W, CHEN Y, QIU R, WANG B, YANG Z F, LI H Y, LIN Y R, XIE Y Y, SHEN R X, CHEN S F, WANG Z, CHEN Y L, GUO J X, CHEN L T, ZHAO X C, DONG Z C, LIU Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants[J]. Molecular Plant, 2015, 8(8): 1274-1284. DOI:10.1016/j.molp.2015.04.007 |
| [9] |
LIU H, DONG S Y, SUN D Y, LIU W, GU F W, LIU Y Z, GUO T, WANG H, WANG J F, CHEN Z Q. CONSTANS-Like 9(OsCOL9) interacts with receptor for activated c-kinase 1(OsRACK1)to regulate blast resistance through salicylic acid and ethylene signaling pathways[J]. PLOS ONE, 2016, 11(11): e0166249. DOI:10.1371/journal.pone.0166249 |
| [10] |
刘维, 刘浩, 董双玉, 古丰玮, 陈志强, 王加峰, 王慧. 利用CRISPR/Cas9技术创建OsCOL9水稻突变体[J]. 华北农学报, 2017, 32(4): 42-48. DOI:10.7668/hbnxb.2017.04.007 LIU W, LIU H, DONG S Y, GU F W, CHEN Z Q, WANG J F, WANG H. MutantedOsCOL9 based on CRISPR/Cas9 technology in rice[J]. Acta Agriculturae Boreali-Sinica, 2017, 32(4): 42-48. DOI:10.7668/hbnxb.2017.04.007 |
| [11] |
SHI T, WU G C, JIN Q Z, WANG X G. Detection of camellia oil adulteration using chemometrics based on fatty acids GC fingerprints and phytosterols GC–MS fingerprints[J]. Food Chemistry, 2021, 352: 129422. DOI:10.1016/j.foodchem.2021.129422 |
| [12] |
SATO Y, TAKEHISA H, KAMATSUKI K, MINAMI H, NAMIKI N, IKAWA H, OHYANAGI H, SUGIMOTO K, ANTONIO B A, NAGAMURA Y. RiceXPro version 3.0: expanding the informatics resource for rice transcriptome[J]. Nucleic Acids Research, 2013, 41: D1206-D1213. DOI:10.1093/nar/gks1125 |
| [13] |
MENG X B, YU H, ZHANG Y, ZHUANG F F, SONG X G, GAO S S, GAO C X, LI J Y. Construction of a genome-wide mutant library in rice using CRISPR/Cas9[J]. Molecular Plant, 2017, 10: 1238-1241. DOI:10.1016/j.molp.2017.06.006 |
| [14] |
CHEN K L, WANG Y P, ZHANG R, ZHANG H W, GAO C X. CRISPR/ Cas genome editing and precision plant breeding in agriculture[J]. Annual Review of Plant Biology, 2019, 70: 667-697. DOI:10.1146/annurev-arplant-050718-100049 |
| [15] |
CAO Y P, SHI J L, LI Z, MING F. Isolation of OsFAD2, OsFAD6 and FAD family members response to abiotic stresses inOryza sativa L[J]. Yi Chuan, 2010, 32(8): 839-847. DOI:10.3724/sp.j.1005.2010.00839 |
| [16] |
TIWARI G J, LIU Q, SHRESHTHA P, LI Z Y, RAHMAN S. RNAi-mediated down-regulation of the expression ofOsFAD2-1: effect on lipid accumulation and expression of lipid biosynthetic genes in the rice grain[J]. BMC Plant Biology, 2016, 16(1): 189. DOI:10.1186/s12870-016-0881-6 |
| [17] |
BAHARIAH B, ABDUL MASANI M Y, RASID O A, AHMAD PARVEEZ G K. Multiplex CRISPR/Cas9-mediated genome editing of theFAD2 gene in rice: a model genome editing system for oil palm[J]. Journal of Genetic Engineering Biotechnology, 2021, 19: 86. DOI:10.1186/s43141-021-00185-4 |
| [18] |
ABE K, ARAKI E, SUZUKI Y, TOKI S, SAIKA H. Production of high oleic/low linoleic rice by genome editing[J]. Plant Physiology and Biochemistry, 2018, 131: 58-62. DOI:10.1016/j.plaphy.2018.04.033 |
| [19] |
LIU H, HONG Y B, LU Q, LI H F, GU J Z, REN L, DENG L, ZHOU B J, CHEN X P, LIANG X Q. Integrated analysis of comparative lipidomics and proteomics reveals the dynamic changes of lipid molecular species in high-oleic acid peanut seed[J]. Journal of Agriculture and Food Chemistry, 2020, 68(1): 426-438. DOI:10.1021/acs.jafc.9b04179 |
| [20] |
SOHAIL M, RAKHA A, BUTT M S, IQBAL M J, RASHID S. Rice bran nutraceutics: A comprehensive review[J]. Critical Reviews in Food Science and Nutrition, 2017, 57(17): 3771-3780. DOI:10.1080/10408398.2016.1164120 |
| [21] |
LIU H, GU J Z, LU Q, LI H F, HONG Y B, CHEN X P, REN L, DENG L, LIANG X Q. Transcriptomic analysis reveals the high-oleic acid feedback regulating the homologous gene expression of stearoyl-ACP desaturase 2(SAD2)in peanuts[J]. International Journal Molecular Sciences, 2019, 20(12): 3091. DOI:10.3390/ijms20123091 |
(责任编辑 崔建勋)
 2021, Vol. 48
2021, Vol. 48