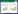文章信息
基金项目
- 广东省农业科学院作物研究所所长基金(201904);广东省农作物遗传改良重点实验室开放基金(201904);广东省科技计划项目(2017B020232002);广东省现代农业产业技术体系创新团队项目(2021KJ111)
作者简介
-
单建伟(1980—),男,博士,助理研究员,研究方向为马铃薯分子生物学及遗传育种,E-mail: chanyujianwei@126.com; 李小波,博士,研究员,广东省甘薯马铃薯产业技术体系创新团队马铃薯育种岗位专家,广东省农业科学院作物研究所马铃薯学科团队学术带头人,华中农业大学和湖南农业大学校外硕士生导师,兼任广东省马铃薯产业技术创新联盟常务副理事长,中国作物学会马铃薯专业委员会委员。
主要从事马铃薯遗传育种和节本增效栽培技术研究与应用。先后主持广东省基础研究重大项目子课题、广东省应用型研发专项、国家重点研发计划子课题、广东省马铃薯良种重大科研联合攻关等各级项目20多项;获广东省农业科学院科技奖二等奖1项,广东省农业技术推广奖一等奖2项、二等奖2项;制定地方标准2项;培育马铃薯品种6个;获得植物新品种保护权1项。培育的马铃薯品种多次被列为广东省和广州市主导品种,推广面积达13万hm2以上,社会经济效益显著。主编书籍2部,发表科技论文40余篇;申请和获授权国家发明专利5件,实用新型专利6件,软件著作权4件.
通讯作者
- 李小波(1981—),男,博士,研究员,研究方向为马铃薯遗传育种及示范推广,E-mail: Lixiaobo1981@163.com.
文章历史
- 收稿日期:2021-11-07
2. 云南省农业科学院经济作物研究所,云南 昆明 650000
2. Industrial Crops Institute, Yunnan Academy of Agricultural Sciences, Kunming 650000, China
【研究意义】马铃薯为茄科(Solanaceae)茄属(Solanum)马铃薯组(Petota)草本植物,原产于南美洲安第斯高原[1]。马铃薯的植物分类学是一个不断更新和完善的过程,Spooner等综合前人有关马铃薯表型、分子标记、系统发生、生殖生物学、倍性、核型等方面的研究将马铃薯组分类为107个野生种和4个栽培种[2]。对于栽培作物而言,通常在其起源地具有最丰富的遗传资源和遗传多样性,而在其传播过程中遗传多样性逐渐降低[3]。20世纪20年代,前苏联、英国、德国和美国学者先后赴南美洲考察和收集马铃薯野生种及栽培种种质资源,为马铃薯的育种和遗传学研究开创了新纪元[4]。当今世界范围内广泛种植的现代马铃薯品种(modern cultivar)是以马铃薯普通栽培种为基础,通过与其他地方栽培种或野生种杂交利用了有利基因的产物,使其能够适应更宽泛的生态条件和地理区域,但目前广泛种植的马铃薯品种遗传多样性依然很低[5]。广泛收集国内外马铃薯品种(系)资源,对其群体结构和遗传多样性进行分析评价,将为我国马铃薯遗传育种提供理论指导。
【前人研究进展】我国马铃薯的栽培历史并不长[6],对其育种起步也较晚,始于20世纪30年代中末期,早期的马铃薯育种工作以引种为主,先后从英国、美国和前苏联等引进一批育成品种和杂交组合。由于马铃薯适应性广、产量高,新中国成立以后为了尽快恢复农业生产,国家有计划地组织调运种薯,扩大马铃薯种植面积,全国开展了马铃薯育种协作,真正开始马铃薯育种工作,促进了马铃薯在我国的迅猛发展,并相继育成了一批有代表性的马铃薯品种,如东农303、克新系列、陇薯系列等[7-8]。当前,我国马铃薯种植面积和总产量均超过全球的1/4,居世界首位。由于我国马铃薯育种工作起步较晚,所用亲本多引自欧洲和北美等国,对种质资源特别是野生种和地方栽培种的收集、研究、创新不够,因此我国栽培的马铃薯品种遗传多样性更低,许多品种之间同质性高,部分品种存在同种异名现象,仅靠表型差异难以分辨。
【本研究切入点】为了指导马铃薯遗传育种,有必要收集国内外马铃薯品种(系)资源,并对其遗传多样性进行评价。【拟解决的关键问题】本研究对收集的国内外马铃薯品种(系)资源,利用限制性酶切位点关联DNA测序(Restrictionsite Associated DNA sequencing,RADseq) 中的ddRADseq技术对其进行简化基因组测序,结合群体结构分析和遗传多样性分析对其进行群体结构和遗传多样性评价,开发马铃薯SNP-Panel,为马铃薯育种、品种鉴定提供理论依据。
1 材料与方法 1.1 试验材料供试材料为广东省农业科学院作物研究所马铃薯研究室收集的185份马铃薯品种(系),包含育成品种以及一些高代品系(表 1)。这些材料分别来自我国的华南、华北、西南、华中地区以及国外的秘鲁和意大利。
1.2 试验方法
1.2.1 基因组DNA制备 取每个马铃薯品种(系)的嫩叶,利用液氮速冻后储存于-80℃超低温冰箱,分离样品基因组DNA后,利用琼脂糖凝胶电泳检测DNA样品完整性及有无RNA污染,并利用分光光度计对DNA样品的质量和浓度进行定量检测。
1.2.2 文库构建、质控及测序 取约200 ng基因组DNA用限制性内切酶MseⅠ和SacⅠ(纽英伦生物技术有限公司)按照产品说明书进行完全酶切;酶切产物连接特异性接头,然后对连接产物进行纯化;用高保真聚合酶KOD-Plus-Neo(东洋纺生物科技有限公司)按照产品说明书进行PCR扩增富集;将所有扩增产物进行低压电泳,切取300~400 bp大小的电泳片段,用琼脂糖凝胶回收试剂盒(QIAGEN,德国)进行纯化;用分光光度计对纯化产物进行初步定量,用安捷伦2100生物分析仪对文库插入片段大小进行检测;采用实时荧光定量PCR手段对文库的有效浓度进行准确定量,有效浓度>2 nmol/L为合格;最后用Illumina HiSeq测序平台进行双末端测序。
1.2.3 测序数据过滤 测序得到的原始数据被称为raw read,含有测序接头序列或低质量的碱基。使用Cutadapt(Version 1.13) 软件去除raw read中的接头序列,用Trimmomatic(Version 0.36)去除测序数据中的低质量碱基,得到clean read用于后续分析;测序read的长度必须大于50 bp。
1.2.4 参考基因组比对 使用BWA(Version 0.7.15-r1140)软件中的MEM算法将测序数据与参考基因组(http://solanaceae.plantbiology.msu.edu/pgsc_download.shtml)进行比对,得到SAM格式的比对结果,然后使用Samtools(Version 1.3.1) 将其转换为BAM格式,使用Picard(Version 1.91) 软件中的SortSam对BAM文件中的序列进行排序,将处理后的BAM文件用于覆盖度和覆盖深度统计以及变异位点识别。
1.2.5 变异位点检测及注释 利用GATK(Version 3.7)软件包中的HaplotypeCaller模块对所有样品生成gvcf格式文件,然后用GenotypeGVCFs模块对所有样品进行SNP和InDel变异检测。并按照测序深度≥ 5、所有样品基因型缺失率≤ 50%、稀有等位基因频率≥ 10%、杂合率≤ 50%、相对杂合率≤ 67% 等标准对变异位点进行筛选,得到的变异位点为有效变异位点。用ANNOVAR (Version 2016Feb1)对变异位点进行注释。依据变异位点在参考基因组上的位置信息以及参考基因组中的基因位置信息,可得到变异位点在基因组中的区域(基因间区、基因区或CDS区等)信息。
1.2.6 马铃薯群体结构分析 通过Structure软件对群体结构进行分析,结果以ΔK值变化图和祖先堆叠图展示,可直观反映个体间的分类关系以及每个样品的“混血”程度。采用PLINK(Version v1.90p)软件对群体结构进行主成分分析(Principal Component Analysis,PCA)。PCA散点图中,两个样品距离越远,表明遗传背景差异越大,而遗传背景相似的个体在PCA散点图中聚类在一起。采用MEGA7(Version 7.0)软件中的邻接法(Neighbor-joining methods)构建系统发生树,并通过ggtree(Version 1.7.10)进行可视化处理。基于分子标记信息,利用Cervus 3.0软件计算多态性信息含量(Polymorphism Information Content,PIC)、观测杂合度(Observed Heterozygosit,Ho)、期望杂合度(Expected Heterozygosity,He)等。
1.2.7 SNP分子标记开发 在综合区分度和检测成本的基础上,结合SNP标记在染色体上的位置信息以及多态性,选择120个在染色体上分布相对均匀的SNP标记,分别在SNP位点上下游开发PCR引物,PCR产物长度介于150~350 bp之间。
2 结果与分析 2.1 变异位点检测及注释通过测序最终获得clean read 7.50×108条、2.04×1011个碱基,平均每个样品4.05×106条clean read,1.10×109个碱基。为了检测变异位点,将测序得到的clean read与马铃薯基因组(http://solanaceae.plantbiology.msu.edu/pgsc_download.shtml)进行比对,并进行群体结构和遗传多样性进行分析。本研究在综合考虑可行性和经济性的基础上对测序覆盖度进行了控制,样品平均测序覆盖度为2.62%。利用GATK软件包共检测到39 038个有效变异位点,其中SNP位点36 267个,InDel位点2 771个。SNP位点在马铃薯12条染色体上的平均分布密度为50.02个/Mb SNP位点,但在不同染色体上的的分布密度不均,其中10号染色体上的SNP位点密度最低(26.22个/Mb),5号染色体上SNP位点密度最高(83.50个/Mb);InDel变异位点在12条染色体上的分布密度介于1.97~6.05个/Mb之间,同样10号染色体分布密度最低,5号染色体分布密度最高(表 2)。同一条染色体不同区域的变异位点密度也存在较大差异,如1号染色体10~20 Mb范围内的变异位点密度最高,每100 kb存在约90个变异位点,而在70~90 Mb范围内变异位点密度较低(图 1)。
|
| 图 1 变异位点在染色体上的分布 Fig. 1 Distribution of variation sites on chromosomes |
利用ANNOVAR软件结合变异位点在参考基因组和自身基因组上的位置信息对变异位点进行注释,结果(表 3)显示,共有26 697个SNP位点位于基因间区,占所有SNP位点的73.65%;1 392个SNP位点位于基因转录起始位点上游1 kb范围内,1 314个SNP位点位于转录终止位点下游1 kb范围内,116个SNP位点位于一个基因转录起始位点上游1 kb范围内并同时在另一个基因的转录终止位点下游1 kb范围内;6 729个SNP位点位于基因内部,占所有SNP位点的18.56%,其中3 172个SNP位点位于内含子区域,2 605个SNP位点位于外显子区;352个SNP位点位于5' UTR区,597个位点位于3' UTR区;3个SNP位点位于可变转录本上。此外,共有1 761个(63.57%)InDel位点位于基因间区;308个InDel位点位于基因编码区上下游1 kb范围内;25.31% 的InDel位点位于基因内部,其中406个位于内含子内,134个位于外显子内;3' UTR和5' UTR区域内的InDel位点数分别为101、56个;4个InDel位点位于可变转录本内。
2.3 马铃薯群体结构分析
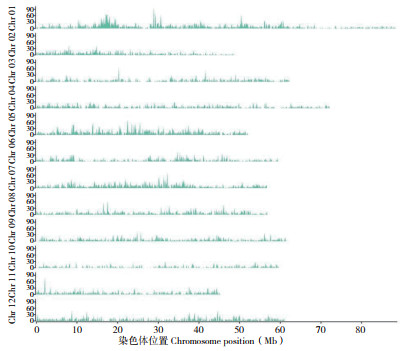
为了明确本研究所用马铃薯的群体结构,通过PCA法对筛选得到的有效SNP标记进行降维处理,从中提取关键信息将分子标记差异信息反映在二维坐标图上,坐标轴为对样品差异性解释度最高的特征向量,两个样品遗传背景差异越小,则样品分子标记位点信息越相似,反映在PCA图中的距离越近。PCA散点图显示,第一特征向量(PC1)和第二特征向量(PC2)可以将本研究所用群体区分为G1和G2两个亚群(图 2A)。利用Structure软件对群体结构进行分析,群体结构分析的ΔK值变化趋势图显示,当K=2时对应的ΔK值最大,因而本研究群体的最佳亚群数为2,这一结果与PCA分析结果一致,即将本研究群体分为两个亚群较为合适(图 2B)。G2亚群包含29个品种(系)全部来自云南省农业科学院,分别为S17-192-12、S17-114-4、S17-188- 13、S17-119-2、S17-188-4、S17-114-2、S17- 166-11、S17-147-12、S17-114-10、S17-1-1、S17-176-1、S17-173-97、S17-4-13、S17-201- 1、S17-173-4、S17-147-23、S17-187-4、S17- 1-9、S17-4-21、S17-173-30、S17-1-11、S17- 129-3、S17-27-1、S17-8-1、S17-1-15、S17- 181-12、S17-172-2、S17-181-1、紫云一号-1;剩余的156个品种(系)构成G1亚群,包括来自我国华南、华北、华中、西南、西北等地的品种(系),以及引自秘鲁、意大利等国的品种(系)。祖先堆叠图(图 2C)显示,两个亚群间存在明显基因交流,部分样本存在混血。群体系统进化分析结果(图 2D)表明,所有G2亚群中的29个品种(系)聚类在一起且其分枝离圆心的距离最长,表明G2亚群中的个体亲缘关系较近,同时G2亚群中的个体在进化上积累了更多变异。
|
| A:群体结构PCA分析;B:群体结构分析的ΔK值变化;C:群体结构祖先堆叠图;D:群体系统发生树 A: PCA analysis of population structure; B: Change in ΔK value of population structure analysis with Structure; C: Stacked diagram of ancestors; D: Phylogenetic tree of population 图 2 马铃薯群体结构分析 Fig. 2 Analysis of potato population structure |
PIC>0.5为高度多态性[9],而本研究群体的平均PIC=0.3107,表明群体遗传多样性中等偏低。同时,群体的期望杂合度He=0.3932,观测杂合度Ho=0.1852,群体自交系数Fis=0.553,也表明群体遗传多样性偏低。
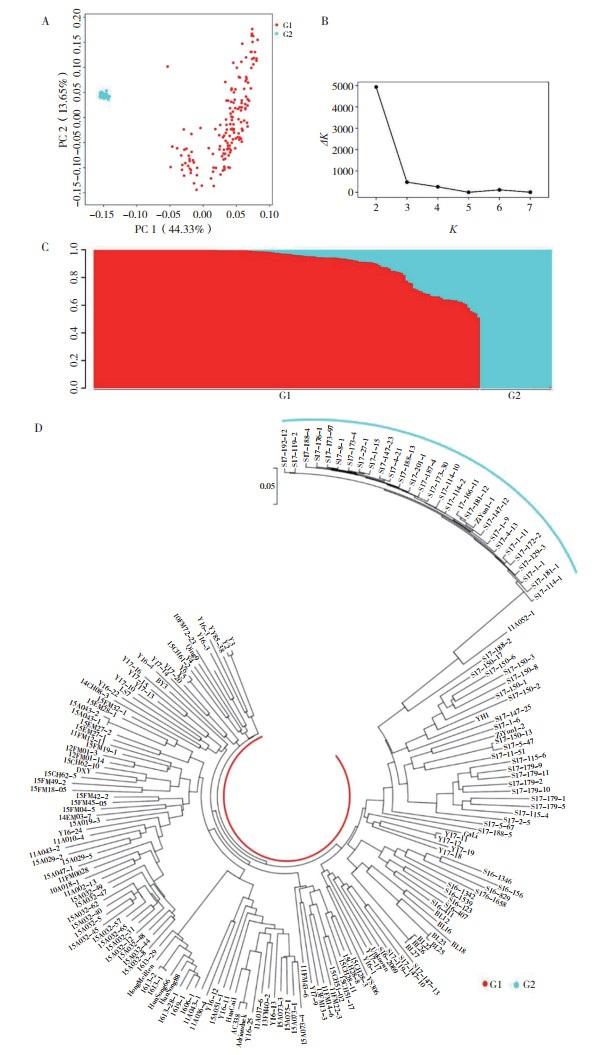
2.4 SNP分子标记开发靶向测序(target-seq)技术通过设计多重PCR引物对目标区域DNA片段进行特异性扩增,再通过双端标签区分不同样本,混样建库,进行多样本多位点高通量测序分析,具有信息量大、针对性强、低成本、效率极高、精准度高等特点,被广泛应用于分子标记辅助选择育种(MarkerAssisted Selection,MAS)、数量性状位点(Quantitative Trait Locus,QTL)精细定位、品种指纹分析、品种鉴定、基因编辑位点检测等领域。为了便于后续开展马铃薯亲缘关系鉴定、品种鉴别、群体遗传分析以及分子标记辅助选择育种,我们开发了包含120个马铃薯SNP标记的SNP-Panel,这些SNP标记均匀分布在马铃薯的12条染色体上(图 3),并针对这120个SNP标记设计了分离目标DNA区域的特异引物用于后续多样本高通量SNP标记检测。为了验证SNP标记的有效性,选取了60个SNP标记,利用与所选SNP标记对应的引物对46份马铃薯品种(系)进行了靶向高通量测序,结果表明,60个SNP标记能够准确将46份马铃薯品种(系)区分开。理论上,所选标记越多,越能区分更多的品种(系),因此需要在区分度和检测成本之间寻求平衡。因此在综合区分度和检测成本的基础上,本研究确定了SNP标记的个数为120。
|
| 图 3 SNP-Panel在马铃薯染色体上的分布 Fig. 3 Distribution of SNP-Panel on potato chromosomes |
3 讨论
RADseq是在第二代测序技术基础上发展而来的一项基于全基因组酶切位点的简化基因组测序技术,该技术具有检测速度快、高通量、成本低等优点,可以检测出上千万的SNP位点,能够满足群体结构分析、遗传图谱构建和QTL定位等研究需求[10-11]。依据所使用的限制性内切酶的数量和种类,RADseq技术可细分为Original RADseq、GBS、2b-RADseq、ezRAD、ddRADseq等技术方法。ddRADseq技术由于检测准确、成本低、SNP位点丰富等优点得到广泛应用[12-13]。本研究采用ddRADseq技术对185个马铃薯品种(系)进行了简化基因组测序分析。基于变异位点在各样品中的测序深度均不小于5、在所有样品中的缺失率不超过50%、低频等位基因频率不低于10%、杂合率不超过50%、相对杂合率不超过67% 等标准共获得39 038个高质量变异位点,其中SNP位点36 267个,InDel位点2 771个。高质量变异位点为后续马铃薯群体结构分析、SNP标记Panel开发、分子标记辅助选择育种、QTL位点定位奠定了基础。
物种的遗传多样性越高对生存环境的适应性越强,抵御生物胁迫、非生物胁迫的能力越强,易于拓展其生存分布范围。本研究通过对来自不同地方的185份马铃薯品种(系)的遗传多样性分析发现,多态性信息含量PIC=0.3107,处于中等偏低水平;群体期望杂合度He=0.3932,而观测杂合度Ho=0.1852,即Ho<He。这一结果表明这些马铃薯品种(系)的遗传多样性较低,即不同品种(系)间的同质性较高,这与前人研究结果较为一致[14-18]。为了提升马铃薯品种的遗传多样性,在未来的马铃薯育种工作中应加强对马铃薯种质资源的收集和创新,特别是对马铃薯野生种的利用。马铃薯种质资源丰富,包含107个野生种和4个栽培种。原始栽培种和野生种含有众多调控马铃薯晚疫病、疮痂病、青枯病、病毒病、黑胫病抗性的等位基因,也含有能够调控马铃薯高淀粉和蛋白质含量、低还原糖含量、耐霜冻、休眠期、早熟等品质和农艺性状的优良等位基因[8]。加速对马铃薯种质资源的收集、评价、研究、创新和利用,有利于突破当前马铃薯育种的瓶颈[19-22]。
本研究结果表明,供试马铃薯种(系)间的遗传多样性较低,工作中也发现一些品种间表型差异很小,如果不借助分子标记技术,仅从表型差异很难区分。鉴于此,本研究还开发了包含120个马铃薯SNP-Panel,这些SNP标记均匀覆盖马铃薯12条染色体,并针对这些SNP标记设计了分离目标DNA区域的特异引物,以用于后续进行多样本高通量靶向SNP标记检测,为后续开展马铃薯亲缘关系鉴定、品种鉴别、群体遗传分析以及分子标记辅助选择育种奠定基础。
4 结论本研究对收集的185份马铃薯品种(系) 种质资源进行简化基因组测序,鉴定了39 038个变异位点,其中SNP位点36 267个,InDel位点2 771个;PCA分析和Structure分析均将供试的185份马铃薯品种(系)划分为两个亚类;遗传多样性分析表明,群体多态性信息含量PIC=0.3107,期望杂合度He=0.3932,观测杂合度Ho=0.1852,自交系数Fis=0.553,群体遗传多样性较低;开发了覆盖马铃薯12条染色体且包含120个SNP标记的SNP-Panel。本研究为后续进行马铃薯遗传多样性研究、分子标记辅助选择育种、分子身份证开发、品种鉴定、遗传图谱构建奠定了一定基础。
| [1] |
谢从华, 柳俊. 中国马铃薯引进与传播之辨析[J]. 华中农业大学学报, 2021, 40(4): 1-7. DOI:10.13300/j.cnki.hnlkxb.2021.04.001 XIE C H, LIU J. Review of introduction and spreed of potato in China[J]. Journal of Huazhong Agricultural University, 2021, 40(4): 1-7. DOI:10.13300/j.cnki.hnlkxb.2021.04.001 |
| [2] |
SPOONERD M, GHISLAIN M, SIMON R, JANSKY SH, GAVRILENKO Y. Systematics, diversity, genetics, and evolution of wild and cultivated potatoes[J]. Botanical Review, 2014, 80(4): 283-383. DOI:10.1007/s12229-014-9146-y |
| [3] |
TURRILL W B. Studies on the origin of cultivated plants[J]. Nature, 1926, 118: 2967. DOI:10.1038/118392a0 |
| [4] |
陈亚兰, 陈鑫. 马铃薯遗传育种技术[M]. 武汉: 武汉大学出版社, 2015. CHEN Y L, CHEN X. Potato genetics and breeding technology[M]. Wuhan: Wuhan University Press, 2015. |
| [5] |
WANG J. Genetic diversity of the selected 64 potato germplasms revealed by AFLP markers[J]. Molecular Plant Breeding, 2011(2): 22-29. DOI:10.5376/mpb.2011.02.0004 |
| [6] |
谷茂, 马慧英, 薛世明. 中国马铃薯栽培史考略[J]. 西北农业大学学报, 1999, 27(1): 80-84. GU M, MA H Y, XUE S M. A study on the cultivation history of potato in China[J]. Journal of Northwest A & F University, 1999, 27(1): 80-84. |
| [7] |
蔡兴奎, 谢从华. 中国马铃薯发展历史、育种现状及发展建议[J]. 长江蔬菜, 2016(12): 30-33. CAI X K, XIE C H. Development history, current breeding situation and development suggestions of chinese potato[J]. Journal of Changjiang Vegetables, 2016(12): 30-33. |
| [8] |
孙慧生. 马铃薯育种学[M]. 北京: 中国农业出版社, 2003. SUN H S. Potato breeding[M]. Beijing: China Agriculture Press, 2003. |
| [9] |
BOTSTEIN D R, WHITE R L, SKOLNICK M H, DAVIS R W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. The American Journal of Human Genetics, 1980, 32(3): 314-31. |
| [10] |
HENNING F, LEE H J, FRANCHINI P, MEYER A. Genetic mapping of horizontal stripes in Lake Victoria cichlid fishes: benefits and pitfalls of using RAD markers for dense linkage mapping[J]. Molecular Ecology, 2014, 23(21): 5224-5240. DOI:10.1111/mec.12860 |
| [11] |
SUTHERLAND B J G, GOSSELIN T, NORMANDEAU E, LAMOTHE M, ISABEL N, AUDET C, BERNATCHEZ L. Salmonid chromosome evolution as revealed by a novel method for comparing RADseq linkage maps[J]. Genome Biology and Evolution, 2016, 8(12): 3600-3617. DOI:10.1101/039164 |
| [12] |
杨琼, 孙娜, 郑敏, 罗除成, 罗光华, 方继朝. 水稻二化螟和三化螟基因组ddRADseq文库的构建[J]. 环境昆虫学报, 2016, 38(6): 1114-1120. YANG Q, SUN N, ZHENG M, LUO C C, LUO G H, FANG J C. Construnction of genomic ddRADseq libraries of Chilo suppressalis and scirpophaga incertulas(Lepidoptera: Pyralidase)[J]. Journal of Environmental Entomology, 2016, 38(6): 1114-1120. |
| [13] |
KONAR A, CHOUDHURY O, BULLIS R, FIEDLER L, KRUSER J M, STEPHENS M T, GAILING O, SCHLARBAUM S, COGGESHALL M V, STATON M E, CARLSON J E, EMRICH S, ROMERO-SEVERSON J. High-quality genetic mapping with ddRADseq in the non-model tree Quercus rubra[J]. BMC Genomics, 2017, 18(1): 417. DOI:10.1186/s12864-017-3765-8 |
| [14] |
韩志刚, 郝文胜, 谢锐, 郭景山, 伊六喜, 侯建华. 基于全基因组重测序SNP标记的148份马铃薯种质遗传多样性分析[J]. 西北植物学报, 2021, 41(8): 1302-1314. HAN Z G, HAO W S, XIE R, GUO J S, YI L X, HOU J H. Analysis of genetic diversity of 148 potato germplasm based on SNP markers from whole genome resequencing[J]. Acta Botanica Boreali-Occidentalia Sinica, 2021, 41(8): 1302-1314. |
| [15] |
宋峥, 王崇, 徐颖华, 雷剑, 柴沙沙, 王连军, 杨新笋. 马铃薯地方种质资源的SSR遗传多样性分析[J]. 分子植物育种, 2021, 1-21. SONG Z, WANG C, XU Y H, LEI J, CHAI S S, WANG L J, YANG X S. SSR analysis on genetic diversity for potato germplasm resources[J]. Molecular Plant Breeding, 2021, 1-21. |
| [16] |
高玉坤, 崔江慧, 项晓冬, 薛薇, 殷丛培, 任根增, 李峰, 王维东, 王志刚, 魏世林, 梁红凯, 常金华. 65个马铃薯品种(系)指纹图谱构建和遗传多样性分析[J]. 农业生物技术学报, 2020, 28(8): 1363-1378. GAO Y K, CUI J H, XIANG X D, XUE W, YIN C P, REN G Z, LI F, WANG W D, WANG Z G, WEI S L, LIANG H K, CHANG J H. Fingerprint construction and genetic diversity analysis for 65 potato (Solanum tuberosum)varieties(lines)[J]. Chinese Journal of Agricultural Biotechnology, 2020, 28(8): 1363-1378. |
| [17] |
王鹏, 李芳弟, 郭天顺, 窦俊焕, 颉炜清, 罗照霞, 齐小东, 杨晨, 赵中梁, 宋怡, 吕汰. SSR标记马铃薯育成品种的遗传多样性分析[J]. 中国马铃薯, 2019, 33(5): 257-266. WANG P, LI F D, GUO T S, DOU J H, JI W Q, LUO Z X, QI X D, YANG C, ZHAO Z L, SONG Y, LU T. Analysis of genetic diversity for potato(Solanum tuberosum L.)varieties using SSR markers[J]. Chinese Potato Journal, 2019, 33(5): 257-266. |
| [18] |
张海雯, 巩檑, 马斯霜, 甘晓燕, 宋玉霞. 基于SSR标记的马铃薯种质遗传多样性及群体结构分析[J]. 分子植物育种, 2020, 18(12): 4144-4152. DOI:10.13271/j.mpb.018.004144 ZHANG H W, GONG L, MA S S, GAN X Y, SONG Y X. Genetic diversity and population structure analysis of potato(Solanum tuberosum)germplasm based on SSR markers[J]. Molecular Plant Breeding, 2020, 18(12): 4144-4152. DOI:10.13271/j.mpb.018.004144 |
| [19] |
HUANG B Q, RUESS H, LIANG Q Q, COLLEONI C, SPOONER D M. Analyses of 202 plastid genomes elucidate the phylogeny of solanum section petota[J]. Scientific Reports, 2019(9): 4454. DOI:10.1038/s41598-019-40790-5 |
| [20] |
GAVRILENKO T, ANTONOVA O, SHUVALOVA A, KRYLOVA E, ALPATYEVA N, SPOONER D, NOVIKOVA L. Genetic diversity and origin of cultivated potatoes based on plastid microsatellite polymorphism[J]. Genetic Resources and Crop Evolution, 2013, 60(7): 1997-2015. DOI:10.1007/s10722-013-9968-1 |
| [21] |
SPOONER D M, RUESS H, ARBIZU C I, RODRIGUEZ F, SOLISLEMUS C. Greatly reduced phylogenetic structure in the cultivated potato clade(Solanum section petota pro parte)[J]. American Journal of Botany, 2018, 105(1): 60-70. DOI:10.1002/ajb2.1008 |
| [22] |
SPOONER D M. Species delimitations in plants: lessons learned from potato taxonomy by a practicing taxonomist[J]. Journal of Systematics and Evolution, 2016, 54(3): 191-203. DOI:10.1111/jse.12203 |
(责任编辑 崔建勋)
 2021, Vol. 48
2021, Vol. 48