文章信息
基金项目
- 广东省农业科学院作物研究所所长基金(202103);广东省农业科学院中青年学科带头人培养项目(R2019PY-JX003);广东省农业科学院优秀博士人才项目(R2021YJ-YB2008)
作者简介
-
张闻婷(1988—),女,博士,研究方向为南药植物基因组学,E-mail:zhangwenting@gdaas.cn
张闻婷,毕业于中国科学院大学,遗传学专业博士学位,上海师范大学环境生物学博士后,2021年8月入职广东省农业科学院作物研究所,优秀博士项目人才。主要从事华南特色作物(南药)基因组学研究工作。参加科技项目5项,发表SCI论文7篇,其中第一作者或共同第一作者4篇; 王继华,博士,研究员,硕士生研究生导师,广东省现代南药(化橘红) 产业技术体系育种岗位科学家,广东省农业科学院金颖之星,中国中药协会中药材种养殖专业委员会技术专家和国家南药科技创新联盟副理事长。
主要从事华南特色作物(南药) 的育种与栽培研究工作。主持和参加科技项目30多项,获广东省自然科学三等奖与广东省农业技术推广奖二等奖各1项。在国内外学术期刊发表研究论文50多篇,获授权发明专利15项,转让4项,出版专著1本.
通讯作者
- 王继华(1979—),男,博士,研究员,研究方向为南药栽培与育种,E-mail:wangjihua@gdaas.cn.
文章历史
- 收稿日期:2021-10-18
2. 华南农业大学生命科学学院,广东 广州 510642
2. College of Life Sciences, South China Agricultural University, Guangzhou 510642, China
药用植物被视为发现和开发新药物和疗法的重要自然资源。据估计,世界上约有75% 的人口,特别是居住在发展中国家的人口,使用植物作为民间药物的来源,以满足他们的初级保健需求[1]。世界卫生组织(WHO)已记录了全球300 000种植物中的21 000种具有药用价值的植物[2]。药用植物是各种生物活性物质的主要来源,包括生物碱、萜类、多酚、香豆素、皂苷等。植物生物活性物质的药用保健功能和治疗效果主要归因于其多种生物学和药学作用,例如抗氧化、抗菌、抗炎、抗癌、抗糖尿病活性。利用草药治疗各种人类疾病是世界性和普遍性的,因其有害反应较少且比现代药物便宜而在制药业中受到越来越多的关注。
基因组学研究阐明了基因组序列中嵌入的遗传组成、结构、组织、功能、多样性和相互作用,从基因组的分子、染色体、生化和表型水平研究变化情况,涵盖基于基因组组装展开的注释功能和非功能元件、识别重复序列、研究基因组区域的表观遗传状态和质体基因组的序列信息等结构基因组方面工作,以及基于基因转录、翻译和调控展开的转录组学、蛋白质组学和代谢组学等功能基因组方面工作。三七作为药用成分人参皂苷的重要原料,种植历史悠久,应用范围广,经济价值显著,目前已先后公布四版三七的基因组序列信息,对三七基因组信息的探索和挖掘可以为异源生产皂苷生物合成途径提供宝贵资源[3-4]。中国罂粟地方品种的基因组测序扩展了这一宝贵药用物种的可用遗传信息,从而能够系统地研究BIA的生物合成、调控和运输,用于农艺和制药目的;基因组组装和分析将成为评估罂粟药理学、化学成分、栽培、性状遗传改良和种群的未来研究的有用资源[5]。基因组序列为传统药材作物遗传改良的进化、变异和性状研究提供宝贵的资源,对药用植物基础生物学研究和应用育种计划都非常有价值。
本文综述了药用植物基因组学的最新研究进展,涵盖药用植物基因组测序、结构基因组学和功能基因组学三方面研究方向,讨论了药用植物遗传作图、全基组组装、质体序列价值以及比较基因组学的应用,并从转录组、蛋白组、代谢组三方面综合分析了药用植物功能基因组的研究策略和关注重点,在此基础上,对药用植物在表观基因组学和泛基因组学方面进行展望,以期推动药用植物基因组学长足发展。
1 基因组测序研究基因组的DNA序列整合了物种起源、发育和表观基因组调控的重要信息。基因组学领域的最新进展很大程度上归因于增加的通量和降低的成本对DNA进行测序的能力[6]。人类基因组计划和其他主要的基因组测序项目推动了测序技术的发展。
1.1 测序技术概述由Frederick Sanger提出的链终止法[7]以及Walter Gibert发明的链降解法[8]被称为第一代测序技术(FGST),是1977年首次成功采用的测序方法,因其操作简单、准确率高、读长较长的优势而被广泛应用,基于该技术启动[9]并完成了人类基因组计划[10]。虽然多种新一代测序技术(the next generation sequencing, NGS)应运而生,但第一代测序技术因其高度的准确性仍然被作为验证新一代测序技术结果的“黄金标准”。
第一代测序技术的读长可以达到700~1 000 bp,但每个反应只能得到一条序列,测序通量低,无法满足大规模的基因测序。鉴于此,第二代测序技术应运而生,可以实现一次对几十万到几百万条核酸分子进行序列测定,也称为高通量测序技术(High-throughput sequencing, HTS),其读长和准确率虽然不及第一代测序技术,但HTS的应用使得对一个物种的转录组和基因组进行细致全貌的分析成为可能,帮助基因组学和分子生物学的研究取得突破性进展。二代测序平台主要有Roche公司的454、Illumina公司的Solexa和Hiseq、ABI公司的Solid、Themo fisher公司的IonTorrent和华大基因的BGISEQ等,它们都基于相似的原理,实现边合成边测序[11-12]。
与二代测序相比,第三代测序技术真正实现了单分子测序,无PCR扩增偏好性和GC偏好性,具有超长的测序读长,平均测序读长达到10~15 kb,最长读长40 kb,同时三代测序技术还可直接检测碱基修饰,如DNA甲基化等,为表观遗传学研究提供了有力手段。PacBio平台的HiFi模式生成DNA模板的长高保真循环连续一致(CCS) 读数,实现了高精度(99.8%)的长序列测序[13]。三代测序技术正被应用于高质量的基因组测序及组装相关工作。
基于纳米孔的测序仪作为第四代DNA测序技术,有可能以不到1 000美元,甚至可能不到100美元的价格快速可靠地对整个人类基因组进行测序,该技术使用的单分子技术使我们能够进一步研究DNA与蛋白质之间,以及蛋白质与蛋白质之间的相互作用[14]。
1.2 药用植物数据库随着下一代测序技术的高速发展,基因组数据库的主要功能已经从数据存储发展成为整合各种基因组信息的基因组门户/ 中心,以及提供在线基因组学分析的网络服务器,引领基因组测序和重测序项目的拼图。迄今为止,专门针对药用植物的基因组数据库很少,金银花(Lonicera japonica, http://www.gzybioinformatics.cn/LjaFGD/index.php)、杜鹃(Rhododendron,http://bioinfor.kib.ac.cn/RPGD/)和百脉根(Lotus japonicus, http://www.kazusa.or.jp/lotus/)等药用植物已陆续建立各自独立的基因组数据库,迫切需要建立一个以药用植物为重点的综合基因组序列信息和药理属性相关的数据库。
2021年8月,陈士林团队在Chinese Medicine和Science China Life Sciences杂志上分别发表了BPGD(Brazilian Pharmacopoeia Genomic Database, http://www.bpgenome.com) [15]和GPGD(Global Pharmacopoeia Genome Database,http://www.gpgenome.com) [16]数据库相关文章,可以说是首个药用植物基因组数据库。这两个数据库是获取和分析药用植物DNA条形码数据、加速药用植物研究并促进其市场监管合理发展的有效平台。
MepmiRDB是第一个药用植物MicroRNA (miRNA) 数据库,其中包含了29种药用植物miRNA的信息(http://mepmirdb.cn/mepmirdb/index.html) [17]。由于生物活性成分的主要部分是通过次级代谢途径产生的,miRNA在调节次生代谢物的生物合成和积累对药用植物尤为重要。
此外,已经建立了大量的药用植物及其衍生植物化学物质的公共数据库,包括UMPDB[18]、MPD3[1]、IMPPAT[19]、Phytochemica [20]、SerpentinaDB[21]、FERN Ethnomedicinal Plant Database [22]、NPACT [23]、HIT [24]、CHCD[25]、NuBBEDB[26]、NANPDB[27]等。这些数据库对于药用植物的传统信息(包括分类学、通用名称、位置、药用部位和用途)、化学信息和基因组信息(依赖NCBI)给予了较为详细的注释,为研究药用植物的药效属性提供便利。
2 结构基因组学研究 2.1 遗传作图遗传连锁图谱的构建是基因组研究中的重要环节,是基因定位与克隆乃至基因组结构与功能研究的基础。传统的利用正向遗传学方法的基因定位一般是通过构建遗传连锁图谱进行,但由于多态性标记鉴定步骤繁琐,而且多数情况下定位精确度低、区间大,导致全过程耗时耗力。大多数药用植物生长周期长,栽培历史短,遗传背景复杂,基因高度杂合,为建立药用植物的遗传图谱带来了较大的困难,所以目前建立的较为完善的药用植物遗传图谱很少。
利用传统的SSR、AFLP、RFLP、RAPD与EST-SSR等分子标记构建的药用植物遗传图谱有红菽草(Trifolium pratense) [28]、洋蓟(Cynara scolymus) [29]、白玉草(Silene vulgaris) [30]和百香果(Passiflora edulis) [31]。随着高通量测序技术的快速发展以及测序成本的不断降低,植物基因组参考序列得以释放促进全基因组范围分子标记的开发,有力推动药用植物分子遗传图谱的研究向着高饱和化、实用化和通用化的方向发展。例如,通过构建雌雄异株植物戟叶酸模(Rumex hastatus)的连锁图谱和染色体规模的基因组组装,发现低重组率可能促进植物性染色体进化[32]。此外,五节芒(Miscanthus floridulus) [33]高密度遗传图谱证实Hi-C组装的物理图谱,两者在染色体分配和顺序方面一致;长叶薄荷(Mentha longifolia(L.)Huds.)全基因组测序和组装,并构建抗黄萎病和感黄萎病亚种间的遗传图谱,最终锚定在12条假染色体上[34]。
2.2 基因组de novo测序药用植物基因组大小变化范围广,目前已知最大的是宽瓣重楼[35],其基因组达到82.55 Gb,而我们所熟知的银杏[36]也有9.87 Gb,此外如罂粟[5]、三七[3]、姜和地黄等物种的基因组也都大于2 Gb;基因组较小的药用植物有掌叶覆盆子(240 Mb)、穿心莲(270 Mb)和菟丝子(270 Mb)等。药用植物多数都是二倍体植物,比较少数的多倍体,如地黄[37]作为同源四倍体其染色体的数目也达到4n=56,广藿香[38]约在631万年前经历了全基因组八倍化(WGO,whole-genome octuplication)复制事件。而染色体数较少药用植物有菘蓝(2n=14)和香椿(2n=16)等。
随着基因组测序技术的飞速发展,已有上百种药用植物获得了全基因组序列(图 1)。近两年三代测序技术广泛普及,利用二代Illumina、三代Pacbio或Nanopore以及Hi-C挂载,完成了大量药用植物染色体水平的基因组测序和组装。本团队通过陆续采集上百种南方道地药材,对其中20多种完成全基因组调查(Genome survey),并完成了巴戟天[39]的全基因组测序和组装。巴戟天作为中国南方岭南四大草药之一,主根入药广泛用于治疗各种疾病,如阳痿、不孕症、月经不调、风湿病、关节痛等。巴戟天高质量基因组组装为了解该物种的进化、活性成分生物合成和遗传改良提供了宝贵的基因组资源。此外,四大南药之一的槟榔[40]基因组也已公布,益智和砂仁的基因组暂时还未破解。
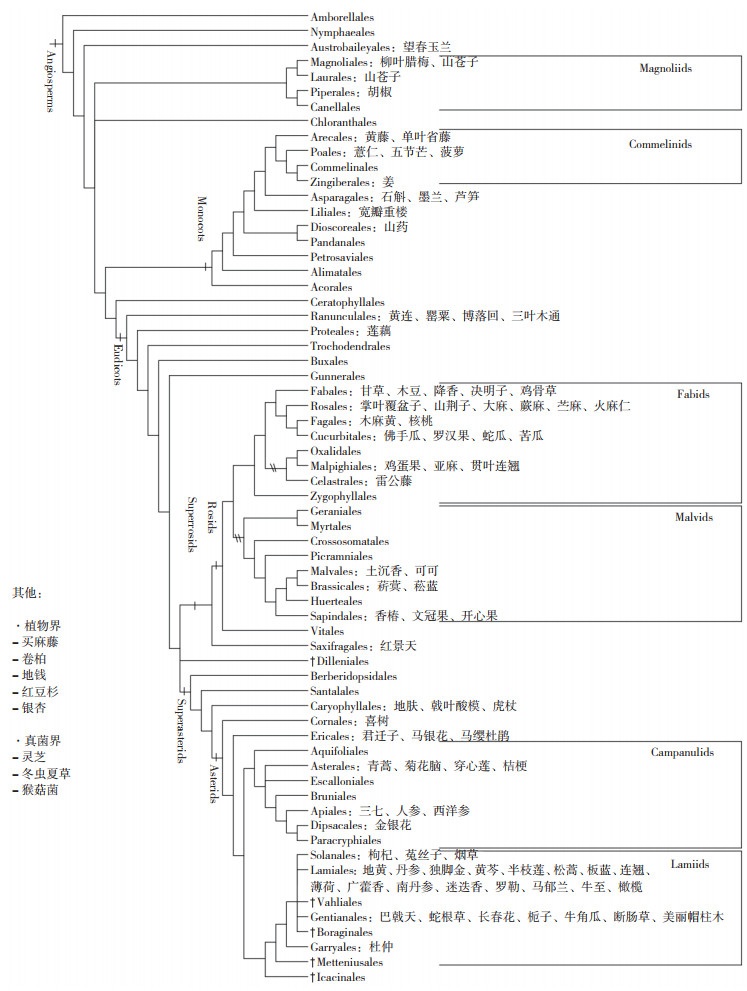
|
| 图 1 完成全基因组测序和组装的药用植物举例 Fig. 1 Examples of medicinal plants that have completed whole-genome sequencing and assembly |
有意思的是药用植物中文名相似的植物亲缘关系可能离得非常远,例如贯叶连翘和连翘其实是两个完全不同目的物种。贯叶连翘[41]是蔷薇类植物,基因组大小在373 Mb左右,2n=2x=16,对治疗抑郁症具有一定的效用。而连翘[42]是菊类植物,基因组达到737.5 Mb,2n=2x=28,清热解毒常用治外感风热。
除了被子植物外,石松门的卷柏、苔藓植物门的地钱、松柏门的红豆杉以及买麻藤门的买麻藤,鉴于经济价值和药用价值突出,基因组序列的公布也受到广泛关注。此外,在我们熟知的一些物种中,例如灵芝、冬虫夏草、猴菇菌,虽然不属于植物但具有悠久的药材使用历史。以上这些特殊的药用物种均已公布基因组序列。基因组测序技术的发展推动了药用植物鉴定、起源、进化、药用化合物的分子表征、非生物胁迫抗性等研究,促进了后续功能基因组的研究,对研究药用植物的育种栽培、代谢产物、功能调控和药理属性提供了大量生物信息和遗传信息数据。
2.3 叶绿体和线粒体基因组叶绿体是在植物、藻类和蓝细菌中发现的具有代谢活性的半自主细胞器,其主要功能是进行光合作用,还参与脂肪酸、氨基酸、激素、维生素、核苷酸和次生代谢产物的生物合成[43]。叶绿体基因组可以母系遗传,基因含量和顺序高度保守,分子进化缓慢,重组率低,使其成为物种鉴定和系统发育研究的理想材料[44-45]。叶绿体非编码区已成功应用于DNA条形码研究[46]。药用植物叶绿体基因组组装对探索叶绿体功能与其适应性和植物化学特征之间的关系具有重要意义。
叶绿体基因组具有保守的四分体结构:一个大的单拷贝(LSC) 区域、一个小的单拷贝(SSC)区域和两个反向重复(IR)区域。叶绿体基因组分析通常从测序确定分子结构、注释获得编码基因、系统发育、进化和比较等方面展开。本团队已陆续发表多种药用植物叶绿体基因组文章,包括台湾榕(Ficus formosana) [47]、梅片树(Dryobalanops aromatica) [48]、凉粉草(Mesona chinensis) [49]、扁桃斑鸠菊(Vernonia amygdalina) [50]、白簕(Acanthopanax trifoliatus) [49]、华山姜(Alpinia chinensis) [51]和龙脷叶(Sauropus spatulifolius) [52]。药用植物丹参的叶绿体基因组序列受到广泛关注,从Salvia miltiorrhiza[53]、S. yangii[54]到S. przewalskii、 S. bulleyana和S. japonica[55],陆续有丹参属的物种叶绿体基因组被公布。2021年6月,有研究提供了跨属的(Cadaba和Maerua)4种山柑科(Capparaceae) 药用植物(即C. farinosa、C. glutenulosa、M. crassifolia和M. oblongifolia) 完整叶绿体基因组[56]。此外,山槐[57]、青蒿[58]、芍药[59]、马兜铃属[60]等药用植物的完整叶绿体基因组也陆续公布。药用植物叶绿体基因组的研究为品种鉴定和高药效植物选育提供了有价值的信息。
与叶绿体基因组不同的是,植物线粒体基因具有许多独特的特征,表现出物种之间的广泛差异,目前为止仅有200多个陆地植物的线粒体基因组被公布[61]。丹参的线粒体基因组上发现长片段的叶绿体基因组序列,暗示两者间的基因转移[54]。通过对人参(Panax ginseng CA Mey)线粒体基因组进行测序和表征,开发了用于阐明人参种质多样性的KASP标记[62]。此外,药用植物毛茛科宽裂北乌头(Aconitum kusnezoffii) [63]和豆科乌拉尔甘草(Glycyrrhiza uralensis)的完整线粒体基因组[64]均于2021年发表公布。
2.4 比较基因组比较基因组学通过比较不同生物的基因组序列,在分子水平上了解不同生命形式的区别,为研究生物体之间的进化与变异提供了强大工具,有助于识别物种间保守或共有的基因,以及赋予每个生物体独特特征的序列变化。随着测序技术的简单化和测序成本的降低,比较基因组学作为一种梳理物种之间微妙差异的工具,在生物学研究中得到广泛应用。
通过对决明子(Senna tora)和其他14个豆科植物的比较基因组分析,发现决明子具有丰富的物种特异基因,其中扩张的基因家族多富集在苯丙烷、异黄酮和萜烯生物合成相关基因,为研究植物中蒽醌生物合成途径提供更多的理论依据[65]。有籽和无籽君迁子(Diospyros lotus) 基因组之间的比较为阐明无籽发育提供分子基础[66]。黄芩(Scutellaria baicalensis)和半枝莲(Scutellaria barbata)的染色体水平基因组组装实现两者比较基因组分析,基于基因共线性发现两物种间的染色体数目变异和结构重排,DNA序列水平的比对显示两者基因组之间大规模结构变异,单拷贝同源蛋白重建的系统发育分析表明两者与丹参(Salvia miltiorrhiza)亲缘关系最密切[67]。佛手瓜(Sechium edule)、蛇瓜和南瓜的染色体共线性分析显示佛手瓜和南瓜间存在大量同义基因块,而佛手瓜和蛇瓜间则发生了更多的染色体间重排事件[68]。
基于罂粟地方品种CHM和先前发表品种HN1基因组比较,结合罂粟科其他物种的LTR逆转录转座子的进化分析,发现Gypsy和Copia家族的大量增殖是最有可能推动罂粟基因组扩张的主要因素;CHM和HN1基因组中特异基因家族分析显示,CHM中的独特基因主要富集在涉及碳固定和代谢的功能类别,表明CHM作为野生种质可以为驯化罂粟的种质改良提供宝贵的遗传资源[5]。基于三代测序技术已经先后完成多套染色体规模的大麻全基因组组装,包括产生高THC的雌性株系PK、雄性株系Finola、产生高CBD的CBDRx(cs10)株系、野生大麻雌性株系JL以及产生平衡CBD: THC大麻素比例的雌性株系Cannbio-2,其基因组大小在600~800 Mb范围,BUSCO评估证实Cannbio-2基因组序列组装的完整性最好[69]。基于对40株大麻后代栽培种的二代重测序数据,与父本基因组比较分析实现了大麻Y染色体的识别;拷贝数变异(CNV)和RNA表达评估确定了多个控制大麻素表达的CNV和抗性相关的基因[70]。除了罂粟和大麻之外,已有多个针对同种药用植物的基因组版本公布,如黄连、姜、穿心莲和鸡蛋果等。
3 功能基因组学研究功能基因组学建立在结构基因组学的基础上,利用转录组和蛋白组等项目产生的大量数据,试图描述基因的功能和相互作用,侧重于基因转录、翻译、表达调控和蛋白质相互作用等动态信息。
3.1 转录组学转录组学技术是用于研究生物体其所有RNA转录本总和的技术,其中mRNA在信息网络中充当瞬时中介分子,而非编码RNA则执行其他各种功能。转录组学研究领域主要有两种技术:微阵列,可以量化一组预定序列;RNA测序(RNASeq),使用高通量测序来捕获所有序列。测量生物体基因在不同组织、环境或时间点的表达,可以提供有关基因调控方式的信息,并揭示生物体生物学的细节,还可以帮助推断以前未注释的基因的功能[71]。虽然越来越多药用植物的基因组序列得到解析公布,但还是有大量的药用植物缺乏基因组信息,在功能基因和遗传机制方面的研究远远不足,因此将转录组测序应用于药用植物至关重要。
药用植物转录组学研究主要从4个方面展开:功能基因挖掘、分子标记开发、次级代谢物生物合成途径的鉴定和药用植物发育机制的鉴定[72]。研究者使用NGS技术对天麻(Gastrodia elata) 进行转录组测序和分析,鉴定出34 322个转录本,其中2 007个(5.85%)转录本包含至少1个SSR,在这些SSR中AG/CT重复基序是最常见的,共检测到498个,该结果为天麻代谢、生长和发育的分子机制研究提供了更深入的了解[73]。三萜皂苷是鸭腱藤(Entada phaseoloides)中的主要生物活性化合物,通过对鸭腱藤的根、茎和叶组织进行比较转录组分析,鉴定了26个细胞色素P450和17个与三萜皂苷生物合成相关的尿苷二磷酸糖基转移酶候选基因,为研究三萜皂苷生物合成的功能基因组学提供便利[74]。李等人使用转录组测序来分析丹参不同组织对中度干旱胁迫响应的分子机制,GO富集分析显示根和叶的差异转录本在代谢过程和催化活性中显着富集,在中等干旱胁迫下,编码参与苯丙烷和萜类化合物生物合成的关键酶的基因表达上调,该研究结果为进一步研究丹参药用成分的生物合成机制和栽培中的有效灌溉方法提供了科学依据[75]。
此外,本团队在解析巴戟天基因组的同时,采集了茎、叶、1年生根(AR)、3年生根(TR) 和6年生根(SR)组织材料,并提取RNA进行转录组测序;通过分析组织特异性表达的基因簇,发现与活性成分的合成、运输和储存以及防御反应相关的基因随着根发育而表现出不断增强的表达模式,这结果为进一步揭示基因表达调控网络和调控巴戟天生物活性代谢物及其衍生物的产生提供了基础[39]。基于大麻Purple Kush和Finola株系的转录组比较分析表明,许多编码参与大麻素和前体途径的蛋白质的基因在Purple Kush中的表达比在Finola中更高,检测到Δ9- 四氢大麻酚酸合酶在Purple Kush中有表达而在Finola中被大麻二酚酸合酶取代,从基因表达水平解释了Purple Kush株系可以而Finola不能产生大麻素Δ9- 四氢大麻酚(THC)的原因[76]。
3.2 蛋白质组学植物蛋白质组巨大的功能、空间和时间多样性受到多种因素的调控,这些因素不断改变蛋白质的丰度、修饰、相互作用、定位和活性,以满足植物的动态需求。剖析蛋白质组的复杂性及其潜在的遗传变异正引起越来越多的研究关注[77]。由于质谱、样品制备程序、分析软件的显著改进以及众多植物物种基因组可用性的增强,现在可以进行大规模基于质谱的植物蛋白质组学研究,并提供更全面的概述[78]。
基因组测序、计算机预测和基于质谱的肽组学的进步促进了越来越高通量的生物活性肽的发现,通过基于MS的蛋白质组学/ 肽组学对三色苋(Amaranthus tricolor)气生组织进行了分析,识别了计算机预测的抗菌肽(AMP),蛋白质组学以高通量的方式鉴定了新型抗菌肽[79]。通过无标记定量蛋白质组学方法对高丽参(Korean Ginseng)的叶、根、芽和果实进行比较蛋白质组分析,最终在4种组织中鉴定出2 604种显著调节的蛋白质,并确定与人参皂苷生物合成相关的差异途径和蛋白质,包括甲基赤藓糖醇4- 磷酸(MEP)途径、甲羟戊酸(MVA)途径、UDP- 糖基转移酶(UGT)和氧化还原酶(CYP450s)[80]。通过对胡黄连(Picrorhiza kurroa)叶和根茎进行特异性蛋白质组学分析,观察到在CO2增强情况下与光合作用和碳代谢相关的蛋白质丰度以组织特异性方式发生变化,该研究为药用植物将如何应对不断变化的环境以及适应性反应提供更好的理解[81]。基于对青蒿的全局蛋白质组学分析以及高(HAP)和低(LAP)青蒿素含量两种化学型的定量靶向亚蛋白质组学分析,分别在质谱的结果和基因组序列注释的基础上鉴定了182和13 403种蛋白质[82]。双向电泳鉴定了群心菜(Lepidium draba)缺水胁迫(6% PEG)下20个与光合作用、能量代谢等功能相关的差异表达蛋白[83]。通过对铁线莲(Clematis florida)耐热株系(PS)和热敏感株系(SG)进行蛋白质组学分析,在蛋白质水平上表征两者对热应激的不同耐热反应[84]。
总之,药用植物的蛋白质组学提供了关于蛋白质的结构、功能和翻译后修饰、蛋白质- 蛋白质相互作用、植物性生物活性药物的蛋白质靶标、分子水平的蛋白质- 药物相互作用以及细胞中各信号通路的信息,这些提供了对药用植物在人类疾病细胞中的治疗机制的深入了解,以及对药用植物可遗传性状和生理状态的复杂理解。在生物学或医学背景下蛋白质组学信息的积累有助于制定药用植物的有效使用方法。
3.3 代谢组学代谢组学是一种用于综合分析植物化学物质的现代组学技术。药用植物生物合成的代谢产物作为活性成分不仅具有药用价值,而且在植物对环境的适应和植物抵抗外界胁迫方面发挥着不可或缺的作用。在不同的生长环境、生长阶段和器官中,代谢产物的积累存在特定差异。尽管在异源宿主中生产药用化合物的合成生物学取得了进展,但天然植物物种通常是其生产的最可靠和最经济的来源。因此,研究药用植物的代谢成分以表征其天然代谢物多样性并确定重要化合物的植物体内生物合成途径,从而制定进一步提高其含量的策略,就变得至关重要。
基于佛手瓜基因组解析,对其3、6、9 d的果实进行代谢组的比较分析,鉴定了一系列差异表达的代谢物,帮助理解果实发育相关的代谢调控途径[68]。研究者使用高效液相色谱技术在板蓝(Strobilanthes cusia)4个发育阶段测量了不同组织(包括根、茎和叶)中3种主要次生代谢物(indicant、indigo、indirubin)的含量,结果表明这3种代谢物主要在叶组织中合成和积累,其次是茎,最后是根[85]。通过对生姜根茎进行发育阶段的划分并利用UHPLC-MS/MS测定各个阶段的活性化合物,发现在从成熟根茎到新发芽根茎的过程中,大部分氨基酸和多半脂类物质的含量升高,而大多数有机酸、核苷酸、维生素和次级代谢物的水平趋于降低[86]。主成分分析(PCA) 显示贯叶连翘(Hypericum perforatum) 和粉色西番莲(Passiflora incarnate) 之间的代谢组学特征相似,而缬草(Valeriana officinalis)和蜜蜂花(Melissa officinalis)具有不同的代谢组学特征,该结果对于分类属性来讲是一致的[87]。利用同位素和质谱相结合的方法开展了短小蛇根草(Ophiorrhiza pumila)的代谢组分析,鉴定出独特且多样的含氮代谢物,其中MIA(monoterpene indole alkaloids)、IA(indole alkaloids) 和蒽醌(anthraquinones)以组织特异性方式积累,在根和毛根中含量最高,在叶组织中含量低[88]。通过对黄芩和半枝莲的根、茎、叶和花组织进行活性化合物检测,发现黄芩素(Baicalein)、去甲汉黄芩素(norwogonin)、汉黄芩素(wogonin)、黄芩苷(baicalin)、去甲汉黄芩苷(norwogonoside) 和汉黄芩苷(wogonoside)主要在根部积累,而野黄芩苷(scutellarin)主要分布在地上部分的器官(茎、叶和花)中,证实了生物活性化合物的器官特异定位[67]。
代谢组获得的数据可以为药用植物活性成分的积累和有效利用信息提供科学依据。GC-MS和LC-MS等基于质谱的平台技术进步有助于分离和鉴定多种代谢物。随着草药基因组计划的实施和中医药合成生物学的发展,药用植物的代谢组学研究将迎来一个蓬勃发展的新时期。
4 展望药用植物在泛基因组学方面的研究面临巨大挑战,很大原因在于药用植物种质资源匮乏,受全球消费需求和环境因素的影响育种工作难以开展,世界范围内物种加速丧失和栖息地破坏增加了药用植物灭绝的风险。药用植物的育种比农作物要复杂得多,需要考虑植物的有效成分含量、产量、药用部位和生长周期,多数药用植物的品质与其栖息地关系紧密[89]。在多方组织和研究机构的共同努力下,希望全球药用植物资源保护和可持续利用战略和方法可以得到广泛普及。药用植物表观基因组学研究也还处于萌芽阶段,从DNA或者RNA修饰水平关注药用植物亚种间表型、活性化合物以及环境适应性等方面的差异,为功能基因组研究提供宝贵的资源。
药用植物具有显著的经济和社会效益。基于结构基因组学和功能基因组学展开的多组学分析,可以更全面、深入和精确地解析药用植物复杂药理属性形成的分子机制和调控机理。未来几年,基因组学的研究将以前所未有的速度揭开药用植物化学多样性和特殊代谢物分子功能的神秘面纱。一旦阐明了涉及药用植物中特定化合物生物合成途径的基因,就可以应用合成生物学或基因组编辑在工程生物中生产。药用植物基因组工程可以极大地促进药用植物生物活性成分生物合成机制的研究,并将中药研究推向生命科学领域的前沿。
| [1] |
MUMTAZ A, ASHFAQ U A, UI QAMAR M T, ANWAR F, GULZAR F, ALI M A, SAARI N, PERVEZ M T. MPD3: a useful medicinal plants database for drug designing[J]. Natural Product Research, 2017, 31(11): 1228-1236. DOI:10.1080/14786419.2016.1233409 |
| [2] |
PANDITA D, PANDITA A, WANI S H, ABDELMOHSEN S A M, ALYOUSEF H A, ABDELBACKI A M M, AL-YAFRASI M A, ALMANA F A, ELANSARY H O. Crosstalk of multi-omics platforms with plants of therapeutic importance[J]. Cells, 2021, 10(6): 1296. DOI:10.3390/cells10061296 |
| [3] |
FAN GY, LIU XC, SUN S, SHI CC, DU X, HAN K, YANG B R, FU Y Y, LIU M H, SEIM I, ZHANG H, XU Q W, WANG J H, SU X S, SHAO L B, ZHU Y F, SHAO Y C, ZHAO Y P, WONG A K C, ZHUANG D, CHEN W B, ZHANG G Y, YANG H M, XU X, TSUI S K, LIU X, LEE S M. The chromosome level genome and genome-wide association study for the agronomic traits of Panax notoginseng[J]. Science, 2020, 23(9): 101538. DOI:10.1016/j.isci.2020.101538 |
| [4] |
JIANG Z Q, TU L C, YANG W F, ZHANG Y F, HU T Y, MA B W, LU Y, CUI X M, GAO J, WU X Y, TONG Y R, ZHOU J W, SONG Y D, LIU Y, LIU N, HUANG L Q, GAO W. The chromosome-level reference genome assembly for Panax notoginseng and insights into ginsenoside biosynthesis[J]. Plant Communications, 2021, 2(1): 100113. DOI:10.1016/j.xplc.2020.100113 |
| [5] |
PEI L, WANG B S, YE J, HU X D, FU L H, LI K, NI Z Y, WANG Z L, WEI Y J, SHI L Y, ZHANG Y, BAI X, JIANG M W, WANG S H, MA C L, LI S J, LIU K H, LI W S, CONG B. Genome and transcriptome of Papaver somniferum Chinese landrace CHM indicates that massive genome expansion contributes to high benzylisoquinoline alkaloid biosynthesis[J]. Horticulture Research, 2021, 8(1): 5. DOI:10.1038/s41438-020-00435-5 |
| [6] |
NIEDRINGHAUS T P, MILANOVA D, KERBY M B, SNYDER M P, BARRON A E. Landscape of next-generation sequencing technologies[J]. Analytical Chemistry, 2011, 83(12): 4327-4341. DOI:10.1021/ac2010857 |
| [7] |
SANGER F, NICKLEN S, COULSON A R. DNA sequencing with chain-terminating inhibitors[J]. Proceedings of the National Academy of Sciences, 1977, 74(12): 5463. DOI:10.1073/pnas.74.12.5463 |
| [8] |
MAXAM A M, GILBERT W. A new method for sequencing DNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(2): 560-564. DOI:10.1073/pnas.74.2.560 |
| [9] |
WATSON J D. The human genome project: past, present, and future[J]. Science, 1990, 248(4951): 44-49. DOI:10.1126/science.2181665 |
| [10] |
LANDER E S, LINTON L M, BIRREN B, NUSBAUM C, ZODY M C, BALDWIN J, DEVON K, DEWAR K, DOYLE M, FITZHUGH W, FUNKE R, GAGE D, HARRIS K, HEAFORD A, HOWLAND J, KANN L, LEHOCZKY J, LEVINE R, MCEWAN P, MCKERNAN K, MELDRIM J, MESIROV J P, MIRANDA C, MORRIS W, NAYLOR J, RAYMOND C, ROSETTI M, SANTOS R, SHERIDAN A, SOUGNEZ C, STANGE-THOMANN N, STOJANOVIC N, SUBRAMANIAN A, WYMAN D, ROGERS J, SULSTON J, AINSCOUGH R, BECK S, BENTLEY D, BURTON J, CLEE C, CARTER N, COULSON A, DEADMAN R, DELOUKAS P, DUNHAM A, DUNHAM I, DURBIN R, FRENCH L, GRAFHAM D, GREGORY S, HUBBARD T, HUMPHRAY S, HUNT A, JONES M, LLOYD C, MCMURRAY A, MATTHEWS L, MERCER S, MILNE S, MULLIKIN J C, MUNGALL A, PLUMB R, ROSS M, SHOWNKEEN R, SIMS S, WATERSTON R H, WILSON R K, HILLIER L W, MCPHERSON J D, MARRA M A, MARDIS E R, FULTON L A, CHINWALLA A T, PEPIN K H, GISH W R, CHISSOE S L, WENDL M C, DELEHAUNTY K D, MINER T L, DELEHAUNTY A, KRAMER J B, COOK L L, FULTON R S, JOHNSON D L, MINX P J, CLIFTON S W, HAWKINS T, BRANSCOMB E, PREDKI P, RICHARDSON P, WENNING S, SLEZAK T, DOGGETT N, CHENG J F, OLSEN A, LUCAS S, ELKIN C, UBERBACHER E, FRAZIER M, GIBBS R A, MUZNY D M, SCHERER S E, BOUCK J B, SODERGREN E J, WORLEY K C, RIVES C M, GORRELL J H, METZKER M L, NAYLOR S L, KUCHERLAPATI R S, NELSON D L, WEINSTOCK G M, SAKAKI Y, FUJIYAMA A, HATTORI M, YADA T, TOYODA A, ITOH T, KAWAGOE C, WATANABE H, TOTOKI Y, TAYLOR T, WEISSENBACH J, HEILIG R, SAURIN W, ARTIGUENAVE F, BROTTIER P, BRULS T, PELLETIER E, ROBERT C, WINCKER P, ROSENTHAL A, PLATZER M, NYAKATURA G, TAUDIEN S, RUMP A, SMITH D R, RUBENFIELD M, WEINSTOCK K, LEE H M, DUBOIS J, YANG H M, YU J, WANG J, HUANG G Y, GU J, HOOD L, ROWEN L, MADAN A, QIN S, DAVIS R W, FEDERSPIEL N A, ABOLA A P, PROCTOR M J, ROE B A, CHEN F, PAN H Q, RAMSER J, LEHRACH H, REINHARDT R, MCCOMBIE W R, DE LA BASTIDE M, DEDHIA N, BLÖCKER H, HORNISCHER K, NORDSIEK G, AGARWALA R, ARAVIND L, BAILEY J A, BATEMAN A, BATZOGLOU S, BIRNEY E, BORK P, BURGE C B, CERUTTI L, CHEN HSIU-CHUANCHURCH D, CLAMP M, COPLEY R R, EDDY S R, EICHLER E E, FUREY T S, GALAGAN J, GILBERT J G R, HARMON C, HAYASHIZAKI Y, HAUSSLER D, HERMJAKOB H, HOKAMP K, JANG W, JOHNSON L S, JONES T A, KASIF S, KASPRYZK A, KENNEDY S, KENT W J, KITTS P, KOONIN E V, KORF I, KULP D, LANCET D, LOWE T M, MCLYSAGHT A, MIKKELSEN T, MORAN J V, MULDER N, POLLARA V J, PONTING C P, SCHULER G, SCHULTZ J, SLATER G, SMIT A F A, STUPKA E, SZUSTAKOWKI J, THIERRY-MIEG D, THIERRYMIEG J, WAGNER L, WALLIS J, WHEELER R, WILLIAMS A, WOLF Y I, WOLFE K H, YANG S, YEH R, COLLINS F, GUYER M S, PETERSON J, FELSENFELD A, WETTERSTRAND K A, MYERS R M, SCHMUTZ J, DICKSON M, GRIMWOOD J, COX D R, OLSON M V, KAUL R, RAYMOND C, SHIMIZU N, KAWASAKI K, MINOSHIMA S, EVANS G A, ATHANASIOU M, SCHULTZ R, PATRINOS A, MORGAN M J. Initial sequencing and analysis of the human genome[J]. Nature, 2001, 409(6822): 860-921. DOI:10.1038/35057062 |
| [11] |
MARDIS E R. DNA sequencing technologies: 2006-2016[J]. Nature Protocols, 2017, 12(2): 213-218. DOI:10.1038/nprot.2016.182 |
| [12] |
SHENDURE J, JI H. Next-generation DNA sequencing[J]. Nature Biotechnology, 2008, 26(10): 1135-1145. DOI:10.1038/nbt1486 |
| [13] |
WENGER A M, PELUSO P, ROWELL W J, CHANG P, HALL R J, CONCEPCION G T, EBLER J, FUNGTAMMASAN A, KOLESNIKOV A, OLSON N D, TÖPFER A, ALONGE M, MAHMOUD M, QIAN Y F, CHIN C, PHILLIPPY A M, SCHATZ M C, MYERS G, DEPRISTO M A, RUAN J, MARSCHALL T, SEDLAZECK F J, ZOOK J M, LI H, KOREN S, CARROLL A, RANK D R, HUNKAPILLER M W. Accurate circular consensus long-read sequencing improves variant detection and assembly of a human genome[J]. Nature Biotechnology, 2019, 37(10): 1155-1162. DOI:10.1038/s41587-019-0217-9 |
| [14] |
FENG Y X, ZHANG Y C, YING C F, WANG D Q, DU C L. Nanoporebased fourth-generation DNA sequencing technology[J]. Genomics, Proteomics & Bioinformatics, 2015, 13(1): 14-16. DOI:10.1016/j.gpb.2015.01.009 |
| [15] |
ZHOU G R, LIAO B S, LI Q S, XU J, CHEN S L. Establishing a genomic database for the medicinal plants in the Brazilian Pharmacopoeia[J]. Chinese Medicine, 2021, 16(1): 71. DOI:10.1186/s13020-021-00484-5 |
| [16] |
LIAO B S, HU H Y, XIAO H M, ZHOU G R, SUN W, CHU Y, MENG X X, WEI J H, ZHANG H, XU J, CHEN S L. Global Pharmacopoeia Genome Database is an integrated and mineable genomic database for traditional medicines derived from eight international pharmacopoeias[J]. Science China Life Sciences, 2021. DOI:10.1007/s11427-021-1968-7 |
| [17] |
YU D L, LU J J, SHAO W S, MA X X, XIE T, ITO H, WANG T Z, XU M, WANG H Z, MENG Y J. MepmiRDB: a medicinal plant microRNA database[J]. Database, 2019. DOI:10.1093/database/baz070 |
| [18] |
KUMAR A, KUMAR R, SHARMA M, KUMAR U, GAJULA M N V P, SINGH K P. Uttarakhand Medicinal Plants Database(UMPDB): A platform for exploring genomic, chemical, and traditional knowledge[J]. Data, 2018, 3(1): 7. DOI:10.3390/data3010007 |
| [19] |
MOHANRAJ K, KARTHIKEYAN B S, VIVEK-ANANTH R P, CHAND R P B, APARNA S R, MANGALAPANDI P, SAMAL A. IMPPAT: A curated database of Indian medicinal plants, phytochemistry and therapeutics[J]. Scientific Reports, 2018, 8(1): 4329. DOI:10.1038/s41598-018-22631-z |
| [20] |
PATHANIA S, RAMAKRISHNAN S M, BAGLER G. Phytochemica: a platform to explore phytochemicals of medicinal plants[J]. Database (Oxford), 2015. DOI:10.1093/database/bav075 |
| [21] |
PATHANIA S, RAMAKRISHNAN S M, RANDHAWA V, BAGLER G. SerpentinaDB: a database of plant-derived molecules of Rauvolfia serpentina[J]. BMC Complement Altern Med, 2015, 15: 262. DOI:10.1186/s12906-015-0683-7 |
| [22] |
SAMBHAJI B T, PRADNYA N G, MANISHA V K, KAILAS D S. FERN Ethnomedicinal Plant Database: Exploring fern ethnomedicinal plants knowledge for computational drug discovery[J]. Current Computer-Aided Drug Design, 2015, 11(3): 266-271. DOI:10.2174/1573409911666151103114738 |
| [23] |
MANGAL M, SAGAR P, SINGH H, RAGHAVA G P, AGARWAL S M. NPACT: Naturally occurring plant-based anti-cancer compoundactivity-target database[J]. Nucleic Acids Res, 2013, 41(D1): 1124-1129. DOI:10.1093/nar/gks1047 |
| [24] |
YE H, YE L, KANG H, ZHANG D, TAO L, TANG K, LIU X, ZHU R, LIU Q, CHEN Y Z, LI Y, CAO Z. HIT: linking herbal active ingredients to targets[J]. Nucleic Acids Res, 2011, 39(S1): 1055-1059. DOI:10.1093/nar/gkq1165 |
| [25] |
E H R M A N T M, BA R L OW D J, H Y L A N D S P J. Hyl a nd s, Phytochemical databases of Chinese herbal constituents and bioactive plant compounds with known target specificities[J]. J Chem Inf Model, 2007, 47(2): 254-63. DOI:10.1021/ci600288m |
| [26] |
PILON A C, VALLI M, DAMETTO A C, PINTO M E F, FREIRE R T, CASTRO-GAMBOA I, ANDRICOPULO A D, BOLZANI V S. NuBBE(DB): an updated database to uncover chemical and biological information from Brazilian biodiversity[J]. Sci Rep, 2017, 7(1): 7215. DOI:10.1038/s41598-017-07451-x |
| [27] |
NTIE-KANG F, TELUKUNTA K K, DÖRING K, SIMOBEN C V, A. MOUMBOCK AF, MALANGE Y I, NJUME L E, YONG J N, SIPPL W, GÜNTHER S. NANPDB. A resource for natural products from Northern African sources[J]. J Nat Prod, 2017, 80(7): 2067-2076. DOI:10.1021/acs.jnatprod.7b00283 |
| [28] |
ISOBE S, KLIMENKO I, IVASHUTA S, GAU M, KOZLOV N N. First RFLP linkage map of red clover(Trifolium pratense L.)based on cDNA probes and its transferability to other red clover germplasm[J]. Theor Appl Genet, 2003, 108(1): 105-112. DOI:10.1007/s00122-003-1412-z |
| [29] |
L A N T E R I S, AC Q UA D R O A, C O M I N O C, M AU R O R, MAUROMICALE G, PORTIS E. A first linkage map of globe artichoke (Cynara cardunculus var. scolymus L.)based on AFLP, S-SAP, M-AFLP and microsatellite markers[J]. Theoretical and Applied Genetics, 2006, 112(8): 1532-1542. DOI:10.1007/s00122-006-0256-8 |
| [30] |
BRATTELER M, LEXER C, WIDMER A. A genetic linkage map of Silene vulgaris based on AFLP markers[J]. Genome, 2006, 49(4): 320-327. DOI:10.1139/g05-114 |
| [31] |
CARNEIRO M S, CAMARGO L E, COELHO A S, VENCOVSKY R, RUI P L, STENZEL N M, VIEIRA M L. RAPD-based genetic linkage maps of yellow passion fruit(Passiflora edulis Sims. f. flavicarpa Deg.)[J]. Genome, 2002, 45(4): 670-8. DOI:10.1139/g02-035 |
| [32] |
RIFKIN J L, BEAUDRY F E G, HUMPHRIES Z, CHOUDHURY B I, BARRETT S C H, WRIGHT S I. Widespread recombination suppression facilitates plant sex chromosome evolution[J]. Molecular Biology and Evolution, 2021, 38(3): 1018-1030. DOI:10.1093/molbev/msaa271 |
| [33] |
ZHANG G B, GE C X, XU P P, WANG S K, CHENG S N, HAN Y B, WANG Y C, ZHUANG Y B, HOU X W, YU T, XU X T, DENG S H, LI Q Q, YANG Y Q, YIN X R, WANG W D, LIU W X, ZHENG C X, SUN X Z, WANG Z L, MING R, DONG S T, MA J X, ZHANG X S, CHEN C X. The reference genome of Miscanthus floridulus illuminates the evolution of Saccharinae[J]. Nature Plants, 2021, 7(5): 608-618. DOI:10.1038/s41477-021-00908-y |
| [34] |
VINING K J, JOHNSON S R, AHKAMI A, LANGE I, PARRISH A N, TRAPP S C, CROTEAU R B, STRAUB S C K, PANDELOVA I, LANGE B M. Draft genome sequence of Mentha longifolia and development of resources for mint cultivar improvement[J]. Molecular Plant, 2017, 10(2): 323-339. DOI:10.1016/j.molp.2016.10.018 |
| [35] |
LI J, LV M Q, DU L, YUNGA A, HAO S J, ZHANG Y L, ZHANG X W, GUO L D, GAO X Y, DENG L, ZHANG X, SHI C C, GUO F, LIU R, FANG B, SU Q X, HU X, SU X S, LIN L, LIU Q, WANG Y H, QIN Y T, ZHANG W W, LI S Y, LIU C N, LI H. An enormous < em > Paris polyphylla < /em > genome sheds light on genome size evolution and polyphyllin biogenesis[J]. BioRxiv, 2020. DOI:10.1101/2020.06.01.126920 |
| [36] |
LIU H L, WANG X B, WANG G B, CUI P, WU S G, AI C, HU N, LI A, HE B, SHAO X J, WU Z C, FENG H, CHANG Y X, MU D S, HOU J, DAI X G, YIN T M, RUAN J, CAO F L. The nearly complete genome of Ginkgo biloba illuminates gymnosperm evolution[J]. Nature Plants, 2021, 7(6): 748-756. DOI:10.1038/s41477-021-00933-x |
| [37] |
MA L G, DONG C M, WANG X L, ZHENG X K, NIU Y, CHEN S L, FENG W S. De novo genome assembly of the potent medicinal plant Rehmannia glutinosa using nanopore technology[J]. Computational and Structural Biotechnology Journal, 2021, 19: 3954-3963. DOI:10.1016/j.csbj.2021.07.006 |
| [38] |
HE Y, PENG F, DENG C, XIONG L, HUANG Z Y, ZHANG R Q, LIU M J, PENG C. Building an octaploid genome and transcriptome of the medicinal plant pogostemon cablin from Lamiales[J]. Scientific Data, 2018, 5(1): 180274. DOI:10.1038/sdata.2018.274 |
| [39] |
WANG J H, XU S Q, MEI Y, CAI S K, GU Y, SUN M Y, LIANG Z, XIAO Y, ZHANG M Q, YANG S H. A high-quality genome assembly of Morinda officinalis, a famous native southern herb in the Lingnan region of southern China[J]. Horticulture Research, 2021, 8(1): 135. DOI:10.1038/s41438-021-00551-w |
| [40] |
YANG Y D, HUANG L Y, XU C Y, QI L, WU Z Y, LI J, CHEN H X, WU Y, FU T, ZHU H, SAAND M A, LI J, LIU L Y, FAN H K, ZHOU H Q, QIN W Q. Chromosome-scale genome assembly of areca palm(Areca catechu)[J]. Molecular Ecology Resources, 2021, 21(7): 2504-2519. DOI:10.1111/1755-0998.13446 |
| [41] |
ZHOU W, WANG Y, LI B, PETIJOVÁ L, HU S Y, ZHANG Q, NIU J F, WANG D H, WANG S Q, DONG Y, ČELLÁROVÁ E, WANG Z Z. Whole-genome sequence data of Hypericum perforatum and functional characterization of melatonin biosynthesis by N-acetylserotonin O-methyltransferase[J]. Journal of Pineal Research, 2021, 70(2): e12709. DOI:10.1111/jpi.12709 |
| [42] |
LI L F, CUSHMAN S A, HE Y X, LI Y. Genome sequencing and population genomics modeling provide insights into the local adaptation of weeping forsythia[J]. Horticulture Research, 2020, 7(1): 130. DOI:10.1038/s41438-020-00352-7 |
| [43] |
DUMSCHOTT K, SCHMIDT M H, CHAWLA H S, SNOWDON R, USADEL B. Oxford Nanopore sequencing: new opportunities for plant genomics[J]. Journal of Experimental Botany, 2020, 71(18): 5313-5322. DOI:10.1093/jxb/eraa263 |
| [44] |
DROUIN G, DAOUD H, XIA J N. Relative rates of synonymous substitutions in the mitochondrial, chloroplast and nuclear genomes of seed plants[J]. Molecular Phylogenetics and Evolution, 2008, 49(3): 827-831. DOI:10.1016/j.ympev.2008.09.009 |
| [45] |
SMITH D R. Mutation rates in plastid genomes: they are lower than you might think[J]. Genome Biology and Evolution, 2015, 7(5): 1227-34. DOI:10.1093/gbe/evv069 |
| [46] |
SCHINDEL D E, MILLER S E. DNA barcoding a useful tool for taxonomists[J]. Nature, 2005, 435(7038): 17. DOI:10.1038/435017b |
| [47] |
XU S Q, GUO S, FAN D D, WANG J H. The complete chloroplast genome sequence of Ficus formosana Maxim(Moraceae)from Guangzhou, China[J]. Mitochondrial DNA Part B, 2021, 6(7): 1895-1896. DOI:10.1080/23802359.2021.1934170 |
| [48] |
WANG J H, HUANG W Z, CAI S, ZHAO J L. The complete chloroplast genome sequence of Dryobalanops aromatica[J]. Mitochondrial DNA Part B, 2021, 6(6): 1687-1688. DOI:10.1080/23802359.2021.1914223 |
| [49] |
XU S Q, LI Y F, ZHAO Y, WANG J H. The complete chloroplast genome sequence of the medicinal plant Mesona chinensis(Labiatae)[J]. Mitochondrial DNA Part B, 2020, 5(3): 2721-2722. DOI:10.1080/23802359.2020.1788433 |
| [50] |
ZHOU F, LAN K K, LI X R, MEI Y, CAI S K, WANG J H. The complete chloroplast genome sequence of Vernonia amygdalina Delile[J]. Mitochondrial DNA Part B, 2021, 6(3): 1134-1135. DOI:10.1080/23802359.2021.1902411 |
| [51] |
LI J Y, ZHOU F, MEI Y, GU Y, XU S Q, CAI S, SUN M Y, WANG J H. The complete chloroplast genome sequence of Acanthopanax trifoliatus (Linn.)Merr[J]. Mitochondrial DNA Part B, 2020, 5(3): 3408-3409. DOI:10.1080/23802359.2020.1823900 |
| [52] |
MEI Y, CAI S, XU S Q, GU Y, ZHOU F, WANG J H. The complete chloroplast genome sequence of medicinal plant Alpinia chinensis (Retz.)Rosc[J]. Mitochondrial DNA Part B, 2020, 5(3): 3328-3329. DOI:10.1080/23802359.2020.1817805 |
| [53] |
CAI S K, ZHOU F, GU Y, HUANG Z N, MEI Y, YANG N B, WANG J H. The complete chloroplast genome sequence of Sauropus spatulifolius Beille[J]. Mitochondrial DNA Part B, 2020, 5(2): 1703-1704. DOI:10.1080/23802359.2020.1748535 |
| [54] |
QIAN J, SONG J Y, GAO H H, ZHU Y J, XU J, PANG X H, YAO H, SUN C, LI X E, LI C Y, LIU J Y, XU H B, CHEN S L. The complete chloroplast genome sequence of the medicinal plant Salvia miltiorrhiza[J]. PLOS ONE, 2013, 8(2): e57607. DOI:10.1371/journal.pone.0057607 |
| [55] |
GAO C W, WU C H, ZHANG Q, ZHAO X, WU M X, CHEN R R, ZHAO Y L, LI Z Q. Characterization of chloroplast genomes from two Salvia medicinal plants and gene transfer among their mitochondrial and chloroplast genomes[J]. Frontiers in Genetics, 2020, 11: 574962. DOI:10.3389/fgene.2020.574962 |
| [56] |
LIANG C L, WANG L, LEI J, DUAN B Z, MA W S, XIAO S M, QI H J, WANG Z, LIU Y Q, SHEN X F, GUO S, HU H Y, XU J, CHEN S L. a comparative analysis of the chloroplast genomes of four Salvia medicinal plants[J]. Engineering, 2019, 5(5): 907-915. DOI:10.1016/j.eng.2019.01.017 |
| [57] |
ALZAHRANI D A, ALBOKHARI E J, YARADUA S S, ABBA A. Comparative analysis of chloroplast genomes of four medicinal Capparaceae Species: genome structures, phylogenetic relationships and adaptive evolution[J]. Plants, 2021, 10(6): 1229. DOI:10.3390/plants10061229 |
| [58] |
WEI F, TANG D F, WEI K H, QIN F, LI L, LIN Y, ZHU Y X, KHAN A, KASHIF M H, MIAO J. The complete chloroplast genome sequence of the medicinal plant Sophora tonkinensis[J]. Scientific Reports, 2020, 10(1): 12473. DOI:10.1038/s41598-020-69549-z |
| [59] |
SHEN X F, LIAO B S, LIU Z X, BAI R, XIAO S M, LI X W, ZHANG B L, XU J, CHEN S L. Complete chloroplast genome sequence and phylogenetic analysis of the medicinal plant Artemisia annua[J]. Molecules, 2017, 22(8): 1330. DOI:10.3390/molecules22081330 |
| [60] |
WU L W, NIE L P, WANG Q, XU Z C, WANG Y, HE C N, SONG J Y, YAO H. Comparative and phylogenetic analyses of the chloroplast genomes of species of Paeoniaceae[J]. Scientific Reports, 2021, 11(1): 14643. DOI:10.1038/s41598-021-94137-0 |
| [61] |
LI X Q, ZUO Y J, ZHU X X, LIAO S, MA J S. Complete chloroplast genomes and comparative analysis of sequences evolution among seven Aristolochia(Aristolochiaceae)medicinal species[J]. International Journal of Molecular Sciences, 2019, 20(5): 1045. DOI:10.3390/ijms20051045 |
| [62] |
WU Z Q, LIAO X Z, ZHANG X N, TEMBROCK L R, BROZ A. Genomic architectural variation of plant mitochondria—A review of multichromosomal structuring[J]. Journal of Systematics and Evolution, 2020. DOI:10.1111/jse.12655 |
| [63] |
JANG W, LEE H O, KIM J, LEE J, HONG C, BANG K, CHUNG J, JO I. Complete mitochondrial genome and a set of 10 novel kompetitive allelespecific PCR markers in Ginseng(Panax ginseng C. A. Mey.)[J]. Agronomy, 2020, 10(12): 1868. DOI:10.3390/agronomy10121868 |
| [64] |
LI S N, YANG Y Y, XU L, XING Y P, ZHAO R, AO W, ZHANG T T, ZHANG D C, SONG Y Y, BAO G H, KANG T G. The complete mitochondrial genome of Aconitum kusnezoffii Rchb.(Ranales, Ranunculaceae)[J]. Mitochondrial DNA Part B, 2021, 6(3): 779-781. DOI:10.1080/23802359.2021.1882894 |
| [65] |
YANG Y Y, LI S N, XU L, XING Y P, ZHAO R, BAO G H, ZHANG T T, ZHANG D C, SONG Y Y, AO W, KANG T G. The complete mitochondrial genome of Glycyrrhiza uralensis Fisch.(Fabales, Leguminosae)[J]. Mitochondrial DNA Part B, 2021, 6(2): 475-477. DOI:10.1080/23802359.2021.1872432 |
| [66] |
KANG S, PANDEY R P, LEE C, SIM J, JEONG J, CHOI B, JUNG M, GINZBURG D, ZHAO K M, WON S Y, OH T, YU Y, KIM N, LEE O R, LEE T, BASHYAL P, KIM T, LEE W, HAWKINS C, KIM C, KIM J S, AHN B O, RHEE S Y, SOHNG J K. Genome-enabled discovery of anthraquinone biosynthesis in Senna tora[J]. Nature Communications, 2020, 11(1): 5875. DOI:10.1038/s41467-020-19681-1 |
| [67] |
MAO W T, YAO G X, WANG S D, ZHOU L, CHEN G S, DONG N G, HU G L. Chromosome-level genomes of seeded and seedless date plum based on third-generation DNA sequencing and Hi-C analysis[J]. Forestry Research, 2021, 1: 9. DOI:10.48130/FR-2021-0009 |
| [68] |
XU Z C, GAO R R, PU X D, XU R, WANG J Y, ZHENG S H, ZENG Y, CHEN J, HE C N, SONG J Y. Comparative genome analysis of Scutellaria baicalensis and Scutellaria barbata reveals the evolution of active flavonoid biosynthesis[J]. Genomics, Proteomics & Bioinformatics, 2020, 18(3): 230-240. DOI:10.1016/j.gpb.2020.06.002 |
| [69] |
FU A Z, WANG Q, MU J L, MA L L, WEN C L, ZHAO X Y, GAO L P, LI J, SHI K, WANG Y X, ZHANG X C, ZHANG X W, GRIERSON D, ZUO J H. Combined genomic, transcriptomic, and metabolomic analyses provide insights into chayote(Sechium edule)evolution and fruit development[J]. Horticulture Research, 2021, 8(1): 35. DOI:10.1038/s41438-021-00487-1 |
| [70] |
BRAICH S, BAILLIE R C, SPANGENBERG G C, COGAN N O I. A new and improved genome sequence of & lt; em & gt; Cannabis sativa < /em & gt[J]. BioRxiv, 2020, 422592. DOI:10.1101/2020.12.13.422592 |
| [71] |
MCKERNAN K J, HELBERT Y, KANE L T, EBLING H, ZHANG L, LIU B, EATON Z, MCLAUGHLIN S, KINGAN S, BAYBAYAN P, CONCEPCION G, JORDAN M, RIVA A, BARBAZUK W, HARKINS T. Sequence and annotation of 42 cannabis genomes reveals extensive copy number variation in cannabinoid synthesis and pathogen resistance genes[J]. BioRxiv, 2020, 894428. DOI:10.1101/2020.01.03.894428 |
| [72] |
LOWE R, SHIRLEY N, BLEACKLEY M, DOLAN S, SHAFEE T. Transcriptomics technologies[J]. PLOS Computational Biology, 2017, 13(5): e1005457. DOI:10.1371/journal.pcbi.1005457 |
| [73] |
GUO J, HUANG Z, SUN J L, CUI X M, LIU Y. Research progress and future development trends in medicinal plant transcriptomics[J]. Frontiers in Plant Science, 2021, 12: 1520. DOI:10.3389/fpls.2021.691838 |
| [74] |
WANG Y, SHAHID M Q, GHOURI F, BALOCH F S. De Novo assembly and annotation of the juvenile tuber transcriptome of a Gastrodia elata hybrid by RNA sequencing: detection of SSR Markers[J]. Biochemical Genetics, 2020, 58(6): 914-934. DOI:10.1007/s10528-020-09983-w |
| [75] |
LIAO W, MEI Z, MIAO L, LIU P, GAO R. Comparative transcriptome analysis of root, stem, and leaf tissues of Entada phaseoloides reveals potential genes involved in triterpenoid saponin biosynthesis[J]. BMC Genomics, 2020, 21(1): 639. DOI:10.1186/s12864-020-07056-1 |
| [76] |
李晓艳, 周敬雯, 严铸云, 陈新. 基于转录组测序揭示适度干旱胁迫对丹参基因表达的调控[J]. 中草药, 2020, 51(6): 1600-1608. DOI:10.7501/j.issn.0253-2670.2020.06.029 LI X Y, ZHOU J W, YAN Z Y, CHEN X. Sequencing and analysis of transcriptome to reveal regulation of gene expression in Salvia miltiorrhiza under moderate drought stress[J]. Chinese Traditional and Herbal Drugs, 2020, 51(6): 1600-1608. DOI:10.7501/j.issn.0253-2670.2020.06.029 |
| [77] |
VAN BAKEL H, STOUT J M, COTE A G, TALLON C M, SHARPE A G, HUGHES T R, PAGE J E. The draft genome and transcriptome of Cannabis sativa[J]. Genome Biology, 2011, 12(10): 102. DOI:10.1186/gb-2011-12-10-r102 |
| [78] |
CHEN Y M, WANG Y, YANG J, ZHOU W B, DAI S J. Exploring the diversity of plant proteome[J]. Journal of Integrative Plant Biology, 2021, 63(7): 1197-1210. DOI:10.1111/jipb.13087 |
| [79] |
MUSTAFA G, KOMATSU S. Plant proteomic research for improvement of food crops under stresses: a review[J]. Molecular Omics, 2021, 17: 860-880. DOI:10.1039/D1MO00151E |
| [80] |
MOYER T B, ALLEN J L, SHAW L N, HICKS L M. Multiple classes of antimicrobial peptides in Amaranthus tricolor revealed by prediction, proteomics, and mass spectrometric characterization[J]. Journal of Natural Products, 2021, 84(2): 444-452. DOI:10.1021/acs.jnatprod.0c01203 |
| [81] |
VAN NGUYEN T, KIM S, MIN C, GUPTA R, LEE G, JANG J, RATHI D, SHIN H, JUNG J, JO I, HONG W, JUNG K, KIM S, KIM Y, KIM S. Optimization of protein isolation and label-free quantitative proteomic analysis in four different tissues of Korean Ginseng[J]. Plants, 2021, 10(7): 1409. DOI:10.3390/plants10071409 |
| [82] |
KUMAR R, JOSHI R, KUMARI M, THAKUR R, KUMAR D, KUMAR S. Elevated CO 2 and temperature influence key proteins and metabolites associated with photosynthesis, antioxidant and carbon metabolism in Picrorhiza kurroa[J]. Journal of Proteomics, 2020, 219: 103755. DOI:10.1016/j.jprot.2020.103755 |
| [83] |
CHEN M H, YAN T X, JI L Y, DONG Y, SIDOLI S, YUAN Z F, CAI C L, CHEN J W, TANG Y L, SHEN Q, PAN Q F, FU X Q, LIAO L J, GARCIA B A, YAN W, TANG K. Comprehensive map of the Artemisia annua proteome and quantification of differential protein expression in chemotypes producing high versus low content of Artemisinin[J]. Proteomics, 2020, 20(10): 1900310. DOI:10.1002/pmic.201900310 |
| [84] |
JAMSHIDI GOHARRIZI K, FATEHI F, NAZARI M, SALEHI F, MALEKI M. Assessment of changes in the content of sulforaphane and expression levels of CYP79F1 and myrosinase genes and proteomic profile of Lepidium draba plant under water-deficit stress induced by polyethylene glycol[J]. Acta Physiologiae Plantarum, 2020, 42(6): 101. DOI:10.1007/s11738-020-03085-1 |
| [85] |
JIANG C H, BI Y, MO J, ZHANG R Y, QU M M, FENG S C, ESSEMINE J. Proteome and transcriptome reveal the involvement of heat shock proteins and antioxidant system in thermotolerance of Clematis florida[J]. Scientific Reports, 2020, 10(1): 8883. DOI:10.1038/s41598-020-65699-2 |
| [86] |
XU W, ZHANG L B, CUNNINGHAM A B, LI S, ZHUANG H F, WANG Y H, LIU A Z. Blue genome: chromosome-scale genome reveals the evolutionary and molecular basis of indigo biosynthesis in Strobilanthes cusia[J]. The Plant Journal, 2020, 104(4): 864-879. DOI:10.1111/tpj.14992 |
| [87] |
LI H L, WU L, DONG Z M, JIANG Y S, JIANG S J, XING H T, LI Q, LIU G C, TIAN S M, WU Z Y, BIN W, LI Z X, ZHAO P, ZHANG Y, TANG J M, XU J B, HUANG K, LIU X, ZHANG W L, LIAO Q H, REN Y, HUANG X Z, LI Q Z, LI C Y, WANG Y, XAVIER-RAVI B, LI H H, LIU Y, WAN T, LIU Q H, ZOU Y, JIAN J B, XIA Q Y, LIU Y Q. Haplotype-resolved genome of diploid ginger(Zingiber offi cinale)and its unique gingerol biosynthetic pathway[J]. Horticulture Research, 2021, 8(1): 189. DOI:10.1038/s41438-021-00627-7 |
| [88] |
GONULALAN E M, NEMUTLU E, BAYAZEID O, KOÇAK E, YALÇIN F N, DEMIREZER L O. Metabolomics and proteomics profiles of some medicinal plants and correlation with BDNF activity[J]. Phytomedicine, 2020, 74: 152920. DOI:10.1016/j.phymed.2019.152920 |
| [89] |
RAI A, HIRAKAWA H, NAKABAYASHI R, KIKUCHI S, HAYASHI K, RAI M, TSUGAWA H, NAKAYA T, MORI T, NAGASAKI H, FUKUSHI R, KUSUYA Y, TAKAHASHI H, UCHIYAMA H, TOYODA A, HIKOSAKA S, GOTO E, SAITO K, YAMAZAKI M. Chromosomelevel genome assembly of Ophiorrhiza pumila reveals the evolution of camptothecin biosynthesis[J]. Nature Communications, 2021, 12(1): 405. DOI:10.1038/s41467-020-20508-2 |
| [90] |
WANG W, XU J F, FANG H Y, LI Z J, LI M H. Advances and challenges in medicinal plant breeding[J]. Plant Science, 2020, 298: 110573. DOI:10.1016/j.plantsci.2020.110573 |
(责任编辑 杨贤智)
 2021, Vol. 48
2021, Vol. 48