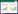文章信息
基金项目
- 国家重点研发计划项目(2018YFD0200706);广东省基础与应用基础研究基金(2019A1515010305);广东省重点领域研发计划项目(2018B020202008);广东省现代农业产业技术体系创新团队项目(2020 KJ106)
作者简介
- 李武(1981—),男,博士,副研究员,研究方向为作物遗传育种及栽培技术,E-mail: liwu@gdaas.cn; 李光玉(1995—),女,研究实习员,研究方向为作物遗传育种,E-mail: liguangyu@gdaas.cn.
通讯作者
- 刘建华(1965—),男,研究员,研究方向为作物遗传育种,E-mail: liujianhua@gdaas.cn.
文章历史
- 收稿日期:2020-12-31
【研究意义】甜玉米是一类果蔬兼用的特用型玉米,其形成是由于淀粉合成代谢途径发生基因突变,导致淀粉合成途径受阻、蔗糖等糖类物质积累。由于甜玉米是普通玉米籽粒淀粉合成基因突变产生,其种子活力普遍较低,这成为制约我国甜玉米生产的主要因素之一。低温发芽率是甜玉米种子活力鉴定的重要指标,但目前有关玉米应对低温胁迫的研究尚不充分[1]。低温胁迫主要分为冷害胁迫(<15 ℃)和冻害胁迫(<0 ℃)。作为C4植物代表的玉米,当温度低于15 ℃或者更低时,容易遭受冷害胁迫而影响发芽率[2]。伴随全球气候变化,极端天气频发,广东近5年来的春季最低气温维持在4~5 ℃左右,平均气温约13 ℃,玉米直播常遭遇低温胁迫,不耐低温发芽的品种往往容易受灾。因此,低温发芽特性好的甜玉米品种对推广直播栽培、实现节本增效具有重要价值[3]。挖掘鉴定低温发芽相关调控基因,将为阐明甜玉米低温发芽的生物学基础、加快育种改良提供重要的理论支撑。
【前人研究进展】玉米耐冷性遗传研究与功能鉴定主要集中在发芽期和苗期。早期研究中,Mc Connell等[4]研究6个耐冷性差异显著的自交系及其F1、F2和回交群体,发现苗期活力受加性和显性效应控制,而发芽能力受加性、显性效应和上位性基因调控。Hodges等[5]研究由4个耐冷能力不同的自交系完全双列杂交得到的群体,结果表明不同群体在发芽期和苗期耐冷性的遗传因素可能不同。Revilla等[6]利用同样的方法研究5个自交系及其后代F1的耐冷性,发现高发芽率和苗期快速生长两个耐冷性状的遗传调控符合加-显性模型。近年来,较多学者利用不同的作图群体对玉米耐低温发芽和苗期耐冷的数量性状遗传位点(Quantitative trait locus,QTL)进行鉴定。例如,利用不同作图群体(F2:4家系、重组自交系群体等)研究玉米耐冷相关QTL,多集中于分析发芽期发芽和苗期光合作用性状,如发芽指数、光合作用效率、根干重、茎含氮量等指标[7]。有学者利用ETH-DH7×ETH-DL3构建的F2:3群体,于第6号染色体定位到低温下控制苗期光合作用的主效QTL,且该位点能在多个环境下被重复检测[8]。随后Nguyen等[9]利用相同的F2:3群体研究低温下苗期光合系统耐冷性,但检测到的主效QTL位于第3号染色体上。Rodriguez[10]利用210份F2:3家系(EP42×A661),以茎干重、存活率等为评价指标,在低温环境下检测到4个QTL(其中1个为存活率QTL)。Hu等[11]利用重组自交系群体,以相对出芽率、初生根长为指标,通过不同温度时间处理(12 ℃ 16 h、18 ℃ 8 h、28 ℃ 24 h)鉴定出15个QTL,主要分布在第4、5、6、7、9号染色体上。李旭辉等[12]利用低温萌发能力高的玉米自交系220和P9-10,以及低温出苗能力弱的自交系Y1518和PH4CV,配制了3个F2:3群体,通过6K玉米SNP芯片做基因型鉴定,确定了67个与低温发芽相关的QTL,其中17个贡献率大于10%。上述研究报道,均以普通玉米种子为分析对象,鲜有甜玉米种子低温萌发相关的分子遗传学研究。
【本研究切入点】根据前期工作对甜玉米种子低温下的发芽率、发芽势、发芽指数、活力指数等指标的综合评价,我们获得了2份低温萌发能力差异显著的甜玉米自交系Scil-H7th和Scil-C4th。【拟解决的关键问题】利用低温萌发能力高的Scil-H7th为供体亲本(DP)、低温萌发能力低的Scil-C4th为轮回亲本(RP),杂交构建近等基因系(Near Iso-genic Lines, NIL),并分析不同品系的低温萌发能力。通过转录组测序分析低温萌发能力不同的材料,以鉴定甜玉米耐低温萌发的相关基因,为后续甜玉米耐低温萌发特异种质资源的创制奠定基础。
1 材料与方法 1.1 试验材料甜玉米自交系Scil-C4th(轮回亲本,RP)和Scil-H7th(供体亲本,DP)于2014年早季在广东省农业科学院白云试验基地种植并杂交获得F1;以Scil-C4th为轮回亲本连续回交4次,经过4代自交得到稳定自交系,并通过种子标准发芽试验进行表型选择,最终获得3个RP背景的NIL品系,分别命名为L6、L18及L41。筛选完整饱满的Scil-C4th、Scil-H7th及NIL品系甜玉米种子作为本试验材料。
1.2 试验方法1.2.1 种子发芽处理 将Scil-C4th、Scil-H7th及NIL不同品系甜玉米种子浸泡12 h后进行种子发芽试验,分别在15 ℃人工气候箱中培养,每个发芽处理3次重复,每个重复100粒种子。样品在12 h光照/12 h黑暗环境中生长,光照强度为250 μmol/m2·s,持续7 d[13]。在吸胀后1 d(时期Ⅰ)和吸胀后3 d(时期Ⅱ),从样本中随机收集5个种子胚,液氮冻存,用于提取RNA进行高通量测序。发芽试验期间,每天计数发芽种子,并在吸胀后7 d测量根长、茎高以及计算发芽率[14],在80 ℃干燥24 h后测定幼苗干重。计算发芽指数(GI)和种子活力指数(VI):
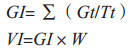
|
式中,Gt 为第t 天发芽种子的数量,Tt 为对应于Gt 的时间(d),W 为幼苗干重。
1.2.2 RNA提取、文库制备及测序 将种子胚用液氮匀浆,用植物核糖核酸提取试剂盒(中国天根生物技术有限公司,北京)提取总RNA。通过在1% 琼脂糖凝胶上电泳分析RNA质量,并用NanoDrop 2000(赛默菲舍科学公司,美国)鉴定RNA的纯度和浓度[15]。随后,每个样品取1.5 μg总RNA,按照HiSeq 2500测序仪(Illumina公司, 美国)说明书进行cDNA文库构建、质量控制和高通量测序。
1.2.3 数据拼装、功能注释与分类 对测序产生的原始数据进行过滤,删除包含poly-N和低质量的reads,以获得干净的reads。然后使用bowtie2 [16]或Tophat [17]将干净的reads比对到参考基因组。每个基因的FPKM值使用Cufflinks计算[16],每个基因的reads数通过HTseq-coun计算[18]。P <0.05被设置为显著差异表达的阈值。对筛选出来的差异表达基因(Differentially expressed genes, DEGs)进行基于R软件包的GO富集和KEGG通路富集分析。
1.2.4 qRT–PCR验证候选基因表达量 采用qRT–PCR技术从转录组DEGs中选择7个候选基因表达量进行验证[15]。基因序列信息从Ensembl Plants(http://plants.ensembl.org)获得,引物采用Primer3设计(https://bioinfo.ut.ee/primer3-0.4.0/)。使用cDNA第一条链反转录超级试剂盒反转录获得样本的第一链cDNA,并根据AceQ qPCR混合试剂盒操作说明对反转录获得的cDNA进行qRTPCR验证。本试验采用Step-One-Plus系统(Applied Biosystems公司,美国)进行测定,每个基因采用3个生物学重复和3个技术重复测定表达水平[15]。Actin 基因被用作获得Ct值标准化的内源性参照基因,通过比较Ct(ΔΔCt)方法计算基因的相对表达值。
2 结果与分析 2.1 甜玉米品系DP、RP及3个NIL的发芽性能为明确不同品系的低温萌发能力,测定自交系Scil-H7th、Scil-C4th及3个NIL的发芽相关指标。由图 1可知,DP(Scil-H7th)的种子发芽和成苗显著快于RP(Scil-C4th);而衍生自DP和RP的NIL中,L18表现出与DP相似的成苗特性(图 1A)。相比随机选择的另外两个NIL(L41和L6),L18表现出种子发芽快和种子活力高的特点;相比亲本,L18在发芽7 d后发芽率达到64.54%,显著高于RP的23.58%,其发芽势为RP的1.20倍,接近DP(图 1B)。由以上指标分析可知,L18的发芽指数和种子活力指数均优于RP。
|
| 图 1 甜玉米品系DP、RP及3个NIL在低温下的萌发特性 Fig. 1 Germination characteristics of sweet corn lines DP, RP and three NILs at low temperature A:DP、RP 及 3 个 NIL 萌发后整株对比;B:DP、RP 及 3 个 NIL 在低温下的发芽相关指标 A: Seedlings of DP, RP and three NILs; B: Germination related indexes of DP, RP and three NILs |
2.2 甜玉米品系DP、RP和L18的RNA测序文库构建、转录组测序及聚类分析
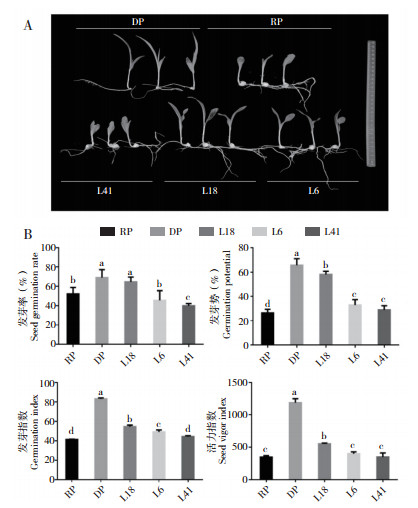
为了筛选与甜玉米低温萌发的相关基因,利用高通量测序对NIL L18及其亲本进行转录组分析。RP、DP和L18种子经低温催芽,在吸胀后1 d(时期Ⅰ,以-1表示)和吸胀后3 d(时期Ⅱ,以-2表示)取样,得到RP-1、DP-1、L18-1和RP-2、DP-2、L18-2样品,提取RNA进行转录组测序,每个时期3次重复,共测序获得18个玉米样品平均8.54 Gb的原始碱基,经质量控制过滤后的干净碱基平均为8.14 Gb,Q30值介于91.71%~92.44% 之间。
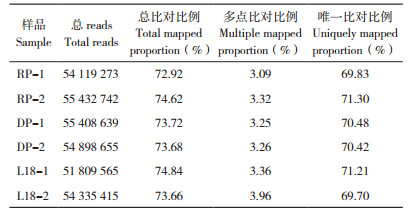
测序原始数据过滤后,总共51 809 565~ 55 432 742个reads用于比对分析。结果(表 1)显示,能够比对到参考基因组的reads比例在72.92%~74.84% 之间。对于RP、DP和L18,具有唯一匹配的reads比例分别为70.57%、70.45% 和70.46%。利用FPKM值(每百万份映射读数转录本的每千碱基片段)进行定量分析,基因表达量分布如图 2A所示,结果表明各样品的基因表达水平分布范围较为一致;基于相似性和主成分分析对每个样本进行区分和聚类,结果(图 2B和图 2C )表明重复样品间相似度高,重复性好。
|

|
| 图 2 RNA测序分析得到甜玉米品系DP、RP和L18的基因表达概况和聚类分析结果 Fig. 2 Overview of gene expression of sweet corn lines of DP, RP and L18 and clustering analysis A:样品 FPKM 值;B:样品相似性聚类分析;C:样品 PCA 主成分分析 A: FPKM values of samples; B: Clustering of samples based on sample-to-sample distance; C: Principal component analysis of samples |
2.3 甜玉米品系DP、RP和L18在不同萌发阶段的差异表达基因
为了筛选与低温萌发相关的基因,对DP、RP和L18在不同萌发时期以及同一萌发时期3个品系之间进行比较分析。自交系内不同萌发时期的分析结果(图 3A)表明,DP-2和DP-1共有1 629个DEGs,其中780个基因表达上调,849个基因表达下调;RP-2和RP-1共有2 834个DEGs,其中731个基因上调表达,2 103个基因表达下调;L18-2和L18-1共有1 823个DEGs,其中1 002个基因表达上调,821个基因表达下调;3个材料在两个萌发时期共有的DEGs为447个,其中RP和L18共有的DEGs有986个,高于L18与DP之间共有的DEGs(630个)。分析DP和RP、L18与RP之间相同萌发时期的DEGs,结果(图 3B)发现,DP与RP在两个萌发时期共有的DEGs达到了3 484个,而L18与RP共有的DEGs只有627个,说明L18遗传背景已与RP较接近;取两者共有基因的交集,获得410个共有DEGs,推定为低温萌发过程中不同材料间表达差异的基因集。
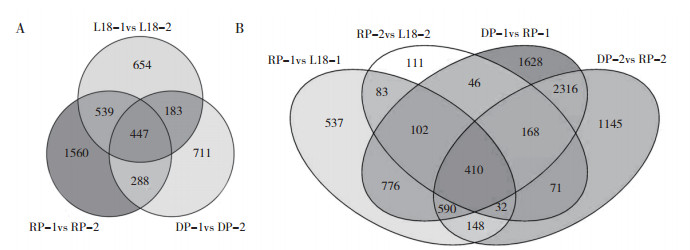
|
| 图 3 甜玉米品系DP、RP和L18在低温萌发过程中的DEGs韦维恩图 Fig. 3 Venn diagram of DEGs in sweet corn of DP, RP and L18 during low-temperature germination result base on RNA sequencing A:在不同萌发时期;B:在同一萌发时期 A: At different germination stages; B: At the same germination stage |
2.4 功能注释、通路富集和低温萌发相关基因筛选
为了进一步分析DEGs所在的通路,采用KEEG数据库对上述DEGs进行功能注释和通路富集分析。结果显示,共有89个和43个涉及的通路被注释在RP-1与L18-1组和RP-2与L18-2组;在3个品系中同时鉴定了6个关键通路,包括zma04712昼夜节律-植物、zma01040不饱和脂肪酸生物合成、zma00945芪类化合物、二芳基庚酸和姜酚生物合成、zma00941类黄酮生物合成、zma00940苯丙酸生物合成和zma00195光合作用。4个比较组(RP-1 vs DP-1、RP-2 vs DP-2、RP-1 vs L18-1、RP-2 vs L18-2)共有的410个DEGs已完成功能注释,其中286个基因被鉴定为功能性蛋白质,124个基因为未鉴定蛋白。Zm00001d005672、Zm00001d015497(线粒体乌头酸水合酶3)、Zm00001d033044(GDSL酯酶/脂肪酶)、Zm00001d033307(肌动蛋白轻链1)、Zm00001d033909、Zm00001d033948(Tubbylike F-box蛋白2)和Zm00001d037001(无静脉样蛋白)显示具有超过100倍的上调表达,Zm00001d029587(锌指蛋白AZF2)是L18中发现的下调超过100倍的蛋白。进一步分析发现,65个DEGs仅在DP和L18中表达,而不在RP中表达,其中32个基因在数据库中被注释。结合自交系在不同萌发时期的DEGs和自交系间相同萌发时期的DEGs,筛选出有交集的20个DEGs(表 2),推测这些基因与低温萌发密切相关。

|
根据注释信息进行分析,进一步筛选出最有可能参与低温萌发过程的7个上调基因(Zm00001d011006、Zm00001d018738、Zm00001d028815、Zm00001d020652、Zm00001d037383、Zm00001d029696、Zm00001d018195),利用qRT-PCR对DP在低温萌发过程2个时期的胚部、根系和嫩叶进行表达水平检测。结果表明,这些基因表达量变化趋势与转录组测序结果中的变化趋势趋于一致(图 4A)。其中,Zm00001d037383(UGT1 基因)随发芽进程表达显著上调,并且在胚部的表达显著高于在其他组织的表达(图 4B)。

|
| 图 4 甜玉米品系DP、RP和L18低温萌发相关基因的qRT-PCR分析 Fig. 4 qRT-PCR analysis of genes related to low-temperature germination of sweet corn lines of DP, RP and L18 A:7 个候选基因的表达模式分析;B:UGT1 基因在不同组织样品中的表达水平 A: Expression pattern of 7 candidate genes; B: Expression level of UGT1 in different tissues |
3 讨论
已有不少报道指出,通过构建近等基因系研究玉米生长发育和生理过程的潜在分子机制[19-20]。本研究通过将高种子活力甜玉米材料Scil-H7的染色体片段导入低种子活力材料Scil-C4th的遗传背景,获得了不同种子活力的近等基因系。其中,L18的种子发芽率比RP高23.58%,发芽势是RP的1.20倍,显著提高发芽指数和种子活力指数。生理实验结果表明,L18很可能携带了有利于低温条件下种子活力和发芽能力的关键基因位点,这为玉米低温萌发相关基础研究提供了有价值的种质资源。
通过Illumina测序获得Scil-H7、Scil-C4th和L18甜玉米胚在两个发芽阶段平均产生的8.54 Gb原始序列数据。甜玉米种子发芽期间已建成了复杂的信号和代谢途径[21-22],影响种子发芽的因素包括不同途径的变化[23]。转录组分析表明,发芽过程中全基因组基因表达发生广泛的变化[24]。许多已鉴定的DEGs编码蛋白质在代谢过程和核苷酸/蛋白质结合中的功能已经得到证明。KEGG富集显示,本研究在3个品系中同时鉴定了6个关键通路,包括zma04712昼夜节律-植物、zma01040不饱和脂肪酸生物合成、zma00945芪类化合物、二芳基庚酸和姜酚生物合成、zma00941类黄酮生物合成、zma00940苯丙酸生物合成和zma00195光合作用。途径分析表明,这些DEGs主要与次级代谢和种子发芽活力相关。转录组分析一共鉴定出447个DEGs存在于不同发芽阶段的Scil-H7、Scil-C4th和L18中,它们可能建立了一个调控网络,并随胚发芽进程而变化。
基于L18和轮回亲本Scil1-C4th之间相似的遗传性状,通过高通量转录组测序的方式捕获与种子活力相关的DEGs作为候选基因。将Scil-H7设为参照,在L18、Scil-H7和Scil1-C4th的两个发芽阶段比较鉴定出410个DEGs,其中65个仅在Scil-H7和L18中表达,而不在Scil1-C4th中表达,这些DEGs被认为是与种子发芽紧密关联的调节因子,可用于后续功能分析。通过对不同组织样本的qRT-PCR验证,发现Zm00001d037383(UGT1 基因)与低温发芽高度相关。
对UGT1 基因进行功能注释与同源基因功能搜索,发现在拟南芥中UGT1可以催化单螺旋藻,促进葡萄糖结合;UGT1的敲除导致木质素单体生物合成,木质素聚合和细胞壁相关转录因子表达水平大幅提高,表明UGT1催化的单螺旋葡萄糖基化对正常细胞壁木质素是必需的,从而在一定程度上阐明了细胞壁发育和细胞壁木质化的分子机制[25]。然而,细胞壁的发育是植物种子早期发芽的重要特征,细胞壁相关基因表达的活跃对植物种子更强的抵抗逆境胁迫能力、快速发芽与出苗能力具有重要意义。因此,推测UGT1 基因可能是Scil-H7中响应低温发芽的重要目标基因。分析UGT1 基因在转录组测序样品中的表达量发现,该基因在玉米种子低温发芽过程中表达量迅速增加,表现为低温胁迫下的迅速响应。UGT1 基因在玉米中耐低温发芽的作用及其分子机制有待进一步探索。
4 结论本研究利用高通量转录组测序技术,对耐低温发芽差异的母本及其自交系后代进行分析,从不同材料在相同时期的4个比较组(RP-1 vs DP-1、RP-2 vs DP-2、RP-1 vs L18-1、RP-2 vs L18-2)鉴定出共有DEGs 410个。其中,65个DEGs仅在Scil-H7和L18中特异表达,32个基因功能注释完整,从中进一步筛选出20个可能与低温萌发密切相关的基因。对其中最有可能参与低温萌发的7个上调表达基因进行表达量验证,其中编码UDP-葡萄糖基转移酶的基因Zm00001d037383(UGT1)在DP胚部表达明显高于其他组织,推测该基因在甜玉米耐低温萌发过程中起关键作用。研究结果,为后续创制低温萌发能力更强的甜玉米种质资源奠定了基础。
| [1] |
PRESTERL T, OUZUNOVA M, SCHMIDT W, MOLLER E M, ROBER F K, KNAAK C, ERNST K, WESTHOFF P, GEIGER H H. Quantitative trait loci for early plant vigour of maize grown in chilly environments[J]. Theoretical and Applied Genetics, 2007, 114(6): 1059-1070. DOI:10.1007/s00122-006-0499-4 |
| [2] |
PETER R, ESCHHOLZ T W, STAMP P, LIEDGENS M. Swiss Flint maize landraces—A rich pool of variability for early vigour in cool environments[J]. Field Crops Research, 2009, 110(2): 157-166. DOI:10.1016/j.fcr.2008.07.015 |
| [3] |
刘蔚楠, 万忠, 甘阳英, 胡建广, 尹艳. 2015年广东甜玉米产业发展形势与对策建议[J]. 广东农业科学, 2016, 4(3): 12-16. DOI:10.3969/j.issn.1004-874X.2013.12.002.issn.1004-874X.2013.12.002 LIU W N, WAN Z, GAN Y Y, HU J G, YIN Y. Development situation and c ountermeasures of Guangdong sweet corn industry in 2015[J]. Guangdong Agricultural Sciences, 2016, 4(33): 12-16. DOI:10.3969/j.issn.1004-874X.2013.12.002.issn.1004-874X.2013.12.002 |
| [4] |
MCCONNELL R, GARDNER C. Inheritance of several cold tolerance traits in Corn 1[J]. Crop Science, 1979, 19(6): 847-852. DOI:10.2135/cropsci1979.0011183X001900060025x |
| [5] |
HODGES D M, ANDREWS C J, JOHNSON D A, HAMILTON R I. Sensitivity of maize hybrids to chilling and their combining abilities at two developmental stages[J]. Crop Science, 1997, 37(3): 850-856. DOI:10.2135/cropsci1997.0011183X003700030026x |
| [6] |
REVILLA P, MALVAR R A, CARTEA M E, BUTRON A, ORDAS A. Inheritance of cold tolerance at emergence and during early season growth in maize[J]. Crop Science, 2000, 40(6): 1579-1585. DOI:10.2135/cropsci2000.4061579x |
| [7] |
HUND A, FRACHEBOUD Y, SOLDATI A, FRASCAROLI E, SALVI S, STAMP P. QTL controlling root and shoot traits of maize seedlings under cold stress[J]. Theoretical and Applied Genetics, 2004, 109(3): 618-629. DOI:10.1007/s00122-004-1665-1 |
| [8] |
JOMPUK C, FRACHEBOUD Y, STAMP P, LEIPNER J. Mapping of quantitative trait loci associated with chilling tolerance in maize(Zea mays L.)seedlings grown under field conditions[J]. Journal of Experimental Botany, 2005, 56(414): 1153-1163. DOI:10.1093/jxb/eri108 |
| [9] |
NGUYEN H T, LEIPNER J, STAMP P, GUERRA-PERAZA O. Low temperature stress in maize(Zea mays L.)induces g enes i nvolved in photosynthesis and signal transduction as studied by suppression subtractive hybridization[J]. Plant Physiology and Biochemistry, 2009, 47(2): 116-122. DOI:10.1016/j.plaphy.2008.10.010 |
| [10] |
Rodríguez V M, Butrón A, Rady M O, et al. Identification of quantitative trait loci involved in the response to cold stress in maize (Zea mays L.)[J]. Molecular Breeding, 2014, 33(2): 363-371. DOI:10.1007/s11032-013-9955-4 |
| [11] |
HU S D, LUEBBERSTEDT T, ZHAO G W, LEE M. QTL mapping of low-temperature germination ability in the maize IBM Syn4 RIL Population[J]. PLOS ONE, 2016, 11(3): e0152795. DOI:10.1371/journal.pone.0152795 |
| [12] |
李旭辉, 王贵华, 贾光耀, 任利沙, 李莉, 付俊杰, 王国英, 顾日良, 王建华. 基于三个相关群体的玉米低温发芽能力相关性状的QTL定位//中国作物学会作物种子专业委员会学术年会论文摘要集[C]. 2017: 21-22. LI X H, WANG G H, JIA G Y, REN L S, LI L, FU J J, WANG G Y, GU R L, WANG J H. QTL mapping for low temperature germination ability in three connected populations in Zea mays L. //Abstract Book of Crop Seed Association of Crop Science Society of China[C].2017: 21-22. |
| [13] |
毛笈华, 文天祥, 于永涛, 李春艳, 祁喜涛, 高磊, 胡建广. 甜玉米种子活力及其杂种优势初步研究[J]. 广东农业科学, 2015, 13(20): 37-41. DOI:10.16768/j.issn.1004-874X.2015.13.020 MAO J H, WEN T X, YU Y T, LI C Y, QI X T, GAO L, HU J G. Study on seed vigor of sweet corn and its heretosis[J]. Guangdong Agricultural Sciences, 2015, 13(20): 37-41. DOI:10.16768/j.issn.1004-874X.2015.13.020 |
| [14] |
CHENG X, HE S, GENG G. Dynamic QTL analysis of seed reserve utilization in sh2 sweet corn germination stages[J]. Genetics and Molecular Research, 2016, 15(3): gmr.15038183. DOI:10.4238/gmr.15038183 |
| [15] |
CHEN L K, GAO W W, CHEN S P, WANG L P, ZOU J Y, LIU Y Z, WANG H, CHEN Z Q, GUO T. High-resolution QTL mapping for grain appearance traits and co-localization of chalkiness-associated differentially expressed candidate genes in rice[J]. Rice, 2016, 9(48): 1-17. DOI:10.1186/s12284-016-0121-6 |
| [16] |
LANGMEAD B, SALZBERG S L. Fast gapped-read alignment with Bowtie 2[J]. Nature Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923 |
| [17] |
KIM D, PERTEA G, TRAPNELL C, PIMENTEL H, KELLEY R, SALZBERG S L. TopHat2: accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions[J]. Genome Biology, 2013, 14(4): 1-13. DOI:10.1186/gb-2013-14-4-r36 |
| [18] |
ANDERS S, PYL P T, HUBER W. HTSeq-a Python framework to work with high-throughput sequencing data[J]. Bioinformatics, 2015, 31(2): 166-169. DOI:10.1093/bioinformatics/btu638 |
| [19] |
PEA G, AUNG H H, FRASCAROLI E, LANDI P, PE M E. Extensive genomic characterization of a set of near-isogenic lines for heterotic QTL in maize(Zea mays L.)[J]. Bmc Genomics, 2013, 14(1): 1-15. DOI:10.1186/1471-2164-14-61 |
| [20] |
CUI M, JIA B, LIU H, KAN X, ZHANG Y, ZHOU R, LI Z, YANG L, DENG D, YIN Z. Genetic mapping of the leaf number above the primary ear and its relationship with plant height and flowering time in maize[J]. Frontiers in Plant Science, 2017, 8: 1437. DOI:10.3389/fpls.2017.01437 |
| [21] |
SANWO M M, DEMASON D A. Characteristics of alpha-Amylase during germination of two high-sugar sweet corn cultivars of Zea mays L[J]. Plant Physiology, 1992, 99(3): 1184-1192. DOI:10.1104/pp.99.3.1184 |
| [22] |
XIANG N, GUO X B, LIU F Y, LI Q, HU J G, BRENNAN C S. Effect of light-and dark-germination on the phenolic biosynthesis, phytochemical profiles, and antioxidant activities in sweet corn(Zea mays L.)sprouts[J]. International Journal of Molecular Sciences, 2017, 18(6): 1246. DOI:10.3390/ijms18061246 |
| [23] |
REVILLA P, BUTRON A, RODRIGUEZ V M, MALVAR R A, ORDAS A. Identification of genes related to germination in aged maize seed by screening natural variability[J]. Journal of Experimental Botany, 2009, 60(14): 4151-4157. DOI:10.1093/jxb/erp249 |
| [24] |
决登伟, 桑雪莲, 舒波, 刘丽琴, 王一承, 石胜友. 玉米WRKY转录因子非生物胁迫的表达分析[J]. 广东农业科学, 2017, 44(1): 15-22. DOI:10.16768/j.issn.1004-874X.2017.01.003.issn.1004-874X.2017.01.003 JUE W D, SANG X L, SHU B, LIU L Q, WANG Y C, SHI S Y. Expressi on a nalysis of maize WRKY transcription factor genes under abioticstress[J]. Guangdong Agricultural Sciences, 2017, 44(1): 15-22. DOI:10.16768/j.issn.1004-874X.2017.01.003.issn.1004-874X.2017.01.003 |
| [25] |
LIN J S, HUANG X X, LI Q, CAO Y P, BAO Y, MENG X F, LI Y J, FU C X, HOU B K. UDP-glycosyltransferase 72B1 catalyzes the glucose conjugation of monolignols and is essential for the normal cell wall lignification in Arabidopsis thaliana[J]. The Plant Journal, 2016, 88(1): 26-42. DOI:10.1111/tpj.13229 |
 2021, Vol. 48
2021, Vol. 48