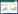文章信息
基金项目
- 国家重点研发计划项目(2016YFD0500402);国家自然科学基金(31972712,31702238)
作者简介
- 赵维荣(1995 -),女,在读硕士生,研究方向为兽医公共卫生,E-mail:2287239319@qq.com.
通讯作者
- 张茂林(1969 -),男,博士,副教授,研究方向为人兽共患病毒病,E-mail:zhre98@163.com.
文章历史
- 收稿日期:2020-03-10
2. 吉林大学动物医学学院,吉林 长春 130062
2. College of Veterinary Medicine, Jilin University, Changchun 130062, China
【研究意义】狂犬病是由狂犬病病毒(Rabies virus,RABV)引起的一种古老的、以神经系统感染为特征的人兽共患病毒性传染病[1]。该病一直是公共卫生关注的重点,感染者一旦发病,死亡率可近乎达到100%[2],迄今为止尚无有效的治疗方法。目前该病主要流行于亚洲和非洲,其中印度和中国较为严重[3]。RABV典型的病毒粒子呈子弹状,基因组为不分节段的单股负链RNA,大小约12 kb,沿3′端向5′端依次编码核蛋白(N)、磷蛋白(P)、基质蛋白(M)、糖蛋白(G)和依赖RNA的RNA聚合酶蛋白(L)。基因组RNA与N、P、L蛋白共同形成核糖核蛋白复合体(RNP),也称核衣壳,是病毒转录和基因组复制的功能性模板[4-6]。M蛋白位于RABV囊膜下,连接RNP和保护性抗原G,在RNP和含糖蛋白囊膜之间起桥梁作用[7]。了解M蛋白的合成分布场所有利于解析RABV病毒粒子的装配过程。
【前人研究进展】M蛋白由202个氨基酸组成,是RABV结构蛋白中长度最小的一种蛋白。已有研究表明,M蛋白在细胞膜上的聚集可以增加其对脂质双分子层的结合亲和力,在膜曲率、RNPs包膜和病毒出芽中发挥重要作用,并且在调节结构蛋白合成及装配的动态平衡中也起重要作用,同时对于RABV致病性及子代病毒粒子的形态发生也至关重要[8-9]。在RABV 5个结构蛋白中,N蛋白、P蛋白和L蛋白主要在胞质内基氏小体中合成,G蛋白主要通过粗面内质网(ER)翻译和高尔基体(Golgi)加工,最后到达出芽部位[10-11]。对于M蛋白的研究更多集中在其作用上,而对于其合成和分布场所的研究则相对较少。
【本研究切入点】研究M蛋白合成和分布场所是揭示RABV病毒粒子转配过程中M蛋白、G蛋白和RNP三者间结合顺序的关键。【拟解决的关键问题】本研究首先利用TCID50法对3种RABV毒株(CVS-11、SRV-9、PB4)在N2A、BHK细胞上的效价进行测定,随后分别接种N2A、BHK细胞,利用免疫荧光和激光共聚焦显微镜观察RABV M蛋白与ER和Golgi在胞内共定位以及M蛋白与G蛋白、M蛋白与N蛋白在这两种细胞器内的共定位,研究结果可望推断M蛋白的合成加工途径以及G蛋白、M蛋白和RNP三者的组装顺序,为RABV粒子的复制与装配过程的解析提供有力证据。
1 材料与方法 1.1 试验材料1.1.1 细胞与毒株 小鼠神经瘤母细胞N2A、乳仓鼠肾细胞BHK由吉林大学人兽共患病研究所病毒病研究室保存并进行传代培养。RABV的实验室标准攻击毒株CVS-11、口服疫苗毒株SRV-9由吉林大学人兽共患病研究所病毒病研究室保存,街毒株PB4由长春军事兽医研究所OIE狂犬病参考实验室涂长春研究员惠赠。
1.1.2 主要试剂与设备 细胞培养基DMEM、内质网标志蛋白抗体〔Anti-Calnexin antibody produced in rabbit,Anti-Protein Disulfide Isomerase(MD-12)antibody produced in rabbit〕和高尔基体标志蛋白抗体(Anti-ERGIC-53/p58 antibody produced in rabbit)购自Sigma公司;胎牛血清FBS购自Biological Industries公司,青链霉素混合液、Hoechs 33258染色液购自Solarbio公司;丙酮购自北京化工厂;PBS粉末试剂购自Monad公司;伊文思兰、Alexa FluorTM 546 goat antirabbit antibody、Alexa FluorTM 647 goat anti-mouse antibody购自Thermo公司;多聚甲醛溶液购自源叶生物公司;Triton X-100购自生工生物工程(上海)股份有限公司;FITC Anti-Rabies Monoclonal Globulin购自FUJIREBIO公司;FITC Anti-RABV G蛋白抗体、FITC Anti-RABV P蛋白抗体由长春军事兽医研究所扈荣良研究员惠赠;鼠源抗RABV M蛋白抗体由吉林大学人兽共患病研究所病毒病研究室制作并保存。96孔板、24孔板购自Corning公司;细胞爬片购自NEST公司;显微镜盖玻片购自江苏世泰实验器材有限公司。
1.2 试验方法1.2.1 病毒培养 用T25细胞培养瓶培养BHK细胞至70%~80% 时,按MOI=1的量将CVS-11、SRV-9接种于细胞瓶中,37℃下感作1 h后弃去上清,加入含1% FBS的DMEM培养基继续培养,96 h后置于-80℃并反复冻融3次,4℃、1 000 r/min离心10 min取上清,分装保存于-80℃。PB4由4周龄小鼠颅内接种培养,待小鼠濒死时无菌取鼠脑,加入含20%FBS的DMEM培养基,组织细胞破碎仪充分破碎研磨后,4℃、1 000 r/min离心10 min取上清,分装保存于-80℃。
1.2.2 TCID50法测定不同RABV毒株效价 取96孔板,将细胞以1×104个/ 孔铺于孔板中,37℃培养12 h待其贴壁后弃去上清,将病毒液用含1%FBS的DMEM培养基按10-1~10-8倍比稀释,4个复孔,100 μL/ 孔。37℃培养48 h后弃去上清,PBS润洗1次,在80% 冷丙酮中室温下固定30 min,PBS润洗3次,将FITC Anti-Rabies Monoclonal Globulin、伊文思蓝均以1 ∶ 200稀释,每孔加入50 μL,37℃避光孵育1 h,PBS清洗3次,每次10 min,于荧光显微镜下观察绿色荧光标记的阳性孔数并记录,应用Spearman-Karber公式计算每种RABV毒株的TCID50。
1.2.3 免疫荧光 将细胞爬片放进24孔板,将细胞以10×104/ 孔铺于爬片上,37℃培养12 h待其贴壁后弃去上清,PBS润洗3次,加入少量不含FBS的DMEM培养基,将CVS-11、SRV-9、PB4按MOI=1进行接毒并设置对照,置37℃感作1 h后弃去上清,PBS润洗3次,加入含1% FBS的DMEM培养基继续培养,CVS-11、SRV-9于培养24 h时收样,PB4于培养72 h时收样;4% PFA室温固定25~30 min,0.1% Triton-X 100室温打孔10 min,10% FBS室温封闭2 h,内质网和高尔基体标志蛋白抗体作为一抗4℃过夜,其荧光二抗、抗RABV M蛋白抗体及其荧光二抗、FITC AntiRabies Monoclonal Globulin、FITC Anti-RABV G蛋白抗体、FITC Anti-RABV P蛋白抗体均于37℃孵育1 h即可,Hoechs 33258染色液染核5 min,90% 甘油封片并用锡纸包裹于4℃下保存,于激光共聚焦显微镜下观察并采集图像。
2 结果与分析 2.1 不同RABV毒株在不同细胞上的效价在进行免疫荧光前,为准确计算细胞接毒量,利用TCID50法测定CVS-11、SRV-9、PB4毒株在N2A、BHK细胞上的效价。结果显示,CVS- 11、SRV-9、PB4毒株的效价有明显差别,感染48 h后CVS-11的效价相对较高,SRV-9的效价相对较低,PB4是一种直接从感染小鼠脑中分离得到的街毒株,只感染N2A细胞,感染72 h后其效价依然很低(表 1)。
2.2 不同RABV毒株M蛋白在不同细胞上与ER、Golgi的共定位
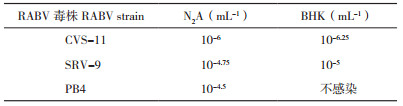
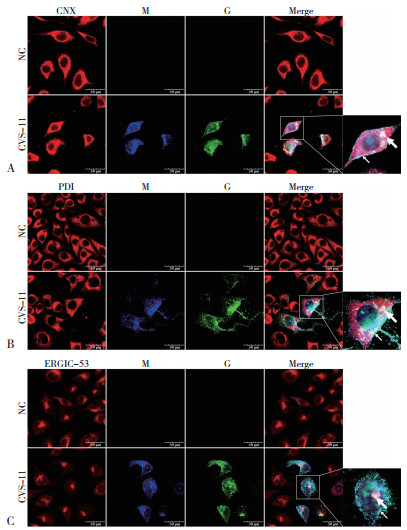
2.2.1 不同RABV毒株M蛋白在N2A细胞上与ER、Golgi的共定位 为探究N2A细胞中ER、Golgi在RABV M蛋白合成加工过程中的作用,结合不同RABV株的感染特性,CVS-11、SRV-9于24 h后进行免疫荧光,PB4于72 h后进行免疫荧光。通过激光共聚焦观察显示,在N2A细胞中CVS-11、SRV-9、PB4的M蛋白与CNX、PDI存在共定位,且共定位主要出现在胞浆内(图 1、图 2);与ERGIC-53也存在共定位,且共定位也主要出现在胞浆内(图 3),说明在N2A细胞中CVS-11、SRV-9、PB4的M蛋白是通过ERGolgi途径合成加工;不同的是CVS-11、SRV-9的M蛋白在胞膜附近有大量表达,而PB4的M蛋白在胞膜附近几乎无表达,说明CVS-11、SRV-9的M蛋白在24 h后已到达细胞膜,而PB4的M蛋白即使在72 h后也很少出现在细胞膜附近。

|
| 图 1 N2A细胞中CVS-11(A)、SRV-9(B)、PB4(C)M蛋白与ER标志蛋白CNX的共定位 Fig. 1 Co-localization of M proteins from CVS-11(A), SRV-9(B), PB4(C)with ER marker protein CNX in N2A cells 共定位处显示粉色;箭头所示为细胞膜附近 The co-localization is shown in pink; The arrow shows the vicinity of the cell membrane |
|
| 图 2 N2A细胞中CVS-11(A)、SRV-9(B)、PB4(C)M蛋白与ER标志蛋白PDI的共定位 Fig. 2 Co-localization of M proteins from CVS-11(A), SRV-9(B), PB4(C)with ER marker protein PDI in N2A cells 共定位处显示粉色;箭头所示为细胞膜附近 The co-localization is shown in pink; The arrow shows the vicinity of the cell membrane |

|
| 图 3 N2A细胞中CVS-11(A)、SRV-9(B)、PB4(C)M蛋白与Golgi标志蛋白ERGIC-53的共定位 Fig. 3 Co-localization of M proteins from CVS-11(A), SRV-9(B), PB4(C)with Golgi marker protein ERGIC-53 in N2A cells 共定位处显示粉色;箭头所示为细胞膜附近 The co-localization is shown in pink; The arrow shows the vicinity of the cell membrane |
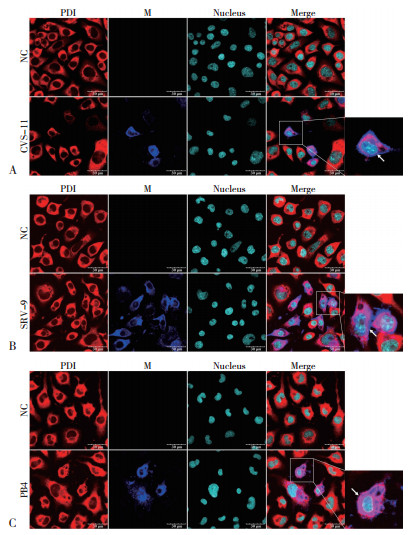
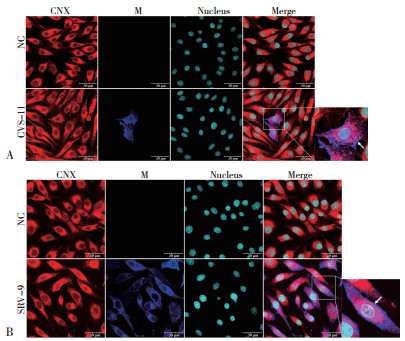
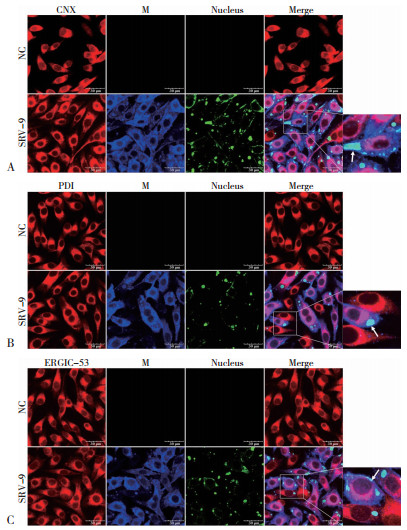
2.2.2 不同RABV毒株M蛋白在BHK细胞上与ER、Golgi的共定位 在N2A细胞内共定位的基础上,进一步探究不同RABV毒株M蛋白在BHK细胞中的共定位情况。通过激光共聚焦观察显示,在BHK细胞中CVS-11、SRV-9的M蛋白与CNX、PDI存在共定位,且共定位主要出现在胞浆内(图 4、图 5);与ERGIC-53也存在共定位,且共定位也主要出现在胞浆内(图 6);而且M蛋白在胞膜附近都有表达。这说明在BHK细胞中CVS-11、SRV-9的M蛋白是通过ER-Golgi途径合成加工,且CVS-11、SRV-9的M蛋白在24 h后都已到达细胞膜附近。
|
| 图 4 BHK细胞中CVS-11(A)、SRV-9(B)M蛋白与ER标志蛋白CNX的共定位 Fig. 4 Co-localization of M proteins from CVS-11(A), SRV-9(B)with ER marker protein CNX in BHK cells 共定位处显示粉色;箭头所示为细胞膜附近 The co-localization is shown in pink; The arrow shows the vicinity of the cell membrane |

|
| 图 5 BHK细胞中CVS-11(A)、SRV-9(B)M蛋白与ER标志蛋白PDI的共定位 Fig. 5 Co-localization of M proteins from CVS-11(A), SRV-9(B)with ER marker protein PDI in BHK cells 共定位处显示粉色;箭头所示为细胞膜附近 The co-localization is shown in pink; The arrow shows the vicinity of the cell membrane |

|
| 图 6 BHK细胞中CVS-11(A)、SRV-9(B)M蛋白与Golgi标志蛋白ERGIC-53的共定位 Fig. 6 Co-localization of M proteins from CVS-11(A), SRV-9(B)with Golgi marker protein ERGIC-53 in BHK cells 共定位处显示粉色;箭头所示为细胞膜附近 The co-localizationt is shown in pink; The arrow shows the vicinity of the cell membrane |
2.3 RABV的M蛋白与G蛋白以及ER、Golgi的共定位(以CVS-11感染N2A细胞为例)
由前述结果可知,3种RABV毒株的M蛋白通过ER-Golgi途径合成加工,G蛋白与M蛋白在细胞内共定位,有利于解析RABV部分结构蛋白的加工及组装。在N2A细胞中以MOI=1的病毒量接种CVS-11,24 h后进行免疫荧光。通过激光共聚焦观察显示,CVS-11的M蛋白、G蛋白与ER、Golgi三者存在共定位(图 7),且三者共定位主要出现在胞浆内,在膜附近M蛋白与G蛋白出现共定位,说明CVS-11的G蛋白也可能通过ER-Golgi途径合成加工,可能在Golgi内M蛋白与G蛋白结合并转运到细胞膜附近。
|
| 图 7 N2A细胞中CVS-11的M蛋白、G蛋白与ER、Golgi的共定位 Fig. 7 Co-localization of M proteins and G proteins from CVS-11 with ER or Golgi in N2A cells A、B:M蛋白、G蛋白与ER的共定位,M蛋白与G蛋白共定位显示灰蓝色(小箭头所示,下同),M蛋白、G蛋白与ER三者共定位显示白色(大箭头所示,下同);C:M蛋白、G蛋白与Golgi的共定位,M蛋白与G蛋白共定位(小箭头所示),M蛋白、G蛋白与Golgi三者共定位(大箭头所示) A and B: Co-localization of CVS-11 M protein, G protein and ER, colocalization of M protein and G protein is shown in gray-blue(shown by small arrow, the same below)while co-localization of M protein, G protein and ER is shown in white(shown by large arrow, the same below); C: Co-localization of CVS-11 M protein, G protein and Golgi, co-localization of M protein and G protein(shown by small arrow), co-localization of M protein, G protein and Golgi(shown by large arrow) |
2.4 RABV的M蛋白与N蛋白以及ER、Golgi的共定位(以SRV-9感染BHK细胞为例)
已有研究表明,N蛋白以RNP形式与G蛋白和M蛋白组装成完整的RABV粒子。在前述结果的基础上,通过研究M蛋白和N蛋白的共定位,探究M蛋白与RNP的结合先后顺序。通过激光共聚焦显示,在BHK细胞中,SRV-9的M蛋白与ER、Golgi有明显共定位,这与前述结果相符,而N蛋白与ER、Golgi无明显共定位;M蛋白与N蛋白的共定位主要在胞浆内,且多出现在细胞膜附近(图 8箭头所示),提示病毒粒子可能处于出芽阶段,同时也说明M蛋白与RNP结合发生于细胞膜附近。
|
| 图 8 BHK细胞中SRV-9的M蛋白和N蛋白与ER、Golgi的共定位 Fig. 8 Co-localization of M proteins and N proteins from SRV-9 with ER or Golgi in BHK cells A、B:M蛋白、N蛋白与ER的共定位,M蛋白与N蛋白共定位显示灰蓝色,M蛋白、N蛋白与ER三者共定位显示白色(下同);C:M蛋白、N蛋白与Golgi的共定位。箭头所示为细胞膜附近 A and B: Co-localization of SRV-9 M protein, N protein and ER, co-localization of M protein and N protein is shown in gray-blue while co-localization of M protein, N protein and ER is shown in white(the same below); C: Colocalization of SRV-9 M protein, N protein and Golgi. Arrows show the vicinity of the cell membrane |
3 讨论
狂犬病是由RABV感染引起的一种以神经系统障碍为特征的高度致死性人畜共患传染病[12]。M蛋白是RABV 5个结构蛋白中氨基酸数量最小但变异却最大的蛋白,在结构蛋白的合成和装配过程中起调控作用。ER是真核细胞中最大的细胞器之一,呈扁平囊状或小管状。研究发现,许多分泌蛋白和膜蛋白都是由ER合成;此外,ER还参与多种蛋白质折叠、脂类和胆固醇合成等许多过程[13]。病毒在感染细胞的过程中也会利用ER使其成为病毒与宿主细胞相互作用的核心[14]。粗面内质网(RER)表面附着大量核糖体,是胞内多种蛋白的合成主要场所。Golgi是细胞内的中央膜结合细胞器,常位于细胞核附近,作为分泌通路的中心枢纽,接收来自ER的蛋白质并进行加工修饰后,将它们输出到其他内膜结构,如内体、溶酶体、质膜和细胞外,以发挥各自功能[15]。Golgi还可以作为许多病毒的膜支架,或者被病毒用作病毒工厂进行病毒复制[16]。ER-Golgi途径是细胞中许多蛋白质的合成加工场所,然后被转运到达各自工作位点。研究发现,多种病毒结构蛋白都由ER-Golgi途径合成加工,例如,冠状病毒(CoV)结构蛋白M、S和E在ER被合成,被折叠并包装进入COP Ⅱ形成的囊泡中,然后从特定的ER出口位点出芽,随后到达Golgi被糖基化修饰[17, 18]。丙型肝炎病毒(HCV)包膜糖蛋白在ER中与载脂蛋白相互作用,形成HCV LVPs后以COP Ⅱ囊泡的形式运输到Golgi,通过Golgi分泌途径最终出口[19]。水泡性口炎病毒(VSV)的糖蛋白G也是通过ER-Golgi途径合成[20]。M蛋白作为RABV结构蛋白之一,在RABV组装过程中将RNP与囊膜糖蛋白G的C端连接起来,是形成完整病毒粒子的关键,并决定了病毒粒子的毒力及下一级神经元的感染。关于RABV的M蛋白合成加工途径的研究较少,我们猜测该蛋白也是通过ER-Golgi途径合成加工。RABV在细胞内进行转录、复制,合成所有结构蛋白后便开始组装新的病毒粒子,其基因组与N蛋白、P蛋白及L蛋白共同形成RNP,但对于G蛋白、M蛋白及RNP三者结合的先后顺序还尚不清楚,它们如何组装成完整的RABV粒子还需进一步探究。为此,本研究应用免疫荧光技术和激光共聚焦显微镜,发现在N2A、BHK细胞中,3种RABV毒株(CVS-11、SRV-9、PB4)的M蛋白均与ER、Golgi存在共定位,同时也发现RABV的M蛋白与G蛋白在ER、Golgi上共定位,而M蛋白与N蛋白则在胞浆中共定位,说明在体外传代细胞中,RABV的M蛋白是通过ER-Golgi途径合成加工,且不同毒株在不同细胞系中的结果具有一致性。此外,在病毒粒子组装过程中M蛋白有可能先与G蛋白结合,随后与N蛋白、P蛋白和L蛋白及RNA形成的RNP结合并出芽。本研究结果主要在体外传代细胞中完成,关于RABV的M蛋白在体内是否也存在同样的合成加工途径以及病毒粒子是否有同样的装配过程还有待进一步探索,而RABV的M蛋白在ER-Golgi途径中的合成修饰过程仍未知,同时核衣壳RNP的位置、数量及颗粒大小也是RABV复制能力的体现,不同毒株间是否存在差异也将是后期研究的重点。
4 结论M蛋白作为RABV结构蛋白之一,无论是在病毒的前期转录复制过程还是后期装配出芽过程都具有重要作用,因此对M蛋白合成和分布场所的研究非常重要。本研究初步探究了不同RABV毒株M蛋白在胞内的合成和分布,发现RABV M蛋白在细胞内主要通过ER-Golgi途径合成,并与G蛋白结合转运到细胞膜附近,随后与RNP结合,且不同RABV毒株在不同细胞系中没有明显差异。
| [1] |
GILBERT A T. Rabies virus vectors and reservoir species[J]. Revue Scientifique et Technique, 2018, 37(2): 371-384. DOI:10.20506/rst.37.2.2808 |
| [2] |
BRUNKER K, MOLLENTZE N. Rabies Virus[J]. Trends in Microbiology, 2018, 26(10): 886-887. DOI:10.1016/j.tim.2018.07.001 |
| [3] |
WUNNER W H, BRIGGS D J. Rabies in the 21st Century[J]. PLoS Neglected Tropical Diseases, 2010, 4(3): e591. DOI:10.1371/journal.pntd.0000591 |
| [4] |
SCHNELL M J, MCGETTIGAN J P, WIRBLICH C, PAPANERI A. The cell biology of rabies virus: using stealth to reach the brain[J]. Nature Reviews, Microbiology, 2010, 8(1): 51-61. DOI:10.1038/nrmicro2260 |
| [5] |
GAO J, WANG X, ZHAO M, LIU E, DUAN M, GUAN Z, GUO Y, ZHANG M. Entry of Challenge Virus Standard(CVS)-11 into N2a cells via a clathrin-mediated, cholesterol-, dynamin-, pHdependent endocytic pathway[J]. Virology Journal, 2019, 16(1): 80. DOI:10.1186/s12985-019-1186-9 |
| [6] |
BERTOUNE M A R, NICKL B, KRIEGER T, WOHLERS L, BONATERRA G A, DIETZSCHOLD B, WEIHE E, BETTE M. The phenotype of the RABV glycoprotein determines cellular and global virus load in the brain and is decisive for the pace of the disease[J]. Virology, 2017, 511: 82-94. DOI:10.1016/j.virol.2017.08.019 |
| [7] |
LUO J, ZHANG Y, ZHANG Q, WU Y, ZHANG B, MO M, TIAN Q, ZHAO J, MEI M, GUO X. The deoptimization of rabies virus matrix protein impacts viral transcription and replication[J]. Viruses, 2019, 12(1): 4. DOI:10.3390/v12010004 |
| [8] |
PULMANAUSAHAKUL R, LI J, SCHNELL M J, DIETZSCHOLD B. The glycoprotein and the matrix protein of rabies virus affect pathogenicity by regulating viral replication and facilitating cellto-cell spread[J]. Journal of Virology, 2008, 82(5): 2330-2338. DOI:10.1128/JVI.02327-07 |
| [9] |
MA X, LI Z. Significantly improved recovery of recombinant sonchus yellow net rhabdovirus by expressing the negative-strand genomic RNA[J]. Viruses, 2020, 12(12): 1459. DOI:10.3390/v12121459 |
| [10] |
FOOKS A R, CLIQUET F, FINKE S, FREULING C, HEMACHUDHA T, MANI R S, MULLER T, NADIN-DAVIS S, PICARD-MEYER E, WILDE H, BANYARD A C. Rabies[J]. Nature Reviews, Disease Primers, 2017, 3: 17091. DOI:10.1038/nrdp.2017.91 |
| [11] |
NIKOLIC J, LE BARS R, LAMA Z, SCRIMA N, LAGAUDRIEREGESBERT C, GAUDIN Y, BLONDEL D. Negri bodies are viral factories with properties of liquid organelles[J]. Nature Communications, 2017, 8(1): 58. DOI:10.1038/s41467-017-00102-9 |
| [12] |
SINGH R, SINGH K P, CHERIAN S, SAMINATHAN M, KAPOOR S, MANJUNATHA REDDY G B, PANDA S, DHAMA K. Rabies - epidemiology, pathogenesis, public health concerns and advances in diagnosis and control: a comprehensive review[J]. Veterinary Quarterly, 2017, 37(1): 212-251. DOI:10.1080/01652176.2017.1343516 |
| [13] |
SCHWARZ D S, BLOWER M D. The endoplasmic reticulum: structure, function and response to cellular signaling[J]. Cellular and Molecular Life Sciences, 2016, 73(1): 79-94. DOI:10.1007/s00018-015-2052-6 |
| [14] |
BAGCHI P. Endoplasmic reticulum in viral infection[J]. International Review of Cell and Molecular Biology, 2020, 350: 265-284. DOI:10.1016/bs.ircmb.2019.10.005 |
| [15] |
WANG Y, SEEMANN J. Golgi biogenesis[J]. Cold Spring Harbor Perspectives Biology, 2011, 3(10): a005330. DOI:10.1101/cshperspect.a005330 |
| [16] |
LI J, AHAT E, WANG Y. Golgi structure and function in health, stress, and diseases[J]. Results and Problems in Cell Differentiation, 2019, 67: 441-485. DOI:10.1007/978-3-030-23173-6_19 |
| [17] |
UJIKE M, TAGUCHI F. Incorporation of spike and membrane glycoproteins into coronavirus virions[J]. Viruses, 2015, 7(4): 1700-1725. DOI:10.3390/v7041700 |
| [18] |
PERR IER A, BONNIN A, DESMAR ET S L, DANNEEL S A, GOFFARD A, ROUILLÉ Y, DUBUISSON J, BELOUZARD S. The C-terminal domain of the MERS coronavirus M protein contains a trans-Golgi network localization signal[J]. The Journal of Biological Chemistry, 2019, 294(39): 14 406-14 421. DOI:10.1074/jbc.RA119.008964 |
| [19] |
SYED G H, KHAN M, YANG S, SIDDIQUI A. Hepatitis C virus lipoviroparticles assemble in the endoplasmic reticulum(ER) and bud off from the ER to the Golgi compartment in COPⅡ vesicles[J]. Journal of Virology, 2017, 91(15): e00499-17. DOI:10.1128/JVI.00499-17 |
| [20] |
LO I C, GUPTA V, MIDDE K K, TAUPIN V, LOPEZ-SANCHEZ I, KUFAREVA I, ABAGYAN R, RANDAZZO P A, FARQUHAR M G, GHOSH P. Activation of Gαi at the Golgi by GIV/Girdin imposes finiteness in Arf1 signaling[J]. Developmental Cell, 2015, 33(2): 189-203. DOI:10.1016/j.devcel.2015.02.009 |
 2021, Vol. 48
2021, Vol. 48