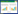文章信息
基金项目
- 国家现代农业产业技术体系专项(CARS-46);广东省科技计划项目(2014A020208021);广东省自然科学基金面上项目(2019A1515011884)
作者简介
- 黄金凤(1995-),女,硕士,研究方向为鱼类生理学,E-mail:2200311715@qq.com.
通讯作者
- 孙彩云(1972-),女,白族,博士,副教授,研究方向为鱼类生理学,E-mail:suncaiy@mail.sysu.
文章历史
- 收稿日期:2021-04-16
【研究意义】几丁质是由β-1, 4糖苷键连接N-乙酰氨基葡萄糖的一种线性多糖,是自然界中唯一存在的阳离子型可食用纤维。几丁质贮量十分丰富,仅海洋生物每年所合成的几丁质已超过10亿t,是自然界中贮量仅次于纤维素的第二大天然多糖[1]。几丁质多存在于真菌细胞壁、昆虫消化道和多数节肢动物的外骨骼中,因此研究关于降解几丁质的酶具有重要意义。几丁质酶(EC 3.2.1.14)是一种糖苷水解酶,催化N-乙酰氨基葡萄糖从几丁质上脱去,从而降解几丁质。几丁质酶根据其催化结构域的氨基酸序列相似性主要分为糖基水解酶(GH)家族18、19、23和48,该酶在自然界分布广泛,在细菌到高等植物和动物等生物体中均有分布,植物的几丁质酶属于GH19,鱼类的几丁质酶均为GH18,GH23和GH48分别在细菌和真菌中分布[2-4]。几丁质酶具有多种生理作用,如促进甲壳类和昆虫类蜕皮,降解鱼类胃肠道里的虾蟹壳帮助消化,在植物里则参与抵抗含有几丁质的致病菌[4],在鱼皮肤粘液中抵抗寄生虫等[5]。
【前人研究进展】鱼类中的几丁质酶最早在海鲷的胃肠道中被发现[6]。随着基因组测序的快速发展,已研究获得斑马鱼、日本比目鱼等多种鱼类的几丁质酶序列[7]。肉食性海水鱼类的天然食谱中含有丰富的几丁质,如含有高结晶度α-几丁质的虾蟹和浮游动物,含有低结晶度β-几丁质的乌贼等[4, 8]。研究表明,鱼类消化道组织具有几丁质降解活性,其中胃中的几丁质酶活性最高,降解几丁质寡糖的活性比其他组织高,如白姑鱼、日本鲭、斑鱾、琉球棘角鱼和梭鱼等。而降解单糖的β-N-乙酰氨基葡萄糖酶的活性在各组织中广泛分布,而非局限于消化道。不同的几丁质降解酶的分布很可能取决于该鱼类品种的饮食习惯和栖息地位置[4, 9]。已有研究表明,从鱼胃分离获得的几丁质酶约38~62 ku,与植物几丁质酶大小(24.5~29 ku)有明显差异。在实验室条件下,鱼胃的丁质酶降解几丁质低聚糖的最适pH值为2.0~5.0,降解几丁质多聚糖的最适pH值为1.5~5.5。这表明鱼类胃中的几丁质酶适应胃组织的酸性环境来降解几丁质底物[4]。
【本研究切入点】迄今为止,有关罗非鱼几丁质酶的研究报道仅有1篇,它揭示了罗非鱼血清、胃和肠中存在几丁质酶降解活性,能够降解4-甲基伞形酮-β-D-N, N-二乙酰壳三糖〔4MU-(GlcNAc)2〕或4-甲基伞形酮-β-D-N, N, N-三乙酰壳三糖〔4MU-(GlcNAc)3〕,其中血清中的几丁质酶具有最高的比活性[10]。但目前仍未见关于罗非鱼几丁质酶序列及其功能的报道。【拟解决的关键问题】本研究克隆了吉富罗非鱼3种预测的酸性几丁质酶,并对克隆结果进行了进化树分析,同时检测几丁质酶在罗非鱼中的组织分布,以期获得相关功能信息。提取罗非鱼胃组织蛋白并纯化其中的几丁质酶,测定酶活,比较罗非鱼和其他鱼类几丁质酶的活性,为罗非鱼几丁质酶的深入研究奠定基础。
1 材料与方法 1.1 试验材料本研究所用的吉富罗非鱼(70~100 g)约为30尾,均为雄性,购自广东省国家罗非鱼良种场。试验前,供试罗非鱼在12 h ︰ 12 h黑暗光照循环条件下,室温循环淡水中驯化1周,每天9:30和16:30进行饱食投喂。采样前24 h内对罗非鱼停食,在第2天早上进行取样。
主要试剂:总RNA提取试剂Trizol Reagent、反转录试剂盒M-MLV First Strand cDNA Synthesis Kit购自英潍捷基(上海)贸易有限公司;聚合酶Blend taq plus和连接试剂Ligation High,购自东洋纺(上海)生物科技有限公司;DNase Ⅰ购自美国纽英伦生物技术公司;E.Z.N.A® Gel Extraction Kit、E.Z.N.A® Plasmid Extraction Kit,购自Omega Bio-Tek公司;SYBR® Green Real-time PCR Master Mix试剂盒、100 bp DNA Ladder,购自广州东盛生物科技有限公司;DEPC、X-gal和IPTG购自生工生物工程(上海)公司。引物由英潍捷基(上海)贸易有限公司合成。所用大肠杆菌DH5α和pUCM-T载体购自英潍捷基(上海)贸易有限公司,感受态细菌则由本实验室制备。其他试剂均为国产分析纯。
主要仪器:凝胶成像分析系统和CFX Connect荧光定量PCR仪购于伯乐生命医学产品有限公司,VeritiTM PCR仪、高速冷冻离心机5810R和移液枪购于德国艾本德股份有限公司,核酸蛋白测定仪NanoDrop2000购于美国应用生物系统公司,三洋制冰机购于日本SANYO公司,微波炉购于广东美的集团有限公司。
1.2 试验方法1.2.1 罗非鱼tChit1a、tChi3和tChit基因cDNA的克隆 根据NCBI的尼罗罗非鱼基因组中3种预测的酸性几丁质酶序列tChit1a(XM_039611991.1)、tChi3(XM_005464320.4)和tChit(XM_003459030.3),采用Primer 5软件设计特异性引物(表 1)。

|
用丁香酚将罗非鱼麻醉,断头处死,取出胃、肠用DEPC水清洗,按照说明书用Trizol Reagent进行总RNA提取,用DNase Ⅰ去除基因组DNA,用反转录试剂盒反转录合成第一链cDNA,并以此为模板按照聚合酶Blend tap plus所要求的反应条件进行PCR扩增,所用引物分别为tChi1a-ORF-F/R、tChi3-ORF-F/R和tChit-ORF-F/R(表 1)。PCR反应程序均为:94 ℃ 3 min;94 ℃ 30 s、56 ℃ 30 s、72 ℃ 45 s,40个循环;72 ℃ 7 min;4 ℃保存。对PCR产物进行胶回收,加入pUCM-T载体和Ligation High室温孵育连接,然后通过热激法转化至大肠杆菌DH5α中,阳性克隆经菌落PCR鉴定后,送至生工生物工程(上海)公司进行测序。
1.2.2 罗非鱼tChit1a、tChi3和tChit基因序列的生物信息学分析 利用SignalP 5.0寻找几丁质酶tChit1a、tChi3和tChit基因的信号肽(http://www.cbs.dtu.dk/services/SignalP/)[11];利用ClustalW2软件进行氨基酸序列同源性分析(http://www.ebi.ac.uk/Tools/msa/clustalo/)[12]。通过NCBI在线网站进行氨基酸序列相似度分析(https://blast.ncbi.nlm.nih.gov/Blast.cgi)[13];采用MEGA6.0软件Neighbor-Joining法进行进化树分析[14]。
1.2.3 罗非鱼tChit1a、tChi3和tChit基因的mRNA组织分布 用丁香酚麻醉罗非鱼,断头处死,迅速取出端脑、中脑、小脑、延髓、脊髓、下丘、垂体、鳃、心脏、肾、脾、脂肪、肝、胃、前肠、中肠、后肠、白肌、红肌和性腺等组织,放入RNase-free的1.5 mL离心管中,立即放入液氮,之后置于-80 ℃冰箱中贮藏。按照1.2.1克隆步骤进行总RNA提取和反转录,以获得的第一链cDNA为模板,按照SYBR® Green说明书进行实时定量PCR,所用引物为tChit1a-qt-F/R、tChi3-qt-F/R和tChit-qt-F/R(表 1)。PCR反应程序为:95 ℃ 5 min;95 ℃ 15 s、56 ℃ 15 s、72 ℃ 30 s,40个循环;每个循环结束后在82 ℃收集荧光15 s;95 ℃ 10 s,然后以0.5 ℃/2 s的递增速度,65~95 ℃下进行温度梯度退火及收集荧光值。所测的值以18S rRNA为内参进行标准化,2-ΔΔCt法分析数据。
1.2.4 罗非鱼胃几丁质酶的纯化 用丁香酚麻醉罗非鱼,断头处死,取出其胃组织进行清洗,剪下内壁粘膜层置于PBS缓冲液中破碎(70 Hz/120 s),随后12 000 g离心20 min后收集上清用于纯化。用几丁质树脂制备重力柱,根据说明书清洗柱子,随后将样品过几丁质柱2次,缓冲液清洗杂蛋白,然后加入洗脱液,关闭出样口,4 ℃过夜。第2天依次收集滤液,并进行蛋白浓度测定和Western blot检测。
1.2.5 SDS-PAGE及Western blot验证 制备12% SDS聚丙烯酰胺凝胶,上样后80 V电泳30 min,120 V电泳60 min。电泳结束,其中一块PAGE胶用于G250染色,另一块PAGE胶则按照正极-海绵-滤纸-NC膜-PAGE胶-滤纸-海绵-负极的顺序夹紧,放在电泳槽中,装置在冰浴中恒流200 mA电泳60 min;电转完的NC膜用TBST缓冲液漂洗后浸入5% 脱脂奶粉中封闭30 min;用1 ︰ 4 000的斜带石斑鱼1型几丁质酶抗体稀释液孵育过夜,最后放入Goat anti-rabbit IgG按1 ︰ 5 000稀释液中孵育1 h,最后滴加ECL荧光混合液到膜上,在化学发光仪中显色并拍照记录。
1.2.6 酶活测定 采用4-甲基伞形酮(4-MU)荧光底物测定酶活。酶活定义为:在37 ℃下,每分钟释放1 μmol 4-MU所需要的酶量,用U表示。比活力用每克蛋白所含的酶活力单位(U/mg)数表示。1 μg待测样品、不同pH的PBS缓冲液和底物0.5 mmol/L的4-甲基伞形酮-β-D-N, N-二乙酰壳三糖〔4MU-(GlcNAc)2〕或4-甲基伞形酮-β-D-N, N, N-三乙酰壳三糖〔4MU-(GlcNAc)3〕,在37 ℃下孵育70 min,然后用0.5 mol/L Na2CO3缓冲液终止反应。用pH 7.4的PBS缓冲液配制梯度浓度0、0.1、0.2、0.4、0.6、0.8、1 nmol的4-MU标准样品,而待测样品是1 μg纯化产物与2.5 nmol的4MU-(GlcNAc)2/3,标准样品和待测样品的反应终体积均为100 μL,均在37 ℃、500 r/min反应70 min,最后在每孔加入100 μL的0.5 mol/L的Na2CO3溶液终止反应,用酶标仪在激发波长370 nm、发射波长448 nm下的荧光强度测定。
2 结果与分析 2.1 罗非鱼3种几丁质酶的cDNA及其编码的氨基酸序列从吉富罗非鱼胃中克隆得到tChit1a的ORF序列,全长1 362 bp,编码453个氨基酸(AA);Expasy在线软件预测蛋白大小为50.03 ku,等电点为6.14;SignalP在线软件预测结果显示,前19 AA为信号肽,紧接着为糖苷酶18家族催化域343 AA,然后为43 AA的连接区域,最后为48 AA的几丁质结合域(图 1)。

|
| 图 1 罗非鱼3种几丁质酶氨基酸序列比较 Fig. 1 Comparison of amino acid sequences of three chitinases of tilapia 相同的氨基酸用“*”表示,高度保守和低相似性的氨基酸分别用“:”和“.”表示,方框表示几丁质酶保守催化活性位点 The identical amino acids are represented by "*", and the highly conservative and less similar amino acids are represented by ": " and ".", and conservative catalytic activity site of chitinases is represented by the box |
从吉富罗非鱼前肠和中肠中克隆得到tChi3的ORF序列,全长1 362 bp,编码453 AA;Expasy在线软件预测蛋白大小为50.03 ku,等电点为5.39;SignalP在线软件预测结果显示,前19 AA为信号肽,紧接着为糖苷酶18家族催化域343 AA,然后是43 AA的连接区域,最后为48 AA的几丁质结合域(图 1)。
从吉富罗非鱼中肠中克隆得到tChit的ORF序列,大小为1 422 bp,编码473AA;Expasy在线软件预测蛋白大小为52.44 ku,等电点为5.68;SignalP在线软件预测结果显示,前20 AA为信号肽,紧接着为糖苷酶18家族催化域343 AA,然后为49 AA的连接区域,最后为61 AA的几丁质结合域(图 1)。
比对吉富罗非鱼3种几丁质酶序列,结果显示tChit1a与tChi3的氨基酸序列相似度为83.66%,而tChit与tChit1a、tChi3的相似度则较低,分别为49.89% 和50.11%。从结构(图 1)来看,3种几丁质酶结构相似,在序列N端具有信号肽和第18家族糖苷催化域,C端具有几丁质结合域,中间由连接域连接。其中,tChit1a和tChi3结构相似,几丁质酶催化活性位点序列一致,而tChit的连接域长于tChit1a和tChi3的连接域,有较多连续的苏氨酸(Thr),且在催化活性位点有2个氨基酸不一样。
2.2 罗非鱼3种几丁质酶的进行树分析本研究供试罗非鱼与其他物种的几丁质酶进化树分析结果(图 2)显示,罗非鱼3种几丁质酶既不属于哺乳动物的酸性几丁质酶和几丁三糖苷酶,也不属于鱼类酸性几丁质酶1和2,可归类为鱼类三型几丁质酶,即非酸性几丁质酶。
|
|
图 2 罗非鱼与其他物种的几丁质酶进化树分析 Fig. 2 Phylogenetic tree analysis of chitinases among tilapia and other species
罗非鱼tChit1a、tChi3和tChit几丁质酶归为FCase 3,位于方框中 The tChit1a, tChi3 and tChit, showed in the box, were classified into fish chitinases 3(FCase 3) 哺乳类酸性几丁质酶:Acidic mammalian chitinase(AMCase 1),鱼类酸性几丁质酶1:acidic fish chitinase 1(AFCase 1), 鱼类酸性几丁质酶2:acidic fish chitinase 2(AFCase 2),壳三糖酶:chitotriosidase 1. 普通牛Bos taurus chitin binding protein(AB051629.1), 2. 日本蟾蜍Bufo japonicus chitinase(AJ345054.1), 3. 斜带石斑鱼Epinephelus coioides chitinase 1(FJ169895.1), 4. 斜带石斑鱼Epinephelus coioides chitinase 2(FJ169894.1), 5. 家马Equus caballus chitotriosidase(EF694759.1), 6. 原鸡Gallus gallus chitin binding protein(AB071038.1), 7. 人Homo sapiens acidic mammalian chitinase(AF290004.1), 8. 人Homo sapiens chitotriosidase(U29615.1), 9. 矛尾鱼Latimeria chalumnae acidic chitinase(AB704869.1), 10. 日本叉牙七鳃鳗Lethenteron japonicum chitinase(EU741679.1), 11. 食蟹猕猴Macaca fascicularis acidic mammalian chitinase(FJ685619.1), 12. 条纹狼鲈Morone saxatilis chitinase(EU048546.1), 13. 小鼠Mus musculus acidic chitinase(EF094027.1), 14. 小鼠Mus musculus chitotriosidase(AY458654.1), 15. 虹鳟Oncorhynchus mykiss chitinase(EU877960.1), 16. 虹鳟Oncorhynchus mykiss chitinase(AJ535688.1), 17. 真鲷Pagrus major chitinase 2(AB678430.1), 18. Paralichthys olivaceus chitinase 1(AB121732), 19. 牙鲆Paralichthys olivaceus chitinase 2(AB121733.1), 20. 牙鲆Paralichthys olivaceus chitinase 3(AB121734.1), 21. 三线矶鲈Parapristipoma trilineatum chitinase 1(AB642677.1), 22. 三线矶鲈Parapristipoma trilineatum chitinase 2(AB642678.1), 23. 白姑鱼Pennahia argentata chitinase(AB605774.1), 24. 白姑鱼Pennahia argentata chitinase(AB605775.1), 25. Prionace glauca chitinase(AB872008.1), 26. Rattus norvegicus chitinase(AY486074.1), 27. 褐家鼠Rattus norvegicus chitotriosidase(DQ286232.1), 28. 斑点盖纹沙丁鱼Sardinops melanostictus chitinase 1(AB985610.1), 29. 斑点盖纹沙丁鱼Sardinops melanostictus chitinase 2(AB985611.1), 30. 白腹鲭Scomber japonicus chitinase 1(AB686657.1), 31. 白腹鲭Scomber japonicus chitinase 2(AB689022.1), 32. 褐菖鲉Sebastiscus marmoratus chitinase 1(AB686658.1), 33. 褐菖鲉Sebastiscus marmoratus chitinase 2(AB686659.1), 34. 太平洋蓝鳍金枪鱼Thunnus orientalis chitinase 3(AB678426.1), 35. 热带爪蟾Xenopus tropicalis chitotriosidase(BC075332.1), 36. 罗非鱼Oreochromis niloticus chitinase tChit1a, 37.罗非鱼Oreochromis niloticus chitinase tChi3, 38. 罗非鱼Oreochromis niloticus chitinase tChit |
2.3 3种几丁质酶在罗非鱼不同组织中的表达
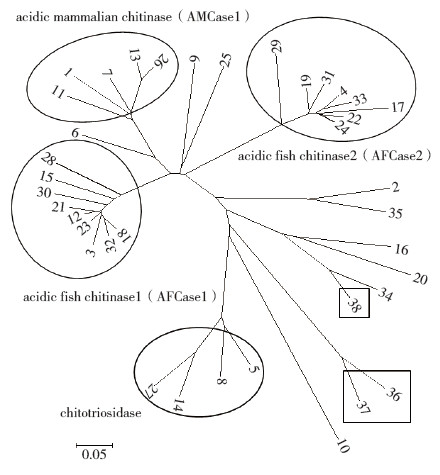
3种几丁质酶在罗非鱼不同组织中的表达结果显示,tChit1a在中肠的表达量最高,其次是前肠(图 3A);而tChi3在前肠的表达量最高,其次是中肠(图 3B);tChit主要在中肠后部表达(图 3C)。可见,罗非鱼3种几丁质酶均主要在肠道中表达,在其他组织中表达量非常低。
|
| 图 3 几丁质酶tChit1a、tChi3和tChit mRNA在罗非鱼不同组织中的分布 Fig. 3 Distribution of tChit1a, tChi3 and tChit in different tissues of tilapia |
2.4 罗非鱼胃几丁质酶的纯化和酶活测定
利用几丁质树脂纯化罗非鱼胃几丁质酶,SDS-PAGE胶在40 ku附近有两条明显条带,50 ku附近没有明显条带(图 4A)。本研究所克隆获得的3种几丁质酶tChit1a、tChi3和tChit预测的成熟肽大小分别为48.01、48.11、50.425 ku,但Western blot验证未在预测大小附近发现明显条带(图 4B)。用实验室前期制备的斜带石斑鱼1型几丁质酶多克隆抗体检测,同样没有检测到同源性高的条带。

|
| 图 4 罗非鱼胃几丁质酶的纯化结果 Fig. 4 Purification of chitinases extracted from the stomach of tilapia A:12% SDS-PAGE;B:Western blot,一抗:斜带石斑鱼1型几丁质酶多克隆抗体(1 ︰ 4 000)M:Marker;1:纯化样品;箭头指示克隆获得的几丁质酶预测大小位置 A: 12% SDS-PAGE; B: Western blot by using polyclonal anti-Chitinase 1 of Epinephelus lanceolatus(1 ︰ 4 000) M: Marker; 1: Purified product; Arrowhead showed the predicted protein of tChit1a, tChi3 and tChit |
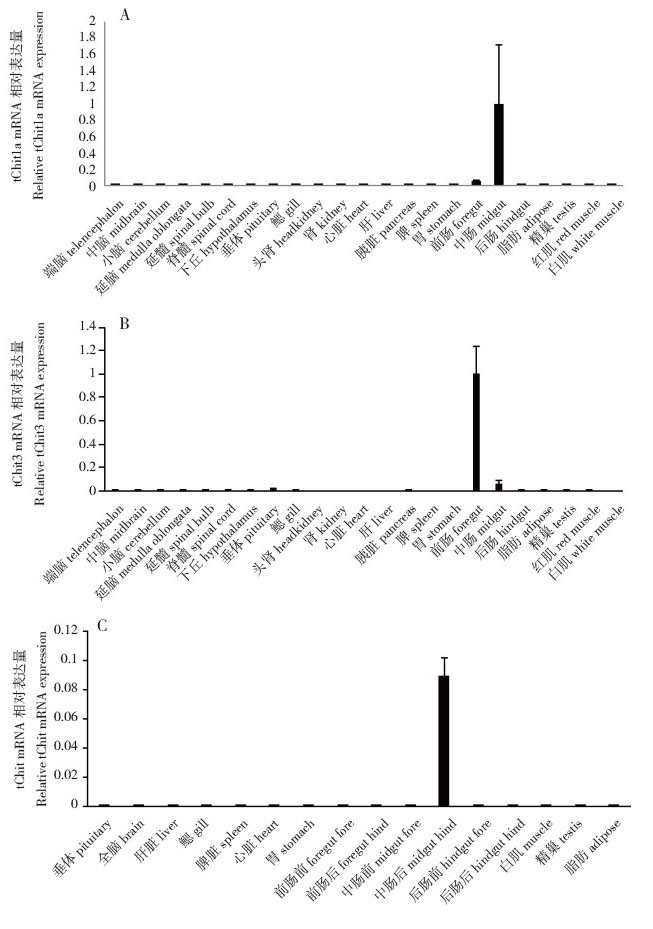
用4-MU法检测纯化产物的几丁质酶活性,结果如图 5所示,纯化产物的几丁质降解酶活性为弱酸性,在pH 4~7之间活性较高,最适pH值为5.0。1 μg纯化产物与2.5 nmol的4MU-(GlcNAc)2/3反应70 min,在最适pH值下,降解4MU-(GlcNAc)2的酶活为1.73 U/g,降解4MU-(GlcNAc)3的酶活为4.89 U/g。
|
| 图 5 罗非鱼胃几丁质酶活性测定结果 Fig. 5 Determination result of enzyme activity of chitinases extracted from the stomach of tilapia A:4-MU标准曲线;B:不同反应pH值与酶活性的关系 A: 4MU standard curve; B: The relationship between pH and enzyme activity |
3 讨论
本研究从吉富罗非鱼中克隆获得3种几丁质酶tChit1a、tChi3和tChit的ORF,这3种几丁质酶主要分布于中肠和前肠。tChit1a、tChi3和tChit结构相似,均属于18家族几丁质酶,预测的成熟肽大小分别为48.01、48.11、50.425 ku。其中tChit1a和tChi3的全长氨基酸序列相似性较高,达83.66%,催化活性位点序列一致,而tChit在该位点与它们则有两个氨基酸差异,表明tChit1a和tChi3亲缘关系更近。进化树分析表明,3种几丁质酶不属于鱼类酸性几丁质酶-1(AFCase-1)和鱼类酸性几丁质酶-2(AFCase-2),而与鱼类几丁质酶-3(FCase-3)亲缘关系更近。其中tChit与金枪鱼几丁质酶3、比目鱼几丁质酶3和虹鳟几丁质酶聚成一起,而tChit1a和tChi3则聚成另一分支,说明它们亲缘关系较近。为更有效降解食物中的几丁质,硬骨鱼鱼类胃中表达两种不同的几丁质酶,即AFCase-1和AFCase-2,它们具有不同的降解模式,AFCase-1优先水解GlcNAc链的非还原端的第二糖苷键,而AFCase-2优先水解第三糖苷键[15]。但关于FCase-3的研究较少。在褐菖鲉肝脏和肾脏表达的一种几丁质酶属于FCase-3[16]。有关FCase-3的降解特性还没有报道。此外,罗非鱼3种几丁质酶的等电点均在5~6.5间,略小于7.0,说明这3种酶均偏中性,推测它们发挥催化作用的环境pH也偏中性,具体功能需要进一步探索。此外,为继续探索罗非鱼酸性几丁质酶,本研究分析了来源于NCBI的尼罗罗非鱼基因组注释的另外12种罗非鱼几丁质酶,包括XM_003440467.5、XM_003459039.4、XM_013266354.2、XM_019349756.2、XM_025901892.1、XM_025901891.1、XM_025901890.1、XM_019349750.2、XM_019349749.2、XM_005448561.3、XM_019355318.2和XM_003459030.3,进化分析结果显示,它们不属于AFCase-1和AFCase-2,均归类为FCase-3。
研究表明,tChit、tChit1a和tChi3主要在罗非鱼前肠和中肠表达,在胃中的表达量相对较低。其他物种研究表明,大菱鲆的3种几丁质酶在肠道高表达,其次是脑、皮肤和脾脏[17];而日本沙丁鱼几丁质酶的高表达组织是性腺[15];褐菖鲉几丁质酶主要在肝和肾脏中表达[16]。不同的几丁质酶同工酶可能发挥不同的作用,包括免疫、消化和蜕皮等[4, 18]。本研究中,tChit、tChit1a和tChi3几丁质酶均在肠道高表达,因此,推测这3种几丁质酶可能主要将上游进行了部分分解的几丁质寡糖再进一步水解,从而帮助机体消化吸收几丁质。
为了解罗非鱼是否存在高活性的酸性几丁质酶,本研究通过纯化胃蛋白以期获得酸性几丁质酶。已有多种具有活性的几丁质酶在鱼类胃中分离出来,且有多种同工酶,例如,在白姑鱼的胃组织中纯化获得两种大小分别为42、56 ku的几丁质酶[19-20];在褐菖鲉胃中发现3种大小分别为46、52、56 ku的几丁质酶[21];在日本沙丁鱼胃中纯化得到大小为45、56 ku的几丁质酶[15]。在六线鱼胃组织中纯化出47、51、62 ku等3种几丁质酶[22]。这些纯化获得的几丁质酶具有较高的酶活性,能够降解pNP-(GlcNAc)n,推测几丁质酶活性与这些鱼类栖息地和食物来源有关,它们均以海洋富含几丁质的贝类、甲壳类或浮游生物为食,胃组织中高活性的几丁质酶有助于它们消化食物中的几丁质。几丁质酶存在于许多鱼类的胃中,但并不局限于胃组织,在其他组织中同样发现几丁质酶的表达和酶活性,例如在褐菖鲉肾脏中发现几丁质酶mRNA的表达[16],在白姑鱼肾脏检测到几丁质酶活性[23],在日本鲭鱼幽门盲囊中检测到几丁质酶mRNA[23]。但还未有研究展示胃以外组织的纯化几丁质酶结果。
在前期研究中,我们成功纯化获得鞍带石斑鱼和赤点石斑鱼胃组织中的几丁质酶,并通过了斜带石斑鱼1型几丁质酶多克隆抗体的Western blot验证。本研究中,我们首先从吉富罗非鱼的胃组织中提取总蛋白,通过几丁质树脂亲和层析法分离纯化几丁质酶,SDS-PAGE结果显示,我们只获得约40 ku的两条条带,且未能用多克隆抗体检测到这两条条带。我们使用了较DNS法[24]更为灵敏的4-MU法分析纯化产物酶活性,结果显示,在最适pH 5的条件下,纯化产物降解4MU-(GlcNAc)2的酶活为1.73 U/g,降解4MU-(GlcNAc)3的酶活为4.89 U/g。以4-硝基苯基-β-D-乙酰氨基葡萄糖〔pNP-(GlcNAc)n〕为底物,从六线鱼胃组织纯化获得3种几丁质酶的最佳酶活分别为3.04、1.25、2.37 U/mg[22],从白姑鱼胃中纯化获得的两种几丁质酶降解pNP-(GlcNAc)3分别为0.109、2.07 U/mg[15]。本研究的罗非鱼纯化产物明显低于这两种鱼的胃几丁质酶。而检测真鲷、海鲈鱼、六线鱼、太平洋鳕鱼和黄尾鰤等胃组织粗酶液的几丁质酶活性在0.24~1.59 U/g之间[9],与本研究获得的纯化产物几丁质酶活性接近。推测罗非鱼胃中的几丁质酶蛋白表达量较低,通过几丁质树脂亲和层析法并不能很好地纯化几丁质酶,后续可能需要用多种纯化方法相结合,如在现基础上再结合阴/阳离子交换层析方法等,以进一步分离天然的几丁质酶,探究它们对不同形式完整几丁质底物的降解特性及反应机制。几丁质在海洋贮藏量非常丰富[25],其中包括大量虾蟹壳。在处理虾蟹壳废料方面,除了鱼类几丁质酶潜在的降解虾蟹壳几丁质的作用外,细菌几丁质降解酶也是重要的潜在候选者。对虾酱中筛选分离出来的细菌进行全基因组测序,灰色链霉菌CS1801包含103个糖苷水解酶家族基因,体现了几丁质的降解潜力[26]。在海鲜工业废物(虾壳倾倒区)分离出的地衣芽孢杆菌(SSCL-10)的最高比活度为3.4 U/mL。对菌株进行化学诱变并通过紫外线照射来筛选潜在的几丁质分解菌群,诱变模型已成功使突变型产生的几丁质酶产量提高了5~6倍[27]。
4 结论本研究发现,不同于以贝类、甲壳类或者浮游生物为食的鱼类,罗非鱼3种几丁质酶tChit1a、tChi3和tChit主要在前肠或中肠中高表达,而在胃中的表达量很低。推测主要原因为罗非鱼是栖息在水体中下层的鱼类,是以植物性饵料为主的杂食性鱼类,其食物中的虾蟹壳所占比例较小,这种食性影响到罗非鱼几丁质酶的表达。因此,这3种在罗非鱼肠道不同位置表达的几丁质酶可能不同程度地参与罗非鱼的消化吸收,但降解几丁质可能不是其主要功能。
| [1] |
陈西广, 刘万顺, 刘晨光. 几丁质的研究与进展[J]. 中国生物工程杂志, 1997, 17(3): 4-8. DOI:10.13523/j.cb.19970302 CHEN X G, LIU W S, LIU C G. The research and progress of chitin[J]. Progress in Biotechnology, 1997, 17(3): 4-8. DOI:10.13523/j.cb.19970302 |
| [2] |
HENRISSAT B, BAIROCH A. New families in the classification of glycosyl hydrolases based on amino acid sequence similarities[J]. Biochemical Journal, 1993, 293(3): 781-788. DOI:10.1042/bj2930781 |
| [3] |
ADRANGI S, FARAMARZI M A. From bacteria to human: A journey into the world of chitinases[J]. Biotechnology Advances, 2013, 31(8): 1786-1795. DOI:10.1016/j.biotechadv.2013.09.012 |
| [4] |
IKEDA M, KAKIZAKI H, MATSUMIYA M. Biochemistry of fish stomach chitinase[J]. International Journal of Biological Mcromolecules, 2017, 104: 1672-1681. DOI:10.1016/j.ijbiomac.2017.03.118 |
| [5] |
FUOCHI V, LI VOLTI G, CAMIOLO G, TIRALONGO F, GIALLONGO C, DISTEFANO A, PETRONIO PETRONIO G, BARBAGALLO I, VIOLA M, FURNERI PM, DI ROSA M, AVOLA R, TIBULLO D. Antimicrobial and anti-proliferative effects of skin mucus derived from Dasyatis pastinaca(Linnaeus, 1758)[J]. Marine Drugs, 2017, 15(11): 1. DOI:10.3390/md15110342 |
| [6] |
SERA H, OKUTANI K. Studies on chitinolytic enzyme present in aquatic animals: Ⅶ. The chitinolytic enzyme present in a sea bream, Acanthopagrus schlegeli[J]. Bulletin of the Japanese Society for the Science of Fish, 1968, 34: 920-924. DOI:10.1007/978-4-431-65907-5_30 |
| [7] |
KUROKAWA T, UJI S, SUZUKI T. Molecular cloning of multiple chitinase genes in Japanese flounder, Paralichthys olivaceus[J]. Comparative Biochemistry and Physiology, 2004, 138(3): 255-264. DOI:10.1016/j.cbpc.2004.03.015 |
| [8] |
JOLLÈS, P, MUZZARELLI R. Native, industrial and fossil chitins[J]. Exs, 1999, 87: 1-6. |
| [9] |
MATSUMIYA M, MOCHIZUKI A. Distribution of chitinase and β-N-Acetylhexosaminidase in the organs of several fishes[J]. Fisheries Science, 1996, 62(1): 150-151. DOI:10.2331/fishsci.62.150 |
| [10] |
MOLINARI LM, PEDROSO RB, SCOARIS DDE O, UEDA-NAKAMURA T, NAKAMURA CV, DIAS FILHO BP. Identification and partial characterisation of a chitinase from Nile tilapia, Oreochromis niloticus[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2007, 146(1): 81-87. DOI:10.1016/j.cbpb.2006.09.004 |
| [11] |
ALMAGRO ARMENTEROS JJ, TSIRIGOS KD, SØNDERBY CK, PETERSEN TN, WINTHER O, BRUNAK S, HEIJNE G, NIELSEN H. SignalP 5.0 improves signal peptide predictions using deep neural networks[J]. Nature Biotechnology, 2019, 37(1): 420-423. DOI:10.1038/s41587-019-0036-z |
| [12] |
LARKIN MA, BLACKSHIELDS G, BROWN NP, CHENNA R, MCGETTIGAN PA, MCWILLIAM H, VALENTIN F, WALLACE IM, WILM A, LOPEZ R, THOMPSON JD, GIBSON TJ, HIGGINS DG. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(1): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
| [13] |
JOHNSON M, ZARETSKAYA I, RAYTSELIS Y, MEREZHUK Y, MCGINNIS S, MADDEN TL. NCBIBLAST: a better web interface[J]. Nucleic Acids Research, 2008, 36(S): 5-9. DOI:10.1093/nar/gkn201 |
| [14] |
TAMURA K, STECHER G, PETERSON D, FILIPSKI A, KUMAR S. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(1): 2725-2729. DOI:10.1093/molbev/mst197 |
| [15] |
KAWASHIMA S, IKEHATA H, TADA C, OGINO T, KAKIZAKI H, IKEDA M, FUKUSHIMA H, MATSUMIYA M. Stomach Chitinase from Japanese Sardine Sardinops melanostictus: Purification, characterization, and molecular cloning of chitinase isozymes with a long linker[J]. Marine Drugs, 2016, 14(1): 22. DOI:10.3390/md14010022 |
| [16] |
WATANABE M, KAKIZAKI H, TSUKAMOTO T, FUJIWARA M, FUKUSHIMA H, UEDA M, MATSUMIYA M. Distribution of chitinolytic enzyme in the organs and molecular cloning of a novel chitinase gene from the kidney of marbled rockfish Sebastiscus marmoratus[J]. Advances in Bioscience and Biotechnology, 2018, 9(1): 36-51. DOI:10.4236/abb.2018.91004 |
| [17] |
GAO C, CAI X, ZHANG Y, SU B, SONG H, WENQI W, LI C. Characterization and expression analysis of chitinase genes(CHIT1, CHIT2 and CHIT3)in turbot(Scophthalmus maximus L.)following bacterial challenge[J]. Fish and Shellfish Immunology, 2017, 64: 357. DOI:10.1016/j.fsi.2017.03.019 |
| [18] |
缪承杜, 赵培静, 夏枫耿, 吴翠玲. 一株产几丁质酶芽孢杆菌分离鉴定及产酶活性初步研究[J]. 广东农业科学, 2013, 40(24): 124-127. DOI:10.16768/j.issn.1004-874X.2013.24.017 MIU C D, ZHAO P J, XIA F G, WU C L. Study on the identification and activity of chitinase produced by Brevibacillus laterosporus[J]. Guangdong Agricultural Sciences, 2013, 40(24): 124-127. DOI:10.16768/j.issn.1004-874X.2013.24.017 |
| [19] |
IKEDA M, MIYAUCHI K, MATSUMIYA M. Purification and characterization of chitinase from the stomach of silver croaker Pennahia argentatus[J]. Bioscience Biotechnology & Biochemistry, 2009, 76(5): 971-979. DOI:10.1016/j.pep.2009.01.015 |
| [20] |
IKEDA M, MIYAUCHI K, MATSUMIYA M. Purification and characterization of a 56 ku chitinase isozyme(PaChiB)from the stomach of the silver croaker, Pennahia argentatus[J]. Bioscience, Biotechnology, and Biochemistry, 2012, 65(2): 214-222. DOI:10.1271/bbb.110989 |
| [21] |
IKEDA M, SHIRASE D, SATO T, UEDA M, HIRABAYASHI S, MATSUMIYA M. Primary structure and enzymatic properties of chitinase isozymes purified from the stomach of the marbled rockfish Sebastiscus marmoratus[J]. Journal of Chitin and Chitosan Science, 2014, 2(2): 106-116. DOI:10.1166/jcc.2014.1048 |
| [22] |
MATSUMIYA M, ARAKANE Y, HAGA A, MUTHUKRISHNAN S, KRAMER KJ. Substrate specificity of chitinases from two species of fish, greenling, Hexagrammos otakii, and common mackerel, Scomber japonicus, and the insect, tobacco hornworm, Manduca sexta[J]. Bioscience Biotechnology & Biochemistry, 2006, 70(4): 971-979. DOI:10.1271/bbb.70.971 |
| [23] |
KAKIZAKI H, IKEDA M, FUKUSHIMA H, MATSUMIYA M. Distribution of chitinolytic enzymes in the organs and cdna cloning of chitinase isozymes from the stomach of two species of fish, chub mackerel(Scomber japonicus)and silver croaker(Pennahia argentata)and Silver Croaker[J]. Open Journal of Marine Science, 2015, 5(4): 398-411. DOI:10.4236/ojms.2015.54032 |
| [24] |
卓侃, 李玉中, 廖金铃. 淡紫拟青霉MD1几丁质酶活性测定[J]. 广东农业科学, 2009(5): 167-170. DOI:10.16768/j.issn.1004-874X.2009.05.019 ZHUO K, LI Y Z, LIAO J L. Determination of chitinase activity of Paecilomyces lilacinus MD1[J]. Guangdong Agricultural Sciences, 2009(5): 167-170. DOI:10.16768/j.issn.1004-874X.2009.05.019 |
| [25] |
戴世鲲, 王广华, 李涛, 吴华莲, 向文洲. 南海北部巴士海峡深海沉积物中几丁质酶基因多样性分析[J]. 广东农业科学, 2015, 42(13): 140-145. DOI: 10.16768/j.issn.1004-874X.2015.13.005. DAI S K, WANG G H, LI T, WU H L. Phylogenetic diversity analysis of chitinase gene in deep-sea sediments from the Bashi Channel in the northern South China Sea[J]. Guangdong Agricultural Sciences, 2015, 42(13): 140-145. DOI: 10.16768/j.issn.1004-874X.2015.13.005. |
| [26] |
XU T, QI M, LIU H, CAO D, QI B. Chitin degradation potential and whole-genome sequence of Streptomyces diastaticus strain CS1801[J]. AMB Express, 2020, 10: 29. DOI:10.1186/s13568-020-0963-6 |
| [27] |
SASI A, DURAIPANDIYAN N, MARIKANI K, DHANASEKARAN S, VENUGOPAL D. Identification and characterization of a newly isolated chitinase-producing strain Bacillus licheniformis SSCL-10 for chitin degradation[J]. Archaea, 2020, 2020(8844811): 9. DOI:10.1155/2020/8844811 |
 2021, Vol. 48
2021, Vol. 48