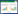文章信息
基金项目
- 国家自然科学基金青年基金(31701895)
作者简介
- 张小乙(2001—),女,在读本科生,研究方向为植物生长发育基因功能鉴定,E-mail: 869619338@qq.com.
通讯作者
- 翟璐璐(1986—),女,博士,副教授,研究方向为园艺植物生长发育与次生代谢,E-mail: zhailulu@jlu.edu.cn.
文章历史
- 收稿日期:2021-05-29
【研究意义】种子不仅是植物生命的起始,也是人类赖以生存的最基本食物及禽畜饲料的来源。高等植物的种子发育过程可分为两个主要阶段:胚和胚乳的形态建成以及种子成熟[1],其中种子成熟是一个特异、复杂的发育过程,起始于心型胚的形成,经历胚的生长与膨大填充、贮藏物质(主要包括贮藏蛋白、脂类和碳水化合物等)及ABA的积累、种子脱水与休眠等过程,终止于种子干燥期结束。种子成熟是种子发育的重要阶段,不仅是决定种子质量和产量的重要时期,还是影响种子贮藏和质量保存的关键环节,因此,越来越多的科研人员将目光投向植物种子成熟的相关研究领域。尽管人们对种子成熟调控网络有一定认识,但全面揭示这种调控网络还需挖掘更多的关键因子。
【前人研究进展】miRNAs是一类广泛存在于真核生物中的内源性、非编码、小分子RNA,其长度为19~24 nt。miRNA于1994年在秀丽隐杆线虫中首次被发现[2]。此后,陆续有真核生物的miRNA被鉴定出。2002年,首个植物中的miRNA在拟南芥中得到鉴定[3-4]此后,关于植物miRNAs的研究开始得到广泛关注。随着近年来高通量测序技术的不断发展与广泛应用,越来越多来自不同物种、组织的miRNA被陆续鉴定和分析,目前为止,已从80余种植物中鉴定出上万个miRNA(miRBase数据库,http://www. mirbase.org/)。相关研究已经证实,miRNA能在转录、转录后及翻译水平上负向调控其靶标基因的表达,从而参与植物生长发育各方面,包括植物器官发育[5]、生理状态转化[6]、植物激素调控[7]、营养平衡[8]以及胁迫响应[9]等,而靶标基因中不乏大量转录因子,如SPL(SQUAMOSA Promoter-Binding Protein-Like)[10]、MYB[11]、NAC[12]、ARF[13]、HD-Zip[14]和AP2[15]等,这也揭示了miRNA在基因调控网络中的核心地位。miR156是高度保守的家族之一[16],最初发现于拟南芥[17],通过调控SBP/SPL基因参与植物的生长发育过程[18-19],如种子萌发[20]、叶片发育形态建成、花期调控[21]、生殖器官发育[22]以及次生代谢物花青苷合成等[23]。Nodine和Bartel在DCL1(DICER-LIKE1)突变体中,证实miR156可在早期胚的分化阶段通过调控SPL10和SPL11的表达阻止分化、促进转录因子的提前表达,从而实现胚的正常发育[24]。其中,早期研究更多关注种子发育过程中胚的形态建成部分,对于种子成熟方面的相关报道较为有限。Huang等[25]利用高通量测序技术分析发现,miR156家族在种子和成熟胚中的表达量最高,其中多数成员在胚发育过程中表达,且表达量随种子成熟而呈稳定上升趋势,是控制油菜种子发育和成熟的主要因子。随后,Chao等[26]分析了miR156在蝴蝶兰叶、根、花、种子等不同组织部位中的表达情况,同样发现miR156在种子中具有较高的表达量。另有研究发现,miR156a在胡麻种子发育后期表达量显著升高,并影响种子含油率[27]。目前,多数相关研究集中在基因鉴定和表达模式层面,关于植物种子成熟过程中miR156的调控机制尚不完整,有待进一步研究。
【本研究切入点】植物种子成熟是一个复杂的发育过程,各环节伴随大量基因选择性的激活和抑制,目前该过程的调控机制着力于转录因子、功能蛋白、表观遗传等类别调控因子的研究,miRNA特别是miR156在这一生命过程中的作用及调控机制尚不清晰。【拟解决的关键问题】为探究miR156及其靶标基因在植物种子发育过程中尤其是成熟阶段的调控功能,本研究选用miR156下调突变体MIM156及SPL过表达突变体材料,利用本实验室建立的β- 葡萄糖苷酸酶(GUS)蛋白构建大豆种子贮藏蛋白基因(Conglycinin)的报告基因检测体系,对miR156及其靶标基因是否参与种子成熟调控进行探究与分析,以期从种子贮藏蛋白积累层面为植物种子成熟调控机制提供理论依据与研究思路。
1 材料与方法 1.1 试验材料本研究使用的拟南芥突变体种子包括miR156基因家族的下调表达突变体MIM156,SPL10过表达突变体CS66330、CS66332以及SPL11过表达突变体CS66334、CS66335的种子均购买自ABRC种子库。其中,下调突变体MIM156通过转入人工合成的miR156相似序列片段获得,该片段能与内源miR156发生竞争性抑制反应,进而间接导致内源miR156表达水平降低。SPL2ox-1和SPL2ox-2为本研究通过蘸花侵染法将SPL2基因成功转入gus23拟南芥中纯合的过表达株系。gus23由加拿大农业部伦敦研究与开发中心崔玉海研究员实验室惠赠。WT即拟南芥(Arabidopsis thaliana)Columbia型。
将野生型拟南芥WT及各种拟南芥突变体的种子在4 ℃黑暗环境下春化3 d,播于蛭石与草炭以1 ∶ 3比例混合的湿润基质中,随后于22 ℃、16 h/8 h(光照/黑暗)条件下培养。
1.2 试验方法1.2.1 拟南芥不同组织部位RNA提取与cDNA获得 采用OminiPlant RNA Kit(Dnase I)全能型植物RNA提取试剂盒对所采集拟南芥播种后生长14 d的幼苗、莲座叶、花和果荚等组织进行总RNA的提取,详细步骤参考试剂盒说明书。反转录详细步骤参照PrimeScript ™ RT reagent Kitwith gDNA Eraser(Perfect Real Time)(TaKaRaBio.,Code No. RR047A)试剂盒说明书。
1.2.2 gus23背景下突变体材料获取 分别将突变体(MIM156、CS66330、CS66332、CS66334和CS66335)与gus23的种子进行春化、消毒并播种,待开花后选择长势好、花蕾茂盛的植株,除去早期几个花蕾后,即可以拟南芥突变体为父本、gus23为母本进行杂交。杂交时间选择在7:00—8:00最佳,此时花粉活力最强,大大增加杂交的成功率。所获得的种子经草铵膦筛选并进行阳性植株的PCR验证,最终筛选出纯合体用于GUS染色试验。
1.2.3 GUS染色分析 将上述杂交后代的种子播种于添加草铵膦的MS培养基中,以gus23为对照播种到MS培养皿中,春化2~3 d,放入培养室中培养并分别收集14 d的幼苗,参照Tang等[28]方法染色,具体操作如下:将去根的幼苗放入配好的X-Glucsolution染色液中,真空抽气10~15 min后置于37 ℃条件下染色16~20 h,随后利用75% 的乙醇37 ℃脱色1 d。最后,利用体式显微镜观察并拍照。
1.2.4 基因表达分析 分别在TAIR网站( www.arabidopsis.org)和miRBase数据库中获取LEC1、ABI3、FUS3以及miR156a-j的序列,利用PrimerSelect和EditSeq软件设计荧光定量引物(表 1),并用NCBI Primer Blast进行引物的特异性比对。以拟南芥不同组织的cDNA为模板,采用SYBR Premix Ex Taq(Tli Rnase H Plus)(TaKaRaBio.,Code No. RR420A)进行实时荧光定量(qRT-PCR)测定不同基因的相对表达量,应用2-ΔΔCt方法进行分析。每个样品进行3次生物学重复,3次技术重复。
1.2.5 AtmiR156靶标基因的预测与分析 使用psRNA Target程序(http://plantgrn.noble.org/psRNA Target/)对TAIR数据库中拟南芥的转录本进行miRNA靶标预测。psRNA Target参数设置为默认值。从TAIR网站中获取SPL基因家族成员在野生型拟南芥不同发育时期的胚组织中的表达水平相关信息及基因序列信息,分别运用GraphPad Prism 6和MEGA5.02软件对SPL基因家族成员进行表达水平与进化树分析。
2 结果与分析 2.1 miR156可能参与种子成熟调控过程为在种子贮藏蛋白积累层面探究miR156是否参与调控种子成熟过程,以来自加拿大农业部伦敦研究与开发中心崔玉海研究员实验室的拟南芥gus23突变体(βCG:GUS的异位表达)为母本,与miR156下调突变体MIM156进行杂交,利用获得的杂交后代进一步GUS染色。gus23突变体gus23具有β- 葡萄糖苷酸酶(GUS)蛋白构建大豆种子贮藏蛋白基因(SSPs,Seed Storage Proteins)的报告基因体系,正常情况下GUS基因只在种子中表达,而在幼苗中有一些种子成熟抑制因子存在,所以GUS不能表达。当幼苗中的种子成熟抑制因子功能缺失或正向的种子成熟调控基因异位表达后,幼苗叶片便会具有GUS表型。如图 1A所示,以14 d苗龄的拟南芥gus23突变体为对照,经X-Glucsolution染色后,所有MIM156-gus23株系中,幼苗真叶呈现出GUS表型,说明种子贮藏蛋白基因在此叶片中得到了异位表达,而调控该基因表达的极有可能就是miR156。由此可推测,miR156负向调控种子贮藏蛋白基因的表达。
|
| 图 1 MIM156-gus23的GUS表型分析及种子成熟关键调控基因的表达分析 Fig. 1 Analysis of GUS phenotype in MIM156-gus23 and expression of key genes during seed maturation |
根据前人报道,LEC1、ABI3和FUS3是调控种子成熟的关键因子,在种子成熟过程中起至关重要的作用。为进一步验证miR156对种子成熟过程的调控,本研究选取开花后7 d的拟南芥果荚材料,采用(qRT-PCR)技术对基因LEC1、ABI3和FUS3进行表达量的测定,此时期果荚内的种子中各种贮藏物质开始合成。结果表明,以野生型拟南芥WT为对照,MIM156下调表达突变体中LEC1、ABI3和FUS3的表达水平均有不同程度升高,且与WT存在极显著差异(图 1B),说明miR156确实可以调控种子成熟过程。
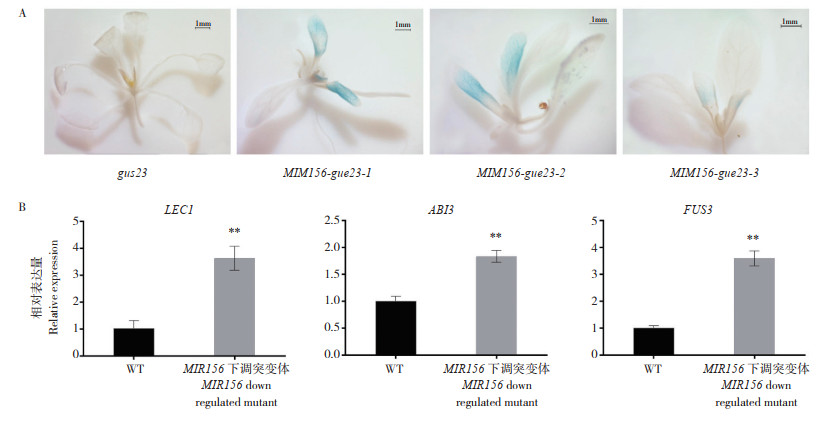
2.2 影响种子发育的miR156家族成员的鉴定拟南芥miR156基因家族共有10个成员,分别为miR156a~j,为确定是哪个成员对拟南芥种子发育产生影响、探明这些miR156在不同组中发挥的作用,本研究收集野生型拟南芥4个组织材料,分别为播种后14d的幼苗、果荚、花和莲座叶,并采用实时荧光定量技术对不同miR156家族成员进行组织表达特异性分析,结果见图 2。以14d幼苗的表达量为参照,miR156a、miR156d、miR156e和miR156j在果荚中的表达量最高,最低为幼苗表达量的20倍,最高可达7 000倍;miR156b、miR156h、miR156i在花中表达量最高,分别约为幼苗表达量的2.5、301.1和25.9倍,而miR156c则是在幼苗中的表达量最高;此外,没有检测到miR156f和miR156g。上述结果表明,miR156家族中大部分成员在拟南芥的果荚和花中有较高表达,特别是果荚中,miR156可能调控拟南芥种子的发育过程,此外miR156c在幼苗期有较高表达,说明其可能抑制与种子发育相关基因的表达。
|
| 图 2 拟南芥miR156基因家族在不同组织部位中的相对表达量分析 Fig. 2 Relative expression analysis of miR156 gene family in different tissues of Arabidopsis |
2.3 miR156靶标基因的生物信息学分析
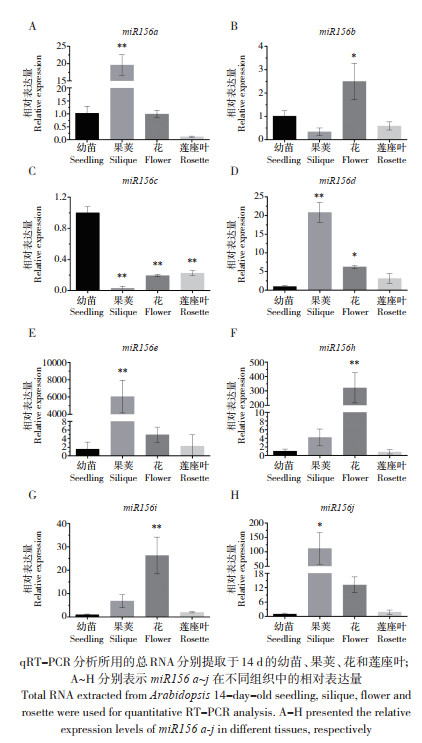
为进一步研究miR156家族的生物学功能,本试验采用psRNA Target程序对TAIR数据库中拟南芥的转录本进行miR156靶标基因的预测,共鉴定到10个SPL基因(图 3A),分别为SPL2、SPL3、SPL4、SPL5、SPL9、SPL10、SPL11、SPL13(A和B)和SPL15。从图 3A可以看出,miR156与上述SPL基因的序列能够较为完美的匹配,仅个别碱基存在错配现象,非常符合植物中miRNA与其靶标基因的序列匹配原则。
|
| 图 3 拟南芥miR156靶基因的预测、表达模式和系统发育分析 Fig. 3 Analysis of miR156 target genes on prediction, expression patterns and phylogenetics in Arabidopsis |
利用TAIR网站查询上述10个SPL基因在野生型拟南芥不同胚胎发育时期的表达量水平,分析结果见图 3B,自胚的八分体时期起发育至完全成熟绿胚的8个阶段中,大部分SPL基因能够展现不同程度的特异性表达,其中,SPL2、SPL10和SPL11的表达量较高,尤其是SPL2和SPL10。在八分体时期,SPL10的表达水平最高、达4.36,为同一发育时期SPL13A表达量的62倍,其次是SPL2、表达量为3.97(图 3B)。在球型胚、早期心型胚、晚期心型胚和早期鱼雷型胚时期,SPL2的表达量均最高,随后的胚发育过程中,其表达量逐渐降低。在胚发育整个过程中,SPL3、SPL4、SPL5、SPL9和SPL15的表达量极低甚至在前期SPL4根本不表达。综上,miR156可能通过靶标SPL2、SPL10和SPL11调控种子的发育过程。
为研究miR156靶标基因间的进化关系,本研究进行了10个SPL基因的系统进化树分析,结果表明,与表达模式相似,SPL3/4/5、SPL2/10/11以及SPL9/15分别被划分到三组,如图 3C,在胚发育时期具有较高表达量的SPL2、SPL10和SPL11的亲缘关系最近,在进化上最为相似,说明它们可能发挥相同的生物调控作用。
2.4 miR156介导SPL2、SPL10和SPL11调控种子成熟过程鉴于SPL2、SPL10和SPL11在种子胚发育过程中具有较高的表达量,且亲源关系很近,我们将这3个SPL基因作为miR156靶标基因重点探究其对植物种子发育的影响。本研究对SPL2进行克隆并利用农杆菌介导的遗传转化法创制了gus23背景下的SPL2ox过表达突变体材料,从中挑选2个株系进行GUS染色分析,经染色后发现SPL2-ox-1和SPL2-ox-2的子叶呈蓝色(图 4)。将CS66330、CS66332、CS66334和CS66335与gus23的杂交后代分别进行染色,结果显示,所有杂交后代均能显现出GUS表型,但组织部位略有差异,其中,CS66330和CS66335在新发叶上有蓝色GUS表型,CS66332的GUS表型集中于真叶上,CS66334则在子叶和新发叶上均表现异位的GUS活性(图 4)。种子贮藏蛋白基因在幼苗中得到表达,即SPL2、SPL10和SPL11基因的过表达影响种子贮藏蛋白基因的表达。

|
| 图 4 拟南芥AtSPL2ox、AtSPL10ox-gus23和AtSPL11ox-gus23突变体中GUS表型分析 Fig. 4 GUS phenotypic analysis of AtSPL2ox, AtSPL10oxgus23 and AtSPL11ox-gus23 mutants of Arabidopsis |
SPL2、SPL10和SPL11作为miR156的靶标基因,受miR156的切割而与miR156呈负调控关系。miR156下调表达突变体MIM156以及靶标基因SPL2、SPL10和SPL11的GUS染色结果显示,miR156的下调表达及上述SPLs的过表达均可影响种子贮藏蛋白基因的表达,由此形成了一条miR156调控种子成熟的调控途径,即拟南芥miR156通过调控SPL2、SPL10和SPL11影响种子贮藏蛋白的表达从而影响种子成熟过程。
3 讨论种子是植物生长发育的基础,也是重要的农业资源,目前我国种业发展处于瓶颈期,挖掘种子发育和成熟过程中的关键基因及其调控机制具有重要意义。大量研究报道表明,miR156作为保守的miRNA家族,在植物种子的胚形态建成、休眠和萌发等生物过程中发挥了重要作用,而其在植物种子成熟过程中的调控机制未见报道,为此,本研究选用拟南芥miR156下调突变体MIM156为父本与种子贮藏蛋白基因启动子驱动GUS报告基因的检测体系(gus23)进行杂交,以其杂交后代14 d幼苗为试验材料进行GUS表型分析。如miR156参与种子成熟的正向调控,则在gus23检测体系中过表达时,可能促使GUS和SSPs在幼苗中异位表达;如果参与种子成熟的负向调控,则在gus23检测体系中功能缺失时会表现出相似结果。本研究结果中,MIM156是miR156的下调表达突变体,且在叶片中表现出蓝色GUS活性,证明了miR156的表达量降低能够促进SSPs基因的异位表达,从而负向参与种子的成熟过程。已知调控拟南芥种子发育的3个重要因子为LEC1、ABI3和FUS3,其在种子发育的许多阶段起重要作用。关于LEC1、ABI3和FUS3调控拟南芥种子发育已有相关报道详细阐述其可能的调控网络[29-30],然而,对于该调控网络的激活因子尚不明确。分析qRT-PCR结果发现,此3个基因在果荚中表达量显著上调,这为miR156作为LEC1、ABI3和FUS3调控拟南芥种子发育的激活因子提供了有力证据。
基因家族各成员在不同组织中表达量差异的测定,常用来分析基因家族在某一生物过程中的主效基因,近年来应用十分广泛。最新研究通过分析鹰嘴豆各组织中miR156各成员表达量差异,进而推断miR156基因家族的表达模式的相关论述[31]。本研究通过对拟南芥播种后14 d的幼苗、果荚、花和莲座叶中miR156家族个成员的表达量分析,发现miR156a、miR156d、miR156e和miR156j在果荚中的表达量最高,其中最高的miR156e,其表达量为在幼苗中的7 000倍,充分证明miR156e在拟南芥种子发育过程中起主要调控作用。同时发现miR156c在幼苗中的表达量最高,表明其可能对种子发育起抑制作用。由此推断miR156家族在拟南芥种子发育过程中可能具有双向调控的复杂系统。
miR156的靶标为SPL基因家族已被公认,但在调控种子萌发过程中,miR156是否同样通过调控SPL基因家族发挥作用,目前还未见相关报道。本研究利用psRNATarget程序进行靶标预测,筛选出10个SPL靶基因,再通过TAIR网站上获取拟南芥种子发育各个阶段的表达量进行分析,研究发现果荚中表达量较高的3个基因分别是SPL2、SPL10和SPL11。进而通过对SPL基因家族系统发育进化树的分析,结果显示SPL2、SPL10和SPL11聚类在同一组中。相似地,在前人研究中,同样证明了SPL10和SPL11的氨基酸相似性高达78%[32],且在对SPL2、SPL10和SPL11的研究中,发现三者N-末端有3个保守结构域,一个是转录激活结构域:AHA-like motif[33],另两个分别是丝氨酸富集结构域和功能未知RTYF结构域,三者都是保守蛋白质结构域,说明这些蛋白在植物生长发育过程中具有重要作用[34],而这3个SPLs基因的功能极有可能存在较高相似性。综上,可推测miR156可能是通过调控SPL2、SPL10和SPL11的表达,进而调控种子发育和成熟过程。
为验证SPLs基因对种子成熟过程中的作用,本研究采用gus23检测体系进一步检测,结果显示CS66330和CS66335在新发叶上有蓝色GUS表型,CS66332在真叶上出现GUS表型,CS66334则在子叶和新发叶上均表现出异位的GUS活性,研究结果充分证明SPL2、SPL10、SPL11对种子贮藏蛋白起正向调控作用,鉴于miR156负向调控种子成熟过程及靶标SPL基因家族,可以推测该调控网络为miR156通过调控SPL2、SPL10、SPL11基因的表达,进而作用于LEC1、ABI3和FUS3调控网络中,再由该调控网络进一步控制种子贮藏蛋白基因的表达,从而影响种子的发育与成熟。
本文通过对miR156与其靶基因SPL2、SPL10、SPL11的相关研究,揭示了由miR156调控拟南芥种子发育和成熟过程中可能的调控网络。但是对于SPL2、SPL10、SPL11基因如何将功能作用于LEC1、ABI3和FUS3调控网络,目前的研究尚不能解决此问题,还需要进一步试验,将这一过程的调控机制阐明和完善。
4 结论本研究利用前期研究建立的报告基因筛选体系,对miR156下调突变体进行了GUS表型分析,表明14 d MIM156-gus23的幼苗中真叶均存在GUS活性,说明miR156基因的下调表达激活了SSPs基因的异位表达,且与种子成熟过程呈负向调控关系。为进一步验证这一结果,本研究对种子成熟过程中的关键调控基因LEC1、ABI3和FUS3在MIM156中进行表达分析,结果显示miR156的下调表达影响了这些基因在种苗中的表达情况,且与野生型拟南芥相比,LEC1、ABI3和FUS3的表达丰度显著升高,同样证实了miR156参与调控种子成熟过程。进一步通过生物信息学的分析及GUS表型分析,结果表明miR156的靶标基因SPL2、SPL10和SPL11正向调控种子贮藏蛋白基因的异位表达。综上所述,本研究推测miR156极有可能作为重要调控因子作用于其靶标基因SPL2、SPL10和SPL11通过影响种子贮藏蛋白基因SSPs的表达从而对种子成熟过程进行调控。
| [1] |
WEST M, HARADA J J. Embryogenesis in higher plants: an overview[J]. Plant Cell, 1993, 5: 1361-1369. DOI:10.2307/3869788 |
| [2] |
LEE R C, FEINBAUM R L, AMBROS V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5): 843-854. DOI:10.1016/0092-8674(93)90529-y |
| [3] |
PARK W, LI J J, SONG R T, MESSING J, CHEN X M. carpel factory, a Dicer homolog, and HEN1, a novel protein, act in microRNA metabolism in Arabidopsis thaliana[J]. Current Biology, 2002, 12(17): 1484-1495. DOI:10.1016/S0960-9822(02)01017-5 |
| [4] |
LLAVE C, KASSCHAU K D, RECTOR M A, CARRINGTON J C. Endogenous and silencing-associated small RNAs in plants[J]. The Plant Cell, 2002, 14(7): 1605-1619. DOI:10.1105/tpc.003210.ruses |
| [5] |
GAO R M, WANG Y, GRUBER M Y, HANNOUFA A. MiR156/SPL10 modulates lateral root development, branching and leaf morphology in Arabidopsis by Silencing AGAMOUS-LIKE 79[J]. Frontiers in Plant Science, 2018, 8: 2226. DOI:10.3389/fpls.2017.02226 |
| [6] |
LIU J, CHENG X L, LIU P, LI D Y, CHEN T, GU X F, SUN J Q. MicroRNA319-regulated TCPs interact with FBHs and PFT1 to activate CO transcription and control flowering time in Arabidopsis[J]. PLOS Genetics, 2017, 13(5): e1006833. DOI:10.1371/journal.pgen.1006833 |
| [7] |
VISENTIN I, PAGLIARANI C, DEVA E, CARACCI A, TURECKOVA V, NOVAK O, LOVISOLO C, SCHUBERT A, CARDINALE F. A novel strigolactone-miR156 module controls stomatal behaviour during drought recovery[J]. Plant Cell Environment, 2020, 43: 1613-1624. DOI:10.1111/pce.13758 |
| [8] |
YU C, CHEN Y T, CAO Y Q, CHEM H M, WANG J C, BI Y M, TIAN F, YANG F H, ROTHSTEIN S J, ZHOU X P, HE C Y. Overexpression of miR169o, an overlapping microRNA in response to both nitrogen limitation and bacterial infection, promotes nitrogen use efficiency and susceptibility to bacterial blight in rice[J]. Plant and Cell Physiology, 2018, 59(6): 1234-1247. DOI:10.1093/pcp/pcy060 |
| [9] |
WANG J W, YE Y J, XU M, FENG L G, XU L A. Roles of the SPL gene family and miR156 in the salt stress responses of tamarisk(Tamarix chinensis)[J]. BMC Plant Biology, 2019, 19: 370. |
| [10] |
WANG L, MING L, LIAO K, XIA C J, SUN S Y, CHANG Y, WANG H K, FU D B, XU C H, WANG Z J, LI X, XIE W B, OUYANG Y D, ZHANG Q L, LI X H, ZHANG Q H, XIAO J H, ZHANG Q F. Bract suppression regulated by the miR156/529-SPLs-NL1-PLA1 module is required for the transition from vegetative to reproductive branching in rice[J]. Molecular Plant, 2021, 14(7): 1168-1184. DOI:10.1016/j.molp.2021.04.013 |
| [11] |
BONAR N, L IN EY M, ZH ANG R X, AUST IN C, DESSOLY J, DAVIDSON D, STEPHENS J, MCDOUGALL G, TAYLOR M, BRYAN G J, HORNYIK C. Potato miR828 is associated with purple tuber skin and flesh color[J]. Frontiers in Plant Science, 2018, 9: 1742. DOI:10.3389/fpls.2018.01742 |
| [12] |
JIANG D G, ZHOU L Y, CHEN W T, YE N H, XIA J X, ZHUANG C X. Overexpression of a microRNA-targeted NAC transcription factor improves drought and salt tolerance in Rice via ABA-mediated pathways[J]. Rice, 2019, 12: 76. DOI:10.1186/s12284-019-0334-6 |
| [13] |
YAO X Z, CHEN J L, ZHOU J, YU H C Z, GE C N, ZHANG M, GAO X H, DAI X H, YANG Z N, ZHAO Y D. An essential role for mirna167 in maternal control of embryonic and seed development[J]. Plant Physiology, 2019, 180(1): 453-464. DOI:10.1104/pp.19.00127 |
| [14] |
RONG F X, CH EN, F F, HUANG L, ZHANG J Y, ZHANG C W, HOU D, CHENG Z H, WENG Y Q, CHEN P, LI Y H. A mutation in class Ⅲ homeodomain-leucine zipper(HD-ZIP Ⅲ)transcription factor results in curly leaf(cul)in cucumber(Cucumis sativus L.)[J]. Theoretical Applied Genetics, 2019, 132: 113-123. DOI:10.1007/s00122-018-3198-z |
| [15] |
CHUNG M Y, NATH U K, VREBALOV J, GAPPER N, LEE J M, LEE D J, KIM C K, GIOVANNONI J. Ectopic expression of miRNA172 in tomato (Solanum lycopersicum)reveals novel function in fruit development through regulation of an AP2 transcription factor[J]. BMC Plant Biology, 2020, 20(1): 283. DOI:10.1186/s12870-020-02489-y |
| [16] |
WILLMANN M R, POETHIG R S. Conservation and evolution of miRNA regulatory programs in plant development[J]. Current Opinion in Plant Biology, 2007, 10(5): 503-511. DOI:10.1016/j.pbi.2007.07.004 |
| [17] |
REINHART B J, WEINSTEIN E G, RHOADES M W, BARTEL B, BARTEL D. MicroRNAs in plants[J]. Genes and Development, 2002, 16(13): 1616-1626. DOI:10.1101/gad.1004402 |
| [18] |
MIAO C B, WANG Z, ZHANG L, YAO J J, HUA K, LIU X, SHI H Z, ZHU J K. The grain yield modulator miR156 regulates seed dormancy through the gibberellin pathway in rice[J]. Nature Communications, 2019, 10: 3822. DOI:10.1038/s41467-019-11830-5 |
| [19] |
ZHENG C F, YE M X, SANG M M, WU R L. A Regulatory Network for miR156-SPL Module in Arabidopsis thaliana[J]. International Journal of Molecular Sciences, 2019, 20(24): 6166. DOI:10.3390/ijms20246166 |
| [20] |
HUO H, WEI S, BRADFORD K J. DELAY of Germination1(DOG1) regulates both seed dormancy and flowering time through microRNA pathways[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(15): E2199-206. DOI:10.1073/pnas.1600558113 |
| [21] |
WANG J W. Regulation of flowering time by the miR156-mediated age pathway[J]. Journal of Experimental Botany, 2014(17): 4723-4730. DOI:10.1093/jxb/eru246 |
| [22] |
XING S, SALINAS M, HÖHMANN S, BERNDTGEN R, HUIJSER P. miR156-targeted and nontargeted SBP-Box transcription factors act in concert to secure male fertility in Arabidopsis[J]. Plant Cell, 2010, 22(12): 3935-3950. DOI:10.1105/tpc.110.079343 |
| [23] |
GOU J Y, FELIPPES F F, LIU C J, WEIGEL D, WANG J W. Negative regulation of anthocyanin biosynthesis in Arabidopsis by a miR156- targeted SPL transcription factor[J]. The Plant Cell, 2011, 23(4): 1512-1522. DOI:10.1105/tpc.111.084525 |
| [24] |
NODINE M D, BARTEL D P. MicroRNAs prevent precocious gene expression and enable pattern formation during plant embryogenesis[J]. Genes and Development, 2010, 24(23): 2678-2692. DOI:10.1101/gad.1986710 |
| [25] |
HUANG D, KOH C, FEURTADO J A, TSANG E W T, CUTLER A J. MicroRNAs and their putative targets in Brassica napus seed maturation[J]. BMC Genomics, 2013, 14(1): 1-25. DOI:10.1186/1471-2164-14-140 |
| [26] |
CHAO Y T, SU C L, JEAN W H, CEHN W C, CHANG T C A, SHIH M C. Identification and characterization of the microRNA transcriptome of a moth orchid Phalaenopsis aphrodite[J]. Plant Molecular Biology, 2014, 84(4/5): 529-548. DOI:10.1007/s11103-013-0150-0 |
| [27] |
张天豹. 胡麻种子发育和脂肪酸代谢相关microRNAs的鉴定和功能分析[D]. 北京: 中国农业科学院, 2020. ZHANG T B. Identification and functional analysis of MicroRNAs involving in seed development and fatty acid metabolism in Linum usiatissimum L[D]. Beijing: Chinese Academy of Agricultural Sciences, 2020. |
| [28] |
WANG J, MENG X, DOBROVOLSKAYA O B, ORLOV Y L, CHEN M. Noncoding RNAs and their roles in stress response in plants[J]. Genomics Proteomics Bioinformatics, 2017, 15(5): 301-312. DOI:10.1016/j.gpb.2017.01.007 |
| [29] |
郭秀芬, 张海龙, 王明晶, 赵振杰, 宋扬, 李立新. LAFL基因在种子发育和萌发中的功能[J]. 植物生理学报, 2019, 55(4): 393-400. GUO X F, ZHANG H L, WANG M J, ZHAO Z J, SONG Y, LI L X. Roles of LAFL genes in seed development and germination[J]. Plant Physiology Journal, 2019, 55(4): 393-400. |
| [30] |
TIAN R, WANG F F, ZHENG Q L, NIZA V, DOWNIE A B, PERRY S E. Direct and indirect targets of the Arabidopsis seed transcription factor ABSCISIC ACID INSENSITIVE3[J]. The Plant Journal, 2020, 103(5): 1679-1694. DOI:10.1111/tpj.14854 |
| [31] |
于月华, 吕宝莲, 倪志勇. 鹰嘴豆miRNA中miR156家族的生物信息学分析及靶基因预测[J]. 分子植物育种, 2021, 19(4): 1055-1060. DOI:10.13271/j.mpb.019.001055 YU Y H, LYU B L, NI Z Y. Bioinformatics analysis of mir156 family in chickpea miRNA and prediction of their target genes[J]. Molecular Plant Breeding, 2021, 19(4): 1055-1060. DOI:10.13271/j.mpb.019.001055 |
| [32] |
CA R DON G H, HÖHM ANN S, NET T E SHEIM K, SAEDL ER H, HUIJSER P. Functional analysis of the Arabidopsis thaliana SBP-box gene SPL3:a novel gene involved in the floral transition[J]. The Plant Journal, 1997, 12: 367-377. DOI:10.1046/j.1365-313X.1997.12020367.x |
| [33] |
RIESE M, HÖHMANN S, SAEDLER H, MÜNSTER T, HUIJSER P. Comparative analysis of the SBP-box gene families in P. patens and seed plants[J]. Gene, 2007, 401(1-2): 28-37. DOI:10.1016/j.gene.2007.06.018 |
| [34] |
S HIKATA M, KOYAMA T, MITSUDA N, OHME-TAKAGI M. Arabidopsis SBP-Box Genes SPL10, SPL11 and SPL2 control morphological change in association with shoot maturation in the reproductive phase[J]. Plant Cell Physiology, 2009, 50(12): 2133-2145. DOI:10.1093/pcp/pcp148 |
 2021, Vol. 48
2021, Vol. 48