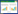文章信息
基金项目
- 国家自然科学基金(31801875);广东省自然科学基金(2020A1515011168);广东省科技计划项目(2020B020220001,2020B0202090002);广东省农业科学院科技创新战略专项资金(高水平农科院建设)—人才项目(R2019PY-QF009,R2018QD-040,R2020PY-JX003)
作者简介
-
宫超(1988—),女,博士,助理研究员,研究方向为茄果类蔬菜分子生物学及新品种选育,E-mail:gongchao@gdaas.cn
宫超,理学博士,广东省农业科学院青年副研究员,主要开展茄果类蔬菜分子育种及抗病相关研究。主持科研项目8项,包括国家自然科学基金青年科学基金、广东省自然科学基金面上项目、广东省农业科学院院长基金、广东省蔬菜新技术重点实验室开放基金等。发表科技论文20余篇,其中SCI收录8篇;以第一作者在Postharvest Biology and Technology、Phytopathology、Theoretical and Applied Genetics、Scientia Horticulturae、Horticulturae、《园艺学报》、《广东农业科学》、《农学学报》杂志发表学术论文10篇,其中中科院1区top期刊3篇;完成计算机软件著作2件;授权国家发明专利1项,实用新型专利1项;参与育成新品种2个。入选“中国热带作物学会青年人才托举工程项目(2020年度)”.
通讯作者
- 李涛(1982—),男,博士,副研究员,研究方向为茄果类蔬菜分子生物学及分子育种,E-mail:tianxing84@163.com.
文章历史
- 收稿日期:2021-07-21
茄科雷尔氏菌侵染能够引起寄主植物出现明显的萎蔫病症,对世界上许多农作物的产量和品质都造成了严重的影响。由于全球范围内集约农业的大力发展,导致茄科雷尔氏菌的宿主范围正在不断扩大[1]。青枯菌是一种好氧、不形成芽孢的革兰氏阴性菌,属于β-变形菌门(β-Proteobacteria),土壤传播,具有游动性,可从植物根部或茎的基部侵入植株导管系统,通过增殖和一系列生化活动堵塞或损坏寄主维管束输导组织,造成植物维管束萎蔫、坏死、甚至整株死亡,病害一旦发生常造成茄科作物严重减产甚至绝收[2-3]。病原菌在患病的植物残体、繁殖器官如块茎、根茎或某些作物的种子中越冬。被侵染的寄主植物死亡率高且缺乏有效的防治方法使青枯病成为世界上为害最大的植物病害之一。目前,随着北方温室大棚面积和复种指数的增加,青枯病已成为我国番茄生产的主要病害之一。最经济有效的防控手段是利用抗青枯病分子标记辅助选育优良的抗病品种。但是,抗青枯病番茄品种表现出的果实小、果软、果皮着色不匀以及畸形果多等劣质连锁遗传的问题一直悬而未决;且番茄的抗性主要是数量遗传,许多基因对抗病能力贡献很小[4]。番茄青枯病防治及抗病作用机制研究仍然是当今世界上植物抗病研究的热点和难点之一。为了有效防控青枯病,必须采取综合的病害管理方法来限制病原菌的存活、传播和侵染。
生物防治因其有效性及环境友好性的特点被认为是防治植物病害的有效手段。植物根际生存着种类繁多的微生物,包括有益菌和病原菌等,越来越多的能够帮助植物抵抗病原菌侵染的根际微生物相继被发现,防治青枯病的作用机制逐渐被解析。研究发现植物免疫系统和根际微生物之间存在密切的相互作用,根际微生物能够影响寄主植物免疫系统相关基因的表达模式,并会对植物代谢产生一定的影响[5];反之,植物体内基因表达及根系分泌物的变化也会对根际微生物群落的形成产生深远的影响。植物根际有益微生物可以帮助植物抵抗病原菌侵染,从而参与植物抗病过程。然而,目前对参与番茄抗青枯病过程的相关根际微生物及其作用机制的了解还非常有限。本文总结了近年来番茄青枯病抗性相关根际微生物的研究进展,重点介绍了抗青枯病根际微生物及其作用机制、抗青枯病番茄根际群落特征及特异性群落形成的影响因素,以及生防微生物参与番茄青枯病抗性遗传,以期为在番茄根际建立一个有益的微生物类群来防治青枯病作有效的指导,并对抗青枯病番茄品种的选育提供新思路。
1 番茄青枯病研究现状 1.1 番茄青枯病的为害青枯病是由薄壁菌门雷尔氏属茄科雷尔氏菌(R. solanacearum)侵染引起的重要细菌性土传病害,寄主范围广,该病原菌能侵染50多科450多种植物,包括番茄(Lycopersicum esculentum L.)、茄子(Solanum melongena L.)、烟草(Nicotina tabacum L.)、马铃薯(Solanum tuberosum L.)、辣椒(Capsicum spp. L.)以及花生(Arachis hypogaea L.)等。R. solanacearum对茄科植物的危害尤为严重,是最难防治的作物细菌性病害, 也是制约热带和亚热带地区蔬菜生产的重要限制性因素[1, 6-7]。青枯菌主要分为3种:R.solanacearum(种系型phylotypeⅡ)、R. pseudosolanacearum(种系型phylotypeⅠandⅡ)和R. syzygii(种系型phylotypeⅣ)[8-9]。青枯菌能够很好地适应土壤环境,即使在缺乏寄主植物的情况下也可以存活数年[10]。病原菌可以在病株或植物残体中越冬,在马铃薯块茎、生姜根茎、香蕉吸盘等营养繁殖器官中越冬,在辣椒、番茄等作物的种子上越冬,也可以在一些杂草寄主的根际越冬。病原菌能够在水生生境中生存,使用被污染的水源进行灌溉是寄主植物青枯病发生的接种源[11]。当遇到易感病的宿主,病原菌可以通过根尖和侧根裂纹或者通过农耕措施以及线虫、木质部取食昆虫攻击造成的自然伤口感染植株。病原菌通过鞭毛介导的游动作用以及对根系分泌物的感应及趋化作用进入伤口[6, 11],在内皮层的细胞间隙迅速发育,并利用寄主本身营养物质迅速增殖,分泌多个致病因子以侵染植物,如胞外多糖(Exopoly Saccharides,EPS)、细胞壁降解酶、3型分泌效应因子,随着木质部中的细菌繁殖,EPS的数量增加,形成致密的生物膜, 阻塞木质部导管、破坏周围组织,致使植物快速萎蔫,最终死亡[12-13]。目前公认的主要的致病因子是EPS,EPS是青枯菌在培养基上和植物体内生长过程中分泌大量粘性物质的重要成分,它可能保护青枯菌避开了植物组织或环境中的有毒物质,随着青枯菌的繁殖而增多,进而直接阻碍植物蒸腾作用的水分运输,也可能是EPS覆盖了青枯菌细胞表面的植物细胞识别基团,防止寄主可能发生的过敏性坏死反应等来阻碍病原菌的入侵[14]。Tiffany等2017年在针对青枯病期间番茄木质部汁液的代谢组学研究中发现,R. solanacearum产生并排泄大量的多胺腐胺(一种植物干旱胁迫信号),用腐胺处理番茄根和叶后再接种青枯病菌,可加速番茄青枯病的发生,增加青枯病菌在番茄木质部中的生长,增强病菌在寄主植物中系统性传播。该项研究表明,腐胺是一种新型的青枯病毒力代谢产物[6]。Hasegawa等研究发现β-D-葡萄糖苷乙酯,是一种从番茄根分泌物中分离到的R. solanacearum的新型趋化剂[15]。与许多植物病原细菌不同,R.solanacearum通过寄主植物上一个侵染位点便有可能使寄主植物产生系统性感染,导致青枯病的发生[16]。
1.2 我国番茄青枯病发生现状在我国,长期连作导致病原菌和植物自身毒素在土壤中积累,土传病害严重发生。连作导致青枯病在我国大面积爆发[17]。我国一直致力于青枯病及其病原菌茄科雷尔氏菌的研究。目前尽管已经运用不同的方法来防控青枯病,但对于大多数寄主植物,包括番茄在内,仍缺乏有效的、环境友好型的防治方法[9]。在我国,已经有30个省份报告了青枯病的发生,其中番茄青枯病主要发生在中南部和东部地区,多于北部和西部冷凉地区。虽然以前雷尔氏菌只在热带和亚热带地区流行,目前其在温带地区流行的趋势也逐渐增强。可能由于全球气候变暖和种植制度的变化,在过去的10年中,青枯病在我国温带和冷凉地区报道也呈显著上升趋势。目前,青枯病是华南地区及长江流域南部省份番茄生产面临的严重障碍,根据生长季节的不同,发病率从10% 到80% 不等[18-19]。广东省农业科学院于20世纪60年代中期从广东省多种植物中分离出了青枯菌,随着农业生产的加剧,青枯病在茄科作物和其他寄主上的流行率不断上升[9]。华南地区高温、多雨、高湿的天气持续时间长,夏秋季的高温暴雨常造成番茄青枯病严重发生[18],目前青枯病已成为华南地区番茄生产的最主要障碍。
1.3 番茄青枯病的防治番茄属于茄科作物,不耐连作,番茄重茬栽培导致的连作障碍现象在全国各大产区普遍发生,土壤退化现象不断加剧,土传病害严重发生。生产中为了解决土传病害危害严重导致减产的问题,大量使用化学肥料和农药,造成环境严重污染、农产品农药大量残留以及品质下降等一系列问题,同时导致土壤中微生物种群结构失衡,有益微生物种群减少,土壤肥力降低,作物减产[20],严重制约了番茄产业持续健康的发展。番茄青枯病病原菌生存力强,适应性强,遗传多样性丰富难以控制。
种植具有青枯病遗传抗性的番茄品种,是番茄稳产的最理想、经济有效且持久的方法[4],然而,抗青枯病的番茄品种能够被新出现的致病菌株侵染,再加上气候变化如高温高湿天气、土壤水分过多、根系受伤、以及环境中CO2浓度的升高均会导致抗病品种失去抗病能力[21]。目前化学杀菌剂和栽培技术广泛应用于青枯病的防治,但不能有效地控制青枯病的发生[10, 22]。此外,杀菌剂的广泛使用不仅能使青枯菌产生抗药性,产生的残留物也会造成严重的环境问题,危害人体健康[23]。轮作是非常有效的措施,但在我国难以大面积实施,而抗病品种和化学防治目前还难以满足生产的需求,嫁接技术可以在一定程度上防治番茄青枯病, 但生产中嫁接砧木质量良莠不齐,嫁接番茄也常表现出感青枯病症状。目前生物防治措施在综合病害管理中越来越普遍,生物防治的效果已经初见端倪。根系是青枯菌与植物互作的最主要场所。根际微生物参与介导植物对营养的吸收、抗病性和耐逆性,对植物健康和生长发育至关重要[24]。
促进植物生长的根际细菌(PGPR)是一类能在植物根组织内、根组织上或根组织周围生长,直接或间接促进植物生长的细菌,众所周知,PGPR的应用是通过减少农业生产中的化学品投入来实现绿色和可持续农业目标的一种非常有潜力的方法[25]。利用PGPR进行生物防治因环境友好的特点为防控植物青枯病提供了很好的替代传统防治技术的方法[26]。因此,PGPR被认为是控制番茄青枯病的最佳生物防治剂。近年来,在实验室、温室环境下已成功地获得了一系列对青枯病具有较好的生防效果的根际细菌,如假单胞菌属(Pseudomonas spp.)[27]、芽孢杆菌属(Bacillus spp.)[28]、黄杆菌(Flavobacterium johnonia)和大青金黄杆菌(Chryseobacterium daecheongense)[29]等。对于番茄,也相继从根际土壤中分离获得了对青枯病有较好的抑制作用的微生物(表 1)。我国地域辽阔各地区气候差异较大,华南地区气候条件具有极强的特异性,温度高、湿度大,每年4~7月33℃以上的高温天气连续几十天,大多时间空气相对湿度超过80%,导致青枯菌类型及栽培品种的选择具有一定的特异性,一些现成的生物防治剂不能有针对性地发挥很好的防治效果。因此,适用于防治华南地区番茄青枯病的生防菌菌种资源还有待于进一步挖掘。
PGPR的作用机制比较复杂的,通常分为两个方面[25]:(1)参与营养循环和植物催化,包括N2固定、不溶性磷的增溶作用、通过铁载体分泌的铁固定、植物激素的形成,以及降低植物乙烯水平;(2)参与由产生抗生素、竞争根际铁以及诱导寄主植物产生系统抗性介导的植物病原菌的生物防治。这些机制实现的基本前提是PGPR必须在根际定殖并维持决定性的种群密度才能有效地发挥作用。另有研究表明,一些PGPR接种到土壤后会刺激根际中其他微生物种群发生变化进而获得对病原菌的生防能力,其本身不会在根际长久定殖但是可以赋予土壤比较长久的抵抗病原菌的能力,不依赖于自身在土壤中较高的相对丰度,PGPR对土壤细菌群落结构的调控是其发挥生防作用的重要机制[25]。随着对PGPR作用机制的解析,很多关于PGPR对根际本土细菌群落产生影响的报道。
2 根际微生物群落参与调控番茄青枯病抗性 2.1 番茄青枯病抗性影响因子寄主植物及其对应的微生物的全基因组作为植物防御的重要组成部分是抗病性的遗传基础[39-40]。已有研究发现,调控免疫系统的相关基因会影响植物根际微生物的组成,添加不同的微生物也会影响寄主植物免疫系统相关基因表达量的变化,大量能够帮助植物抵抗病原菌侵染的细菌、真菌相继被发现,表明植物免疫系统和根际微生物之间是相互影响的[31]。根际微生物调控植物抗性和生理响应的机制是错综复杂的。
青枯病抗性是由数量性状位点控制的,但在作物上的表现是可变的,受环境因素的影响,包括植物微生物群落[41]。番茄栽培品种‘Hawaii7996’是一个著名的抗青枯病番茄品种[7],其通过几个主要和次要的数量性状位点(Quantitative trait loci,QTL)产生稳定的青枯病抗性。以上研究表明,主要抗青枯病数量性状位点(QTL)Bwr-6位于6号染色体、Bwr-12位于12号染色体。然而,两个QTL的遗传特性尚未被解析。Kim等利用全基因组重测序的方法,分析全基因组范围内可以区分抗病群体和感病群体的单核苷酸多态性(Single Nucleotide Polymorphisms, SNPs), 结果表明主要的SNPs在6号和12号染色体上,选择既携带SNPs又接近Bwr-6和Bwr-12位点的基因,并从中筛选获得一个可以鉴定抗青枯病番茄品种的单核苷酸多态性标记[42]。番茄的青枯病抗性主要是数量遗传,许多基因对抗病能力贡献很小[4],在Hawaii7996等主要作物中,对青枯病的数量抗性尚不完全清楚,抗病基因和功能也没有完全解析,其具体的抗病通路和分子机制亟待研究。Kwak等[25]根据研究结果做出假设,抗病品种Hawaii7996上的一个或多个数量性状位点可能在募集能够提高青枯病抗性的特定微生物方面发挥作用,但是根际募集的参与提高青枯病抗性的根际微生物与植物青枯病抗性位点(基因)之间的关系犹未可知。Hawaii7996的数量抗性表现经常受到环境条件的影响,如病原菌菌株、温度和土壤条件[41],这可能与环境变化改变其根际微生物群落结构和组成存在相关性。目前为止,植物基因型特异性造成的根际微生物群落的特异性是否由寄主植物的一个或多个抗性位点调控、青枯病抗性相关根际微生物参与植物诱导抗性机制还有待于进一步验证。
2.2 根际微生物群落调节寄主植物青枯病抗性大量研究利用有益微生物接种于作物根际,用于克服连作引起的微生物群落结构失调产生的不良影响,有利于抑制土传病害的爆发。青枯菌入侵导致番茄根际微生物群落多样性和组成发生变化[43],生防制剂可以调节土壤微生物群落结构,减轻植物青枯病的发生。土壤和植物的健康水平在很大程度上取决于土壤微生物群落的组成和多样性,土壤微生物群落的多样性对土壤抑制土传病害的能力至关重要[44-45]。土壤微生物是调节植物抗性和病原菌致病力的重要因子,在调节土壤肥力、养分循环、促进植物健康和防治青枯病等方面发挥着重要作用。研究表明,有益微生物相对丰度与土壤质量,包括植物长势,病害发生率,养分含量、土壤酶活性和土壤pH值显著相关[46]。Kwak等研究发现青枯病发生的严重程度与土壤微生物群落组成密切相关[30]。众所周知,土壤中自然存在的微生物群落能够显著降低植物青枯病的严重程度[47]。健康的土壤能够抑制番茄、烟草青枯病的发生[17, 30]。大量研究发现,与健康土壤相比,感染青枯病的土壤微生物群落的组成和结构发生了改变[46, 48]。另外,研究表明嫁接可以增强番茄抗青枯病能力,很大程度上取决于嫁接改变了番茄根际微生物群落[49-50]。Kwak等2018研究发现,抗青枯病番茄品种Hawaii7996和易感青枯病番茄品种Moneymaker的根际微生物群落存在显著差异,将易感青枯病番茄品种Moneymaker移植到之前种过抗病品种Hawaii7996的土中能够缓解易感病番茄植株的病症,并且证明根际微生物群落结构的改变使番茄具有青枯病抗性,具体表现为黄杆菌(Flavobacterium)在抗病品种中具有明显富集的现象[30]。
2.3 根际微生物诱导植物抗青枯病反应在寄主植物根际建立一个对于自身有利的根际微生物群落对植物的抗病性和病原菌致病性都至关重要。植物募集有益的微生物来刺激植物生长,激活植物的系统防御,产生对病原菌的抗性[51]。植物进化出复杂的先天免疫系统,当植物受到一个或多个病原菌进攻时,免疫系统被激活,植物第一道防线依赖于特异的细胞表面模式识别受体(PRRs)识别病原相关分子模式(PAMP),从而产生PAMPs触发免疫(PTI)反应,往往会引发植物系统获得抗病性(SAR),限制病原菌的定殖[52]。非致病微生物也会以类似的方式被识别。存在于根际的PGPR对病原菌存在直接的拮抗作用,但通常也会使植物产生微生物相关分子模式(MAMPs)触发免疫(MTI)反应,在JA、ET等植物激素介导下,使寄主植物产生诱导系统抗性(ISR)[53]。假单胞菌(Pseudomonas spp.)和其他革兰氏阴性菌已经被证实能够激活植物诱导系统抗性。一些解淀粉芽胞杆菌(Bacillus amyloliquefaciens)、枯草芽胞杆菌(B. subtilis)、巴氏芽胞杆菌(B. pasteurii)、蜡样芽胞杆菌(B. cereus)、短小芽胞杆菌(B. pumilus)、蕈状芽胞杆菌(B. mycoides)和球形芽胞杆菌(B. sphaericus)菌株能够诱导宿主植物产生诱导系统抗性,显著降低多种宿主植物包括番茄、辣椒、烟草等作物病害的发病率和严重程度。
培育具有适当抗性的作物品种是青枯病综合治理的关键。根据土壤类型、气候条件等环境因子选择适合种植的番茄品种及栽培方式,规范田间管理,合理、高效地充分利用根际微生态系统的区域优势来提高番茄生产力和土壤营养水平,将有效的改变番茄青枯病的发生。
3 抗青枯病根际微生物群落特征及特定微生物募集 3.1 抗青枯病土壤根际微生物群落特征研究证实,能够抑制土传病原菌的微生物群落对植物病害防控意义重大,土壤微生物群落结构的变化与土传青枯病的发生密切相关[54-55]。Wang等研究表明,相对于发生烟草青枯病的土壤,健康烟草土壤中的有益微生物更为丰富,包括芽胞杆菌属(Bacillus)、壤霉菌属(Agromyces)、小单孢菌属(Micromonospora)、假诺卡氏菌属(Pseudonocardia)、支顶孢属(Acremonium)、溶杆菌属(Lysobacter)、根瘤菌属(Mesorhizobium)、微枝形杆菌属(Microvirga)、慢生根瘤菌属(Bradyrhizobium)、枝顶孢属(Acremonium)和毛壳菌属(Chaetomium)等[46]。Lee等研究发现,相对于发病植株根际土壤,变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、疣微菌门(Verrucomicrobia)在未发生青枯病的土样样品中丰度更高[47]。Kwak等[30]在关于微生物是否参与植物对病原菌的抗性的研究表明,抗青枯病番茄品种Hawaii7996和易感青枯病番茄品种Moneymaker的根际微生物结构和组成存在差异。在番茄生长的第一阶段,相对于Moneymaker而言,Hawaii7996根际土壤中的拟杆菌门(Bacteroidetes)生长尤为显著;在纲和科分类水平上,黄杆菌纲(Flavobacteriia)、黄杆菌科(Flavobacteriaceae),鞘氨醇单胞菌科(Sphingomonadaceae)和假单胞菌科(Pseudomonadaceae)在Hawaii7996根际土壤中含量较高。
Kwak等研究不仅证实了根际微生物确实参与了番茄抗青枯病过程,抗病番茄植株根系存在有益菌能够帮助番茄抵抗青枯菌的入侵,并以此为指导,将分离得到的黄杆菌添加到番茄根际后能够增强青枯病抗性,番茄抗青枯病能力的提高主要是黄杆菌在抗病品种Hawaii7996中富集的结果。Marian等[10]研究表明,在番茄根际分离到的Ralstonia sp. TCR112和Mitsuaria sp. TWR114可以通过直接的拮抗作用和/或间接作用(如竞争营养物质和诱导番茄产生抗性)来阻止病原菌增殖和侵染进而达到抑制番茄青枯病的目的。刘琼光等[57]研究表明,抗青枯病番茄品种根际微生物的数量高于感青枯病品种,并且抗青枯病能力越强的番茄, 其根系青枯菌越少。我们前期研究表明,采集自不同地点的番茄根际土壤样品中健康番茄根际土壤和感病番茄根际土壤中根际细菌群落均有所差异[58]。我们的研究还表明,EM菌肥的使用可以在一定程度上减轻青枯病的危害或与其增强番茄生长势有关(未发表数据),但是,存在于番茄根际对青枯菌有较好拮抗作用有益微生物参与提高植株抗青枯病的作用机制还有待于进一步探索。
3.2 根际微生物群落形成的影响因素植物与土壤微生物的相互作用是一个动态的、复杂的生物过程,涉及微生物和植物代谢途径之间的相互联系,同时会受到环境因素的影响。无论土壤类型或地理位置如何,植物种类、栽培品种、基因型和不同生育期都会影响根际微生物群落。植物种类或基因型对微生物群落的影响程度通常小于环境因子和土壤因子的影响。尽管植物种类或基因型对微生物群落的影响程度相对较小,但植物基因型对微生物群落组成的影响尤为重要,报道称通过选择特定的作物基因型可以间接地从其根际微生物群落中获益。具有理想表型的植物基因型可以作为筛选有益微生物类群的有效工具,例如,与高产、抗病的作物基因型直接相关的微生物群落可能为生物肥料或生物防治剂的研发提供候选菌株,而探索与微生物群落相关的寄主植物基因可能为作物育种提供新的见解[24]。寄主植物根系分泌物组成的变化会直接影响根际土壤微生物群落的组成。寄主植物的健康状况会通过影响根系分泌物来改变根际微生物群落的组成[52]。植物通过在根际释放分泌物,选择性地募集有利于植物生长的微生物或抑制对植物有害的微生物,从而主动选择和培育特定的根际微生物群落[59]。
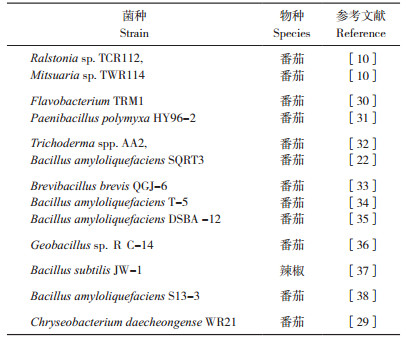
土壤因素对根际微生物富集影响较为显著,比如,土壤的类型、含水量、土壤pH以及有机质含量等。土壤理化性质影响植物生理特征以及植物根系分泌物的种类和水平,植物生理特征以及植物根系分泌物的种类和水平又会对植物根际微生物的组成产生影响。植物生长环境变化会进一步引起根系分泌物的迅速变化,造成了微生物的聚集在时间和空间上存在不同[60]。农业生产管理方式,比如在田间生产管理时施用有机肥料、无机肥料、土壤改良剂、生防菌剂等增强植物抗逆性和产量等相关的措施均会造成根际微生物群落产生不同程度的变化。土壤微生物群落中不同微生物种类之间存在着复杂的相互作用(如共生、寄生、空间或养分竞争、捕食),微生物与寄主植物之间也存在着复杂的相互作用(如营养供应、诱导寄主抗病性、产生抗生素或抗菌物质),根际微生物与寄主植物间形成了一个复杂的相互关联的生态网络[61],这种复杂的相互作用关系也是影响根际微生物群落形成的一个重要因素。在根际土壤中,病原菌入侵也可以通过间接影响寄主植物来改变根际微生物群落[62]。此外,季节性波动或气候条件变化、寄主植物与其他植物间相互作用也会对根际微生物群落产生一定程度的影响。根际微生物群落组成模式的形成和微生物活性的改变通常是一个甚至多个影响因素共同作用的结果,在农业生态系统中,根际微生物多样性的影响因素如图 1所示,影响因素会因具体情况有所不同,作用强度也会存在主次之分。植物能够促使一些根际微生物分类群的丰度增加,而减少其他分类群的丰度,植物也会通过提供碳等能源来影响微生物的活动。无论是在自然生态系统中还是农业生态系统中根际微生物群落多样性的形成依赖于植物释放到根际的根系分泌物。植物根际微生物在植物健康和疾病中起着重要作用[63-64]。微生物区系受植株健康状况影响显著,也许是因为健康状况直接影响着根系分泌物,而根系分泌物对土壤微生物尤其是根际土壤微生物具有很强的选择作用,能够募集特定的微生物类群而形成独特的微生物区系[65],根际微生物结构与组成能够在一定程度上反映植物的健康状况。根际微生物群落中的有益菌对植物生长和生物学功能的发挥具有重要调节作用[66]。土壤微生态平衡和微生物多样性是抑制植物土传病害的必要条件[67]。土壤微生物群落在植物生长过程中起着重要作用。一方面,植物可以通过根系分泌物改变土壤微生物,另一方面,土壤微生物也能够影响植物的生产力、多样性和健康状况[68]。研究发现,通过改良根际微生物群落,可以增强对土传病害的抑制作用[67, 69]。根际微生物与番茄青枯病的发生有着密切的关系。然而,特定的地区及特定的番茄品种根际微生物群落特征及健康与发病株系特异性募集的差异菌群与青枯病抗性差异之间的关系有待于进一步解析。
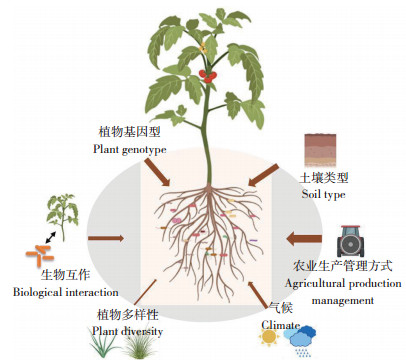
|
| 箭头的相对厚度代表每个因素影响根际微生物多样性的重要性 The relative thicknesses of arrows show the importance of each factor influencing the diversity of rhizosphere micro organism 图 1 农业生态系统中根际微生物多样性影响因素 Fig. 1 Factors influencing the diversity of rhizosphere micro organism in agricultural ecosystems |
4 展望
综上所述,根际微生物群落的结构与青枯病抗性息息相关。研究发现天然的微生物在保护植物免受病原微生物侵害方面具有重要作用,为开发有益微生物来改善植物病害指明了道路。华南地区番茄抗青枯病品种根际微生物群落中青枯病抗性主要贡献菌群犹未可知,这也是我们研究的主要方向之一。另外,番茄青枯病的防治与抗病机制研究是当今世界上植物抗病研究的热点和难点之一。作为青枯病抗性的重要调控者,根际微生物参与诱导番茄青枯病抗性的作用机制尚不明确,并且根际拮抗微生物诱导番茄产生对青枯菌抗性机制仍不清楚,这将为下一步挖掘番茄抗青枯病基因指明方向,也为利用基因编辑技术(CRISPR/Cas9)改良番茄青枯病抗性奠定基础。对健康番茄根际拮抗微生物诱导番茄青枯菌抗性机制的深入研究可为番茄抗青枯病机理的解析及抗病品种选育奠定理论基础。因此,随着利用高通量测序技术结合生物信息学分析方法的普及,针对挖掘健康番茄根际中微生物群落的指示类群(根际拮抗微生物)在番茄抗青枯病过程中发挥的作用,及其在诱导番茄青枯病抗性过程中可能的作用机制问题的研究将为番茄青枯病抗性机制的解析和抗病基因的挖掘奠定坚实的基础,对番茄青枯病的防治与品种的合理布局及抗病品种的选育具有重要的理论与实践意义。
| [1] |
DEVENDRA K C, AUNDY K, SAJAD U N N. In-vitro evaluation of Arabidopsis thaliana ecotypes against Ralstonia solanacearum Race 4[J]. International Journal of Current Microbiology and Applied Sciences, 2017, 6(5): 575-579. DOI:10.20546/ijcmas.2017.605.066 |
| [2] |
FRENCH E, KIM B S, RIVERA-ZULUAGA K, IYER-PASCUZZI S A. Whole root transcriptomic analysis reveals a role for auxin pathways in resistance to Ralstonia solanacearum in tomato[J]. Molecular Plant-Microbe interaction, 2018, 31(4): 432-44. DOI:10.1101/176685 |
| [3] |
REMO F, LUBOOBIL S, MABALAWATAI S, NANNYONGA B K. A mathematical model for the dynamics and MCMC analysis of tomato bacterial wilt disease[J]. International Journal of Biomathematics, 2018, 11(1): 1850001. DOI:10.1264/jsme2.ME16136 |
| [4] |
K IM, B S, FRENCH E, CALDWELL D, HARRINGTON E J, IYER-PASCUZZI A S. Bacterial wilt disease: host resistance and pathogen virulence mechanisms[J]. Physiological & Molecular Plant Pathology, 2016, 95: 37-43. DOI:10.1016/j.pmpp.2016.02.007 |
| [5] |
MARTÍNEZ-CASTRO E, JARQUIN-GÁLVEZ R, ALPUCHE-SOLÍS Á G, VALLEJO-PÉREZ M R, COLLI-MULL J G, LARA-ÁVILA J P. Bacterial wilt and canker of tomato: fundamentals of a complex biological system[J]. Euphytica, 2018, 214(4). DOI:10.1007/s10681-018-2140-4 |
| [6] |
LOWE-POWER T M, HENDRICH C G, VON ROEPENACK-LAHAYE E, L IB, WU D, MITRA R, DALSING B L, RICCA P, NAIDOO J, COOK D, JANCEWICZ A, MASSON P, THOMMA B, LAHAYE T, MICHAEL A J, ALLEN C. Metabolomics of tomato xylem sap during bacterial wilt reveals Ralstonia solanacearum produces abundant putrescine, a metabolite that accelerates wilt disease[J]. Environmental Microbiology, 2017, 20(4). DOI:10.1111/1462-2920.14020 |
| [7] |
CHOI K, CHOI J, LEE P A, ROY N, KHAN R, LEE H J, WEON H Y, KONG H G, LEE S W. Alteration of bacterial wilt resistance in tomato plant by microbiota transplant[J]. Frontiers in Plant Science, 2020, 11: 1186. DOI:10.3389/fpls.2020.01186 |
| [8] |
PRIOR P, AILLOUD F, DALSING B L, REMENANT B, SANCHEZ B, ALLEN C. Genomic and proteomic evidence supporting the division of the plant pathogen Ralstonia solanacearum into three species[J]. BMC Genomics, 2016, 17: 90. DOI:10.1186/s12864-016-2413-z |
| [9] |
JIANG G, WEI Z, XU J, CHEN H, ZHANG Y, SHE X, MACHO A P, DING W, LIAO B. Bacterial wilt in china: history, current status, and future perspectives[J]. Frontiers in Plant Science, 2017, 8: 1549. DOI:10.3389/fpls.2017.01549 |
| [10] |
MARIAN M, NISHIOKA T, KOYAMA H, SUGA H, SHIMIZU M. Biocontrol potential of Ralstonia sp. TCR112 and Mitsuaria sp. TWR114 against tomato bacterial wilt[J]. Applied Soil Ecology, 2018, 128: 71-80. DOI:10.1016/j.apsoil.2018.04.005 |
| [11] |
DEVENDRA K C, SAJAD UN N, MOHAMMAD S D, KAMRAN A K. Ralstonia solanacearum: A wide spread and global bacterial plant wilt pathogen[J]. Journal of Pharmacognosy and Phytochemistry, 2018, 7(2): 85-90. |
| [12] |
MILLING A, BABUJEE L, ALLEN C. Ralstonia solanacearum extracellular polysaccharide is a specific elicitor of defense responses in wilt-resistant tomato plants[J]. PLoS One, 2011, 6(1): e15853. DOI:10.1371/journal.pone.0015853 |
| [13] |
TRAN T M, MACINTYRE A, HAWES M, ALLEN C. Escaping underground nets: Extracellular Dnases degrade plant extracellular traps and contribute to virulence of the plant pathogenic bacterium Ralstonia solanacearum[J]. PLOS Pathogens, 2016, 12: e1005686. DOI:10.1371/journal.ppat.1005686 |
| [14] |
CALDWELL D, KIM B S, IYER-PASCUZZI A S. Ralstonia solanacearum differentially colonizes roots of resistant and susceptible tomato plants[J]. Phytopathology, 2017, 107(5): 52-536. DOI:10.1094/PHYTO-09-16-0353-R |
| [15] |
HASEGAWA T, OKABE A, KATO Y, OOSHIRO A, KAWAIDE H, NATSUME M. Ethyl β-d-glucoside: A novel chemoattractant of Ralstonia solanacearum isolated from tomato root exudates by a bioassay-guided fractionation[J]. Bioscience, Biotechnology, and Biochemistry, 2018, 82(12): 1-4. DOI:10.1080/09168451.2018.1515618 |
| [16] |
DENNY T P. Plant pathogenic Ralstonia species//Gnanamanickam, S S. Plant-Associated Bacteria[C]. 2006: 573-644. DOI: 10.1007/978-1-4020-4538-7_16.
|
| [17] |
QI G, MA G, CHEN S, LIN C, ZHAO X. Microbial network and soil properties are changed in bacterial wilt-susceptible soil[J]. Applied and Environmental Microbiology, 2019, 85(13). DOI:10.1128/AEM.00162-19 |
| [18] |
黎振兴, 李植良, 孙保娟, 李涛, 宫超, 衡周. 华南地区番茄和茄子研究历史、现状与展望[J]. 广东农业科学, 2020, 47(12): 42-52. DOI:10.16768/j.issn.1004-874X.2020.12.005 LI Z X, LI Z L, SUN B J, LI T, GONG C, HENG Z. History, present situation and prospect of researches on tomato and eggplant in south china[J]. Guangdong Agricultural Sciences, 2020, 47(12): 42-52. DOI:10.16768/j.issn.1004-874X.2020.12.005 |
| [19] |
WEI Z, HUANG J, YANG T, JOUSSET A, XU Y, SHEN Q, FRIMAN V P. Seasonal variation in the biocontrol efficiency of bacterial wilt is driven by temperature-mediated changes in bacterial competitive interactions[J]. Journal of Applied Ecology, 2017, 54(5). DOI:10.1111/1365-2664.12873 |
| [20] |
顾松松, 熊兴耀, 谭琳, 胡新喜, 汤心砚, 程予奇, 胡秋龙. 土壤微生态与马铃薯连作障碍机制的研究进展[J]. 中国农学通报, 2018, 34(30): 42-45. DOI:10.11924/j.issn.1000-6850.casb17110026 GU S S, XIONG X Y, TAN L, HU X X, TANG X Y, CHENG Y Q, HU Q L. Soil microorganisms and the mechanism of potato continuous cropping obstacle: research progress[J]. Chinese Agricultural Science Bulletin, 2018, 34(30): 42-45. DOI:10.11924/j.issn.1000-6850.casb17110026 |
| [21] |
HONG J K, JANG S J, LEE Y H, JO Y S, YUN J G, JO H, PARK C J, KIM H J. Reduced bacterial wilt in tomato plants by bactericidal peroxyacetic acid mixture treatment[J]. The Plant Pathology Journal, 2018, 34(1): 78-84. DOI:10.5423/PPJ.NT.06.2017.0131 |
| [22] |
LI C Y, HU W U, PAN B, LIU Y, YUAN S F, DING Y Y, LI R, ZHENG X Y, SHEN B, SHEN Q R. Rhizobacterium Bacillus amyloliquefaciens strain SQRT3-mediated induced systemic resistance controls bacterial wilt of tomato[J]. Pedosphere, 2017, 27(6): 1135-1146. DOI:10.1016/S1002-0160(17)60406-5 |
| [23] |
VU T T, KIM H, TRAN V K, VU H D, HOANG T X, HAN J W, CHOI Y H, JANG K S, CHOI G J, KIM J C. Antibacterial activity of tannins isolated from Sapium baccatum extract and use for control of tomato bacterial wilt[J]. PLoS ONE, 2017, 12(7): e0181499. DOI:10.1371/journal.pone.0181499 |
| [24] |
POUDEL R, JUMPPONEN A, KENNELLY M M, RIVARD C L, GOMEZ-MONTANO L, GARRETT K A. Rootstocks shape the rhizobiome: rhizosphere and endosphere bacterial communities in the grafted tomato system[J]. Applied and Environmental Microbiology, 2019, 85: e01765-18. DOI:10.1101/375444 |
| [25] |
KANG Y J, SHEN M, WANG H L, ZHAO Q X. A possible mechanism of action of plant growth-promoting rhizobacteria(PGPR)strain Bacillus pumilus WP8 via regulation of soil bacterial community structure[J]. The Journal of General and Applied Microbiology, 2013, 59(4): 267-277. DOI:10.2323/jgam.59.267 |
| [26] |
PASTOR N, CARLIER E, ANDRÉS J, ROSAS S B, ROVERA M. Characterization of rhizosphere bacteria for control of phytopathogenic fungi of tomato[J]. Journal of Environmental Management, 2012, 95: S332-S337. DOI:10.1016/j.jenvman.2011.03.037 |
| [27] |
LEMESSA F, ZELLER W. Screening rhizobacteria for biological control of Ralstonia solanacearum in Ethiopia[J]. Biological Control, 2007, 42: 336-344. DOI:10.1016/j.biocontrol.2007.05.014 |
| [28] |
KURABACHEW H, WYDRA K. Characterization of plant growth promoting rhizobacteria and their potential as bioprotectant against tomato bacterial wilt caused by Ralstonia solanacearum[J]. Biological Control, 2013, 67: 75-83. DOI:10.1016/j.biocontrol.2013.07.004 |
| [29] |
HUANG J, WEI Z, TAN S, MEI X, YIN S, SHEN Q, XU Y. The rhizosphere soil of diseased tomato plants as a source for novel microorganisms to control bacterial wilt[J]. Applied Soil Ecology, 2013, 72: 79-84. DOI:10.1016/j.apsoil.2013.05.017 |
| [30] |
KWAK M J, KONG H G, CHOI K, KWON S K, SONG J Y, LEE J, LEE J, LEE P A, CHOI S Y, SEO M, LEE H J, JUNG E J, PARK H, ROY N, KIM H, LEE M M, RUBIN E M, LEE S W, KIM J F. Rhizosphere microbiome structure alters to enable wilt resistance in tomato[J]. Nature biotechnology, 2018, 36: 1100. DOI:10.1038/nbt.4232 |
| [31] |
LUO Y C, CHENG Y J, YI J C, ZHANG Z J, LUO Q, ZHANG D J, LI Y G. Complete genome sequence of industrial biocontrol strain Paenibacillus polymyxa HY96-2 and further analysis of its biocontrol mechanism[J]. Frontiers in Microbiology, 2018, 9: 1-14. DOI:10.3389/fmicb.2018.01520 |
| [32] |
YENDYO S, RAMESH G C, PANDEY B R. Evaluation of Trichoderma spp., Pseudomonas fluorescence and Bacillus subtilis for biological control of Ralstonia wilt of tomato[J]. F1000Research, 2017, 6: 2028. DOI:10.12688/f1000research.12448.1 |
| [33] |
伍善东, 雷平, 郭照辉, 黄军, 程伟, 付祖姣, 单世平. 1株番茄青枯病内生拮抗细菌的分离鉴定及盆栽防效[J]. 湖南农业大学学报(自然科学版), 2016, 42(6): 627-630. DOI:10.13331/j.cnki.jhau.2016.06.008 WU S D, LEI P, GUO Z H, HUANG J, CHENG W, FU Z J, SHAN S P. Isolation, identification and potted control efficacy of an endophytic bacerial against Ralstonia solanacearum[J]. Journal of Hunan Agricultural University(Natural Sciences), 2016, 42(6): 627-630. DOI:10.13331/j.cnki.jhau.2016.06.008 |
| [34] |
RAZA W, WANG J, WU Y, LING N, WEI Z, HUANG Q, SHEN Q. Effects of volatile organic compounds produced by Bacillus amyloliquefaciens on the growth and virulence traits of tomato bacterial wilt pathogen Ralstonia solanacearum[J]. Applied Microbiology and Biotechnology, 2016, 100: 7639-7650. DOI:10.1007/s00253-016-7584-7 |
| [35] |
SINGH D, YADAV D K, CHAUDHARY G, RANA V S, SHARMA R K. Potential of Bacillus amyloliquefaciens for biocontrol of bacterial wilt of tomato incited by Ralstonia solanacearum[J]. Journal of Plant Pathology & Microbiology, 2016, 7: 327. DOI:10.4172/2157-7471.1000327 |
| [36] |
韦中, 胡洁, 董月, 杨天杰, 沈其荣, 徐阳春. 基于菜粕有机肥筛选番茄青枯病高效生防菌的研究[J]. 南京农业大学学报, 2015, 38(3): 424-430. DOI:10.7685/j.issn.1000-2030.2015.03.011 WEI Z, HU J, DONG Y, YANG T J, SHEN Q R, XU Y C. Screen of antagonists against Ralstonia solanacearum by using rapeseed cake compost as a selected medium[J]. Journal of Nanjing Agricultural University, 2015, 38(3): 424-430. DOI:10.7685/j.issn.1000-2030.2015.03.011 |
| [37] |
KWON J W, KIM S D. Characterization of an antibiotic produced by Bacillus subtilis JW-1 that suppresses Ralstonia solanacearum[J]. Journal of Microbiology and Biotechnology, 2014, 24(1): 13-18. DOI:10.4014/jmb.1308.08060 |
| [38] |
YAMAMOTO S, SHIRAISHI S, KAWAGOE Y, MOCHIZUKI M, SUZUKI S. Impact of Bacillus amyloliquefaciens S13-3 on control of bacterial wilt and powdery mildew in tomato[J]. Pest Management Science, 2014, 71(5): 722-727. DOI:10.1002/ps.3837 |
| [39] |
BORDENSTEIN S R, THEIS K R. Host biology in light of the microbiome: ten principles of holobionts and hologenomes[J]. PLoS Biology, 2015, 13: e1002226. DOI:10.1371/journal.pbio.1002226 |
| [40] |
ARNOLD C. The hologenome: A new view of evolution[J]. The New Scientist, 2013, 217(2899): 30-34. DOI:10.1016/S0262-4079(13)60115-3 |
| [41] |
WANG J F, HO F I, TRUONG H T H, HUANG S M, BALATERO C H, DITTAPONGPITCH V, HIDAYATI N. Identification of major QTLs associated with stable resistance of tomato cultivar'Hawaii 7996'to Ralstonia solanacearum[J]. Euphytica, 190(2): 1-12. DOI:10.1007/s10681-012-0830-x |
| [42] |
KIM B, HWANG I S, LEE H J, LEE J M, SEO E, CHOI D, OH C S. Identification of a molecular marker tightly linked to bacterial wilt resistance in tomato by genome-wide SNP analysis[J]. Theoretical and Applied Genetics, 2018, 131(5): 1017-1030. DOI:10.1007/s00122-018-3054-1 |
| [43] |
WEI Z, HU J, GU Y, YIN S, XU Y, JOUSSET A, SHEN Q, FRIMAN V P. Ralstonia solanacearum pathogen disrupts bacterial rhizosphere microbiome during an invasion[J]. Soil Biology and Biochemistry, 2018, 118: 8-17. DOI:10.1016/j.soilbio.2017.11.012 |
| [44] |
VAN ELSAS J D, CHIURAZZI M, MALLON C A, ELHOTTOVA D, KRISTUFEK V, SALLES J F. Microbial diversity determines the invasion of soil by a bacterial pathogen[J]. Proceedings of the National Academy of Sciences, 2012, 109(4): 1159-1164. DOI:10.1073/pnas.1109326109 |
| [45] |
GARBEVA P, POSTMA J, VAN VEEN JA, VAN ELSAS J D. Effect of above-ground plant species on soil microbial community structure and its impact on suppression of Rhizoctonia solani AG3[J]. Environmental Microbiology, 2006, 8(2): 233-246. DOI:10.1111/j.1462-2920.2005.00888.x |
| [46] |
WANG R, ZHANG H C, SUN L G, QI G F, CHEN S, ZHAO X Y. Microbial community composition is related to soil biological and chemical properties and bacterial wilt outbreak[J]. Scientific Reports, 2017, 7(1): 343. DOI:10.1038/s41598-017-00472-6 |
| [47] |
LEE C G, ⅡDA T, INOUE Y, MURAMOTO Y, WATANABE H, NAKAHO K, OHKUMA M. Prokaryotic communities at different depths between soils with and without tomato bacterial wilt but pathogen-present in a single greenhouse[J]. Microbes and Environments, 2017, 32(2): 118-124. DOI:10.1264/jsme2.ME16136 |
| [48] |
SHIOMI Y, NISHIYAMA M, ONIZUKA T, MARUMOTO T. Comparison of bacterial community structures in the rhizoplane of tomato plants grown in soils suppressive and conducive towards bacterial wilt[J]. Applied and Environmental Microbiology, 1999, 65(9): 3996-4001. DOI:10.1002/(SICI)1097-0290(19990905)64:5<620::AID-BIT13>3.3.CO;2-N |
| [49] |
ZHANG H, WANG R, CHEN S, QI G, HE Z, ZHAO X. Microbial taxa and functional genes shift in degraded soil with bacterial wilt[J]. Scientific Reports, 2017, 7: 39911. DOI:10.1038/srep39911 |
| [50] |
赵文宗, 郑旭阳, 张映卿, 钟川, 阳燕娟, 于文进. 两种砧木对樱桃番茄青枯病抗性及根际微生物数量的影响[J]. 广西植物, 2019, 39(10): 1307-1316. DOI:10.11931/guihaia.gxzw201812009 ZHAO W Z, ZHENG X Y, ZHANG Y Q, ZHONG C, YANG Y J, YU W J. Effects of two rootstocks on bacterial wilt resistance and rhizospheric microorganisms of cherry tomato[J]. Guihaia, 2019, 39(10): 1307-1316. DOI:10.11931/guihaia.gxzw201812009 |
| [51] |
刘娜, 朱为民, 鲁博, 何璐, 朱龙英, 张辉. 嫁接番茄抗青枯病特性及对根际土壤微生物数量的影响[J]. 上海农业学报, 2013, 29(5): 79-83. DOI:10.3969/j.issn.1000-3924.2013.05.018 LIU N, ZHU W M, LU B, HE L, ZHU L Y, ZHANG H. Effects of grafting on the bacterial wilt resistance and rhizospheric microorganism population of tomato[J]. Acta Agriculturae Shanghai, 2013, 29(5): 79-83. DOI:10.3969/j.issn.1000-3924.2013.05.018 |
| [52] |
WEN T, ZHAO M, LIU T, HUANG Q, YUAN J, SHEN Q. High abundance of Ralstonia solanacearum changed tomato rhizosphere microbiome and metabolome[J]. BMC Plant Biology, 2020, 20: 166. DOI:10.1186/s12870-020-02365-9 |
| [53] |
GONG C, CHENG M Z, LI J F, CHEN H Y, ZHANG Z Z, QI H N, ZHANG Y, LIU J Y, CHEN X L, WANG A X. Alpha-subunit of the chloroplast atp synthase of tomato reinforces the resistance to grey mold and broad-spectrum resistance in transgenic tobacco[J]. Phytopathology, 2020, 111(3). DOI:10.1094/PHYTO-06-20-0242-R |
| [54] |
RODRIGUEZ P A, ROTHBALLER M, CHOWDHURY S P, NUSSBAUMER T, GUTJAHR C, FALTER-BRAUN P. Systems biology of plant-microbiome interactions[J]. Molecular Plant, 2019, 12(6). DOI:10.1016/j.molp.2019.05.006 |
| [55] |
WANG R, ZHANG H, SUN L, QI G, CHEN S, ZHAO X. Microbial community composition is related to soil biological and chemical properties and bacterial wilt outbreak[J]. Scientific Reports, 2017(71): 343. DOI:10.1038/s41598-017-00472-6 |
| [56] |
YANG H, LI J, XIAO Y, GU Y, LIU H, LIANG Y, LIU X, HU J, MENG D, YIN H. An integrated insight into the relationship between soil microbial community and tobacco bacterial wilt disease[J]. Frontiers in Microbiology, 2017, 8: 2179. DOI:10.3389/fmicb.2017.02179 |
| [57] |
刘琼光, 杨艳. 番茄品种抗性与青枯菌和土壤微生物的关系[J]. 仲恺农业技术学院学报, 2006(3): 31-34. LIU Q G, YANG Y. The relationship between tomato resistance and the quantity of Ralstonia solanacearum and rhizosphere microbes[J]. Journal of Zhongkai University of Agriculture and Technology, 2006(3): 31-34. |
| [58] |
宫超, 李涛, 黎振兴, 衡周, 李植良, 孙保娟. 健康的与感染青枯病的番茄砧木根际细菌群落结构特征[J]. 园艺学报, 2019(S1): 26-35. GONG C, LI T, LI Z X, HENG Z, LI Z L, SUN B J. Characteristics of bacterial community structure in rhizosphere of healthy and bacterial wilt-diseased tomato rootstocks[J]. Acta Horticulturae Sinica, 2019(S1): 2635. |
| [59] |
ANTONIOU A, TSOLAKIDOU M D, STRINGLIS I A, PANTELIDES I S. Rhizosphere microbiome recruited from a suppressive compost improves plant fitness and increases protection against vascular wilt pathogens of tomato[J]. Frontiers in Plant Science, 2017, 8: 2022. DOI:10.3389/fpls.2017.02022 |
| [60] |
DILKES N B, JONES D L, FARRAR J. Temporal dynamics of carbon partitioning and rhizodeposition in wheat[J]. Plant Physiology, 134(2): 706-15. DOI:10.1104/pp.103.032045 |
| [61] |
SCHMITT S, TSAI P, BELL J, FROMONT J, ILAN M, LINDQUIST N, PEREZ T, RODRIGO A, SCHUPP P J, VACELET J, WEBSTER N, HENTSCHEL U, TAYLOR M W. Assessing the complex sponge microbiota: core, variable and speciesspecific bacterial communities in marine sponges[J]. The ISME Journal, 2012, 6(3): 564-576. DOI:10.1038/ismej.2011.116 |
| [62] |
SCHARF B E, HYNES M F, ALEXANDRE G M. Chemotaxis signaling systems in model beneficial plant-bacteria associations[J]. Plant Molecular Biology, 2016, 90(6): 549-59. DOI:10.1007/s11103-016-0432-4 |
| [63] |
LEACH J E, TRIPLETT L R, ARGUESO C T, PANKAJ T. Communication in the phytobiome[J]. Cell, 2017, 169(4): 587-596. DOI:10.1016/j.cell.2017.04.025 |
| [64] |
CASTRILLO G, TEIXEIRA P J P L, PAREDES S H, LAW T F, DE LORENZO L, FELTCHER M E, FINKEL O M, BREAKFIELD N W, MIECZKOWSKI P, JONES C D, PAZ-ARES J, DANGL J L. Root microbiota drive direct integration of phosphate stress and immunity[J]. Nature, 2017, 543: 513-518. DOI:10.1038/nature21417 |
| [65] |
CHAPARRO J M, BADRI D V, VIVANCO J M. Rhizosphere microbiome assemblage is affected by plant development[J]. The ISME Journal, 2014, 8: 790-803. DOI:10.1038/ismej.2013.196 |
| [66] |
KIM J F, JEONG H, PARK S Y, KIM S B, PARK Y K, CHOI S K, RYU C M, HUR C G, GHIM S Y, OH T K, KIM J J, PARK C S, PARK S H. Genome sequence of the polymyxin-producing plant-probiotic rhizobacterium Paenibacillus polymyxa E681[J]. Journal of Bacteriology, 2010, 192(22): 6103-6104. DOI:10.1128/JB.00983-10 |
| [67] |
SHEN Z, RUAN Y, CHAO X, ZHANG J, LI R, SHEN Q. Rhizosphere microbial community manipulated by 2 years of consecutive biofertilizer application associated with banana Fusarium wilt disease suppression[J]. Biology and Fertility of Soils, 2015, 51: 553-562. DOI:10.1007/s00374-015-1002-7 |
| [68] |
CHAPARRO J M, SHEFLIN A M, MANTER D K, VIVANCO J M. Manipulating the soil microbiome to increase soil health and plant fertility[J]. Biology and Fertility of Soils, 2012, 48(5): 489-499. DOI:10.1007/s00374-012-0691-4 |
| [69] |
QIU M, ZHANG R, XUE C, ZHANG S, LI S, ZHANG N, SHEN Q. Application of bio-organic fertilizer can control Fusarium wilt of cucumber plants by regulating microbial community of rhizosphere soil[J]. Biology and Fertility of Soils, 2012, 48: 807-816. DOI:10.1007/s00374-012-0675-4 |
(责任编辑 杨贤智)
 2021, Vol. 48
2021, Vol. 48