文章信息
基金项目
- 广东省农业科学院科技人才引进专项(R2021YJ-QG008);广东省农业科学院农业生物基因研究中心创新基金重点项目
作者简介
-
余界石,博士,广东省农业科学院青年科技骨干,美国南达科他州立大学微生物专业博士,现任职于广东省农业科学院农业生物基因研究中心微生物资源研究室。主要从事丁型流感病毒、猪流行性腹泻病毒等畜禽传染病病原生物学的研究以及新型动物疫苗的研发与应用等研究。在国际期刊发表论文17篇,其中以第一作者在《npj Vaccines》《Emerging Microbes & Infections》《Journal of Virology》《Journal of Medical Virology》《mSphere》等学术期刊上发表SCI论文8篇。主持国家自然科学基金青年项目、广东省农业科学院科技人才引进专项、广东省农业科学院农业生物基因研究中心创新基金重点项目各1项。
余界石(1987—),男,博士,研究方向为流感病毒,E-mail:jieshi_yu@outlook.com; 魏文康,博士,研究员,硕士研究生导师,现任广东省农业科学院农业生物基因研究中心主任,兼任广东省畜禽疫病监测与防控共性关键技术研发创新团队首席专家、国家动物疫病预防控制中心规模养殖场疫病净化专家。主要从事动物疫病诊断与综合防控、微生物资源等研究及科技管理工作。先后承担国家、广东省重大课题专项、省部市级各级课题50余项,参加国家“十三五”重点研发项目2项。在《Emerging Infectious Diseases》(Top1区)、《Transboundary and Emerging Diseases》(Top1区)等科技期刊上发表论文80余篇,其中SCI论文20余篇,参与出版著作4部。获得省部级成果奖励13项,其中中华农业科技奖1项、广东省科技进步奖一等奖1项、二等奖2项,广东省农业技术推广奖一等奖2项;获得授权专利、国家版权局软件著作权13项、兽药证书2件,转化产品20余个已在生产中广泛应用,获经济社会效益显著。2016年被评为广东省农业科学院“十二五”科技创新先进个人;2017年获广东省畜牧兽医学会首届广东省杰出畜牧兽医科技工作者称号.
通讯作者
- 魏文康(1971—),男,博士,研究员,研究方向为预防兽医,E-mail:weiwenkang@gdaas.cn.
文章历史
- 收稿日期:2022-09-09
流感病毒属于正粘病毒科,包含4个属,分别是α流感病毒属、β流感病毒属、γ流感病毒属和δ流感病毒属,其中每个属只包括1种类型,分别为甲型流感病毒、乙型流感病毒、丙型流感病毒和丁型流感病毒[1]。甲型流感病毒是所有类型流感病毒中最具攻击性和最值得关注的病毒,其宿主范围最广,感染造成的发病率和死亡率最高,并且可以引发严重的呼吸道疾病,是季节性流感和以往所有流感大流行的元凶[2]。乙型流感病毒感染可引起轻度至重度呼吸道疾病,也会造成季节性流感,但通常仅限于人类[3]。一般情况下,丙型流感病毒感染只引起轻微的呼吸道症状[4]。虽然大多数人在幼儿时期就产生了丙型流感病毒的抗体(表明曾接触过这种病毒),但由丙型流感病毒引起的临床疾病相关报告相对较少,因此对其的关注度也相对较低。
丁型流感病毒是正粘病毒家族的最新成员。2011年,美国科研人员首次从表现出流感样症状的猪只中分离出1株新型流感病毒。由于该病毒与人类丙型流感病毒有50% 的基因同源性,因此其早期被认为是一种新亚型的猪丙型流感病毒[5]。进一步研究发现,这种新型流感病毒在与甲型、乙型或丙型流感病毒共同感染的情况下不会发生基因组重配,而且其基因组组成和一些生物学特性与已知类型的流感病毒存在明显差异[6]。第一,在受体识别和结合方面,丁型流感病毒除了与丙型流感病毒一样结合唾液酸分子Neu5, 9Ac2作为进入细胞的受体,其还可以结合唾液酸分子Neu5Gc9Ac作为它的受体[7-8]。丁型流感病毒表现出更强和更广谱的受体结合能力[7]。第二,丁型流感病毒基因组节段M的剪切方式不同于其他类型流感病毒。甲型和乙型流感病毒的M1蛋白由未拼接的mRNA转录而成,而丙型和丁型流感病毒的M1蛋白由拼接的mRNA转录而成[6, 9]。但丙型流感病毒M基因组节段剪接转录产生1个终止密码子形成M1 mRNA,而丁型流感病毒M基因组节段的剪接转录产生1个额外的4- 氨基酸肽,添加到其M1的外显子中[9]。目前,该额外的4- 氨基酸肽在丁型流感病毒生命周期中的作用还未阐明。第三,丁型流感病毒具有独特的耐热、耐酸特性,在4种类型流感病毒中最稳定[10]。国际病毒分类委员会于2016年正式将该病毒命名为丁型流感病毒[9]。
大量研究证实,丁型流感病毒以牛为主要天然宿主,尚未发现其与人类的临床感染有关[11]。但有机会接触这种病毒的特殊人群(如牛场工作人员)具有很高水平的针对该病毒的抗体阳性率[12]。近年来,随着丁型流感病毒在牛群和猪群中的检出率不断升高,其会否成为新的人畜共患病病毒备受关注。目前,根据病毒主要表面抗原HEF的序列差异,已发现5个遗传演化谱系的丁型流感病毒,分别是D/OK、D/660、D/Yama2016、D/Yama2019和D/CA2019[13]。在欧洲和美洲,主要流行D/OK、D/660和D/CA2019谱系的丁型流感病毒;D/Yama2016和D/Yama2019谱系的丁型流感病毒主要在日本流行[14-16]。对许多国家来说,丁型流感病毒的流行状况依然不清楚。
我国研究人员于2014、2017、2021、2022年陆续报道了丁型流感病毒的核酸阳性病例。但丁型流感病毒在我国的总体流行情况、遗传多样性信息,以及对我国畜牧业的影响依然不明确。本综述系统性地总结了丁型流感病毒在我国的分布情况和研究进展,并且分析了丁型流感病毒对我国畜牧业的潜在威胁,为我国相关科研人员深入研究有潜在致病性的丁型流感病毒提供参考和借鉴。
1 丁型流感病毒流行情况及致病性概述 1.1 丁型流感病毒具有宽泛的宿主范围且分布广泛虽然丁型流感病毒的首次分离源于猪,但越来越多的研究发现,该病毒主要在牛群中流行[6, 17-19]。研究人员在牛群中发现了丁型流感病毒的高阳性率(>80%) [9]。因此,牛被认为是丁型流感病毒的主要天然宿主[6, 9, 11]。此外,回顾性血清学研究发现,最早于2003年的牛血清中检测到丁型流感病毒抗体[20],说明该病毒存在的时间可能比较长。除了牛和猪外,在马、绵羊、山羊、骆驼、野猪和鹿血清中也检测到丁型流感病毒抗体[5, 9, 21-26]。马来西亚研究人员在养禽场的气溶胶中检测到丁型流感病毒基因组核酸[27]。可见,丁型流感病毒具有宽泛的宿主范围。到目前为止,丁型流感病毒已广泛分布于全球数十个国家,包括美洲的美国、墨西哥和加拿大,亚洲的中国、日本和土耳其,欧洲的法国、爱尔兰、意大利、卢森堡、英国等,以及非洲的埃塞俄比亚、多哥、摩洛哥、肯尼亚、纳米比亚等[9, 28]。
1.2 丁型流感病毒是牛呼吸系统疾病综合征的重要关联因子考虑到丁型流感病毒的广泛分布与传播,充分了解其致病性显得尤为重要。宏基因组学研究表明,丁型流感病毒与牛呼吸系统疾病综合征密切相关[29-31]。牛呼吸系统疾病综合征是一种多因子疾病,由宿主、病原和环境因素之间的复杂相互作用引起[32]。通常在有压力和不利环境条件下的牛更容易受到原发性病毒感染,而病毒感染可能继发细菌入侵,从而导致严重疾病甚至死亡[32]。目前与牛呼吸系统疾病综合征直接关联的病毒包括牛病毒性腹泻病毒、牛传染性鼻气管炎病毒、牛呼吸道实验室合胞病毒、牛副流感病毒3型和丁型流感病毒[33]。研究表明,丁型流感病毒可在牛中引起轻度至中度呼吸道症状,而且病毒能够在牛的上、下呼吸道中复制[33-35]。一项最新研究发现,与单独感染相比,丁型流感病毒和牛支原体合并感染会增加犊牛呼吸道疾病的严重程度[36-37]。丁型流感病毒感染促进牛分枝杆菌在下呼吸道的定殖,增加白细胞向气道腔的汇集[36]。由于牛是丁型流感病毒的自然宿主,因此在牛群中加强对该病毒的持续监测和风险评估十分必要。
1.3 丁型流感病毒具备潜在人畜共患风险丁型流感病毒是否具备人畜共患潜能仍无定论,迄今为止没有直接证据表明丁型流感病毒可在人体内致病或在人群之间传播。然而,有实验证明丁型流感病毒能够通过豚鼠和雪貂的直接接触传播,而这两种动物都是已建立的人类流感病毒感染模型[5, 38]。此外,美国早期的一项血清学调查显示,1.3%(n=316)的普通人群具有丁型流感病毒抗体[5]。来自美国佛罗里达州的一项血清学研究显示,在有牛接触史的工人血清中,丁型流感病毒抗体阳性率高达98%[12]。意大利一项更全面的血清学研究显示,2005—2017年期间,所调查区域内携带丁型流感病毒抗体的人群占比5.1%~46.0% 不等[39]。美国环境公共卫生学相关研究发现,在公共场所(如医院和机场)收集的气溶胶样品中存在丁型流感病毒基因组核酸[40-41]。上述研究结果提示丁型流感病毒具有人畜共患风险。虽然丁型流感病毒被证明只在牛中引起轻度或中度呼吸道疾病,但是丁型流感病毒在全球数十个国家持续传播,并具有人畜共患传播的可能性,因此,系统性地分析和了解丁型流感病毒的流行与演化及致病性与传播,对评估和预防该病毒对畜牧业的危害至关重要。
2 丁型流感病毒在中国的流行与分布继美国研究人员在俄克拉荷马州首次分离出丁型流感病毒[5]后不久,美国其他地区[18, 21, 42]以及一些国家,如中国[23, 43]、日本[44]、意大利[19, 45]、法国[17]、加拿大[21]、爱尔兰[46]、卢森堡[47]、英国[48]和埃塞俄比亚[49],也陆续报道了该病毒的核酸或血清阳性病例。20 14年,研究人员在我国山东省的牛群中检测到丁型流感病毒。研究人员从15个养牛场的表观健康牛群中采集了453份鼻拭子样本,通过RT-PCR检测,发现其中有3份(0.66%)呈丁型流感病毒核酸阳性,并且获得了它们的基因组序列[43]。这些毒株分别被命名为D/bovine/Shandong/Y127/2014、D/bovine/Shandong/Y125/2014、D/bovine/Shandong/Y217/2014。这是继欧洲和美洲之后,首次在亚洲报道丁型流感病毒的阳性病例。
随后,科研人员在我国南方牛群中也检测到丁型流感病毒。该研究团队收集了来自伴有临床症状的不同动物共607份临床样本和250份无症状动物的鼻拭子样本[23]。被采集样本的动物包括牛(奶牛、黄牛和水牛)、猪和山羊,主要分布于广东省广州、清远、河源和江门4市的16个农场[23]。检测结果显示,奶牛、黄牛和猪鼻拭子样品中丁型流感病毒的核酸阳性率分别为12.8%(20/156)、7.3%(4/55) 和36.8%(7/19)[23]。该团队研究人员还首次在奶牛、水牛和山羊的血清样本中检测到丁型流感病毒基因组核酸,阳性率分别为7.8%(15/193)、5.9%(3/51) 和33.8%(27/80);他们还发现猪肺样本中丁型流感病毒阳性率也比较高、为28.9%(13/45)[23]。
为了监测丁型流感病毒在我国的流行与分布,研究人员检测了来自安徽(124)、广东(88)、福建(460)、山东(17)、江苏(235) 和河南(21)7个省份共945份具有呼吸道疾病的猪肺组织样本,结果发现,其中6份呈丁型流感病毒核酸阳性,这些毒株分别被命名为D/swine/China/Fujian706/2018、D/swine/China/Anhui7/2018、D/swine/China/Anhui8/2018、D/swine/China/Anhui119/2018、D/swine/China/JiangsuQF54/2018和D/swine/China/JiangsuQF52/2018[50]。该研究结果表明,在我国南方的安徽、江苏和福建省份的猪群中存在丁型流感病毒流行。尽管丁型流感病毒最早是从猪身上被分离到,但其在猪群中的流行率低于其在牛群中的流行率。该研究首次报道了丁型流感病毒在亚洲的猪群中流行。
在最新的一项丁型流感病毒的监测研究中,研究人员从广东的一个肉牛屠宰场采集了250份表观健康牛的鼻拭子样本,其中10份呈丁型流感病毒核酸阳性[51]。这些丁型流感病毒阳性样品分别来自甘肃、内蒙古、安徽、陕西、辽宁、宁夏、河北、青海和广东的养牛场[51]。以上研究结果提示,丁型流感病毒可能已在我国广泛流行。
3 丁型流感病毒中国毒株的遗传多样性丁型流感病毒与丙型流感病毒一样含有7种基因组节段[5],其中最长的3个节段分别编码病毒聚合酶复合体的3个组分PB2、PB1和P3,第4长节段编码病毒唯一的表面糖蛋白HEF,第5长节段编码病毒核蛋白NP,最短的两个基因组节段(M和NS)分别通过不同的策略产生病毒基质蛋白M1、离子通道蛋白M2以及非结构蛋白NS1与NS2。HEF是丁型流感病毒中最易变异的基因组节段,主要用于该病毒的系统演化分析。根据HEF的序列差异,丁型流感病毒被分为5个不同的遗传演化谱系,分别为D/OK、D/660、D/Yama2016、D/Yama2019和D/CA2019。
D/OK谱系是目前被报道最多的谱系[9]。在D/OK谱系中,来自中国的毒株与来自美国和意大利的毒株在遗传演化关系上彼此分离,表明不同国家的丁型流感病毒可能存在亚谱系。如图 1所示,D/OK谱系的丁型流感病毒中国毒株自成一个小分支[23, 43, 50]。此外,来自美国和意大利的D/OK谱系中的猪源毒株和牛源毒株通常聚集在一起,表明D/OK谱系的丁型流感病毒在猪和牛之间可能相互传播。然而,已报道的丁型流感病毒中国毒株表现出非常低的遗传多样性(遗传距离 < 0.005)。在D/660谱系中,来自美国的毒株不断分化,演化出新的亚谱系毒株(图 1)。最近在日本出现的丁型流感病毒株被认为属于新的遗传演化谱系,分别是D/Yama2016和D/Yama2019 (图 1),这两个谱系的毒株只存在于日本,并且与其他国家流行的D/OK和D/660谱系的毒株有很大差异[9, 16]。目前D/CA2019谱系的丁型流感病毒只在美国被报道[13]。值得重视的是,研究人员最近在中国牛群中发现了D/Yama2019谱系的丁型流感病毒[51]。该研究是首次在日本以外的国家检测到D/Yama2019谱系的丁型流感病毒。这说明丁型流感病毒中国毒株也存在遗传多样性。
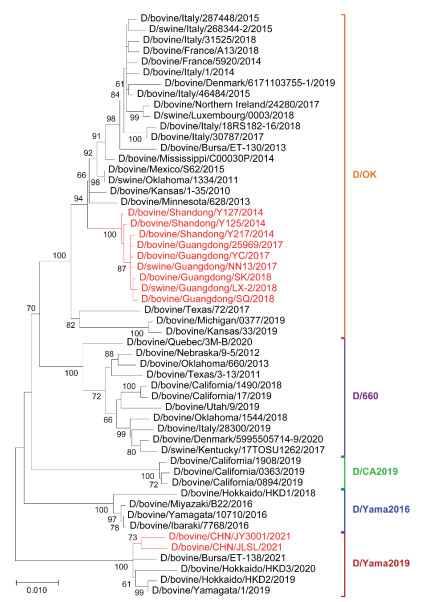
|
| 红色部分为丁型流感病毒中国毒株 Influenza D virus strains from China are labelled by red color 图 1 丁型流感病毒HEF基因组节段遗传演化分析 Fig. 1 Phylogenetic analysis of HEF segments of influenza D virus |
在美洲和欧洲,D/OK和D/660谱系的丁型流感病毒在牛群中共同传播且频繁重配。不同谱系丁型流感病毒之间的基因组重配加快了病毒的演化与变异。新产生的变异毒株会突破原有的群体免疫,对农业动物健康构成一定威胁。根据已有研究结果,未发现丁型流感病毒中国毒株之间发生了基因组重配[51]。但由于已报道的毒株数量有限,目前还不能确定基因组重配事件是否真的没有发生。尤其是当D/Yama2019谱系的丁型流感病毒也在中国牛群中流行时,这会大大增加其与D/OK谱系的丁型流感病毒之间发生基因组重配的概率。我们推测,这两个谱系之间的丁型流感病毒频繁重组可能导致新的抗原变异株产生,进而对动物健康构成潜在威胁。因此,加强对中国丁型流感病毒的监测研究十分必要。
4 丁型流感病毒对我国畜牧养殖业的潜在风险分析 4.1 丁型流感病毒在我国的总体流行情况还不明确在我国,丁型流感病毒最早于2014年在山东的养牛场被发现[43]。随后,研究人员在南方一些省份(广东、江苏、安徽和福建)的牛群或猪群中检测到该病毒[23, 50]。在最新的一项丁型流感病毒流行病学调查研究中,研究人员通过屠宰场采集了来自不同省份的牛鼻拭子样品,发现来自甘肃、内蒙古、安徽、陕西、辽宁、宁夏、河北、青海和广东的养牛场存在丁型流感病毒核酸阳性的牛[51]。但以上数据不足以反映丁型流感病毒在我国的总体流行情况。我国的养牛产业(包括奶牛和肉牛)主要分布在四大区域:西北产区(新疆、甘肃、陕西和宁夏)、西南产区(四川、重庆、云南、贵州和广西)、东北产区(内蒙古、吉林和黑龙江)和中原产区(山东、河南、河北和安徽)。从目前仅有的流行病学调查数据来看,四大产区可能都存在丁型流感病毒的分布或传播。此外,伴随着不同产区之间、规模化养牛场与小型牛场或散养场之间、产地与消费地之间活牛的频繁交易及运输,丁型流感病毒很有可能广泛流行于全国各地的牛群中。为了明确丁型流感病毒在我国的总体流行情况,亟需更大规模的流行病学调查研究。
4.2 丁型流感病毒对我国畜牧动物健康的影响依然不清楚已报道的牛丁型流感病毒监测研究表明,与健康牛相比,患有呼吸道疾病(包括肺炎)的牛更容易检测到丁型流感病毒[9, 11]。一项早期的丁型流感病毒监测研究发现,在55头患有呼吸道疾病综合征的犊牛中有23.6% 出现了丁型流感病毒感染,但在相同牛场内的82头健康犊牛中只有2.4% 出现了丁型流感病毒感染[18]。在我国牛群中,无症状牛的丁型流感病毒阳性率普遍比患病牛低[23, 41, 43]。值得注意的是,我国研究人员在患病的牛和山羊的血清样本中检测到丁型流感病毒基因组核酸,这意味着该病毒感染可能会诱发病毒血症,并随着机体循环系统侵染其他组织器官[23]。此外,该研究团队还在山羊直肠拭子中检测到丁型流感病毒,表明丁型流感病毒可能类似于甲型和乙型流感病毒可以在动物肠道内复制[23]。为了明确丁型流感病毒的临床致病性,有研究团队对犊牛鼻内接种了丁型流感病毒,与对照相比,感染组犊牛均表现出呼吸道感染的相关症状,但都较轻[35]。然而,最新研究证实,与丁型流感病毒或牛支原体单独感染相比,两者合并感染会加重犊牛呼吸道症状[36-37]。总的来说,丁型流感病毒单独感染会引起牛轻微的呼吸到症状,但感染牛可能更易被其他病原侵袭,进而导致严重的呼吸道症状。由于牛是丁型流感病毒的自然宿主和主要传播源,因此应加强对牛群该病毒的持续监测和风险评估。
丁型流感病毒分布于全球各地,可感染多种动物物种[9]。我国和其他国家的研究人员都在猪肺样本中检测到丁型流感病毒基因组核酸,表明该病毒可以感染猪下呼吸道[23, 45, 50]。对野猪进行该病毒感染实验,发现丁型流感病毒能够在下呼吸道复制,并且在受感染的野猪中观察到丁型流感病毒血症[24]。由于流感病毒很容易从猪传播给人类和其他动物物种,因此需要进一步研究丁型流感病毒在猪中的传播与发病机制。丁型流感病毒还可以感染羊、马、骆驼和鹿[9, 26],但是对这些动物的致病风险依然不清楚。
5 展望近年来,丁型流感病毒越来越受到病毒学领域研究者的关注。首先,丁型流感病毒与甲型流感病毒相似,具有比乙型和丙型流感病毒更宽泛的宿主谱。这说明丁型流感病毒可能与甲型流感病毒类似,具备快速传播和演化的能力。其次,丁型流感病毒在四大类型流感病毒中表现出许多独特性,例如其拥有优越的耐热耐酸特性[10]、可识别多样化的唾液酸分子作为其进入细胞的受体[7-8]、特殊的基因组组成与配置模式[52]、不一样的M1蛋白产生方式[6]等。此外,丁型流感病毒表现出人畜共患的潜能。我国是畜牧业大国,对有潜在风险的新发丁型流感病毒进行前瞻性研究十分有必要。目前,我国相关研究人员对丁型流感病毒的研究十分有限,只在2014、2017、2021、2022年分别报道了丁型流感病毒的核酸阳性病例。丁型流感病毒在我国的总体流行情况、遗传多样性信息,以及对我国动物养殖业的影响依然不明确,而且至今我国还未有分离出丁型流感病毒的报道。这严重影响了相关科研人员对丁型流感病毒的深入研究。要进一步了解丁型流感病毒对养殖业的影响,研究人员需要对其进行较大规模和较大范围的流行病学调查,包括我国丁型流感病毒的总体阳性率、地域分布情况及与疾病的关联性等。同时,通过对某一区域丁型流感病毒的跟踪性调查,可以进一步了解其传播动力学及其感染对养殖动物的影响;丁型流感病毒中国毒株的分离鉴定及生物学特性研究(如病毒致病性和跨宿主传播能力)也十分必要,这些研究将为该病毒的防控提供指导。
| [1] |
HUTCHINSON E C. Influenza virus[J]. Trends in Microbiology, 2018, 26(9): 809-810. DOI:10.1016/j.tim.2018.05.013 |
| [2] |
KRAMMER F, SMITH G J D, FOUCHIER R A M, PEIRIS M, KEDZIERSKA K, DOHERTY P C, PALESE P, SHAW M L, TREANOR J, WEBSTER R G, GARCIA-SASTRE A. Influenza[J]. Nature Reviews Disease Primers, 2018, 4(1): 3. DOI:10.1038/s41572-018-0002-y |
| [3] |
BERGERON H C, RENEER Z B, ARORA A, REYNOLDS S, NAGY T, TRIPP R A. Influenza B virus (IBV) immune-mediated disease in C57BL/6 mice[J]. Vaccines (Basel), 2022, 10(9). DOI:10.3390/vaccines10091440 |
| [4] |
MATSUZAKI Y, SUGAWARA K, ABIKO C, IKEDA T, AOKI Y, MIZUTA K, KATSUSHIMA N, KATSUSHIMA F, KATSUSHIMA Y, ITAGAKI T, SHIMOTAI Y, HONGO S, MURAKI Y, NISHIMURA H. Epidemiological information regarding the periodic epidemics of influenza C virus in Japan (1996-2013) and the seroprevalence of antibodies to different antigenic groups[J]. Journal of Clinical Virology, 2014, 61(1): 87-93. DOI:10.1016/j.jcv.2014.06.017 |
| [5] |
HAUSE B M, DUCATEZ M, COLLIN E A, RAN Z, LIU R, SHENG Z, ARMIEN A, KAPLAN B, CHAKRAVARTY S, HOPPE A D, WEBBY R J, SIMONSON R R, LI F. Isolation of a novel swine influenza virus from Oklahoma in 2011 which is distantly related to human influenza C viruses[J]. PLOS Pathogens, 2013, 9(2): e1003176. DOI:10.1371/journal.ppat.1003176 |
| [6] |
HAUSE B M, COLLIN E A, LIU R, HUANG B, SHENG Z, LU W, WANG D, NELSON E A, LI F. Characterization of a novel influenza virus in cattle and swine: proposal for a new genus in the Orthomyxoviridae family[J]. mBio, 2014, 5(2): e00031-00014. DOI:10.1128/mBio.00031-14 |
| [7] |
LIU R, SREENIVASAN C, YU H, SHENG Z, NEWKIRK S J, AN W, SMITH D F, CHEN X, WANG D, LI F. Influenza D virus diverges from its related influenza C virus in the recognition of 9-O-acetylated N-acetyl- or N-glycolyl-neuraminic acid-containing glycan receptors[J]. Virology, 2020, 545: 16-23. DOI:10.1016/j.virol.2020.02.007 |
| [8] |
SONG H, QI J, KHEDRI Z, DIAZ S, YU H, CHEN X, VARKI A, SHI Y, GAO G F. An open receptor-binding cavity of hemagglutinin-esterase-fusion glycoprotein from newly-identified influenza D virus: basis for its broad cell tropism[J]. PLOS Pathogens, 2016, 12(1): e1005411. DOI:10.1371/journal.ppat.1005411 |
| [9] |
YU J, LI F, WANG D. The first decade of research advances in influenza D virus[J]. Journal of General Virology, 2021, 102(1): e001529. DOI:10.1099/jgv.0.001529 |
| [10] |
YU J, HIKA B, LIU R, SHENG Z, HAUSE B M, LI F, WANG D. The hemagglutinin-esterase fusion glycoprotein is a primary determinant of the exceptional thermal and acid stability of influenza D virus[J]. mSphere, 2017, 2(4): e00254-17. DOI:10.1128/mSphere.00254-17 |
| [11] |
LIU R, SHENG Z, HUANG C, WANG D, LI F. Influenza D virus[J]. Current Opinion in Virology, 2020, 44: 154-161. DOI:10.1016/j.coviro.2020.08.004 |
| [12] |
WHITE S K, MA W, MCDANIEL C J, GRAY G C, LEDNICKY J A. Serologic evidence of exposure to influenza D virus among persons with occupational contact with cattle[J]. Journal of Clinical Virology, 2016, 81: 31-33. DOI:10.1016/j.jcv.2016.05.017 |
| [13] |
HUANG C, YU J, HAUSE B M, PARK J Y, SREENIVASAN C, UPRETY T, SHENG Z, WANG D, LI F. Emergence of new phylogenetic lineage of influenza D virus with broad antigenicity in California, United States[J]. Emerging Microbes & Infections, 2021, 10(1): 739-742. DOI:10.1080/22221751.2021.1910078 |
| [14] |
MEKATA H, YAMAMOTO M, HAMABE S, TANAKA H, OMATSU T, MIZUTANI T, HAUSE B M, OKABAYASHI T. Molecular epidemiological survey and phylogenetic analysis of bovine influenza D virus in Japan[J]. Transboundary and Emerging Diseases, 2018, 65(2): e355-e360. DOI:10.1111/tbed.12765 |
| [15] |
HAYAKAWA J, MASUKO T, TAKEHANA T, SUZUKI T. Genetic and antigenic characterization and retrospective surveillance of bovine influenza D viruses identified in Hokkaido, Japan from 2018 to 2020[J]. Viruses, 2020, 12(8): 877. DOI:10.3390/v12080877 |
| [16] |
MURAKAMI S, SATO R, ISHIDA H, KATAYAMA M, TAKENAKA-UEMA A, HORIMOTO T. Influenza D virus of new phylogenetic lineage, Japan[J]. Emerging Infectious Diseases, 2020, 26(1): 168-171. DOI:10.3201/eid2601.191092 |
| [17] |
DUCATEZ M F, PELLETIER C, MEYER G. Influenza D virus in cattle, France, 2011-2014[J]. Emerging Infectious Diseases, 2015, 21(2): 368-371. DOI:10.3201/eid2102.141449 |
| [18] |
FERGUSON L, ECKARD L, EPPERSON W B, LONG L P, SMITH D, HUSTON C, GENOVA S, WEBBY R, WAN X F. Influenza D virus infection in Mississippi beef cattle[J]. Virology, 2015, 486: 28-34. DOI:10.1016/j.virol.2015.08.030 |
| [19] |
CHIAPPONI C, FACCINI S, DE MATTIA A, BAIONI L, BARBIERI I, ROSIGNOLI C, NIGRELLI A, FONI E. Detection of influenza D virus among swine and cattle, Italy[J]. Emerging Infectious Diseases, 2016, 22(2): 352-354. DOI:10.3201/eid2202.151439 |
| [20] |
LUO J, FERGUSON L, SMITH D R, WOOLUMS A R, EPPERSON W B, WAN X F. Serological evidence for high prevalence of influenza D viruses in cattle, Nebraska, United States, 2003-2004[J]. Virology, 2017, 501: 88-91. DOI:10.1016/j.virol.2016.11.004 |
| [21] |
QUAST M, SREENIVASAN C, SEXTON G, NEDLAND H, SINGREY A, FAWCETT L, MILLER G, LAUER D, VOSS S, POLLOCK S, CUNHA C W, CHRISTOPHER-HENNINGS J, NELSON E, LI F. Serological evidence for the presence of influenza D virus in small ruminants[J]. Veterinary Microbiology, 2015, 180(3/4): 281-285. DOI:10.1016/j.vetmic.2015.09.005 |
| [22] |
SALEM E, COOK E A J, LBACHA H A, OLIVA J, AWOUME F, APLOGAN G L, HYMANN E C, MULOI D, DEEM S L, ALALI S, ZOUAGUI Z, FEVRE E M, MEYER G, DUCATEZ M F. Serologic evidence for influenza C and D virus among ruminants and camelids, Africa, 1991-2015[J]. Emerging Infectious Diseases, 2017, 23(9): 1556-1559. DOI:10.3201/eid2309.170342 |
| [23] |
ZHAI S L, ZHANG H, CHEN S N, ZHOU X, LIN T, LIU R, LV D H, WEN X H, WEI W K, WANG D, LI F. Influenza D virus in animal species in Guangdong Province, Southern China[J]. Emerging Infectious Diseases, 2017, 23(8): 1392-1396. DOI:10.3201/eid2308.170059 |
| [24] |
FERGUSON L, LUO K, OLIVIER A K, CUNNINGHAM F L, BLACKMON S, HANSON-DORR K, SUN H, BAROCH J, LUTMAN M W, QUADE B, EPPERSON W, WEBBY R, DELIBERTO T J, WAN X F. Influenza D virus infection in feral swine populations, United States[J]. Emerging Infectious Diseases, 2018, 24(6): 1020-1028. DOI:10.3201/eid2406.172102 |
| [25] |
OLIVA J, EICHENBAUM A, BELIN J, GAUDINO M, GUILLOTIN J, ALZIEU J P, NICOLLET P, BRUGIDOU R, GUENEAU E, MICHEL E, MEYER G, DUCATEZ M F. Serological evidence of influenza D virus circulation among cattle and small ruminants in France[J]. Viruses, 2019, 11(6): 516. DOI:10.3390/v11060516 |
| [26] |
GUAN M, JACOBSON O, SARAFIANOS G, BAROCH J, DELIBERTO T J, WAN X F. Exposure of white-tailed deer in North America to influenza D virus[J]. Virology, 2022, 573: 111-117. DOI:10.1016/j.virol.2022.06.007 |
| [27] |
BAILEY E S, FIELDHOUSE J K, ALARJA N A, CHEN D D, KOVALIK M E, ZEMKE J N, CHOI J Y, BORKENHAGEN L K, TOH T H, LEE J S Y, CHONG K S, GRAY G C. First sequence of influenza D virus identified in poultry farm bioaerosols in Sarawak, Malaysia[J]. Tropical Diseases, Travel Medicine and Vaccines, 2020, 6: 5. DOI:10.1186/s40794-020-0105-9 |
| [28] |
MOLINI U, CURINI V, JACOBS E, TONGO E, BERJAOUI S, HEMBERGER M Y, PUGLIA I, JAGO M, KHAISEB S, CATTOLI G, DUNDON W G, LORUSSO A, DI GIALLONARDO F. First influenza D virus full-genome sequence retrieved from livestock in Namibia, Africa[J]. Acta Tropica, 2022, 232: 106482. DOI:10.1016/j.actatropica.2022.106482 |
| [29] |
MITRA N, CERNICCHIARO N, TORRES S, LI F, HAUSE B M. Metagenomic characterization of the virome associated with bovine respiratory disease in feedlot cattle identified novel viruses and suggests an etiologic role for influenza D virus[J]. Journal of General Virology, 2016, 97(8): 1771-1784. DOI:10.1099/jgv.0.000492 |
| [30] |
NG T F, KONDOV N O, DENG X, VAN EENENNAAM A, NEIBERGS H L, DELWART E. A metagenomics and case-control study to identify viruses associated with bovine respiratory disease[J]. Journal of Virology, 2015, 89(10): 5340-5349. DOI:10.1128/JVI.00064-15 |
| [31] |
ZHANG M, HILL J E, FERNANDO C, ALEXANDER T W, TIMSIT E, VAN DER MEER F, HUANG Y. Respiratory viruses identified in western Canadian beef cattle by metagenomic sequencing and their association with bovine respiratory disease[J]. Transboundary and Emerging Diseases, 2019, 66(3): 1379-1386. DOI:10.1111/tbed.13172 |
| [32] |
MOSIER D. Review of BRD pathogenesis: the old and the new[J]. Animal Health Research Reviews, 2014, 15(2): 166-168. DOI:10.1017/S1466252314000176 |
| [33] |
ZHANG X, OUTLAW C, OLIVIER A K, WOOLUMS A, EPPERSON W, WAN X F. Pathogenesis of co-infections of influenza D virus and Mannheimia haemolytica in cattle[J]. Veterinary Microbiology, 2019, 231: 246-253. DOI:10.1016/j.vetmic.2019.03.027 |
| [34] |
FERGUSON L, OLIVIER A K, GENOVA S, EPPERSON W B, SMITH D R, SCHNEIDER L, BARTON K, MCCUAN K, WEBBY R J, WAN X F. Pathogenesis of influenza D virus in cattle[J]. Journal of Virology, 2016, 90(12): 5636-5642. DOI:10.1128/JVI.03122-15 |
| [35] |
SALEM E, HAGGLUND S, CASSARD H, CORRE T, NASLUND K, FORET C, GAUTHIER D, PINARD A, DELVERDIER M, ZOHARI S, VALARCHER J F, DUCATEZ M, MEYER G. Pathogenesis, host innate immune response, and aerosol transmission of influenza D virus in cattle[J]. Journal of Virology, 2019, 93(7). DOI:10.1128/JVI.01853-18 |
| [36] |
LION A, SECULA A, RANCON C, BOULESTEIX O, PINARD A, DESLIS A, HAGGLUND S, SALEM E, CASSARD H, NASLUND K, GAUDINO M, MORENO A, BROCCHI E, DELVERDIER M, ZOHARI S, BARANOWSKI E, VALARCHER J F, DUCATEZ M F, MEYER G. Enhanced pathogenesis caused by influenza D virus and Mycoplasma bovis coinfection in calves: a disease severity linked with overexpression of IFN-gamma as a key player of the enhanced innate immune response in lungs[J]. Microbiology Spectrum, 2021, 9(3): e0169021. DOI:10.1128/spectrum.01690-21 |
| [37] |
GAUDINO M, NAGAMINE B, DUCATEZ M F, MEYER G. Understanding the mechanisms of viral and bacterial coinfections in bovine respiratory disease: a comprehensive literature review of experimental evidence[J]. Veterinary Research, 2022, 53(1): 70. DOI:10.1186/s13567-022-01086-1 |
| [38] |
SREENIVASAN C, THOMAS M, SHENG Z, HAUSE B M, COLLIN E A, KNUDSEN D E, PILLATZKI A, NELSON E, WANG D, KAUSHIK R S, LI F. Replication and transmission of the novel bovine influenza D virus in a guinea pig model[J]. Journal of Virology, 2015, 89(23): 11990-12001. DOI:10.1128/JVI.01630-15 |
| [39] |
TROMBETTA C M, MARCHI S, MANINI I, KISTNER O, LI F, PIU P, MANENTI A, BIUSO F, SREENIVASAN C, DRUCE J, MONTOMOLI E. Influenza D virus: serological evidence in the Italian population from 2005 to 2017[J]. Viruses, 2019, 12(1). DOI:10.3390/v12010030 |
| [40] |
BAILEY E S, CHOI J Y, ZEMKE J, YONDON M, GRAY G C. Molecular surveillance of respiratory viruses with bioaerosol sampling in an airport[J]. Tropical Diseases, Travel Medicine and Vaccines, 2018, 4: 11. DOI:10.1186/s40794-018-0071-7 |
| [41] |
CHOI J Y, ZEMKE J, PHILO S E, BAILEY E S, YONDON M, GRAY G C. Aerosol sampling in a hospital emergency room setting: a complementary surveillance method for the detection of respiratory viruses[J]. Frontiers in Public Health, 2018, 6: 174. DOI:10.3389/fpubh.2018.00174 |
| [42] |
COLLIN E A, SHENG Z, LANG Y, MA W, HAUSE B M, LI F. Cocirculation of two distinct genetic and antigenic lineages of proposed influenza D virus in cattle[J]. Journal of Virology, 2015, 89(2): 1036-1042. DOI:10.1128/JVI.02718-14 |
| [43] |
JIANG W M, WANG S C, PENG C, YU J M, ZHUANG Q Y, HOU G Y, LIU S, LI J P, CHEN J M. Identification of a potential novel type of influenza virus in bovine in China[J]. Virus Genes, 2014, 49(3): 493-496. DOI:10.1007/s11262-014-1107-3 |
| [44] |
HORIMOTO T, HIONO T, MEKATA H, ODAGIRI T, LEI Z, KOBAYASHI T, NORIMINE J, INOSHIMA Y, HIKONO H, MURAKAMI K, SATO R, MURAKAMI H, SAKAGUCHI M, ISHⅡ K, ANDO T, OTOMARU K, OZAWA M, SAKODA Y, MURAKAMI S. Nationwide distribution of bovine influenza D virus infection in Japan[J]. PLoS One, 2016, 11(9): e0163828. DOI:10.1371/journal.pone.0163828 |
| [45] |
FONI E, CHIAPPONI C, BAIONI L, ZANNI I, MERENDA M, ROSIGNOLI C, KYRIAKIS C S, LUINI M V, MANDOLA M L, BOLZONI L, NIGRELLI A D, FACCINI S. Influenza D in Italy: towards a better understanding of an emerging viral infection in swine[J]. Scientific Reports, 2017, 7(1): 11660. DOI:10.1038/s41598-017-12012-3 |
| [46] |
FLYNN O, GALLAGHER C, MOONEY J, IRVINE C, DUCATEZ M, HAUSE B, MCGRATH G, RYAN E. Influenza D virus in cattle, Ireland[J]. Emerging Infectious Diseases, 2018, 24(2): 389-391. DOI:10.3201/eid2402.170759 |
| [47] |
SNOECK C J, OLIVA J, PAULY M, LOSCH S, WILDSCHUTZ F, MULLER C P, HUBSCHEN J M, DUCATEZ M F. Influenza D virus circulation in cattle and swine, Luxembourg, 2012-2016[J]. Emerging Infectious Diseases, 2018, 24(7): 1388-1389. DOI:10.3201/eid2407.171937 |
| [48] |
DANE H, DUFFY C, GUELBENZU M, HAUSE B, FEE S, FORSTER F, MCMENAMY M J, LEMON K. Detection of influenza D virus in bovine respiratory disease samples, UK[J]. Transboundary and Emerging Diseases, 2019, 66(5): 2184-2187. DOI:10.1111/tbed.13273 |
| [49] |
MURAKAMI S, ODAGIRI T, MELAKU S K, BAZARTSEREN B, ISHIDA H, TAKENAKA-UEMA A, MURAKI Y, SENTSUI H, HORIMOTO T. Influenza D virus infection in dromedary camels, Ethiopia[J]. Emerging Infectious Diseases, 2019, 25(6): 1224-1226. DOI:10.3201/eid2506.181158 |
| [50] |
HE W T, LU M, XING G, SHAO Y, ZHANG M, YANG Y, LI X, ZHANG L, LI G, CAO Z, SU S, VEIT M, HE H. Emergence and adaptive evolution of influenza D virus[J]. Microbial Pathogenesis, 2021, 160: 105193. DOI:10.1016/j.micpath.2021.105193 |
| [51] |
YU J, LI T, WEN Z, WU S, WANG Z, ZHENG J, CHEN M, CHEN F, WEI W K, ZHAI S L, LIAO M. Identification of D/Yama2019 lineage-like influenza D virus in Chinese cattle[J]. Frontiers in Veterinary Science, 2022, 9: 939456. DOI:10.3389/fvets.2022.939456 |
| [52] |
NAKATSU S, MURAKAMI S, SHINDO K, HORIMOTO T, SAGARA H, NODA T, KAWAOKA Y. Influenza C and D viruses package eight organized ribonucleoprotein complexes[J]. Journal of Virology, 2018, 92(6). DOI:10.1128/JVI.02084-17 |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49




