文章信息
基金项目
- 华南应用微生物国家重点实验室开放基金(SKLAM002-2021);华南地区乡村振兴专项资金(2021KJ382);茂名实验室科研启动项目(2021TDQD002);华南地区农业科学院地方分院和专家工作站工作经费项目(2022支撑11)
作者简介
-
董博,博士,主要开展畜禽水产肠道微生物组学、培养组学和益生机制方面的研究,以及畜禽水产微生态制剂研发及技术推广服务工作。主持国自然联合基金子课题,留基委、深圳科创委和微生物重点实验室开放课题等多项项目,获得授权专利2项,在《Microbiology Spectrum》《mSys》《Clinical Microbiology and Infection》《Aquaculture Research》《Animals》
《动物营养学报》等期刊以第一作者或通讯作者发表学术论文10余篇。获广东省科学技术三等奖1项(排名第1)、广东省农业科学院科学技术二等奖1项(排名第1)、广东省农业技术推广奖一等奖1项(排名第10)、广东省自然科学三等奖2项(分别排名第4、第6)。
董博(1982—),男,博士,助理研究员,研究方向为海洋微生物组学,E-mail:dongbo@gdaas.cn; 陈庄,研究员,学科带头人,现任广东省农业科技战略专家,国家、广东省和浙江省自然科学基金评审专家。主要从事畜牧胃肠道微生物、畜牧营养调控研究。在农业微生态制剂研究开发和动物基因编辑等方面有深入研究,曾担任畜牧育种国家重点实验室副主任,是业内公认学术造诣高、工作业绩突出的专业技术人员。主持省部级以上科技项目10余项,作为首席科学家主持国家“973”重大项目农业生物技术及农产品加工科学问题,在国内外期刊累计发表论文80余篇,申请发明专利18项。参编广东省第一部动物转基因专著《转基因动物》(1995)。获科技成果多项,其中省部级以上成果奖励4项,包括农业部农业科技创新团队奖、广东省农业技术推广二等奖、广东省自然科学三等奖和广东省科技进步三等奖.
通讯作者
- 陈庄(1963—),男,博士,研究员,研究方向为畜禽水产微生物组学,E-mail:chenzhuang@gdaas.cn.
文章历史
- 收稿日期:2022-09-30
2. 深圳市海洋生物基因组学重点实验室,广东 深圳 518083;
3. 深圳大学生命科学与海洋学院,广东 深圳 518061
2. Shenzhen Key Laboratory of Marine Genomics, Shenzhen 518083, China;
3. College of Life Science and Oceanology, Shenzhen University, Shenzhen 518061, China
【研究意义】弹涂鱼是一类可以在水中和陆地上生存的硬骨鱼类,栖息于海洋潮间带滩涂,低潮时暴露在空气中。3.6亿年前,鱼类成功从水生生物转变为陆地生物或四足动物,这一过渡期间的形式尚存在化石中。两栖鱼类成为了解脊椎动物水陆转变相关遗传变化提供了有用的研究模型[1]。
【前人研究进展】依据基因组数据分析得到的系统发育树结果对弹涂鱼分类,大弹涂鱼属(Boleophthalmus) 和青弹涂鱼属(Scartelaos) 构成一个类群,而齿弹涂鱼属(Periophthalmodon) 和弹涂鱼属(Periophthalmus)构成另一类群。这个分群与它们的水生习性一致,前一类群水生习性强,而后一类群陆生习性特别强,其中大弹涂鱼(Boleophthalmus pectinirostris)栖息于中低潮带的泥滩,植食性,刮食底栖硅藻,水生习性较强;而大鳍弹涂鱼(Periophthalmus magnuspinnatus) 栖息于中高潮带的水草或红树林滩涂区域,肉食性,主要捕食水生昆虫及小型甲壳生物等,一生中约2/3的时间离水生活,陆生习性最强。作为从水生鱼类进化到陆生四肢动物过渡环节的代表,弹涂鱼及其两栖适应性一直受到国内外研究者的关注,其陆地适应包括通过空气呼吸、有更高的氨耐受性、使用改良的胸鳍进行陆地运动、抗病能力强以抵抗水陆两种生境中众多不同的病源微生物等[2]。这些进化的表型被认为是选择压力驱动的遗传进化的结果[1]。然而,目前对胃肠道微生物群宏基因组这一生物体第二基因组所赋予的两栖适应性知之甚少。
免疫是两栖鱼类在从水到陆地过渡期间应对多样化环境和各种病原体的关键因素。研究发现,与其他硬骨鱼分离后,弹涂鱼的先天免疫系统基因显著扩增[1]。在已测序的脊椎动物中,弹涂鱼拥有最多数量(11拷贝)的TLR13[1-2],这有利于防御陆地病原体。除了提供陆地适应的基因组外,弹涂鱼肠道微生物群在免疫等生理特征中起重要作用[3-7]。胃肠道微生物群与宿主共同从水到陆地的过渡期间进行了数百万年进化,鱼类的许多生理功能由它们维持或参与,包括病原体的拮抗作用、肠上皮细胞的增殖和免疫系统的成熟等[8-10]。
【本研究切入点】长期以来,人们认为所有鱼类的宿主遗传背景和环境因素使其选择“核心微生物群”来维持所有鱼类成员共有的一些基本功能[11]。这个假设可以推广到两栖动物和陆生脊椎动物。而弹涂鱼的独特生活习性将为这一假设以及核心微生物群如何从水生变为陆地[12-13]提供参考。【拟解决的关键问题】本研究采用16S扩增子测序和宏基因组技术,通过比较分析两种水陆生习性差异较大的弹涂鱼代表种与具有代表性的水生鱼类的胃肠道微生物群组成、丰度、多样性和功能,以期得出有益于其两栖适应性的标志胃肠道微生物群,为弹涂鱼对两栖生活的适应性提供研究参考。
1 材料与方法 1.1 试验材料供试的大弹涂鱼和大鳍弹涂鱼分别于2016年7月和2017年7月在广东省珠海市淇澳岛野外捕获。供试的水生鱼类为草鱼、鲢鱼和鳙鱼,于2016年8月在广东省广州市一孵化场收集,每尾重约1~2 kg,采用深圳澳华农牧有限公司的商业饲料喂养。选择大而健康的个体,用无菌蒸馏水冲洗鱼体表,并使用70% 乙醇消毒,然后作无菌解剖取出后肠,挤出肠道内容物和上皮粘膜,于-80 ℃下保存备用。所有试验均按照动物伦理委员会的指导方针开展。
1.2 宏基因组DNA提取本研究选取4条大弹涂鱼、3条大鳍弹涂鱼、8条草鱼、5条鲢鱼、1条鳙鱼的肠道内容物和上皮粘膜样品共21份,冰上解冻后采用DNA提取试剂盒(CTAB法)提取总细菌DNA[14]。在1% 琼脂糖凝胶上检测DNA的完整性和纯度,通过Qubit荧光计(Thermo Fisher Scientific,USA)测量DNA浓度。将提取的宏基因组DNA保存于-80 ℃备用。
1.3 文库构建和验证通过超声波基因剪切仪Covaris将宏基因组DNA分解成350 bp片段用于电泳测定。随后将片段化的DNA与End Repair Mix(NEB,USA) 混合于20 ℃下温育30 min。用QIAquick PCR纯化试剂盒(Qiagen,USA)纯化末端修复的DNA,然后加入A-Tailing Mix(NEB,USA)。连接后,通过2% 琼脂糖凝胶选择接头连接的DNA以回收靶片段。纯化凝胶并进行PCR扩增以富集接头连接的DNA片段。然后通过2% 琼脂糖凝胶选择PCR产物,并使用QIAquick PCR纯化试剂盒纯化以回收靶片段。构建的文库以两种方式定量,包括使用Agilent 2100生物分析仪与Agilent DNA 1000试剂测定平均分子长度,以及通过定量实时PCR仪(Agilent,USA)对文库进行定量。
1.4 文库测序首先在cBot仪器(Hiseq 4000 PE Cluster Kit,Illumina,USA)的流动池内扩增合格的文库用于生成簇。随后将聚集的流动细胞加载到Hiseq 4000测序仪(Hiseq 4000 SBS Kit,Illumina)上进行双末端测序,推荐的测序基因长度为100 bp或150 bp。
1.5 宏基因组的组装和注释通过Illumina Hiseq PE150平台对总共21个样品进行测序。宏基因组由MEGAHIT软件组装,分析由深圳倍森基因科技有限公司实施[14-15]。推断的氨基酸序列的注释通过蛋白质分子序列比对搜索工具BLASTP针对NR数据库进行(E ≤ 1e-3,KAIJU) [16]。为了确定肠道微生物群的准确系统发育组成,将所有宏基因组测序基因比对到原核参考基因组,使用具有默认参数的核酸分子序列比对搜索工具BLASTN提交至NCBI的基因组数据库,针对每个相应宿主的基因组过滤序列相似性≥ 75% 的测序基因。
1.6 OTU统计和维恩图绘制通过软件USEARCH(v7.0.1090) [17]将标签聚类到OTU(操作分类单元):(1) 使用UPARSE将标签聚集为97% 阈值,得到OTU唯一代表序列。每个样本的OTU数主要代表样本多样性程度。(2)使用UCHIME(v4.2.40)过滤掉嵌合体。(3)使用USEARCH GLOBAL(扩增子分析软件)将所有标签映射到每个OTU代表性序列,然后将每个样品中每个OTU的标签号总结为OTU丰度表。使用核糖体数据库RDP(v.2.2)分类器对序列进行分类(在Greengenes数据库上训练,以0.8信任值作为阈值)。
基于OTU丰度列出每组的OTU用于比较。维恩图由软件R(v3.1.1)绘制,其中汇总了公共或特定的OTU ID。不同的颜色代表不同的样本或组,每个圆圈的内部象征性地表示观察到的OTU数量,重叠区域或交叉点表示公共OTU集,单层区域表示在某个样本/组中唯一标识的OTU数量。
2 结果与分析 2.1 鱼类宏基因组数据集的总结本研究选取4条大弹涂鱼、3条大鳍弹涂鱼、8条草鱼、5条鲢鱼、1条鳙鱼的肠道内容物和上皮粘膜样本进行16S rRNA测序,分别生成29 Mb、17.39 Mb、22 Gb、46.7 Gb和12 Gb的原始数据。由于宿主污染占原始数据的大部分,因此对所有序列数据过滤以去除宿主污染并生成鱼类宏基因组数据。最终从宏基因组数据中,本研究注释共4 966种、1 453属、378科、178目、76类和54门。
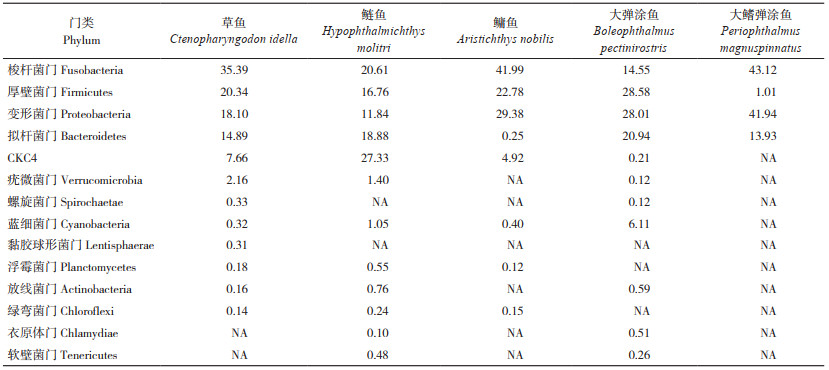
草鱼的胃肠道微生物群(表 1)由梭杆菌门(35.39%)、厚壁菌门(20.34%)、拟杆菌门(18.10%)、变形菌门(14.89%) 和CKC4门(7.66.%)主导,这与之前的研究结果一致[12, 18],但丰度可能因饲养条件和鱼类年龄不同而有所不同。代表性的属分别是气单胞菌属(19.02%)、希瓦氏菌属(3.79%)、鲸杆菌属(2.96%)、拟杆菌属(1.60%)和梭菌属(0.81%)。
|
2.2 弹涂鱼和草鱼、鲢鱼、鳙鱼之间微生物物种及丰度的比较
2.2.1 弹涂鱼胃肠道微生物组成 在弹涂鱼的宏基因组中,本研究观察到胃肠道微生物群的厚壁菌门、变形菌门、拟杆菌门、梭杆菌门占比较大(表 1),然而每个菌的丰度在大弹涂鱼和大鳍弹涂鱼之间显著不同。例如,厚壁菌门在大弹涂鱼中占胃肠道微生物约35%,而在大鳍弹涂鱼中仅为1%;相反,梭杆菌门在大鳍弹涂鱼中占43%,远远超过其在大弹涂鱼中的比例。此外,大弹涂鱼的胃肠道微生物门水平上的多样性显著高于大鳍弹涂鱼。胃肠道细菌的多样性从杂食性动物到食草性动物一般是增加的[19-20],草食性的大弹涂鱼胃肠道细菌多样性高于杂食性的大鳍弹涂鱼也可以证明这一事实[2]。
这两种弹涂鱼物种胃肠道微生物组成的另一个显著区别是蓝细菌在大弹涂鱼中所占比例为6.11%,而在大鳍弹涂鱼中则未发现,这种差异与两者间较大的水陆习性差异相符合。前者更偏水生且更多地摄取海水中的藻类(包括蓝藻) [19-20]等。
2.2.2 草鱼、鲢鱼和鳙鱼胃肠道微生物组成 鳙鱼、鲢鱼和草鱼的宏基因组结果(表 1)与之前的报道[21-25]一致,其中4种主要门类厚壁菌门、变形菌门、拟杆菌门、梭杆菌门丰度相对较高。厚壁菌门和拟杆菌门的比例从这3种鲤科鱼类到大弹涂鱼,再到大鳍弹涂鱼逐渐增加,与上述3种水生性鲤科鱼类及偏水生性的大弹涂鱼相比,偏陆生性的大鳍弹涂鱼胃肠道中厚壁菌门和拟杆菌门比例更高[20]。
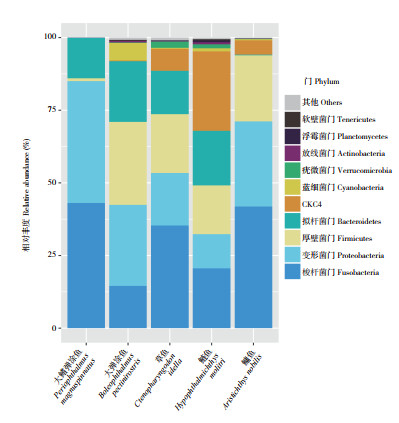
2.2.3 弹涂鱼和草鱼、鲢鱼、鳙鱼之间胃肠道微生物差异 胃肠道微生物群在3种鲤科鱼类与弹涂鱼中的最大差异在于CKC4门,在鲤科鱼类中占比从4%~27% 不等,而在大弹涂鱼中仅为0.21%,在大鳍弹涂鱼中则未发现(图 1)。目前关于CKC4门细菌群的信息很少,其最早在斑马鱼胃肠道中被发现。之前的研究表明CKC4门可能对雌激素及其类似物敏感,推测内分泌暴露显著改变了斑马鱼肠道微生物群中CKC4门的丰度[26],CKC4门可能与宿主脂质代谢的变化有关。
|
| 图 1 弹涂鱼和草鱼、鲢鱼、鳙鱼宏基因组中注释的胃肠道微生物门水平相对丰度比较 Fig. 1 Comparison of relative abundance of the gastrointestinal phyla annotated in the metagenomes of mudskippers and Ctenopharyngodon idellus, Hypophthalmichthys molitrix and Aristichthys nobilis |
2.3 不同物种之间的微生物群种类组成和丰度比较
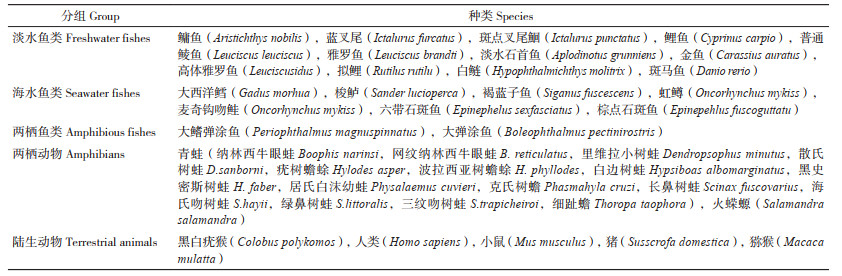
本研究在NCBI上下载了38种物种的16S rRNA测序数据,以及可用的宏基因组结果。对收集的不同物种微生物组数据加以对比,可反映每个进化群体中胃肠道微生物群的整体变化和细微变化。将不同物种分为5类,分别为两栖鱼类、海水鱼类、淡水鱼类、两栖动物和陆生动物(表 2)。
|
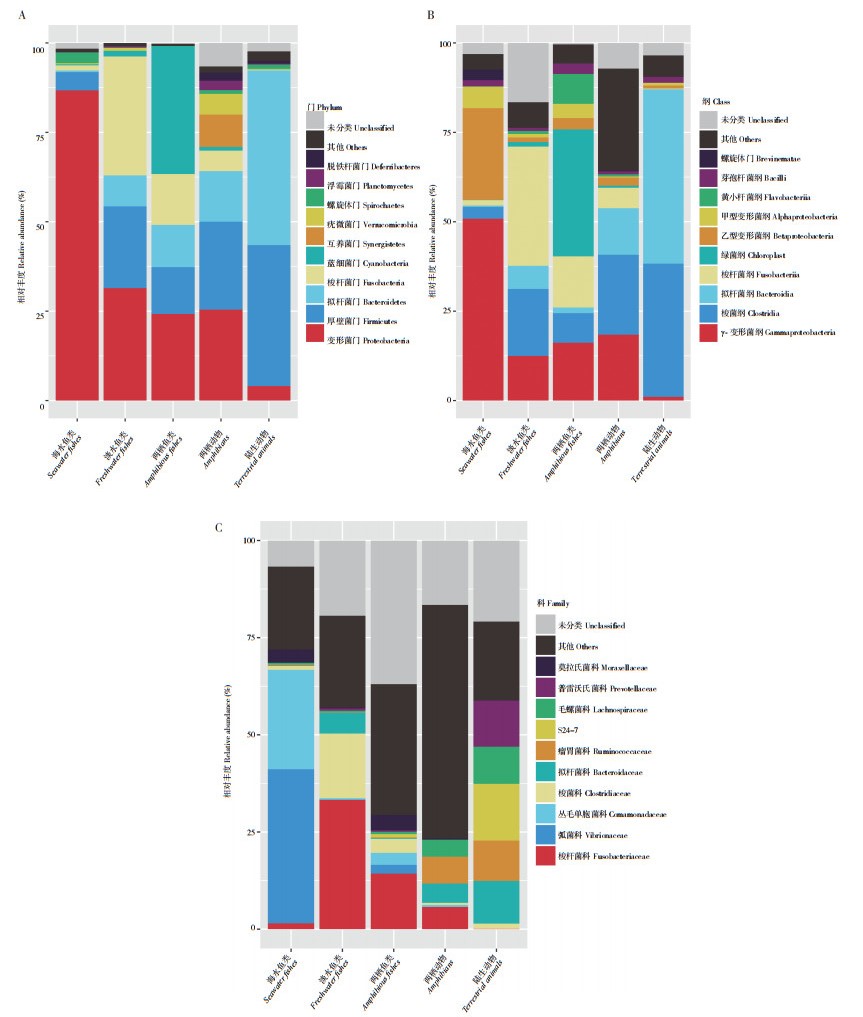
弹涂鱼和草鱼、鲢鱼和鳙鱼的胃肠道微生物组成与丰度在图 1中的分布与表 1呈现的结果一致。根据5类鱼类中纲水平微生物群组成可知,弹涂鱼中的黄杆菌含量远远高于其他鱼类。事实上,黄杆菌属于条件致病菌,广泛分布于水和陆地,并对野生鱼类和人工繁殖的鱼类种群构成严重威胁[27]。本研究通过搜索相应的宏基因组注释结果,在大弹涂鱼的胃肠道微生物群中发现肠球菌菌株(0.07%)。这些革兰氏阳性细菌通常存在于陆生哺乳动物中[28]。
由图 2可知,瘤胃菌科Ruminococcaceae和毛螺菌科Lachnospiraceae在弹涂鱼、两栖动物和陆地哺乳动物中相对丰富(分别为0.16% 和0.61%,7.0% 和4.4%,10.3% 和11.9%)。特别是S24-7家族,在陆地动物胃肠道微生物群(14.60%)中特别丰富,在弹涂鱼(0.78%)中也有少量,但不存在于水生鱼类和两栖动物中。
|
| 图 2 5类物种的胃肠道微生物群的门(A)、纲(B)、科(C)水平分类组成 Fig. 2 Taxonomic compositions of gastrointestinal microbiota in five species at the phylum (A), class (B) and family (C) level |
同样,梭菌科Clostridiaceae、莫拉氏科Moraxella、梭杆菌科Fusobacteriaceae在淡水鱼类中最为丰富(分别为16.6%、3.3% 和33.2%);在弹涂鱼中比较丰富(分别为3.6%、4.0% 和14.2%),但在其他3组中低于1.5%、0.3%、6.0%。已有研究发现,酪酸梭菌可以改善南美白对虾的生长性能,增加体内粗蛋白含量,调节肠道消化能力,增强肠道对氨胁迫的免疫功能[29];产气荚膜梭菌是鱼类中一种重要的食源性病原体,被认为是鱼类的致病微生物[30]。
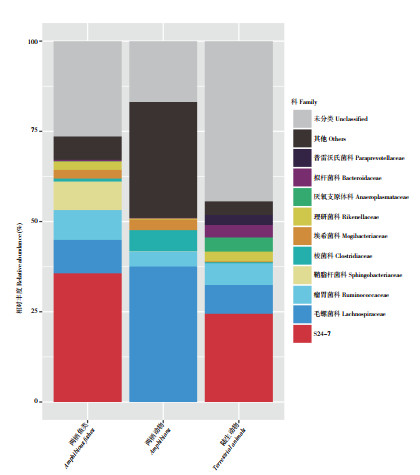
2.4 弹涂鱼与陆生动物胃肠道微生物比较分析维恩图可以显示多样本/组中的共同/唯一OTU的数量。如果与OTU代表性物种结合,可以获得不同环境的核心微生物组。通过文献找到40种物种的可用胃肠道宏基因组数据。OTU集的相关维恩图显示将有助于确定弹涂鱼和陆生动物之间的关系。本研究发现,两栖弹涂鱼和陆生动物(包括两栖动物和陆栖哺乳动物)肠菌之间有759个OTU重叠;不同于鱼类的弹涂鱼与陆生动物的OTU大部分来自S24-7以及毛螺菌Lachnospiraceae、瘤胃菌科Ruminococcaceae、理研菌科Rikenellaceae(图 3)。这些菌株的详细功能值得进一步探索。
|
| 图 3 弹涂鱼、两栖动物和陆生动物胃肠道微生物中不同典型OTU科水平的分类组成 Fig. 3 Taxonomic compositions of various typical OTU in gastrointestinal microbiota of mudskippers, amphibians and terrestrial animals at family level |
2.5 大弹涂鱼与草鱼宏基因组功能注释比较
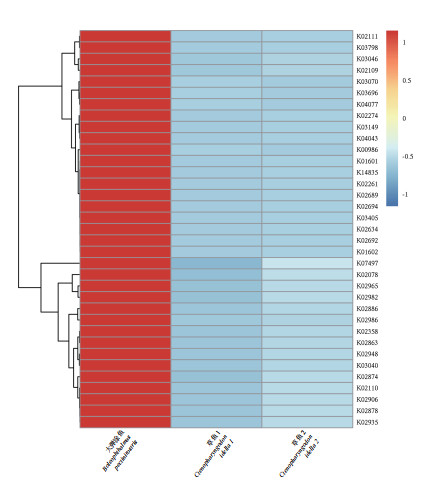
基于所有弹涂鱼样本的宏基因组数据的功能注释和丰度信息,选择每个样本中前35个功能类别及其丰度信息来绘制热图并聚类以获得功能差异。从图 4可见,大弹涂鱼中宏基因组功能特征与草鱼的功能特征具有显著差异,最显著的差异是氨通道蛋白AmtB,其在大弹涂鱼微生物群中比在草鱼中更丰富,并且其高氨运输能力与弹涂鱼适于潮间生活的氨抗性特征相关[1-2]。
|
| 图 4 大弹涂鱼与草鱼宏基因组的功能注释热图 Fig. 4 Heat map of function annotation from the metagenomes of Boleophthalmus pectinirostris and Ctenopharyngodon idellus |
3 讨论
鱼类微生物组研究目前在鱼类研究中的代表性较低,特别是与其他动物宿主相比具有独特生理和生化差异的两栖弹涂鱼。因此,本研究首次研究了鱼类、两栖动物和陆地脊椎动物之间的胃肠道微生物群差异或重叠。两栖鱼类、海水鱼类、淡水鱼类、两栖动物和陆生动物的进化谱系虽然清晰,但胃肠道微生物群和相应的宏基因组的进化仍然不明确。
3.1 大弹涂鱼和大鳍弹涂鱼之间的胃肠道微生物群组成的差异为了研究弹涂鱼胃肠道微生物在两栖习性中的作用,本研究选择大部分陆栖物种大鳍弹涂鱼和相对偏水生大弹涂鱼来收集胃肠道微生物并对其进行后续的16S rDNA扩增子测序。尽管这两种弹涂鱼物种具有密切的遗传关系,但它们在地理标志微生物类别、多样性和丰富程度之间存在显著差异,这可能是由于不同的栖息地和饲养偏好。本研究发现,大弹涂鱼胃肠道微生物中蓝细菌丰度较高,这可能与其更多的水生生活方式和对藻类的偏好有关[1-2]。另外,大弹涂鱼中胃肠道微生物群多样性较大鳍弹涂鱼高,而这与偏植食性动物的胃肠道微生物多样性通常较高是一致的[19-20]。
3.2 弹涂鱼、水生鱼类和陆地动物的胃肠道微生物群特征比较鱼类(弹涂鱼和草鱼、鲢鱼、鳙鱼)胃肠道微生物类别在门的水平上相对稳定。尽管每种门的丰度在不同鱼类中变化很大,但最丰富的门是厚壁菌门、拟杆菌门、梭杆菌门和变形菌门。然而,一个显著的例外是CKC4门,它仅在草鱼、鲢鱼、鳙鱼的鲤科鱼类中普遍存在,在弹涂鱼中仅存在0.21%。为了仔细检查家族层面的差异,本研究发现许多微生物菌株只存于两栖弹涂鱼和陆生动物,如瘤胃菌科Ruminococcaceae和毛螺菌科Lachnospiraceae,特别是S24-7,表明两栖弹涂鱼通过胃肠道微生物来适应陆地生活。研究还发现,大弹涂鱼中的黄杆菌比其他鱼类多,检测到胃肠道微生物群中的肠球菌菌株丰度为0.07。据报道,肠球菌菌株产生AMP样物质并且对从病鱼中分离的病原菌表现出广谱抑制,特别是对革兰氏阴性菌冷黄杆菌Flavobacterium frigidarium、果胶弧菌Vibrio pectenicida、对虾弧菌V.penaeicida和美人鱼发光杆菌Photobacterium damselae[31]。在弹涂鱼胃肠道中肠球菌菌株和致病性黄杆菌之间可能存在平衡关系,因此弹涂鱼可以在这种不利环境中忍受相对高丰度的黄杆菌。在健康的弹涂鱼个体中,这种病原体的相对高丰度表明两栖鱼可能已经对不同生活栖息地的致命病原体产生了特殊的抗性。弹涂鱼这种强大的免疫特性有待进一步研究。
3.3 核心微生物群和影响胃肠道微生物群的关键因素许多因素促成了硬骨鱼类胃肠道微生物群的组成,包括宿主遗传、环境、胃肠道生理、细菌共生体和营养[32]。核心微生物组,即环境中所有鱼类个体中存在的微生物群落成员,已经成为研究胃肠道微生物群落科研人员的主要目标[32-34]。
总体而言,包括海水鱼类[34]和淡水鱼类[26, 35-38]在内的胃肠道微生物群主要是变形菌门,其次是梭杆菌门和厚壁菌门,以及拟杆菌门、放线菌门和疣粒菌门[38],占总菌群的90%,每个主要门的含量在不同物种中都有显著差异。本研究发现弹涂鱼的胃肠道微生物群同时拥有陆地、淡水、海洋和两栖群体的典型家族,这与弹涂鱼的水陆过渡和咸淡水界面栖息的特征情况非常吻合[2]。在一定程度上,这一发现也为生态环境和栖息特点影响核心微生物菌群的假设提供了证据,即在过渡阶段两栖性的弹涂鱼依靠多样的肠道微生物来应对不同环境。
3.4 与弹涂鱼两栖适应性相关微生物菌株弹涂鱼是两栖鱼类,但很少有关于其先天免疫系统成分的系统研究,尤其是当免疫力来自胃肠道微生物群对病原体屏障的研究。本研究首次分析了两种代表性两栖弹涂鱼(大弹涂鱼和大鳍弹涂鱼)的胃肠道微生物群,并对其中的微生物进行注释,以探讨胃肠道中是否存在特殊菌株或抗菌肽以适应潮间带复杂多变的不利环境(如高氨低氧)。
本研究结果表明,弹涂鱼中不同OTU最多来自S24-7、毛螺菌Lachnospiraceae、瘤胃菌Ruminococcaceae、理研菌Rikenellaceae,这提示以上菌株对陆地生物独特的功能值得研究。从纲水平菌群比较大弹涂鱼和草、鲢、鳙鱼的胃肠道微生物组成,黄杆菌在大弹涂鱼中含量高于其他海洋鱼类和淡水鱼类。另外,瘤胃菌Ruminococcaceae和毛螺菌Lachnospiraceae在弹涂鱼、两栖类和陆栖类群中相对丰富。
值得注意的是,S24-7家族在陆地动物肠道微生物群中占有独特地位(14.60%),在弹涂鱼(0.78%)中也可以找到,但在鱼类和两栖动物中均不存在,该家族表现出对于恒温性肠道的强烈偏好性,核心代谢为发酵及厌氧细菌[38]。这使得该类研究中大部分为陆生动物群体。据报道,具有脲酶活性的S24-7产生脲酶,能够分解尿素产生氨,增加血液循环中的氨含量,由此推测弹涂鱼的氨耐受能力强,与胃肠道中的S24-7存在一定关联。另外,多项研究报道了S24-7家族成员丰度的改变与不同的环境条件有关,且至少有一些成员是先天免疫系统的目标,该家族涉及宿主-微生物相互作用,并且能够影响肠道功能和健康,具体表现在其可以改变宿主肠道中的局部免疫系统,细菌细胞在正常生理状态下不与肠细胞接触;然而它们可能会将DNA释放到粘液层中,通过特异性受体(如Toll样受体9)影响宿主先天免疫细胞[39]。据报道,S24-7可通过与肠粘液层相互作用调节免疫功能[40]。
4 结论弹涂鱼胃肠道微生物群具有典型的陆生、淡水、海洋和两栖动物复合微生物群落,这与弹涂鱼陆地-水过渡的特殊生态位以及在淡水和海水界面生存的特点有关。这一发现也为核心肠道微生物群的概念提供了支持,因为不同的生态环境和生活习惯选择了相应的微生物群,而在过渡阶段的两栖动物将两套胃肠道微生物群集合起来应对两个不同的环境。本研究展示了5个类群OTU集(淡水鱼类、海洋鱼类、陆地动物、两栖动物和弹涂鱼)。韦恩图的对比关系进一步反映了从水生到两栖进化过程中两栖动物(如弹涂鱼)胃肠道微生物群的变化。结果表明,有243个OTU在陆地脊椎动物中被发现,但在淡水和海洋鱼类中均不存在,这些物种可能涉及陆地代谢、氨抗性和干暴露等细菌或真菌,其中13个OTU出现在两栖动物中,其余230个出现在陆生动物中。此外,两栖动物和弹涂鱼中存在27种OTU,但在淡水鱼类和海洋鱼类中不存在,提示这些OTU可能与两栖生活方式有关。
与水生鱼类相比,弹涂鱼胃肠道微生物群的多样性更高,即弹涂鱼拥有硬骨鱼类中最大的TLR基因多样性(11个重复成员) [1]。更多样性的TLR谱系将通过PRR(模式识别受体)识别更多的病原体类别,并促进免疫因子和AMP表达的下游激活[41],这将极大提高肠上皮的免疫力和健康[42-43]。结合前人有关TLR扩张的发现,支持有关两栖物种具有高免疫抵抗力而适于恶劣和多样潮间下生活的结论。
| [1] |
YOU X, BIAN C, ZAN Q, XU X, LIU X, CHEN J, WANG J, QIU Y, LI W, ZHANG X, SUN Y, CHENG S, HONG W, LI Y, CHENG S, FANG G, SHI C, LIANG J, Tom Tang Y, YANG C, RUAN Z, BAI J, PENG C, MU Q, LU J, FAN M, YANG S, HUANG Z, JIANG X, FANG X, ZHANG G, ZHANG Y, POLGA G, YU H, LI J, LIU Z, ZHANG G, Ravi V, Con S L, WANG J, YANG H, Venkatesh B, WANG J, SHI Q. Mudskipper genomes provide insights into the terrestrial adaptation of amphibious fishes[J]. Nature Communication, 2014, 5594: 2-3. DOI:10.1038/ncomms6594 |
| [2] |
YOU X, SUN M, LI J, BIAN C, CHEN J, YI Y, YU H, SHI Q. Mudskippers and their genetic adaptations to an amphibious lifestyle[J]. Animals, 2018, 8(2): 24. DOI:10.3390/ani8020024 |
| [3] |
游欣欣, 卞超, 陈洁明, 孙颖, 石琼. 弹涂鱼两栖习性的基因组学研究进展[J]. 海洋渔业, 2015, 37(5): 479-484. DOI:10.13233/j.cnk.mar.fish.2015.05.012 YOU X X, BIAN C, CHEN J M, SUN Y, SHI Q. Recent advances in genomic study on amphibious mudskippers[J]. Marine Fisheries, 2015, 37(5): 479-484. DOI:10.13233/j.cnk.mar.fish.2015.05.012 |
| [4] |
CEREZUELA R, MESEGUER J, ESTEBAN M. Effects of dietary inulin, Bacillus subtilis and microalgae on intestinal gene expression in gilthead seabream (Sparusaurata L.)[J]. Fish & Shellfish Immunology, 2013, 234: 843-848. DOI:10.1016/j.fsi.2012.12.026 |
| [5] |
NEU J. The developing intestinal microbiome: probiotics and prebiotics[J]. World Review Nutrition Diettetics, 2014, 110: 167-176. DOI:10.1159/000358465 |
| [6] |
DAWOOD M, KOSHIO S, ISHIKAWA M, YOKOYAMA S, BASUINI M F E, HOSSAIN M S, NHU T H, DOSSOU S, MOSS A S. Effects of dietary supplementation of Lactobacillus rhamnosus or/and Lactococcuslactis on the growth, gut microbiota and immune responses of red sea bream, Pagrus major[J]. Fish & Shellfish Immunology, 2016, 49: 275-285. |
| [7] |
FLINT H, BAYER E, RINCON M, LAMED R, WHITE B. Polysaccharide utilization by g ut bacteria: potential for new insights from genomic analysis[J]. Nature Review Microbiology, 2008, 6: 121-131. DOI:10.1038/nrmicro1817 |
| [8] |
DESANTIS T, HUGENHOLTZ P, LARSEN N, ROJAS M, BRODIE E L, KELLER K, HUBER T, DALEVI D, HU P, ANDERSEN G L. Green genes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Applied and Environmental Microbiology, 2006, 72: 5069-5072. DOI:10.1128/aem.03006-05 |
| [9] |
FAKRUDDIN M, MANNAN K, ANDREWS S. Viable but nonculturable bacteria: food safety and public health perspective[J]. ISRN Microbiology, 2013, 26: 703-813. DOI:10.1155/2013/703813 |
| [10] |
RAWLS J, SAMUEL B, GORDON J. Gnotobiotic zebrafish reveal evolutionarily conserved responses to the gut microbiota[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101: 4596-4601. DOI:10.1073/pnas.0400706101 |
| [11] |
TURNBAUGH P J, LEY R E, MAHOWALD M A, MAGRINI V, MARDIS E R, GORDON J I. An obesity-associated gut microbiome with increased capacity for energy harvest[J]. Nature, 2006, 444: 1027-1031. DOI:10.1038/nature05414 |
| [12] |
LARSEN A, MOHAMMED H, ARIAS C. Characterization of the gut microbiome of three commercially valuable warmwater fish species[J]. Journal of Applied Microbiology, 2014, 116: 1396-1404. DOI:10.1111/jam.12475 |
| [13] |
YE L, AMBERG J, CHAPMAN D, GAIKOWSKI M, LIU W. Fish gut microbiota analysis differentiates physiology and behavior of invasive Asian carp and indigenous American fish[J]. The ISME Journal, 2014, 8: 541-551. DOI:10.1038/ismej.2016.71 |
| [14] |
DONG B, YI Y, LIANG L. High throughput identification of antimicrobial peptides from fish gastrointestinal microbiota[J]. Toxins, 2017, 9: 266. DOI:10.3390/toxins9090266 |
| [15] |
LI D, LUO R, LIU C M, LEUNG, C M, TING H F, SADAKANE K, YAMASHITA H, LAM T W. MEGAHIT v1.0: A fast and scalable metagenome assembler driven by advanced methodologies and community practices[J]. Methods, 2016, 102: 3-11. DOI:10.1016/j.ymeth.2016.02.020 |
| [16] |
MENZEL P, NG K, KROGH A. Fast and sensitive taxonomic classification for metagenomics with Kaiju[J]. Nature Communications, 2016, 7: 11257. DOI:10.1038/ncomms11257 |
| [17] |
EDGAR R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10: 996-998. DOI:10.1038/nmeth.2604 |
| [18] |
NI J, YAN Q, YU Y. Factors inf luencing the grass carp gut microbiome and its effect on metabolism[J]. FEMS Microbiology Ecology, 2014, 87: 704-714. DOI:10.1111/1574-6941.12256 |
| [19] |
LEY R E, LOZUPONE C A, HAMADY M, KNIGHT R, GORDON J I. Worlds within worlds: evolution of the vertebrate gut microbiota[J]. Nature Review Microbiology, 2008, 6: 776-788. DOI:10.1038/nrmicro1978 |
| [20] |
KOHL K D, AMAYA J, PASSEMENT C A, DEARING M D, MCCUE M D. Unique and shared responses of the gut microbiota to prolonged fasting: a comparative study across five classes of vertebrate hosts[J]. FEMS Microbiology Ecology, 2014, 90: 883-894. DOI:10.111/1574-6941.12442 |
| [21] |
WU S G, REN Y, PENG C, HAO Y, XIONG F, WANG G, LI W, ZOU H, ANGERT E R. Metatranscriptomic discovery of plant biomass-degrading capacity from grass carp intestinal microbiomes[J]. FEMS Microbiology Ecology, 2015, 91: fiv107. DOI:10.1093/femsec/fiv107 |
| [22] |
YE L, AMBERG J, CHAPMAN D, GAIKOWSKI M, LIU W T. Fish gut microbiota analysis differentiates physiology and behavior of invasive Asian carp and indigenous American fish[J]. The ISME Journal, 2013, 8: 541-551. DOI:10.1038/ismej.2016.71 |
| [23] |
NI J J, YAN Q Y, Y Y H, ZHANG T L. Factors influencing the grass carp gut microbiome and its effect on metabolism[J]. FEMS Microbiology Ecology, 2014, 87: 704-714. DOI:10.1111/1574-6941.12256 |
| [24] |
KESSEL M, DUTILH B, NEVELING K, KWINT M, VELTMAN J A, FLIK G, JETTEN M, KLAREN P, CAMP H. Pyrosequencing of 16S rRNA gene ampliconsto study the microbiota in the gastrointestinal tract of carp (Cyprinuscarpio L.)[J]. AMB Express, 2011, 1: 41. DOI:10.1186/2191-0855-1-41 |
| [25] |
LI T T, LONG M, GATESOUPE F J, ZHANG Q Q, LI A H, GONG X N. Comparative analysis of the intestinal bacterial communities in different species of carp by pyrosequencing[J]. Microbial Ecology, 2014, 69: 25-36. DOI:10.1007/s00248-014-0480-8 |
| [26] |
LIU H, GUO X W, GOONERATNE R, LAI R F, ZENG C, ZHAN F B, WANG W M. The gut microbiome and degradation enzyme activity of wild freshwater fishes influenced by their trophic levels[J]. Scientific Reports, 2016, 6: 24340. DOI:10.1038/srep24340 |
| [27] |
CHEN W G, LIU F L, LING Z X, TONG X J, XIANG C. Human intestinal lumen and mucosa-associated microbiota in patients with colorectal cancer[J]. PLoS One, 2012, 7: e39743. DOI:10.1371/journal.pone.0039743 |
| [28] |
FISHER K, PHILLIPS C. The ecology, epidemiology and virulence of Enterococcus[J]. Microbiology, 2009, 155: 749-757. DOI:10.1099/mic.0.026385-0 |
| [29] |
DUAN Y F, ZHANG Y, DONG H B, WANG Y, ZHENG X T, ZHANG J S. Effect of dietary Clostridium butyricum on growth, intestine health status and resistance to ammonia stress in Pacific white shrimp Litopenaeus vannamei[J]. Fish & Shellfish Immunology, 2017, 65: 25-33. DOI:10.1016/j.fsi.2017.03.048 |
| [30] |
SABRY M, EL-MOEIN K, HAMZA E. Occurrence of Clostridium perfringens types A, E, and C in fresh fish and its public health significance[J]. Journal of Food Protection, 2016, 9: 994-1000. DOI:10.4315/0362-028x.jfp-15-569 |
| [31] |
GHOMRASSI H, BRAIEK O B, CHOISET Y, HAERTLÉ T, HANI K. Evaluation of marine bacteriocinogenic enterococci strains with inhibitory activity against fish-pathogenic gram-negative bacteria[J]. Diseases of Aquatic Organisms, 2016, 118: 31-43. DOI:10.3354/dao02953 |
| [32] |
WILSON B, DANILOWICZ B S, MEIJER W G. The diversity of bacterial communities associated with Atlantic cod Gadus morhua[J]. Microbial Ecology, 2008, 55: 425-434. DOI:10.1007/s00248-007-9288-0 |
| [33] |
TURNBAUGH P J, LEY R E, HAMADY M, LIGGETT F, KNIGHT R, GORDON J L. The human microbiome project[J]. Nature, 2007, 449: 804-810. DOI:10.1038/nature06244 |
| [34] |
ROESELERS G, MITTGE E K, STEPHENS W Z, PARICHY D M, CAVANAUGH C M, GUILLEMIN K, RAWLS J F. Evidence for a core gut microbiotain the zebrafish[J]. The ISME Journal, 2011, 5: 1595-1608. DOI:10.1038/ismej.2011.38 |
| [35] |
HENNERSDORF P, KLEINERTZ S, THEISEN S, THEISEN S, ABDUL-AZIZ M A, MROTZEK G, PALM H W, SALUZ H P. Microbial diversity and parasitic load in tropical fish of different environmental conditions[J]. PLoS One, 2016, 11: e0151594. DOI:10.1371/journal.pone.0151594 |
| [36] |
SULLAM K E, ESSINGER S D, LOZUPONE C A, O'CONNOR M P, ROSEN G L, KNIGHT R, KILHAM S S, RUSSELL J A. Environmental and ecological factors that shape the gut bacterial communities of fish: a meta-analysis[J]. Molecular Ecology, 2012, 21: 3363-3378. DOI:10.1111/j.1365-294x.2012.05552.x |
| [37] |
LARSEN A M, MOHAMMED H H, ARIAS C R. Characterization of the gut microbiota of three commercially valuable warmwater fish species[J]. Journal of Applied Microbiology, 2014, 116: 1396-1404. DOI:10.1111/jam.12475 |
| [38] |
EICHMILLER J J, HAMILTON M J, STALEY C, SADOWSKY M J, SORENSEN P W. Environment shapes the fecal microbiome of invasive carp species[J]. Microbiome, 2016, 4: 44. DOI:10.1186/s40168-016-0190-1 |
| [39] |
LLEWELLYN M S, BOUTIN S, HOSEINIFAR S H, DEROME N. Teleost microbiomes: the state of the art in their characterisation, manipulation and impor tance in aquaculture and fisheries[J]. Frontiers in Microbiology, 2014, 5: 1-17. DOI:10.3389/fmicb.2014.00207 |
| [40] |
LIU J H, BIAN G R, SUN D M, ZHU W Y, MAO S Y. Starter feeding supplementation alters colonic mucosal bacterial communities and modulates mucosal immune homeostasis in newborn lambs[J]. Frontiers in Microbiolog y, 2017, 8: 429. DOI:10.3389/fmicb.2017.00429 |
| [41] |
SABRY M, EL-MOEIN K, HAMZA E. Occurrence of Clostridium perfringens types A, E, and C in fresh fish and its public health significance[J]. Journal of Food Protection, 2016, 79: 994-1000. DOI:10.4315/0362-028x.jfp-15-569 |
| [42] |
YI Y H, YOU X X, BIAN C, CHEN S X, LYU A, QIU L M, SHI Q. High-throughput identification of antimicrobial peptides from amphibious mudskippers[J]. Marine Drugs, 2017, 15: E364. DOI:10.3390/md15110364 |
| [43] |
FOUREAU D M, MIELCARZ D W, MENARD L C, SCHULTHESS J, WERTS C, RYFFEL B, KASPER L H, GATLE D B. TLR9-dependent induction of intestinal alpha-defensins by Toxoplasma gondii[J]. Journal of Immunology, 2010, 184: 7022-7029. DOI:10.4049/jimmunol.0901642 |
| [44] |
STOCKINGER S, ALBERS T, DUERR C U, MÉNARD S, PÜTSEP K, ANDERSSON M D, HORNEF M W. Interleukin-13-mediated paneth cell degranulation and antimicrobial peptide release[J]. Journal of Innate Immunity, 2014, 6: 530-541. DOI:10.1159/000357644 |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49




