文章信息
基金项目
- 广东省基础与应用基础研究区域联合基金-重点项目(2019B515120061);广东省重点领域研发计划项目(2018B020202006);广东省农业科学院学科团队建设项目(202131TD);广东省科技计划项目(2020B121201013)
作者简介
- 江桐欣(1997—),女,在读硕士生,研究方向为植物抗病基因编辑,E-mail:740150563@qq.com; 刘文华,副研究员,分子育种技术研究室副主任,美国宾夕法尼亚州立大学植物生物学博士。主要从事农业生物功能基因组学和新育种技术方面的研究工作。主持或参与国家转基因生物新品种培育重大专项、广东省粤穗联合基金、广东省现代种业和广东省自然科学基金等基因组学和基因编辑育种相关项目12项,申请基因编辑国家发明专利6项,其中授权2项,发表学术论文4篇。建立了水稻、番茄和猪等农业生物无外源DNA的CRISPR RNP基因组编辑平台,获得了水稻抗白叶枯病和稻瘟病、番茄抗青枯病、猪抗蓝耳病和猪基因组编辑文库等农业生物新种质和育种材料.
通讯作者
- 刘文华(1968—),男,博士,副研究员,研究方向为基因编辑育种,E-mail:1827624353@qq.com.
文章历史
- 收稿日期:2022-09-08
2. 仲恺农业工程学院农业与生物学院,广东 广州 510225
2. College of Agriculture and Biology, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China
成簇的有规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)及其关联蛋白(CRISPR associated,Cas)是细菌和古细菌的获得性免疫体系。自CRISPR体系被改造成以RNA为导向、以Cas蛋白为效应蛋白的可程序化设计的核酸酶后,便取代以往的锌指核酸酶(Zinc finger nuclease,ZFN)和转录激活子类效应子核酸酶(Transcription activator-like effector nuclease,TALEN),成为基因编辑领域最常用的工具。在农业领域中,CRISPR基因编辑通常用来敲除基因,达到验证基因功能或创制新种质的目的。CRISPR核酸酶在基因位点产生的双链切口(DNA double strand break,DSB),经非同源末端连接修复(Non-homologous end joining,NHEJ)后,在切口处往往插入或缺失一定数目的核苷酸,引起移码突变而提前出现终止密码子,从而使基因编码产物失去正常功能。因为插入或缺失的核苷酸完全是随机性的,因而CRISPR基因敲除是一种非精准的基因编辑。但是,经过长期驯化形成的作物精英品种,往往会丢失一些优良基因,而要从野生种或者地方品种中引入无遗传拖拽(Genetic drag)的优良基因,需要在精英品种基因组的安全位点进行基因插入或等位基因位点进行基因替换。与CRISPR介导的基因敲除相比,CRISPR介导的基因插入和基因替换属于更为精准的基因编辑,是实现基因编辑自由的终极目标。
CRISPR精准基因编辑包括单碱基编辑(Base editing,BE)、引导编辑(Prime editing,PE)和基于同源重组(Homologous recombination,HR)的基因插入或替换。单碱基编辑是对碱基C或A进行置换产生T或G的编辑[1-6],引导编辑是对任意数个到几十个核苷酸进行突变、插入或替换的编辑[7-8]。由于单碱基和引导编辑利用的脱氨酶和逆转录酶被限制在CRISPR产生的双链切口附近,因而只能对切口附近的一个至数十个碱基进行编辑,不能满足长片段的插入或替换。基于同源重组的CRISPR精准编辑,即基因打靶(Gene targeting,GT),可在CRISPR靶位点产生双链切口后,在含有靶位点同源序列的供体DNA或RNA存在的情况下,利用细胞内在的同源定向修复(Homology directed repair,HDR)机制,在靶位点进行DNA大片段的插入或替换。本文从HDR修复机制、CRISPR组分(Cas9/12和sgRNA/crRNA)及修饰、供体DNA/RNA及修饰、CRISPR组分和供体DNA/RNA在植物遗传转化中的递送等方面进行综述,并对基于同源重组的CRISPR精准基因编辑技术在农作物基因功能分析和新技术育种中的应用前景进行展望。
1 DNA双链切口同源重组修复 1.1 SDSA途径介导的同源重组修复在植物中,DNA产生双链切口(DSB)后,细胞中存在两种主要修复机制,即非同源性末端连接修复(NHEJ)和同源定向修复(HDR),且发生NHEJ的概率远远高于HDR[9-11]。合成依赖链退火(Sequence-dependent strand anneling,SDSA)途径是植物体细胞中主要的基于同源重组的修复机制,其过程如下:基因组DNA产生双链切口后,在MRN复合体(Mre11、Rad50、Nbs1)等的作用下形成3' 突出末端。在含有与3' 突出末端同源的外源供体DNA存在的情况下,突出末端侵入供体DNA,与供体DNA同源臂互补配对,并以供体DNA为模板合成DSB缺失部分核苷酸,当合成到另一个同源臂时脱离供体DNA,返回与基因组DNA另一条链退火配对,这样外源供体DNA替换DSB处的基因,或在DSB处插入新的基因(图 1A)。此途径中CtIP、RAD52、RAD54等蛋白起到关键作用[12-14]。在植物减数分裂形成配子时,有一条与SDSA途径前半部分重叠的特殊同源重组修复途径,称为双链切口修复(Double-strand break repair,DSBR),双链切口修复除在切口处产生基因的插入或替换外,有时也会产生基因的交换和重组[15]。
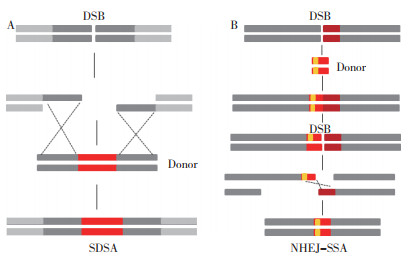
|
| A:SDSA途径;B:NHEJ-SSA途径 A: SDSA pathway; B: NHEJ-SSA pathway 图 1 DNA双链切口同源重组修复 Fig. 1 DNA double-strand break(DSB)repair via homology recombination |
1.2 NHEJ辅助的SSA途径介导的同源重组修复
DNA产生双链切口后,如果在切口两侧出现重复序列,细胞会采用单链退火修复(Single-strand annealing,SSA)连接两个断口。SSA的修复过程如下:在MRN复合体等的作用下,形成3' 突出末端,切口两侧的3' 突出末端在重复序列处退火配对,切除不配对的近3' 末端序列,最后在连接酶的作用下形成丢失重复序列之间序列的DNA[16]。有别于微同源介导的末端连接(Microhomology mediated end joining,MMEJ),SSA最短同源序列的长度一般在63~89 bp[17]。在植物中,RAD51家族蛋白XRCC2、RAD51B和RAD51D在SSA途径中起着重要作用[18]。
借助NHEJ修复,SSA也可被用于在基因组中插入或替换DNA片段[19],其过程如下:在细胞中引入含有待插入或替换序列和DSB一侧同源序列的供体DNA,当基因组DNA产生双链切口后,依赖NHEJ途径,使供体DNA无缝插入在DSB处,并重构DSB位点。当再次产生DSB后,SSA修复机制发生作用,最后实现DNA序列的插入或替换(图 1B)。
2 用于同源重组精准基因编辑的CRISPR核酸酶 2.1 Cas及融合蛋白Cas蛋白是CRISPR基因编辑系统的核心组分,这类来自细菌和古细菌病毒免疫系统的蛋白,自从被改造为基因编辑工具后,在植物基因插入或替换中便得到广泛应用。Li等[20]用Cas9和供体DNA在烟草原生质体中获得9% 的基因打靶(Gene targeting,GT)效率,但在拟南芥原生质体中未获得成功。Li等[21]利用Cas9和供体DNA对水稻内源的除草剂敏感基因5-烯醇丙酮莽草酸-3-磷酸合酶(EPSPS)基因进行替换,效率为2.0%。Shi等[22]利用Cas9和供体DNA在玉米中进行基因替换和插入,其效率分别为1% 和2.5%~4.1%。Wolter等[23]利用Cas12和供体DNA在拟南芥中进行基因打靶,效率为1.47%。Vu等[24]利用Cas12和供体DNA在番茄中也成功进行基因打靶,其效率因光照和温度的变化而变化,介于6%~10%。
使用原始的Cas9和供体DNA进行基因打靶,虽然其效率在不同植物中有所不同,但一般都在5% 以内,还有许多提升空间。考虑到DNA双链切口重组修复途径中的一些关键蛋白以及Cas9和供体DNA的共位性(Colocalization)可能对HDR效率提高产生的影响,近年来相继出现许多Cas9融合蛋白(图 2)。CtIP在DSB修复早期与5' 端切除有关,开启了基于同源重组的SDSA途径。在动物中,Charpentier等[25]采用Cas9-CtIP融合蛋白将基于同源重组的转基因整合效率提高近2倍。在HDR途径中,RAD52有助于3' 突出末端侵入供体DNA,Shao等[26]利用RAD52-Cas9融合蛋白,将人细胞和猪细胞中同源重组效率分别提高3倍和2.2倍。根据动物和植物HDR途径的相似性,在植物中使用Cas9-CtIP和Cas9-RAD52也有可能提高HR效率。与该推论一致的是,在杨树中过量表达CtIP并同时抑制NHEJ途径,可使基于同源重组的基因敲入效率提高48%[27]。除此类HDR途径关键蛋白外,另一类可直接结合或催化反应后结合双链或单链供体DNA的蛋白或酶,也可与Cas9形成融合蛋白,从而提高HR效率。VirD2是来自根瘤农杆菌(Agrobacterium tumefaciens)的毒性因子,能识别T-DNA 5' 端边界序列,继而通过其松弛酶活性产生单链切口,并与产生的单链DNA 5' 端共价结合,在T-DNA由农杆菌向植物细胞转位及其在植物细胞基因组整合过程中起着重要作用。利用VirD2与单链DNA的结合活性,Ali等[28]使用Cas9-VirD2融合蛋白在水稻中成功实现OsALS和OsCCD7基因的替换以及OsHDT基因HA标签的无缝整合,HR效率与Cas9相比提高4倍。链霉亲和素(Steptavidin,SA)是自然界中存在的能非共价结合4个生物素(Biotin)分子的蛋白,水溶性的单价链霉亲和素(mono-Streptavidin,mSA)也具有较强的生物素单分子结合活性[29]。我们实验室采用Cas9-mSA融合蛋白,在猪细胞基因组中进行Rosa26位点Green1基因的无缝整合,效率为6%,在水稻中也成功获得OsALS基因的替换植株,HR效率约为4%。逆转录子(Retron)是细菌中发现的病毒免疫相关操纵子,包括逆转录酶(Reverse transciptase,RT)基因和一段非编码RNA,非编码RNA能被RT逆转录成多拷贝单链DNA(Multicopy single-stranded DNA,msDNA),并形成RNA-msDNA共价复合体[30]。Kong等[31]在人细胞基因组中,利用Cas9-RTEMX1和HEK3位点进行替换,其效率高达10%。同样,Zhao等[32]在人基因组中,利用Cas9-RT进行精准基因编辑,HDR效率最高可达11.4%。借鉴动物中利用Cas9-RT提高HDR效率的成功经验,在植物中利用Cas9-RT进行精准基因编辑,有望获得相同效果。
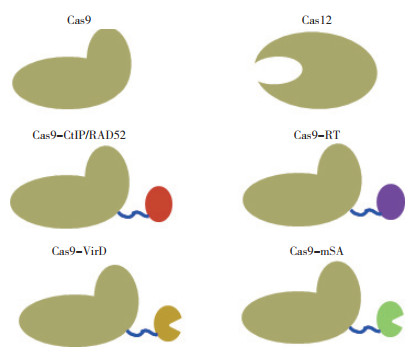
|
| 图 2 Cas及融合蛋白 Fig. 2 Cas and fusion proteins |
2.2 sgRNA/crRNA及修饰
CRISPR核酸酶的另一组分是与Cas蛋白结合的小分子RNA,称为sgRNA(single guide RNA)或crRNA(CRISPR RNA)。与Cas9蛋白结合的是sgRNA,与Cas12结合的是crRNA。sgRNA包括5' 端与靶序列互补配对的含20个核苷酸的引导序列以及3' 端具有茎环结构的83个或93个核苷酸的骨架序列(图 3)。为提高sgRNA在细胞中的表达量和稳定性,研究者对sgRNA进行序列和结构的优化,衍生出几种新型sgRNA,包括U-A碱基对换的sgRNA(sgRNA flipped)、茎环结构中配对碱基数增加10个的sgRNA(sgRNA extended)、结合两种修饰类型的sgRNA(sgRNA flipped and extended)[33]。crRNA包括5' 端能形成茎环机构的20、24或29个核苷酸的骨架序列,以及3' 端与靶序列互补配对的21个核苷酸的向导序列(图 3)。同样,对crRNA 5' 端进行延长,增加4个、9个等数目的核苷酸,均可提高基于NHEJ和HDR的基因编辑效率[34]。
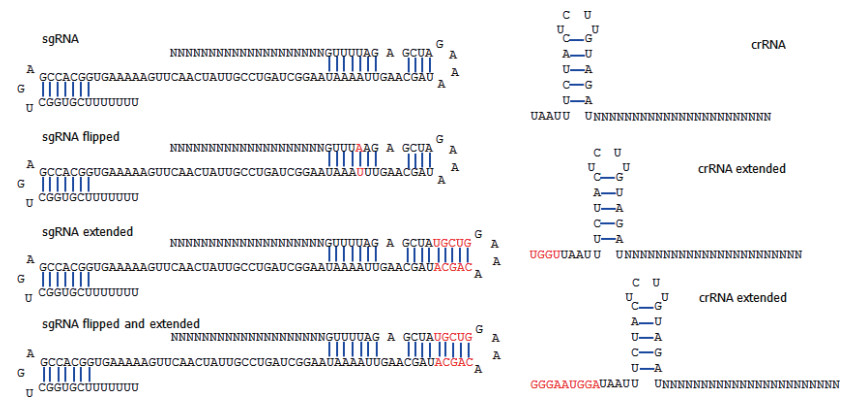
|
| 图 3 sgRNA/crRNA及修饰 Fig. 3 sgRNA/crRNA and modifiers |
3 用于同源重组精准基因编辑的供体DNA/RNA 3.1 双链供体DNA及修饰
用于同源重组精准基因编辑的双链供体DNA具有如下结构:5' 端和3' 端与靶位点同源的序列;中间为需要替换或插入的目标序列。与靶位点同源的序列也称为同源臂,其为基因组双链缺口产生后的3' 突出端侵入供体DNA提供互补配对序列,同源臂的长度为数百到数千碱基不等[35]。供体DNA可进行多种修饰,产生多种可提高HR效率的衍生体(图 4)。为防止细胞中脱氧核酸酶对双链供体DNA的降解,对双链供体DNA末端2~3个磷酸酯键进行硫代磷酸化(Phosophorothioation)修饰,有利于提高双链供体DNA在细胞中的稳定性。Gutierrez-Triana等[36]采用硫代硫酸化的供体DNA注射动物胚胎,极大地提高了HR效率。在水稻中,Lu等[19]硫代磷酸化双链供体DNA 4个末端,并磷酸化双链供体DNA 2个5' 末端,通过NHEJ辅助的SSA途径,获得6.1% 的基因替换效率。5' 端生物素标记(Biotinylation)的双链供体DNA,能结合Cas9-mSA融合蛋白,使供体DNA和DSB共位,从而提高HDR效率。我们实验室应用此方法成功获得OsALS基因的替换水稻,HR效率为4%。此外,有研究表明染色质及其结构影响HDR效率[37]。Cruz-Becerra等[38]将供体双链DNA染色质化并引入人细胞,与“裸露”双链DNA相比,HR介导的基因插入效率提高2.3~7.4倍。

|
| RB: T-DNA right border sequence; msDNA: Multi-copy single-stranded DNA 图 4 供体DNA/RNA及修饰 Fig. 4 Donor DNA/RNA and modifiers |
3.2 单链供体DNA/RNA及修饰
单链供体DNA与双链供体DNA一样,含有5' 和3' 末端同源臂,但对HDR效率的影响表现出不对称性(Asymmetry)的特点,实验发现PAM(Protospacer adjacent motif)远端同源臂较短、PAM近端同源臂较长,可提高HDR效率60%[39]。单链RNA也可作为HDR模板,Butt等[40]在水稻中表达sgRNA和模板RNA相连的嵌合RNA,获得OsALS基因替换的水稻植株。同样地,在水稻中,Li等[41]利用Cas12和在细胞核中表达crRNA和供体RNA,使HDR介导的OsALS基因替换效率达到1.7%~4.6%。为提高同源重组效率,单链供体DNA或RNA也可进行适当修饰(图 4)。在水稻中,Ali等[28]利用5' 端带有T-DNA右边界序列(Right border,RB)的供体单链DNA和Cas9-VirD2蛋白,通过VirD2识别和共价结合RB序列,使单链供体DNA和基因组DNA双链切口共位,从而使HR介导的基因插入或替换效率提高4倍。在逆转录子(Retron)基因编辑体系中,供体DNA为多拷贝单链DNA(msDNA),每份拷贝供体DNA的5' 端通过逆转录子结构性RNA(msr)间接连接在sgRNA上,这样当Cas9-逆转录酶(Cas9-RT)在细胞中产生双链切口时,多份拷贝单链供体DNA同时出现在双链切口处,极大地提高了HDR效率,在人细胞中HDR介导的基因替换效率大于10%[31-32]。逆转录子基因编辑在动物细胞中的成功应用为植物基因编辑工具的开发提供了一条崭新的途径。
4 植物遗传转化及CRISPR组分和供体DNA/RNA递送 4.1 依赖于组织培养的遗传转化及CRISPR组分和供体DNA/RNA递送像植物所有基因转化一样,植物基因插入或替换等精准基因编辑往往需要通过组织培养,在植物细胞中同时递送CRISPR组分及供体DNA或RNA,待细胞基因被编辑后,再诱导编辑细胞进行胚胎发生,获得再生植株(图 5A)。CRISPR组分及供体DNA或RNA可通过根癌农杆菌侵染、基因枪轰击和PEG/Ca2+转化植物愈伤组织或原生质体进入植物细胞,且它们在细胞中的位置和数量直接影响基因插入或替换的效率。
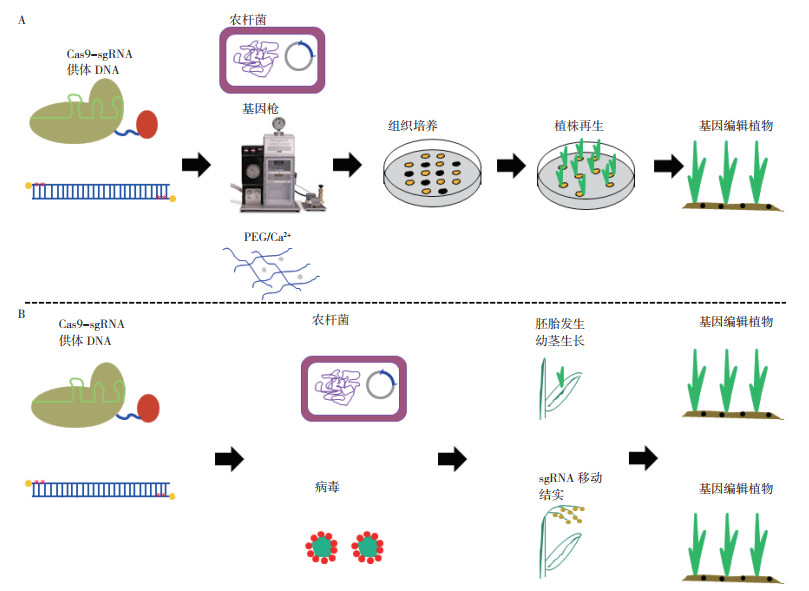
|
| A:依赖于组织培养的遗传转化;B:无组织培养过程的遗传转化 A: Tissue-culture dependent genetic transformation; B: Tissue-culture-free genetic transformation 图 5 植物遗传转化和CRISPR组分和供体DNA/RNA的递送 Fig. 5 Plant genetic transformation and delivery of CRISPR reagents and donor DNA/RNA |
将Cas9、sgRNA和两端含有sgRNA识别位点的供体DNA编码序列置于同一个农杆菌双元表达载体上,通过农杆菌侵染进入植物细胞并整合到植物基因组后,植物细胞核中就同时存在翻译产生的Cas9、转录产生的sgRNA以及CRISPR组分酶切释放的供体DNA,当CRISPR组分在靶基因处产生双链切口后,可保障在切口处有供体DNA的存在,从而实现靶基因的插入或替换,这个过程被称为植物原位基因打靶(in planta Gene targeting)[42]。植物原位基因打靶利用的供体DNA只有一份,在农杆菌双元载体中插入双生病毒(Geminivirus)复制子组分编码序列,可使供体DNA通过滚环复制形成多份拷贝,从而提高植物原位打靶的效率。Cermak等[43]利用含有豆类黄矮病毒(Bean yellow dwarf virus,BeYDV)复制子组分编码序列的双元载体,通过农杆菌转化,成功在ANT1基因编码序列前插入35S启动子,在获得的转化植株中,有2/3是HDR介导的精准插入。使用同样的供体DNA扩增策略,Wang等[44]利用小麦矮化病毒(Wheat dwarf virus,WDV)复制子,在水稻ACT1和GST位点分别实现19.4% 和7.7% 的精准替换。
利用基因枪也可将Cas9、sgRNA和供体DNA递送到组织细胞,再通过组织培养诱导分化产生基因编辑植株。基因枪转化可根据需要以DNA、RNA或核酸蛋白(Ribonucleoprotein,RNP)的形式引入CRISPR组分和供体DNA或RNA,且不受引入数量的限制,因而在水稻和玉米等作物中广泛使用。Ali等[28]使用基因枪转化法,将Cas9-VirD2、sgRNA和带有RB序列的供体DNA成功引入水稻愈伤组织细胞,分别获得OsALS基因被替换、OsCCD7基因被替换及OsHDT基因后无缝整合HA编码序列的水稻再生植株。Sun等[45]利用基因枪转化法,将含有Cas9、sgRNA和供体DNA编码序列的双元载体以及游离供体DNA,一同引入水稻愈伤组织细胞,成功获得OsALS的替换植株。Lu等[19]利用基因枪转化法,将Cas9和sgRNA编码质粒以及供体DNA引入水稻愈伤组织细胞,利用双链切口的NHEJ辅助的SSA途径,在基因组中多个位点进行基因插入或替换。Svitshev等[46]用基因枪法转化玉米幼胚,引入CRISPR组分和供体双链DNA或单链DNA,也成功获得ALS2替换植株。
PEG/Ca2+转化法可将Cas9和sgRNA组成的RNP以及供体DNA引入植物原生质体,实现基因的插入或替换等精准基因编辑。但对大多数植物来说,原生质体再生植株难度较高。目前,在莴苣和番茄等植物中,已有通过原生质体转化获得CRISPR基因敲除植株的报道[47-48],将来有望在这些植物中实施基于同源重组的更为精准的基因编辑。
4.2 无组织培养过程的遗传转化及CRISPR组分和供体DNA/RNA递送组织培养是限制植物基因转化或基因编辑效率的瓶颈之一,成熟植株从头诱导分生组织分化形成幼茎的方法可消除这一瓶颈的影响。在双子叶植物中,生长调节因子WUSCHEL2(Wus2)、SHOOT MERISTEMLESS(STEM)及植物细胞分裂素生物合成基因-异戊基转移酶基因(Isopentenyl transferase,Ipt)在分生组织发生和维护及幼茎分化中起着关键的作用。Maher等[49]将CRISPR组分及Wus2、STEM和Ipt的不同组合,通过农杆菌侵染幼苗或成熟植株,在植株上产生靶基因编辑的幼茎,切下幼茎,进一步在培养基中诱导生根,可获得基因敲除的植株。目前,这一避开植物组织培养、直接在幼苗和成熟植株上进行的基因递送,在Nicotiana benthamiana、番茄、马铃薯和葡萄中得以实现。
利用病毒将sgRNA递送到植物顶端分生组织,以期在后代种子中获得基因编辑植株,是避开组织培养的另一种有效方法。Ellison等[50]用烟草脆裂病毒(Tobacco rattle virus)递送融合了Flowering Locus T(FT)RNA的sgRNA到稳定表达Cas9的Nicotiana benthamiana植株顶端分生组织,在后代植株中有30% 发生多位点基因编辑。在小麦中,Li等[51]用大麦条纹花叶病毒(Barley strip mosaic virus)递送融合了FT RNA的sgRNA到植株顶端分生组织,在后代植株中有12.9%~100.0% 发生基因编辑。由于病毒递送受到载体容量影响,基因插入或替换需要递送除sgRNA外的供体DNA或RNA,因此将该技术用于基于同源重组的精准基因编辑具有一定的挑战性。
5 基于同源重组的CRISPR精准基因编辑技术应用前景展望在开发出基于NHEJ辅助的SSA介导的CRISPR基因插入或替换技术后,Lu等[52]呼吁在全球协作开展水稻蛋白标签计划(Rice Protein Tagging Project,RPTP),即在5年内对水稻全基因组编码蛋白进行原位FLAG或HA标签。预见在不久的将来,随着这一计划的顺利实施,水稻蛋白定位,蛋白与蛋白互作,蛋白与DNA、RNA、碳水化合物和脂肪等互作的研究,将获得长足发展。这种系统性地解析水稻全基因组编码蛋白功能的研究,将为水稻重要农艺性状的改良提供坚实的基础。
基于同源重组的CRISPR精准基因编辑技术还可直接应用于作物育种中。Dong等[53]利用该技术在水稻基因组的安全位点(Genomic safe harbor,GSH)插入类胡萝卜素生物合成途径中的两个关键酶基因(5.2 kb),获得类胡萝卜素在水稻种子中高水平表达而其他性状不受影响的“黄金大米”。作物中的许多复杂生命过程,如次生代谢产物合成、C3和C4光合作用、生物固氮等,往往涉及到多基因的协同表达和时空调控。随着同源重组介导的CRISPR精准基因编辑技术不断优化,可望在作物代谢产物流向控制、C3向C4光合作用转变及固氮效能提高等方面有所突破。在作物遗传改良中,许多性状是由具有不同的编码序列或启动子序列的等位基因决定的,利用基于同源重组的CRISPR基因编辑技术可克服传统育种的遗传拖拽现象,达到在精英品种中迅速引入并固定野生植物或农家品种中优良等位基因的目的。在作物杂交育种中,如果选育的目标性状是由隐型突变基因控制的,可利用有性生殖过程中的同源重组机制,构建隐性突变基因的CRISPR基因驱动(Gene drive),使隐性突变基因在杂种F1代中进行超孟德尔遗传,从而使优良性状在杂种F1代中迅速稳定下来。
生物基因组的自由撰写(编辑)是自基因组完整阅读(测序)以来合成生物学领域一直追求的目标,基于同源重组的CRISPR精准基因编辑技术在动植物中的成功应用,无疑是向这一目标迈进的一大步。随着对植物同源定向重组修复机制研究的更加深入和CRISPR基因编辑技术的不断革新,基于同源重组的CRISPR精准编辑技术必将在农作物功能基因组学研究和新技术育种中发挥越来越重要的作用。
| [1] |
KOMOR A C, KIM Y B, PACKER A S, ZURIS J A, LIU D R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533: 420-424. DOI:10.1038/nature17946 |
| [2] |
GAUDELLI N M, KOMOR A C, REES H A, PACKER M, BADRAN A H, BRYSON D I, LIU D R. Programmable base editing of A · T to G · C in genomic DNA without DNA cleavage[J]. Nature, 2017, 551: 467-471. DOI:10.1038/nature24644 |
| [3] |
ZONG Y, WANG Y P, LI C, ZHANG R, CHEN K L, RAN Y D, QIU J L, WANG D W, GAO C X. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion[J]. Nature Biotechnology, 2017, 35: 438-440. DOI:10.1038/nbt.3871 |
| [4] |
LU Y, ZHU J K. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system[J]. Molecular Plant, 2017, 10: 523-525. DOI:10.1016/j.molp.2016.11.013 |
| [5] |
LI J, SUN Y, DU J, ZHAO Y, XIA L. Generation of targeted point mutations in rice by a modified CRISPR/Cas9 system[J]. Molecular Plant, 2017, 10: 526-529. DOI:10.1016/j.molp.2016.12.001 |
| [6] |
SHIMATANI Z, KASHOJIYA S, TAKAYAMA M, TERADA R, ARAZOE T, ISHⅡ H, TERAMURA H, YAMAMOTO T, KOMATSU H, EZURA H, NISHIDA K, ARⅡZUMI T, KONDO A. Targeted base editing in rice and tomato using a CRISPR-Cas9 cytidine deaminase fusion[J]. Nature Biotechnology, 2017, 35: 441-443. DOI:10.1038/nbt.3833 |
| [7] |
ANZALONE A, RANDOLPH P, DAVIS J, SOUSA A, KOBLAN L W, LEVY J M, CHEN P J, WILSON C, NEWBY G A, RAGURAM A, LIU D R. Search-and-replace genome editing without double-strand breaks or donor DNA[J]. Nature, 2019, 576: 149-157. DOI:10.1038/s41586-019-1711-4 |
| [8] |
LIN Q, ZONG Y, XUE C, WANG S, JIN S, ZHU Z, WANG Y, ANZALONE A V, DOMAN J L, LIU D R, GAO C X. Prime genome editing in rice and wheat[J]. Nature Biotechnology, 2020, 38: 582-585. DOI:10.1038/s41587-020-0455-x |
| [9] |
JOHNSON R D, JASIN M. Double-strand-break-induced homologous recombination in mammalian cells[J]. Biochemical Society Transactions, 2001, 29: 196-201. DOI:10.1042/0300-5127:0290196 |
| [10] |
MANOVA V, GRUSZKA D. DNA damage and repair in plants – From models to crops[J]. Frontiers in Plant Science, 2015, 6: 885. DOI:10.3389/fpls.2015.00885 |
| [11] |
SCHMIDT C, PACHER M, PUCHTA H. DNA break repair in plants and its application for genome engineering[J]. Methods in Molecular Biology, 2019, 1864: 237-266. DOI:10.1007/978-1-4939-8778-8_17 |
| [12] |
CHARPENTIER M, KHEDHER A H Y, MENORET S, BRION A, LAMRIBET K, DARDILLAC E, BOIX C, PERROUAULT L, TESSON L, GENY S, CIAN A D, ITIER J M, ANEGON I, LOPEZ B, GIOVANNANGELI C, CONCORDET J P. CtIP fusion to Cas9 enhances transgene integration by homology-dependent repair[J]. Nature Communications, 2018, 9: 1133. DOI:10.1038/s41467-018-03475-7 |
| [13] |
SAMACH A, MELAMED-BESSUDO C, AVIVI_RAGOLSKI N, PIETROKOVSKI, LEVY A A. Identification of plant RAD52 homologs and characterization of the Arabidopsis thaliana RAD52-like genes[J]. Plant Cell, 2011, 23: 4266-4279. DOI:10.1105/tpc.111.091744 |
| [14] |
MANNUSS A, DUKOWIC-SCHULZE S, SUER S, HARTUNG F, PACHER M, PUCHTA H. RAD5A, RECQ4A, and MUS81 have specific functions in homologous recombination and define different pathways of DNA repair in Arabidopsis thaliana[J]. Plant Cell, 2010, 22: 3318-3330. DOI:10.1105/tpc.110.078568 |
| [15] |
OSMAN K, HIGGINS J D, SANCHEZ-MORAN E, ARMSTRONG S J, FRANKLIN F C H. Pathways to meiotic recombination in Arabidopsis thaliana[J]. The New Phytologist, 2011, 190: 523-544. DOI:10.1111/j.1469-8137.2011.03665.x |
| [16] |
FAUSER F, PUCHTA H. Gene targeting in plants: 25 years later[J]. The international journal of developmental biology, 2013, 57: 629-637. DOI:10.1387/ijdb.130194hp |
| [17] |
SUGAWARA N, IRA G, HABER J E. DNA length dependence of the single-strand annealing pathway and the role of Saccharomyces cerevisiae RAD59 in double-strand break repair[J]. Molecular and Cellular Biology, 2000, 20: 5300-5309. DOI:10.1128/MCB.20.14.5300-5309.2000 |
| [18] |
SERRA H, DA INES O, DEGROOTE F, GALLEGO M E, WHITE C I. Roles of XRCC2, RAD51B and RAD51D in RAD51-independent SSA recombination[J]. PLoS Genetics, 2013, 9: e1003971. DOI:10.1371/journal.pgen.1003971 |
| [19] |
LU Y, TIAN Y, SHEN R, YAO Q, WANG M, CHEN M, DONG J, ZHANG T, LI F, LEI M, ZHU J. Targeted, efficient sequence insertion and replacement in rice[J]. Nature Biotechnology, 2020, 38: 1402-1407. DOI:10.1038/s41587-020-0581-5 |
| [20] |
LI J, NORVILLE J E, AACH J, McCORMACK M, ZHANG D, BUSH J, CHURCH G M, SHEEN J. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nature Biotechnology, 2013, 31: 688-691. DOI:10.1038/nbt.2654 |
| [21] |
LI J, MENG X, ZONG Y, CHEN K, ZHANG H, LIU J, LI J, GAO C. Gene replacements and insertions in rice by intron targeting using CRISPR-Cas9[J]. Nature Plants, 2016, 2: 16139. DOI:10.1038/nplants.2016.139 |
| [22] |
SHI J, GAO H, WANG H, LAFITTE H R, ARCHBALD R L, YANG M, HAKIMI S M, MO H, HABBEN J E. ARGOS8 variants generated by CRISPR-Cas9 improve maize grain yield under field drought stress[J]. Plant Biotechnology Journal, 2017, 15: 207-216. DOI:10.1111/pbi.12603 |
| [23] |
WOLTER F, PUCHTA H. In planta targeting can be enhanced by the use of CRISPR/Cas12a[J]. The Plant Journal, 2019, 100: 1083-1094. DOI:10.1111/tpj.14488 |
| [24] |
VU T V, SIVANKALYANI V, KIM E, DOAN D T H, TRAN M T, KIM J, SUNG Y W, PARK M, KANG Y J, KIM J. Highly efficient homology-directed repair using CRISPR/Cpf1-geminiviral replicon in tomato[J]. Plant Biotechnology Journal, 2020, 18: 2133-2143. DOI:10.1111/pbi.13373 |
| [25] |
CHARPENTIER M, KHEDHER A H Y, MENORET S, BRION A, LAMRIBET K, DARDILLAC E, BOIX C, PERROUAULT L, TESSON L, GENY S, DE CIAN A, ITIER J M, ANEGON I, LOPEZ B, GIOVANNANGELI C, CONCORDET J P. CtIP fusion to Cas9 enhances transgene integration by homology-dependent repair[J]. Nature Communications, 2018, 9: 1133. |
| [26] |
SHAO S, REN C, LIU Z, BAI Y, CHEN Z, WEI Z, WANG X, ZHANG Z, XU K. Enhancing CRISPR/Cas9-mediated homology-directed repair in mammalian cells by expression Saccharomyces cerevisiae Rad52[J]. The International Journal of Biochemistry and Cell Biology, 2017, 92: 43-52. DOI:10.1016/j.biocel.2017.09.012 |
| [27] |
MOVAHEDI A, WEI H, ZHOU X, FOUNTAIN J C, CHEN Z, MU Z, SUN W, ZHANG J, LI D, GUO B, VARSHNEY R, YANG L, ZHUGE Q. Precise exogenous insertion and sequence replacements in poplar by simultaneous HDR overexpression and NHEJ suppression using CRISPR-Cas9[J]. Horticulture Research, 2022, 9: 154. DOI:10.1093/hr/uhac154 |
| [28] |
ALI Z, SHAMI A, SEDEEK K, KAMEL R, ALHABSI A, TEHSEEN M, HASSAN N, BUTT H, KABABJI A, HAMDAN S, MAHFOUZ M M. Fusion of the Cas9 endonuclease and the VirD2 relaxase facilitates homology-directed repair for precise genome engineering in rice[J]. Coummunication Biology, 2020, 3: 44. DOI:10.1038/s42003-020-0768-9 |
| [29] |
LIM K H, HUANG H, PRALLE A, PARK S. Stable, high-affinity streptavidin monomer for protein labeling and monovalent biotin detection[J]. Biotechnology and Bioengineering, 2013, 110: 57-67. DOI:10.1002/bit.24605 |
| [30] |
SIMON A, ELLINGTON A D, FINKELSTEIN I J. Retrons and their applications in genome engineering[J]. Nucleic Acids Research, 2019, 47: 11007-11019. DOI:10.1093/nar/gkz865 |
| [31] |
KONG X, WANG Z, ZHANG R, WANG X, ZHOU Y, SHI L, YANG H. Precise genome editing without exogenous donor DNA via retron editing system in human cells[J]. Protein Cell, 2021, 12: 899-902. DOI:10.1007/s13238-021-00862-7 |
| [32] |
ZHAO B, CHEN S, LEE J, FRASER H. Bacterial retrons enable precise gene editing in human cells[J]. The CRISPR Journal, 2022, 5: 31-39. DOI:10.1089/crispr.2021.0065 |
| [33] |
CHEN B, GILBERT L, CIMINI B A, SCHNITZBAUER J, ZHANG W, LI G, PARK J, BLACKBURN E H, WEISSMAN J S, QI L S, HUANG B. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system[J]. Cell, 2013, 155: 1479-1491. DOI:10.1016/j.cell.2013.12.001 |
| [34] |
PARK H M, LIU H, WU J, CHONG A, MACKLEY V, FELLMANN C, RAO A, JIANG F, CHU H, MURTHY N, LEE K. Extension of the crRNA enhances Cpf1 gene editing in vitro and in vivo[J]. Nature Communications, 2018, 9: 3313. DOI:10.1038/s41467-018-05641-3 |
| [35] |
SHY B R, MACDOUGALL M S, CLARKE R, MERRILL B. Co-incident insertion enables high efficiency genome engineering in mouse embryonic stem cells[J]. Nucleic Acids Research, 2016, 44: 7997-8010. DOI:10.1093/nar/gkw685 |
| [36] |
GUTIERREZ-TRIANA J A, TAVHELIDSE T, THUMBERGER T, THOMAS I, WITTBRODT B, KELLNER T, ANLAS K, TSINGOS E, WITTBRODT J. Efficient single-copy HDR by 5' modified long dsDNA donors[J]. Elife, 2018, 7: e39468. DOI:10.7554/eLife.39468 |
| [37] |
JANSSEN J M, CHEN X, LIU J, GONCALVES M A F V. The chromatin structure of CRISPR-Cas9 target DNA controls the balance between mutagenic and homology-directed fgene-editing events[J]. Molecular Therapy, Nucleic Acids, 2019, 16: 141-154. DOI:10.1016/j.omtn.2019.02.009 |
| [38] |
CRUZ-BECERRA G, KADONAGA J T. Enhancement of homology-directed repair with chromatin donor templates in cells[J]. Elife, 2020, 9: e55780. DOI:10.7554/eLife.55780 |
| [39] |
RICHARDSON C D, Ray G J, DEWITT M A, CURIE G L, CORN J E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA[J]. Nature Biotechnology, 2016, 34: 339-344. DOI:10.1038/nbt.3481 |
| [40] |
BUTT H, EID A, ALI Z, ATIA M A M, MOKHTAR M M, HASSAN N, LEE C M, BAO G, MAHFOUZ M M. Efficient CRISPR/Cas9-mediated genome editing using a chimeric single-guide RNA molecule[J]. Frontiers in Plant Science, 2017, 8: 1441. DOI:10.3389/fpls.2017.01441 |
| [41] |
LI S, LI J, HE Y, XU M, ZHANG J, DU W, ZHAO Y, XIA L. Precise gene replacement in rice by RNA transcript-templated homologous recombination[J]. Nature Biotechnology, 2019, 37: 445-450. DOI:10.1038/s41587-019-0065-7 |
| [42] |
SCHIML S, FAUSER F, PUCHTA H. The CRISPR/Cas system can be used as nuclease for in planta gene targeting and as paired nickases for directed mutagenesis in Arabidopsis resulting in heritable progeny[J]. The Plant Journal, 2014, 80: 1139-1150. DOI:10.1111/tpj.12704 |
| [43] |
CERMAK T, BALTES N J, CEGAN R, ZHANG Y, VOYTAS D F. High-frequency, precise modification of the tomato genome[J]. Genome Biology, 2015, 16: 232. DOI:10.1186/s13059-015-0796-9 |
| [44] |
WANG M, LU Y, BOTELLA J R, MAO Y, HUA K, ZHU J. Gene targeting by homology_directed repair in rice using a geminivirus-based CRISPR/Cas9 system[J]. Molecular Plant, 2017, 10: 1007-1010. DOI:10.1016/j.molp.2017.03.002 |
| [45] |
SUN Y, ZHANG X, WU C, HE Y, MA Y, HOU H, GUO X, DU W, ZHAO Y, XIA L. Engineering herbicide-resistant rice plants through CRISPR/Cas9-mediated homologous recombination of acetolactate synthase[J]. Molecular Plant, 2016, 9: 628-631. DOI:10.1016/j.molp.2016.01.001 |
| [46] |
SVITASHEV S, YOUNG J K, SCHWARTZ C, GAO H, FALCO S C, CIGAN A M. Targeted mutagenesis, precise gene editing, and site-specific gene insertion in maize using Cas9 and guide RNA[J]. Plant Physiol, 2015, 169: 931-945. DOI:10.1104/pp.15.00793 |
| [47] |
CHOI S H, AHN W S, JIE E Y, CHO H, KIM S W. Development of late-bolting plants by CRISPR/Cas9-mediated genome editing from mesophyll protoplasts of lettuce[J]. Plant Cell Reports, 2022, 41: 1627-1630. DOI:10.1007/s00299-022-02875-w |
| [48] |
LIN C, HSU C, YUAN Y, ZHENG P, WU F, CHENG Q, WU Y, WU T, LIN S, YUE J, CHENG Y, LIN S, SHIH M, SHEEN J, LIN Y. DNA-free CRISPR-Cas9 gene editing of wild tetraploid tomato Solanum peruvianum using protoplast regeneration[J]. Plant Physiology, 188: 1917-1930. DOI:10.1093/plphys/kiac022 |
| [49] |
MAHER M F, NASTI R A, VOLLBRECHT M, STARKER C G, CLARK M D, VOYTAS D F. Plant gene editing through de novo induction of meristems[J]. Nature Biotechnology, 2020, 38: 84-89. DOI:10.1038/s41587-019-0337-2 |
| [50] |
ELLISON E E, NAGALAKSHMI U, GAMO M E, HUANG P, DINESH-KUMAR S, VOYATAS D F. Multiplexed heritable gene editing using RNA viruses and mobile single guide RNAs[J]. Nature Plants, 2020, 6: 620-624. DOI:10.1038/s41477-020-0670-y |
| [51] |
LI T, HU J, SUN Y, LI B, ZHANG D, LI W, LIU J, LI D, GAO C, ZHANG Y, WANG Y. Highly efficient heritable genome editing in wheat using an RNA virus and bypassing tissue culture[J]. Molecular Plant, 2021, 11: 1787-1798. DOI:10.1016/j.molp.2021.07.010 |
| [52] |
LU Y, RONALD P C, HAN B, LI J, ZHU J. Rice protein tagging project: a call for international collaborations on genome-wide in-locus tagging of rice proteins[J]. Molecular Plant, 2020, 12: 1663-1665. DOI:10.1016/j.molp.2020.11.006 |
| [53] |
DONG O X, YU S, JAIN R, ZHANG N, DUONG P Q, BUTLER C, LI Y, LIPZEN A, MARTIN J A, BARRY K W, SCHMUTZ J, TIAN L, RONALD P C. Marker-free carotenoid-enriched rice generated through targeted gene insertion using CRISPR-Cas9[J]. Nature Communications, 2020, 11: 1178. DOI:10.1038/s41467-020-14981-y |
(责任编辑 张辉玲)
 2022, Vol. 49
2022, Vol. 49




