文章信息
基金项目
- 福建省财政专项-福建省农业科学院创新团队建设项目(CXTD2021-2-14,STIT2017-2-8)
作者简介
- 钟礼义(1968—),男,推广研究员,研究方向为食药用菌育种,E-mail:fjzly99@163.com.
通讯作者
- 陈体强(1968—),男,硕士,研究员,研究方向为药用真菌学,E-mail:chen_tiqiang@189.cn.
文章历史
- 收稿日期:2021-09-02
2. 武平县食用菌技术推广服务站,福建 武平 364300;
3. 福建农林大学菌物研究中心,福建 福州 350002
2. Wuping Mushroom Technical Advice Station, Wuping 364300, China;
3. Mycological Research Center, Fujian Agriculture and Forestry University, Fuzhou 350002, China
【研究意义】紫芝是福建、广东、广西等华南地区较为常见的灵芝科灵芝属药用真菌[1]。已知人工栽培的紫芝菌种主要为野外分离和驯化的菌株,如福建的闽紫96、武芝2号(原名紫芝S2),广东的HR-G32、育山1号,海南的HN-1[2-6]。其中武芝2号是从福建龙岩梁野山自然保护区多个野生分离株中,经多年驯化栽培选育得到的优良菌株,菌盖大,多数可达20 cm以上;经多年多点区域试验和Finlay稳定性分析鉴定,武芝2号稳定性好、适应性强,受栽培环境因素影响较小,平均单产(干品)可达25.05 kg/m3,2012年获得福建省非主要作物新品种认定[7-8]。实际上,在获得新品种认定后的推广应用中,从大田棚栽培到林下种植,从福建龙岩到广东河源、广西灌阳等产区,都观察到武芝2号子实体在形态发育上存在一定的表型可塑性(菌柄中生、近中生或假中生),对进一步申请品种权做DUS测试带来一定难度。因此,选择简单而可靠的分子标记进行新品种分子鉴定显得非常重要。【前人研究进展】据报道,从我国不同产地采集的被鉴定为同种的20份野生紫芝标本具有高度表型可塑性,单纯从形态分类上有可能是属于紫芝组的不同种类,但基于ITS、SSU、LSU(18S、28S rRNA基因)等多基因序列联合标记被鉴定为紫芝Ganoderma sinense同一种类[9]。上述菌株HR-G32和育山1号的栽培形态虽有较大差别(菌盖大小分别为4.3~5.2 cm和5~17 cm),但其ITS序列与紫芝G. sinense的同源性均达100%[6]。苏春丽等[10]利用ITS序列聚类分析,可以准确把34个中国栽培灵芝菌株中属于紫芝组Section Phaeonema的菌株基本上各自聚成一组。可见,利用ITS分子标记对灵芝保藏菌种进行分类是一种比形态学特征分类更准确有效的方法。已知真菌核糖体DNA(Ribosomal DNA,rDNA)的内转录间隔区ITS为中度保守区域,其保守性基本上表现为种内相对一致,种间差异比较明显,其种间特异性分子标记可用于灵芝种类的鉴定[11-12]。
【本研究切入点】本研究通过Sanger法测序得到武芝2号的ITS序列,针对灵芝属相关种类的ITS序列构建系统进化树进行分类鉴定,然后从三代测序技术得到的全基因组中找出rDNA-ITS序列(包含部分18S rRNA基因序列、ITS1全序列、5.8S rRNA基因序列、ITS2全序列及部分28S rRNA基因序列),通过比对选出用于分类鉴定的有效分子标记。【拟解决的关键问题】定位武芝2号基因组rDNA-ITS的ITS1和ITS2区域,验证相关PCR引物扩增测序得到的ITS序列的准确性及紫芝特异性扩增引物的有效性,为紫芝栽培品种或菌株鉴定提供科学依据。
1 材料与方法 1.1 试验材料武芝2号由福建武平县食用菌技术推广服务站提供,现以Ganoderma sp. TQC-2021a strain Zizhi S2在NCBI数据库中注册并获得分类号(NCBI: txid2816325)。
1.2 试验方法1.2.1 ITS序列扩增、测序 武芝2号菌丝体培养、DNA提取纯化等步骤参照文献[13]。采用真菌通用引物ITS1_19bp(5–′TCCGTAGGTGAACCTGCGG–3)′和ITS4_20bp(5′–TCCTCCGCTTATTGATATGC–3′)进行PCR扩增。扩增产物经凝胶电泳检测后回收并纯化,进行Sanger双脱氧法测序[14-15]。所得序列经BioEdit v7.0.9.0软件编辑后,向NCBI网站提交申请GenBank登录号。
1.2.2 ITS序列分析 下载同期已发布的灵芝属相关种类与菌株的ITS序列,采用Bioedit v7.0中的ClustalW2软件进行序列多重比对,MEGA v5.10软件计算遗传距离,Neighbor-Joining构建系统发育进化树,运行1 000次Bootstrap检验评价系统发育树拓扑结构的可靠性[16],根据Kimura-2法计算各序列所代表菌株之间的进化距离[17]。
1.2.3 rDNA-ITS结构分析与序列比对 针对武芝2号核基因组71条Scaffolds(PacBio SMART第三代测序技术获得)[18],采用Barrnap v0.9软件预测rRNA基因簇(18S rRNA、5.8S rRNA、28S rRNA)所在位置、数目及分布情况;使用在线软件GNU-GPL(http://www.emerencia.org/FungalITSextractor.html)提取ITS1、ITS2区域[19];采用GSDS v2.0软件[20]绘制武芝2号rDNA片段的基因结构图。同时,针对紫芝分子鉴定的2个特异性引物GS1_22bp(5′-ACGGA CTGTG GAGCG GGCTC TG-3′)、GS2_22bp(5′-GTCAT A AGCT TTGTC TCCAT AC-3′)序列[21],通过BLAST比对定位出rDNA所在位置。
1.2.4 紫芝ITS2序列二级结构的比较 将上述提取得到的武芝2号ITS2序列(207 bp)输入ITS2数据库(http://its2.bioapps.biozentrum.uni-wuerzburg.de/)进行二级结构的预测,并与数据库中已有的紫芝G. sinense的ITS2二级结构进行比较。
2 结果与分析 2.1 ITS分子鉴定武芝2号菌株的分类归属经PCR扩增和凝胶电泳检测,结果发现1条长约650 bp、明亮清晰的DNA条带(图 1)。测序后编辑得到武芝2号的ITS序列全长649 bp,提交NCBI获得GenBank登录号为MG282563.1。

|
| 图 1 PCR扩增产物的凝胶电泳结果 Fig. 1 Gel electrophoresis result of amplified PCR products |
将灵芝属20个菌株的ITS序列,以猴头菇Hericium erinaceum的ITS序列为外群,构建系统发育进化树。结果(图 2)显示,武芝2号(Zizhi S2)与AY593864、AY593865、HQ235633、HQ235634等所对应的4个紫芝G. sinense菌株(XZ-G-C1、XZ-G-C2、AS5.69 type 1、AS5.69 type 2)处于同一分支,而与同属的其他灵芝种类如紫铜灵芝G. cupreum、树舌灵芝G. applanatum、赤芝G. lucidum和橡胶灵芝G. pseudoferreum则属于不同分支。由此可知,武芝2号菌株在分类上属于紫芝G. sinense,与其他4个紫芝菌株同属一个种群。
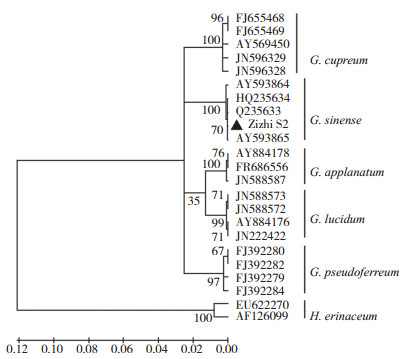
|
| AY593864.1、AY593865.1对应物种名称Ganoderma japonicum,是Ganoderma sinense的同物异名 The species name corresponding to the sequences AY593864.1 and AY593865.1 is Ganoderma japonica, a synonym of Ganoderma sinense 图 2 基于ITS构建的武芝2号与其他灵芝种类的NJ进化树 Fig. 2 NJ phylogenetic tree of Wuzhi No. 2 and other species of Ganoderma constructed based on ITS |
2.2 ITS序列在武芝2号基因组上的分布
通过比对发现,武芝2号ITS序列(MG282563.1)出现在基因组的Scaffold No.49(序列长度248 643 bp)上,共有4个重复序列(6 161~6 810、6 345~16 994、27 559~ 28 208、37 751~38 400 bp),序列相似度高达99.54%~100%,仅在第4个ITS区域中检出1个碱基的差异。结果表明,采用第一代测序技术Sanger法引物扩增测序得到的ITS序列,与第三代测序技术(PacBio SMART)长片段非扩增测序得到的4个串联ITS序列高度一致。
在GenBank中注册的武芝2号ITS序列(Sanger法测序),包含部分18S rRNA基因序列、ITS1全序列、5.8S rRNA基因序列、ITS2全序列及部分28S rRNA基因序列。武芝2号基因组rDNA-ITS结构如图 3所示,其中ITS1位于18S rRNA与5.8S rRNA基因序列之间,ITS2位于5.8S rRNA与28S rRNA基因序列之间,串联构成一个转录单元,而其后的5S rRNA基因为单独的转录单元。
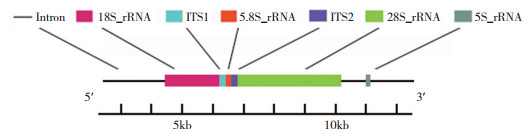
|
| 从18S rRNA 5′端到5S rRNA 3′端的序列全长约6 670 bp Total length of the sequence from 5′ end of 18S rRNA to 3′ end of 5S rRNA is about 6 670 bp 图 3 武芝2号的rDNA片段结构 Fig. 3 rDNA fragment structure of Wuzhi No. 2 |
2.3 紫芝分子鉴定特异性引物序列的验证
通过Blast比对发现,文献报道的用于紫芝分子鉴定的2个特异性引物短序列(GS1_22bp和GS2_22bp)[21]分别出现在本研究所得武芝2号的ITS序列119~140 bp和588~567 bp(反向)位置(图 4),比对结果验证了该紫芝特异性扩增引物的有效性。
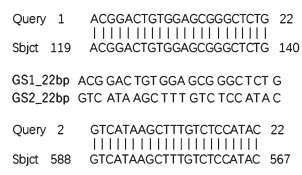
|
| 图 4 紫芝特异性引物与武芝2号ITS序列的比对 Fig. 4 Comparison of specific primers of Ganoderma sinense with ITS sequences of Wuzhi No. 2 |
2.4 紫芝ITS序列相似性及二级结构差异
BLAST在线比对结果进一步证实武芝2号(MG282563.1)与已知紫芝G. sinense菌株Zizhi的ITS序列(KT906369.1)完全一致,并与其他4个紫芝菌株的ITS序列相似度极高(99.53%~99.69%),仅有的差异出现在2~3个单碱基位点。而从基因组序列中提取得到的4个ITS2序列为207 bp(5′端TGAAA,3′端CCTC),即Sanger法测序得到的ITS序列的385~591 bp(5′端TGAAA,3′端CCTT),仅第4个ITS2序列存在1个单碱基位点的差异。
通过ITS2 Database在线预测获得ITS2序列的二级结构(图 5),武芝2号(Zizhi S2)与数据库中紫芝G. sinense的ITS2分子形态特征均由螺旋Ⅰ、Ⅱ、Ⅲ和Ⅳ区组成,其中两者的螺旋Ⅰ、Ⅱ、Ⅲ区一致性均为100%,螺旋Ⅳ区为75%,两者差异在于螺旋Ⅳ间夹角。
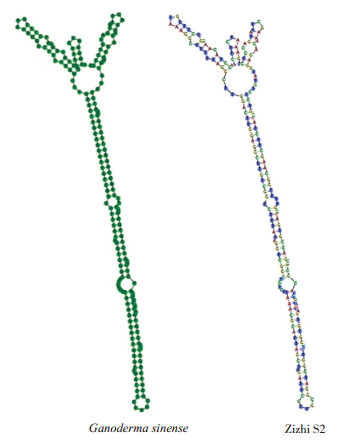
|
| 图 5 武芝2号与紫芝ITS2序列的二级结构比较 Fig. 5 Comparison of secondary structures of ITS2 sequence of Ganoderma sinense and Wuzhi No. 2 |
3 讨论
近年来,紫芝栽培新品种武芝2号在福建、广东和广西等地推广应用,已经形成规模化的栽培(年产量达3 000 t以上),但尚未通过DUS测试申请新品种权保护。由于表型可塑性的存在,选择有适度变化的子实体发育形态作为品种性状进行一致性评价显然不妥。本研究前期筛选得到的1个ISSR和2个RAPD-PCR引物短序列(ISSR13_14bp、S1326_10bp和S1506_10bp)可以作为鉴别紫芝栽培菌株的有效分子标记[7];其中S1326_10bp出现在武芝2号基因组中48条Scaffold上的158个位点,仅rDNA-ITS所在的Sacfold_49上就有8个位点(分布在非rDNA序列区),该位点丰富的简单重复序列区域具有很高的多态性。本研究基于第三代测序技术得到的武芝2号基因组Sacfold_49上找到4个串联分布的ITS序列,与Sanger法测序得到的武芝2号ITS序列(GenBank登录号:MG282563.1)及在GenBank上注册的紫芝ITS序列高度一致,可以用于紫芝栽培品种的分子鉴定。
本研究基于武芝2号基因组序列,针对文献报道的2个紫芝特异性引物序列(GS1_22bp、GS2_22bp)[21],通过Primer-BLAST反向验证,出现在武芝2号基因组Scaffold_49的rDNA-ITS序列上,其中GS1_22b分别位于ITS1区域的3′端和5.8S rRNA之前(即ITS_649bp的119~140 bp),GS2_22bp出现在ITS2区域的3′端与28S rRNA的5′端(即ITS_649bp的588~567 bp),可以认为是一种具有序列相关扩增多态性的有效分子标记。由此可见,基于ITS序列设计引物,建立快速鉴定灵芝属菌株种间特异性SRAP-PCR是可行的。
紫芝是我国的特有种类,长期混淆使用Ganoderma japonicum这个学名,直到1979年才作为新种Ganoderma sinense Zhao, Xu et Zhang正式定名,而Ganoderma japonicum则被视为Ganoderma sinense的同物异名[22-23]。本研究基于ITS序列比对结果,武芝2号G. sp. strain Zizhi S2(MG282563.1)与G. japonicum AS5.69 type 1(AY593864)、G. sinense strain XZ-G-C1(HQ235633)、G. sinense strain XZ-G-C2 (HQ235634)、G. japonicum AS5.69 type 2(AY593865)等4个紫芝菌株在ITS序列进化树上聚在同一个分支。
Sanger法测序得到的ITS序列,通常包括18S rRNA基因的3′端部分序列,ITS1、5.8S rRNA基因和ITS2全部序列,以及28S rRNA基因的5′端部分序列。其PCR扩增常用的一对引物为ITS1_19bp和ITS4_20bp,或ITS1和ITS5_22bp(5′-GGAAG GAGAA GTCGT AACAA GG-3′),其中ITS4_20bp位于28S rRNA上,而ITS5_22bp位于18S rRNA上。因此,不同研究者得到的ITS序列长度存在差异;如不同地区59份灵芝G. lucidum样品上得到的ITS序列在625~673 bp之间[24]。本研究构建进化树使用的紫芝ITS序列长度在649~675 bp之间,而在线BLAST比对的另外3个紫芝ITS序列长度KF494998为647 bp、KT906369为649 bp、KC415760为789 bp)。这是由于不同紫芝ITS序列中所包含的18S rRNA基因的3′端部分序列和28S rRNA基因的5′端部分序列的长度存在较大差异。
已知ITS2呈现更丰富的分子多样性,可以提供更多关于灵芝属种间和种内差异的序列信息[24];已知具有高度多样化群落的真菌,其ITS2序列区域受外界环境的影响较小,与编码区(18S和28S rRNA基因)相比具有进化速度快的特点[25]。本研究首次揭示了武芝2号与紫芝G. sinense的ITS2在二级结构上的分子形态特征。
以我国特有种类G. sinense J. D. Zhao, L. W. Hsu et X. Q. Zhang为代表的紫芝是灵芝科灵芝属的重要类群,其特征是菌肉呈均匀褐色、深褐色至栗褐色。在我国灵芝科中记载的紫芝组Sect. Phaeonema种类有26种,这个大种群以紫芝G. sinense为中心[22-23, 26]。因此,广义上的紫芝是灵芝科灵芝属紫芝组种类的统称,狭义上的紫芝是指G. sinense这个代表种。武芝2号在获得新品种认定之前,基于形态分类学特征(盖近圆形,中央略下凹,或似漏斗状) 曾被鉴定为紫芝的近缘种——硬孔灵芝G. duropora(标本号HMAS 95275,中国科学院菌物标本馆)[3]。但其担孢子形态与纹饰特征(宽卵圆形,顶端脐突较大,表面有纵向分布或长或短的沟、嵴和数条环状波纹) 与紫芝G. sinense基本相同[2]。本研究通过ITS/ITS2分子鉴定初步确认武芝2号与紫芝G. sinense同属一种。
紫芝全基因组(GenBank登录号:AYKW00 000000.1)早于2015年公布[27],其后续研究工作如通过挖掘紫芝基因组数据并利用某些新基因的研究近期才见报道[28-29]。本研究关于武芝2号的de novo测序计划始于2018年(基因组计划编号D1801921),组装、注释后的分析工作直至2021年才完成[18]。基于紫芝基因组的研究成果将为分子育种培育优良品种奠定坚实基础。
4 结论本研究采用Sanger法测序得到武芝2号的ITS序列全长649 bp(GenBank登录号:MG282563.1),由灵芝属不同种类20个菌株或实物标本ITS序列构建的系统发育进化树中,武芝2号与4个同物异名的紫芝菌株聚为一个分支,同属一个种群。通过比对发现,武芝2号ITS序列相应出现在基因组Scaffold No.49上rDNA中的4个串联重复区域,序列相似度高达99.54%~100%;采用第一代扩增测序技术得到的单个ITS序列,与第三代长片段非扩增测序技术得到的4个ITS序列高度一致。通过Blast比对发现,紫芝已知的2个特异性引物短序列(GS1_22bp、GS2_22bp)出现在武芝2号ITS序列(649 bp)的119~140 bp正向和588~567 bp反向位置,验证了基于ITS序列设计的特异性扩增引物是正确、有效的。比对结果还证实武芝2号与GenBank中5个紫芝ITS序列(KT906369.1)高相似度(99.53%~100%)。武芝2号ITS2序列的二级结构与ITS2数据库中紫芝G. sinense的4个螺旋区中,仅螺旋Ⅳ区间的夹角存在差异。
| [1] |
吴兴亮, 戴玉成. 中国灵芝科彩色图鉴[M]. 北京: 科学出版社, 2005. WU X L, DAI Y C. Colored Illustrations of Ganodermataceae of China[M]. Beijing: Science Press, 2005. |
| [2] |
陈体强, 吴锦忠, 李晔, 黄启东, 余德亿, 林兴生, 朱培根, 徐洁. 福建野生紫芝资源开发利用(Ⅱ').闽紫96'(中国灵芝)[J]. 菌物研究, 2006, 4(4): 27-32. DOI:10.3969/j.issn.1672-3538.2006.04.005 CHEN T Q, WU J Z, LI Y, HUANG Q D, YU D Y, LIN X S, ZHU P G, XU J. Exploitation and utilization of wild Zizhi resource(Sect. Phaeonema) in Fujian (Ⅱ) G. sinense 'Minzi 96'[J]. Journal of Fungal Research, 2006, 4(4): 27-32. DOI:10.3969/j.issn.1672-3538.2006.04.005 |
| [3] |
陈体强, 吴锦忠, 钟礼义, 邱福平, 林兴生, 朱培根. 福建野生紫芝资源开发利用(Ⅰ). 硬孔灵芝[J]. 福建农林大学学报(自然科学版), 2006, 35(3): 324-328. DOI:10.16768/j.issn.1671-5470.2006.03.022 CHEN T Q, WU J Z, ZHONG L Y, QIU F P, LIN X S, ZHU P G. Exploitation and utilization of wild Zizhi resources(Sect. Phaeonema) in Fujian Province (Ⅰ). Ganoderma duropora[J]. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2006, 35(3): 324-328. DOI:10.16768/j.issn.1671-5470.2006.03.022 |
| [4] |
陈逸湘, 宋斌, 李挺, 曾振基, 凌宏通. 紫芝研究进展[J]. 广东农业科学, 2011, 38(24): 36-39. DOI:10.16768/j.issn.1004-874X.2011.24.014 CHEN Y X, SONG B, LI T, ZENG Z J, LING H T. Research on advances of Ganoderma sinense[J]. Guangdong Agricultural Science, 2011, 38(24): 36-39. DOI:10.16768/j.issn.1004-874X.2011.24.014 |
| [5] |
刘月廉, 吴钿, 陈晓春. 来自广东河源两个灵芝菌株的比较鉴定[J]. 广东农业科学, 2011, 38(4): 123-125. DOI:10.3969/j.issn.1004-874X.2011.04.047 LIU Y L, WU D, CHEN X C. Comparative identification of two Ganoderma strains from Heyuan, Guangdong[J]. Guangdong Agricultural Science, 2011, 38(4): 123-125. DOI:10.3969/j.issn.1004-874X.2011.04.047 |
| [6] |
金鑫, 熊川, 李萍, 李强, 陈祖琴, 张璐, 黄文丽. 三株海南岛野生灵芝的鉴定, 多糖组成及其抗氧化活性研究[J]. 天然产物研究与开发, 2020, 32(2): 190-199. DOI:10.16333/j.1001-6880.2020.2.003 JIN X, XIONG C, LI P, LI Q, CHEN Z Q, ZHANG L, HUANG W L. Identification, polysaccharide composition and antioxidant activity of three wild Ganoderma strains in Hainan Island[J]. Natural Product Research and Development, 2020, 32(2): 190-199. DOI:10.16333/j.1001-6880.2020.2.003 |
| [7] |
钟礼义. 紫芝新品种武芝2号区域试验[J]. 中国食用菌, 2013, 32(6): 25-27. ZHONG L Y. The regional test on new varieties of ZIZHI(Ganoderma sinense)[J]. Edible Fungi of China, 2013, 32(6): 25-27. |
| [8] |
刘新锐, 王圣铕, 谢宝贵, 柯宾榕, 吴小平. 紫芝不亲和性因子分析[J]. 菌物学报, 2014, 33(2): 464-468. DOI:10.13346/j.mycosystema.130077 LIU X R, WANG S Y, XIE B G, KE B R, WU X P. Incompatibility factors of Ganoderma sinense[J]. Mycosystema, 2014, 33(2): 464-468. DOI:10.13346/j.mycosystema.130077 |
| [9] |
HAPUARACHCHI K, KARUNARATHNA S C, MCKENZIE E, KAKUMYAN P, WEN T C. High phenotypic plasticity of Ganoderma sinense (Ganodermataceae, Polyporales) in China[J]. Asian Journal of Mycology, 2019, 2(1): 1-47. DOI:10.5943/ajom/2/1/1 |
| [10] |
苏春丽, 唐传红, 张劲松, 陈明杰, 潘迎捷. 基于rDNA序列探讨中国栽培灵芝菌株的亲缘关系[J]. 微生物学报, 2007, 47(1): 11-16. DOI:10.3321/j.issn:0001-6209.2007.01.004 SU C L, TANG C H, ZHANG J S, CHEN M J, PAN Y J. The phylogenetic relationship of cultivated isolates of Ganoderma in China inferred from nuclear ribosomal DNA ITS sequences[J]. Acta Microbiologica Sinica, 2007, 47(1): 11-16. DOI:10.3321/j.issn:0001-6209.2007.01.004 |
| [11] |
林剑伟, 阙友雄, 陈天生, 许莉萍, 张木清. 核糖体DNA的内转录间隔区序列标记在真菌分类鉴定中的应用[J]. 生物技术通讯, 2007, 18(2): 292-294. DOI:10.3969/j.issn.1009-0002.2007.02.035 LIN J W, QUE Y X, CHEN T S, XU L P, ZHANG M Q. Application of ribosomal DNA internal transcribed spacer in Fungi Taxonomy[J]. Letters in Biotechnology, 2007, 18(2): 292-294. DOI:10.3969/j.issn.1009-0002.2007.02.035 |
| [12] |
PARK Y J, KWON O C, SON E S, YOON D E, HAN W, NAM J Y, YOO Y B, LEE C S. Genetic diversity analysis of Ganoderma species and development of a specific marker for identification of medicinal mushroom Ganoderma lucidum[J]. African Journal of Microbiology Research, 2012, 6(25): 5417-5425. DOI:10.5897/AJMR12.846 |
| [13] |
钟礼义, 陈体强, 刘新锐, 应正河, 杨驰. 鉴别紫芝菌株的PCR引物筛选及其序列比对验证[J]. 福建农业学报, 2020, 35(7): 1-6. DOI:10.19303/j.issn.1008-0384.2020.07.005 ZHONG L Y, CHEN T Q, LIU X R, YING Z H, YANG C. Selection and sequence alignment of PCR primers for identifying Zizhi strain[J]. Fujian Journal of Agricultural Sciences, 2020, 35(7): 1-6. DOI:10.19303/j.issn.1008-0384.2020.07.005 |
| [14] |
黄龙花, 杨小兵, 张智, 胡惠萍, 张一帆, 吴清平. 基于ITS序列分析鉴定灵芝属菌种[J]. 中国食用菌, 2010, 9(1): 55-57. DOI:10.3969/j.issn.1003-8310.2010.01.017 HUANG L H, YANG X B, ZHANG Z, HU H P, ZHANG Y F, WU Q P. Identification of Ganoderma spawns based on ITS sequence analysis[J]. Edible Fungi of China, 2010, 9(1): 55-57. DOI:10.3969/j.issn.1003-8310.2010.01.017 |
| [15] |
贾定洪, 王波, 彭卫红, 黄忠乾, 谭伟, 甘炳成, 郑林用. 4个野生灵芝菌株的ITS序列分析[J]. 西南农业学报, 2012, 25(4): 1414-1416. DOI:10.3969/j.issn.1001-4829.2012.04.059 JIA D H, WANG B, PENG W H, HUANG Z Q, TAN W, GAN B C, ZHENG L Y. Analysis of ITS sequence in four wild Ganoderma lucidum strains[J]. Southwest China Journal of Agricultural Sciences, 2012, 25(4): 1414-1416. DOI:10.3969/j.issn.1001-4829.2012.04.059 |
| [16] |
FELSENSTEIN J. Confidence limits on phylogenies: An approach using the bootstrap[J]. Evolution, 1985, 39(4): 783-791. DOI:10.1111/j.1558-5646.1985.tb00420.x |
| [17] |
KIMURA M. Estimation of evolutionary distances between homologous nucleotide sequences[J]. Proceedings of the National Academy of Sciences, 1981, 78(1): 454-458. DOI:10.1073/pnas.78.1.454 |
| [18] |
陈体强, 徐晓兰, 石林春, 钟礼义. 紫芝栽培品种'武芝2号'(紫芝S2) 全基因组测序及分析[J]. 生物技术通报, 2021, 37(11): 39-53. DOI:10.13560/j.cnki.biotech.bull.1985.2021-1158 CHEN T Q, XU X L, SHI L C, ZHONG L Y. Sequencing and analysis of the whole genome of Zizhi cultivar'Wuzhi No.2'(Ganoderma sp. strain Zizhi S2)[J]. Biotechnology Bulletin, 2021, 37(11): 39-53. DOI:10.13560/j.cnki.biotech.bull.1985.2021-1158 |
| [19] |
NILSSON R H, VLDRE V, HARTMANN M, UNTERSEHER M, AMEND A, BERGSTEN J, KRISTIANSSON E, RYBERG M, JUMPPONEN A, ABARENKOV K. An open source software package for automated extraction of ITS1 and ITS2 from fungal ITS sequences for use in high-throughput community assays and molecular ecology[J]. Fungal Ecology, 2010, 3(4): 284-287. DOI:10.1016/j.funeco.2010.05.002 |
| [20] |
郭安源, 朱其慧, 陈新, 罗静初. GSDS: 基因结构显示系统[J]. 遗传, 2007, 29(8): 1023-1026. DOI:10.3321/j.issn:0253-9772.2007.08.021 GUO A Y, ZHU Q H, CHEN X, LUO J C. GSDS: a gene structure display server[J]. Hereditas, 2007, 29(8): 1023-1026. DOI:10.3321/j.issn:0253-9772.2007.08.021 |
| [21] |
苏春丽, 唐传红, 张劲松. 基于ITS序列的紫芝分子鉴定[J]. 成都医学院学报, 2014, 9(2): 121-123. DOI:10.3969/j.issn.1674-2257.2014.02.004 SU C L, TANG C H, ZHANG J S. Molecular detection of Ganoderma sinense based on nuclear ribosomal DNA ITS sequence[J]. Journal of Chengdu Medical College, 2014, 9(2): 121-123. DOI:10.3969/j.issn.1674-2257.2014.02.004 |
| [22] |
赵继鼎, 徐连旺, 张小青. 中国灵芝亚科的分类研究[J]. 微生物学报, 1979, 19(3): 265-279. ZHAO J D, XU L W, ZHANG X Q. Taxonomic studies on the subfamily Ganodermoideae of China[J]. Acta Microbiologica Sinica, 1979, 19(3): 265-279. |
| [23] |
赵继鼎. 中国灵芝科的分类研究XI.灵芝亚属紫芝组[J]. 菌物学报, 1987, 7(4): 205-211. ZHAO J D. Studies on the taxonomy of Ganodermataceae in China X. Subgen. Ganoderma Sect. Phaenema[J]. Mycosystema, 1987, 7(4): 205-211. |
| [24] |
WANG D M, YAO Y J. Intrastrain internal transcribed spacer heterogeneity in Ganoderma species[J]. Canadian Journal of Microbiology, 2005, 51(2): 113-121. DOI:10.1139/w04-118 |
| [25] |
BAZZICALUPO AL, BÁLINT M, SCHMITT I. Comparison of ITS1 and ITS2 rDNA in 454 sequencing of hyperdiverse fungal communities[J]. Fungal Ecology, 2013, 6(1): 102-109. DOI:10.1016/j.funeco.2012.09.003 |
| [26] |
赵继鼎, 张小青. 中国真菌志(第十八卷)·灵芝科[M]. 北京: 科学出版社, 2000. ZHAO J D, ZHANG X Q. Chinese fungi Chronicles (Tomus 18·Ganodermataceae)[M]. Beijing: Science Press, 2000. |
| [27] |
ZHU YJ, XU J, SUN C, QIAN J, SONG J Y, LUO H M, LI X, LI Y, XU Z C, JI A J, WANG L Z, LU S F, CHEN SL. Chromosome-level genome map provides insights into diverse defense mechanisms in the medicinal fungus Ganoderma sinense[J]. Scientific Reports, 2015(5): 11087. DOI:10.1038/srep11087 |
| [28] |
WANG L Z, WANG H Y, PU X D, SUN S J, WEI J H, SUN C. Cloning and characterization of sesquiterpene synthase genes from the Ganoderma sinense genome[J]. Scientia Sinica Vitae, 2018, 48(4): 447-454. DOI:10.1360/N052017-00190 |
| [29] |
褚丽华, 王丽芝, 陈士林, 曾欣宜, 徐江, 李滢, 孙超. 一个紫芝多产物倍半萜合酶的功能鉴定[J]. 中国实验方剂学杂志, 2019, 25(12): 151-157. DOI:10.13422/j.cnki.syfjx.20191216 CHU L H, WANG L Z, CHEN S L, ZENG X Y, XU J, LI Y, SUN C. Functional identification of a multi-product sesquiterpene synthase from Ganoderma sinense[J]. Chinese Journal of Experimental Traditional Medical Formulae, 2019, 25(12): 151-157. DOI:10.13422/j.cnki.syfjx.20191216 |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49




