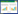文章信息
基金项目
- 深圳市科技计划项目(CJGJZD20210408092401004)
作者简介
- 王宏乐(1984—),女,博士,农艺师,研究方向为植物病理学和数字农业,E-mail: teajam@163.com.
文章历史
- 收稿日期:2022-03-23
2. 深圳市丰农控股有限公司,广东 深圳 518055;
3. 深圳市五谷网络科技有限公司,广东 深圳 518055;
4. 深圳市宇众物联科技有限公司,广东 深圳 518126
2. Shenzhen Fengnong Holding Co., ltd, Shenzhen 518055, China;
3. Shenzhen Wugu Network Technology Co., Ltd, Shenzhen 518055, China;
4. Shenzhen Yuzhong Union Science and Technology Co., Ltd, Shenzhen 518126, China
【研究意义】在植物病害智能识别技术的研究中,传统的机器学习技术已有了不错的识别效果,新出现的深度学习技术进一步推进了植物病害识别技术的发展,并成为该领域研究的热点。关于深度学习技术用于植物病害识别已有诸多报道,目前已报道了约20种作物上的数百种植物病害的智能识别,识别准确率高达90% 以上[1-3]。但是,这类模型多数使用实验室场景下的标准病害图片作为训练集,其建立的病害识别模型应用到田间识别时,识别准确率降至6%~45% 之间[1, 4-5]。作为训练集的实验室场景图片大多背景一致,拍摄角度差别不大,采集方法和数据相对标准一致,这类图片具有清晰的病害特征,有利于模型训练中的病害特征学习;而应用场景的田间场景图片往往情况复杂,存在不同光线、角度和病害特征清晰度不同等问题,采集技术和标准较难统一,病害特征不显著,背景因子干扰较大,因而对田间场景图片的识别率较低[1, 6]。如何优化植物病害识别深度学习模型,从而跨越实验室和田间不同场景的鸿沟,增强模型鲁棒性,以实现深度学习模型在田间实际场景下的稳定且准确识别,成为迫切需要解决的问题。
【前人研究进展】关于模型优化的相关报道有很多,目前普遍认为可以通过两个方面对深度学习训练的病害识别模型进行优化,包括对算法的优化及训练数据的优化。至今,已有诸多关于通过优化算法提高识别率的报道,其中主要包括数据增强、数据均衡、训练模型和参数优化等方法。Fuentes等[7]通过对靶标植物病虫害数据进行标注和特征提取来进行数据增强,提高模型识别率约30%,可同时解决实际应用场景中的9种番茄常见病虫害识别问题,该方法可以降低背景干扰,同时解决田间识别中存在的多标签问题。Perez等[8]建立了一个基于神经网络的数据增强模型,可以提高约7% 的识别率。Montalbo等[9]对大粒咖啡病虫害识别模型进行优化,通过调整深度学习中的参数和超参数来解决过拟合问题。Fuentes等[10]采取前人报道的方法对网络结构进行修改,大大减少了识别结果的假阳性,识别率提升达13%。还有研究者通过运用双边分支网络(Bilateral-Branch Netwo rk, BBN)解决了数据不均衡问题,可提升精度5% 以上[11-12]。在训练数据优化方面也有一些报道:Monhanty等[1]研究发现,使用彩色图片或是去除背景的图片训练集得到的模型较使用灰度图片训练集得到的模型的识别率高3%~6%;Lee等[5]尝试通过优化数据集,合并不同作物相同病害的数据用于模型训练,模型对实验室场景图片的识别率虽略有降低,但对田间场景图片的识别率远高于传统作物-病害模型,并且可以识别未经训练作物的相同病害。总体而言,通过优化数据集实现病害识别模型优化的研究较少,在该方向还可开展更多的探索。
【本研究切入点】农作物病虫害发生存在周期性。从采集困难程度来说,实验室场景图片收集往往较为容易,收集成本低廉,但是病症的多样性反映不足;田间场景图片可以较好反映病害的症状表现和多样性特征,但采集周期性较长,场景复杂,费用高昂。目前普遍反映基于实验室场景图片建立的病害识别模型准确率存在严重不足,无法满足农业生产和数字农业的需求。【拟解决的关键问题】本研究结合已报道的人工智能深度学习技术在植物病虫害识别的应用方法,通过调整模型训练集不同来源数据组成用于模型训练来优化模型,从而减少深度学习模型对田间场景数据的依赖,缩短模型建立初期对田间数据采集周期,降低田间数据采集成本,同时提高深度学习病虫害识别模型在实际运用中的准确率、适用性和稳定性,为人工智能深度学习技术进一步运用于智慧农业的病虫害防治提供理论和实践基础。
1 材料与方法 1.1 数据集本研究所用数据集为包含3组感染病害的叶片和1组用于组内对比的健康叶片,共计5 720张图片,其中5 200张用于训练集,520张为测试集。训练集包含柑橘黄龙病1 200张,苹果黑星病1 200张,芒果细菌性斑点病800张,以及健康芒果叶片2 000张;测试集包含病害图片数量为该类别所对应的训练集图片数量的10%;训练集和测试集中,来源于实验室和田间采集的数量相等。图片部分来源于Plant Village数据集[13],其余为田间和实验室采集,图片情况见图 1。所有数据集的图片压缩为224×224像素大小后用于训练、测试及验证。

|
| 图 1 训练集图片样例 Fig. 1 Image samples of training datasets |
1.2 深度学习模型训练实验设计
不同的神经网络模型在植物病害分类任务中的表现有所不同,选择合适的神经网络模型架构可提高模型的识别率。本研究结合前人研究成果[1-7],选择Split-Attention Networks(RestNeSt-50)[14]、Visual Geometry Group Network-16(VGG-16)[15]和Residual Network-50(ResNet-50)[16] 3种深度学习模型架构用于模型训练。这3种模型均在ImageNet数据集上进行预训练。本研究使用迁移学习(Transfer Learning)的方式进一步对模型进行训练。为避免训练过程中数据分布发生改变,采取全部采样的方法,即全部数据均参与训练,每张图片仅采样1次。
1.2.1 图片总数不变的训练集内2种场景数据分布对模型识别率的影响 在训练集图片总张数不变的情况下,通过调整训练集的实验室场景和田间场景图片的比例,探索训练集内不同来源数据集分布对模型识别率的影响。本实验设计5组图片数量相同而数据分布不同的训练集:F0数据分布为实验室场景图片占比100% 的情况;F30为实验室场景图片占比70%、田间场景图片占比30% 的情况;F50为实验室场景与田间图片占比各50% 的情况;F70为实验室场景图片占比30%、田间场景图片占比70% 的情况;F100的数据分布为田间场景图片占比100% 的情况。
1.2.2 图片总数增加的训练集内2种场景数据的分布对模型识别率的影响 通过在一种场景图片条件下逐渐增加另一种场景的数据集图片,探索数据集分布改变对识别率的影响。本实验分别设计了在训练集的全部实验室场景图片中逐渐添加田间场景图片的情况(LF),以及在训练集的全部田间场景图片中逐渐添加实验室场景图片的情况(FL),每组设计5个添加梯度。在实验室场景图片中添加田间场景图片,添加图片数量的梯度占比分别为0%(LF0)、30%(LF30)、50%(LF50)、70%(LF70)和100%(LF100);在田间场景图片中添加实验室场景图片,添加图片数量的梯度分别为0%(FL0)、30%(FL30)、50%(FL50)、70%(FL70)和100%(FL100)。
本研究使用3种不同的深度学习网络结构进行模型训练,根据数据添加情况不同,将通过在实验室场景图片中梯度添加田间场景图片的数据集训练得到的模型归类为ResNeSt50-LF、VGG16-LF和ResNet50-LF;以及将通过在田间场景图片中梯度添加实验室场景图片的数据集训练得到的模型归类为ResNeSt50-FL、VGG16-FL和ResNet50-FL。
1.2.3 训练参数 本研究使用PyTorch深度学习框架,计算平台为一台台式计算机,操作系统为Ubuntu 18.04 LTS 64位系统,配有单块型号为NVIDIA V100s的图形处理器(GPU),搭载Intel Core i5-7500 CPU,内存为32 GB。训练过程中所有层的参数均设为可学习,批处理设为32,输入图像统一为224×224分辨率,总训练周期数为150,同时使用衰减权重为0.0005,动量为0.9的随机梯度下降(Stochastic Gradient Descent)作为优化器。初始的学习率设置为0.01,学习率调整策略使用带热重启的随机梯度下降(Stochastic Gradient Descent with Warm Restart)[17-19],退火方式使用余弦退火[20]。为使结果便于相互比较,所有模型训练使用的超参数(hyper-parameters)相同。
1.3 模型评价为了评价模型的优劣,以及评价模型是否存在过拟合,我们将实际训练集按80-20(训练集的80% 用于训练,剩余20% 用于验证)分为训练集-验证集,验证集的数据分布情况与训练集一致。本研究另外准备520张测试集,由相同数量的实验室场景图片及田间场景图片组成。本研究对每组实验的平均F1分数、平均损失值(Loss)、准确率以及总精度进行计算[1]。
2 结果与分析 2.1 图片总数不变的训练集内2种场景数据的分布对模型识别率的影响分别使用RestNeSt-50、VGG-16和ResNet-50等3种网络结构对5组图片总张数相同而场景分布不同的训练集数据进行训练,并使用与训练数据分布一致的验证集数据进行评价。结果表明,在3种网络结构中,使用100% 由实验室场景图片组成的训练集F0训练得到的模型的平均F1分数皆最高,平均损失值皆最小,在RestNeSt-50、VGG16和ResNet-50上平均F1分数分别为0.9988、0.9981和0.9979,平均损失值分别为0.0011、0.0048和0.0022(图 2)。
|
| 图 2 不同植物病害识别模型在验证集上的平均F1分数与损失值 Fig. 2 Mean F1 score and loss of different plant diseases recognition models on validation sets |
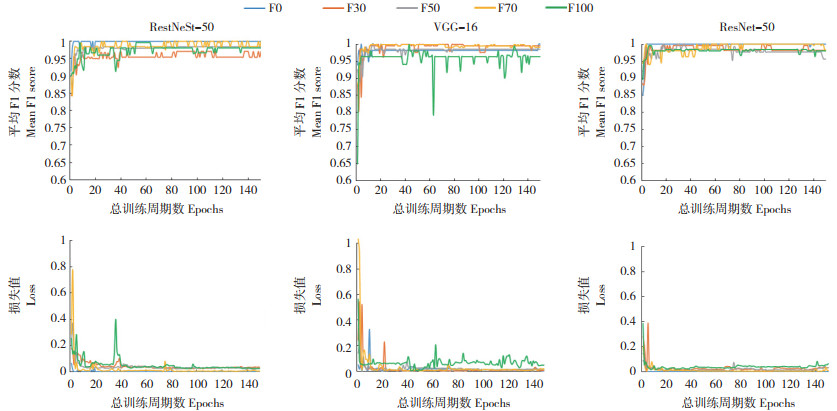
使用实验室场景与田间场景图片比例为1∶ 1的测试集对训练得到的模型进行评价,结果显示,准确率最低的为仅使用实验室场景图片训练集(F0)训练的模型,在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为73.00%、78.67% 和73.33%;3组模型在实验室场景图片的测试集上表现较好,准确率均在90% 以上,但对田间场景的测试集的识别准确率约为50%。随着田间场景图片的比例增加,模型对测试集数据的识别准确率提升缓慢。除训练集F0训练的模型外,其他各组的总精度都在90% 以上。使用实验室场景图片和田间场景图片比例为7 ∶ 3的训练集(F30)训练的模型,在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为96.67%、97.33% 和93.33%,相对于F0有大幅度提升。使用实验室场景图片和田间场景图片比例相等的训练集(F50)训练的模型,在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为96.00%、98.67% 和97.67%。使用实验室场景图片和田间场景图片比例为3 ∶ 7的训练集(F70)训练的模型,在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为98.00%、98.33% 和98.00%。使用仅含有田间场景图片的训练集(F100)训练的模型,在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为95.67%、97.67% 和95.67%。综上所述,使用仅含有实验室场景的图片作为训练集时,模型对田间场景图片识别准确率很低,且总精度最低;仅使用田间场景来源的图片作为训练集时,模型对实验室场景图片识别准确率可达90% 以上;训练集内田间场景的图片比例增加可以提高模型的准确率,训练集含30% 的田间场景图片比例即可大幅度提升模型在测试集上的准确度(图 3)。
|
| Field:模型识别测试集中田间场景图片的准确率;Lab:模型识别测试集中实验室场景图片的准确率;Total:模型识别测试集中所有图片的准确率 Field: Accuracy of field-condition images on test sets recognized by models; Lab: Accuracy of lab-condition images on test sets recognized by models; Total: Accuracy of all images on test sets recognized by models 图 3 不同植物病害识别模型在测试集上的识别准确率 Fig. 3 Accuracies of different plant diseases recognition models on test sets |
2.2 图片总张数增加的训练集内2种场景数据的分布对模型识别率的影响
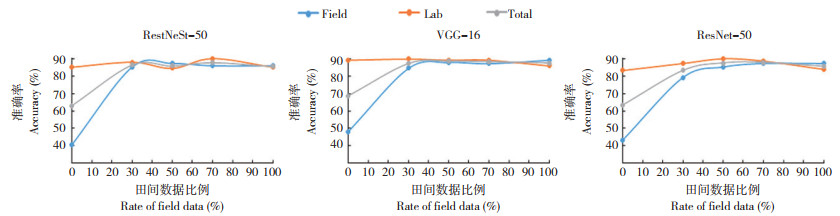
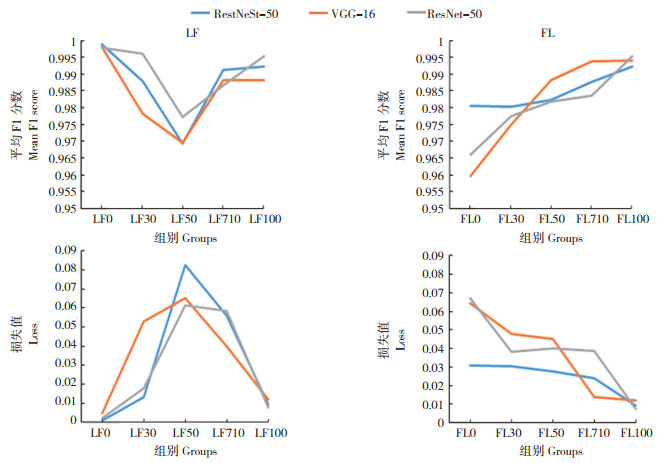
在实验来源数据中按比例添加田间场景数据,F1分数先下降再上升,损失值先上升后下降。当添加田间场景图片为实验室场景图片数量的0% 时(LF0),在RestNeSt-50、VGG-16和ResNet-50上平均F1分数分别为0.9988、0.9981和0.9979, 损失值分别为0.0011、0.0048和0.0022。当添加田间场景图片数量为实验室场景图片数量的50% 时(LF50),RestNeSt-50、VGG-16和ResNet-50的平均F1分数达到最低值,分别为0.9693、0.9695和0.9773;损失值达到最高,分别为0.0826、0.0652和0.0612。而后随着训练集内田间场景图片数量的增加,F1分数开始上升,损失值开始下降。当添加田间场景图片数量为实验室场景图片数量的100% 时(LF100),在RestNeSt-50、VGG-16和ResNet-50上平均F1分数分别为0.9922、0.9881和0.9953, 损失值分别为0.0094、0.0119和0.0075(图 4)。
|
| 图 4 训练集数据集改变对植物病害识别模型在验证集上的平均F1分数与损失值的影响 Fig. 4 Mean F1 score and loss of plant diseases recognition models of the training with different datasets on validation sets |
在田间来源数据中添加实验室场景数据,F1分数随图片数量增加上升,损失值也随之下降。当添加实验室场景图片数量为田间场景图片数量的0% 时(FL0),在RestNeSt-50、VGG-16和ResNet-50上平均F1分数分别为0.9806、0.9597和0.9659, 损失值分别为0.0309、0.0643和0.0673。随着添加的实验室场景图片数量增加,3组模型的平均F1分数均随图片数量增加上升,损失值也均随之下降。当添加实验室场景图片数量为田间场景图片数量的100% 时(FL100),在RestNeSt-50、VGG-16和ResNet-50上平均F1分数分别为0.9922、0.9881和0.9953, 平均损失值分别为0.0094、0.0119和0.0075(图 4)。
使用实验室场景与田间场景来源图片比例为1 ∶ 1的测试集对上述模型进行评价。LF0在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为73.00%、78.67% 和73.33%;LF30在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为97.33%、96.00% 和97.33%,相比LF0有大幅度提升。FL0在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为95.67%、97.67% 和95.67%;FL30在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为97.33%、97.00% 和97.00%;FL100在RestNeSt-50、VGG-16和ResNet-50上的总精度分别为99.33%、98.00% 和99.33%,相比FL0有小幅度提升。总体而言,模型的识别总精度随图片数量增加而增加;当在100% 实验室场景图片训练集内增加30% 的田间场景图片,模型总精度明显提高;当训练集增加的另一种场景图片数量为原先场景图片数量的70%~100% 时,总精度趋于稳定(图 5)。

|
| 添加比例为添加的另一种场景图片的数量和原 100% 的实验室或田间场景数据集的图片数量的百分比值 Rate of addition is percentage between the number of images of another condition and the number of images of a 100% dataset in laboratory or field condition 图 5 训练集数据集改变的不同网络结构在测试集上的识别准确率 Fig. 5 Recognition accuracies of different network structures change by the training datasets on test sets |
3 讨论
诸多研究表明,建立高精度的深度学习病害识别模型,需要大量的作物病害及健康数据,其中健康作物的数据有利于增强深度学习模型对病害特征的学习[1, 5],如已报道的Plant Village数据集[13]包含38种作物感病-健康配对数据。但健康作物的数据并非必要存在,以Plant Village数据集为例,其中的柑桔黄龙病以及南瓜白粉病并无相应健康作物数据与之配对。另有报道称将不同作物的相同病害或健康数据进行合并,能够提高深度学习模型对田间病害识别的准确率[5]。本实验设计时,曾考虑病害识别模型对不同作物不同病害的特征识别(柑桔、苹果、芒果),以及同一作物健康与感病情况的区分(健康芒果与芒果细菌性斑点病)。本实验结果表明,通过调整数据分布的方法,模型对不同的作物病害以及同一作物的不同类别的识别均有提升。目前本方法所应用的数据集偏小,有待进一步放大数据集,同时扩大应用和优化范围。
实验室场景采集到的图片往往具有一定的采集标准,其图片的病害特征清晰,且大多背景一致,拍摄角度差别不大,而田间场景图片往往情况复杂,采集标准较难统一,采集时存在不同光照强度及角度,背景复杂多变,因而模型对田间场景图片识别的准确率往往较低。诸多报道表明,仅使用以实验室场景图片训练的模型,对实验室场景图片识别的准确率多在90% 以上[1, 3-7, 9-10],而对田间场景图片识别的准确率大大低于实验室场景图片[1, 4-5],本研究也得到相同结果。Lee等[5]研究表明,使用以田间场景图片训练的模型用于实验室场景图片识别,其准确率约为60%,本研究获得的准确率略高于该报道。基于此,本研究认为,训练集场景过于单一不利于建立农业生产复杂环境下的识别模型。调整训练集内不同场景图片数据的比例,是提升病害识别模型准确率和鲁棒性的一个行之有效的方案。
因而,在基于深度学习的植物病害识别模型中,训练集的图片类型对模型的识别准确率十分重要。Fuentes等报道了可识别29种植物病害模型训练及准确度的测试结果,通过比较训练集内不同病害种类不同场景图片的分布,认为训练集为混合场景图片数据的,病害类别识别准确率整体优于单一场景照片训练集数据[10]。本研究也得到相似的结果,进一步证实了训练集使用混合场景图片数据更有利于提升病害识别模型在田间场景下的识别准确率。
在植物病害识别模型研究中,ResNet-50和VGG-16是最为常见的深度学习网络结构[1-7]。ResNet-50解决了梯度弥散问题,比VGG-16具有更高的预测精度[21-25]。ResNeSt-50为2020年报道的拆分注意力深度学习网络结构,具有不错的预测精度[16]。近年来,该网络结构逐渐运用于植物病虫害识别领域,已报道的有木薯病害识别、无人机采集的玉米草地贪夜蛾危害状识别及水稻病害识别等,预测精度均优于其他网络[25-27]。在本研究结果中,3种网络结构的总精度差别不大,变化趋势基本一致,VGG-16的预测总精度稍低于ResNet-50和ResNeSt-50,ResNeSt-50的预测总精度略优于ResNet-50。本研究侧重于研究不同场景的数据分布对深度学习模型准确率的影响,获得了较好的效果,为建立更加科学、高效的病害识别模型提供了重要参考和借鉴。今后还将结合实际生产应用需要,增加数据量和场景差异性,进一步对病害识别模型进行改进、优化与验证。
4 结论本实验通过调整实验室和田间场景的病害图片的配比,使用3种不同的网络结构分别进行训练,以了解数据结构变化对病虫识别模型准确率的影响,结果表明:(1)在数据集数量不增加的情况下,通过调整不同场景数据比例,对训练得到的模型的准确率存在影响,当田间场景图片比例达到30% 时即可大幅度提升模型的准确率。(2)训练图片数量的增加可以提高模型的识别准确率。在实验室场景图片中增加30% 的田间场景图片,即可大幅度提升模型准确率。在田间场景图片中增加实验室场景图片,对模型的准确率有一定提升,但提升幅度不大。(3)通过在训练集为实验室场景图片中增加田间场景图片的方法,得到的病害识别模型对实验室场景和田间场景图片均具有相当的识别准确率。
综上所述,该方法适用于农业复杂环境下的高准确度植物病害识别模型的快速建立,可以减少深度学习模型对田间场景数据的依赖,缩短田间数据采集周期,降低田间数据采集成本,同时提高深度学习植物病害识别模型在实际运用中的准确率、适用性和稳定性,为人工智能深度学习技术进一步运用于智慧农业的病虫害防治提供理论和实践基础。
| [1] |
MOHANTY S P, HUGHES D P, SALATH M. Using deep learning for Image-based plant disease detection[J]. Frontiers in Plant Science, 2016, 7: 1419-1429. DOI:10.1515/9783110691276-017 |
| [2] |
KAMILARIS A, PRENAFETA-BOLDú F X. Deep learning in agriculture: A survey[J]. Computers and Electronics in Agriculture, 2018, 147: 70-90. DOI:10.1016/j.compag.2018.02.016 |
| [3] |
王聃, 柴秀娟. 机器学习在植物病害识别研究中的应用[J]. 中国农机化学报, 2019, 40(9): 171-180. DOI:10.13733/j.jcam.issn.2095-5553.2019.09.30 WANG D, CHAI X J. Application of machine learning in plant diseases recognition[J]. Journal of Chinese Agricultural Mechanization, 2019, 40(9): 171-180. DOI:10.13733/j.jcam.issn.2095-5553.2019.09.30 |
| [4] |
FERENTINOS K P. Deep learning models for plant disease detection and diagnosis[J]. Computers and Electronics in Agriculture, 2018, 145: 311-318. DOI:10.1016/j.compag.2018.01.009 |
| [5] |
LEE S H, GOËAU H, BONNET P, JOLY A. New perspectives on plant disease characterization based on deep learning[J]. Computers and Electronics in Agriculture, 2020, 170. DOI:10.1016/j.compag.2020.105220 |
| [6] |
T ODA Y, OKU R A F. H ow c onvoluti on a l n eur a l n etworks diagnose plant disease[J]. Plant Phenomics, 2019, 1(1). DOI:10.1155/2019/9237136 |
| [7] |
FUENTES A, YOON S, KIM S C, PARK D S. A robust deep-learningbased detector for real-time tomato plant diseases and pests recognition[J]. Sensors, 2017, 17: 2022. DOI:10.3390/s17092022 |
| [8] |
PEREZ L, WANG J. The effectiveness of data augmentation in image classification using deep learning[J]. arXiv, 2017. DOI:10.48550/arXiv.1712.04621 |
| [9] |
MONTALBO F, HERNANDEZ A A. An optimized classification model for Coffea liberica disease using deep convolutional neural networks//IEEE[C]. International Colloquium on Signal Processing&Its Applications (CSPA), 2020: 213-218. DOI: 10.1109/CSPA48992.2020.9068683.
|
| [10] |
FUENTES A, YOON S, LEE J, PARK D S. High-performance deep learning neural network-based tomato plant diseases and pests diagnosis system with refinement filter bank[J]. Frontiers in Plant Science, 2018, 9: 1162. DOI:10.3389/fpls.2018.01162 |
| [11] |
ZHOU B Y, CUI Q, WEI X S, ZHAO M C. BBN: bilateral-branch network with cumulative learning for long-tailed visual recognition[C]. Conference on Computer Vision and Pattern Recognition (CVPR), 2020: 9716-9725. DOI: 10.1109/CVPR42600.2020.00974.
|
| [12] |
LIANG Y, QIAN T. Recommending accurate and diverse items using bilateral branch network[J]. arXiv, 2021. DOI:10.48550/arXiv.2101.00781 |
| [13] |
Hughes D P, Salathé M. An open access repository of images on plant health to enable the development of mobile disease diagnostics[J]. arXiv, 2015. DOI:10.48550/arXiv.1511.08060 |
| [14] |
ZHANG H, WU C, ZHANG Z, ZHU Y, ZHANG Z, LIN H B, SUN Y, HE T, MUELLER J, MANMATHA R, LI M, SMOLA A. ResNeSt: Split-Attention Networks[J]. arXiv, 2020. DOI:10.48550/arXiv.2004.08955 |
| [15] |
SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[J]. Computer Science, 2014, 14. |
| [16] |
HE K M, ZHANG X Y, REN S Q, SUN J. Deep Residual Learning for Image Recognition//IEEE[C]. Conference on Computer Vision and Pattern Recognition (CVPR), 2016: 770-778. DOI: 10.1109/CVPR.2016.90.
|
| [17] |
LOSHCHILOV I, HUTTER F. SGDR: stochastic gradient descent with warm restarts//ICLR. International Conference on Learning Representations[C]. 2017. DOI: 10.48550/arXiv.1608.03983
|
| [18] |
GOTMARE A, KESKAR N S, XIONG C M, SOCHER R. A closer look at deep learning heuristics: learning rate restarts, warmup and distillation[J]. arXiv, 2018. DOI:10.48550/arXiv.1810.13243 |
| [19] |
魏靖, 王玉亭, 袁会珠, 张梦蕾, 王振营. 基于深度学习与特征可视化方法的草地贪夜蛾及其近缘种成虫识别[J]. 智慧农业, 2020, 2(3): 75-85. DOI:10.12133/j.smartag.2020.2.3.202008-SA001 WEI J, WANG Y T, YUAN H Z, ZHANG M L, WANG Z Y. Identification and morphological analysis of adult Spodoptera frugiperda and its close related species using deep learning[J]. Smart Agriculture, 2020, 2(3): 75-85. DOI:10.12133/j.smartag.2020.2.3.202008-SA001 |
| [20] |
SMITH L N. Cyclical learning rates for training neural networks//IEEE[C]. Winter Conference on Applications of Computer Vision (WACV), 2017: 464-472. DOI: 10.1109/WACV.2017.58.
|
| [21] |
ZHANG K.WU Q, LIU A, MENG X. Can deep learning identify tomato leaf disease[J]. Advances in Multimedia, 2018, 6710865. DOI:10.1155/2018/6710865 |
| [22] |
RAMCHARAN A, BARANOWSKI K, MCCLOSKEY P, AHMED B, LEGG J, HUGHES D P. Deep learning for image-based cassava disease detection[J]. Frontiers in Plant Science, 2017, 8: 1852. DOI:10.3389/fpls.2017.01852 |
| [23] |
FUENTES A, YOON S, KIM S, PARK D. A robust deep-learningbased detector for real-time tomato plant diseases and pests recognition[J]. Sensors, 2017, 17(9): 2022. DOI:10.3390/s17092022 |
| [24] |
SALEEM M H, POTGIETER J, ARIF K M. Plant disease detection and classification by deep learning[J]. Plants, 2019, 8(11): 468. DOI:10.3390/plants8110468 |
| [25] |
FENG J, SUN Y, ZHANG K, ZHAO Y, REN Y, CHEN Y, ZHUANG H, CHEN S. Autonomous detection of Spodoptera frugiperda by feeding symptoms directly from UAV RGB imagery[J]. Applied Sciences, 2022, 12(5): 2592. DOI:10.3390/app12052592 |
| [26] |
DENG R, TAO M, XING H, YANG X, LIU C, LIAO K, QI L. Automatic diagnosis of rice diseases using deep learning[J]. Frontiers in Plant Science, 2021, 1691. DOI:10.3389/fpls.2021.701038 |
| [27] |
CHEN Y, XU K, ZHOU P, BAN X, HE D. Improved cross entropy loss for noisy labels in vision leaf disease classification[J]. IET Image Processing, 2022, 16(6): 1511-1519. DOI:10.1049/ipr2.12402 |
(责任编辑 杨贤智)
 2022, Vol. 49
2022, Vol. 49