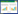文章信息
基金项目
- 国家重点研发计划项目子课题(2021YFA0805901-3);国家自然科学基金(82070199);广东省普通高校重点领域专项(2021ZDZX2050);广东省重点领域研发计划重大科技专项(2018B020203003)
作者简介
- 盘家圣(1995—),男,在读硕士生,研究方向为动物分子遗传与育种,E-mail:243699325@qq.com.
通讯作者
- 唐冬生(1962—),男,博士,教授,研究方向为动物分子遗传与育种,E-mail:tangdsh@163.com.
文章历史
- 收稿日期:2022-03-17
2. 佛山科学技术学院医学院/广东省基因编辑工程技术研究中心,广东 佛山 528225
2. School of Medicine, Foshan University/Guangdong Research Center of Gene Editing Engineering Technology, Foshan 528225, China
生猪养殖是事关我国国民经济健康发展和粮食安全的重要产业。同时,猪由于其机体结构、解剖生理特征等方面与人类相似,因此具有重要医学研究价值。对猪进行基因修饰,能够快速改良其经济性状,同时还可以构建用于生物医学研究的动物模型。传统基因修饰技术(如转基因和基因打靶等)虽然已在基因修饰猪研究上应用20余年,但实践表明,传统猪基因修饰技术效率低、精度差、周期长、成本高。2020年Charpentier和Doudna因成功开发CRISPR/Cas9基因编辑技术荣获诺贝尔化学奖[1]。CRISPR/Cas9基因编辑技术作为一种革命性的基因修饰技术,推动了包括猪在内的动植物基因修饰进程。CRISPR/Cas9基因编辑技术通过在靶点处产生DNA双链断裂(Double-strand break, DSB),激活胞内同源重修复(Homology directed repair, HDR)或非同源末端连接(Non-homologous end joning, NHEJ)修复,实现对DNA的定点插入/ 删除或替换[2]。由于具有简便、高效和经济优势,CRISPR/Cas9基因编辑技术已广泛应用于包括猪在内的众多物种上[3-8]。但近年研究表明,CRISPR/Cas9引发的DNA双链断裂可能导致基因编辑后的细胞出现不可预期的情况,如经NHEJ所致的插入/ 缺失(Indels)不可控,还能引发脱靶效应、基因组大范围删除或重排,以及诱导抑癌基因p53介导DNA损伤等[9-11]。为了避免DSB引发副作用,科学家开发出不依赖DSB、仅介导单个碱基突变的基因编辑工具(Base editors, BEs),如腺嘌呤碱基编辑器(Adenine base editors,ABE)和胞嘧啶碱基编辑器(Cytidine base editors,CBE)等[12-13]。研究表明,由于BEs不介导DSB,能有效避免CRISPR/Cas9引发的副作用,使单个碱基定向转换,且具有更高基因编辑精准性,因而在农业和医学领域得到广泛应用[14-22]。
本文对碱基编辑技术发展进程和碱基编辑技术介导的人类疾病模型与基因治疗、异种器官移植、猪遗传育种等相关研究的最新进展进行综述,以期为今后进一步利用碱基编辑技术开展疾病模型构建和猪遗传育种提供理论指导。
1 碱基编辑系统原理与研究进展碱基编辑技术主要将灭活的或有单链切割活性的Cas蛋白与碱基脱氨酶融合,在sgRNA(guide RNA)的引导下,在不引入DSB、无需供体DNA的情况下,实现DNA序列中碱基的定向编辑,提高基因编辑的效率、准确性和特异性,是加速基因遗传病治疗和动植物遗传改良的新工具[13]。目前,成熟应用的碱基编辑器主要有4种:CBE,可实现C → T(G → A)突变;ABE,可实现A → G(T → C)突变;CBE/ABE双碱基编辑器,可同时实现C → T(G → A)与A → G(T → C)突变;胞嘧啶到鸟嘌呤基础编辑器(C-to-G base editor, CGBE),可实现C → G突变[12-13, 23-24]。
1.1 胞嘧啶碱基编辑器(CBE)在胞嘧啶脱氨酶的催化下C脱氨生成具有T碱基配对性质的尿嘧啶U,在碱基修复(Mismatch repair, MMR)过程中形成A-T碱基对。Komor等[13]将鼠源的胞嘧啶脱氨酶APOBEC1融合到无双链切割功能的Cas9蛋白(dead Cas9, dCas9)的C端,构建了第一代CBE,即BE1;为了进一步提高碱基编辑效率,Komor等将尿嘧啶DNA糖苷酶抑制因子UGI融合到BE1的C端,并恢复了Cas9 HNH结构域的His残基,产生第三代碱基编辑器rAPOBEC1-XTEN-nCas9-UGI,即BE3(图 1A)。尽管BE3在编辑效率方面得到不同程度的改善,但其仍存在潜在脱靶效应、产物纯度不佳的问题,限制了CBE在生物工程中的应用。针对这些缺陷,研究者们对CBE进行不断改进。
|
| A:以BE3[13]为例,sgRNA识别DNA特异性位点,rAPOBEC1结合非互补链,nCas9(d10A)切割互补链,BE3诱导C-G碱基对变成T-A碱基对B:以ABE7.10[12]为例,sgRNA识别DNA特异性位点,Tad+Tad*结合非互补链,nCas9(d10A)切割互补链,ABE7.10诱导A-T碱基对变成G-C碱基对 A: Taking BE3[13] as an example, sgRNA recognizes DNA-specific sites, rAPOBEC1 binds to non-complementary strands, and nCas9 (d10A) cleaves the complementary strands, thus BE3 induces C-G base pairs to become T-A base pairs B: Taking ABE7.10[12] as an example, sgRNA recognizes DNA-specific sites, Tad+Tad* binds to non-complementary strands, and nCas9 (d10A) cleaves complementary strands, thus ABE7.10 induces A-T base pairs to become G-C base pairs 图 1 CBE和ABE的作用机制[12-13] Fig. 1 Mechanism of CBE and ABE[12-13] |
BE3被广泛用于生物研究中[21, 25-37]。但在部分位点上,BE3可能会产生副产物,其中目标C-G碱基对被转换为G-C或A-T碱基对,不符合预期[33-37]。为了进一步抑制尿嘧啶DNA糖基化酶(Uracil DNA Glycosylase, UNG)活性,Komor等[38]在BE3的CAS9序列C端附加一个额外的UGI拷贝,经优化后形成BE4。与BE3相比,BE4在工程细胞中对C → T编辑的准确率大幅增加。为了进一步提高编辑效率,Koblan等[39]在BE4主要蛋白rAPOBEC1-Cas9的N端和C端分别添加核定位信号(Nuclear localization signal, NLS),再将绿色荧光蛋白基因GFP和2A肽基因P2A序列连接在rAPOBEC1-Cas9的C端,接着优化密码子,最后得到BE4max;同时,该团队在先天性1f型糖基化障碍疾病治疗研究中发现,BE4max对致病性SNP的校正效率为BE4的2倍。
CBE编辑工具在小鼠胚胎中应用存在大量非靶点脱靶现象[40],在植物的应用上也发现类似状况。Jin等[41]用BE3、HF1-BE3和ABE分别处理水稻,其全基因组测序分析结果显示,BE3和HF1-BE3诱发大量全基因组脱靶情况,这些突变大多是C → T型单核苷酸变种(Single nucleotide variation, SNVs)。随着蛋白工程技术的发展,科学家们进一步对CBE进行优化以降低其脱靶效应。改良的碱基编辑工具YE1-BE4、YE2-BE4、YEE-BE4、R33A+K34A-BE4、YE1-BE4-CP1028、YE1-BE4-NG和AALN-BE4在保持原有靶向碱基编辑活性的同时,能够最大限度降低DNA脱靶率[42]。
1.2 腺嘌呤碱基编辑器(ABE)在CBE的启发下,ABE应运而生。ABE的原理是腺苷脱氨化会产生肌苷,肌苷在复制和转录过程中被识别为G,从而实现A → G转换。但自然存在的腺嘌呤脱氨酶不能结合DNA,学者试图研发一种以DNA为底物的腺嘌呤脱氨酶。Gaudelli等[12]采取一种细菌选择策略,遴选出1种大肠杆菌源tRNA腺苷酸脱氨酶(ecTadA)变体,经优化,ABE7.10能有效将目标A-T碱基对转换为G-C碱基对(在人体细胞中约50%),具有高产物纯度(≥ 99.9%)和低Indels(≤ 0.1%)的特点,且极少发生DNA脱靶现象(图 1B)。
为进一步优化ABE7.10,Koblan等[39]用2个bpNLS替代ABE7.10中的SV40 NLS,优化其密码子,得到ABEmax;为了对比ABEmax与ABE7.10,在HEK293T细胞中设计2对sgRNA分别对HBG1和HBG2启动子(γ-globin)区域激活突变,结果显示,对于第1对sgRNA,ABEmax编辑活性均约为ABE7.10的2倍;对于第2对sgRNA,ABEmax编辑效率分别提高5.2倍和7.1倍。
ABE显示出极少的脱靶效应,但ABE对细胞RNA的脱靶效应尚未得到深入研究。Rees等[43]证明了ABEmax在细胞RNA中产生了编辑效率低但可检测到的腺苷到肌苷编辑;为了筛选最小化RNA编辑活性,Rees等比较了一系列ABEmax突变体,发现在TadA中引入E59A或E59Q突变、在TadA* 中引入V106W突变,可保持DNA编辑活性的同时使得RNA编辑频率显著极低。ABEmaxAW或ABEmaxQW保持了有效的靶DNA编辑,大大减少了RNA编辑,提高了DNA特异性,并减少了Indels的形成。
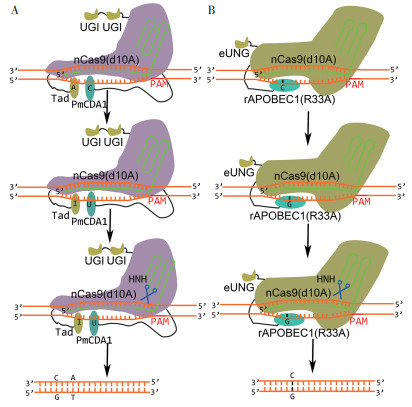
1.3 CBE/ABE双碱基编辑器CBE和ABE虽然是精准的基因编辑工具,但只能催化单一类型的核苷酸转换。最近,有两项研究介绍了一种能同时编辑C和A两种碱基的双碱基编辑器[23, 44],这种双碱基编辑系统由nCas9与胞嘧啶脱氨酶和腺嘌呤脱氨酶融合而成,能同时诱导C → T、A → G突变。双碱基编辑器的开发进一步拓宽了碱基编辑器的功能(图 2A)。
|
| A:以SPACE[44]为例,sgRNA识别DNA特异性位点,Tad+PmCDA1结合非互补链,nCas9(d10A)切割互补链,SPACE诱导C-G碱基对变成T-A碱基对以及A-T碱基对变成G-C碱基对B:以CGBE[24]为例,sgRNA识别DNA特异性位点,rAPOBEC1 (R33A) 结合非互补链,nCas9(d10A)切割互补链,eUNG取代UGI发挥作用,CGBE诱导C-G碱基对变成G-C碱基对 A: Taking SPACE[44] as an example, sgRNA recognizes DNA-specific sites, Tad + PmCDA1 binds to non-complementary strands, and nCas9 (d10A) cleaves complementary strands, thus SPACE induces C-G base pairs to T-A base pairs and A-T base pairs to become G-C base pairs B: Taking CGBE[24] as an example, sgRNA recognizes DNA-specific sites, rAPOBEC1(R33A) binds non-complementary strands, nCas9 (d10A) cuts complementary strands, and eUNG replaces UGI. So CGBE induces C-G base pairs to become G-C base pairs 图 2 ACBE和CGBE的作用机制[24, 44] Fig. 2 Mechanism of ACBE and CGBE |
Zhang等[23]在ABE7.10的N端添加胞嘧啶脱氨酶hAID得到融合体ABE7.10-N-AID,然后优化其密码子并增加二部核定位信号(bpNLS),添加2个拷贝的UGI得到了A&C-BEmax。在HEK293T细胞中试验28个内源性靶点,A&CBEmax与ABEmax相比,A→G的编辑效率相似或略有降低,而与AID-BE4max相比,C→T的编辑效率则有所提高。此外,与同时采用ABEmax和AID-BE4max相比,A&C-BEmax同时引导的A/C突变率更高。Grunewald等[44]将腺嘌呤碱基编辑器miniABEmax-V82G中的腺嘌呤脱氨酶和胞嘧啶碱基编辑器Target-AID中的胞嘧啶脱氨酶PmCDA1分别融合到nCas9的N端和C端,并融入2个拷贝UGI,开发出一种双碱基编辑器SPACE。SPACE的C→T编辑效率与CBE相当,对A → G编辑效率则略有降低,总体来看,SPACE双碱基编辑器效率高于CBE+ABE。双碱基编辑器的开发解决了同时采用2个单碱基编辑器的低效编辑问题,能实现更加复杂的碱基编辑,在动植物遗传改良、遗传病模型以及校正疾病相关等位基因方面具有潜在应用价值。
1.4 胞嘧啶到鸟嘌呤基础编辑器(CGBE)CBE和ABE可分别实现C → T(G → A)、A → G(T → C)的转换,但在农业和医学上的应用受限。Kurt等[24]在研究碱基编辑器时发现C → G突变,将BE4max(R33A)删除2个UGI,增加1个大肠杆菌尿嘧啶DNA N-糖基化酶eUNG,形成了CGBE(图 2B),实现C → G(互补链为G → C)的转换;通过在人HEK293T细胞中选取18个靶点测试,结果发现CGBE具有高效的C → G编辑效率(41.7%~71.5%)。为了指导CGBE应用,科学家们还基于机器学习开发了编辑效率预测系统,Yuan等[45]开发的深度学习模型能够准确预测目标位点的CGBE编辑结果,为推广CGBE提供了助力。虽然CGBE在工程细胞中的部分靶点产生了较理想的C → G编辑效率,突破了碱基颠换编辑的可能,但仍需继续探索优化,并在特定细胞、特定基因靶点中进一步验证,以拓展其应用范围。
2 碱基编辑技术在基因修饰猪模型构建中的应用许多人类遗传病都与SNP相关,而碱基编辑技术为建立单个碱基突变的遗传病动物模型、异种器官移植以及治疗人类遗传病提供了极大便利。而猪作为遗传病模型和异种器官移植供体有优势。一方面,猪可以作为鼠和非人灵长类动物疾病模型研究的过渡模型。相比小鼠,猪作为动物模型的优势在于遗传、身体构造和生理特性与人类相似;相比猴子,猪全年可发情繁殖,而且性成熟时间早,妊娠周期短,产仔数多,取材容易且饲养难度较低,碱基编辑技术的面世推动了遗传疾病模型、异种器官移植、基因疗法的研究进程。
2.1 异种器官移植供体猪近年来,人类生活质量和医疗系统的改善大大提高了人类的预期寿命,但仍存在慢性病和终末期器官衰竭,而器官供体资源紧缺,异种移植被认为是一种很有前途的解决方案。但由于抗体介导的排斥反应(Antibody-mediated rejection, AMR)和猪内源性逆转录病毒(Porcine endogenous retrovirus, PERV)传播的潜在风险,导致猪器官的临床应用受阻。
pol基因有多个基因拷贝,是猪与人异种器官移植安全性的关键基因之一[46]。敲除pol基因可避免病毒的跨物种感染,可解决异种移植的生物安全问题。Xie等[21]将BE3和pol-sgRNA共转染巴马猪胎儿成纤维细胞(Porcine fetal fibroblasts, PFFs),在pol多基因拷贝中提前引入终止密码子,结果表明,BE3最高可减少84.9% 的PERV拷贝数,为进一步提高对PERV灭活效率以及生产pol基因精准修饰猪提供可行性方案。
目前,与AMR相关的主要异种抗原包括α-1, 3-半乳糖(α-Gal)、猪异种抗原SD(A)和N-羟基神经氨酸(Neu5Gc),分别由α-1, 3半乳糖基转移酶(GGTA1基因编码)、β-1, 4-N-乙酰半乳糖胺转移酶2(β4GalNT2基因编码)和CMPNeu5Ac羟化酶(CMAH基因编码)催化合成[47-48]。Yuan等[49]将BE4-GAM与GGTA1-sgRNA、B4galNT2-sgRNA和CMAH-sgRNA等3个sgRNA通过电穿孔法共同转染巴马猪PFFs,获得4个(4/52)三基因编辑单克隆细胞,结合体细胞核移植(Somatic cell nuclear transfer, SCNT)技术,得到1只三基因精准修饰的仔猪;免疫荧光试验(Immuno fluorescence assay, IFA)结果显示,与WT巴马猪细胞相比,三基因编辑猪细胞的3种相关酶活性显著降低。这进一步克服了超急性免疫排斥反应,使异种器官移植研究得到极大进展。碱基编辑工具将为猪异种器官移植提供精准修饰,避免可能存在的异种移植风险,如染色体断裂或不必要的功能蛋白失调等。
2.2 白化病模型猪白化病是机体无法合成黑色素所造成的疾病,分析与白化病相关的色素突变有助于更好地理解黑色素形成的机制。研究发现,酪氨酸酶是黑色素生物合成途径中的关键酶[50]。人TYR基因的遗传缺陷可导致眼皮肤白化病1型(OCA1),这是一种常染色体隐性遗传病,其特征是头发、皮肤和眼睛中的黑色素减少[51]。Li等[28]用BE3构建TYR基因编辑PFFs,通过SCNT技术生产TYR基因精准修饰克隆猪,得到的4头白化病模型猪都表现出典型的白化病表型,酪氨酸酶在模型猪的心脏、肝脏、肺或肾脏中不表达。该疾病模型猪精准模拟了人类白化病疾病的表征,为白化病治疗提供了生物材料。
2.3 瓦登伯革氏综合征(Waardenburg syndrome, WS)模型猪WS是一种显性的遗传性听觉-色素综合征,以听力损失和色素沉着减退为特征,患病率约为1/42 000,在我国也发现散发病例[52-53]。Hai等[54]利用N-乙基-N-亚硝基脲诱变技术建立了WS2A小型猪模型。其中单等位基因突变(MITF c.740T>C突变)与WS2A的病理相似,纯合突变表现出脉络膜和视网膜退化。此模型为更好地理解人类听力损失的病因和开发新的治疗策略提供新线索。Yao等[55]以上述MITF c.740T>C(L247S/ L247S)基因编辑猪模型为基础,利用HA3A-eBE-Y130F对突变猪早期胚胎进行了校正,基因分型表明,杂合子、纯合子胚胎的纠正成功率分别为16.13%、10.87%,可通过产前基因治疗实现对该致病性突变的彻底纠正。但hA3AeBE-Y130F对该猪成纤维细胞产生的SNVs脱靶效应是CRISPR/Cas9介导的HDR的5倍,碱基编辑工具仍需要不断优化改进。
2.4 杜氏肌肉营养不良症(Duchenne muscular dystrophy, DMD)模型猪DMD是一种X连锁遗传性肌营养不良症,肌营养不良蛋白的缺失损害了肌肉纤维周围肌膜的稳定性,导致肌肉细胞膜破裂和渗漏,从而导致肌原纤维坏死,随后发生组织纤维化,被脂肪取代并丧失功能[56-58]。小鼠是模拟人类疾病的重要模式动物,但DMD小鼠模型却并不表现典型的DMD症状[5]。基于猪与人类的相似性,有望通过猪模拟人类DMD。Xie等[21]用BE3结合SCNT技术共构建了400个克隆猪胚胎,并移植到代孕母猪,得到了1头健康的DMD单等位基因精准修饰母猪,有待与WT公猪交配以产生表现DMD症状的猪。
2.5 免疫缺陷模型猪研究表明,RAG1或RAG2基因功能缺失的实验小鼠没有成熟的B和T细胞[59],IL2RG在T细胞分泌中起重要作用[60]。Xie等[21]用BE3结合SCNT技术得到5头免疫缺陷症仔猪,但2头RAG1、RAG2、IL2RG多基因编辑仔猪在出生后12、49 d相继因肺部感染而死亡,2头RAG1、RAG2双基因精准修饰仔猪也因相同原因在出生后41、75 d死亡,只有IL2RG基因精准修饰仔猪正常生长。与同年龄WT仔猪相比,多基因编辑仔猪的胸腺小叶萎缩,脾脏小而薄。上述研究成功制备免疫缺陷猪模型,为治疗和研究免疫缺陷疾病提供了生物材料。
2.6 人类巨口综合征(Ablepharon macrostomia syndrome, AMS)模型猪AMS和Barber-Say综合征(Barber-Say syndrome, BSS)是罕见的先天性外胚层发育不良,具有相似的临床特征:无眼睑、大口、小耳、皮肤松弛、毛发稀疏、鼻子和耳朵变形,以及乳头、生殖器、手指和手的各种异常,大部分智力和运动功能发育正常[61]。虽然AMS和BSS是由同一氨基酸Glu的错义突变引致,但表型取决于替代的氨基酸。TWIST2第75位的赖氨酸导致AMS,而谷氨酰胺或丙氨酸产生BSS[61]。TWIST2基因的1个碱基改变是导致AMS的主要原因。Li等[28]用BE3构建TWIST2基因编辑猪成纤维细胞,通过SCNT技术生产TWIST2基因编辑克隆猪,构建AMS模型猪,该模型猪复制了人类疾病的表型。
2.7 儿童早衰综合征(Hutchinson giford progeria syndrome,HGPS)模型猪HGPS是一种罕见的散发性常染色体显性综合征,涉及早衰,患者通常在约13岁时因心肌梗死或中风而死亡。大多数这种综合征的遗传基础是核纤层蛋白A/C(Lamin A/C,LMNA)基因的第608位氨基酸对应三联体密码子从GGC变为GGT,激活1个剪接供体位点,产生异常的核纤层蛋白A/C,出现皮肤硬化、关节挛缩、关节活动范围缩小、骨骼异常、脱发、生长障碍、凝血酶原时间延长、血清磷水平升高、低频传导性听力损失和功能性口腔缺损的现象[62]。Xie等[21]将体外转录的BE3 mRNA和LMNA-sgRNA,结合胚胎注射得到8头LMNA G608G基因精准修饰仔猪,其中有1头C → T纯合突变仔猪,但出生2 d内死亡,经耳朵总RNA的qPCR试验和心、肝、脾、肺、肾的Western blot检测,LMNA G608G基因精准修饰仔猪中存在核纤层蛋白A突变体(WT仔猪没有);进一步基因测序发现,有4头仔猪靶点处存在Indels;使用Cas-OFFinder预测脱靶位点,选择7个风险位点分析,8头仔猪中有1头存在脱靶突变,这表明BE3在生产基因精准修饰猪中存在安全性问题。
3 碱基编辑技术在猪遗传改良中的应用以CRISPR/Cas9为代表的基因编辑技术可以高效地对猪细胞基因组进行改造,将携带目标基因的改造细胞作为核供体用于体细胞克隆,只需1个世代就能稳定获得1种或多种优良的生产性状,因此在提高猪生长、肉质、繁殖能力和抗病能力上被广泛应用。但脱靶效应、基因大片段缺失、染色体结构异常乃至染色体整体缺失等,引发了学者对CRISPR/Cas9有关生物安全的顾虑[9-11, 63]。与CRISPR/Cas9相比,碱基编辑技术对基因组的有害切割更小,具有更高的精准性和生物安全性,因而有望在猪遗传改良上发挥更大作用。
3.1 改良猪的生长性状猪肉是重要的肉类食品,提高猪的产肉量和瘦肉率是猪遗传改良的重要目标。MSTN基因是介导哺乳动物骨骼肌发育的负调控因子,抑制其表达可以增加瘦肉率和促进肌肉生长[64-66]。早期研究发现,MSTN SNP会导致比利时蓝牛产生双肌臀的表型[67]。由于MSTN在骨骼肌生长发育中的特殊功能,MSTN基因成为提高家畜瘦肉率、降低脂肪率、改善肉质的基因编辑技术靶点。已有多个研究团队利用CRISPR/Cas9技术通过敲除MSTN基因以提升猪的产肉性能。2020年,Li等[68]和我们研究团队[69]分别得到敲除MSTN的小耳花猪和巴马小型猪,均表现出肌纤维数量增加和肌纤维长度减小。基因编辑介导的MSTN敲除能够显著提高猪的产肉性状,提高猪的经济价值,但CRISPR/Cas9的DSB可能引发安全性问题[9-11, 63]。为减少安全性问题,减少Indels的产生,模拟MSTN自然突变过程十分重要。Wang等[70]和我们研究团队[71]在MSTN的PAM位点对应序列设计sgRNA进行C → T转换,产生提前终止密码子,导致猪成纤维细胞中产生MSTN的无义突变。该研究结果在动物遗传育种改良中具有很大应用潜力,但还需通过构建活体基因编辑猪进行性能和生物安全验证。
3.2 提高猪的抗病性能我国生猪的存栏量约占世界的50%,高传染性、高致死性猪病不仅会给畜牧业带来巨大的经济损失,一旦流行甚至会带来粮食安全问题。疫苗的研发应用可使某些传染性猪病得到一定程度的控制,但由于对野猪群体控制难度大、疫苗具有局限性等,很多猪传染病难以消灭。猪呼吸与繁殖综合征是蓝耳病病毒(Porcine reproductive and respiratory syndrome virus, PRRSV)引发的具有较高发病率和死亡率的传染病,是世界范围内养猪业最重要的经济疾病之一[72-74]。2014年起,Whitworth等[75]针对PRRSV的受体生产基因敲除猪。Burkard等[76]利用CRISPR/Cas9技术敲除PRRSV入侵机体的细胞受体CD163,成功获得能够抵抗PRRSV感染的基因编辑猪。为了避免安全性问题,减少Indels的产生和模拟CD163自然突变十分重要。Wang等[70]在CD163的PAM位点对应序列设计多个sgRNA进行C → T转化,导致猪成纤维细胞中产生CD163的无义突变。这项研究为预防猪病毒性传染疾病提供了新思路。除蓝耳病外,还存在非洲猪瘟、流行性腹泻和圆环病毒病等一系列猪病[77]。氨基肽酶-N(Aminopeptidase, APN)是一种多功能蛋白,参与多肽代谢、细胞粘附和冠状病毒进入等多种生物学过程的调节,其广泛存在猪小肠上皮细胞中[78]。应用基因编辑技术对相关基因进行改良,能有效降低猪的不良性状和疾病发生率。
3.3 多性状联合改良随着功能基因组学和生物信息学的发展,学者在家畜中鉴定出越来越多有关经济性状的SNP。MC4R基因型与许多品系的背部脂肪、生长速率和总体采食量之间存在显著相关[79],MC4R c.893G>A会导致猪MC4R功能显著改变。农业经济性状主要是由多个SNP联合调控。开发高效、精准的基因组修饰工具,对多个经济性状基因位点或多个功能基因进行同时修饰,对加速猪的遗传改良十分重要。Wang等[70]将SpCas9-NG突变体与hA3A-BE3融合构建hA3A-BE3-NG系统,该融合系统可大大提高可编辑范围,在高甲基化背景下也能保持一定水平的编辑效率,适用于改良猪的经济性状。Wang等[70]在CD163、APN、MSTN和MC4R基因中NG的PAM位点对应序列设计sgRNA进行C → T转换,导致猪成纤维细胞中产生多个经济性状相关基因的无义和错义突变。值得注意的是,由于hA3A-BE3-NG编辑窗口较宽,旁观者效应(Bystander effect)导致的C → T较多,且存在一定量的非C → T突变、脱靶突变和Indels。采用碱基编辑技术能够改良猪育种,但目前已经确定能够单一控制性状的基因少,而且受到PAM和编辑窗口窄的限制,碱基编辑技术用于某些特定基因的特定靶点较少。
4 碱基编辑系统在猪成纤维细胞中的编辑效率和脱靶情况赵建国研究团队和我们研究团队较关注碱基编辑系统在猪成纤维细胞中的编辑效率。鉴于将碱基编辑系统用于猪细胞的目的是后续生产基因克隆猪,因此未有在其他细胞系的相关试验。赵建国研究团队用hA3A-BE3、hA3A-BE3-Y130F、hA3A-eBE3-Y130F分别对猪胎儿肾脏成纤维细胞的IGF2进行编辑,碱基编辑效率分别28.57%、23.53%、19.23%,纯合突变率分别为16.67%、1.96%、1.92%;Indels分别为42.86%、31.37%、21.15%,旁侧突变率分别为28.57%、7.84%、9.60%,通过优化碱基编辑工具,在维持一定编辑效率的前提下可逐步降低Indels和旁侧突变率;此外,使用hA3A-BE3-Y130F对猪胎儿肾脏成纤维细胞的IGF2进行编辑还存在非C → T突变[80]。该研究团队还用hA3A-BE3-NG对猪胎儿肾脏成纤维细胞的APN、CD163、MC4R、MSTN进行多基因编辑,结果显示,对APN的碱基编辑效率为33.33%,旁侧突变率为35.19%,Indels为9.26%,非C → T突变率为1.85%;对CD163的碱基编辑效率为35.19%,旁侧突变率为12.96%,Indels为5.56%,非C → T突变率为9.26%;对MC4R的碱基编辑效率为5.56%,旁侧突变率、Indels、非C → T突变率均为0%;对MSTN的碱基编辑效率为46.30%,旁侧突变率为18.52%,Indels为1.85%,非C → T突变率为11.11%[70]。在碱基编辑效率较高的靶点伴有一定的旁侧突变、Indels以及非C → T突变。
我们研究团队改良的PX-YE1-BE4MaxNG和PX-ABEmaxAW碱基编辑工具,分别针对C → T和A → G转换,前者大多用于提前引入终止密码子,后者大多用于破坏起始密码子,引入外显子跳跃,以及部分点突变引起性状修复或损坏。我们使用PX-YE1-BE4Max-NG对猪胎儿肾脏成纤维细胞MSTN site1-5、ApoA5、CD163、LDLR进行编辑效率测试,其编辑效率为1%~30.1%[71];使用PX-ABEmaxAW对猪胎儿肾脏成纤维细胞IRX3、MSTN site1-2、DMD进行编辑效率测试,其编辑效率为27.4%~49.4%[81]。后续利用PX-YE1-BE4Max-NG、PX-ABEmaxAW分别对猪胎儿肾脏成纤维细胞的MSTN、GHR进行基因编辑,碱基编辑效率分别为28.6%、33.33%,纯合突变率分别为9.5%、33.33%。我们研究团队改良的碱基编辑工具涵盖了C → T和A → G突变,在靶点上保持碱基突变效率的同时无旁侧突变、Indels以及非C → T突变,采用CRISPOR(http://crispor.tefor.net)选择最有可能脱靶的位点进行脱靶测试,发现均无Indels和潜在碱基突变,后续可采用更完善的脱靶鉴定方法进行检测。
5 展望与利用HDR的CRISPR/Cas9技术相比,ABE、CBE等碱基编辑系统更精准、更便捷、更高效;与仅能用于功能验证的转染过表达载体相比,ABE、CBE等在疾病模型构建、异体器官移植和精准遗传改良方面更符合自然规则。鉴于猪的生理特性与人非常相似,猪的人类疾病模型对人类临床前研究非常有价值。研究人员先后培育出免疫缺陷症、白化病、杜氏肌营养不良症、儿童早衰症的碱基编辑疾病模型猪,为疾病发病机制和治疗研究提供了理想的大动物模型。而在农业上,CD163、APN、MSTN和MC4R等靶点碱基编辑猪的成功构建,为高效精准的性状改良和抗病育种提供了新方案。
目前,在猪精准修饰中碱基编辑技术的限制主要体现在以下方面:(1)仍不能确保不产生任何副产物、脱靶突变和Indels;(2)由于受PAM限制,以及编辑活性窗口窄、不同动物不同类型细胞的编辑活性差异大的限制,碱基编辑技术仍不能对任意细胞中的任何位点实现高效编辑,因此仍有许多常见的人类疾病模型难以建立,仍有许多猪优良性状难以通过碱基编辑技术培育;(3)大动物研究存在生产成本高、养殖和维护设施昂贵,以及动物福利等相关难题;(4)大部分情况下,研究者只能采用导入质粒编辑体细胞+ 体细胞核移植的方法,试验步骤繁琐。此外,传统的显微注射方法具有持续的Cas9切割活性,其产生的大部分基因编辑胚胎或动物都存在基因型嵌合。随着世界各研究团队的深耕,碱基编辑技术不断发展,许多不足正在逐步改善。单碱基编辑技术的不断发展将推动猪精准修饰在医学和农业上的应用,推动遗传病治疗、异种器官移植、精准农产品改良快速发展,造福人类。
| [1] |
JINEK M, CHYLINSKI K, FONFARA I, HAUER M, DOUDNA J A, CHARPENTIER E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [2] |
CONG L, RAN F A, COX D, LIN S, BARRETTO R, HABIB N, HSU P D, WU X, JIANG W, MARRAFFINI L A, ZHANG F. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [3] |
FENG Z Y, ZHANG B T, DING W N, LIU X D, YANG D L, WEI P L, CAO F Q, ZHU S H, ZHANG F, MAO Y F, ZHU J K. Efficient genome editing in plants using a CRISPR/Cas system[J]. Cell Research, 2013, 23(10): 1229-1232. DOI:10.1038/cr.2013.114 |
| [4] |
MIAO J, GUO D S, ZHANG J Z, HUANG Q P, QIN G J, ZHANG X, WAN J M, GU H Y, QU L J. Targeted mutagenesis in rice using CRISPR-Cas system[J]. Cell Research, 2013, 23(10): 1233-1236. DOI:10.1038/cr.2013.123 |
| [5] |
NAKAMURA K, FUJII W, TSUOI M, TANIHATA J, TERAMOTO N, TAKEUCHI S, NAITO K, YAMANOUCHI K, NISHIHARA M. Generation of muscular dystrophy model rats with a CRISPR/Cas system[J]. Scientifi c Reports, 2014(4): 5635. DOI:10.1038/srep05635 |
| [6] |
GAO J P, WANG G H, MA S Y, XIE X D, WU X W, ZHANG X T, WU Y Q, ZHAO P, XIA Q Y. CRISPR/Cas9-mediated targeted mutagenesis in Nicotiana tabacum[J]. Plant Molecular Biology, 2015, 87(1/2): 99-110. DOI:10.1007/s11103-014-0263-0 |
| [7] |
单奇伟, 高彩霞. 植物基因组编辑及衍生技术最新研究进展[J]. 遗传, 2015, 37(10): 953-973. DOI:10.16288/j.yczz.15-156 SHAN Q W, GAO C X. Research progress of genome editing and derivative technologies in plants[J]. Hereditas, 2015, 37(10): 953-973. DOI:10.16288/j.yczz.15-156 |
| [8] |
YU H H, ZHAO H, QING Y B, PAN W R, JIA B Y, ZHAO H Y, HUANG X X, WEI H J. Porcine zygote injection with Cas9/sgRNA results in DMD-modified pig with muscle dystrophy[J]. International Journal of Molecular Sciences, 2016, 17(10): 1668. DOI:10.3390/ijms17101668 |
| [9] |
HAAPANIEMI E, BOTLA S, PERSSON J, SCHMIERER B, TAIPALE J. CRISPR-Cas9 genome editing induces a p53-mediated DNA damage response[J]. Nature Medicine, 2018, 24(7): 927-930. DOI:10.1038/s41591-018-0049-z |
| [10] |
IHRY R J, WORRINGER K A, SALICK M R, FRIAS E, HO D, THERIAULT K, KOMMINENI S, CHEN J, SONDEY M, YE C, RANDHAWA R, KULKARNI T, YANG Z, MCALLISTER G, RUSS C, REECE-HOYES J, FORRESTER W, HOFFMAN G R, DOLMETSCH R, KAYKAS A. p53 inhibits CRISPR-Cas9 engineering in human pluripotent stem cells[J]. Nature Medicine, 2018, 24(7): 939-946. DOI:10.1038/s41591-018-0050-6 |
| [11] |
KOSICKI M, TOMBERG K, BRADLEY A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements[J]. Nature Biotechnology, 2018, 36(8): 765-771. DOI:10.1038/nbt.4192 |
| [12] |
GAUDELLI N M, KOMOR A C, REES H A, PACKER M S, BADRAN A H, BRYSON D I, LIU D R. Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage[J]. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644 |
| [13] |
KOMOR A C, KIM Y B, PACKER M S, ZURIS J A, LIU D R. Programmable editing of a target base in genomic DNA without doublestranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946 |
| [14] |
LIANG P P, DING C H, SUN H W, XIE X W, XU Y W, ZHANG X Y, SUN Y, XIONG Y Y, MA W B, LIU Y X, WANG Y L, FANG J P, LIU D, ZHOU S Y, ZHOU C Q, HUANG J J. Correction of beta-thalassemia mutant by base editor in human embryos[J]. Protein Cell, 2017, 8(11): 811-822. DOI:10.1007/s13238-017-0475-6 |
| [15] |
REN B, YAN F, KUANG Y J, LI N, ZHANG D W, LIN H H, ZHOU H B. A CRISPR/Cas9 toolkit for efficient targeted base editing to induce genetic variations in rice[J]. Science China Life Sciences, 2017, 60(5): 516-519. DOI:10.1007/s11427-016-0406-x |
| [16] |
ZONG Y, WANG Y P, LI C, ZHANG R, CHEN K L, RAN Y D, QIU J L, WANG D W, GAO C X. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion[J]. Nature Biotechnology, 2017, 35(5): 438-440. DOI:10.1038/nbt.3811 |
| [17] |
LIU Z, LU Z Y, YANG G, HUANG S S, LI G L, FENG S J, LIU Y J, LI J N, YU W X, ZHANG Y, CHEN J, SUN Q, HUANG X X. Efficient generation of mouse models of human diseases via ABE-and BEmediated base editing[J]. Nature Communications, 2018, 9(1): 2338. DOI:10.1038/s41467-018-04768-7 |
| [18] |
RYU S M, KOO T, KIM K, LIM K, BAEK G, KIM S T, KIM H S, KIM D E, LEE H, CHUNG E, KIM J S. Adenine base editing in mouse embryos and an adult mouse model of duchenne muscular dystrophy[J]. Nature Biotechnology, 2018, 36(6): 536-539. DOI:10.1038/nbt.4148 |
| [19] |
SASAGURI H, NAGATA K, SEKIGUCHI M, FUJIOKA R, MATSUBA Y, HASHIMOTO S, SATO K, KURUP D, YOKOTA T, SAIDO T C. Introduction of pathogenic mutations into the mouse Psen1 gene by base editor and target-AID[J]. Nature Communications, 2018, 9(1): 2892. DOI:10.1038/s41467-018-05262-w |
| [20] |
ZENG Y T, LI J N, LI G L, HUANG S S, YU W X, ZHANG Y, CHEN D J, CHEN J, LIU J Q, HUANG X X. Correction of the marfan syndrome pathogenic FBN1 mutation by base editing in human cells and heterozygous embryos[J]. Molecular Therapy, 2018, 26(11): 2631-2637. DOI:10.1016/j.ymthe.2018.08.007 |
| [21] |
XIE J K, GE W K, LI N, LIU Q S, CHEN F B, YANG X Y, HUANG X Y, OUYANG Z, ZHANG Q J, ZHAO Y, LIU Z M, GOU S X, WU H, LAI C D, FAN N N, JIN Q, SHI H, LIANG Y H, LAN T, QUAN L Q, LI X P, WANG K P, LAI L X. Efficient base editing for multiple genes and loci in pigs using base editors[J]. Nature Communications, 2019, 10(1): 2852. DOI:10.1038/s41467-019-10421-8 |
| [22] |
LEVY J M, YEH W H, PENDSE N, DAVIS J R, HENNESSEY E, BUTCHER R, KOBLAN L W, COMANDER J, LIU Q, LIU D R. Cytosine and adenine base editing of the brain, liver, retina, heart and skeletal muscle of mice via adeno-associated viruses[J]. Nature Biomedical Engineering, 2020, 4(1): 97-110. DOI:10.1038/s41551-019-0501-5 |
| [23] |
ZHANG X H, ZHU B Y, CHEN L, XIE L, YU W S, WANG Y, LI L X, YIN S M, YANG L, HU H D, HAN H H, LI Y M, WANG L R, CHEN G, MA X Y, GENG H Q, HUANG W F, PANG X F, YANG Z Z, WU Y X, SIWKO S, KURITA R, NAKAMURA Y, YANG L, LIU M Y, LI D L. Dual base editor catalyzes both cytosine and adenine base conversions in human cells[J]. Nature Biotechnology, 2020, 38(7): 856-860. DOI:10.1038/s41587-020-0527-y |
| [24] |
KURT I C, ZHOU R, IYER S, GARCIA S P, MILLER B R, LANGNER L M, GRUNEWALD J, JOUNG J K. CRISPR C-to-G base editors for inducing targeted DNA transversions in human cells[J]. Nature Biotechnology, 2021, 39(1): 41-46. DOI:10.1038/s41587-020-0609-x |
| [25] |
CHADWICK A C, WANG X, MUSUNURU K. In Vivo base editing of PCSK9 as a therapeutic alternative to genome editing[J]. Arteriosclerosis Thrombosis And Vascular Biology, 2017, 37(9): 1741-1747. DOI:10.1161/ATVBAHA.117.309881 |
| [26] |
ZHANG Y H, QIN W, LU X C, XU J S, HUANG H G, BAI H P, LI S, LIN S. Programmable base editing of zebrafish genome using a modified CRISPR-Cas9 system[J]. Nature Communications, 2017, 8(1): 118. DOI:10.1038/s41467-017-00175-6 |
| [27] |
LI Y F, MA S Y, SUN L, ZHANG T, CHANG J S, LU W, CHEN X X, LIU Y, WANG X G, SHI R, ZHAO P, XIA Q Y. Programmable single and multiplex base-editing in Bombyx mori using RNA-guided cytidine deaminases[J]. G3(Bethesda), 2018, 8(5): 1701-1709. DOI:10.1534/g3.118.200134 |
| [28] |
LI Z F, DUAN X Y, AN X M, FENG T, LI P, LI L L, LIU J, WU P X, PAN D K, DU X G, WU S. Efficient RNA-guided base editing for disease modeling in pigs[J]. Cell Discovery, 2018(4): 64. DOI:10.1038/s41421-018-0065-7 |
| [29] |
LIU Z Q, CHEN M, CHEN S Y, DENG J C, SONG Y N, LAI L X, LI Z J. Highly efficient RNA-guided base editing in rabbit[J]. Nature Communications, 2018, 9(1): 2717. DOI:10.1038/s41467-018-05232-2 |
| [30] |
YANG G, ZHU T Y, LU Z Y, LI G L, ZHANG H, FENG S J, LIU Y J, LI J N, ZHANG Y, CHEN J, GUO X J, HUANG X X. Generation of is ogenic single and multiplex gene knockout mice by base editinginduced STOP[J]. Science Bulletin, 2018, 63(17): 1101-1107. DOI:10.1016/j.scib.2018.07.002 |
| [31] |
LI G W, ZHOU S W, LI C, CAI B, YU H H, MA B H, HUANG Y, DING Y G, LIU Y, DING Q, HE C, ZHOU J K, WANG Y, ZHOU G X, LI Y, YAN Y, HUA J L, PETERSEN B, JIANG Y, SONSTEGARD T, HUANG X X, CHEN Y L, WANG X L. Base pair editing in goat: nonsense codon introgression into FGF5 results in longer hair[J]. FEBS Journal, 2019, 286(23): 4675-4692. DOI:10.1111/febs.14983 |
| [32] |
ZHOU S W, CAI B, HE C, WANG Y, DING Q, LIU J, LIU Y, DING Y G, ZHAO X E, LI G W, LI C, YU H H, KOU Q F, NIU W Z, PETERSEN B, SONSTEGARD T, MA B, CHEN Y L, WANG X L. Programmable base editing of the sheep genome revealed no genome-wide off-target mutations[J]. Frontiers in Genetics, 2019, 10: 215. DOI:10.3389/fgene.2019.00215 |
| [33] |
HESS G T, FRESARD L, HAN K, LEE C H, LI A, CIMPRICH K A, MONTGOMERY S B, BASSIK M C. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells[J]. Nature Methods, 2016, 13(12): 1036-1042. DOI:10.1038/nmeth.4038 |
| [34] |
MA Y Q, ZHANG J Y, YIN W J, ZHANG Z C, SONG Y, CHANG X. Targeted AID-mediated mutagenesis (TAM) enables efficient genomic diversification in mammalian cells[J]. Nature Methods, 2016, 13(12): 1029-1035. DOI:10.1038/nmeth.4027 |
| [35] |
KIM K, RYU S M, KIM S T, BAEK G Y, KIM D, LIM K, CHUNG E, KIM S, KIM J S. Highly efficient RNA-guided base editing in mouse embryos[J]. Nature Biotechnology, 2017, 35(5): 435-437. DOI:10.1038/nbt.3816 |
| [36] |
KOMOR A C, BADRAN A H, LIU D R. CRISPR-based technologies for the manipulation of eukaryotic genomes[J]. Cell, 2017, 168(1/2): 20-36. DOI:10.1016/j.cell.2016.10.044 |
| [37] |
LU Y M, ZHU J K. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system[J]. Molecular Plant, 2017, 10(3): 523-525. DOI:10.1016/j.molp.2016.11.013 |
| [38] |
KOMOR A C, ZHAO K T, PACK ER M S, GAUDEL L I N M, WATERBURY A L, KOBLAN L W, KIM Y B, BADRAN A H, LIU D R. Improved base excision repair inhibition and bacteriophage Mu Gam protein yields C: G-to-T: A base editors with higher efficiency and product purity[J]. Science Advances, 2017, 3(8): eaao4774. DOI:10.1126/sciadv.aao4774 |
| [39] |
KOBLAN L W, DOMAN J L, WILSON C, LEVY J M, TAY T, NEWBY G A, MAIANTI J P, RAGURAM A, LIU D R. Improving cytidine and adenine base editors by expression optimization and ancestral reconstruction[J]. Nature Biotechnology, 2018, 36(9): 843-846. DOI:10.1038/nbt.4172 |
| [40] |
ZUO E W, SUN Y D, WEI W, YUAN T L, YING W Q, SUN H, YUAN L Y, STEINMETZ L M, LI Y X, YANG H. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos[J]. Science, 2019, 364(6437): 289-292. DOI:10.1126/science.aav9973 |
| [41] |
JIN J, ZONG Y, GAO Q, ZHU Z X, WANG Y P, QIN P, LIANG C Z, WANG D W, QIU J L, ZHANG F, GAO C X. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice[J]. Science, 2019, 364(6437): 292-295. DOI:10.1126/science.aaw7166 |
| [42] |
DOMAN J L, RAGURAM A, NEWBY G A, LIU D R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors[J]. Nature Biotechnology, 2020, 38(5): 620-628. DOI:10.1038/s41587-020-0414-6 |
| [43] |
REES H A, WILSON C, DOMAN J L, LIU D R. Analysis and minimization of cellular RNA editing by DNA adenine base editors[J]. Science Advances, 2019, 5(5): eaax5717. DOI:10.1126/sciadv.aax5717 |
| [44] |
GRUNEWALD J, ZHOU R H, LAREAU C A, GARCIA S P, IYER S, MILLER B R, LANGNER L M, HSU J Y, ARYEE M J, JOUNG J K. A dual-deaminase CRISPR base editor enables concurrent adenine and cytosine editing[J]. Nature Biotechnology, 2020, 38(7): 861-864. DOI:10.1038/s41587-020-0535-y |
| [45] |
YUAN T L, YAN N N, FEI T Y, ZHENG J T, MENG J, LI N N, LIU J, ZHANG H H, XIE L, YING W Q, LI D, SHI L, SUN Y S, LI Y Y, LI Y X, SUN Y D, ZUO E W. Optimization of C-to-G base editors with sequence context preference predictable by machine learning methods[J]. Nature Communications, 2021, 12(1): 4902. DOI:10.1038/s41467-021-25217-y |
| [46] |
NIU D, WEI H J, LIN L, GEORGE H, WANG T, LEE I H, ZHAO H Y, WANG Y, KAN Y N, SHROCK E, LESHA E, WANG G, LUO Y L, QING Y B, JIAO D L, ZHAO H, ZHOU X Y, WANG S Q, WEI H, GUELL M, CHURCH G M, YANG L H. Inactivation of porcine endogenous retrovirus in pigs using CRISPR-Cas9[J]. Science, 2017, 357(6357): 1303-1307. DOI:10.1126/science.aan4187 |
| [47] |
ZHANG R J, WANG Y, CHEN L, WANG R G, LI C, LI X X, FANG B, REN X Y, RUAN M M, LIU J Y, XIONG Q, ZHANG L N, JIN Y, ZHANG M L, LIU X R, LI L, CHEN Q, PAN D K, LI R F, COOPER D K C, YANG H Y, DAI Y F. Reducing immunoreactivity of porcine bioprosthetic heart valves by genetically-deleting three major glycan antigens, GGTA1/β4GalNT2/CMAH[J]. Acta Biomater, 2018, 72: 196-205. DOI:10.1016/j.actbio.2018.03.055 |
| [48] |
PETERSEN B, FRENZEL A, LUCAS-HAHN A, HERRMANN D, HASSEL P, KLEIN S, ZIEGLER M, HADELER K G, NIEMANN H. Efficient production of biallelic GGTA1 knockout pigs by cytoplasmic microinjection of CRISPR/Cas9 into zygotes[J]. Xenotransplantation, 2016, 23(5): 338-346. DOI:10.1111/xen.12258 |
| [49] |
YUAN H M, YU T T, WANG L Y, YANG L, ZHANG Y Z, LIU H, LI M J, TANG X C, LIU Z Q, LI Z J, LU C, CHEN X, PANG D X, OUYANG H S. Efficient base editing by RNA-guided cytidine base editors (CBEs) in pigs[J]. Cellular and Molecular Life Sciences, 2020, 77(4): 719-733. DOI:10.1007/s00018-019-03205-2 |
| [50] |
OETTING W S, KING R A. Molecular basis of albinism: mutations and polymorphisms of pigmentation genes associated with albinism[J]. Human Mutation, 1999, 13(2): 99-115. |
| [51] |
OETTING W S. The tyrosinase gene and oculocutaneous albinism type 1(OCA1): A model for understanding the molecular biology of melanin formation[J]. Pigment Cell Research, 2000, 13(5): 320-325. DOI:10.1034/j.1600-0749.2000.130503.x |
| [52] |
SONG J, FENG Y, ACKE F R, COUCKE P, VLEMINCKX K, DHOOGE I J. Hearing loss in Waardenburg syndrome: a systematic review[J]. Clinical Genetics, 2016, 89(4): 416-425. DOI:10.1111/cge.12631 |
| [53] |
OHNO N, K I YOSAWA M, MOR I H, WA NG W F, TA K A SE H, MOCHI ZUK I M. Clinical findings in Japanese patients with Waardenburg syndrome type 2[J]. Japanese Journal of Ophthalmology, 2003, 47(1): 77-84. DOI:10.1016/s0021-5155(02)00629-9 |
| [54] |
HAI T, GUO W W, YAO J, CAO C, LUO A, QI M, WANG X L, WANG X, HUANG J J, ZHANG Y, ZHANG H Y, WANG D Y, SHANG H T, HONG Q L, ZHANG R, JIA Q T, ZHENG Q T, QIN G S, LI Y S, ZHANG T, JIN W W, CHEN Z Y, WANG H M, ZHOU Q, MENG A M, WEI H, YANG S M, ZHAO J G. Creation of miniature pig model of human Waardenburg syndrome type 2A by ENU mutagenesis[J]. Human Genetics, 2017, 136(11/12): 1463-1475. DOI:10.1007/s00439-017-1851-2 |
| [55] |
YAO J, WANG Y, CAO C W, SONG R G, BI D F, ZHANG H Y, LI Y S, QIN G S, HOU N P, ZHANG N, ZHANG J, GUO W W, YANG S M, WANG Y F, ZHAO J G. CRISPR/Cas9-mediated correction of MITF homozygous point mutation in a Waardenburg syndrome 2A pig model[J]. Molecular Therapy Nucleic Acids, 2021, 24: 986-999. DOI:10.1016/j.omtn.2021.04.009 |
| [56] |
HOFFMAN E P, JR R H B, KUNKEL L M. Dystrophin: the protein product of the Duchenne muscular dystrophy locus[J]. Cell, 1987, 51(6): 919-928. DOI:10.1016/0092-8674(87)90579-4 |
| [57] |
KOENIG M, MONACO A P, KUNKEL L M. The complete sequence of dystrophin predicts a rod-shaped cytoskeletal protein[J]. Cell, 1988, 53(2): 219-228. DOI:10.1016/0092-8674(88)90383-2 |
| [58] |
KINALI M, ARECHAVALA-GOMEZA V, CIRAK S, GLOVER A, GUGLIERI M, FENG L, HOLLINGSWORTH K G, HUNT D, JUNGBLUTH H, ROPER H P, QUINLIVAN R M, GOSALAKKAL J A, JAYAWANT S, NADEAU A, HUGHES-CARRE L, MANZUR A Y, MERCURI E, MORGAN J E, STRAUB V, BUSHBY K, SEWRY C, RUTHERFORD M, MUNTONI F. Muscle histology vs MRI in duchenne muscular dystrophy[J]. Neurology, 2011, 76(4): 346-353. DOI:10.1212/WNL.0b013e318208811f |
| [59] |
SCHWARZ K, GAUSS G H, LUDWIG L, PANNICKE U, LI Z, LINDNER D, FRIEDRICH W, SEGER R A, HANSEN-HAGGE T E, DESIDERIO S, LIEBER M R, BARTRAM C R. RAG mutations in human B cell-negative SCID[J]. Science, 1996, 274(5284): 97-99. DOI:10.1126/science.274.5284.97 |
| [60] |
NOGUCHI M, YI H, ROSENBLATT H M, FILIPOVICH A H, ADELSTEIN S, MODI W S, MCBRIDE O W, LEONARD W J. Interleukin-2 receptor gamma chain mutation results in X-linked severe combined immunodeficiency in humans[J]. Cell, 1993, 73(1): 147-157. DOI:10.1016/0092-8674(93)90167-o |
| [61] |
MARCHEGIANI S, DAVIS T, TESSADORI F, HAAFTEN G V, BRANCATI F, HOISCHEN A, HUANG H G, VALKANAS E, PUSEY B, SCHANZE D, VENSELAAR H, SILFHOUT A T V, WOLFE L A, TIFFT C J, ZERFAS P M, ZAMBRUNO G, KARIMINEJAD A, SABBAGH-KERMANI F, LEE J, TSOKOS M G, LEE C R, FERRAZ V, SILVA E M D, STEVENS C A, ROCHE N, BARTSCH O, FARNDON P, BERMEJO-SANCHEZ E, BROOKS B P, MADURO V, DALLAPICCOLA B, RAMOS F J, CHUNG H B, CAIGNEC C L, MARTINS F, JACYK W K, MAZZANTI L, BRUNNER H G, BAKKERS J, LIN S, MALICDAN M C V, BOERKOEL C F, GAHL W A, VRIES B B A D, HAELST M M V, ZENKER M, MARKELLO T C. Recurrent mutations in the basic domain of TWIST2 cause ablepharon macrostomia and barber-say syndromes[J]. American Journal of Human Genetics, 2015, 97(1): 99-110. DOI:10.1016/j.ajhg.2015.05.017 |
| [62] |
MERIDETH M A, GORDON L B, CLAUSS S, SACHDEV V, SMITH A C M, PERRY M B, BREWER C C, ZALEWSKI C, KIM H J, SOLOMON B, BROOKS B P, GERBER L H, TURNER M L, DOMINGO D L, HART T C, GRAF J, REYNOLDS J C, GROPMAN A, YANOVSKI J A, GERHARD-HERMAN M, COLLINS F S, NABEL E G, CANNON R O, GAHL W A, INTRONE W J. Phenotype and course of Hutchinson-Gilford progeria syndrome[J]. New England Journal of Medicine, 2008, 358(6): 592-604. DOI:10.1056/NEJMoa0706898 |
| [63] |
ZUCCARO M V, XU J, MITCHELL C, MARIN D, ZIMMERMAN R, RANA B, WEINSTEIN E, KING R T, PALMEROLA K L, SMITH M E, TSANG S H, GOLAND R, JASIN M, LOBO R, TREFF N, EGLI D. Allele-specific chromosome removal after Cas9 cleavage in human embryos[J]. Cell, 2020, 183(6): 1650-1664. DOI:10.1016/j.cell.2020.10.025 |
| [64] |
MCPHERRON A C, LAWLER A M, LEE S J. Regulation of skeletal muscle mass in mice by a new TGF-beta superfamily member[J]. Nature, 1997, 387(6628): 83-90. DOI:10.1038/387083a0 |
| [65] |
BURRONE J, O'BYRNE M, MURTHY V N. Multiple forms of synaptic plasticity triggered by selective suppression of activity in individual neurons[J]. Nature, 2002, 420(6914): 414-418. DOI:10.1038/nature01242 |
| [66] |
LEE S J. Quadrupling muscle mass in mice by targeting TGF-beta signaling pathways[J]. PLoS One, 2007, 2(8): e789. DOI:10.1371/journal.pone.0000789 |
| [67] |
MCPHERRON A C, LEE S J. Double muscling in cattle due to mutations in the myostatin gene[J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(23): 12457-12461. DOI:10.1073/pnas.94.23.12457 |
| [68] |
LI R Q, ZENG W, MA M, WEI Z X, LIU H B, LIU X F, WANG M, SHI X, ZENG J H, YANG L F, MO D L, LIU X H, CHEN Y S, HE Z Y. Precise editing of myostatin signal peptide by CRISPR/Cas9 increases the muscle mass of Liang Guang Small Spotted pigs[J]. Transgenic Research, 2020, 29(1): 149-163. DOI:10.1007/s11248-020-00188-w |
| [69] |
ZHU X X, ZHAN Q M, WEI Y Y, YAN A F, FENG J, LIU L, LU S S, TANG D S. CRISPR/Cas9-mediated MSTN disruption accelerates the growth of Chinese Bama pigs[J]. Reproduction in Domestic Animals, 2020, 55(10): 1314-1327. DOI:10.1111/rda.13775 |
| [70] |
WANG Y, BI D F, QIN G S, SONG R S, YAO J, CAO C W, ZHENG Q T, HOU N P, WANG Y F, ZHAO J G. Cytosine base editor (hA3ABE3-NG)-mediated multiple gene editing for pyramid breeding in pigs[J]. Frontiers in Genetics, 2020(11): 592623. DOI:10.3389/fgene.2020.592623 |
| [71] |
PAN J S, LIN Z S, WEN J C, GUO J F, WU X H, LIU Y Y, LAI W J, LIANG Q Y, XIE Y S, CHEN Y R, CHEN Y H, YAN A F, FENG J, LIU L, GONG D Y, ZHU X X, LU J H, TANG D S. Application of the modified cytosine base-editing in the cultured cells of bama minipig[J]. Biotechnology Letters, 2021, 43(9): 1699-1714. DOI:10.1007/s10529-021-03159-1 |
| [72] |
COLLINS J E, BENFIELD D A, CHRISTIANSON W T, HARRIS L, HENNINGS J C, SHAW D P, GOYAL S M, MCCULLOUGH S, MORRISON R B, JOO H S. Isolation of swine infertility and respiratory syndrome virus (isolate ATCC VR-2332) in North America and experimental reproduction of the disease in gnotobiotic pigs[J]. Journal of Veterinary Diagnostic Investigation, 1992, 4(2): 117-126. DOI:10.1177/104063879200400201 |
| [73] |
DONE S H, PATON D J, WHITE M E. Porcine reproductive and respiratory syndrome (PRRS): a review, with emphasis on pathological, virological and diagnostic aspects[J]. The British Veterinary Journal, 1996, 152(2): 157-174. DOI:10.1016/s0007-1935(96)80071-6 |
| [74] |
PEJSAK Z, STADEJEK T, MARKOWSKA-DANIEL I. Isolation of swine infertility and respiratory syndrome virus (isolate ATCC VR-2332) in North America and experimental reproduction of the disease in gnotobiotic pigs[J]. Veterinary Microbiology, 1997, 55(1-4): 317-322. DOI:10.1016/s0378-1135(96)01326-0 |
| [75] |
WHITWORTH K M, ROWLAND R R R, EWEN C L, TRIBLE B R, KERRIGAN M A, CINO-OZUNA A G, SAMUEL M S, LIGHTNER J E, MCLAREN D G, MILEHAM A J, WELLS K D, PRATHER R S. Geneedited pigs are protected from porcine reproductive and respiratory syndrome virus[J]. Nature Biotechnology, 2016, 34(1): 20-22. DOI:10.1038/nbt.3434 |
| [76] |
BURKARD C, LILLICO S G, REID E, JACKSON B, MILEHAM A J, AIT-ALI T, WHITELAW C B A, ARCHIBALD A L. Precision engineering for PRRSV resistance in pigs: Macrophages from genome edited pigs lacking CD163 SRCR5 domain are fully resistant to both PRRSV genotypes while maintaining biological function[J]. PLoS Pathogens, 2017, 13(2): e1006206. DOI:10.1371/journal.ppat.1006206 |
| [77] |
MADSON D M, ARRUDA P H E, MAGSTADT D R, BURROUGH E R, HOANG H, SUN D, BOWER L P, BHANDARI M, GAUGER P C, STEVENSON G W, WILBERTS B L, WANG C, ZHANG J, YOON K J. Characterization of porcine epidemic diarrhea virus isolate US/Iowa/18984/2013 infection in 1-day-old cesarean-derived colostrumdeprived piglets[J]. Veterinary Pathology, 2016, 53(1): 44-52. DOI:10.1177/0300985815591080 |
| [78] |
WHITWORTH K M, ROWLAND R R R, PETROVAN V, SHEAHAN M, CINO-OZUNA A G, FANG Y, HESSE R, MILEHAM A, SAMUEL M S, WELLS K D, PRATHER R S. Resistance to coronavirus infection in amino peptidase N-deficient pigs[J]. Transgenic Research, 2019, 28(1): 21-32. DOI:10.1007/s11248-018-0100-3 |
| [79] |
KIM K S, LARSEN N, SHORT T, PLASTOW G, ROTHSCHILD M F. A missense variant of the porcine melanocortin-4 receptor (MC4R) gene is associated with fatness, growth, and feed intake traits[J]. Mammalian Genome, 2000, 11(2): 131-135. DOI:10.1007/s003350010025 |
| [80] |
王煜, 宋瑞高, 赵建国, 王彦芳. 碱基编辑器介导的猪IGF2基因高效定点突变[J]. 中国畜牧兽医, 2020, 47(11): 3427-3435. DOI:10.16431/j.cnki.1671-7236.2020.11.002 WANG Y, SONG R G, ZHAO J G, WANG Y F. Base editor-mediated highly efficient site-directed mutation of porcine IGF2 gene[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(11): 3427-3435. DOI:10.16431/j.cnki.1671-7236.2020.11.002 |
| [81] |
ZHU X X, PAN J S, LIN T, YANG Y C, HUANG Q Y, YANG S P, QU Z X, LIN Z S, WEN J C, YAN A F, FENG J, LIU L, ZHANG X L, LU J H, TANG D S. Adenine base-editing-mediated exon skipping induces gene knockout in cultured pig cells[J]. Biotechnology Letters, 2022, 44(1): 59-76. DOI:10.1007/s10529-021-03214-x |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49