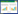文章信息
基金项目
- 广东省自然科学基金(2019A1515011825)
作者简介
- 林春姿(1996—),女,在读硕士生,研究方向为水稻抗稻瘟病育种,E-mail:1162825005@qq.com.
通讯作者
- 王加峰(1979—),男,博士,副研究员,研究方向为水稻抗病分子育种,E-mail:jfwang@scau.edu.cn.
文章历史
- 收稿日期:2022-05-25
【研究意义】钙调素(Calmodulin, CaM)作为细胞内主要的Ca2+传感蛋白,通过与不同的钙调素结合蛋白(Calmodulin Binding Proteins, CaMBPs)结合传递钙信号,调控细胞生理和生长发育过程。IQD家族蛋白属于一类CaMBPs,是Ca2+信号传导的重要感受器[1],在各种陆地植物上都很常见[2]。已发现的IQD家族蛋白的结构、生物学功能不尽相同,但大多在植物生长发育和调控生物胁迫与非生物胁迫过程中发挥重要作用,且它们的亚细胞定位模式各不相同[3]。AtIQD1与KLCR1和CaM相互作用,从而将驱动蛋白与Ca2+第二信使信号联系起来[4-5]。其他IQD家族蛋白也可能介导不同的驱动蛋白转运信号通路,例如蛋白质分选或细胞壁形成[6]。IQD家族最早在拟南芥和水稻中发现,已发现拟南芥、水稻、大豆、番茄、玉米中有33、29、67、34、26个IQD基因[2, 7]。目前绝大多数IQD1基因的具体功能尚不明确,利用CRISPR/Cas9编辑技术对水稻IQD基因家族成员OsIQD1进行定点编辑,获得的突变体对探究OsIQD1基因调控水稻生长发育与逆境响应的作用机制等均具有重要意义。【前人研究进展】IQD家族蛋白均含有IQ基序(IQxxxRGxxxR),其功能的深入研究是阐明Ca2+/CaM介导的胞内钙信号转导通路的关键[2, 8]。拟南芥AtIQD1定位于细胞核和微管,与CaM以依赖于Ca2+的方式互作,并与多种蛋白质同时相互作用以整合Ca2+信号防御生物胁迫[9-10],而AtIQD22表达受赤霉素抑制,被DELLA基因诱导,表明AtIQD22参与植物防御反应和生长发育的调节[11]。过表达番茄IQD12 /SUN会导致番茄果实变长,茎和叶穗轴扭曲生长,推测与生长素的分布改变有关,可见IQD12 /SUN参与调节植物的生长发育[12-13],而ZmIQDs和PtIQDs对干旱胁迫起关键作用[14-15]。虽然IQD家族蛋白很早就在水稻中被发现,但它们的具体生物学功能尚未见报道。基因编辑是利用核酸酶(Cas9)对基因组的目标基因进行剪切编辑,从而使目的基因发生突变而改变某一特定性状的技术。利用基因编辑技术准确有效地对水稻基因组进行修饰,对水稻重要性状的改良具有重要意义。CRISPR/Cas9系统是继锌指核酸酶(Zinc Finger Nucleases,ZFN)技术和类转录激活样效应因子核酸酶(Transcription Activator-Like Effector Nucleases,TALEN)技术之后的新一代基因定点编辑技术,利用CRISPR/Cas9技术可对目标基因进行插入或者删除式的基因敲除[16-18]。CRISPR/Cas9编辑系统作为植物遗传改良的有效途径,自2013年以来在水稻、小麦、玉米、番茄、烟草、大豆等农作物中成功实现了对目标基因的定点编辑[19-23]。【本研究切入点】本研究团队前期通过酵母双杂筛选实验发现Pik-H4和OsIQD1存在互作关系,进一步解析水稻OsIQD1的功能对揭示其在水稻生长发育、逆境胁迫调节过程中的作用机制具有重要意义。【拟解决的关键问题】以水稻OsIQD1为目的基因,通过CRISPR/Cas9基因编辑与转基因技术相结合,获得水稻osiqd1突变体材料,以期为后续开展OsIQD1参与的抗稻瘟病具体调控通路的功能解析奠定重要基础。
1 材料与方法 1.1 试验材料供试的水稻材料为粳稻Pik-H4 NIL,是华南农业大学国家植物航天育种工程技术研究中心利用诱变株系H4与LTH构建的含有抗稻瘟病基因Pik-H4的近等基因系。Cas9-gRNA表达盒载体pGTR和pRGEB32购自Addgene公司。编辑靶点引物、核酸测序服务分别由生工生物工程(上海)股份有限公司、北京擎科生物科技有限公司完成。Bsa I内切酶、T4 DNA连接酶、Phanta Super-Fidelity DNA Polymerase高保真酶分别购自NEB(北京)有限公司、南京诺唯赞生物科技股份有限公司。
1.2 靶点设计利用OsIQD1的CDS序列在CRISPR/Cas9在线靶点设计网站(http://skl.scau.edu.cn/ targetdesign/)设计2个特异性靶点。对2个靶点的编码区序列进行扫描分析,以原间隔子邻近序列(Protospacer Adjacent Motifs,PAM)前20个碱基作为候选靶点,以日本晴全基因组为参考序列,选择特异性高的序列〔第1外显子和第5外显子(DUF4005结构域)区域〕为靶位点,设计2个20 bp的编辑靶点,旨在提高无功能蛋白的发生概率从而实现目标基因敲除。为保证OsIQD1靶点序列的一致性,在Pik-H4 NIL中对两个靶点区域序列分别进行PCR扩增、测序,以分析靶点序列是否与日本晴序列一致,所用引物如表 1所示。
1.3 基因编辑载体构建
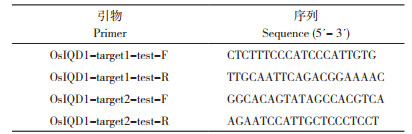
敲除载体构建参考Kabin等[24]的方法。根据分别来自第1外显子、第5外显子的2个靶点设计引物(表 2)。以pGTR载体为模板,分别用3对引物(L5AD5-F和OSIQD1-target1-R、OSIQD1-target1-F和OSIQD1-target2-R、OSIQD1-target2-F和L3AD5-R)扩增相应片段。各片段进行凝胶回收纯化后用Bsa I进行酶切,并用T7连接酶组装3个片段,然后将组装产物用引物(S5AD5-new-F和S3AD5-new-R)再进行一轮PCR扩增。PCR反应体系:2 μL模板DNA、25 μL 2×Phanta Max Buffer、1 μL dNTP、1 μL Phanta Max Super-Fidelity DNA Polymerase,S5AD5-new-F和S3AD5-new-R各2 μL,17 μL ddH2O。PCR反应程序:95℃预变性5 min;95℃变性15 s、57℃退火15 s、72℃延伸15 s,35个循环;72 ℃终延伸5 min。PCR产物纯化后用Fok I进行酶切,最终得到gRNA-靶点1-tRNA-gRNA-靶点2-tRNA片段。将Cas9蛋白表达载体pRGEB32用Bsa I进行酶切,利用T4连接酶将带有靶点的纯合片段与酶切载体进行连接重组,将重组载体转化大肠杆菌DH5α后进行测序分析,阳性转化子转化农杆菌EHA105感受态,并侵染转化Pik-H4 NIL愈伤组织,经2次筛选、分化、生根,将转基因幼苗进行种植。用CTAB法提取T0代转基因植株叶片DNA,用抗潮霉素基因(HPT II)特异引物(HPT-F:5'-ATTTGTGTACGCCCGACAGT-3'和HPT-R:5'-GTGCTTGACATTGGGGAGTT-3')进行PCR检测以确定转基因阳性植株。
1.4 基因编辑株系的靶点分析
为检测OsIQD1基因靶点的突变情况,利用引物OsIQD1-target1-test-F、OsIQD1-target1-test-R、OsIQD1-target2-test-F、OsIQD1-target2-test-R(表 1)分别对转基因阳性植株的OsIQD1基因2个靶点区域的DNA序列进行PCR扩增。PCR反应体系:1 μL模板DNA、12.5 μL 2×Taq Plus Master Mix,引物各1 μL,9.5 μL ddH2O。PCR反应程序:95℃预变性5 min;95℃变性15 s、57℃退火15 s、72℃延伸1 min,27个循环;72 ℃终延伸5 min。获得PCR扩增产物分别为280、357 bp,将靶点区域位置的扩增片段进行测序,并与野生型区域序列进行比对分析,鉴定突变植株的基因变异类型。
1.5 OsIQD1敲除植株的病程相关基因表达量分析基于前期研究已知Pik-H4与OsIQD1存在互作关系,Pik-H4又受稻瘟病菌诱导,因此进一步检测野生型和突变体中的病程相关(Pathogenesis-related, PR)基因表达量。PR1a和PR1b是最早报道的水稻PR1基因家族成员[25-26],其编码的蛋白质剩余类丝氨酸羧肽酶(Serine Carboxypeptidase-Like Proteins, SCPLs)[27]。剪取野生型和突变体植株的叶片,检测稻瘟病菌病程相关基因的表达量,引物序列如表 3所示。
|
2 结果与分析 2.1 水稻IQD1同源蛋白分析
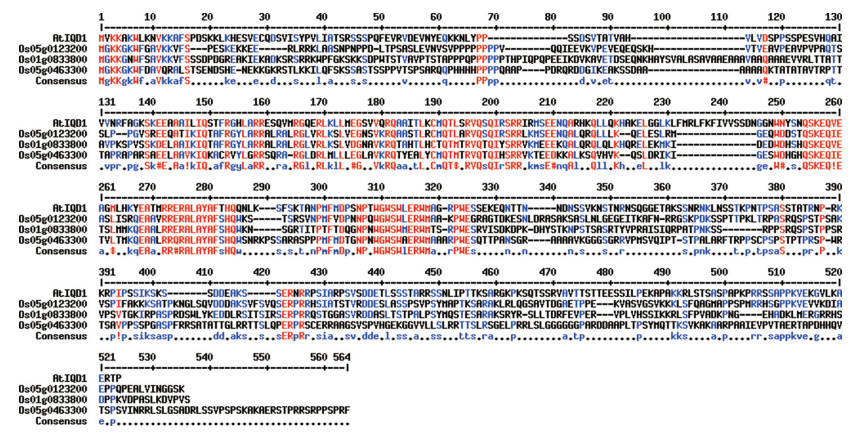
为获得水稻IQD1的候选蛋白,用AtIQD1蛋白序列在美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)数据库进行同源搜索,结果(图 1)表明,水稻中存在3个与AtIQD1同源性较高的候选蛋白,分别为Os01g0833800(41.67%)、Os05g0123200(44.27%)和Os05g0463300(46.77%),Os01g0833800基因包含7个外显子,CDS全长1 503 bp,编码501个氨基酸,蛋白质结构C端有1个DUF4005结构域。Os05g0463300基因包含5个外显子,CDS全长1 617 bp,编码539个氨基酸,蛋白质结构C端含有1个IQ基序。Os05g0123200含有6个外显子,CDS全长1 425 bp,编码475个氨基酸,蛋白质结构的N端含有1个IQ基序,C端含有1个DUF4005结构域。已知IQ基序(IQxxxRGxxxR)主要是少数几个CaM与CaMBPs结合的结构域之一[28],而DUF4005结构域是一种由59个氨基酸组成的肽,在体外直接与微管结合[29]。已知OsIQD1的TIGR Rice Genome Project ID为Os05m00240[2],且根据比对氨基酸序列结果推测,Os05g0123200为AtIQD1的同源基因OsIQD1。
|
| 图 1 OsIQD1与AtIQD1同源性分析 Fig. 1 Homology analysis of OsIQD1 and AtIQD1 |
2.2 pRGEB32-OsIQD1 -gRNA双元载体构建
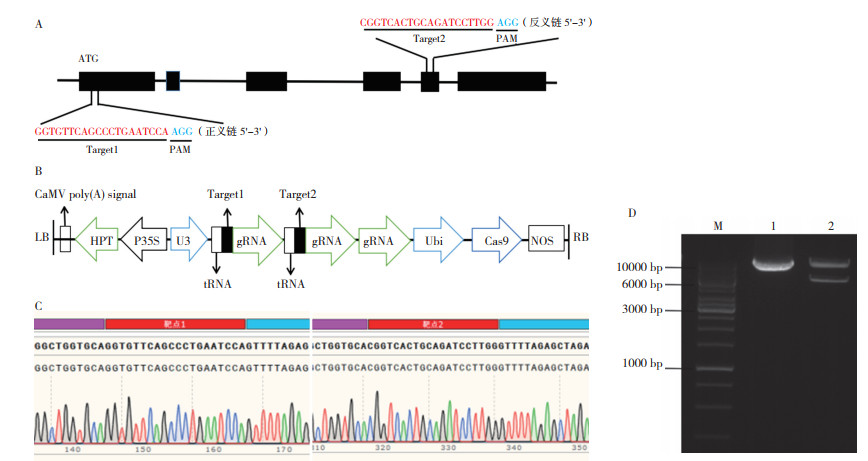
为获得OsIQD1定点编辑突变体,在OsIQD1基因第1外显子、第5外显子各设计1个靶点,将组装好的纯合片段连接到pRGEB32载体骨架中,得到pRGEB32-OsIQD1 -gRNA双元载体(图 2 A、B)。利用OsU3启动子测序引物(pRGEB32-CX-F和pRGEB32-CX-R)对构建载体上的靶点进行测序,结果如图 2 C所示,2个靶点序列与所设计靶点序列一致。抽提相应的质粒DNA,利用Nhe Ⅰ和Hin d Ⅲ进行酶切鉴定,获得大小约为5.57 kb和10.69 kb的目的片段(图 2 D),表明OsIQD1的2个靶点正确组装到pRGEB32载体上。本研究构建的载体适用于后续由农杆菌介导的水稻遗传转化中,为获得OsIQD1水稻突变体奠定基础。
|
| A:OsIQD1的2个gRNA靶点位置;B:pRGEB32-OsIQD1-gRNA表达载体组装示意图;C:pRGEB32-OsIQD1-gRNA表达载体中2个靶点序列的测序结果;D:Nhe Ⅰ和Hin d Ⅲ酶切鉴定pRGEB32-OsIQD1-gRNA载体,M为DNA Marker,1为pRGEB32-OsIQD1 -gRNA载体质粒,2为Nhe Ⅰ和Hin d Ⅲ酶切pRGEB32-OsIQD1 -gRNA载体质粒产物 A: Position of two gRNA targets in the OsIQD1 gene locus; B: Assembly diagram of pRGEB32-OsIQD1-gRNA expression vector; C: Sequencing results for the two target sequences inserted in pRGEB32-OsIQD1-gRNA recombinant vector; D: Identification of the pRGEB32-OsIQD1-gRNA plasmid digested with Nhe Ⅰ and Hin d Ⅲ; M represents maker, lane 1 represents pRGEB32-OsIQD1 -gRNA vector plasmid and lane 2 represents the product of pRGEB32-OsIQD1 -gRNA vector plasmid digested by Nhe Ⅰ and Hin d Ⅲ 图 2 gRNA靶位点、pRGEB32-OsIQD 1 -gRNA表达载体组装示意图及阳性克隆鉴定结果 Fig. 2 gRNA target sites, assembly diagram of pRGEB32-OsIQD1 -gRNA expression vector and positive clone identification results |
2.3 转基因植株鉴定
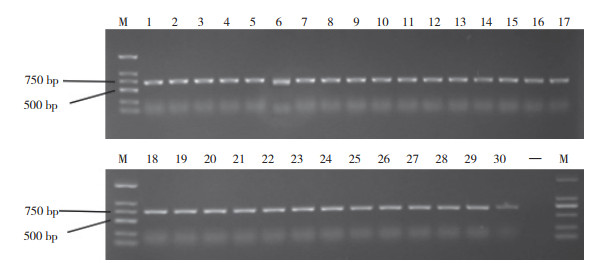
利用农杆菌介导法将pRGEB32-OsIQD1 -gRNA表达载体转入水稻材料Pik-H4 NIL愈伤组织中,获得30株转基因植株。用筛选标记抗潮霉素基因HPT II特异引物(HPT-F和HPT-R)对转基因植株进行PCR鉴定,以载体质粒为模板作为阳性对照,野生型日本晴DNA为模板作为阴性对照。结果(图 3)表明,本研究成功获得30株阳性转基因植株。
|
| M为marker;1~30代表OsIQD1对应的第1~30株转基因植株; “-”代表以ddH2O为模板的阴性对照 M represents marker, lane 1-30 represent the 1st-30th transgenic plants corresponding to OsIQD1; "-" represents negative control with ddH2O as template 图 3 转基因株系抗潮霉素基因HPT II的PCR鉴定 Fig. 3 Identification of hygromycin resistance gene HPT II of transgenic line by PCR |
2.4 突变类型分析
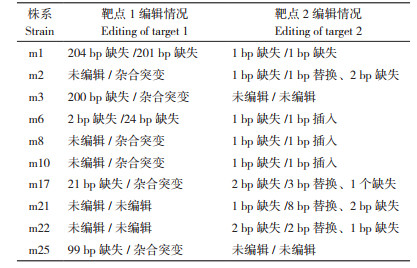
为明确OsIQD1基因靶点是否发生定点编辑,进一步对经潮霉素检测鉴定为转基因阳性植株的突变位点进行分析。由于OsIQD1基因的2个靶点在序列上相距2 587 bp,利用两对引物(表 1)扩增各靶位点区域DNA片段。以获得的转基因阳性植株基因组DNA为模板,用相应的引物对靶位点序列进行PCR扩增并测序。以野生型序列作为参考,分析比较OsIQD1转基因株系中各靶位点附近序列的测序峰图,并利用解码网站(http://skl.scau.edu.cn/dsdecode/)对全部转基因植株进行测序分析。结果(表 4)表明,30个转基因阳性株系中,有10个株系的OsIQD1基因发生了定点编辑现象,其中有6个株系(m1、m2、m6、m8、m10和m17)的两个靶点区域均发生了编辑;有8个株系(m1、m2、m3、m6、m8、m10、m17和m25)的靶点1被编辑,共有13个编辑事件,编辑效率为21.67%,如m1中存在大片段缺失导致蛋白翻译提前终止,而m6中一条染色体存在2 bp缺失引起移码而导致翻译提前终止(图 4A、B);有8个株系(m1、m2、m6、m8、m10、m17、m21和m22)的靶点2被编辑,共有16个编辑事件,编辑效率为26.67%,如m1已经是纯合突变,两条染色体中均有1 bp缺失导致翻译提前终止,而m6中一条染色体也有1 bp缺失引起移码导致翻译提前终止(图 4C、D)。综上分析可推测,部分突变株系(如m1)在T0代时目的基因的功能就丧失,而杂合突变需要在T1、T2代继续测序比较分析。
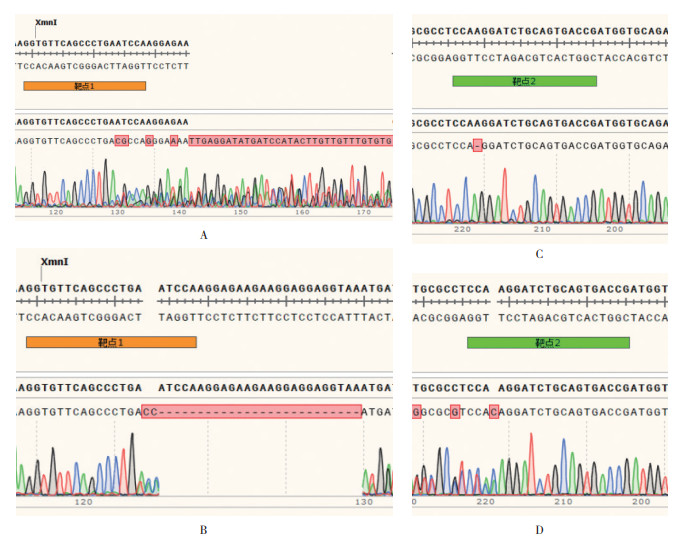
|
| A、B: 分别为m1、m6靶点1测序结果;C、D: 分别为m1、m6靶点2测序结果 A, B: Sequencing results of target 1 in m1 and m6; C, D: Sequencing results of target 2 in m1 and m6 图 4 部分转基因株系中两个靶点的测序结果比对 Fig. 4 Sequencing comparison of two targets in some transgenic lines |
本试验收获部分T0代株系的种子进行扩繁得到T1代植株,经检测发现3种突变类型:(1)敲除靶点1存在2 bp缺失,敲除靶点2存在1 bp缺失,并且测序峰图并未出现严重的双峰现象,表明osiqd1-1为纯合株系;(2)敲除靶点1存在一条链缺失1 bp、另一条链缺失两个大片段的现象,敲除靶点2存在1 bp插入、测序峰图靶点1有双峰现象,靶点2并未出现严重的双峰现象,表明osiqd1-2尚未完全纯合,但仍可作为后续试验材料;(3)敲除靶点1存在1 bp插入,敲除靶点2存在1 bp替换且1 bp插入,并且测序峰图并未出现严重的双峰现象,表明osiqd1-3为纯合株系(图 5A、B、C)。通过荧光定量PCR检测osiqd1-1、osiqd1-2、osiqd1-3等3个独立株系的OsIQD1基因表达量,结果(图 5D)显示,这些突变株系的基因表达量显著低于野生型的表达水平。本研究获得了具有稳定遗传的CRISPR/Cas9敲除OsIQD1的转基因植株。
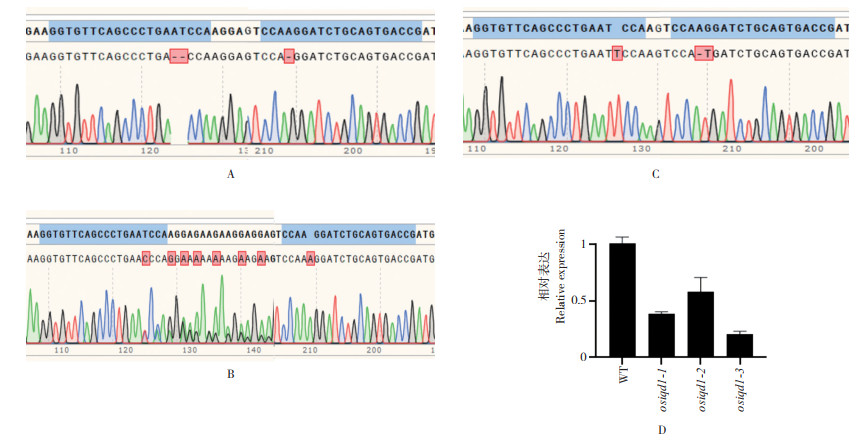
|
| A~C:osiqd1转基因T1代植株靶点检测结果;D:osiqd1-1、osiqd1-2、osiqd1-3植株的OsIQD1相对表达水平 A-C: Target detection result of osiqd1 transgenic plant (T1 generation); D: Relative expression levels of OsIQD1 in osiqd1-1, osiqd1-2 and osiqd1-3 plants 图 5 OsIQD1的CRISPR/Cas9敲除植株验证结果 Fig. 5 Verification of OsIQD1 knock-outed plants via CRISPR/Cas9 |
2.5 OsIQD1敲除植株的病程相关基因表达量分析
病程相关基因一般作为免疫响应的标记基因。本研究检测了野生型和突变体中稻瘟病菌病程相关基因PR1a和PR1b的表达量,结果(图 6)显示,野生型植株PR1a和PR1b基因表达量相较于osiqd1突变体均上调,说明OsIQD1参与植物的免疫反应过程。
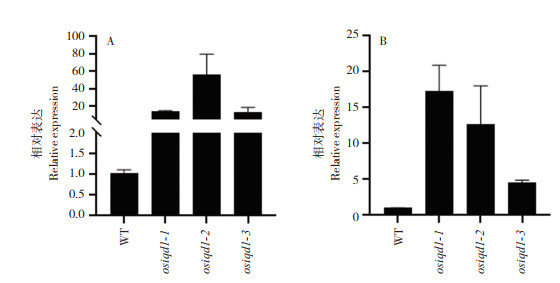
|
| 图 6 病程相关基因PR1 a(A)、PR1 b(B) 在野生型WT和osiqd1-1、osiqd1-2、osiqd1-3植株中的相对表达水平 Fig. 6 Relative expression levels of disease-course related genes PR1a and PR1b in WT and osiqd1-1, osiqd1-2 and osiqd1-3 plants |
2.6 OsIQD1转基因T1代植株的获得
经前期鉴定获得OsIQD1转基因3种突变类型植株(图 7),与野生型Pik-H4 NIL相比,OsIQD1转基因T1代幼苗植株生长相对一致,但分蘖数增多。敲除OSIQD1后对水稻抗病的影响需要进一步检测。

|
| 图 7 osiqd1-1、osiqd1-2、osiqd1-3与野生型WT幼苗植株形态特征比对 Fig. 7 Morphological characteristics of osiqd1-1, osiqd1-2 and osiqd1-3 compared to the WT |
3 讨论
水稻作为全球主要的粮食作物之一,养育世界一半以上的人口。随着人口持续增长、耕地面积减少、极端环境频发等问题的出现,粮食产量和安全引起了国家重点关注[30]。解决当前粮食短缺的有效措施之一是在现有耕地的基础上,突破传统育种手段瓶颈,借助分子手段,实现精准、高效的分子设计育种,提高水稻产量[31]。但传统育种手段需要经过多代回交筛选,育种工作量巨大,周期较长,效率低下,且容易受连锁基因影响。而CRISPR/Cas9是一种高效且简便的基因定点编辑技术,能快速获得目标性状改善并能稳定遗传的纯合突变植株,极大地缩短了育种周期[32-33]。
目前已经报道的水稻IQD基因有29个,但只有OsIQD14得到进一步研究,而其他家族成员未得到深入研究。OsIQD14在水稻籽壳细胞中高度表达,调节微管细胞骨架动力学以控制稻粒大小。除了定位到细胞核和细胞质外,OsIQD14还沿着微管分布。当OsIQD14耗尽时,谷物变得更宽更短,作物产量增加;当OsIQD14过表达时,晶粒变得更长更窄,不会影响总产量[34]。本研究利用CRISPR/Cas9技术,通过双靶点编辑方式,对水稻OsIQD1进行定点突变,相对于单靶点编辑得到较多单一碱基突变,极大提高了对OsIQD1的编辑效率。其中target1位点的编辑效率为21.67%,target2位点的编辑效率为26.67%,表明同一基因的不同靶位点会影响编辑效率,与已报道的细胞质分裂关键基因OsMAP65-3一致,载体的编辑效率与不同的靶序列相关性较大[35]。因此,为确保高效编辑,对目的基因编辑时应选用2个以上的靶位点。
本研究T0代植株突变种类较为丰富,极大提高了OsIQD1变异的多样性,但鉴于CRISPR/Cas9编辑植株的T0代转基因株系会产生纯合突变、杂合突变、双等位突变,以及未发生突变等4种基因型植株,纯合突变株系比例相对较少,要获得较多的变异类型则需要对杂合突变、双等位突变等转基因植株后代的分离情况进行分析与鉴定。目前本研究利用CRISPR/Cas9基因编辑技术对水稻钙调蛋白结合蛋白基因OsIQD1编码区序列进行定点编辑,获得了能够稳定遗传的种质材料,为后续开展水稻抗稻瘟病育种奠定了基础。
研究发现,过表达AtIQD1可以使拟南芥显著增强对坏死型真菌Botrytis cinerea的抗性,敲除AtIQD1则表现感病,下调一些与植物生物防御相关基因的表达[36]。水稻OsIQD1是否具有与AtIQD1类似的功能仍有待进一步研究。本研究通过创制水稻OsIQD1突变体,旨在研究IQD1在参与水稻抗病具体调控通路中的作用机理。病程相关蛋白质是植物受病原物胁迫后诱导产生并积累的一类蛋白质,是植物防卫体系的重要组成部分,它们往往在植物抗病反应的下游发挥作用,在很多情况下是直接限制病原物生长的主要因素。最早报道的水稻PR1基因家族成员PR1a和PR1b,受稻瘟病菌(Magnaporthe oryzae)、水杨酸(Salicylic Acid, SA)、茉莉酸(Jasmonic Acid, JA)等诱导,对光、伤害、磷酸酶抑制剂等胁迫和化学处理作出反应[25-26]。本研究结果表明,与野生型Pik-H4 NIL相比,PR1a和PR1b在osiqd1突变体植株中的表达量明显升高,初步推测OsIQD1参与植物的基础免疫反应过程。
4 结论鉴于CRISPR/Cas9基因编辑可特异性敲除目标DNA序列,本研究利用该技术,设计了分别靶向起始密码子(ATG)下游与DUF4005结构域的两个靶点,对OsIQD1基因进行定点编辑,并获得了osiqd1纯合突变体材料。与野生型Pik-H4 NIL相比,osiqd1突变体在幼苗时期长势大致相同,但分蘖增多;同时,PR1a和PR1b在osiqd1突变体中表达量明显升高,初步推测OsIQD1参与到植物的基础免疫过程中,为水稻抗稻瘟病的研究提供了丰富的遗传材料,也为水稻的抗病育种奠定了重要基础。
| [1] |
ABEL S, BURSTENBINDER K, MULLER J. The emerging function of IQD proteins as scaffolds in cellular signaling and trafficking[J]. Plant Signaling & Behavior, 2013, 8(6): e24369. DOI:10.4161/psb.24369 |
| [2] |
ABEL S, SAVCHENKO T, LEVY M. Genome-wide comparative analysis of the IQD gene families in Arabidopsis thaliana and Oryza sativa[J]. BMC Evolutionary Biology, 2005(5): 72. DOI:10.1186/1471-2148-5-72 |
| [3] |
GUO C, ZHOU J, LI D. New insights into functions of IQ 67-domain proteins[J]. Frontiers in Plant Science, 2020(11): 614851. DOI:10.3389/fpls.2020.614851 |
| [4] |
STEINHORST L, KUDLA J. Calcium -a central regulator of pollen germination and tube growth[J]. Biochimica et Biophysica Acta, 2013(7): 1573-1581. DOI:10.1016/j.bbamcr.2012.10.009 |
| [5] |
BI L, WENG L, JIANG Z, XIAO H. The tomato IQD gene SUN24 regulates seed germination through ABA signaling pathway[J]. Planta An International Journal of Plant Biology, 2018(4): 919-931. DOI:10.1007/s00425-018-2950-6 |
| [6] |
KONG Z, IOKI M, BRAYBROOK S, LI S, YE Z H, JULIE LEE Y R, HOTTA T, CHANG A, TIAN J, WANG G D, LIU B. Kinesin-4 functions in vesicular transport on cortical microtubules and regulates cell wall mechanics during cell elongation in plants[J]. Molecular Plant, 2015(8): 1011-1023. DOI:10.1016/j.molp.2015.01.004 |
| [7] |
FENG L, CHEN Z, MA H, CHEN X, LI Y, WANG Y, XIANG Y. The IQD gene family in soybean: structure, phylogeny, evolution and expression[J]. PLoS ONE, 2014(9): e110896. DOI:10.1371/journal.pone.0110896 |
| [8] |
VAN DER KNAAP E, CHAKRABARTI M, CHU Y H, CLEVENGER J P, ILLA-BERENGUER E, HUANG Z, KEYHANINEJAD N, MU Q, SUN L, WANG Y, WU S. What lies beyond the eye: the molecular mechanisms regulating tomato fruit weight and shape[J]. Frontiers in Plant Science, 2014(5): 227. DOI:10.3389/fpls.2014.00227 |
| [9] |
LEVY M, WANG Q, KASPI R, PARRELLA M P, ABEL S. Arabidopsis IQD1, a novel calmodulin-binding nuclear protein, stimulates glucosinolate accumulation and plant defense[J]. The Plant Journal, 2005, 43: 79-96. DOI:10.1111/j.1365-313X.2005.02435.x |
| [10] |
BURSTENBINDER K, SAVCHENKO T, MULLER J, ADAMSON A W, STAMM G, KWONG R, ZIPP B J, DINESH D C, ABEL S. Arabidopsis calmodulin-binding protein IQ67-domain 1 localizes to microtubules and interacts with kinesin light chain-related protein-1[J]. Journal of Biological Chemistry, 2013(3): 1871-1882. DOI:10.1074/jbc.M112.396200 |
| [11] |
ZENTELLA R, ZHANG Z L, PARK M. Global analysis of DELLA direct targets in early gibberellin signaling in Arabidopsis[J]. Plant Cell, 2007(19): 3037-3057. DOI:10.1105/tpc.107.054999 |
| [12] |
XIAO H, JIANG N, SCHAFFNER E, STOCKINGER E J, VAN DER KNAAP E. A retrotransposon-mediated gene duplication underlies morphological variation of tomato fruit[J]. Science, 2008, 319: 1527-1530. DOI:10.1126/science.1153040 |
| [13] |
WU S, XIAO H, CABRERA A, MEULIA T, VAN DER KNAAP E. SUN regulates vegetative and reproductive organ shape by changing cell division patterns[J]. Plant Physiology, 2011, 157: 1175-1186. DOI:10.1104/pp.111.181065 |
| [14] |
MA H, FENG L, CHEN Z, CHEN X, ZHAO H, XIANG Y. Genome-wide identification and expression analysis of the IQD gene family in Populus trichocarpa[J]. Plant Science, 2014, 229: 96-110. DOI:10.1016/j.plantsci.2014.08.017 |
| [15] |
CAI R, ZHANG C, ZHAO Y, ZHU K, WANG Y, JIANG H, XIANG Y, CHENG B. Genome-wide analysis of the IQD gene family in maize[J]. Molecular Genetics and Genomics, 2016, 291: 543-558. DOI:10.1007/s00438-015-1122-7 |
| [16] |
GA J T, GERSBACH C A, BARBAS C F. ZFN, TALEN, and CR I SPR/Cas-based methods for genome eng ineer ing[J]. Trends in Biotechnology, 2013, 31(7): 397-405. DOI:10.1016/j.tibtech.2013.04.004 |
| [17] |
HAURWITZ R E, JINEK M, WIEDENHEFT B, ZHOU K, DOUDNA J A. Sequence-and structure-specific RNA processing by a CRISPR endonuclease[J]. Science, 2010, 329: 1355-1358. DOI:10.1126/science.1192272 |
| [18] |
WYMAN C, KANAAR R. DNA double-strand break repair: All's well that ends well[J]. Annual Review of Genetics, 2006, 40: 363-383. DOI:10.1146/annurev.genet.40.110405.090451 |
| [19] |
SHAN Q W, WANG Y P, LI J, GAO C X. Genome editing in rice and wheat using the CRISPR/Cas system[J]. Nature Protocols, 2014, 9(10): 2395-2410. DOI:10.1038/nprot.2014.157 |
| [20] |
LI J, ZHANG H W, SI X M, TIAN Y H, CHEN K L, LIU J X, CHEN H B, GAO C X. Generation of thermosensitive male-sterile maize by targeted knockout of the ZmTMSS gene[J]. Journal of Genetics and Genomics, 2017, 44(9): 465-468. DOI:10.1016/j.jgg.2017.02.002 |
| [21] |
XIE K B, YANG Y N. RNA-Guided Genome editing in plants using a CRISPR-Cas System[J]. Molecular Plant, 2013(6): 1975-1983. DOI:10.1093/mp/sst119 |
| [22] |
NEKRASOV V, STASKAWICZ B, WEIGEL D, JONES J D, KAMOUN S. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease[J]. Nature Biotechnology, 2013, 31: 691-693. DOI:10.1038/nbt.2655 |
| [23] |
LI J F, NORVILLE J, AACH J, MCCORMACK M, ZHANG D, BUSH J, CHURCH G M, SHEEN J. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nature Biotechnology, 2013(8): 688-691. DOI:10.1038/nbt.2654 |
| [24] |
KABIN X, BASTIAN M, YINONG Y. Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(11): 3570-3575. DOI:10.1073/pnas.1420294112 |
| [25] |
GANESH K A, NAM-SOO J, RANDEEP R. A Novel Rice (Oryza sativa L.) Acidic PR1 gene highly responsive to cut, phytohormones, and protein phosphatase inhibitors[J]. Biochemical and Biophysical Research Communications, 2000, 274: 157-165. DOI:10.1006/bbrc.2000.3114 |
| [26] |
GANESH K A, RANDEEP R, NAM-SOO J. Rice (Oryza sativa L.) OsPR1b gene is phytohormonally regulated in close interaction with light signals[J]. Biochemical and Biophysical Research Communications, 2000, 278: 290-298. DOI:10.1006/bbrc.2000.3781 |
| [27] |
窦世娟, 关明俐, 李莉云, 刘国振. 水稻的病程相关基因[J]. 科学通报, 2014, 59(3): 245-258. DOI:10.1360/972012-1831 DOU S J, GUAN M L, LI L Y, LIU G Z. Pathogenesis-related genes in rice[J]. Chinese Science Bulletin, 2014, 59(3): 245-258. DOI:10.1360/972012-1831 |
| [28] |
田长恩, 周玉萍. 植物具IQ基序的钙调素结合蛋白的研究进展[J]. 植物学报, 2013, 48(4): 447-460. DOI:10.3724/SP.J.1259.2013.00447 TIAN C E, ZHOU Y P. Research progress in plant IQ Motif-containing Calmodulin-binding proteins[J]. Chinese Bulletin of Botany, 2013, 48(4): 447-460. DOI:10.3724/SP.J.1259.2013.00447 |
| [29] |
LI Y, HUANG Y, WEN Y, WANG D, LIU H, LI Y, ZHAO J, AN L, YU F, LIU X. The domain of unknown function 4005 (DUF4005) in an Arabidopsis IQD protein functions in microtubule binding[J]. Journal of Biological Chemistry, 2021, 297: 100849. DOI:10.1016/j.jbc.2021.100849 |
| [30] |
郭韬, 余泓, 邱杰, 李家洋, 韩斌, 林鸿宣. 中国水稻遗传学研究进展与分子设计育种[J]. 中国科学: 生命科学, 2019, 49(10): 1185-1212. DOI:10.1360/SSV-2019-0209 GUO T, YU H, QIU J, LI J Y, HAN B, LIN H X. Advances in rice genetics and breeding by molecular design in China[J]. Scientia Sinica(Vitae), 2019, 49(10): 1185-1212. DOI:10.1360/SSV-2019-0209 |
| [31] |
凌启鸿, 张洪程, 丁艳锋, 张益彬. 水稻高产技术的新发展: 精确定量栽培[J]. 中国稻米, 2005(1): 3-7. LING Q H, ZHANG H C, DING Y F, ZHANG Y B. New development of high yield technique in ric: precise quantitative cultivation[J]. China Rice, 2005(1): 3-7. |
| [32] |
黄忠明, 周延彪, 唐晓丹, 赵新辉, 周在为, 符星学, 王凯, 史江伟, 李艳锋, 符辰建, 杨远柱. 基于CRISPR/Cas9技术的水稻温敏不育基因tms5突变体的构建[J]. 作物学报, 2018, 44(6): 844-851. DOI:10.3724/SP.J.1006.2018.00844 HUANG Z M, ZHOU Y B, TANG X D, ZHAO X H, ZHOU Z W, FU X X, WANG K, SHI J W, LI Y F, FU C J, YANG Y Z. Construction of tms5 mutants in rice based on CRISPR/Cas9 technology[J]. Acta Agronomica Sinica, 2018, 44(6): 844-851. DOI:10.3724/SP.J.1006.2018.00844 |
| [33] |
季新, 李飞, 晏云, 孙红正, 张静, 李俊周, 彭廷, 杜彦修, 赵全志. 基于CRISPR/Cas9系统的水稻光敏色素互作因子OsPIL15基因编辑[J]. 中国农业科学, 2017, 50(15): 2861-2871. DOI:10.3864/j.issn.0578-1752.2017.15.002 JI X, LI F, YAN Y, SUN H Z, ZHANG J, LI J Z, PENG T, DU Y X, ZHAO Q Z. CRISPR/Cas9 System-Based Editing of phytochrome-interacting factor OsPIL15[J]. Scientia Agricultura Sinica, 2017, 50(15): 2861-2871. DOI:10.3864/j.issn.0578-1752.2017.15.002 |
| [34] |
YANG B, WENDRICH J R, DE RYBEL B, WEIJERS D, XUE H W. Rice microtubule-associated protein IQ67-DOMAIN14 regulates grain shape by modulating microtubule cytoskeleton dynamics[J]. Plant Biotechnology Journal, 2020, 18(5): 1141-1152. DOI:10.1111/pbi.13279 |
| [35] |
林小丽, 姜志树, 贺榕, 胡嘉敏, 朱昌兰, 周大虎, 贺浩华, 徐杰. 基于CRISPR/Cas9技术对水稻细胞质分裂关键基因OsMAP65-3s的编辑[J]. 核农学报, 2022, 36(4): 716-727. DOI:10.11869/j.issn.100-8551.2022.04.0716 LIN X L, JIANG Z S, HE R, HU J M, ZHU C L, ZHOU D H, HE H H, XU J. Editing of OsMAP65-3s, a key gene for cytoplasmic division in rice, based on CRISPR/Cas9 technology[J]. Journal of Nuclear Agricultural Sciences, 2022, 36(4): 716-727. DOI:10.11869/j.issn.100-8551.2022.04.0716 |
| [36] |
BARDA O, LEVY M. IQD1 involvement in hormonal signaling and general defense responses against Botrytis cinerea[J]. Frontiers in Plant Science, 2022, 13: 845140. DOI:10.3389/fpls.2022.845140 |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49