文章信息
基金项目
- 广东省科技计划项目(2019B030316006)
作者简介
-
吴琳华,2021年获中国农业大学作物遗传育种专业博士学位,研究生在读期间参与国家自然科学基金项目2项。2021年7月进入广东省农业科学院水稻研究所从事博士后研究工作,主要研究方向为水稻穗型相关基因的克隆与鉴定,水稻穗型调控的分子机理,稻种资源的收集保存及种质创新和利用。参与广东省自然科学基金项目1项、广东省科技计划项目(水稻种质资源的收集、保存、鉴定评鉴与分发利用)1项
吴琳华(1990—),女,博士,研究方向为水稻穗型调控的分子机理,E-mail:wulinhua361@sina.com.
通讯作者
- 陈文丰(1966—),男,研究员,研究方向为水稻种质资源,E-mail:18-cwf@163.com.
文章历史
- 收稿日期:2022-07-19
粮食安全是维持社会稳定,保证国家安全的重要基础,但是当今全球范围内粮食安全生产面临巨大危机,粮食短缺问题日益加剧。随着人类社会工业化水平的提高,城市不断发展,环境污染愈发严重,可用耕地面积逐年减少,极端气候频发,粮食安全生产受到严重威胁。近年来,国际冲突加剧,新冠疫情大流行也严重冲击着粮食系统的稳定性。世界人口持续增长,预测到2050年,全球人口将接近100亿。全球粮食总产需要提高1倍,才能满足巨大的人口需求[1]。水稻是世界上第二大粮食作物,为超过35亿人口提供口粮,水稻稳产高产对于保证粮食安全解决人口饥饿问题至关重要[2]。全球范围内,每年因气候变化造成的粮食产量波动在32%~39% 之间,水稻的总产也因此造成的产量波动在每年300万t左右[3]。因此培育高产、优质、适应性强的水稻品种势在必行。
从20世纪中叶开始,从矮化育种到杂交育种,水稻单产经历了两次大飞跃,但自此以后水稻单产却没有新的重大突破。传统的育种方法主要依赖于优良农艺性状的确定和杂交,而水稻栽培种的遗传背景狭窄,大多数栽培种之间具有相似的遗传来源,因此拓宽遗传背景,挖掘和利用优异的遗传资源,才能打破这种困境。近来,科学家提出了“绿色超级稻”(Green Super Rice, GSR)的概念,旨在减少投入,环境友好,产量提高。新株型或者理想株型是提高作物产量潜力的一种比较好的方式,通过组合更好的抗倒伏性状提高资源的利用效率,国际水稻研究中心和中国的科学家将理想株型作为提高水稻产量的一种行之有效的方式。高效的管理与应用已经存在于栽培稻品种和野生近缘种之间的遗传变异是实现这一目标的重要途径,IPA1作为经典的控制水稻理想株型的基因,在水稻育种中发挥了重要作用,甚至被认为是新的“绿色革命基因” [2, 4-7]。拓宽栽培稻的遗传背景,打破栽培种资源狭窄的瓶颈,通过分子设计育种组合有利基因,能更好地对现有品种进行遗传改良或者培育超高产品种。水稻穗型是株型的重要组成部分,也是驯化和遗传改良的重要目标之一,因此研究穗发育过程的调控机制对于分子设计育种,提高水稻产量具有重要意义。
在经历了6 500万年前的生物大灭绝后,动物进入了“哺乳动物时期”,同时植物也进入了“禾本科植物时期”。在超过2 000万年的进化压力下,禾本科植物已形成了强大的适应能力,尤其是禾本科植物的花序结构,取得了巨大的生殖成功,这也为后期人类长期依赖禾本科植物得以生存繁衍提供了能量来源。禾本科植物的花序主要有圆锥花序(Panicle,复总状花序)和穗状花序(Spike,单总状花序)两种类型,水稻和小麦分别拥有这两种典型的花序形态。小穗发育是禾本科植物特有的生殖结构,它们直接着生在花序轴上或者着生在其他花序分枝上(图 1)[8]。

|
| 图 1 水稻花序发育模式 Fig. 1 Model of rice inflorescence development |
水稻生长过程分为营养生长阶段、生殖生长阶段以及成熟期3个阶段[9]。稻穗的发育发生在生殖生长阶段,受环境和遗传背景等多重因素调控。水稻产量很大程度上依赖花的数目,而花的数目直接受到花序结构的调控。水稻的花序形态取决于分枝模式和花的位置。水稻圆锥花序的花序轴(Rachis)上形成一系列的分枝(Branch),在分枝上附着小穗(Spikelet),每个小穗包含1个可育小花(Floret),但是花序轴顶端不能形成小花,只留下一个退化的痕迹。本文从水稻花序的形成开始到花序抽出的整个生殖生长阶段,以及驯化和改良等对花序的形态建成影响因素进行综述。
1 成花素蛋白起始SAM向花序分生组织转变水稻是一种典型的短日作物,表现出强烈的光周期敏感性,在短日照诱导条件下,成花素在叶片中合成,转运到顶端分生组织(Shoot apical meristem,SAM),促进顶端分生组织转变为花序分生组织(Inflorescence meristem,IM)。水稻中有两个成花素基因Hd3a和RFT1,它们编码的成花素蛋白可以作为开花移动信号,促进水稻开花。水稻中存在Ehd1和Hd1两条开花途径,Ehd1编码一个B类型的响应调控因子,在无Hd1的短日照条件下促进开花,而无论日照长短,该基因都能上调成花素基因的表达;而在Ehd1突变体背景下,Hd3a完全不表达,水稻抽穗完全依赖低水平表达的RFT1;当RFT1功能丧失后,在Ehd1背景下,无论是短日照还是长日照条件下都不能诱导花序转化。在长日照和短日照条件下,Hd1分别促进和抑制水稻开花,是栽培稻开花的另一个重要决定基因,在Ehd1功能缺失的长日照条件下,Hd1强烈抑制开花[10-15]。
2 水稻花序分生组织向其他分生组织转变水稻花序发育受到环境因素和众多基因的精细调控,形成一个复杂的调控网络,任何一个发育节点受到影响都会导致穗型发生变化。Ikeda等[16]、Suzaki等[17]根据发育进程,将水稻花序发育大致分成9个时期(Staging of inflorescence development,Stage In1-9):(1)从SAM转变为花序轴分生组织(Rachis meristem)并形成第一个苞叶原基;(2)形成第二、第三苞叶原基和第一个一次枝梗原基,并且花序轴分生组织达到最大值;(3)形成的一次枝梗(Primary branch)原基按空间顺序排列,且花序轴分生组织失去活性;(4)虽然起始的时间不同,但所有的一次枝梗同向伸长;(5)一次枝梗上产生二次枝梗(Secondary branch),但是并不能产生小穗(Spikelet),并且最上部的茎节开始伸长;(6)一次枝梗和二次枝梗分生组织转变成末端小穗分生组织并形成最初的颖苞;(7)在颖苞形成后,花器官(Floral organ)开始分化,形成2个浆片、6个雄蕊和1个心皮,但花序轴仍然不伸长;(8)分生组织失去活性,花序轴和枝梗快速伸长,花药和胚珠发育完全;(9)花序从剑叶的叶鞘中伸出,完成抽穗和开花[16-19]。
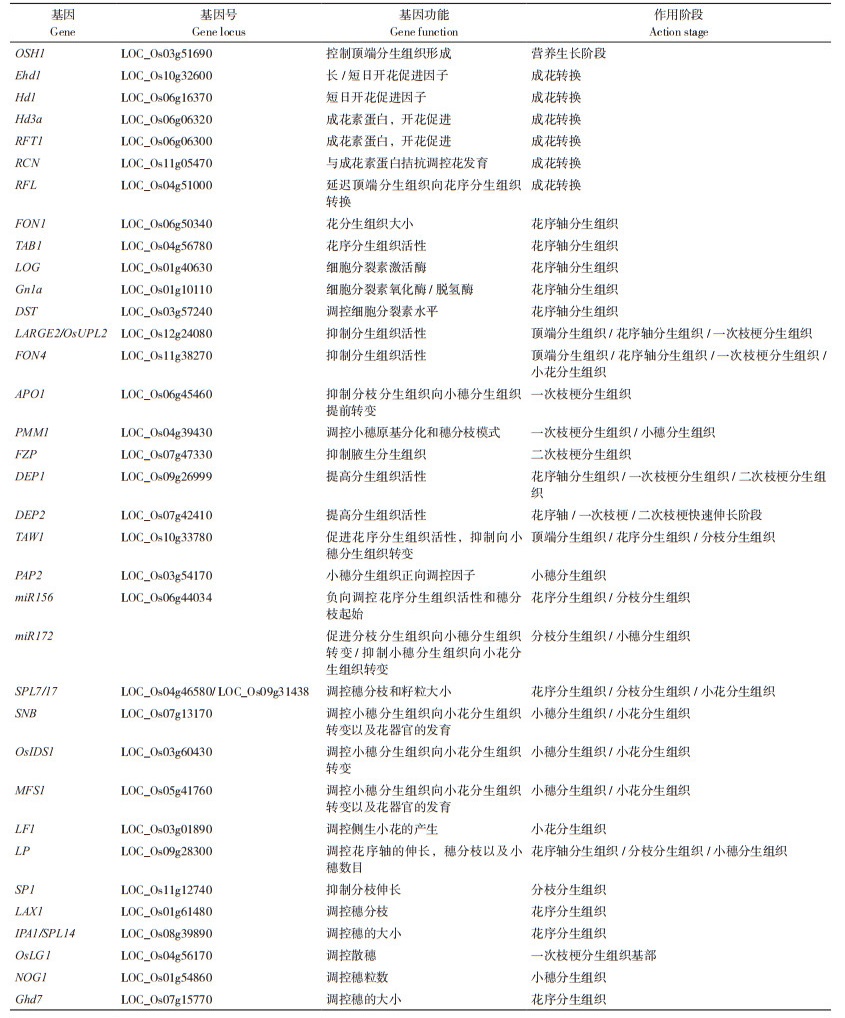
2.1 花序轴分生组织在顶端分生组织转变为花序分生组织后,分生组织立即增大,花序特异基因表达,花序轴分生组织特征建成。拟南芥中,CLAVATA(CLV)- WUSCHEL(WUS)负反馈调节环是保持分生组织干细胞群的主要遗传机制,二者分别是干细胞维持的负向和正向调控因子。尽管不同的植物花序结构有所不同,但是CLV信号途径在单子叶植物和双子叶植物中的功能是部分保守的。水稻CLV1同源基因FLORAL ORGAN NUMBER1(FON1)编码一个LRR类型受体激酶,该基因突变后会造成花分生组织变大,同时花器官数目增多[22]。OsWUS/TAB1优先在分枝分生组织L1层细胞和花分生组织中转录。WUS能够通过促进CLV3的表达限制自身活性,从而形成一个反馈环来保持干细胞群的大小[20-23]。
在tab1突变体中,圆顶状的分生组织变成一个平面结构,生殖腋生分生组织,如分枝分生组织和小穗分生组织,出现活性缺陷,导致花序发育受限,小穗数目变少。当KNOX基因OSH1功能缺失后,种子发芽后顶端分生组织消失,而再生植株在含有高浓度细胞分裂素的培养基上能够生长至生殖生长阶段,但是只能产生少量的小穗,由于花药不正常融合导致小花完全不育[24-26]。
水稻LONELY GUY(LOG)基因编码一个细胞分裂素激活酶,在合成有活性的细胞分裂素的最后一步发挥作用,从而保持分生组织活性,基因突变后导致顶端分生组织活性降低,顶端分生组织和穗变小,穗粒数减少[27]。Gn1a/OsCKX2编码一个细胞分裂素氧化酶/脱氢酶,该基因表达降低能够导致细胞分裂素在花序分生组织中积累,使得小穗数目增加,从而提高水稻产量[28]。DST基因直接调控OsCKX2在生殖分生组织中的表达,从而调控细胞分裂素在顶端分生组织的积累控制生殖器官的数目。DSTreg1是DST的一个半显性等位基因,它能上调生殖分生组织中的细胞分裂素水平而增强分生组织的活性,导致穗分枝数目增加以提高穗粒数[29]。
2.2 一次枝梗分生组织一次枝梗原基从花序轴分生组织上突出,当花序轴分生组织达到最大时,一次枝梗原基停止形成。LARGE2/OsUPL2能够抑制分生组织的活性,负向调控穗大小和穗粒数;在large2突变体中,顶端分生组织和花序轴分生组织变大,一次枝梗分生组织数目增多,最终使得穗子变大,穗粒数增多,粒宽变宽[30]。FLORAL ORGAN NUMBER4(FON4)编码一个拟南芥CLAVATA3同源基因,该基因突变后,会造成顶端分生组织和花序分生组织、小花分生组织增大,使得fon4突变体的一次枝梗数和小花器官数目增加。
ABERRANT PANICLE ORGANIZATION 1(APO1)编码一个与拟南芥UNUSUAL FLORAL ORGANS同源基因,能够抑制分枝分生组织向小穗分生组织的提前转变,其功能缺失后,apo1突变体只分化出少量的一次枝梗分生组织便开始向小穗分生组织转变,导致一次枝梗数目和小穗数目减少,而apo1-D突变体与APO1表达升高的转基因植株,由于APO1抑制IM向SM转变,使得一次枝梗数目和小穗数目增加。
Panicle morphology mutant 1(PMM1)基因编码一个细胞色素P450基因,它是D11的一个新等位基因,参与油菜素内酯的生物合成,在幼穗的分枝原基和小穗原基中表达,从而影响小穗原基的分化和穗分枝模式;功能缺失突变体pmm1的一次枝梗成簇存在,形成不正常的穗部结构[31]。
2.3 二次枝梗分生组织普通野生稻没有二次枝梗的分化,一次枝梗上的小穗数目较少,因而产量较低;而亚洲栽培稻在经历了驯化过程以后,穗子变大,一次枝梗和二次枝梗数目增多,使得栽培稻产量大幅提高。二次枝梗分生组织在一次枝梗分生组织上突起,在这一时期一次枝梗已经伸长,但还没有小穗分化。FRIZZY PANICLE(FZP)基因在普通野生稻向亚洲栽培稻驯化过程中受到选择,是调控二次枝梗数目发生改变的一个重要基因。在发育的花序中,FZP能够抑制腋生分生组织的形成,栽培稻中该基因的顺式调控区发生变化能够降低其表达,延长花序分枝形成的时间,使得花序中的二次枝梗分生组织增多,最终栽培稻中二次枝梗数和穗粒数显著增多,产量大幅提高;但是,当FZP基因功能完全丧失后,突变体会过度生成连续的二次枝梗分生组织,且二次枝梗分生组织不会向小穗分生组织转变,从而不断产生二次分枝,却无种子生成[32-33]。FZP mRNA的3’UTR区域有一个CU-rich元件(CURE),它是多聚尿嘧啶区结合蛋白OsPTB1和OsPTB2的靶标;OsPTB1和OsPTB2在幼穗中大量表达,敲除任一基因都会导致FZP的翻译效率升高,并造成二次枝梗数和穗粒数减少[34]。
水稻RICE CENTRORADIALIS 1(RCN1)和RCN2基因是拟南芥TERMINAL FLOWER1(TFL1)同源基因。水稻中Hd3a成花素蛋白能够与14-3-3蛋白和OsFD蛋白形成“成花素激活复合体”(FAC)以诱导开花;RCN蛋白能够与Hd3a竞争性结合14-3-3,从而与成花素蛋白拮抗以调控花序发育。RCN基因的组成性过表达会导致向生殖生长阶段转变的延迟,使得二次枝梗数量增多,形成密穗。RFL基因影响腋生分生组织的形成而影响分蘖,也可以促进从顶端分生组织向花序分生组织的转变,该基因表达降低会大大延迟向花序分生组织转变的时间,极端情况下会导致无法开花,而过表达该基因能够使开花提前。敲除RFL基因会导致RCN2转录本增加,也就延迟了水稻生殖阶段的转变,说明RCN1对RFL功能具有拮抗作用[35-36]。
DENSE AND ERECT PANICLE 1(DEP1)的显性等位基因是一个功能获得型突变,编码一个截断的磷脂酰乙醇胺结合蛋白,它能够提高分生组织活性,导致花序节间变短,一次枝梗数,尤其是二次枝梗数极显著增多,最终穗粒数增加,大幅提高水稻产量。DEP2编码一个未知功能的植物特异蛋白,它主要影响花序轴、一次枝梗和二次枝梗的快速伸长,但是并不破坏穗原基的起始或者形成[37-39]。
2.4 小穗分生组织一次枝梗分生组织和二次枝梗分生组织转变为末端小穗分生组织,并形成退化颖壳,随后侧生分生组织变为侧生小穗分生组织。TAWAWA1(TAW1)基因编码一个功能未知的核蛋白,在顶端分生组织、花序分生组织和分枝分生组织中表达较高,在小穗分生组织形成初期,TAW1的表达消失。在功能获得型突变体taw1-D中,花序分生组织活性延长,小穗特化延迟,导致分枝延长,小穗数目增加;当TAW1活性降低时,花序分生组织早熟退化,小穗提前形成。因此TAW1是一个独特的分生组织活性调控因子,通过促进花序分生组织活性,抑制向小穗分生组织特征转变以调控花序发育[40]。
PANICLE PHYTOMER 2(PAP2)编码一个MADS-box SEPALLATA(SEP)亚家族蛋白(MADS34),是小穗分生组织身份的一个正向调控因子,该基因突变后会导致分生组织起始模式混乱,形成小穗分生组织的能力下降,早期形成的小穗转变为分枝,而且退化颖壳,不育外稃,以及小穗的最外围器官都伸长,成为叶片的形态,说明该基因也能调控小花器官的形成。在pap2-1突变体中,一次枝梗和二次枝梗的数目显著增加,从而导致小穗数目增加。miR156/miR529/SPL和miR172/AP2途径共同调控水稻分蘖和穗分枝。过表达OsSPL7/14/17能够显著降低分蘖数、穗分枝和小穗数目,说明一次枝梗上早期出现的侧生分生组织提前转变成了小穗。miR156能够负向调控花序分生组织的活性和穗分枝起始,幼苗期将新出分蘖去掉,野生型能够获得更大的穗,但是过表达miR156不能获得大穗,说明过表达miR156转基因植株的穗子变小并不是因为分蘖增多造成的,miR156控制分蘖和穗分枝是两个截然不同的途径。miR172能够促进分枝分生组织向小穗分生组织的转变,但是抑制小穗分生组织向小花分生组织的转变,说明其在小穗分生组织的建成和保持中具有双重功能。miR156和miR529能够精细调控SPL表达水平,精细调控穗的大小;SPL能够直接调控miR172/AP2和PAP2/Rice TFL1/CEN homolog 1(RCN1)途径直接调控穗分枝[41-42]。
2.5 小花分生组织小穗分生组织形成后开始向小花分生组织转变,此时小花分生组织转变为确定性分生组织,花器官开始分化,形成2个浆片、6个雄蕊和1个心皮。SUPERNUMERARY BRACT(SNB)基因编码一个包含2个AP2结构域的转录因子,能够控制小穗分生组织向小花分生组织的转变以及花器官的发育,敲除该基因后会导致小穗分生组织向小花分生组织转变延迟,产生多重退化的颖壳,这种额外苞片的形成,扰乱了小花结构,某些情况下,雄蕊和心皮的数目也会发生变化,在小穗轴的下方还会出现异位小花。在snb osids1双突变体中,分枝和小穗的数目显著降低,与snb单突变体相比,小穗分生组织向小花分生组织转变进一步延迟。过表达miR172能降低SNB和OsIDS1的转录本水平,使得转基因植株呈现比snb osids1双突变体更严重的表型,说明由miR172调控的AP2家族基因表达的降低在调控花序分枝和花器官形成过程中起到重要作用[43]。
MULTI-FLORET SPIKELET1(MFS1)基因编码一个AP2/ERF转录因子,在调控小穗分生组织和小花器官特征过程中起到重要作用,能正向调控LONG STERILE LEMMA(G1)以及SNB和OsIDS1的表达。在突变体mfs1中,小穗分生组织向小花分生组织转变延迟,并出现了额外的颖壳状器官,且小穗轴伸长[44]。
LATERAL FLORET 1(LF1)基因编码一个HD-ZIP Ⅲ蛋白,该基因的miR165/166结合位点发生突变后,lf1突变体的小穗中,侧生小花分生组织被促进,产生相对正常且能够正常开放的小花。在lf1小穗的不育外稃原基中,LF1和OSH1异位表达,导致不育外稃腋下的侧生分生组织起始,生成侧生小花。有关LF1基因的这一调控作用,为水稻“三花小穗”假设提供了有力支撑[45]。
3 驯化和遗传改良调整水稻花序结构 3.1 驯化调整水稻花序结构发育机制限制了选择过程中遗传和表型变化之间的范围,而选择影响发育进程的进化。花序结构多样性在作物驯化过程中起到重要作用,花序结构的调整有利于增加花的数目,从而提高作物产量[46-47]。
普通野生稻呈现典型的散穗表型,这有利于自身异花授粉,成熟后种子的传播以及减少病害侵染,这是对自然生境的一种适应性;而经历驯化后,亚洲栽培稻穗型变紧,这种表型更有利于水稻高密度种植,穗分枝更容易结实增多,提高收获效率。OsLG1基因编码一个SBP家族转录因子,上游调控区域DNA序列的变异直接导致了穗型的转变[48-49]。水稻frizzy panicle(fzp)突变体的小花被连续形成的分枝替代,形成卷曲穗型。FRIZZY PANICLE(FZP)能够抑制穗分枝,并正向影响花分生组织的形成。普通野生稻几乎不存在二次枝梗,在驯化过程中FZP受到选择,基因上游1个4-bp串联重复序列的缺失能够影响生长素响应因子的结合活性,降低其表达,从而显著增加栽培稻的二次枝梗,大幅提高产量[32-33]。普通野生稻的穗粒数较少,产量很低,在驯化过程中选择NUMBER OF GRAINS 1(NOG1)基因能显著增加栽培稻的穗粒数。NOG1增加穗粒数的同时,并不会对有效穗数或者粒重产生负面影响,也不会改变抽穗期或者着粒率,是水稻驯化过程中水稻产量增加的一个重要基因(表 1)[50]。
3.2 遗传改良优化水稻穗型
在水稻的遗传改良过程中,人们对不同穗型进行选择,也直接导致了不同水稻类型中穗型的变化,出现了诸如直立穗、弯曲穗、波浪穗、密穗、稀穗、长穗、短穗、散穗、紧穗等不同穗型。育种家通过传统育种手段以及现代分子设计育种将它们应用到生产实践中,促进了现代水稻高产育种的发展。
通常情况下,灌浆中期水稻穗开始弯曲,冠层消光系数(k-value)增加,从而影响对产量最为关键的顶端3个叶片冠层光合效率降低,直立穗能够减少冠层的遮荫面积,增加光合效率,还能改变栽培条件下的生理状态,包括冠层的光照、温度、湿度、二氧化碳浓度等,从而增加群体生长速率,成为培育高产水稻品种一个非常重要的农艺性状。前文提到直立穗基因DEP1的一个功能获得性等位突变,能提高分生组织的活性,导致花序节间变短,每穗粒数增加,使得单株产量大幅提高;DEP2能够使主穗轴、一次枝梗和二次枝梗快速伸长,该基因突变后植株表现出密穗、直立穗的表型,虽然突变体的株型比较紧凑,但是相比野生型产量并没有显著的变化。DEP3编码一个PLA2亚家族结构域蛋白;dep3突变体有更多的维管束,茎秆更厚,能够提供更有力的支撑,使得穗从开花到成熟一直保持直立。有报道称直立穗类型的粳稻品种更抗倒伏,耐水肥,下位叶片的光合效率更高,并通过增加二次枝梗数及二次枝梗粒数来提高水稻产量,基于此,在中国种植的很多高产品种都具备直立穗的表型[37-39, 51]。
育种过程中选择合适的穗长对于优化穗型,获得理想株型提高产量至关重要。一般来说,籼稻比粳稻穗长更长,小穗密度更为松散;即使同一亚种的不同品种之间,穗长也会因为遗传背景的变化而有所不同,通常水稻的穗长在12~40 cm之间。
LARGER PANICLE(LP)编码一个Kelch repeat-containing F-box蛋白,该蛋白定位于内质网上;该基因突变后,lp突变体穗子明显变大,产生更多的花序分枝,尤其是一次枝梗结实增多,而且抗倒伏能力更强,说明突变体植株对于高产育种非常具有应用潜力。相反,某些基因突变后可能会造成花序伸长障碍。short panicle1(sp1)突变体的一次枝梗原基、二次枝梗原基和花器官都能够正常形成;当野生型中所有的穗轴和小穗原基形成后,穗分枝原基开始生长和伸长,但是突变体中的分枝原基,尤其是靠近基部的位置,生长受到显著甚至完全的抑制,最终导致分枝的伸长抑制和退化,形成短穗,这种表型对产量造成极其不利的影响。SP3编码一个具有Dof结构域的DNA结合转录激活因子,在幼穗中特异表达,尤其是在分枝原基区域。SP3通过上调APO2/RFL,负向调控花序分生组织退化。突变体sp3的幼穗中,细胞分裂素的浓度降低,导致分枝和小穗数目减少,穗子变小。SP3通过调节细胞分裂素的动态平衡调节穗的结构[52]。
密穗表型已在水稻高产育种中被广泛应用,但也有地方种穗型极为松散,着粒密度很低,非常不利于水稻遗传改良。LAX PANICLE(LAX)和SMALL PANICLE(SPA)是水稻腋生分生组织形成的重要调控因子。LAX调控水稻花序腋生分生组织起始和保持,也是小花分生组织特征调控基因,特化末端小穗分生组织,该基因突变后,会导致侧生小穗发育受限;在spa突变体中,穗分枝和小穗数目也显著降低[53]。
IPA1/WFP/OsSPL14是影响水稻株型和产量的一个现象级基因,也被称为“理想株型”基因,甚至被认为是新的“绿色革命基因” [5]。IPA1上游启动子区域的表观调控能够增加IPA1的表达,从而促进穗分枝;而营养生长阶段OsSPL14能够抑制芽分枝[54]。ipa1-2D是上调IPA1的弱等位基因,它能够很大程度上影响穗的大小,又不会过度降低分蘖数,通过优化不同等位基因的组合精细调控IPA1的表达,将会最大程度获得理想株型。IPA1与其他控制株型、穗型的基因存在直接的互作。IPA1能够直接结合在分蘖芽生长负向调控因子OsTB1的启动子区域,从而抑制水稻的分蘖;能够直接正向调控DEP1的表达影响株高和穗长[55]。IPI1能够促进IPA1在穗部的降解,却能够保持它在芽顶端的稳定性。单独利用IPA1时,过表达IPA1会造成穗变大,但是分蘖数降低,从而限制IPA1在育种上的应用;但是IPI1能够组织特异的调控IPA1的丰度,将IPI1敲除,能够同时增加分蘖数和穗大小,从而增加IPA1应用的可行性[56]。
光温条件对水稻的穗型也有很大影响。水稻是一个典型的短日照作物,表现出强烈的光周期敏感性,在短日照诱导条件下能够促进开花。提前开花会缩短营养生长阶段,导致生物量积累不足,难以获得高产,而在恶劣种植条件下,生育期缩短可以最大限度满足生产需求;而开花延迟,更容易获得较大的生物量,但很容易降低结实率,并且随着季节转换,环境不再适合生长,很容易造成减产,甚至延误下一个生长季[57]。Ghd7编码一个CCT结构域蛋白,长日条件下,该基因高表达能够延迟抽穗,株高变高,穗子变大;而该基因功能降低的自然突变体使得水稻能够在温带和更为凉爽的地区种植,该基因在增加产量以及水稻全球适应性中都起到非常重要的作用[58-59]。
4 展望水稻花序结构与产量直接相关,对穗型调控的相关研究,具有重要的应用前景,已成为科学家的重点研究方向。在水稻育种进程中,通过优化穗型培育的栽培种,对促进水稻高产、农民增收、保证粮食安全产生了重要的影响。在过去几十年中,传统水稻栽培技术需要投入大量的资源,化肥和农药的使用在一定程度上促进了水稻增产,但加剧了环境污染,严重破坏生态平衡。通过分子设计育种培育投入减少、环境友好、资源利用率高、抗逆性强、产量高的“绿色超级稻”势在必行,而穗型改良是水稻分子设计育种的一个重要目标,研究花序发育过程的影响因素以遗传调控网络对实现这一目标具有重要意义。花序发育依赖各种类型的分生组织活性。花序轴分生组织,分枝分生组织,小穗分生组织和小花分生组织,是构成水稻复杂花序的必要条件。花序结构直接影响水稻穗粒数和最终产量,其多样性主要依赖于分枝模式和花的位置。在驯化过程中,穗型受到选择,水稻花序调整为更有利于增加小穗数量的结构;在改良过程中,相关优异等位基因的利用,更有利于水稻穗型优化。
在过去数十年中,参与水稻穗型调控的相关因子及遗传调控通路已经取得了重大进展。例如很多参与水稻花序发育调控的基因通过生长素、细胞分裂素等植物激素信号通路调控分生组织的大小,从而调控水稻的穗型。当内源激素不利于花序发育时,通过外施激素类似物或者阻遏物,干预穗的发育过程,有利于保证水稻的高产稳产。但是植物激素调控是一个极其复杂的调控网络,无论是外源或是内源调控,都有可能对其他性状产生不利影响。掌握激素调控的规律,才能有的放矢精准调控水稻各个器官的发育,这类调控还需进一步的研究。
水稻作为禾本科作物研究的模式植物,与玉米、小麦、大麦、高粱等其他禾谷类作物的同源度较高,相关发育机制和调控网络具有部分保守性,这可为其他作物的相关研究提供重要参考。随着基因组学和现代分子生物学技术的快速发展,相关遗传调控机制越来越清晰,这都有助于解析生长发育的调控网络,对优异等位基因的利用效率更高,将加快分子设计育种的进程,为实现作物高产稳产,保证粮食安全提供理论支撑。
| [1] |
RAY D K, MUELLER N D, WEST P C, FOLEY J A. Yield trends are insufficient to double global crop production by 2050[J]. Plos One, 2013, 8: e66428. DOI:10.1371/journal.pone.0066428 |
| [2] |
WING R A, PURUGGANAN M D, ZHANG Q F. The rice genome revolution: from an ancient grain to Green Super Rice[J]. Nature Reviews Genetics, 2018, 19: 505-517. DOI:10.1038/s41576-018-0024-z |
| [3] |
RAY D K, GERBER J S, MACDONALD G K, WEST P C. Climate variation explains a third of global crop yield variability[J]. Nature Communications, 2015, 6: 5989. DOI:10.1038/ncomms6989 |
| [4] |
ZHANG Q F. Strategies for developing Green Super Rice[J]. Proceedings of the National Academy of Sciences, 2007, 104: 16402-16409. DOI:10.1073/pnas.0708013104 |
| [5] |
WANG B B, WANG H Y. IPA1: A new"Green Revolution"gene[J]. Molecular Plant, 2017, 10: 779-781. DOI:10.1016/j.molp.2017.04.011 |
| [6] |
ORDONIO R L, MATSUOKA M. New path towards a better rice architecture[J]. Cell Research, 2017, 27: 1189-1190. DOI:10.1038/cr.2017.115 |
| [7] |
WANG S S, WU K, QIAN Q, LIU Q, LI Q, PAN Y J, YE Y F, LIU X Y, WANG J, ZHANG J Q, LI S, WU Y J, FU X D. Non-canonical regulation of SPL transcription factors by a human OTUB1-like deubiquitinase defines a new plant type rice associated with higher grain yield[J]. Cell Research, 2017, 27: 1142-1156. DOI:10.1038/cr.2017.98 |
| [8] |
KOPPOLU R, CHEN S L, SCHNUR BUSCH T. Evolution of inflorescence branch modifications in cereal crops[J]. Current Opinion in Plant Biolog y, 2022, 65: 102168. DOI:10.1016/j.pbi.2021.102168 |
| [9] |
WANG Y H, LI J Y. The Plant Architecture of Rice (Oryza sativa)[J]. Plant Molecular Biology, 2005, 59: 75-84. DOI:10.1007/s11103-004-4038-x |
| [10] |
HU Z, YANG Z P, ZHANG Y, ZHANG A H, LU Q T, FANG Y, LU C M. Autophagy targets Hd1 for vacuolar degradation to regulate rice flowering[J]. Molecular Plant, 2022, 15: 1137-1156. DOI:10.1016/j.molp.2022.05.006 |
| [11] |
SUN K L, HUANG M H, ZONG W B, XIAO D D, LEI C, LUO Y Q, SONG Y G, LI S T, HAO Y, LUO W N, XU B Q, GUO X T, WEI G L, CHEN L T, LIU Y G, GUO J X. Hd1, Ghd7, and DTH8 synergistically determine the rice heading date and yield-related agronomic traits[J]. Journal of Genetics and Genomics, 2022, 49: 437-447. DOI:10.1016/j.jgg.2022.02.018 |
| [12] |
SHRESTHA R, G-MEZ-ARIZA J, BRAMBILLA V, FORNARA F. Molecular control of seasonal flowering in rice, Arabidopsis and temperate cereals[J]. Annals of Botany, 2014, 114: 1445-1458. DOI:10.1093/aob/mcu032 |
| [13] |
ZHAO J, CHEN H Y, REN D, TANG H W, QIU R, FENG J L, LONG Y M, NIU B X, CHEN D P, ZHONG T Y, LIU Y G, GUO J X. Genetic interactions between diverged alleles of Early heading date 1 (Ehd1) and Heading date 3a (Hd3a)/RICE FLOWERING LOCUS T1 (RFT1) control differential heading and contribute to regional adaptation in rice (Oryza sativa)[J]. New Phytologist, 2015, 208: 936-948. DOI:10.1111/nph.13503 |
| [14] |
ENDO-HIGASHI N, IZAWA T. Flowering time genes heading date 1 and early heading date 1 together control panicle development in rice[J]. Plant and Cell Physiology, 2011, 52: 1083-1094. DOI:10.1093/pcp/pcr059 |
| [15] |
NEMOTO Y, NONOUE Y, YANO M, IZAWA T. Hd1, a CONSTANS ortholog in rice, functions as an Ehd1 repressor through interaction with monocot-specific CCT-domain protein Ghd7[J]. The Plant Journal, 2016, 86: 221-233. DOI:10.1111/tpj.13168 |
| [16] |
IKEDA K, SUNOHARA H, NAGATO Y. Developmental course of inflorescence and spikelet in rice[J]. Breeding Science, 2004, 54: 147-156. DOI:10.1270/jsbbs.54.147 |
| [17] |
SUZAKI T, SATO M, ASHIKARI M, MIYOSHI M, NAGATO Y, HIRANO HY. The gene FLORAL ORGAN NUMBER1 regulates floral meristem size in rice and encodes a leucine-rich repeat receptor kinase orthologous to Arabidopsis CLAVATA1[J]. Development, 2004, 131: 5649-5657. DOI:10.1242/dev.01441 |
| [18] |
ZHANG D B, YUAN Z. Molecular control of grass inflorescence development[J]. Annual Review of Plant Biology, 2014, 65: 553-578. DOI:10.1146/annurev-arplant-050213-040104 |
| [19] |
淳雁, 李学勇. 水稻穗型的遗传调控研究进展[J]. 植物学报, 2017, 52(1): 19-29. DOI:10.11983/CBB16213 CHUN Y, LI X Y. Research progress in genetic regulation of rice panicle architecture[J]. Chinese Bulletin of Botany, 2017, 52(1): 19-29. DOI:10.11983/CBB16213 |
| [20] |
PAUTLER M, TANAKA W, HIRANO H, JACKSON D. Grass meristems I: shoot apical meristem maintenance, axillary meristem determinacy and the floral transition[J]. Plant and Cell Physiology, 2013, 54: 302-312. DOI:10.1093/pcp/pct025 |
| [21] |
翟雪, 张栩, 连光倩, 王建飞. 水稻穗型相关性状的QTLs定位[J]. 江苏农业科学, 2019, 47(13): 59-63. DOI:10.15889/j.issn.1002-1302.2019.13.016 ZHAI X, ZHANG X, LIAN G Q, WANG J F. Mapping of QTLs for panicle architecture in rice[J]. Jiangsu Agricultural Sciences, 2019, 47(13): 59-63. DOI:10.15889/j.issn.1002-1302.2019.13.016 |
| [22] |
NARDMANN J, WERR W. The shoot stem cell niche in angiosperms: expression patterns of WUS orthologues in rice and maize imply major modifications in the course of mono-and dicot evolution[J]. Molecular Biology and Evolution, 2006, 23: 2492-2504. DOI:10.1093/molbev/msl125 |
| [23] |
SOYARS C L, JAMES S R, NIMCHUK Z L. Ready, aim, shoot: stem cell regulation of the shoot apical meristem[J]. Current Opinion in Plant Biology, 2016, 29: 163-168. DOI:10.1016/j.pbi.2015.12.002 |
| [24] |
TANAKA W, OHMORI Y, USHIJIMA T, MATSUSAKA H, MATSUSHITA T, KUMAMARU T, KAWANO S, HIRANO H. Axillary Meristem formation in rice requires the WUSCHEL ortholog TILLERS ABSENT1[J]. The Plant Cell, 2015, 27: 1173-1184. DOI:10.1105/tpc.15.00074 |
| [25] |
TSUDA K, ITO Y, SATO Y, KURATA N. Positive autoregulation of a KNOX gene is essential for shoot apical meristem maintenance in rice[J]. The Plant Cell, 2011, 23: 4368-4381. DOI:10.1105/tpc.111.090050 |
| [26] |
TSUDA K, KURATA N, OHYANAGI H, HAKE S. Genome-wide study of KNOX regulatory network reveals brassinosteroid catabolic genes important for shoot meristem function in rice[J]. The Plant Cell, 2014, 26: 3488-3500. DOI:10.1105/tpc.114.129122 |
| [27] |
KURAKAWA T, UEDA N, MAEKAWA M, KOBAYASHI K, KOJIMA M, NAGATO Y, SAKAKIBARA H, KYOZUKA J. Direct control of shoot meristem activity by a cytokinin-activating enzyme[J]. Nature, 2007, 445: 652-655. DOI:10.1038/nature05504 |
| [28] |
ASHIKARI M, SAKAKIBARA H, LIN S, YAMAMOTO T, TAKASHI T, NISHIMURA A, ANGELES ER, QIAN Q, KITANO H, MATSUOKA M. Cytokinin oxidase regulates rice grain production[J]. Science, 2005, 309: 741-745. DOI:10.1126/science.1113373 |
| [29] |
LI S Y, ZHAO B R, YUAN D Y, DUAN M J, QIAN Q, TANG L, WANG B, LIU X Q, ZHANG J, WANG J, SUN J Q, LIU Z, FENG Y Q, YUAN L P, LI C Y. Rice zinc finger protein DST enhances grain production through controlling Gn1a/OsCKX2 expression[J]. Proceedings of the National Academy of Sciences, 2013, 110: 3167-3172. DOI:10.1073/pnas.1300359110 |
| [30] |
HUANG L J, HUA K, XU R, ZENG D L, WANG R C, DONG G J, ZHANG G Z, LU X L, FANG N, WANG D K, DUAN P G, ZHANG B L, LIU Z P, LI N, LUO Y H, QIAN Q, YAO S G, LI Y H. The LARGE2-APO1/APO2 regulatory module controls panicle size and grain number in rice[J]. The Plant Cell, 2021, 33: 1212-1228. DOI:10.1093/plcell/koab041 |
| [31] |
LI Y, LI X M, FU D B, WU C Y. Panicle Morphology Mutant 1 (PMM1) determines the inflorescence architecture of rice by controlling brassinosteroid biosynthesis[J]. BMC Plant Biology, 2018, 18(1): 348. DOI:10.1186/s12870-018-1577-x |
| [32] |
HUANG Y Y, ZHAO S S, FU Y C, SUN H D, MA X, TAN L B, LIU F X, SUN X Y, SUN H Y, GU P, XIE D X, SUN C Q, ZHU Z F. Variation in the regulatory region of FZP causes increases in secondary inflorescence branching and grain yield in rice domestication[J]. The Plant Journal, 2018, 96: 716-733. DOI:10.1111/tpj.14062 |
| [33] |
BAI X F, HUANG Y, HU Y, LIU H Y, ZHANG B, SMACZNIAK C, HU G, HAN Z M, XING Y Z. Duplication of an upstream silencer of FZP increases grain yield in rice[J]. Nature Plants, 2017, 3: 885-893. DOI:10.1038/s41477-017-0042-4 |
| [34] |
CHEN Q, TIAN F A, CHENG T T, JIANG J E, ZHU G L, GAO Z Y, LIN H Y, HU J, QIAN Q, FANG X H, CHEN F. Translational repression of FZP mediated by CU‐rich element/OsPTB interactions modulates panicle development in rice[J]. The Plant Journal, 2022, 110: 1319-1331. DOI:10.1111/tpj.15737 |
| [35] |
KANEKO-SUZUKI M, KURIHARA-ISHIKAWA R, OKUSHITA-TERAKAWA C, KOJIMA C, NAGANO-FUJIWARA M, OHKI I, TSUJI H, SHIMAMOTO K, TAOKA KI. TFL1-like proteins in rice antagonize rice FT-like protein in inflorescence development by competition for complex formation with 14-3-3 and FD[J]. Plant and Cell Physiology, 2018, 59: 458-468. DOI:10.1093/pcp/pcy021 |
| [36] |
RAO N N, PRASAD K, KUMAR P R, VIJAYRAGHAVAN U. Distinct regulatory role for RFL, the rice LFY homolog, in determining flowering time and plant architecture[J]. Proceedings of the National Academy of Sciences, 2008, 105: 3646-3651. DOI:10.1073/pnas.0709059105 |
| [37] |
葛中英, 张杰, 刘正, 易军, 李亚男, 赵祥, 胡运高. 几个籼型直立穗水稻突变体穗部性状分析[J]. 广东农业科学, 2015, 42(15): 7-11. DOI:10.16768/j.issn.1004-874X.2015.15.002 GE Z Y, ZHANG J, LIU Z, YI J, LI Y N, ZHAO X, HU Y G. Analysis on panicle traits of a few erect panicle mutants in Indica rice[J]. Guangdong Agricultural Sciences, 2015, 42(15): 7-11. DOI:10.16768/j.issn.1004-874X.2015.15.002 |
| [38] |
HUANG X Z, QIAN Q, LIU Z B, SUN H Y, HE S Y, LUO D, XIA G M, CHU C C, LI J Y, FU X D. Natural variation at the DEP1 locus enhances grain yield in rice[J]. Nature Genetics, 2009, 41: 494-497. DOI:10.1038/ng.352 |
| [39] |
LI F, LIU W B, TANG J Y, CHEN J F, TONG H N, HU B, LI C L, FANG J, CHEN M S, CHU C C. Rice DENSE AND ERECT PANICLE 2 is essential for determining panicle outgrowth and elongation[J]. Cell Research, 2010, 20: 838-849. DOI:10.1038/cr.2010.69 |
| [40] |
YOSHIDA A, SASAO M, YASUNO N, TAKAGI K, DAIMON Y, CHEN R, YAMAZAKI R, TOKUNAGA H, KITAGUCHI Y, SATO Y, NAGAMURA Y, USHIJIMA T, KUMAMARU T, ⅡDA S, MAEKAWA M, KYOZUKA J. TAWAWA1, a regulator of rice inflorescence architecture, functions through the suppression of meristem phase transition[J]. Proceedings of the National Academy of Sciences, 2013, 110: 767-772. DOI:10.1073/pnas.1216151110 |
| [41] |
LIU C, TEO Z W N, BI Y, SONG S Y, XI W Y, YANG X B, YIN Z C, YU H. A conserved genetic pathway determines inflorescence architecture in Arabidopsis and rice[J]. Developmental Cell, 2013, 24: 612-622. DOI:10.1016/j.devcel.2013.02.013 |
| [42] |
WANG L, SUN S Y, JIN J Y, FU D B, YANG X F, WENG X Y, XU C G, LI X H, XIAO J H, ZHANG Q F. Coordinated regulation of vegetative and reproductive branching in rice[J]. Proceedings of the National Academy of Sciences, 2015, 112: 15504-15509. DOI:10.1073/pnas.1521949112 |
| [43] |
LEE D, AN G. Two AP 2 family genes, SUPER NUMER ARY BRACT (SNB) and OsINDETERMINATE SPIKELET 1 (OsIDS1), synergistically control inflorescence architecture and floral meristem establishment in rice[J]. The Plant Journal, 2012, 69: 445-461. DOI:10.1111/j.1365-313X.2011.04804.x |
| [44] |
REN D Y, LI Y F, ZHAO F M, SANG X C, SHI J Q, WANG N, GUO S, LING Y H, ZHANG C W, YANG Z L, HE G H. MULTI-FLORET SPIKELET1, which encodes an AP2/ERF protein, determines spikelet meristem fate and sterile lemma identity in rice[J]. Plant Physiology, 2013, 162: 872-884. DOI:10.1104/pp.113.216044 |
| [45] |
ZHANG T, LI Y F, MA L, SANG X C, LING Y H, WANG Y T, YU P, ZHUANG H, HUANG J Y, WANG N, ZHAO F M, ZHANG C W, YANG Z L, FANG L K, HE G H. LATERAL FLORET 1 induced the threeflorets spikelet in rice[J]. Proceedings of the National Academy of Sciences, 2017, 114: 9984-9989. DOI:10.1073/pnas.1700504114 |
| [46] |
DIXON L E, GREENWOOD J R, BENCIVENGA S, ZHANG P, COCKRAM J, MELLERS G, RAMM K, CAVANAGH C, SWAIN S M, BODEN S A. TEOSINTE BRANCHED1 regulates inflorescence architecture and development in bread wheat (Triticum aestivum)[J]. The Plant Cell, 2018, 30: 563-581. DOI:10.1105/tpc.17.00961 |
| [47] |
PRUSINKIEWICZ P, ERASMUS Y, LANE B, HARDER LD, COEN E. Evolution and development of inflorescence architectures[J]. Science, 2007, 316: 1452-1456. DOI:10.1126/science.1140429 |
| [48] |
ZHU Z F, TAN L B, FU Y C, LIU F X, CAI H W, XIE D X, WU F, WU J Z, MATSUMOTO T, SUN C Q. Genetic control of inflorescence architecture during rice domestication[J]. Nature Communications, 2013, 4: 2200. DOI:10.1038/ncomms3200 |
| [49] |
ISHⅡ T, NUMAGUCHI K, MIURA K, YOSHIDA K, THANH PT, HTUN TM, YAMASAKI M, KOMEDA N, MATSUMOTO T, TERAUCHI R, ISHIKAWA R, ASHIKARI M. OsLG1 regulates a closed panicle trait in domesticated rice[J]. Nature Genetics, 2013, 45: 462-465. DOI:10.1038/ng.2567 |
| [50] |
HUO X, WU S, ZHU Z F, LIU F X, FU Y C, CAI H W, SUN X Y, GU P, XIE D X, TAN L, SUN C Q. NOG1 increases grain production in rice[J]. Nature Communications, 2017, 8: 1497. DOI:10.1038/s41467-017-01501-8 |
| [51] |
QIAO Y L, PIAO R H, SHI J X, LEE S I, JIANG W Z, KIM B K, LEE J Y, HAN L Z, MA W B, KOH H J. Fine mapping and candidate gene analysis of dense and erect panicle 3, DEP3, which confers high grain yield in rice (Oryza sativa L.)[J]. Theoretical and Applied Genetics, 2011, 122: 1439-1449. DOI:10.1007/s00122-011-1543-6 |
| [52] |
HUANG Y, BAI X F, LUO M F, XING Y Z. Short Panicle 3 controls panicle architecture by upregulating APO2/RFL and increasing cytokinin content in rice[J]. Journal of Integrative Plant Biology, 2018, 61: 987-999. DOI:10.1111/jipb.12729 |
| [53] |
PARIDA A K, SEKHAR S, PANDA B B, SAHU G, SHAW B P. Effect of panicle morphology on grain filling and rice yield: genetic control and molecular regulation[J]. Frontiers in Genetics, 2022, 13. DOI:10.3389/fgene.2022.876198 |
| [54] |
MIURA K, IKEDA M, MATSUBARA A, SONG X, ITO M, ASANO K, MATSUOKA M, KITANO H, ASHIKARI M. OsSPL14 promotes panicle branching and higher grain productivity in rice[J]. Nature Genetics, 2010, 42: 545-549. DOI:10.1038/ng.592 |
| [55] |
LU Z F, YU H, XIONG G S, WANG J, JIAO Y Q, LIU G F, JING Y H, MENG X B, HU X M, QIAN Q, FU X D, WANG Y H, LI J Y. Genomewide binding analysis of the transcription activator IDEAL PLANT ARCHITECTURE1 reveals a complex network regulating rice plant architecture[J]. The Plant Cell, 2013, 25: 3743-3759. DOI:10.1105/tpc.113.113639 |
| [56] |
WANG J, YU H, XIONG G S, LU Z F, JIAO Y Q, MENG X B, LIU G F, CHEN X W, WANG Y H, LI J Y. Tissue-specific ubiquitination by IPA1 INTERACTING PROTEIN1 modulates IPA1 protein levels to regulate plant architecture in rice[J]. The Plant Cell, 2017, 29: 697-707. DOI:10.1105/tpc.16.00879 |
| [57] |
SUN C H, CHEN D, FANG J, WANG P R, DENG X J, CHU C C. Understanding the genetic and epigenetic architecture in complex network of rice flowering pathways[J]. Protein & Cell, 2014, 5: 889-898. DOI:10.1007/s13238-014-0068-6 |
| [58] |
XUE W Y, XING Y Z, WENG X Y, ZHAO Y, TANG W J, WANG L, ZHOU H J, YU S B, XU C G, LI X H, ZHANG Q F. Natural variation in Ghd7 is an important regulator of heading date and yield potential in rice[J]. Nature Genetics, 2008, 40: 761-767. DOI:10.1038/ng.143 |
| [59] |
苏菁, 陈深, 朱小源. 水稻广谱抗病分子机理研究进展[J]. 广东农业科学, 2020, 47(11): 84-92. DOI:10.16768/j.issn.1004-874X.2020.11.010 SU J, CHEN S, ZHU X Y. Research progress in molecular mechanism of broad-spectrum disease resistance in rice[J]. Guangdong Agricultural Sciences, 2020, 47(11): 84-92. DOI:10.16768/j.issn.1004-874X.2020.11.010 |
(责任编辑 邹移光)
 2022, Vol. 49
2022, Vol. 49