文章信息
基金项目
- 国家自然科学基金(32072027);广东省重点建设学科科研能力提升项目(KA23YY385)
作者简介
- 赖瑞强(1991—),男,博士,讲师,研究方向为分子遗传育种,E-mail:1033980612@qq.com.
通讯作者
- 齐永文(1978—),男,博士,教授,研究方向为分子遗传育种,E-mail:yongwen2001@163.com.
文章历史
- 收稿日期:2023-08-30
蛋白质是生物体的基本组分,属于有机大分子之一,其作为细胞功能的最终执行者,参与了细胞和生物体内全部的生命活动。因此,蛋白质正常表达和运作是生命活动有序且高效进行的关键。众所周知,不同的蛋白质具有不同的结构,且往往执行着不同的功能,例如酶类一般为球蛋白,可执行催化功能;而纤维蛋白可参与生物骨架的构建。然而,多数蛋白质在执行功能的过程中,常常依赖不同的翻译后修饰,继而调控不同的生命活动。
蛋白质翻译后修饰包括磷酸化、乙酰化、泛素化、脂酰化和SUMO化等;修饰过程涉及一系列的酶促反应,最终通过共价键将相应的化学基团或小分子蛋白连接到底物蛋白质分子上,继而发挥相应的功能。其中,SUMO蛋白在生命活动的正常运行中扮演着重要角色[1-2],且在多种植物中已见报道,包括拟南芥[3]、水稻[4]、石斛[5]、玉米[2]、苹果[6]、番茄[7]、荔枝[8]、墨西哥玉米草[9]、大豆[10]、花生[11]、马铃薯[12-13]和辣椒[14]。通过研究,在探索SUMOylation参与植物的生长发育和胁迫响应方面取得了一定的进展。此外,研究者们发现SUMOylation也在转录活性的调控[15-16]、蛋白质的稳定性和细胞周期的调节[17-18]等方面参与调控。因此,SUMO作为一种重要的蛋白质修饰标签,且在真核生物中普遍存在,越来越受到广大研究者的关注。
值得一提的是,玉米(Zea mays L.)作为重要的粮食和饲料作物,由于含有丰富的营养成分,如维生素、蛋白质和微量元素等,有“长寿食品”的美誉,备受广大消费者的喜爱[19]。因此,玉米也是研究者普遍关注的一种作物。尤其玉米B73[20]的基因组信息被公布后,给SUMOylation研究带来了便捷;但相比拟南芥,玉米中与SUMOylation相关的研究报道仍相对匮乏,且多数[21-26]仅对SUMOylation过程中涉及到的部分基因功能进行探究,鲜见对相关蛋白质受SUMOylation后的分子机理进行深入研究,不利于了解SUMO化修饰参与玉米生命活动的规律。
综上,了解SUMO蛋白、SUMOylation相关酶类、SUMOylation过程和了解鉴定SUMOylation底物蛋白的方法,有利于后续研究SUMOylation参与生命活动的分子机理,更有助于后续优化生物的表型。因此,本综述通过总结SUMO蛋白及其相关酶类、蛋白质的SUMOylation过程、SUMOylation底物的鉴定方法和玉米蛋白质的SUMOylation研究进展,旨在获悉SUMOylation的作用规律和当前的研究技术,为玉米育种提供指导基础,甚至为其他植物研究提供方向。
1 SUMO蛋白特征及类型SUMO蛋白在真核生物中广泛存在,且由于其结构与泛素相似得名小泛素。虽然相比泛素的研究,SUMO蛋白的解析起步较晚、研究较浅,但随着生物技术的发展,SUMO蛋白被发现参与了多种生命活动,且与泛素在某些方面存在相互作用,而使其逐渐被关注和研究。有意思的是,虽然它们之间作用于靶蛋白的过程类似,但对靶蛋白稳定性的影响却存在差异。
1.1 SUMO蛋白及其与泛素的联系SUMO蛋白在生命活动的正常运行中扮演着关键角色[1-2]。这类蛋白于1995年首次在酿酒酵母(Saccharomyces cerevisiae)中被发现[27-29]。随后,Matunis等[29]发现有一段小分子多肽,即SUMO蛋白与核孔复合体蛋白质RanGAP1结合后,可以改变其定位和功能,而确定了SUMO蛋白能修饰细胞中的蛋白质。
据报道,SUMO蛋白均具有β-grasp(SUMO like)蛋白结构域,其包含的氨基酸数量在100个左右,属于小分子多肽[30]。与泛素相比,它们的氨基酸序列相似性仅有19% 左右[1],且SUMO蛋白被转录翻译后,需要被SUMO特异性蛋白酶切割掉C端一个小片段并暴露出双甘氨酸基序(diGly),才能成为具有功能的成熟SUMO蛋白(Free-SUMO),而泛素被转录翻译后即具有diGly。然而,它们行使修饰功能时均依赖于C端暴露的diGly,且均作用于靶蛋白的赖氨酸上[15, 31-32]。再者,它们还具有相似的三维结构[1],且修饰的驱动过程均涉及3种酶,分别为活化酶E1、结合酶E2和连接酶E3。此外,它们在生物体内调控蛋白质的情况也存在差异,与泛素介导蛋白质降解相比,SUMOylation可能趋向于稳定蛋白质[15]。由于SUMO蛋白与泛素在某些靶蛋白中可能共享同一个赖氨酸[33],它们也常存在拮抗关系,例如在拟南芥中,泛素E3连接酶RHA2b和SUMO E3连接酶SIZ1均会作用于转录因子MYB30的K283位点,从而影响MYB30蛋白的稳定性[34]。因此,了解SUMO蛋白的特征和功能,将有利于更充分解析植物的生命活动规律,促进作物育种进程。
1.2 SUMO蛋白的类型现阶段可将SUMO蛋白分为4大类[30-32, 35],分别为保守型(Canonical)、非保守型(Noncanonical)、双SUMO(diSUMO-like,DSUL)型和SUMO-Variant(SUMO-v)型。其中,保守型、DSUL型和大多数非保守型SUMO蛋白的C端通常可被SUMO特异性蛋白酶切割而暴露出diGly,暗示它们可对靶蛋白进行SUMOylation继而行使功能。然而,植物体内还有一些非保守型的SUMO蛋白,如拟南芥中AtSUMO4和AtSUMO6的C端仅能暴露出单个Gly,但将AtSUMO1 C端的diGly(Gly-Gly)换成Ala-Gly时AtSUMO1仍具有SUMO化修饰活性,而换成Ala-Ala后则散失了活性,这些数据暗示了倒数第二个Gly发生变化并不一定会损害SUMO化的功能[31]。SUMO-v型虽然具有SUMO-like结构域,但其C端却不能暴露出diGly或单个Gly,暗示其不能对靶蛋白进行SUMOylation。
随着生物技术的发展,越来越多生物的SUMO蛋白被鉴定出来。现已知在酵母和无脊椎动物中仅有一种 SUMO基因[1];而在脊椎动物中却存在多种编码SUMO蛋白的基因,如人体中存在 SUMO-1、SUMO-2、SUMO-3和 SUMO-4等基因[1, 36];一些作物体内的 SUMO基因数量更多,如玉米包含5个 SUMO基因[2],而水稻和大豆多达8个 SUMO基因[2, 36-37],所以植物体内涉及的SUMO蛋白功能可能更加复杂。因此,了解植物体内蛋白质SUMOylation的工作模式,有助于后续更充分地解析植物生长发育及应对外界胁迫的分子机理。
2 蛋白质SUMO化修饰的相关酶类及其过程蛋白质的SUMO化修饰过程是一个动态、可逆的修饰过程,而实现该过程涉及到4种酶,分别为SUMO特异性蛋白酶(SUMO-specific proteases)、SUMO活化酶E1(SUMO activating enzymes)、SUMO结合酶E2(SUMO conjugating enzymes) 和SUMO连接酶E3(SUMO ligases)。
2.1 SUMO化修饰的相关酶类植物中的SUMO活化酶E1是异源二聚体,通常小亚基被命名为SAE1,而大亚基被命名为SAE2。这些亚基的数量在不同物种间或在不同的品种间均可能存在差异。如拟南芥中有2个SAE1蛋白,分别为AtSAE1a和AtSAE1b,但功能冗余,而大亚基只有1个,即AtSAE2,若其功能缺失将导致胚胎致死[38];在大豆中,小亚基也有2个,分别为GmSAE1a和GmSAE1b,而其大亚基有3个,分别为GmSAE2a~c[39];在花生中,不同品种(Ad/Ai)的E1酶数量也存在差异,小亚基包括AdSAE1a、AdSAE1b和AiSAE1,而大亚基为AdSAE2和AiSAE2[11]。因此,通过不同大小亚基的组合,植物体内可能存在多个不同的E1酶,继而可能造成部分功能差异。
此外,SUMO结合酶E2在植物中一般被命名为SCE,其在不同物种和品种中的数量存在差异;在拟南芥中只有1个,即AtSCE1[38],但在大豆中有4个,分别为GmSCE1a ~ d[39];在花生中则包括AdSCE1a/b和AiSCE1a ~ c[11];在水稻中已被鉴定的E2酶有3个,分别为OsSCE1~3[40];而在土豆中多达9个,分别为StSCE1~9[12]。因此,除拟南芥外,多数植物体内存在多个E2酶,暗示它们可能承担着不同的功能、具有更复杂的调控网络。
在植物中的SUMO连接酶E3可以分为3类,分别为SIZ1、MMS21和SIZ/PIAS同源蛋白(如PIAL1和PIAL2),它们均具有SIZ/PIAS RING(SP-RING)结构域[2, 41]。其中,SIZ1和MMS21在植物中普遍存在,且报道较多。相比 MMS21,SIZ1在植物中表达的丰度更高[42],且介导了绝大多数靶蛋白的SUMO化修饰[43]。此外,MMS21仅有SP-RING结构域,而SIZ1是典型的SIZ/PIAS家族蛋白,除SP-RING结构域外还包括其他4个保守结构域[44],分别为SAP(Scaffold attachment factor A/B/acinus/PIAS domain)、PHD(Plant homeo domain)、PINIT(Proline-Isoleucine-Asparagine-Isoleucine-Threonine)、SP-RING(SIZ/PIAS-RING)和SXS(Serine-X-Serine)。其中,SAP主要与DNA的结合相关,而PHD为植物所特有,具有核小体结合活性,且与磷脂酰肌醇的识别和植物响应热胁迫有关;PINIT则是SIZ1定位于细胞核所必需的结构;SP-RING与SCE的结合相关;而SXS是SUMO蛋白的结合区域,可提高SUMO蛋白与连接酶E3的结合效率。
SUMO化修饰过程还涉及一类特殊的酶,即SUMO特异性蛋白酶或泛素样特异性蛋白酶。这类酶属于半胱氨酸蛋白酶,除参与SUMO前体蛋白的加工,使其成为Free-SUMO外,也参与靶蛋白的脱SUMO化,是维持SUMO化修饰动态、可逆平衡过程中必不可少的酶。这类酶在植物中的数量也相当丰富;在拟南芥中至少存在7个[45-46]。然而,相比拟南芥,一些植物中SUMO特异性蛋白酶的数量可能更加丰富,如在大豆中该酶至少有13个,分别为GmPROa、GmOTSa、GmOTSb1/2、GmB2a~e、GmESD4a~d[35]。因此,植物体内具有丰富的SUMO特异性蛋白酶,说明SUMO化修饰的动态、可逆平衡具有庞大的调控网络,有待研究探索。
2.2 SUMO化修饰的过程蛋白质的SUMOylation过程涉及一系列的酶促反应。首先,在ATP的作用下E1酶腺苷化Free-SUMO C端的甘氨酸,形成Free-SUMO~AMP中间体,并执行硫酯酰化功能,使得Free-SUMO与SAE2的半胱氨酸(Cys)结合并释放出AMP。随后,被E1酶活化后的Free-SUMO从SAE2的Cys上转移给结合酶E2,并与其Cys形成硫脂键,形成Free-SUMO~E2中间体。最后,在E3酶的作用下将Free-SUMO呈递给靶蛋白的赖氨酸(Lys)完成SUMOylation。然而,体外实验表明E2酶本身具有识别靶蛋白的能力,在E3酶不存在的情况下也可将Free-SUMO呈递给底物完成SUMOylation(图 1)[47-48]。
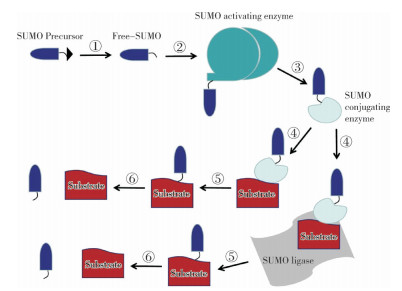
|
| ① SUMO特异性蛋白酶切割前体SUMO;② Free-SUMO被SUMO活化酶活化并与之结合;③活化后的Free-SUMO被呈递给SUMO结合酶;④和⑤ SUMO结合酶直接识别底物或在SUMO连接酶的促进下将FreeSUMO呈递给底物,使底物受到SUMO化修饰;⑥ SUMO特异性蛋白酶去除底物上的SUMO分子 ① SUMO-specific proteases digest SUMO precursors; ② Free-SUMO is activated and bound by SUMO activating enzymes; ③ Activated Free-SUMO is presented to SUMO conjugating enzymes; ④ and ⑤ SUMO conjugating enzymes directly recognize the substrates or present Free-SUMO to the substrates facilitated by SUMO ligases for SUMO modification; ⑥ SUMO-specific proteases remove SUMO molecules on the substrates 图 1 蛋白质SUMO化修饰过程[32] Fig. 1 Process of protein SUMOylation[32] |
目前,认为E2酶具有这种靶蛋白识别能力的原因主要有2种[49]:一是E2酶可特异性识别靶蛋白的空间结构;二是E2酶的C端富含正电荷,使其Cys可与靶蛋白的位点相结合。但至今还未有体内的实验证明,底物在体内的SUMO化修饰不需要E3酶介导,而有些底物在体外的SUMOylation仍依赖于E3酶。因此,E3酶在识别并促进底物SUMO化的过程中也起了至关重要的作用;此外,由于E3酶识别的底物通常含有一个保守特征序列[1],即yKXE/D (y代表疏水氨基酸,K是赖氨酸,X代表任意氨基酸,E是谷氨酸,D是天门冬氨基酸),可利用该特征预测蛋白质的SUMO化位点。不过,靶蛋白的SUMO化是一个动态、可逆的过程,即靶蛋白不仅能被SUMO化,也可被SUMO特异性蛋白酶催化脱SUMO化;如在酵母中存在2个SUMO特异性蛋白酶,分别为Ulp1和Ulp2,它们均可以在体外催化加工SUMO的羧基末端,去除以异肽键连接于靶蛋白上的SUMO分子,使靶蛋白脱SUMO化[17]。
综上,目前靶蛋白的SUMOylation动态、可逆过程可被概括为图 1:首先,SUMO蛋白被转录翻译为前体蛋白Pre-SUMO,并在SUMO特异性蛋白酶的作用下切割掉C端的一个小片段,暴露出diGly,成为有活性的Free-SUMO;接着,Free-SUMO先后通过硫酯键与E1酶和E2酶的Cys结合;随后,一些靶蛋白在体外可以不通过E3酶而直接被E2酶识别执行SUMOylation;但体外有些蛋白质或者在体内时,蛋白质的SUMOylation需要在E3酶的作用下才能将Free-SUMO从E2酶上转移到靶蛋白的Lys的ε-氨基上;此外,在SUMOylation过程中,SUMO特异性蛋白酶也可去除与靶蛋白结合的SUMO蛋白分子,使其脱SUMO化修饰。
3 SUMO化修饰底物的鉴定方法蛋白质翻译后修饰丰富了蛋白质的功能,而生物体内存在多种翻译后修饰。其中,泛素化和磷酸化等已被深入研究。然而,SUMOylation虽然在生物体内广泛存在,参与了多种多样的生命活动,如基因表达和代谢调控,但对其参与的主要生物途径尚不明确。因此,获悉生物体内SUMOylation的靶蛋白,可为了解SUMOylation参与的主要生物途径提供方向。
研究中首先需要鉴定出生物体内能被SUMOylation的蛋白质,而鉴定过程中利用的主要分析方法为质谱分析。但是,质谱分析过程中低丰度蛋白的鉴定成为难题,特别是具有SUMOylation的蛋白质相对于生物体内的总蛋白,大多数仍属于低丰度蛋白。因此,多位研究者利用了一些方法来富集SUMOylation的蛋白质,以期提高质谱分析过程中低丰度蛋白的量;也利用到多种涉及SUMOylation的蛋白质组学分析方法,继而挖掘更多相关信息。
3.1 亲和纯化鉴定SUMOylation底物目前,SUMO化修饰底物的富集方法以利用相关标签进行亲和纯化为主;如有研究以小鼠睾丸为材料,并利用SUMO1 C端连接在靶蛋白赖氨酸ε-氨基上的支链肽段作为抗原制备了SUMO1的多克隆抗体,继而通过该抗体富集被Trypsin/Lys-C酶消化后的SUMO1修饰肽段[50]。
然而,这些研究报道主要集中在动物领域,仅有少数在拟南芥中见到报道;包括Miller等[51]通过获取带His6或Flag或Tap标记的H98R(或T91R)突变型 SUMO1回补 atsumo1/atsumo2突变体的转基因植物,发现过表达 His6-AtSUMOH89R的 atsumo1/atsumo2突变体的转基因植物可正常生长发育、且完全可育,在热胁迫下的SUMO共轭物积累的量也与野生型最为接近,回补效果好,功能正常。因此,该团队选用过表达 His6-AtSUMOH89R的回补植物和野生型进行热处理和氧化胁迫处理;随后,提取正常生长、热处理和氧化胁迫处理的回补植物幼苗总蛋白作为试验组,而热胁迫处理的野生型幼苗总蛋白作为负对照;在变性条件下先后利用Ni柱子—AtSUMO1内源抗体—Ni柱子进行3步纯化,获取蛋白质洗脱液,并进行质谱分析,最后在去除背景下共发现357个候选蛋白,而利用 AtSUMO1H89R突变使得AtSUMO1H89R在胰蛋白酶处理后仅剩下~QTGG基序,以便于鉴定这些蛋白质的SUMO化修饰位点;但仅发现17个位点在14个靶蛋白分子上,而其中还包括了SUMO1和SIZ1,且它们贡献的位点达到5个,位点的鉴定效果不理想。
此外,Rytz等[43]也利用了同样的富集方法进行底物筛选,即通过Ni柱子—AtSUMO1内源抗体—Ni柱子的3步纯化法对热处理或无处理的拟南芥野生型、过表达 His6-AtSUMOH89R到 atsiz1-2、atmms21和 atsumo1/atsumo2突变体的转基因植物总蛋白进行提取和纯化,继而进行质谱分析来鉴定SUMO化修饰蛋白中属于AtSIZ1或者AtMMS21的靶蛋白,通过改进蛋白组学的分析仪器如质谱分析系统,使得鉴定到的SUMO化修饰候选蛋白达到1 058个,其中包含105个AtSIZ1的候选靶蛋白,而未发现到AtMMS21的靶蛋白。至此,罕见有报道进行类似的研究。
3.2 基于诱饵蛋白鉴定SUMOylation底物为鉴定与SUMO化修饰相关的底物,研究人员利用酵母双杂交的方法,并以SUMOylation过程中的相关酶作为诱饵蛋白进行筛选。例如,Garcia-Dominguez等[52]用截短的AtSIZ1(保留SAP、PHD和SP-RING结构域)作为诱饵蛋白鉴定到2个底物,GET3(Global transcription factor group E3)和GET5,并纯化了SUMO化修饰过程中的酶组分进行实验,发现GET3/5均是依赖于SIZ1的SUMO化修饰底物;而Elrouby等[53]以拟南芥的SUMO E2结合酶AtSCE1和SUMO特异性蛋白酶ESD4为诱饵,筛选拟南芥cDNA文库中的互作蛋白,结果鉴定到238个互作蛋白,并对其中134个蛋白质的SUMO化修饰情况进行检测,发现约92.54% 的蛋白质可被SUMOylation。
综上所述,虽然以往研究为鉴定生物体内的SUMOylation底物,开发了一些方法,并由此鉴定到部分SUMOylation底物,为系统了解SUMOylation的生物功能奠定了理论基础,但相比生物体内大量的蛋白质,其鉴定到的数量仍较有限,尤其是在作物方面的研究更是匮乏;此外,在植物体内鉴定SUMOylation底物时,若均通过构建转基因植物的方法[51],显然在多数植物中实现起来比较困难且耗时长,而利用酵母双杂交的方法[53],虽然缓解了时间限制,但这种属于间接的鉴定方式,选用诱饵蛋白对SUMOylation的底物进行筛选时,仅可鉴定到与之互作且同时具有SUMOylation的底物,对于不具备互作但能被SUMOylation的底物难以鉴定到,而一些存在互作但不被SUMOylation的蛋白质也会被包括在内,表明该方法也具有局限性。因此,开发一种适合普遍植物,且能对SUMOylation底物进行高效鉴定和分析的方法,对系统、准确地解析SUMO化修饰功能至关重要。
4 玉米蛋白质的SUMO化修饰研究概况目前,蛋白质SUMO化修饰的相关研究以拟南芥为主,但在玉米中类似的研究报道仍较少,究其原因是在玉米中还未有一套高效的蛋白质SUMO化修饰检测体系。因此,了解玉米体内的SUMO蛋白及其SUMO化修饰过程的相关酶类信息,尤其是梳理现阶段它们被报道的功能,将有助于厘清SUMO化修饰影响玉米生长发育及响应胁迫的规律,为后续玉米表型优化、选育提供指导基础。
4.1 玉米中的SUMO蛋白及其相关酶类玉米中的 SUMO基因[2]包括 ZmSUMO1a、ZmSUMO1b、ZmSUMO2、ZmDSUL和 ZmSUMO-v等5个(表 1),但 ZmSUMO1a和 ZmSUMO1b编码的蛋白氨基酸序列一致。因此,玉米中存在4种类型的SUMO蛋白,但每种类型仅有1个(图 2A),即ZmSUMO1(保守型)、ZmSUMO2(非保守型)、ZmDSUL(双SUMO型)和ZmSUMO-v(SUMO-v型)。其中,ZmSUMO1、ZmSUMO2和ZmDSUL的C端可暴露出diGly(图 2A),具有对底物进行SUMO化修饰的潜在能力。此外,ZmSUMO1编码的蛋白质与拟南芥的AtSUMO1同源性较高、三维结构类似(图 2B),已在体外被证明能对底物进行SUMOylation[2]。玉米中不同的SUMO蛋白除了C端diGly存在差异外,它们的三维结构也存在较大差异,暗示在玉米体内它们可能各自承担了特异功能。
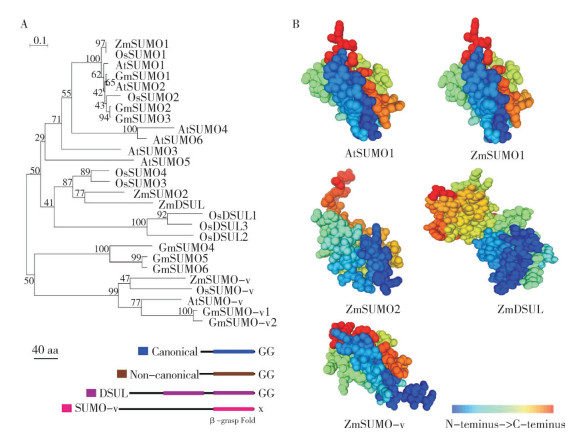
|
| A:玉米和其他物种的SUMO蛋白的系统发育树;不同类型的SUMO蛋白大小比例以玉米的4个SUMO蛋白为例绘制[2];B:利用SWISS-MODEL模型预测(http://swissmodel.expasy.org)预测蛋白质的三维结构;At、Gm、Os和Zm分别表示拟南芥、大豆、水稻和玉米的蛋白质 A: Phylogenetic tree of SUMO proteins in maize and other species; The size scales of different types of SUMO are set based on four SUMO proteins from maize; B: 3D structure prediction with SWISS-MODEL (http://swissmodel.expasy.org); Proteins with At, Gm, Os and Zm indicate proteins in Arabidopsis, soybean, rice and maize, respectively 图 2 ZmSUMOs的分子特征 Fig. 2 Molecular characterization of ZmSUMOs |
玉米中的E1酶大亚基 SAE2有2个转录本[2],分别编码了ZmSAE2a和ZmSAE2b这2个蛋白质,且体外实验表明ZmSAE2a在SUMOylation过程中具有功能;而E2酶包括了ZmSCE1a、ZmSCE1b、ZmSCE1c、ZmSCE1d、ZmSCE1e、ZmSCE1f和ZmSCE1g等7个[2],体外分析发现E2酶ZmSCE1b和ZmSCE1d参与了ZmSUMO1对底物的修饰过程,但ZmSCE1f需进行密码子优化后才能在大肠杆菌中正常工作,继而发挥功能。此外,E3酶[2]包括ZmSIZ1(ZmSIZ1a、ZmSIZ1b和ZmSIZ1c)、ZmMMS21和ZmPIAL1,而SUMO特异性蛋白酶有2种类型,分别为ZmESD4(ZmESD4a、ZmESD4b、ZmESD4c和ZmESD4d)以及ZmOST1(ZmOST1a、ZmOST1b、ZmOST1c、ZmOST1d和ZmOST1e)。随后,关于玉米SUMO化修饰相关的SUMO蛋白以及相关酶类的功能陆续被报道。
4.2 玉米SUMO蛋白及其相关酶类功能的研究进展通过构建 ZmDSUL-RNAi,并将其引入到玉米的精细胞中,发现精细胞的胞质分裂受阻,在合子中会出现未分离的子核,且胚胎细胞也会由于缺少细胞板而出现多核的现象,表明ZmDSUL对玉米早期的胚胎发育有着重要影响[21]。另有研究指出,过表达 ZmSUMO1会导致玉米植株矮小和花器官结构异常;因此,玉米的营养生长和生殖发育需要高度保守的ZmSUMO1与目标蛋白的精确微调缀合,过表达或敲除引起的错误调控对玉米的育性均有影响[22]。近年,玉米E2酶的功能也陆续被研究报道,如过表达 ZmSCE1d的转基因拟南芥植物和过表达 ZmSCE1e的转基因烟草植物,均增加了植物体内的SUMO共轭物,并均可提高植物的抗旱性[24];过表达 ZmSCE1b的转基因拟南芥植物相比拟南芥野生型对除草剂百草枯更加耐受[25]。此外,玉米SUMO化修饰过程中一个重要的酶组分SUMO E3连接酶ZmSIZ1[26, 55]] 和ZmMMS21[56]也被认为参与了玉米的生长发育、病害防御和胁迫响应过程;而Liang等[57]在对玉米小肽进行全基因组鉴定时,发现这些小肽在玉米的生长发育和非生物胁迫响应中均可能发挥了重要作用,这些小肽中也包括了ZmSUMO1,表明ZmSUMO1可能参与了玉米多种多样的生命活动,对调控玉米正常生长发育至关重要。至此,罕见其他玉米SUMO蛋白及其相关酶类的功能报道,尤其是关于玉米蛋白质SUMO化修饰底物的研究更少。
4.3 玉米SUMO化底物的研究进展现阶段,在玉米中鲜见对相关蛋白质受SUMOylation后的分子机理进行深入研究,且SUMOylation靶蛋白的信息缺乏,仅1篇报道确定了玉米SUMO化修饰底物的信息,即Zhang等[56]发现ZmMMS21可与染色质组织体SMC5/6复合物中的ZmSMC5作用并促使其SUMO化修饰,参与DNA损伤修复过程。
为快速鉴定玉米SUMOylation底物,Lai等[26, 54]构建了玉米SUMOylation蛋白质体外检测体系,并以ZmSUMO1作为SUMO蛋白分子为例,形成了基于大肠杆菌高通量鉴定玉米cDNA文库中潜在SUMOylation底物的体系,并鉴定到200多个候选底物,且经SUMO化修饰预测,有93% 以上的蛋白具有SUMO化修饰位点。此外,为进一步检验研究方法的可靠性,对20个候选底物进行了体外验证,准确率达到95%,表明该方法可靠、高效。作为一种新型的鉴定方法,该方法克服了以往底物鉴定方法的一些局限性,可适用于更多物种的SUMO化修饰底物鉴定,将为SUMO化修饰底物鉴定提供重要的指导方向。
5 结语与展望玉米作为我国种植面积最大的作物,提高其产量和品质是育种者一致秉承的目标,而SUMOylation参与了植物许多重要的生命活动,对其正常的生长发育和应对外界胁迫至关重要。但玉米的SUMOylation研究力度尚浅,且不仅是玉米,其他植物中关于SUMOylation底物的信息报道较少。有幸的是,目前关于SUMOylation相关酶类的功能相继被报道,同时也涌现了一些底物鉴定方法;尤其是基于大肠杆菌高通量鉴定玉米cDNA文库中潜在SUMOylation底物体系的成功建立,为未来不限于玉米在内的其他物种的SUMOylation研究提供了新方向;该方法具有以下优势:(1)不需要构建转基因植物和使用特异性抗体,可以广泛应用到其他植物研究中;(2)基于cDNA库,可能结合单细胞和空间转录组对在一个特定的组织,甚至在一个细胞中的SUMO化底物进行针对性鉴定;(3)由于大肠杆菌中不存在SUMO蛋白酶,因此SUMO共轭物,在目前的系统中会更稳定,有利于进行后续的纯化。因此,未来利用该方法将研究者挖掘到更多的SUMOylation底物。
再者,运用多组学分析时,可利用各种高通量技术,如基因组学、转录组学、蛋白质组学和代谢组学等来展开蛋白质研究。通过整合不同层面的数据,可获得更全面的信息,并揭示不同层次之间的相互关系和调控网络。因此,虽然目前SUMOylation中利用多组学研究的信息较少,但随着越来越多的SUMOylation底物被发掘到,利用多组学分析来阐明这些蛋白之间的联系将是一种重要的研究手段,可为我们揭示SUMOylation在生物生命活动中扮演的主要角色提供重要线索[58-59]。例如,Wang等[60]利用转录组和代谢组分析的整合揭示了氨基酸和碳水化合物代谢途径在OsDRAP1介导水稻耐盐性中起到关键作用;而在花生中,Li等[61]对粉红色和紫色种皮花生品种中的差异代谢物进行分析,并通过多组学联合分析鉴定到唯一差异积累的代谢产物氰化氢3-O-葡萄糖苷(Kuromanin,pmb0550),为深入研究花生种皮花青素代谢机制提供了重要资料;类似地,Cai等[62]基于转录组学、蛋白质组学和代谢组学,在玉米中鉴定到与苯丙烷生物合成相关的基因,并暗示这些基因可能与tc19玉米品种的大颗粒表型有关。
综上,多组学联合分析在某些方面已经取得了成果,未来利用Lai等[26, 54]] 建立的SUMOylation鉴定体系,并将多组学联合分析应用到SUMOylation研究中是值得被考虑的手段,而这也将有助于我们解析SUMOylation影响玉米生命活动的规律,为玉米、甚至其他植物表型优化提供重要的指导。
| [1] |
STERNSDORF T, JENSEN K, FREEMONT P S. Sumo[J]. Current Biology, 2003, 13(7): R258-R259. DOI:10.1016/S0960-9822(3)00193-3 |
| [2] |
AUGUSTINE R C, YORK S L, RYTZ T C, VIERSTRA R D. Defining the SUMO System in Maize: SUMOylation is up-regulated during endosperm development and rapidly induced by stress[J]. Plant Physiology, 2016, 171(3): 2191-2210. DOI:10.1104/pp.16.00353 |
| [3] |
HONG Y, CHEN Y, SHI H, KONG X, YAO J, LEI M, ZHU J K, WANG Z. SUMO E3 ligase SIZ1 negatively regulates arsenite resistance via depressing GSH biosynthesis in Arabidopsis[J]. Environmental and Experimental Botany, 2022, 2(1): 118-124. DOI:10.1007/s44154-021-00029-8 |
| [4] |
JIANG J, XIE Y, DU J, YANG C, LAI J. A SUMO ligase OsMMS21 regulates rice development and auxin response[J]. Journal of Plant Physiology, 2021, 263: 153447. DOI:10.1016/j.jplph.2021.153447 |
| [5] |
LIU F, WANG X, SU M, YU M, ZHANG S, LAI J, YANG C, WANG Y. Functional characterization of DnSIZ1, a SIZ/PIAS-type SUMO E3 ligase from dendrobium[J]. BMC Plant Biology, 2015, 15(1): 1-14. DOI:10.1186/s12870-015-0613-3 |
| [6] |
ZHANG C L, WANG G L, ZHANG Y L, HU X, ZHOU L J, YOU C X, LI Y Y, HAO Y J. Apple SUMO E3 ligase MdSIZ1 facilitates SUMOylation of MdARF8 to regulate lateral root formation[J]. New Phytologist, 2021, 229(4): 2206-2222. DOI:10.1111/nph.16978 |
| [7] |
SALMAN A, KOTB A, GHAZY A I, IBRAHIM E I, AL-ATEEQ T K. Structural and functional characterization of tomato SUMO1 gene[J]. Saudi Journal of Biological Sciences, 2020, 27(1): 352-357. DOI:10.1016/j.sjbs.2019.10.004 |
| [8] |
吕科良, 程春振, 陈裕坤, 林玉玲, 郑诚乐, 陈桂信, 赖钟雄. 荔枝SUMO活化酶基因 LcSAE2的克隆及表达分析[J]. 园艺与种苗, 2016(11): 35-38. DOI:10.16530/j.cnki.cn21-1574/s.2016.11.012 LYU K L, CHENG C Z, CHEN Y K, LIN Y L, ZHENG C L, CHEN G X, LAI Z H. Cloning and expression analysis of LcSAE2, a SUMO-activating enzyme gene from Lychee[J]. Horticulture and Seedling, 2016(11): 35-38. DOI:10.16530/j.cnki.cn21-1574/s.2016.11.012 |
| [9] |
LU X, YANG L, YU M, LAI J, WANG C, MCNEIL D, ZHOU M, YANG C. A novel Zea mays ssp. mexicana L. MYC-type ICE-like transcription factor gene ZmmICE1, enhances freezing tolerance in transgenic Arabidopsis thaliana[J]. Plant Physiology Biochemistry, 2017, 113: 78-88. DOI:10.1016/j.plaphy.2017.02.002 |
| [10] |
GUO J, WANG S, WANG G, LU R, WANG Y, GUO Y, JI W. Overexpression of GmSUMO2 gene confers increased abscisic acid sensitivity in transgenic soybean hairy roots[J]. Molecular Biology Reports, 2020, 47(5): 3475-3484. DOI:10.1007/s11033-020-05433-3 |
| [11] |
LIU Y, ZHU J, SUN S, CUI F, HAN Y, PENG Z, ZHANG X, WAN S, LI G. Defining the function of SUMO system in pod development and abiotic stresses in Peanut[J]. BMC plant biology, 2019, 19(1): 1-15. DOI:10.1186/s12870-019-2136-9 |
| [12] |
GHIMIRE S, TANG X, ZHANG N, LIU W, QI X, FU X, SI X. Genomic analysis of the SUMO-conjugating enzyme and genes under abiotic stress in potato (Solanum tuberosum L.)[J]. International Journal of Genomics, 2020, 9703638. DOI:10.1155/2020/9703638 |
| [13] |
ZHANG Y, LYU S, HU Z, YANG X, ZHU H, DENG S. Identification and functional characterization of the SUMO system in sweet potato under salt and drought stress[J]. Plant Science, 2023, 330: 111645. DOI:10.1016/j.plantsci.2023.111645 |
| [14] |
LEI S, WANG Q, CHEN Y, SONG Y, ZHENG M, HSU Y. Capsicum SIZ1 contributes to ABA-induced SUMOylation in pepper[J]. Plant Science, 2022, 314: 111099. DOI:10.1016/j.plantsci.2021.111099 |
| [15] |
MÜLLER S, HOEGE C, PYROWOLAKIS G, JENTSCH S. SUMO, ubiquitin's mysterious cousin[J]. Nature Reviews Molecular Cell Biology, 2001, 2(3): 202-210. DOI:10.1038/35056591 |
| [16] |
SCHWARTZ D C, HOCHSTRASSER M. A superfamily of protein tags: ubiquitin, SUMO and related modifiers[J]. Trends in Biochemical Sciences, 2003, 28(6): 321-328. DOI:10.1016/S0968-0004(03)00113-0 |
| [17] |
LI S J, HOCHSTRASSER M. A new protease required for cellcycle progression in yeast[J]. Nature, 1999, 398(6724): 246-251. DOI:10.1038/18457 |
| [18] |
TRULSSON F, VERTEGAAL A. Site-specific proteomic strategies to identify ubiquitin and SUMO modifications: Challenges and opportunities[J]. Seminars in Cell & Developmental Biology, 2021, 132: 97-108. DOI:10.1016/j.semcdb.2021.11.006 |
| [19] |
MAMATHA H, MEENA M K, KUMAR P C. Quality protein maize (QPM) as balance nutrition for human diet[J]. Advances in Plants and Agriculture Research, 2017, 6(2): 33-35. DOI:10.15406/apar.2017.06.00205 |
| [20] |
SCHNABLE P S, WARE D, FULTON R S, STEIN J C, WEI F, PASTERNAK S, LIANG C, ZHANG J, FULTON L, GRAVES T A, et al. The B73 maize genome: Complexity, diversity, and dynamics[J]. Science, 2009, 326(5956): 1112-1115. DOI:10.1126/science.1178534 |
| [21] |
CHEN J, KALINOWSKA K, MÜLLER B, MERGNER J, DEUTZMANN R, SCHWECHHEIMER C, HAMMES U Z, DRESSELHAUS T. DiSUMO-LIKE interacts with RNA-binding proteins and affects cell-cycle progression during maize embryogenesis[J]. Current Biology, 2018, 28(10): 1548-1560. DOI:10.1016/j.cub.2018.03.066 |
| [22] |
CHEN J, MÜLLER B, WANG H, DRESSELHAUS T. Overexpression of SUMO1 located predominately to euchromatin of dividing cells affects reproductive development in maize[J]. Plant Signaling & Behavior, 2019, 14(5): e1588664. DOI:10.1080/15592324.2019.1588664 |
| [23] |
WANG H, WANG M, XIA Z. The maize class-I SUMO conjugating enzyme ZmSCE1d is involved in drought stress response[J]. International Journal of Molecular Sciences, 2019, 21(1): 29-43. DOI:10.3390/ijms21010029 |
| [24] |
WANG H, WANG M, XIA Z. Overexpression of a maize SUMO conjugating enzyme gene (ZmSCE1e) increases Sumoylation levels and enhances salt and drought tolerance in transgenic tobacco[J]. Plant Science, 2019, 281: 113-121. DOI:10.1016/j.plantsci.2019.01.020 |
| [25] |
WANG H, XU D, ZHU X, WANG M, XIA Z. The maize SUMO conjugating enzyme ZmSCE1b protects plants from paraquat toxicity[J]. Ecotoxicology and Environmental Safety, 2021, 211: 111909. DOI:10.1016/j.ecoenv.2021.111909 |
| [26] |
LAI R, JIANG J, WANG J, DU J, LAI J, YANG C. Functional characterization of three maize SIZ/PIAS-type SUMO E3 ligases[J]. Journal of Plant Physiology, 2022, 268: 153588. DOI:10.1016/j.jplph.2021.153588 |
| [27] |
MELUH P B, KOSH L D. Evidence that the MIF2 gene of Saccharomyces cerevisiae encodes a centromere protein with homology to the mammalian centromere protein CENP-C[J]. Molecular Biology of the Cell, 1995, 6(7): 793-807. DOI:10.1091/mbc.6.7.793 |
| [28] |
YOSHIMITSU T, JUNYA M, AKIO T E, YOSHIKO K. Yeast Ulpl, an Smt3-specific protease, associates with nucleoporins[J]. The Journal of Biochemistry, 2000, 128(5): 723-725. DOI:10.1093/oxfordjournals.jbchem.a022807 |
| [29] |
MATUNIS M J, COUTAVAS E, BLOBEL G. A novel ubiquitin-like modification modulates the partitioning of the Ran-GTPase-activating protein RanGAP1 between the cytosol and the nuclear pore complex[J]. The Journal of Cell Biology, 1996, 135(6): 1457-1470. DOI:10.1083/jcb.135.6.1457 |
| [30] |
NOVATCHKOVA M, BACHMAIR A, EISENHABER B, EISENHABER F. Proteins with two SUMO-like domains in chromatin-associated complexes: the RENi (Rad60-Esc2-NIP45) family[J]. BMC Bioinformatics, 2005, 6(1): 1-9. DOI:10.1186/1471-2105-6-22 |
| [31] |
NOVATCHKOVA M, BUDHIRAJA R, COUPLAND G, EISENHABER F. SUMO conjugation in plants[J]. Planta, 2004, 220: 1-8. DOI:10.1007/s00425-004-1370-y |
| [32] |
NOVATCHKOVA M, TOMANOV K, HOFMANN K, STUIBLE H P, BACHMAIR A. Update on sumoylation: defining core components of the plant SUMO conjugation system by phylogenetic comparison[J]. New Phytologist, 2012, 195(1): 23-31. DOI:10.1111/j.1469-8137.2012.04135.x |
| [33] |
ULMAN A, LEVIN T, DASSA B, JAVITT A, KACEN A, SHMUELI M D, EISENBERG-LERNER A, SHEBAN D, FISHLLEVICH S, LEVY ED, MERBL Y. Altered protein abundance and localization inferred from sites of alternative modification by ubiquitin and SUMO[J]. Journal of Molecular Biology, 2021, 433(21): 167219. DOI:10.1016/j.jmb.2021.167219 |
| [34] |
ZHENG Y, CHEN Z, MA L, LIAO C. The ubiquitin E3 ligase RHA2b promotes degradation of MYB30 in abscisic acid signaling[J]. Plant Physiology, 2018, 178: 428-440. DOI:10.1104/pp.18.00683 |
| [35] |
LI Y, WANG G, XU Z, LI Z, SUN M, GUO Z, JI W. Organization and regulation of soybean SUMOylation system under abiotic stress conditions[J]. Frontiers in Plant Science, 2017, 8: 1458. DOI:10.3389/fpls.2017.01458 |
| [36] |
BOHREN K M, NADKARNI V, SONG J H, GABBAY K H, OWERBACH D. A M55V polymorphism in a novel SUMO gene (SUMO-4) differentially activates heat shock transcription factors and is associated with susceptibility to type I diabetes mellitus[J]. Journal of Biological Chemistry, 2004, 279(26): 27233-27238. DOI:10.1074/jbc.M402273200 |
| [37] |
JOO J, DONG H C, SANG H K, SANG I S. Cellular Localization of rice SUMO/SUMO conjugates and in vitro sumoylation using rice components[J]. Rice Science, 2020, 27(1): 1-4. DOI:10.1016/j.rsci.2019.02.003 |
| [38] |
SARACCO S A, MILLER M J, KUREPA J, VIERSTRA R D. Genetic analysis of SUMOylation in Arabidopsis: conjugation of SUMO1 and SUMO2 to nuclear proteins is essential[J]. Plant Physiology, 2007, 145(1): 119-134. DOI:10.1104/pp.107.102285 |
| [39] |
LI S, LIN M, WANG J, ZHANG L, LIN M, HU Z, QI Z, JIANG H, FU Y, XIN D, LIU D, CHEN Q. Regulation of soybean SUMOylation system in response to Phytophthora sojae infection and heat shock[J]. Plant Growth Regulation, 2019, 87: 69-82. DOI:10.1007/s10725-018-0452-y |
| [40] |
JOO J, DONG H C, LEE Y H, SEO H S, SONG S I. The rice SUMO conjugating enzymes OsSCE1 and OsSCE3 have opposing effects on drought stress[J]. Journal of Plant Physiology, 2019, 240: 152993. DOI:10.1016/j.jplph.2019.152993 |
| [41] |
HAN Y, ZHAO Q, DANG L, LUO Y, CHEN S, SHAO C, HUANG H, LI Y, LI L, CAI T, CHEN S, HE X. The SUMO E3 ligase-like proteins PIAL1 and PIAL2 interact with MOM1 and form a novel complex required for transcriptional silencing[J]. The Plant Cell, 2016, 28: 1215-1229. DOI:10.1105/tpc.15.00997 |
| [42] |
ISHIDA T, YOSHIMURA M, MIURA K, SUGIMOTO K. MMS21/HPY2 and SIZ1, two Arabidopsis SUMO E3 ligases, have distinct functions in development[J]. PLoS ONE, 2012, 7: e46897. DOI:10.1371/journal.pone.0046897 |
| [43] |
RYTZ T C, MILLER M J, MCLOUGHLIN F, AUGUSTINE R C, MARSHALL R S, JUAN Y T, CHARNG Y, SCALF M, SMITH L M, VIERSTRA R D. SUMOylation profiling reveals a diverse array of nuclear targets modified by the SUMO ligase SIZ1 during heat stress[J]. The Plant Cell, 2018, 30(5): 1077-1099. DOI:10.1105/tpc.17.00993 |
| [44] |
秦圣豪, 肖丽娜, 李荣冲, 田丽彬, 韩燕, 崔凤, 李国卫, 刘译阳, 万书波. 花生 AhSIZ1基因的克隆及表达特性分析[J]. 分子植物育种, 2020, 18(23): 7648-7653. DOI:10.13271/j.mpb.018.007648 QIN S H, XIAO L N, LI R C, TIAN L B, HAN Y, CUI F, LI G W, LIU Y Y, WAN S B. Cloning and expressing analysis of AhSIZ1 gene in peanut[J]. Molecular Plant Breeding, 2020, 18(23): 7648-7653. DOI:10.13271/j.mpb.018.007648 |
| [45] |
CONTI, PRICE G, O'DONNELL E, SCHWESSINGER B, DOMINY P, SADANANDOM A. Small ubiquitin-like modifier proteases overly tolerant tO SALT1 and -2 regulate salt stress responses in Arabidopsis[J]. The Plant Cell, 2008, 20: 2894-2908. DOI:10.1105/tpc.108.058669 |
| [46] |
MIURA K., HASEGAWA P M. SUMOylation and other ubiquitin-like post-translational modifications in plants[J]. Trends in Cell Biology, 2010, 20(4): 223-232. |
| [47] |
HAY R T. Protein modification by SUMO[J]. Trends in Biochemical Sciences, 2001, 26: 332-333. DOI:10.1146/annurev.biochem.73.011303.074118 |
| [48] |
DESTERRO J M P, RODRIGUEZ M S, KEMP G D, HAY R T. Identification of the enzyme required for activation of the small ubiquitin-like protein SUMO-1[J]. Journal of Biological Chemistry, 1999, 274(15): 10618-10624. DOI:10.1016/S1097-2765(00)80133-1 |
| [49] |
SAMPSON D A, WANG M, MATUNIS M J. The small ubiquitin-like modifier-1(SUMO-1) consensus sequence mediates Ubc9 binding and is essential for SUMO-1 modification[J]. Journal of Biological Chemistry, 2001, 276(24): 21664-21669. DOI:10.1074/jbc.M100006200 |
| [50] |
CAI L, TU J, SONG L, GAO Z, LI K. Proteome-wide map of endogenous SUMOylation sites in mouse testis[J]. Molecular & Cellular Proteomics, 2017, 16(5): 717-727. DOI:10.1074/mcp.M116.062125 |
| [51] |
MILLER M J, BARRETT-WILT G A, HUA Z, VIERSTRA R D. Proteomic analyses identify a diverse array of nuclear processes affected by small ubiquitin-like modifier conjugation in Arabidopsis[J]. Proceedings of the National Academy of Sciences, 2010, 107(38): 16512-16517. DOI:10.1073/pnas.1004181107 |
| [52] |
GARCIA-DOMINGUEZ M, MARCH-DIAZ R, REYES J C. The PHD domain of plant PIAS proteins mediates sumoylation of bromodomain GTE proteins[J]. Journal of Biological Chemistry, 2008, 283(31): 21469-21477. DOI:10.1074/jbc.M708176200 |
| [53] |
ELROUBY N, COUPLAND R. Proteome-wide screens for small ubiquitin-like modifier (SUMO) substrates identify Arabidopsis proteins implicated in diverse biological processes[J]. Proceedings of the National Academy of Sciences, 2010, 107(40): 17415-17420. DOI:10.1073/pnas.1005452107 |
| [54] |
LAI R, LI W, XU Z, LIU W, ZENG Q, LIN W, JIANG J, LAI J, YANG C. A robust method for identification of plant SUMOylation substrates in a library-based reconstitution system[J]. Plant Communications, 2023. DOI:10.1016/j.xplc.2023.100573 |
| [55] |
LIAO X, SUN J, LI Q, DING W, ZHAO B, WANG B, ZHOU S, WANG H. ZmSIZ1a and ZmSIZ1b play an indispensable role in resistance against Fusarium ear rot in maize[J]. Molecular Plant Pathology, 2023, 1-14. DOI:10.1111/mpp.13297 |
| [56] |
ZHANG J, AUGUSTINE R C, SUZUKI M, FENG J, CHAR S N, YANG B, MCCARTY D R, VIERSTRA R D. The SUMO ligase MMS21 profoundly influences maize development through its impact on genome activity and stability[J]. PLoS Genetics, 2021, 17(10): e1009830. DOI:10.1371/journal.pgen.1009830 |
| [57] |
LIANG Y, ZHU W, CHEN S, QIAN J, LI L. Genome-wide identification and characterization of small peptides in maize[J]. Frontiers in Plant Science, 2021, 12: 695439. DOI:10.3389/fpls.2021.695439 |
| [58] |
李洁, 姚晓华. 多组学关联分析作物耐逆境胁迫研究进展[J]. 广东农业科学, 2019, 46(8): 22-28. DOI:10.16768/j.issn.1004-874X.2019.08.004 LI J, YAO X H. Progress in the study of crop tolerance to adversity stress by multi-omics correlation analysis[J]. Guangdong Agricultural Sciences, 2019, 46(8): 22-28. DOI:10.16768/j.issn.1004-874X.2019.08.004 |
| [59] |
季元, 于冰, 陈偲学. 多组学技术在植物应答非生物胁迫中的研究进展[J]. 中国农学通报, 2023, 39(23): 1-7. DOI:10.11924/j.issn.1000-6850.casb2023-0059 JI Y, YU B, CHEN S X. Research progress of multi-omics technologies in plant response to abiotic stress[J]. Chinese Agricultural Science Bulletin, 2023, 39(23): 1-7. DOI:10.11924/j.issn.1000-6850.casb2023-0059 |
| [60] |
WANG Y, HUANG L, DU F, WANG J, ZHAO X, LI Z, WANG W, XU J, FU B. Comparative transcriptome and metabolome profiling reveal molecular mechanisms underlying OsDRAP1-mediated salt tolerance in rice[J]. Scientific Reports, 2021, 11(1): 5166. DOI:10.1038/s41598-021-84638-3 |
| [61] |
LI J, MA Y, HU M, ZHAO Y, LIU B, WANG C, ZHANG M, ZHANG L, YANG X, MU G. Multi-omics and mirna interaction joint analysis highlight new insights into anthocyanin biosynthesis in peanuts (Arachis hypogaea L.)[J]. Frontiers in Plant Science, 2022, 13: 818345. DOI:10.3389/fpls.2022.818345 |
| [62] |
CAI Q, JIAO F, WANG Q, ZHANG E, SONG X, PEI Y, LI J, ZHAO M, GUO X. Multiomics comparative analysis of the maize large grain mutant tc19 identified pathways related to kernel development[J]. BMC Genomics, 2023, 24(1): 537. DOI:10.1186/s12864-023-09567-z |
(责任编辑 马春敏)
 2023, Vol. 50
2023, Vol. 50