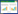文章信息
基金项目
- 海南省自然科学基金(319MS009);海南省教育厅项目(Hys2020-242);海南大学横向课题(HDKYH-2023093);襄阳市农业科学院特色产业科普基地项目(XAAS2022025)
作者简介
- 张婷婷(1997—),女,硕士,研究实习员,研究方向为园艺植物抗逆分子生物学,E-mail:18327852733@163.com.
通讯作者
- 凌冬(1973—),男,正高级农艺师,研究方向为作物育种学,E-mail:125845861@qq.com.
文章历史
- 收稿日期:2023-08-09
2. 海南大学热带农林学院(农业农村学院、乡村振兴学院)/海南省热带园艺作物品质调控重点实验室,海南 海口 570228
2. School of Tropical Agriculture and Forestry (School of Agricultural and Rural Affairs, School of Rural Revitalization), Hainan University/Key Laboratory for Quality Regulation of Tropical Horticultural Crops of Hainan Province, Haikou 570228, China
【研究意义】植物因其固着的特性,在生长过程中经常会遭受来自外界不利环境条件的影响。随着土地盐渍化的加重,盐害已成为威胁植物生长发育的因素之一,使得植物很难吸收养分和水分,种子萌发和植物生长受到抑制、发育缓慢,从而引发离子毒害、低钾胁迫等,最终造成植物死亡[1-3]。铁皮石斛(Dendrobium catenatum Lindl.)是兰科石斛属植物,药用价值高[4],被誉为“九大仙草”之首,其生长发育、活性成分的积累以及药用价值受非生物胁迫的影响[5]。研究发现在盐浓度过高(≥ 200 mmol/L)时,铁皮石斛次生代谢物和总多糖含量减少,影响其营养价值和药用价值[6]。因此,研究铁皮石斛在高盐胁迫下的耐盐机理及耐盐调控途径,对培育铁皮石斛耐盐新品种、提升其药用价值有重要意义。【前人研究进展】Ca2+作为植物体内的第二信使可感应外界胁迫信号并在植物体内进行传导,植物在受到非生物胁迫时,细胞内的钙离子迅速积累,激活钙离子传感器(Calcineurin B-like,CBL),CBL蛋白通过与靶蛋白激酶CIPK(CBL-interacting protein kinase)结合,进而传递信号,参与植物应答非生物胁迫。CBL-CIPK信号通路在植物应答非生物胁迫过程中具有重要作用[7]。CIPK24蛋白激酶与类钙调磷酸酶B蛋白CBL4共同组成的SOS途径是植物中最早发现的参与调控盐胁迫的CBL-CIPK信号途径[8],之后又发现AtCBL10能够与AtCIPK24相互作用激活AtSOS1参与植物响应盐胁迫[9]。胡杨PeCBL1能够与PeCIPK24相互作用参与植物Na+/K+稳态的调节[10];通过转基因技术发现小麦TaCIPK24能够通过增强转基因拟南芥活性氧清除能力来提高转基因拟南芥的耐盐能力[11];同样在苹果中发现,MdSOS2L1能够通过清除活性氧、诱导原花青素和苹果酸的合成来增强苹果的耐盐性,并将其应用至番茄耐盐中[12];而在番茄中利用BiFC和pull-down实验证明SlCBL4和SlCIPK24能够相互作用,过表达 SlCIPK24的植株与野生型相比具有更高的耐盐性[13]。【本研究切入点】前期研究发现,铁皮石斛 DcCIPK24在盐胁迫下呈上调表达[14],在拟南芥中过表达 DcCIPK24基因,发现在干旱胁迫和盐胁迫下,转基因拟南芥生长状态要明显好于野生型拟南芥,进一步研究发现 DcCIPK24能够通过清除活性氧正向调控转基因拟南芥耐旱与耐盐能力[15],但关于其参与耐盐性的调控机制,尤其是对其下游调控基因的研究尚不清楚。【拟解决的关键问题】本文在前期获得转基因拟南芥的基础上,对盐胁迫下两个转基因拟南芥株系和野生型拟南芥进行转录组测序,挖掘 DcCIPK24在拟南芥中过表达后盐胁迫响应途径中的上调表达基因,为深入研究DcCIPK24响应盐胁迫的调控机制提供指导。
1 材料与方法 1.1 试验材料转 DcCIPK24拟南芥种子由海南省热带园艺作物品质调控重点实验室保存,选取遗传稳定性较好的2个T3代纯系转基因拟南芥株系(标记为T1、T2)与野生型(WT)拟南芥种子春化后,播种于1/2 MS固体培养基上,平放至光照培养箱(22±1 ℃,光照/ 黑暗时长16 h/8 h)中培养8~10 d,移至基质(营养土与蛭石1∶2混合)中,继续培养1个月(22±1 ℃,光照/ 黑暗时长16 h/8 h),以备后续处理。
1.2 盐胁迫下拟南芥转录组测序选取长势一致的1月龄野生型和转基因拟南芥各8盆(每盆4株),放于同一托盘中,处理组每5 d浇1.5 L 200 mmol/L NaCl溶液,对照组每5 d浇1.5 L自来水,3次重复。处理10 d时,取野生型、转基因拟南芥叶片各2份,每份0.5 g。一份送往北京诺禾致源科技股份有限公司进行转录组测序,一份于液氮中速冻后置于-80 ℃冰箱中保存,以备提取RNA。每个株系每个处理3次重复。转录组测序所需的RNA提取、建库均在公司完成,采用illumina NovaSeq 6000(Illumina, USA)测序平台进行测序分析。
1.3 数据分析利用DESeq2(1.20.0)软件筛选不同处理下表达水平显著差异的基因,以|log2FC|≥1且 P ≤ 0.05作为标准,最终在不同组合之间筛选出差异表达基因,用于后续分析[16];使用TBtools软件绘制样本间相关热图、差异基因表达热图和维恩图[17];利用clusterProfiler(3.8.1)软件进行差异表达基因的GO功能富集分析和KEGG通路分析[16];使用Excel 2011进行绘图。
1.4 荧光定量PCR验证采用植物总RNA提取试剂盒(天根生化科技(北京) 有限公司,DP437)提取转基因拟南芥和野生型拟南芥叶片RNA,利用反转录试剂盒〔宝生物工程(大连) 有限公司,RR047A〕合成cDNA第一链[18]。根据盐处理下转基因株系与野生型株系的差异基因表达水平变化,选择受盐胁迫诱导明显上调〔AtGAPT5(AT3G11430)、AtSIRK(AT2G19190)、AtMEE25(AT2G34850)、AtUGE5(AT4G10960)〕或下调〔AtAMT1-2(AT1G64780)、AtATTI3(AT2G43530)、AtCYP71A25(AT3G48280)、AtLHCB4.3(AT2G40100)〕的差异基因,以 AtACT2基因作为内标基因[19],利用ChamQTM Universal SYBR qPCR Master Mix(诺唯赞生物科技有限公司,7E280C8)进行qRT-PCR验证,基因相对表达量按照2-ΔΔCT法进行计算,结果利用Excel 2011软件绘图。引物序列见表 1。
2 结果与分析 2.1 转录组数据组装与检测
为保证数据的可靠性,使用fastp(Version 0.19.7)软件对数据进行过滤,去除带有测序接头的序列、无法确定碱基信息的序列以及低质量序列。过滤后reads在39 703 424~49 318 124之间,过滤后碱基数在5.96~7.40 G之间,GC含量在45.5%~46.71% 之间,整体错误率均小于0.05(表 2),表明数据可靠。
2.2 样本相关性分析
为证明样本基因表达数据的可靠性,对样本间基因表达水平进行相关性分析。以皮尔逊相关系数的平方(R2)作为检验标准,R2越接近1,则说明样品之间表达模式的相似性越高。如图 1所示,样品组内 R2均大于0.8,说明数据相关性较高,数据可靠。
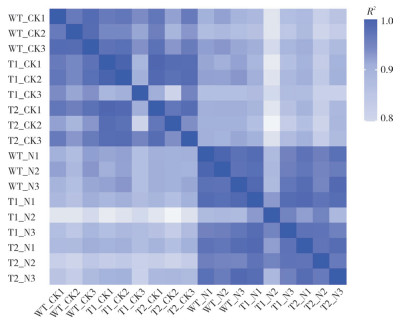
|
| WT-CK:野生型拟南芥正常培养;T1-CK、T2-CK:转基因株系T1、T2正常培养;WT-N:200 mmol/L NaCl处理的野生型拟南芥;T1-N、T2-N:200 mmol/L NaCl处理的转基因株系T1、T2;样本名称中的数字代表重复 WT-CK: Wide-type Arabidopsis cultured under normal conditions; T1-CK, T2-CK: Transgenic Arabidopsis lines T1 and T2 cultured under normal conditions; WT-N: Wide-type Arabidopsis cultured under 200 mmol/L NaCl treatment; T1-N, T2-N: Transgenic Arabidopsis lines T1 and T2 cultured under 200 mmol/L NaCl treatment; Numbers in the sample name represent the repetition 图 1 样本间相关性热图 Fig. 1 Correlation heatmap among the samples |
2.3 差异表达基因分析
由图 2可知,盐处理前后转基因拟南芥和野生型拟南芥中的基因表达变化差异显著。不同组合之间差异表达基因总数、上调和下调数统计如 图 3所示,T1-N vs T1-CK、T2-N vs T2-CK、WT-N vs WT-CK 3个组合的差异基因数较多,说明盐胁迫引起了拟南芥中基因表达量的变化。通过比较转基因拟南芥(T1和T2)和野生型拟南芥中的差异基因,发现在正常条件下,转基因株系T1与野生型拟南芥WT相比,有107个差异基因,其中80个上调表达,27个下调表达;转基因株系T2与野生型拟南芥WT相比,有833个差异,其中550个上调表达,283个下调表达,说明 DcCIPK24过表达引起了拟南芥中基因表达量的变化。而在盐胁迫下,转基因株系T1与野生型拟南芥WT相比,有497个差异基因,其中477个差异基因上调表达,20个差异基因下调表达;转基因株系T2与野生型拟南芥WT相比有250个差异基因,其中上调和下调表达的基因分别有176和74个,说明盐胁迫下部分基因的表达受到 DcCIPK24基因的调控。
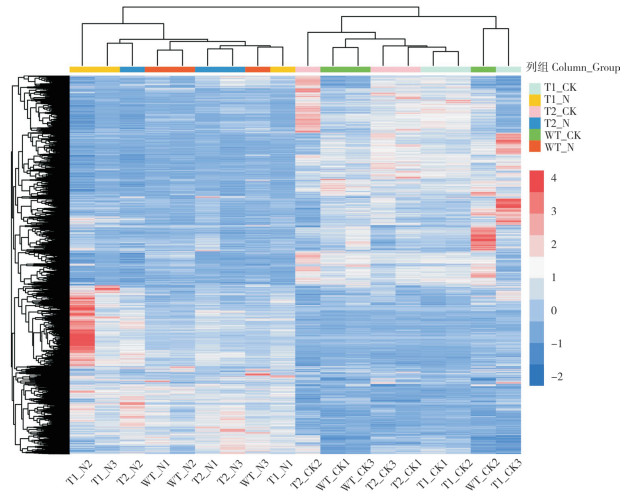
|
| WT-CK:野生型拟南芥正常培养;T1-CK、T2-CK:转基因株系T1、T2正常培养;WT-N:200 mmol/L NaCl处理的野生型拟南芥;T1-N、T2-N:200 mmol/L NaCl处理的转基因株系T1、T2;样本名称中的数字代表重复;蓝色到红色表示基因表达量由低到高 WT-CK: Wide-type Arabidopsis cultured under normal conditions; T1-CK, T2-CK: Transgenic Arabidopsis line T1 and T2 cultured under normal conditions; WT-N: Wide-type Arabidopsis cultured under 200 mmol/L NaCl treatment; T1-N, T2-N: Transgenic Arabidopsis lines T1 and T2 cultured under 200 mmol/L NaCl treatment; Numbers in the sample name represent the repetition; Blue and red represent low and high expression levels, respectively 图 2 差异基因表达热图 Fig. 2 Heat map of differential genes expression |
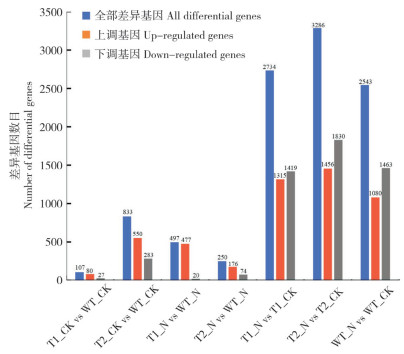
|
| WT-CK:野生型拟南芥正常培养;T1-CK、T2-CK:转基因株系T1、T2正常培养;WT-N:200 mmol/L NaCl处理的野生型拟南芥;T1-N、T2-N:200 mmol/L NaCl处理的转基因株系T1、T2;样本名称中的数字代表重复 WT-CK: Wide-type Arabidopsis cultured under normal conditions; T1-CK, T2-CK: Transgenic Arabidopsis lines T1 and T2 cultured under normal conditions; WT-N: Wide-type Arabidopsis cultured under 200 mmol/L NaCl treatment; T1-N, T2-N: Transgenic Arabidopsis lines T1 and T2 cultured under 200 mmol/L NaCl treatment; Numbers in the sample name represent the repetition 图 3 差异基因数目统计 Fig. 3 Statistics of the number of differential genes |
2.4 盐胁迫下差异表达基因GO分类
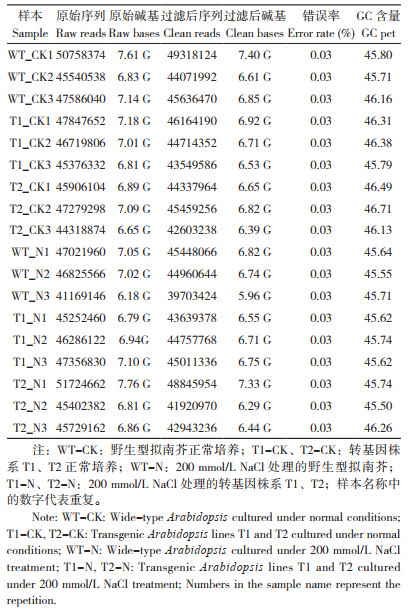
对盐胁迫下转基因株系(T1和T2)与野生型拟南芥(WT)的差异基因进一步分析,发现在T1-N vs WT-N和T2-N vs WT-N的比较组合中,共筛选到96个共同差异表达基因(图 4A)。对这96个差异基因进行GO注释,发现有54% 的差异基因被富集在分子功能(Molecular function,MF),36% 的差异基因被富集在生物过程(Biological process,BP),8% 的差异基因被注释到细胞组分(Cellular constituent,CC)(图 4B)。根据 P≤0.05的标准,分别在T1-N vs WT-N和T2-N vs WT-N中选择前10条GO注释通路进一步分析(图 4C),在T1-N vs WT-N和T2-N vs WT-N中富集均较明显的通路均为分子功能相关通路,主要包括血红素结合、四吡咯结合、铁离子结合、胺基裂解酶、异胡豆苷合成酶。对这5条通路分析发现,在T1-N vs WT-N中有15个差异基因富集在血红素结合通路和四吡咯结合通路中,12个差异基因被注释在铁离子结合通路,3个差异基因被注释在胺基裂解酶和异胡豆苷合成酶通路中;在T2-N vs WT-N中有8个差异基因均注释到血红素结合、四吡咯结合、铁离子结合通路中,2个差异基因注释到胺基裂解酶和异胡豆苷合成酶通路中(表 3)。
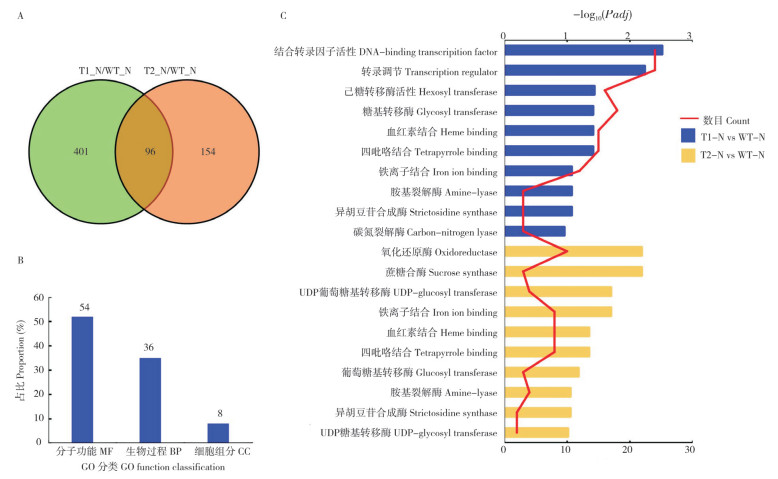
|
| WT-N:200 mmol/L NaCl处理的野生型拟南芥;T1-N、T2-N:200 mmol/L NaCl处理的转基因株系T1、T2 WT-N: Wide-type Arabidopsis cultured under 200 mmol/L NaCl treatment; T1-N, T2-N: Transgenic Arabidopsis lines T1 and T2 cultured under 200 mmol/L NaCl treatment 图 4 盐胁迫下转基因拟南芥和野生型拟南芥Venn图(A)分析和GO富集(B、C)分析 Fig. 4 Venn diagrams (A) and GO ontology analysis (B, C) of transgenic and wild-type Arabidopsis under salt stress |
2.5 盐胁迫下差异表达基因KEGG通路分析
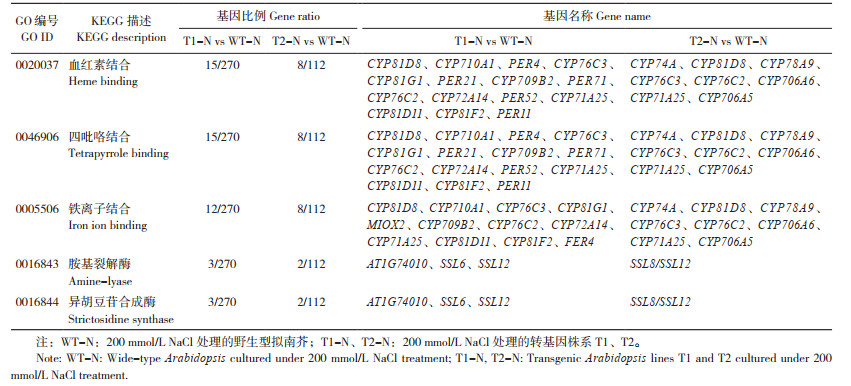
使用clusterProfiler软件将差异基因注释到KEGG数据库,分别选择T1-N vs WT-N和T2-N vs WT-N的前10条KEGG通路进行分析(图 5)。结果发现在盐处理下,T1-N vs WT-N和T2-N vs WT-N差异基因集均注释到氨基糖和核苷酸糖代谢、植物MAPK信号途径通路和甘油酯代谢通路。对这3条通路中的差异基因统计分析,显示T1-N vs WT-N中有8个差异基因(AT4G20460、UGE5、ChiC、EP3、MEE25、GLCAK2、RGP1、MUR4)被注释到氨基糖和核苷酸糖代谢通路中,有6个差异基因(ACS2、SIRK、OXI1、HAI3、MAPKKK18、ERF1B)被注释到植物MAPK信号途径中,有4个差异基因(GPAT1、ALDH2B7、GPAT7、GPAT5)被注释到甘油酯代谢通路中;而T2-N vs WT-N组合中有3个差异基因(MEE25、BXL4、UGE5)被注释到氨基糖和核苷酸糖代谢通路中,有7个差异基因(MAPKKK18、HAI3、ACS2、SAG113、AHG1、MAPKKK17、SIRK)被注释到植物MAPK信号途径中,有2个差异基因(DGAT1、GPAT5)被注释到甘油酯代谢通路中(表 4)。
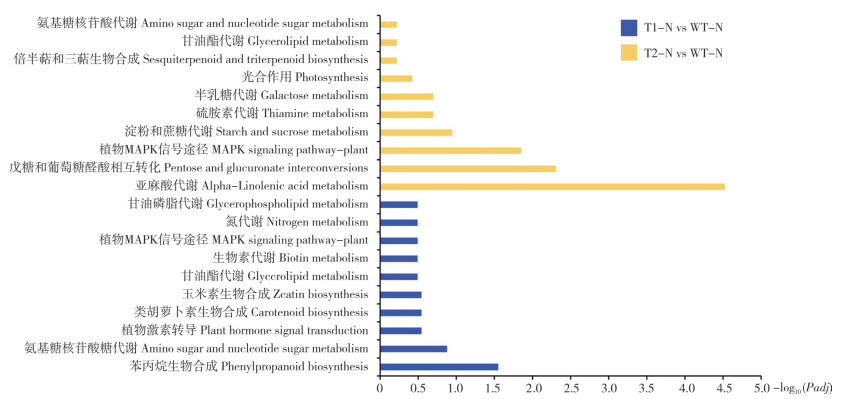
|
| WT-N:200 mmol/L NaCl处理的野生型拟南芥;T1-N、T2-N:200 mmol/L NaCl处理的转基因株系T1、T2 WT-N: Wide-type Arabidopsis cultured under 200 mmol/L NaCl treatment; T1-N. T2-N: Transgenic Arabidopsis lines T1 and T2 cultured under 200 mmol/L NaCl treatment 图 5 盐胁迫下差异表达基因KEGG富集分析 Fig. 5 KEGG enrichment of DEGs under salt stress |
2.6 荧光定量PCR验证
为验证转录组结果可靠性,从筛选到的96个差异基因中选择变化较为明显的8个差异基因进行荧光定量PCR验证。结果(图 6)显示,这些差异基因的表达模式与转录组数据一致,均受盐胁迫诱导。盐胁迫下,AtUGE5、AtGPAT5、AtMEE25、AtSIRK在转基因拟南芥中明显上调表达,AtATTI3、AtLHCB4.3、AtAMT1-2、AtCYP71A25明显下调表达。
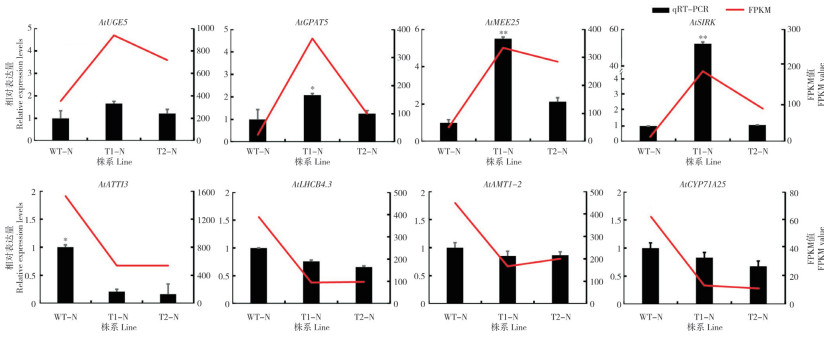
|
| WT-N:200 mmol/L NaCl处理的野生型拟南芥;T1-N、T2-N:200 mmol/L NaCl处理的转基因株系T1、T2 WT-N: Wide-type Arabidopsis cultured under 200 mmol/L NaCl treatment; T1-N, T2-N: Transgenic Arabidopsis lines T1 and T2 cultured under 200 mmol/L NaCl treatment 图 6 盐胁迫相关基因qPCR验证 Fig. 6 qPCR verification of salt stress-related genes |
3 讨论
CBL-CIPK信号途径是植物响应非生物胁迫过程中的重要通路,CBL、CIPK基因家族成员均不同程度的参与非生物胁迫的响应,研究发现,盐胁迫下转 DcCIPK24基因拟南芥长势优于野生型拟南芥[14]。本文在前期研究基础上,对 DcCIPK24的耐盐调控途径进行研究,并对盐胁迫下转 DcCIPK24拟南芥和野生型拟南芥进行转录组比较分析。GO富集分析发现盐胁迫下转基因拟南芥和野生型拟南芥的差异基因主要富集在血红素结合、四吡咯结合、铁离子结合、胺基裂解酶、异胡豆苷合成酶等分子功能相关通路中。
KEGG分析则将差异基因注释到氨基糖和核苷酸糖代谢、植物MAPK信号途径和甘油酯代谢通路中。已有研究表明氨基糖和核苷酸糖代谢通路中UDP糖、GDP糖和ADP糖的合成代谢途径与细胞壁的形成的有关[20]。对通路中的基因表达模式进行荧光定量PCR验证,发现基因表达趋势与转录组数据基本一致,说明转录组数据结果可靠。基因表达分析表明,盐胁迫下氨基糖和核苷酸糖代谢通路中的 AtUGE5和 AtMEE25在转基因拟南芥中呈现上调表达。AtUGE5是UDP- 葡萄糖-4- 差向异构酶(UDP-glucose 4-epimerases,UGEs)基因,UGE5能够调控糖基水解酶基因的表达[21],影响细胞壁中半乳糖的合成[22],在水分胁迫下UDP-葡萄糖会在UGE5的催化下生成UDP- 半乳糖,而UDP- 半乳糖是构成细胞壁的底物[23];AtMEE25是UDP- 阿拉伯糖变位酶MUR4的同源蛋白,能够催化阿拉伯糖吡喃糖和呋喃糖形式之间的相互转化,参与植物细胞壁的合成[24],说明DcCIPK24在盐胁迫下能够通过细胞壁防御机制增强转基因拟南芥的耐盐能力。MAPK通路是依赖于ABA途径参与到植物响应盐胁迫以及干旱胁迫的重要信号途径[25],荧光定量验证表明,盐胁迫下该通路中的 AtSIRK1和 AtGPAT5在转基因拟南芥中呈现上调表达。AtSIRK(也称FRK1)是一种核定位的丝裂原激活蛋白激酶,马铃薯ScFRK1参与了调节雄性和雌性配子发育的信号级联反应[26],番茄FRK1能够特异性的促进开花[27],在拟南芥中 SIRK的转录水平受WRKY6的调控,从而参与调控植物叶片衰老和植物防御[28-29],表明在盐胁迫下 DcCIPK24可能通过植物MAPK信号途径参与耐盐调控。甘油酯代谢通路是植物次生代谢过程中的重要通路,GPAT5(甘油-3- 磷酸酰基转移酶5)主要功能是催化脂肪酰基的酯化反应,影响甘油的生物合成。过表达 AtGPAT5与植物中的木质素和角质素生物合成有关,研究发现高盐胁迫下 gpat5突变体拟南芥种子的发芽率和幼苗的耐盐性降低[30];AtGPAT5的表达同时还受ABA的诱导[31]。荧光定量显示盐胁迫后 GPAT5在转基因拟南芥中呈现上调表达,推测转 DcCIPK24基因拟南芥能够通过甘油酯代谢通路响应盐胁迫。
4 结论本研究对盐胁迫下转 DcCIPK24拟南芥和野生型拟南芥的转录组比较分析,发现盐胁迫处理前后植株中的差异基因数量较多,且上调基因数量显著多于下调基因数量,说明DcCIPK24参与这些差异基因的调控。从盐胁迫下2个转基因株系与野生型植株的比较组合中共获得96个共同差异表达基因,GO注释分析发现54% 的差异基因富集在血红素结合、四吡咯结合、铁离子结合、胺基裂解酶、异胡豆苷合成酶等分子功能相关通路中;KEGG代谢通路和荧光定量PCR分析结果表明,盐胁迫下DcCIPK24可通过调控氨基糖和核苷酸糖代谢通路、植物MAPK信号和甘油酯代谢途径中的 AtUGE5、AtMEE25、AtSIRK和 AtGPAT5等基因的上调表达来响应拟南芥耐盐。
| [1] |
RODRIGUEZ-NAVARRO A. Potassium transport in fungi and plants[J]. Biochimica et Biophysica Acta, 2000, 1469(1): 1-30. DOI:10.1016/s0304-4157(99)00013-1 |
| [2] |
ZHANG X, LIU L, CHEN B, QIN Z, XIAO Y, ZHANG Y, YAO R, LIU H, YANG H. Progress in understanding the physiological and molecular responses of Populus to salt stress[J]. International Journal of Molecular Sciences, 2019, 20(6): 1312. DOI:10.3390/ijms20061312 |
| [3] |
詹振楠, 王文娟. 种子引发对盐胁迫枸杞种子萌发的影响[J]. 广东农业科学, 2018, 45(6): 14-18. DOI:10.16768/j.issn.1004-874X.2018.06.003 ZHAN Z N, WANG W J. Effects of seed priming on Lycium barbarum seed germination under salt stress[J]. Guangdong Agricultural Sciences, 2018, 45(6): 14-18. DOI:10.16768/j.issn.1004-874X.2018.06.003 |
| [4] |
CHO J H, SIM S C, KIM K N. Calcium sensor SlCBL4 associates with SlCIPK24 protein kinase and mediates salt tolerance in Solanum lycopersicum[J]. Plants(Basel), 2021, 10(10): 2173. DOI:10.3390/plants10102173 |
| [5] |
何梅, 章金辉, 王再花, 李杰, 叶广英, 叶庆生. CO2倍增对铁皮石斛光合特性和生长的影响[J]. 广东农业科学, 2020, 47(2): 17-23. DOI:10.16768/j.issn.1004-874X.2020.02.003 HE M, ZHANG J H, WANG Z H, LI J, YE G Y, YE Q S. Effects of doubling the CO2 concentration on photosynthetic characteristics and growth of Dendrobium officinale[J]. Guangdong Agricultural Sciences, 2020, 47(2): 17-23. DOI:10.16768/j.issn.1004-874X.2020.02.003 |
| [6] |
丁久玲, 席刚俊, 郑凯, 高大响, 史俊. NaCl浓度对铁皮石斛次生代谢物的影响[J]. 中南农业科技, 2023, 44(6): 44-47. DING J L, XI G J, ZHENG K, GAO D X, SHI J. Effect of NaCl concentration on secondary metabolites of Dendrobium offi cinale[J]. South-Central Agricultural Science and Technology, 2023, 44(6): 44-47. |
| [7] |
ZHANG H, YANG B, LIU W Z, LI H, WANG L, WANG B, DENG M, LIANG W, DEYHOLOS M K, JIANG Y Q. Identification and characterization of CBL and CIPK gene families in canola (Brassica napus L.)[J]. BMC Plant Biology, 2014, 14: 8. DOI:10.1186/1471-2229-14-8 |
| [8] |
ZHOU Y, YIN X C, DUAN R J, HAO G P, GUO J C, JIANG X Y. SpSOS1 and SpAHA1 work in coordinate in transgenic yeast and improve salt tolerance[J]. PLoS ONE, 2015, 10(9): e0137447. DOI:10.1371/journal.pone.0137447 |
| [9] |
KIM B G, WAADT R, CHEONG Y H, PANDEY G K, DOMINGUEZSOLIS J R, SCHULTKE S, LEE S C, KUDLA J, LUAN S. The calcium sensor CBL10 mediates salt tolerance by regulating ion homeostasis in Arabidopsis[J]. The Plant Journal, 2007, 52(3): 473-484. DOI:10.1111/j.1365-313X.2007.03249.x |
| [10] |
ZHANG H C, LV F L, HAN X, XIA X L, YIN W L. The calcium sensor PeCBL1, interacting with PeCIPK24/25 and PeCIPK26, regulates Na (+)/K (+) homeostasis in Populus euphratica[J]. Plant Cell Reports, 2013, 32(5): 611-621. DOI:10.1007/s00299-013-1394-5 |
| [11] |
SUN T, WANG Y, WANG M, LI T T, ZHOU Y, WANG X T, WEI S Y, HE G Y, YANG G X. Identification and comprehensive analyses of the CBL and CIPK gene families in wheat (Triticum aestivum L.)[J]. BMC Plant Biology, 2015, 15: 269. DOI:10.1186/s12870-015-0657-4 |
| [12] |
HU D G, MA Q J, SUN C H, SUN M H, YOU C X, HAO Y J. Overexpression of MdSOS2L1, a CIPK protein kinase, increases the antioxidant metabolites to enhance salt tolerance in apple and tomato[J]. Physiologia Plantarum, 2016, 156(2): 201-214. DOI:10.1111/ppl.12354 |
| [13] |
司灿. 铁皮石斛对干旱胁迫的生理响应及DNA甲基化研究[D]. 厦门: 华侨大学, 2016. SI C. The physiological response and DNA-methylation research of Dendrobium officinale under drought stress[D]. Xiamen: Huaqiao University, 2016. |
| [14] |
ZHANG T T, LI Y X, WANG P, LUO Q, FU S Y, KANG Y Q, ZHOU Y. Characterization of Dendrobium catenatum CBL-CIPK signaling networks and their response to abiotic stress[J]. International Journal of Biological Macromolecules, 2023, 236: 124010. DOI:10.1016/j.ijbiomac.2023.124010 |
| [15] |
ZHANG T T, LI Y X, KANG Y Q, WANG P, LI W, YU W G, WANG J, WANG J, SONG X Q, JIANG X Y, ZHOU Y. The Dendrobium catenatum DcCIPK24 increases drought and salt tolerance of transgenic Arabidopsis[J]. Industrial Crops & Products, 2022, 187: 115375. DOI:10.1016/j.indcrop.2022.115375 |
| [16] |
LI Y, ZHANG T, KANG Y, WANG P, YU W, WANG J, LI W, JIANG X, ZHOU Y. Integrated metabolome, transcriptome analysis, and multiflux full-length sequencing offer novel insights into the function of lignin biosynthesis as a Sesuvium portulacastrum response to salt stress[J]. International Journal of Biological Macromolecules, 2023, 237: 124222. DOI:10.1016/j.ijbiomac.2023.124222 |
| [17] |
CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y, XIA R. TBtools-an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [18] |
张婷婷, 李雨欣, 张德遥, 康宇乾, 王健, 宋希强, 周扬. 铁皮石斛蛋白磷酸酶PP2C家族基因鉴定及其表达分析[J]. 园艺学报, 2021, 48(12): 2458-2470. DOI:10.16420/j.issn.0513-353x.2021-0382 ZHANG T T, LI Y X, ZHANG D Y, KANG Y Q, WANG J, SONG X Q, ZHOU Y. Genome-wide identification and expression analyses of PP2C gene family in Dendrobium catenatum[J]. Acta Horticulturae Sinica, 2021, 48(12): 2458-2470. DOI:10.16420/j.issn.0513-353x.2021-0382 |
| [19] |
VOLHOV R A, PANCHUK I I, SCHÖFFL F. Heat-stress-dependency and developmental modulation of gene expression: the potential of house-keeping genes as internal standards in mRNA expression profiling using real-time RT-PCR[J]. Journal of Experimental Botany, 2003, 54(391): 2343-2349. DOI:10.1093/jxb/erg244 |
| [20] |
赵琪君. 水稻稻瘟病菌响应柠檬醛的关键基因分析[D]. 贵阳: 贵州大学, 2021. ZHAO Q J. The analysis of key genes in Magnaporthe oryzae in response to citral[D]. Guiyang: Guizhou University, 2021. |
| [21] |
KUGA M, SHIROYANAGI H, KAWAGUCHI T, TANI S. A new function of a putative UDP-glucose 4-epimerase on the expression of glycoside hydrolase genes in Aspergillus aculeatus[J]. Applied Microbiology and Biotechnology, 2023, 107(2-3): 785-795. DOI:10.1007/s00253-022-12337-8 |
| [22] |
GONDOLF V M, STOPPEL R, EBERT B, RAUTENGARTEN C, LIWANAG A J, LOQUÉ D, SCHELLER H V. A gene stacking approach leads to engineered plants with highly increased galactan levels in Arabidopsis[J]. BMC Plant Biology, 2014, 14: 344. DOI:10.1186/s12870-014-0344-x |
| [23] |
魏伟. 大豆GMDREB1转录因子调控植物抗逆反应的分子及生理机制[D]. 呼和浩特: 内蒙古农业大学, 2011. WEI W. Soybean GmDREB1 transcription factors regulation of wheat molecular and physiological reaction tolerance mechanism[D]. Hohhot: Inner MongoliaAgricultural University, 2011. |
| [24] |
BURGET E G, VERMA R, MØLHØJ M, REITER W D. The biosynthesis of L-arabinose in plants: Molecular cloning and characterization of a Golgi-localized UDP-D-xylose 4-epimerase encoded by the MUR4 gene of Arabidopsis[J]. Plant Cell, 2003, 15(2): 523-31. DOI:10.1105/tpc.008425 |
| [25] |
BAI G, XIE H, YAO H, LI F, CHEN X, ZHANG Y, XIAO B, YANG J, LI Y, YANG D H. Genome-wide identification and characterization of ABA receptor PYL/RCAR gene family reveals evolution and roles in drought stress in Nicotiana tabacum[J]. BMC Genomics, 2019, 20(1): 575. DOI:10.1186/s12864-019-5839-2 |
| [26] |
LAFLEUR E, KAPFER C, JOLY V, LIU Y, TEBBJI F, DAIGLE C, GRAY-MITSUMUNE M, CAPPADOCIA M, NANTEL A, MATTON D P. The FRK1 mitogen-activated protein kinase kinase kinase (MAPKKK) from Solanum chacoense is involved in embryo sac and pollen development[J]. Journal of Experimental Botany, 2015, 66(7): 1833-1843. DOI:10.1093/jxb/eru524 |
| [27] |
ODANAKA S, BENNETT A B, KANAYAMA Y. Distinct physiological roles of fructokinase isozymes revealed by gene-specific suppression of Frk1 and Frk2 expression in tomato[J]. Plant Physiology, 2002, 129(3): 1119-1126. DOI:10.1104/pp.000703 |
| [28] |
ROBATZEK S, SOMSSICH I E. Targets of AtWRKY6 regulation during plant senescence and pathogen defense[J]. Genes Development, 2002, 16(9): 1139-1149. DOI:10.1101/gad.222702 |
| [29] |
TOUM L, CONTI G, GUERRIERO F C, CONFORTE V P, GAROLLA F A, ASURMENDI S, VOJNOV A A, GUDESBLAT G E. Singlestranded oligodeoxynucleotides induce plant defence in Arabidopsis thaliana[J]. Annals of Botany, 2020, 126(3): 413-422. DOI:10.1093/aob/mcaa061 |
| [30] |
BEISSON F, LI Y, BONAVENTURE G, POLLARD M, OHLROGGE J B. The acyltransferase GPAT5 is required for the synthesis of suberin in seed coat and root of Arabidopsis[J]. Plant Cell, 2007, 19(1): 351-368. DOI:10.1105/tpc.106.048033 |
| [31] |
BARBERON M, VERMEER J E, DE BELLIS D, WANG P, NASEER S, ANDERSEN T G, HUMBEL B M, NAWRATH C, TAKANO J, SALT D E, GELDNER N. Adaptation of root function by nutrient-induced plasticity of endodermal differentiation[J]. Cell, 2016, 164(3): 447-459. DOI:10.1016/j.cell.2015.12.021 |
(责任编辑 马春敏)
 2023, Vol. 50
2023, Vol. 50