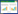文章信息
基金项目
- 河南省高等学校重点科研项目(23A230011);河南省高校国家级大学生创新创业训练计划项目(202210467033);河南省高校大学生创新创业训练计划项目(202210467013);河南省科技攻关项目(232102110086)
作者简介
- 徐彦召(1984—),男,博士,副教授,研究方向为动物病原和新兽药,E-mail:56893346@qq.com.
通讯作者
- 王青(1978—),女,博士,高级实验师,研究方向为预防兽医学,E-mail:wangqing@hist.edu.cn.
文章历史
- 收稿日期:2023-03-15
【研究意义】犬细小病毒(Canine parvovirus,CPV)在自然界分布非常广泛,具有高致病性和高传染性,除感染犬外还可感染犬亚科的其他动物,如浣熊、土狼、灰狼、条纹臭鼬等[1-3]。早在1978年该病毒首次在美国被发现,随即在世界范围内快速传播。CPV对机体的侵入性非常强,往往对机体造成非常严重的危害。2~4月龄的幼龄犬是CPV的最易感宿主。患病犬主要表现为呕吐、腹泻、便血、精神萎靡等,根据表现特征及发病部位将其分为出血性胃肠炎和急性心肌炎[4]。一般感染CPV的犬很难治愈,且治疗期长、费用高。研究表明,即使预后良好的犬仍可向外界环境长期散毒,对其他犬的健康造成很大威胁,是制约养犬业发展的一大因素[5-6]。因此,研究CPV的流行特点及遗传变异规律对今后研究CPV的变异及开展更有效的疫苗防控具有重要意义。【前人研究进展】研究证实,CPV极易发生突变,其基因组替代率与RNA病毒较为相似[7],致使各地区的流行株不一致,甚至同一地区存在多种流行株,从而导致商品疫苗的保护力大大降低[8-9]。国外有关CPV的研究领先国内很多年,我国直到1982年才有CPV的相关报道,随后陆续有新毒株出现。有研究者提出猜想,免疫失败可能是不断出现的新型变异株所致,传统疫苗免疫对不同变异株的中和能力降低,导致临床上接种疫苗后的犬感染CPV仍时有发生[10]。CPV变异株不断出现,同时伴随复杂的病毒流行态势,犬只CPV免疫失败时有发生,这些问题为CPV的预防控制带来非常大的挑战。【本研究切入点】随着我国社会的日益发展和繁荣,宠物的角色逐渐转型,猫和狗越来越多地被当作家庭中不可或缺的亲密伴侣。但CPV变异性强,短时间内很难消灭,且其传播速度非常快、传播范围广。因此,及时检测出CPV、采取有针对性的治疗方案及有效控制其传播是研究者共同面临的重大难题[11]。及时接种疫苗是目前预防该病毒传播的最有效方法,目前临床也研发了针对该病毒的疫苗,但随着CPV的不断变异,现有疫苗是否对变异的病毒具有保护力存在争议,研发新的疫苗仍然是各国研究者共同关注的问题,而获得地区性的种毒是研究者共同关注的焦点。【拟解决的关键问题】为了解新乡地区CPV流行株的生物学特性和遗传进化情况,对分离得到的CPV毒株进行多方面鉴定并分析其遗传变异规律,为新乡地区CPV的预防、治疗及新疫苗的研发提供参考。
1 材料与方法 1.1 试验材料1.1.1 临床病样和细胞 临床样品23份,来自2021年和2022年河南省新乡地区宠物医院收集的疑似患病犬的粪便和眼、鼻、口、肛拭子;猫肾细胞CRFK由中国农业科学院上海兽医研究所惠赠,DH5α感受态细胞购自宝生物工程(大连)有限公司。
1.1.2 主要试剂和仪器 胎牛血清、质粒小量抽提试剂盒、DNA纯化试剂盒购自上海碧云天生物技术有限公司;细菌基因组DNA小剂量提取试剂盒购自北京庄盟国际生物基因科技有限公司;DMEM高糖培养基购自西格玛奥德里奇(上海)贸易有限公司、胰蛋白酶购自赛默飞世尔科技有限公司;pMD19-T载体购自宝生物工程(大连)有限公司。PCR仪(型号:Applied Biosystems® 2720)为赛默飞世尔(上海)仪器有限公司产品,透射电子显微镜(型号:Hitachi HT7700)为日本日立公司产品。
1.1.3 引物设计与合成 根据CPV全基因组序列信息,针对高度保守区利用Premier5.0软件设计3对引物(表 1)用于扩增CPV全基因组序列,引物由通用生物(安徽)股份有限公司进行合成。
1.2 试验方法
1.2.1 病样的PCR鉴定 分别称取1.0 g粪便样品,加入9 mL灭菌超纯水进行溶解;将不同检测对象的眼、鼻、口、肛拭子插入1 mL灭菌超纯水中浸泡。参考基因组DNA小剂量提取试剂盒说明书分别提取病毒DNA。以提取的基因组DNA为模板,以DOG-1引物对建立PCR反应体系。PCR扩增体系为20 μL:2 × Fast Taq Premix 10 μL,上、下游引物各1 μL,病毒DNA 1 μL,加ddH2O至20 μL。PCR产物经1% 琼脂糖凝胶电泳分析。
1.2.2 病毒的分离培养 分别用DMEM高糖培养基稀释眼、鼻、口、肛拭子浸泡液,吹打混匀后用一次性孔径0.22 μm滤膜过滤,并接种于正常培养的单层CRFK细胞,放入细胞培养箱内37 ℃下进行培养,每天定时观察是否出现细胞病变,盲传3代,收取病毒液反复冻融3次,鉴定后保存于-80℃冰箱备用,以添加DMEM培养基的正常细胞作为空白对照。
1.2.3 病毒的透射电镜观察 取上述第3代细胞培养物用一次性孔径0.22 μm滤膜过滤去除杂质,用0.1% 磷钨酸负染,在透射电子显微镜下进行观察。
1.2.4 病毒的序列扩增及连接转化 以DOG-1为鉴定引物,以1.2.2中收集的病毒液为模板建立PCR反应体系,对每代培养的病毒进行鉴定,弃去阴性者。分别以DOG-2与DOG-3两对引物建立PCR体系,对CPV全长基因分两段进行扩增,同时设阴性对照。扩增产物经琼脂糖凝胶电泳检测,并回收纯化目的基因片段。按照pMD19-T载体说明书将上述纯化的2段DNA分别与pMDTM-19T载体在4 ℃条件下过夜连接,将连接产物转化DH5α感受态细胞。将转化后初步鉴定为阳性的菌送往通用生物(安徽)股份有限公司进行测序。利用DNAStar软件将2段DNA序列进行拼接,并通过多次测序校正结果。
1.2.5 分离病毒的遗传进化分析 将拼接好的病毒全基因组序列与NCBI上有代表性的其他22个CPV参考毒株(国内株13株,国外株9株)进行核苷酸比对分析,通过Clustalx1.83软件构建完整基因组的系统发育树,分析分离株与其他参考毒株之间的进化关系。
1.2.6 分离病毒 VP2基因的氨基酸进化分析 将测序所获得的 VP2基因序列与NCBI上的其他15株CPV毒株(国内毒株12株,国外毒株3株)的 VP2基因序列用Megalign 5.0软件进行氨基酸序列对比,并分析分离株与参考病毒VP2的氨基酸变异位点。
2 结果与分析 2.1 病样的PCR鉴定分别将送检的疑似患病犬临床粪便样品和眼、鼻、口、肛拭子样品进行PCR鉴定,琼脂糖凝胶电泳结果(图 1)显示,在451 bp处有1条特异性条带,与预期结果大小一致。
|
| M:DL2 000 DNA Marker;1:眼、鼻、口、肛拭子;2:粪便样本;3:阴性对照 M: DL2 000 DNA Marker; 1: Swab from eyes, nose, mouth, anus; 2: Stool sample; 3: Negative control 图 1 病料的PCR鉴定结果 Fig. 1 PCR identification results of pathological materials |
2.2 病毒的分离培养
将处理后的样品接种于正常的单层CRFK细胞。接种前细胞贴壁生长,呈典型上皮细胞形态,细胞边界清晰(图 2A);而接种72 h后的细胞出现拉网、皱缩、凝聚成片等典型的细胞病变(图 2B)。

|
| A:接种前;B:接种后 A: Before inoculation; B: After inoculation 图 2 接种CPV前后的CRFK细胞 Fig. 2 CRFK cells before and after CPV inoculation |
2.3 病毒的形态观察
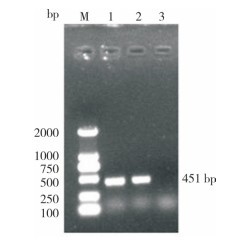
将第3代细胞培养液负染后于透射电子显微镜下进行观察。在电镜下可观察到多个正二十面体结构、直径约25 nm、无囊膜包被的病毒结构体,与经典CPV形态相符(图 3),将所分离的病毒命名为XX-2022株。
|
| 图 3 分离病毒的透射电镜观察 Fig. 3 Transmission electron microscopic observation of isolated virus |
2.4 XX-2022株病毒的全基因组序列扩增
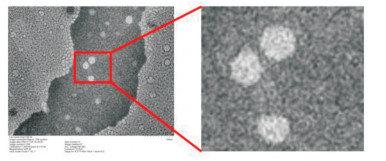
用XX-2022株病毒的第3代细胞培养物作为模板分2段进行PCR扩增。经琼脂糖凝胶电泳后可见2条与预期片段大小一致的目的条带(图 4)。分别将2段目的基因与pMDTM-19T载体连接转化DH5α细胞。将初步鉴定为阳性的菌进行测序并拼接,获得一条长为4 588 bp的序列。
|
| M:DL5000 DNA Marker;1、2:分别为DOG-2引物的PCR扩增和阴性对照;3、4:分别为DOG-3引物的PCR扩增和阴性对照 M: DL5000 DNA Marker; 1, 2: PCR amplification and negative control with DOG-2 primers, respectively; 3, 4: PCR amplification and negative control with DOG-3 primers, respectively 图 4 CPV全基因组序列PCR扩增结果 Fig. 4 PCR amplification results of CPV complete genome sequence |
2.5 XX-2022株病毒的遗传进化分析
将拼接后的XX-2022株病毒序列与22个CPV参考毒株的全基因组序列进行对比分析,并构建系统发育树,发现该毒株的全基因组与国外毒株的同源性为97.3%~99.2%,与国内毒株同源性为96.9%~99.3%(图 5);由系统发育树(图 6)可见,XX-2022株与上海株Canine/SH/1/2019(MN840830.1)处于同一进化小分支中,并且XX-2022株与近10年来我国各地分离得到的毒株同处于一个大分支内,亲缘关系较近,与国外毒株的亲缘关系较远。

|
| 图 5 XX-2022株CPV同源性分析 Fig. 5 Homology analysis of XX-2022 strains of CPV |

|
| 图 6 CPV毒株全长核苷酸序列系统发育树 Fig. 6 Phylogenetic tree of CPV strain with full length nucleotide sequence |
2.6 XX-2022株病毒 VP2基因的氨基酸进化分析
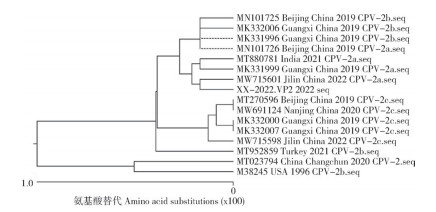
利用Megalign 5.0软件将分离株的 VP2基因序列与NCBI上其他15个CPV参考毒株的 VP2基因序列进行翻译获得相应氨基酸序列,通过Lipman-Pearson Method进行氨基酸分析,结果(图 7)显示,该毒株与MW715601在同一小分支内,氨基酸同源性为99.7%。根据CPV的分型标准,可确定XX-2022株为CPV-2a亚型。
|
| 图 7 XX-2022株病毒 VP2基因的推导氨基酸序列系统发育树 Fig. 7 Phylogenetic tree of amino acid sequence derived from VP2 gene of XX-2022 strain virus |
XX-2022株与其他参考毒株基因的氨基酸变异位点结果如表 2所示,与其他基因型为CPV-2a的毒株相比,XX-2022株也存在5(A) → 5(G)的变异。
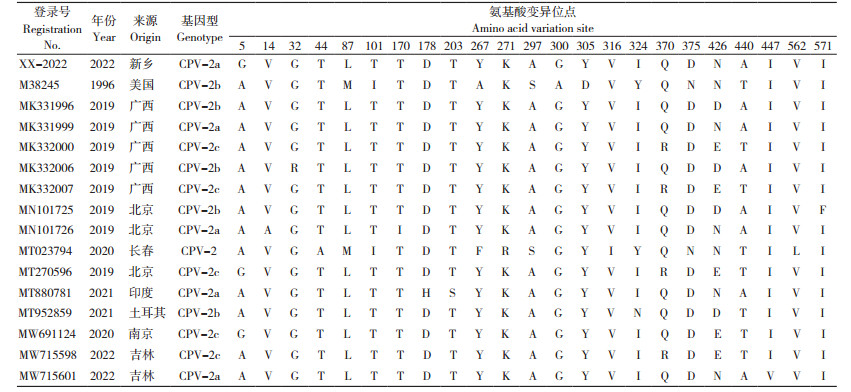
|
3 讨论
CPV于1977年首次由美国学者Eugster和Nairn从出血性肠炎病犬的粪便中分离得到,该病毒可造成犬科动物感染[12-13]。CPV只有1个抗原型、即CPV-2,一直以来RNA病毒通常以高突变率进行免疫逃避,而CPV-2的基因突变与RNA病毒相似,表现出高的遗传异质性,随着变异的积累,目前已形成多个变异亚型,即CPV-2a、CPV-2b、CPV-2c (a) 和CPV-2c (b),但不同亚型之间的抗原性有所不同,从而导致实验室和兽医临床需不断对当地流行毒株进行监测,以评估疫苗的保护力[14-15]。本研究从河南省新乡市宠物医院送检的样品中分离得到1株CPV,对分析新乡地区CPV的流行具有重要的参考意义,为客观评价当地使用疫苗的保护力提供参考。
由于CPV-2不断进行抗原漂移,在抗原性、宿主范围和血凝性上都发生变化,导致其宿主范围不断扩大[16-17]。在自然环境和人工饲养环境下,新的CPV-2a和CPV-2b毒株均被发现可引起猫感染,并可形成犬、猫互相感染,这与其基因组变异导致编码氨基酸发生变异有关[18-19]。从系统遗传学角度来看,CPV-2最初与CPV-2a在蛋白上仅有少量氨基酸变化,CPV-2b亚型是由CPV-2a上的N426D突变引起的,而CPV-2c亚型是CPV-2b上D426E突变引起的[20-21]。本研究对分离的CPV全基因组序列进行进化分析,结果显示,该毒株与登录号为MN840830的CPV毒株处于同一进化分支上,基因组同源性为99.3%;氨基酸序列分析表明,该毒株与登录号为MW715601的CPV VP2在同一小分支内,氨基酸同源性为99.7%。对CPV进行同源性判断,可以研究不同患病动物病原体的遗传序列变化情况,在分子生物学层面上揭示传播方式,分析进化来源,研究致病机制与机理,为控制疫病的发生提供科学依据。
VP2基因是CPV最主要的毒力基因,其基因片段上包含CPV最易变异的区段,其中VP2蛋白的第426位氨基酸具有CPV分型标尺的作用[22]。因此,本研究对分离的XX-2022株病毒的 VP2基因序列进行分析,以期为更深入研究CPV的流行、疫苗研发提供理论参考,为更好地制定CPV预防措施奠定基础。本研究对 VP2基因推导的氨基酸序列进行比对发现,该毒株的基因分型为CPV-2a型。不同基因型之间存在多个氨基酸突变位点,同种基因型之间也存在个别氨基酸突变位点,这些氨基酸突变可能导致病毒表面抗原发生重大变化。因此,根据病毒的流行和变异进化制备疫苗将逐渐成为趋势[23]。但目前用于制备疫苗的CPV毒株更新较慢,导致经常出现免疫失败的现象。因此对CPV进行分子生物学分析,了解其流行情况和抗原变异情况,对CPV引起的疾病预防和控制有重要意义。
4 结论本试验从临床病例中成功分离出1株CPV病毒,命名为XX-2022株,对其基因组序列及 VP2基因推导的氨基酸序列进行分析,结果显示,XX-2022株CPV与Canine/SH/1/2019(MN840830.1)株的亲缘关系较近;该毒株的VP2氨基酸序列与YANJI-5(MW715601)的VP2氨基酸序列在同一小分支,氨基酸同源性为99.7 %。根据CPV病毒的基因分型原则,最终确定XX-2022株CPV属于CPV-2a型。XX-2022株CPV的成功分离为进一步制备有效的CPV疫苗,从而对预防、控制CPV病毒流行具有重大研究意义,为后续研究河南地区乃至全国CPV的流行特性和遗传变异规律提供参考。
| [1] |
莫若. 犬细小病毒2c亚型的分离鉴定及马抗CPV-2c精制免疫球蛋白的制备[D]. 长春: 吉林农业大学, 2021. DOI: 10.27163/d.cnki.gjlnu.2021.000037. MO R. Isolation and identification of canine parvovirus 2c and preparation of equine immunoglobulin against CPV-2c[D]. Changchun: Jilin Agricultural University, 2021. DOI: 10.27163/d.cnki.gjlnu.2021.000037. |
| [2] |
ALLISON A B, KOHLER D J, FOX K A, BROWN J D, GERHOLD R W, SHEARN-BOCHSLER V I, DUBOVI E J, PARRISH C R, HOLMES E C. Frequent cross-species transmission of parvoviruses among diverse carnivore hosts[J]. Journal of Virology, 2013, 87(4): 2342-2347. DOI:10.1128/JVI.02428-12 |
| [3] |
ZHANG L N, WANG K, AN F Y, ZHANG D L, ZHANG H L, XU X L, GUO C, YAN H M, KUANG Y J, ZHANG Z D, LU R G, HUA Y. Fatal canine parvovirus type 2a and 2c infections in wild Chinese pangolins (Manis pentadactyla) in southern China[J]. Transboundary and Emerging Diseases, 2022, 69(6): 4002-4008. DOI:10.1111/tbed.14703 |
| [4] |
周宏专, 苏霞, 徐福洲, 杨兵. 犬细小病毒研究进展[J]. 动物医学进展, 2019, 40(12): 79-84. DOI:10.16437/j.cnki.1007-5038.2019.12.016 ZHOU H Z, SU X, XU F Z, YANG B. Research progress of canine parvovirus[J]. Progress in Veterinary Medicine, 2019, 40(12): 79-84. DOI:10.16437/j.cnki.1007-5038.2019.12.016 |
| [5] |
TAKANO T, HAMAGUCHI S, HASEGAWA N, DOKI T, SOMA T. Predominance of canine parvovirus 2b in Japan: An epidemiological study during 2014-2019[J]. Archives of Virology, 2021, 166(11): 3151-3156. DOI:10.1007/s00705-021-05200-0 |
| [6] |
张玉香, 李改茹, 纪森林, 檀济敏, 王茹怡, 粟硕. 犬细小病毒分子生物学研究进展[J]. 中国预防兽医学报, 2016, 38(12): 993-997. ZHANG Y X, LI G R, JI S L, TAN J M, WANG R Y, SU S. Progress in molecular biology of canine parvovirus[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(12): 993-997. |
| [7] |
CANUTI M, MIRA F, SORENSEN R G, RODRIGUES B, BOUCHARD E, WALZTHONI N, HOPSON M, GILROY C, WHITNEY H G, LANG A S. Distribution and diversity of dog parvoviruses in wild, free-roaming and domestic canids of Newfoundland and Labrador, Canada[J]. Transboundar y and Emerg ing Diseases, 2022, 69: e2694-e2705. DOI:10.1111/tbed.14620 |
| [8] |
鲁荣光, 刘维全, 闫喜军. 犬细小病毒与宿主互作及跨种传播机制研究概况[J]. 动物医学进展, 2019, 40(11): 113-116. DOI:10.16437/j.cnki.1007-5038.2020.11.022 LU R G, Liu W Q, YAN X C. Advance in mechanisms of canine parvovirus interaction with host and cross-species transmission[J]. Progress in Veterinary Medicine, 2019, 40(12): 79-84. DOI:10.16437/j.cnki.1007-5038.2020.11.022 |
| [9] |
LI S H, CHEN X, HAO Y F, ZHANG G Z, LYU Y, WANG J K, LIU WQ, QIN T. Characterization of the VP2 and NS1 genes from canine parvovirus type 2 (CPV-2) and feline panleukopenia virus (FPV) in Northern China[J]. Frontiers in Veterinary Science, 2022(9): 934849-934849. DOI:10.3389/FVETS.2022.934849 |
| [10] |
孙明洁, 董国英, 张雪竹, 胡桂学. 犬细小病毒国内外流行病学研究进展[J]. 中国兽医学报, 2019, 39(5): 1035-1039. DOI:10.19619/j.issn.1007-1938.2017.04.007 SUN M H, DONG G Y, ZHANG X Z, HU G X. Advances on canine parvorirus epidemiology at home and abroad[J]. Chinese Journal of Veterinary Science, 2019, 39(5): 1035-1039. DOI:10.19619/j.issn.1007-1938.2017.04.007 |
| [11] |
孙明洁, 董国英, 张雪竹, 胡桂学. 犬细小病毒检测方法研究进展[J]. 中国兽药杂志, 2018(8): 68-73. SUN M H, DONG G Y, ZHANG X Z, HU G X. Progress of diagnostic methods for canine parvovirus disease[J]. Chinese Journal of Veterinary Drug, 2018(8): 68-73. |
| [12] |
CHEN Y Z, WANG J Y, BI Z W, TAN Y P, LV L X, ZHAO H, XIA X X, ZHU Y M, WANG Y S, QIAN J. Molecular epidemiology and genetic evolution of canine parvovirus in East China, during 2018-2020[J]. Infection Genetics and Evolution, 2021(7): 90. DOI:10.1016/j.meegid.2021.104780 |
| [13] |
陈卫红, 陈永忠. 犬细小病毒病的综合治疗[J]. 广东农业科学, 2009(8): 185-187. DOI:10.16768/j.issn.1004-874X.2009.08.071 CHEN H W, CHEN Y Z. Comprehensive treatment of canine parvovirus disease[J]. Guangdong Agricultural Sciences, 2009(8): 185-187. DOI:10.16768/j.issn.1004-874X.2009.08.071 |
| [14] |
ALLISON A B, KOHLER D J, ORTEGA A, HOOVER E A, GROVE D M, HOLMES E C, PARRISH C R. Host-specific parvovirus evolution in nature is recapitulated by in vitro adaptation to different carnivore species[J]. Plos Pathogens, 2014, 10(11): e1004475. DOI:10.1371/journal.ppat.1004475 |
| [15] |
KHATRI R, POONAM, MOHAN H, MINAKSHI, PUNDIR C S. Epidemiology, pathogenesis, diagnosis and treatment of canine parvovirus disease in dogs: A mini review[J]. Journal of Veterinary Science and Medical Diagnosis, 2017(6): 3. DOI:10.4172/2325-9590.1000233 |
| [16] |
ZHOU P, ZENG W J, ZHANG X, LI S J. The genetic evolution of canine parvovirus-A new perspective[J]. PLoS One, 2017, 12(3): e0175035. DOI:10.1371/journal.pone.0175035 |
| [17] |
张乐, 于小航, 王义鹤, 杨佳美, 赵丽丽, 段铭. 2019—2020年河南与河北部分地区犬瘟热流行病学调查及病毒分离鉴定[J]. 广东农业科学, 2023, 50(2): 95-103. DOI:10.16768/j.issn.1004-874X.2023.02.011 ZHANG L, YU X H, WANG Y H, YANG J M, ZHAO L L, DUAN M. Epidemiological investigation, isolation and identification of canine distemper virus in parts of henan and hebei provinces from 2019 to 2020[J]. Guangdong Agricultural Sciences, 2023, 50(2): 95-103. DOI:10.16768/j.issn.1004-874X.2023.02.011 |
| [18] |
黄冠雄, 许丹宁, 杨舒展, 郭斯璇, 曾依翎, 黄运茂, 田允波, 曹楠. 广东省鹅细小病毒的分离及序列分析[J]. 广东农业科学, 2018, 45(5): 98-104. DOI:10.16768/j.issn.1004-874X.2018.05.017 HUANG G X, XU D N, YANG S Z, GUO S X, ZENG Y L, HUANG Y M, TIAN Y B, CAO N. Isolation and phylogenetic analysis of goose parvovirus isolated in Guangdong province[J]. Guangdong Agricultural Sciences, 2018, 45(5): 98-104. DOI:10.16768/j.issn.1004-874X.2018.05.017 |
| [19] |
WU H, LI X, WANG L, LIU Y, TIAN K. Molecular epidemiological survey of canine parvovirus in domestic dogs in four provinces, China[J]. Virus Disease, 2018, 29(1): 113-117. DOI:10.1007/s13337-018-0427-7 |
| [20] |
邓永, 孔冬妮, 侯力丹, 毛娅卿, 王嘉. 犬细小病毒2c亚型毒株的分离与鉴定[J]. 中国兽药杂志, 2019, 53(9): 19-26. DENG Y, KONG D N, HOU L D, MAO Y Q, WANG J. Isolation and identification of canine parvovirus 2C subtype[J]. Chinese Journal of Veterinary Drug, 2019, 53(9): 19-26. |
| [21] |
WANG K, DU S, WANG Y, WANG S, LUO X, ZHANG Y, LIU C, WANG H, PEI Z, HU G. Isolation and identification of tiger parvovirus in captive siberian tigers and phylogenetic analysis of VP2 gene[J]. Infection, Genetics and Evolution, 2019, 75: 103957-103957. DOI:10.1016/j.meegid.2019.103957 |
| [22] |
DUARTE R B, FELIZARDA S M, OLIVEIRA M P, JÚLIA M S, BRAGA S A. Persistence of vaccine immunity against canine parvovirus and canine distemper virus for determination of vaccine protocol in dogs: impacts and challenges in Brazil[J]. Research Society and Development, 2021, 10(11): e127101119472. DOI:10.33448/rsd-v10i11.19472 |
| [23] |
LI C, TANG J, CHEN Z, NIU G, LIU G. A divergent canine par-vovirus type 2c (CPV-2c) isolate circulating in China[J]. Infection, Genetics and Evolution, 2019, 73: 242-247. DOI:10.1016/j.meegid.2019.05.004 |
(责任编辑 崔建勋)
 2023, Vol. 50
2023, Vol. 50