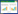文章信息
基金项目
- 国家自然科学基金(32360744);云南省生物种业与精深加工重大专项(202202AE090031)
作者简介
- 张明先(2000—),女,在读硕士生,研究方向为蔬菜种质创新及利用,E-mail:zmx11234566@163.com.
通讯作者
- 吕俊恒(1989—),男,博士,副教授,研究方向为蔬菜种质创新及利用,E-mail:junhenglv@ynau.edu.cn.
文章历史
- 收稿日期:2023-09-27
2. 云南省农业科学院园艺作物研究所, 云南 昆明 650205
2. Horticultural Crop Research Institute, Yunnan Academy of Agricultural Sciences, Kunming 650205, China
【研究意义】类胡萝卜素是植物中重要的次生代谢产物,是类异戊二烯代谢物的1个亚类,在所有光合作用植物、藻类、蓝藻和几种非光合作用真菌和原核生物中产生[1]。类胡萝卜素使园艺植物呈现出鲜艳的色泽,同时还可以散发出香气[2]。类胡萝卜素可以保护叶绿素,使其不受强光引起的光氧化损伤,保证光合作用正常进行[3]。成熟的辣椒(Capsicum annuum)果实中含有α-胡萝卜素、β-胡萝卜素、玉米黄质、叶黄素、隐黄质、辣椒红素和辣椒玉红素等多种类胡萝卜素,辣椒果实含有的各种类胡萝卜素具有重要保健功能及经济价值。类胡萝卜素合成与调控机制发生在多个水平,转录调控是胡萝卜素生成的第一个主要的控制机制[4],通过各种机制如转录调节、突变和自然遗传变异改变基因表达途径会影响许多作物中的类胡萝卜素含量或组成。目前对辣椒类胡萝卜素生物合成的调控,主要是从类胡萝卜素代谢途径中关键基因的结构表达和蛋白产物等方面展开,从而揭示辣椒成熟果实颜色变化的遗传调控机理[5]。【前人研究进展】MADS-box转录因子作用于植物的许多生理和生化过程,尤其在果实成熟方面[6]。研究表明,番茄(Solanum lycopersicum)的SlCMB1基因可以影响果实成熟过程中乙烯的合成,从而影响类胡萝卜素的积累[7]。TAGL1是果实成熟过程中的重要调控因子[8],TAGL1的转录活性可促进乙烯产生和叶绿素分解,从而促进果实成熟,影响类胡萝卜素和番茄红素的积累[9]。RIN在番茄果实采后乙烯生成、番茄红素累积及叶绿素降解中起作用[10]。FUL1和FUL2在果实成熟过程中起到冗余功能,作为成熟调控因子CNR、NOR和RIN的下游,以一种独立方式对ACS2和ACS4进行调控,从而对果实成熟产生影响[7]。番茄SlMADS1在果实成熟过程中起抑制作用,在SlMADS1沉默的番茄中类胡萝卜素的积累、乙烯的产生与相关基因均得到增强[11]。番茄SlFYFL可降低乙烯生物合成及响应基因的表达,从而延缓果实成熟[12]。SlMBP8是一种抑制性调节剂,沉默SlMBP8可促进果实成熟、类胡萝卜素积累和细胞壁代谢,负调控果实成熟[13]。辣椒CaBBX20基因沉默会导致类胡萝卜素积累减少、CaCCS表达下调,对辣椒果实类胡萝卜素代谢具有积极作用[14]。CaPSY1基因的沉默导致成熟辣椒果实中叶黄素含量增加和玉米黄质含量降低,参与成熟辣椒果实类胡萝卜素的代谢[15]。CHYB1是一种催化β-隐黄质形成的基因,在不同发育阶段的红辣椒和橙辣椒果实中均具有较高表达,对成熟阶段辣椒红色形成具有重要作用[16]。【本研究切入点】辣椒果实颜色的形成是复杂的生物过程,色泽的形成由类胡萝卜素相关基因的表达及各种类型胡萝卜素的积累决定[17]。尽管目前对辣椒果实中类胡萝卜素生物合成相关基因有一定研究,但对其代谢调控机理仍不明确。【拟解决的关键问题】本试验克隆皱皮红和皱皮黄辣椒MADS-box基因家族中的CMB1基因,进一步分析CMB1在调控类胡萝卜素合成过程中不同时期的表达模式,初步探究CMB1对辣椒类胡萝卜素的调控作用,为辣椒的果色调控和品种选育提供参考。
1 材料与方法 1.1 试验材料选取2021年7月种植于云南农业大学实验大棚(海拔1 800~2 000 m)内的昆明地方品种红色皱皮辣(皱皮红)与黄色皱皮辣(皱皮黄),根据果实不同成熟时期(绿熟期、转色期、完熟期)进行取样。分别采取3个时期辣椒果实样品,用锡箔纸包严后置于-80 ℃超低温冰箱中保存、备用。
快速通用植物RNA提取试剂盒3.0,购自北京华越洋生物科技有限公司;反转录试剂盒Hifairs®Ⅱ 1st Strand cDNA Synthesis SuperMix for qPCR(gDNA digesterplus)、荧光定量试剂盒Hiefr® qPCR SYBR Green Master Mix(Low Rox Plus),购自翌圣生物科技(上海)股份有限公司;引物于北京擎科生物科技股份有限公司昆明分公司合成。
1.2 试验方法1.2.1 RNA提取和cDNA合成 取辣椒果实为材料,按照快速通用植物RNA提取试剂盒3.0的使用说明书进行RNA提取。用1% 琼脂糖凝胶对各样品的RNA进行电泳检测[18],用逆转录试剂盒进行cDNA的第一条链的合成,并将获得的cDNA置于-20 ℃冰箱中保存、备用[19]。
1.2.2 CMB1克隆 参照NCBI上辣椒基因组的CMB1序列,使用Primer Premier 6.0[20]进行引物设计(CF:5'-ACACAATGGGTAGAGGGA-3',CR:5'-TGCCAGACACTTGTTGAC-3'),以辣椒转色期果实的cDNA作为克隆模板进行PCR反应。PCR扩增体系:2×Phanta Max Master Mix (Dye Plus) 12.5 μL,上/ 下游引物各1 μL,模板DNA 1 μL,添加双蒸水补充至25 uL。PCR扩增程序:98 ℃ 2 min;98 ℃ 10 s、52 ℃ 30 s、72 ℃ 29 s,35个循环;72 ℃ 2 min。用1% 琼脂糖凝胶电泳检测扩增产物,随后将产物送至生工生物工程(上海)股份有限公司测序。
1.2.3 CMB1序列分析 通过NCBI(https://www.ncbi.nlm.nih.gov)进行序列比对和查找,使用ORFfinder(https://www.ncbi.nlm.nih.gov/orffinder/)进行开放阅读框及氨基酸预测,使用DNAMAN进行蛋白序列比对和跨膜结构预测。利用SignalP-5.0(https://services.healthtech.dtu.dk/services/SignalP-5.0/)对CMB1进行信号肽预测;使用ExPasy-ProtParam tool(http://web.expasy.org/protparam/)对CMB1蛋白质的相对分子质量、等电点、稳定性、平均疏水性进行分析[21];使用NetPhos-3.1(https://services.healthtech.dtu.dk/services/NetPhos-3.1/)对CMB1进行蛋白磷酸化位点预测;使用SOPM(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)、Swiss Model(https://www.expasy.org/resources/swiss-model)软件对CMB1的二级、三级结构进行分析预测;使用CDD(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)查看CMB1的保守结构域[22];使用MEGA 11.0软件和iTOL(https://itol.embl.de/)进行进化树分析[23]。
1.2.4 qRT-PCR分析 使用反转录得到的cDNA为模板,按照荧光定量试剂盒使用说明进行qPCR检测,以探究CMB1在皱皮红与皱皮黄果实3个时期的表达模式。qPCR反应体系为:10 μL Hieff® qPCR SYBR Green Master Mix(Low Rox Plus),上/ 下游引物各0.4 μL,2 μL cDNA模板,加RNase-Free H2O至20 μL。qPCR反应体系为:预变性95 ℃ 2 min;变性95 ℃ 10 s、退火55 ℃ 20 s、延伸72 ℃ 20 s,40个循环;溶解曲线阶段按照仪器的默认设置。试验设2个生物学重复、3个技术性重复。在NCBI primer-BLAST设计特异性引物QCF(5'-ATCATGGCACTCTGGGCCTG-3')和QCR(5'-GTGCTGCATATTCCGGTGGC-3'),以辣椒Actin基因为内参,内参引物序列为:ActinF(5'-GCCTAGAAATTTGAGCCTCATT-3')和ActinR(5'-GGTAGTCGTTGTTATGGTAG AA-3')[24]。
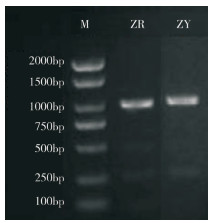
2 结果与分析 2.1 辣椒CMB1基因克隆CMB1 PCR产物经1%琼脂糖凝胶检测,显示条带长度与预期片段大小一致,约968 bp(图 1)。测序结果显示,克隆所得皱皮红与皱皮黄辣椒2个CMB1基因完整的ORF全长均为732 bp,均编码243个氨基酸残基。保守结构域预测结果显示,皱皮红CMB1和皱皮黄CMB1均具有典型的MADS-MEF2-like结构域(2~78区间)和K-box结构域(82~172区间)(图 2)。
|
| M:Maker;ZR:皱皮红;ZY:皱皮黄 M: Maker; 1: Red pepper with wrinkled skin; 2: Yellow pepper with wrinkled skin 图 1 辣椒CMB1基因全长序列PCR产物 Fig. 1 PCR products of full-length sequences of CMB1 gene in pepper |
|
| A:皱皮红CMB1;B:皱皮黄CMB1 A: CMB1 of red pepper with wrinkled skin; B: CMB1 of yellow pepper with wrinkled skin 图 2 辣椒CMB1基因编码蛋白质的保守区域 Fig. 2 Conserved region of protein encoded by the CMB1 gene in pepper |
2.2 辣椒CMB1编码蛋白的理化性质预测及结构分析
利用ExPASyProtparam预测皱皮红CMB1基因编码蛋白的分子式为C1224H1938N356O381S9,分子质量为28 025.55,理论等电点为7.87,不稳定系数为53.77,推测其为不稳定蛋白,具有32个酸性氨基酸残基(Arg+Lys)、碱性氨基酸残基(Asp+Glu)21个,脂肪指数为73.00,亲水性总平均值(重力)指数为-0.817,推测其为疏水蛋白;皱皮黄CMB1基因编码蛋白的分子式为C1220H1929N353O381S10,分子质量为27 958.47,理论等电点为7.10,不稳定系数为53.54,推测其为不稳定蛋白,具有酸性氨基酸残基(Arg+Lys)31个、碱性氨基酸残基(Asp+Glu)31个,脂肪指数为71.81,亲水性总平均值(重力)指数为-0.809,推测其为疏水蛋白。
通过SOPMA分析蛋白二级结构,结果(图 3A)发现,在皱皮红CMB1蛋白二级结构中α螺旋占56.38%,β折叠占4.53%,无规则卷曲占30.45%,延伸链占8.64%;皱皮黄CMB1蛋白二级结构中,α螺旋占53.50%,β折叠占3.70%,无规则卷曲占33.33%,延伸链占9.47%。使用Swiss-model预测皱皮黄、皱皮红CMB1蛋白三级结构(图 3B),利用GMQE对预测结果进行打分,所得到的值在0~1之间,越接近1表示越可信。皱皮红CMB1的评分为0.77,蛋白序列的一致性为70.39%;皱皮黄CMB1的评分为0.77,蛋白序列的一致性为70.94%,预测结果较为可靠。
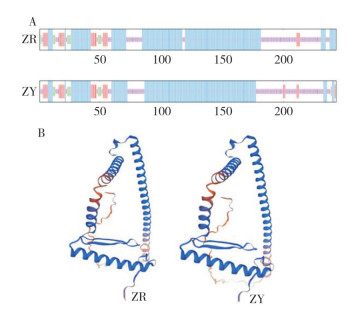
|
| A:二级结构,B:三级结构;ZR:皱皮红;ZY:皱皮黄 A: Secondary structure, B: Tertiary structure; ZR: Red pepper with wrinkled skin; ZY: Yellow pepper with wrinkled skin 图 3 辣椒CMB1蛋白结构预测 Fig. 3 Protein structure prediction of pepper CMB1 |
2.3 CMB1序列比对分析
利用DNAMAN对皱皮红、皱皮黄进行CMB1碱基序列比对,结果(图 4)发现,与皱皮黄相比,皱皮红CMB1在碱基序列中第11个位点发生突变,G突变为T,导致在氨基酸序列中第4个位点发生突变(G突变为V)。再利用DANMAN将皱皮红和皱皮黄CMB1蛋白序列与其他植物同源蛋白序列进行多序列比对,结果(图 5)表明,皱皮红、皱皮黄与遵辣1号(Capsicum annuum L.)的蛋白序列相似度最高(96.34%),与番茄(Solanum lycopersicum L.)的相似度为94.04%,与毛酸浆(Physalis pubescens L.)的相似度为93.77%,与烟草(Nicotiana attenuate Torr.)的相似度为88.89%,与三裂叶薯(Ipomoea triloba L.)相似度最低(87.40%)。

|
| 图 4 辣椒CMB1碱基序列比对 Fig. 4 CMB1 base sequence alignment in pepper |

|
| 图 5 辣椒CMB1与高同源蛋白质序列的比对结果 Fig. 5 Alignment result of CMB1 with high homologous protein sequences in pepper |
2.4 辣椒CMB1蛋白信号肽、跨膜结构与磷酸化位点预测
利用SignalP-5.0与DNAMAN进行辣椒CMB1信号肽、跨膜结构预测,结果显示,CMB1蛋白无信号肽和跨膜结构。使用NetPhos-3.1在线软件对CMB1蛋白磷酸化位点进行预测,同时对预测位点在0~1区间进行打分,结果预测位点可信度随得分从0.5~1增加而增加[25]。对皱皮红CMB1蛋白评分≥ 0.5,且以同一位点最高得分的氨基酸残基类型为准进行筛选,共得到38个磷酸化位点,其中T(苏氨酸Threonine)型11个、S(丝氨酸Serine)型23个、Y(酪氨酸Tyrosine)型4个。皱皮黄CMB1蛋白中磷酸化位点的数量和类型均与皱皮红相同。
2.5 CMB1系统进化树分析以皱皮红、皱皮黄CMB1的蛋白序列为模板进行BLAST,从BLAST结果中筛选出7个科共38条同源性较高的氨基酸序列,采用邻接法构建发育树,以分析皱皮红、皱皮黄辣椒中的CMB1与其他科各成员之间的系统进化关系。系统发育树聚类结果(图 6)显示,CMB1在科的水平上具有保守性,在亚支上,即同科不同属之间,虽然存在保守性,但也存在进化差异。茄科植物与旋花科植物亲缘关系最近,与豆科植物最远;在茄科内与茄属亲缘关系最近,与烟草属关系最远。
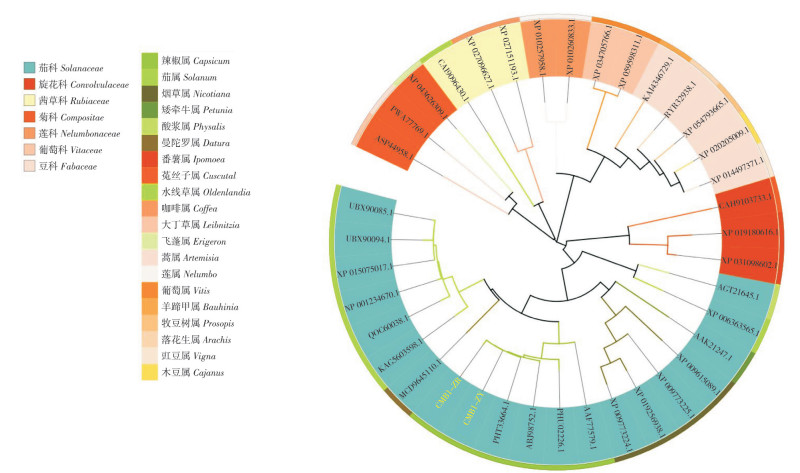
|
| ZR:皱皮红;ZY:皱皮黄 ZR: Red pepper with wrinkled skin; ZY: Yellow pepper with wrinkled skin 图 6 CMB1系统进化树分析 Fig. 6 Evolutionary tree analysis of CMB1 System |
2.6 CMB1在辣椒果实不同发育时期的差异表达
为分析CMB1基因在辣椒果实不同发育时期的表达量,采用qPCR对辣椒不同发育阶段果实中的CMB1基因表达特性进行分析。结果(图 7)表明,CMB1基因表达量在完熟期最高;在绿熟期(S1)、转色期(S2)、完熟期(S2)3个时期中的表达量呈逐渐上升趋势;在皱皮红中的表达量高于皱皮黄中的表达量,与前期研究中辣椒红素与辣椒玉红素合成的关键基因CCS的表达趋势一致[26]。
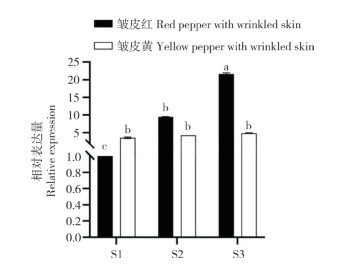
|
|
S1:绿熟期;S2:转色期;S3:完熟期 柱上小写英文字母不同表示差异显著 S1: Green ripening stage; S2: Color change stage; S3: Mature stage Different lowercase letters above the column indicate significant differences 图 7 CMB1基因在辣椒果实不同发育时期的相对表达量 Fig. 7 Relative expression levels of CMB1 gene in pepper fruits at different developmental stages |
3 讨论
MADS-box基因是真核生物中一类非常重要的转录调控因子,它们在细胞的生长发育及信号传导过程中起着至关重要的作用。MADS-box基因家族包括多个成员,它们可以结合并激活多种不同的信号传导途径,从而影响各种细胞的功能[27]。此外,MADS-box基因广泛存在于许多植物物种中,在不同发展过程中扮演不同角色,有多个属于MADS-box的蛋白参与果实成熟与类胡萝卜素积累的调控,包括TAG1、RIN、FUL1、FUL2、MADS1、CMB1、FYFL、MBP8[28]。为进一步研究CMB1基因在辣椒果实发育中的其他潜在作用,本研究从皱皮红、皱皮黄两种辣椒果实中克隆该基因并进行不同时期表达分析。
在本试验中,通过保守结构域分析发现,皱皮红和皱皮黄CMB1均具有两个高度保守的结构域。在2~78区间存在1个MADS-MEF2-like型结构域,在82~172区间有1个K-box结构域,是典型的Ⅱ型MADS-box基因MEF2-1ike转录因子。通过生物信息学分析,结果显示皱皮黄和皱皮黄CMB1均不含信号肽,也无跨膜结构,因此,推测此类蛋白质合成后就在合成处发挥作用,没有任何运输情况,不是定位于生物膜的膜蛋白。
调节植物类胡萝卜素合成的转录因子,主要通过调节类胡萝卜素合成途径中的酶基因表达来实现,进而控制通路代谢流量[29]。在番茄植物中,对CMB1进行RNA干扰处理,会导致果实中的乙烯产量降低大约50%、PSY1和PDS下调[7]。CMB1通过对PSY1和PDS基因的调控,可以影响植物中类胡萝卜素含量的积累,从而影响植物的营养价值和商品价值。本试验的qPCR结果表明,CMB1基因在辣椒果实中的表达量从绿熟期、转色期到完熟期表达量呈递增趋势,完熟期时表达量最高。类胡萝卜素含量随着果实发育成熟而增加,成熟后期类胡萝卜素积累量达到最高值,且随着辣椒果实成熟度逐渐加深,果实中的红色类胡萝卜素积累量大幅度高于黄色类胡萝卜素积累量[30]。本研究皱皮红和皱皮黄的CMB1表达量随着果实成熟度加深也呈上升趋势,变化规律与辣椒中类胡萝卜素积累趋势相似,故推测CMB1转录因子与辣椒果实中辣椒红素等类胡萝卜素的合成相关。前期研究表明,辣椒红素与辣椒玉红素在皱皮黄辣椒果实绿熟期、转色期和完熟期中几乎检测不到,而在皱皮红辣椒果实中的含量则随着发育时期呈现出不断上升的趋势;此外,辣椒红素与辣椒玉红素合成的关键基因CCS与CMB1的表达趋势一致,进一步证明CMB1可能参与调控辣椒红素与辣椒玉红素的合成。[26]
4 结论本试验成功克隆了皱皮红皱皮黄辣椒CMB1基因,全长分别为966、968 bp,两者均含有732 bp完整的ORF,编码243个氨基酸残基。序列比对发现,与皱皮黄CMB1相比,皱皮红CMB1碱基序列第11个位点存在突变,G突变为T,导致蛋白序列第4个位点G突变为V。保守结构域分析发现,皱皮红和皱皮黄CMB1均具有两个高度保守的结构域(2~78区间,MADS-MEF2-like型结构域;82~172区间,K-box结构域)。通过生物信息学分析,预测它们均不含信号肽,也无跨膜结构,是一类不稳定的疏水蛋白,含磷酸化位点38个。在二级结构中主要以α螺旋为主;三级结构预测GMQE值均为0.77。进化树系统分析显示,皱皮红和皱皮黄CMB1与茄科亲缘关系最近,与豆科亲缘关系最远。实时荧光定量PCR检测结果表明,CMB1基因在辣椒果实完熟期表达量最高,在绿熟期、转色期、完熟期3个时期中的表达量呈逐渐上升趋势,且在皱皮红中的表达量高于皱皮黄中的表达量。
| [1] |
HERMANNS A S, ZHOU XS, XU Q, TADMOR Y, LI L. Carotenoid pigment accumulation in horticultural plants[J]. Horticultural Plant Journal, 2020, 6(6): 343-360. DOI:10.1016/j.hpj.2020.10.002 |
| [2] |
张威, 张丽平, 吴丹, 成善汉, 汪志伟, 刘平武. 黄灯笼辣椒MYB4转录因子的克隆与序列分析[J]. 分子植物育种, 2023, 21(6): 1801-1810. DOI:10.13271/j.mpb.021.001801 ZHANG W, ZHANG L P, WU D, CHENG S H, WANG Z W, LIU P W. Yellow lantern chili MYB4 transcription factor of cloning and sequence analysis[J]. Journal of Molecular Plant Breeding, 2023, 21(6): 1801-1810. DOI:10.13271/j.mpb.021.001801 |
| [3] |
NIYOGI K K, TRUONG T B. Evolution of flexible non-photochemical quenching mechanisms that regulate light harvesting in oxygenic photosynthesis[J]. Current Opinion in Plant Biology, 2013, 16(3): 307-314. DOI:10.1016/j.pbi.2013.03.011 |
| [4] |
STANLEY L, YAO W Y. Transcriptional regulation of carotenoid biosynthesis in plants: so many regulators, so little consensus[J]. Frontiers in Plant Science, 2019, 10: 1017. DOI:10.3389/fpls.2019.01017 |
| [5] |
樊宝莲, 王晓云. 转录因子调控植物类胡萝卜素合成途径的研究进展[J]. 分子植物育种, 2021, 19(13): 4401-4408. DOI:10.13271/j.mpb.019.004401 FAN B L, WANG X Y. Research progress of transcription factors regulating carotenoid synthesis pathway in plant[J]. Molecular Plant Breeding, 2021, 19(13): 4401-4408. DOI:10.13271/j.mpb.019.004401 |
| [6] |
MASAKI F, TOSHITSUGU N, YOKO S, YASUHIRO I. A large-scale identification of direct targets of the tomato MADS box transcription factor RIPENING INHIBITOR reveals the regulation of fruit ripening[J]. The Plant Cell, 2013, 25(2): 371-386. DOI:10.1105/tpc.112.108118 |
| [7] |
ZHANG J L, HU Z L, YAO Q Y, GUO X H, NGUYEN V, LI F F. A tomato MADS-box protein, SlCMB1, regulates ethylene biosynthesis and carotenoid accumulation during fruit ripening[J]. Scientific Reports, 2018, 8(1): 3413. DOI:10.1038/s41598-018-21672-8 |
| [8] |
芦旺, 席万鹏. MADS-box转录因子在果实成熟及品质形成中的调控作用研究进展[J]. 园艺学报, 2018, 45(9): 1802-1812. DOI:10.16420/j.issn.0513-353x.2018-0266 LU W, XI W P. MADS-box transcription factors are involved in regulation for fruit ripening and quality development[J]. Acta Horticulturae Sinica, 2018, 45(9): 1802-1812. DOI:10.16420/j.issn.0513-353x.2018-0266 |
| [9] |
JULIA V, IRVIN L P, ANTONIO J M A, RYAN M Q, MI Y C, MERVIN P, JOCELYN R, GRAHAM S, SILVANA G, JAMES G, VIVIAN F I. Fleshy fruit expansion and ripening are regulated by the tomato SHATTERPROOF gene TAGL1[J]. The Plant Cell, 2009, 21(10): 3041-3062. DOI:10.1105/tpc.109.066936 |
| [10] |
LI S, ZHU B, PIRRELLO J, XU C J, ZHANG B, MONHDER B Z. Roles of RIN and ethylene in tomato fruit ripening and ripening‐associated traits[J]. New Phytologist, 2020, 226(2): 460-475. DOI:10.1111/nph.16362 |
| [11] |
DONG T T, HU Z L, DENG L, WANG Y, ZHU M K, ZHANG J L, CHEN G P. A tomato MADS-box transcription factor, SlMADS1, acts as a negative regulator of fruit ripening[J]. Plant Physiology, 2013, 163(2): 1026-1036. DOI:10.1104/pp.113.224436 |
| [12] |
XIE Q L, HU Z L, ZHU Z G, DONG T T, ZHAO Z P, CUI B L, CHENG G P. Overexpression of a novel MADS-box gene SlFYFL delays senescence, fruit ripening and abscission in tomato[J]. Scientific Reports, 2014, 4(1): 4367. DOI:10.1038/srep04367 |
| [13] |
YIN W C, HU Z L, CUI B L, GUO X H, HU J T, ZHU Z G, CHENG G P. Suppression of the MADS-box gene SlMBP8 accelerates fruit ripening of tomato (Solanum lycopersicum)[J]. Plant Physiology and Biochemistry, 2017, 118: 235-244. DOI:10.1016/j.plaphy.2017.06.019 |
| [14] |
MA J, DAI J, LIU X W, DUO L. The transcription factor CaBBX20 regulates capsanthin accumulation in pepper (Capsicum annuum L.)[J]. Scientia Horticulturae, 2023, 314: 111907. DOI:10.1016/j.scienta.2023.111907 |
| [15] |
WEI X C, MENG C Y, YUAN Y X, NATH U K, ZHAO Y Y, WANG Z Y, YANG S J, LI L, NIU L J, YAO Q J, WEI F, ZHANG X W. CaPSY1 gene plays likely the key role in carotenoid metabolism of pepper (Capsicum annuum) at ripening[J]. Functional Plant Biology, 2020, 48(2): 141-155. DOI:10.1071/fp19287 |
| [16] |
LI Q H, YANG X P, YU Y N, KHAN A, FENG P L, ALI M H, SHAO D K, WANG Y Y, ZHANG R X, GAI W X, HAN R, MA X, HOU Q G, GONG Z H. Comprehensive transcriptome-based characterization of differentially expressed genes involved in carotenoid biosynthesis of different ripening stages of Capsicum[J]. Scientia Horticulturae, 2021, 288: 110311. DOI:10.1016/j.scienta.2021.110311 |
| [17] |
谢炳春, 黄俊霖, 温松森, 李涛, 李颖, 徐小万, 徐晓美, 吴智明, 衡周. 中国辣椒BCAT基因家族鉴定、表达分析及克隆[J]. 广东农业科学, 2022, 49(12): 34-43. DOI:10.16768/j.issn.1004-874X.2022.12.004 XIE B C, HUANG J L, WEN S S, LI T, LI Y, XU X W, XU X M, WU Z M, HENG Z. Identification, expression analysis and cloning of Capsicum chinense BCAT gene family[J]. Guangdong Agricultural Sciences, 2022, 49(12): 34-43. DOI:10.16768/j.issn.1004-874X.2022.12.004 |
| [18] |
谭政委, 鲁丹丹, 李磊, 余永亮, 许兰杰, 董薇, 杨红旗, 杨青, 李春明, 安素妨, 梁慧珍. 红花类黄酮3-O-糖基转移酶基因CtUF3GT的克隆及功能鉴定[J]. 药学学报, 2022, 57(8): 2543-2551. DOI:10.16438/j.0513-4870.2022-0378 TAN Z W, LU D D, LI L, YU Y L, XU L J, DONG W, YANG H Q, YANG Q, LI C M, AN S W, LIANG H Z. Cloning and functional identification of safflower flavonoid 3-O-glycosyltransferase gene CtUF3GT[J]. Chinese Journal of Pharmaceutical Sciences, 2022, 57(8): 2543-2551. DOI:10.16438/j.0513-4870.2022-0378 |
| [19] |
侯传明, 郑雅文, 王正加, 徐英武. 山核桃MADS-like基因的克隆与分析[J]. 浙江农林大学学报, 2015, 32(1): 33-39. DOI:10.11833/j.issn.2095-0756.2015.01.005 HOU C M, ZHENG Y W, WANG Z J, XU Y W. Cloning and analysis of a MADS-like gene in Carya cathayensis[J]. Journal of Zhejiang A & F University, 2015, 32(1): 33-39. DOI:10.11833/j.issn.2095-0756.2015.01.005 |
| [20] |
张祥, 刘雨婷, 张婧柔, 李平平, 谢志和, 张芮豪, 邓明华. 涮辣与昆明皱皮椒苯丙氨酸解氨酶(PAL) 的生物信息学及表达分析[J]. 云南农业大学学报(自然科学), 2022, 37(2): 320-329. DOI:10.12101/j.issn.1004-390X(n).202103102 ZHANG X, LIU Y T, ZHANG J R, LI P P, XIE Z H, ZHANG R H, DENG M H. Bioinformatics and expression analysis of Phenylalanine Ammonia-lyase (PAL) from Shuanla (Capsicum chinense) and Kunming wrinkled pepper (C. annuum)[J]. Journal of Yunnan Agricultural University (Natural Science), 2022, 37(2): 320-329. DOI:10.12101/j.issn.1004-390X(n).202103102 |
| [21] |
廖铭宇, 肖佳林, 李丽缘, 宋钰, 黄湖荣, 杨博智. 辣椒雄性不育系和保持系CaMADS6基因克隆及表达分析[J]. 华北农学报, 2023, 38(1): 46-52. DOI:10.7668/hbnxb.20193440 LIAO M Y, XIAO J L, LI L Y, SONG Y, HUANG H R, YANG B Z. Cloning and expression analysis of CaMADS6 in cytoplasmic male sterile line and maintainer line of pepper[J]. Acta Agriculturae Boreali-Sinica, 2023, 38(1): 46-52. DOI:10.7668/hbnxb.20193440 |
| [22] |
杨晓琳, 段迎, 蔡苏云, 贺润丽, 尹桂芳, 王艳青, 卢文洁, 孙道旺, 王莉花. 苦荞FtHCT的基因克隆与生物信息学分析[J]. 分子植物育种, 2023, 21(1): 102-109. DOI:10.13271/j.mpb.021.000102 YANG X L, DUAN Y, CAI S Y, HE R L, YIN G F, WANG Y Q, LU W J, SUN D W, WANG L H. Cloning and bioinformatics analysis of gene FtHCT from tatary buckwheat[J]. Molecular Plant Breeding, 2023, 21(1): 102-109. DOI:10.13271/j.mpb.021.000102 |
| [23] |
周荣, 刘嘉超, 杨凤玺. 墨兰CsAP1-A基因克隆及表达分析[J]. 广东农业科学, 2023, 50(9): 99-107. DOI:10.16768/j.issn.1004-874X.2023.09.010 ZHOU R, LIU J C, YANG F X. Analysis on cloning and expression of CsAP1-A gene in Cymbidium sinense[J]. Guangdong Agricultural Sciences, 2023, 50(9): 99-107. DOI:10.16768/j.issn.1004-874X.2023.09.010 |
| [24] |
刀梅, 刘琅, 杨自云, 王娟, 吴田 '. 凤丹'牡丹PoKAS基因的克隆及表达分析[J]. 西北植物学报, 2023, 43(2): 211-219. DOI:10.7606/j.issn.1000-4025.2023.02.0211 DAO M, LIU L, YANG Z Y, WANG J, Wu T. 'Cloning and expression analysis of PoKAS gene in Pontiana peonica[J]. Northwest Journal of Botany, 2023, 43(2): 211-219. DOI:10.7606/j.issn.1000-4025.2023.02.0211 |
| [25] |
朱云娜, 杨景辉, 王斌王玉昆, 周裕荣, 卢威宇, 刘建国. 菜心BcCIPK23基因克隆及表达特性分析[J]. 广东农业科学, 2023, 50(9): 89-98. DOI: 10.16768/j.issn.1004-874X.2023.09.009. ZHU Y N, YANG J H, WANG B, WANG Y K, ZHOU Y R, LU W Y, LIU J G. Cloning and expression characteristics analysis of BcCIPK23 in flowering Chinese cabbage[J]. Guangdong Agricultural Sciences, DOI: 10.16768/j.issn.1004-874X.2023.09.009. |
| [26] |
LV J H, ZHANG R H, MO Y R, ZHOU H D, LI M J, WU R, CHENG H, ZHANG M X, WANG H S, HUA W, DENG Q L, ZHAO K, DENG M H. Integrative metabolome and transcriptome analyses provide insights into carotenoid variation in different-colored peppers[J]. International Journal of Molecular Sciences, 2023, 24(23): 16563. DOI:10.3390/ijms24231653 |
| [27] |
LI C X, LU X F, XU J R, LIU Y Z. Regulation of fruit ripening by MADS-box transcription factors[J]. Scientia Horticulturae, 2023, 314: 111950. DOI:10.1016/j.scienta.2023.111950 |
| [28] |
LI C X, HOU X M, QI N N, LIU H W, LI Y H, HUANG D J, WANG C L, LIAO W B. Insight into ripening-associated transcription factors in tomato: A review[J]. Scientia Horticulturae, 2021, 288: 110363. DOI:10.1016/j.scienta.2021.110363 |
| [29] |
王莹, 穆艳霞, 王锦. MADS-box基因家族调控植物花器官发育研究进展[J]. 浙江农业学报, 2021, 33(6): 1149-1158. DOI:10.3969/j.issn.1004-1524.202.06.21 WANG Y, MU Y X, WANG J. Research progress of floral development regulation by MADS-box gene family[J]. Acta Agriculturae Zhejiangensis, 2021, 33(6): 1149-1158. DOI:10.3969/j.issn.1004-1524.202.06.21 |
| [30] |
陈奕聪, 杜奋澄, 许杰纯, 杨雨欣, 黎祖瑜, 吴智明. 不同果色辣椒类胡萝卜素累积差异分析[J]. 仲恺农业工程学院学报, 2018, 31(4): 1-6, 14. DOI:10.3969/j.issn.1674-5663.2018.04.001 CHENG Y C, DU F C, XU J C, YANG Y X, LI Z Y, WU Z M. Carotenoids accumulative difference in pepper fruit with different colour[J]. Journal of Zhongkai University of Agriculture and Engineering, 2018, 31(4): 1-6, 14. DOI:10.3969/j.issn.1674-5663.2018.04.001 |
(责任编辑 崔建勋)
 2023, Vol. 50
2023, Vol. 50