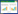文章信息
基金项目
- 浙江省科技厅财政专项(2022CZZX005);国家淡水水产种质资源库长江流域种质资源分库建设项目(2022)
作者简介
- 刘士力(1985—),男,博士,助理研究员,研究方向为水生动物遗传育种,E-mail:liushili1212@126.com.
通讯作者
- 李飞(1986—),男,硕士,副研究员,研究方向为水产种质资源和新品种选育,E-mail:lifeibest1022@163.com.
文章历史
- 收稿日期:2023-06-21
2. 上海海洋大学水产与生命学院/农业农村部淡水水产种质资源重点实验室,上海 201306;
3. 湖州师范学院生命科学学院,浙江 湖州 313001
2. College of Fisheries and Life, Shanghai Ocean University/Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Agriculture and Rural Affairs, Shanghai 201306, China;
3. College of Life Sciences, Huzhou University, Huzhou 313001, China
【研究意义】黄尾鲴(Xenocypris davidi)隶属于鲤科(Cyprinidae)鲴亚科(Xenocyprinae)鲴属(Xenocypris)[1-2],是一种广泛分布于我国长江、珠江流域的淡水经济鱼类,以藻类及植物碎片、有机物碎屑为饵料,兼食浮游动物和底栖动物,具有较高的生态价值与经济意义[3]。黄尾鲴可有效抑制水体富营养化、控制藻类异常增殖,是净化水质的主要鱼种之一,我国浙江省将其作为增殖放流的主要鱼类之一[4]。了解人工繁育对黄尾鲴遗传多样性的影响,可为自然水域黄尾鲴的增殖放流策略设计和实施提供参考。【前人研究进展】增殖放流策略设计和实施需要基础数据的支持,种群遗传学分析能够为鱼类种质资源保护与开发提供基本信息[5]。线粒体DNA(Mitochondrial DNA,mtDNA)是鱼类群体遗传结构和系统演化关系分析的重要工具,具有结构简单、进化速度快、母性遗传等特点[6-13]。其中,mtDNA中的D-loop序列,作为非编码区还拥有受进化压力小、遗传变异率高等优势,是mtDNA中进化速率最快的序列,被广泛用于鱼类的物种鉴定、种群遗传结构分析等研究[14-15]。当前国内有关黄尾鲴的研究报道多集中在其生物学特性、增殖放流、人工繁殖和苗种繁育等方面[16-17],而关于黄尾鲴遗传资源的分子研究还不多。张峻德等[16]利用线粒体COⅠ基因序列,对千岛湖水库共栖的细鳞鲴和黄尾鲴的遗传资源状况进行研究,发现黄尾鲴4个单倍型分为两个较为清晰的分支,同时发现各种群未因距离或千岛湖复杂的水文条件形成隔离。张宏等[17]基于线粒体COⅠ基因对千岛湖和长江黄尾鲴5个地理种群的遗传多样性进行分析,其中泾县群体与不同水系的钱塘江群体和同一水系的南昌群体均存在显著遗传分化。
【本研究切入点】近年来,利用COⅠ等基因序列对黄尾鲴群体进行遗传多样性分析已有较多应用,但采用mtDNA D-loop序列对黄尾鲴群体进行遗传多样性研究尚未见报道。【拟解决的关键问题】本试验对4个黄尾鲴养殖群体的线粒体D-loop序列进行扩增和测序,并对4个群体的遗传结构进行分析,旨在揭示4个群体之间的遗传关系,同时对黄尾鲴种质数据进行补充,为黄尾鲴的种质资源保护与开发提供理论依据。
1 材料与方法 1.1 试验材料试验于2022年在农业农村部淡水渔业健康养殖重点实验室开展。供试黄尾鲴采自浙江长兴、八里店、双浦和湖南醴陵4个黄尾鲴人工繁育基地,分别随机选取32尾,样本共计128尾。剪取适量尾鳍组织加入无水乙醇进行固定,于4 ℃下保存备用。
1.2 DNA提取供试样本DNA的提取采用酚-氯仿抽提法[18],经裂解、萃取和纯化获得的DNA通过1% 琼脂糖凝胶电泳检测质量后于-20 ℃下低温保存。
1.3 线粒体D-loop序列的扩增与测序PCR所用Premix TaqTM酶购自宝日医生物技术(北京)有限公司,所用引物依据黄尾鲴mtDNA基因组序列(GenBank登录号:KF039718)设计。上下游引物序列分别为D-loop-F:5'-CACCCCTGGCTCCCAAAGCCA-3',D-loop-R:5'-ATTGCGGAGACTTGCATGTGTAA-3'。PCR反应体系和反应程序采用常规方法[7]。使用1.0% 琼脂糖凝胶电泳检测PCR产物完整性后送生工生物工程(上海)股份有限公司进行双向测序。
1.4 数据处理及分析利用BioEdit 7.0软件[19]比对mtDNA D-loop区域序列。应用Mega 7.0软件[20]计算碱基含量和转换/颠换比值(TS/TV),并基于邻接法中的Kimura’s 2-Parameter模型构建系统进化树。运用DnaSP 5.0软件[21]确定各群体mtDNA D-loop区域的单倍型数,计算4个群体的变异位点数、单倍型多样性指数(h)和核苷酸多样性指数(π),并进行软件分组,通过Arlequin 3.1软件[22]分子方差(AMOVA)分析计算遗传分化系数(Fst)。
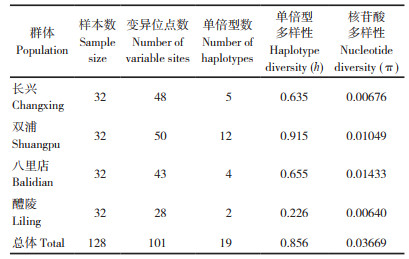
2 结果与分析 2.1 黄尾鲴D-loop序列碱基组成与变异分析本试验共获得来自4个黄尾鲴养殖群体的128条大小为1 038~1 093 bp的mtDNA D-loop区域同源序列,上传至GenBank(登录号:OQ599733~OQ599860)。除去5' 端11 bp的tRNA-Pro片段、3'端93 bp的tRNA-Phe和12S ribosomal RNA片段。选择其中934~989 bp的D-loop序列用于下一步分析。其中醴陵群体的长度为988~989 bp,另外3个群体的长度为934~937 bp,这个差异是由于醴陵群体有一段52 bp的插入片段。
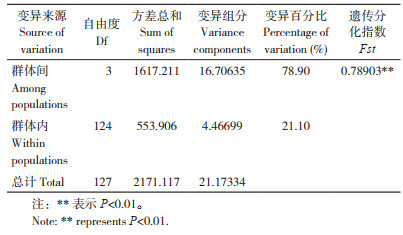
结果(表 1)显示,黄尾鲴除去空位后在932 bp序列中共检测到101个变异位点,突变率达10.8%,包含97个简约信息位点。序列中4种碱基A、G、T和C平均含量分别为33.8%、14.0%、31.5% 和20.7%,且A+T平均含量(65.3%)显著高于C+G平均含量(34.7%),碱基G的含量最低,存在反G偏倚性。计算转换/颠换比值(TS/TV)为4.6。湖南的醴陵群体与浙江的3个群体碱基含量存在一定差异。
|
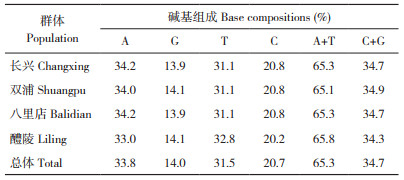
基于D-loop序列,在4个群体128个个体中检测出19种单倍型。其中,浙江双浦群体有12种单倍型,其中11种为该群体独有的单倍型,另有与长兴和八里店群体共享1种单倍型。长兴群体的单倍型数目为5种,其中特有单倍型1种,长兴群体包含八里店群体的所有4种单倍型。湖南醴陵群体仅有2种单倍型,且均为该群体独有的单倍型(表 2)。
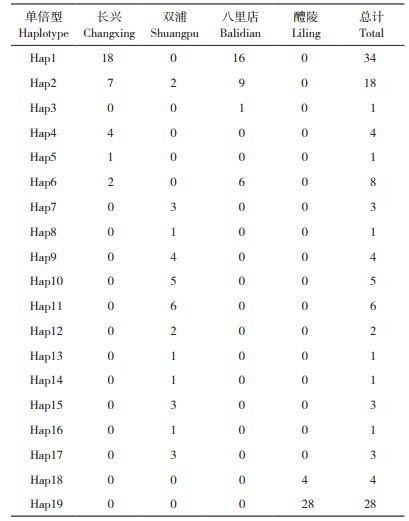
|
2.2 黄尾鲴D-loop序列遗传结构分析
遗传多样性分析结果(表 3)显示,4个黄尾鲴养殖群体的平均单倍型多样性为0.608,其中双浦群体最高,醴陵群体最低;4个黄尾鲴养殖群体单倍型多样性为0.226~0.915;核苷酸多样性为0.00640~0.01433,其中八里店群体最高(0.01433),醴陵仍为最低。结果表明,4个黄尾鲴养殖群体具有不同的遗传多样性。
|
AMOVA分析结果(表 4)显示,4个黄尾鲴养殖群体的群体间变异和群体内变异分别占整个遗传变异的78.90% 和21.10%,遗传分化系数Fst为0.78903,呈极显著(P < 0.01)。可见,整体上4个黄尾鲴养殖群体间拥有较高程度的遗传分化水平。
2.3 黄尾鲴群体遗传距离分析
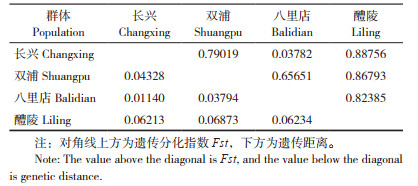
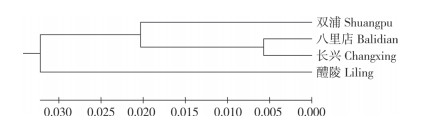
根据K2P模型利用Mega 7.0软件计算得到4个黄尾鲴群体间的遗传距离为0.01140~0.06873,各群体间遗传距离差距较大(表 5)。其中,浙江长兴与八里店群体的遗传距离最近、为0.01140,浙江双浦和湖南醴陵群体的遗传距离最远、为0.06873(图 1)。各群体间的Fst值介于0.03782~0.88756之间。其中,长兴与八里店群体之间的Fst(0.03782)最低,其他任意2个群体间的Fst均在0.5以上。
|
|
| 图 1 基于D-loop序列单倍型构建的黄尾鲴系统树 Fig. 1 Phylogenetic tree of Xenocypris davidi based on D-loop sequences haplotype |
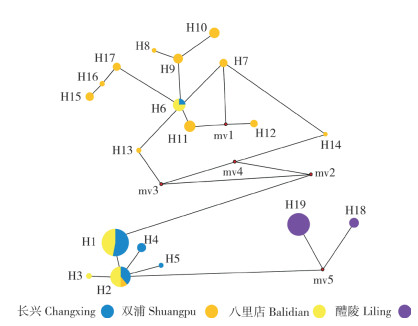
黄尾鲴群体的单倍型网络关系(图 2)显示,4个黄尾鲴群体的单倍型存在一定分化。醴陵、长兴、双浦和八里店群体的单倍型主要形成3个部分。单倍型H1~H5形成第一部分,主要以长兴和八里店群体中的个体为主;第二部分包含单倍型H6~H17,主要以双浦群体中的个体为主;醴陵群体中的个体均位于第三部分。
|
| 图 2 4个黄尾鲴养殖群体D-loop序列单倍型的网络关系 Fig. 2 Median-joining network for haplotypes of D-loop sequences in four cultured populations of Xenocypris davidi |
3 讨论
本研究通过对4个黄尾鲴养殖群体128条D-loop序列进行分析,结果表明,序列碱基出现了反G偏倚性现象;A+T含量(65.3%)显著高于G+C含量(34.7%),且各群体碱基差异较小。这与其他鱼类D-loop区域序列分析结果类似[23-28],也符合脊椎动物mtDNA碱基含量分布情况[29]。其中,碱基G的含量为所有碱基中最低(仅为14.0%),与鱇浪白鱼(Anabarilius grahami)线粒体D-loop区域G碱基含量少的现象相近[24],表明该现象属于鱼类线粒体基因序列的共同特征。这种高A/T的碱基组成现象可能是D-loop序列快速变异进化造成的[25-26]。
本研究分析结果表明,4个黄尾鲴养殖群体、128个个体中存在19种单倍型。长兴群体包含了八里店群体的所有单倍型,有1种为其特有,可能是因为长兴群体种源来自于八里店,其亲本是由历年多次采购的苗种培育而成。而双浦群体具有独有的11种单倍型,双浦群体种质来源明显有别于长兴和八里店群体。此外,32尾醴陵群体黄尾鲴遗传多样性较低,仅有2种单倍型,且其中1种单倍型占该群体的87.5%,说明本研究供试的醴陵人工养殖群体可能由少量亲本繁殖而来,若逃逸到自然水域中可能会对野生种质资源产生破坏。在动物遗传多样性分析中,单倍型多样性指数(h)和核酸多样性指数(π)通常作为评估动物群体遗传多样性的重要指标[6-9]。杨子萍等[27]运用D-loop区序列分析遗传多样性发现,洪泽湖6个群体233尾翘嘴鲌(Ulter alburnus)为“高h低π”模式,表明群体间遗传多样性较低,遗传分化不显著。周其椿等[28]对贵州青山、新店河流中的普安银鲫D-loop序列进行多态性分析,结果发现其呈现“高h高π”模式,遗传多样性高。在本研究中,湖南醴陵养殖群体呈现“低h和高π”模式,可能是人工繁殖过程中出现过瓶颈效应。浙江3个养殖群体均出现较高的单倍型遗传多样性(h=0.635~0.915)和相对较低的核酸多样性(π=0.00676~0.01433),呈现出“高h高π”模式,表明这3个养殖群体的多样性程度均较高[30]。
评估种群多态性的重要指标包括群体间的遗传分化指数(Fst)和遗传距离,这些指标也可以反映种群间的遗传相关性[31-32]。本次试验中,各群体间遗传距离为0.01140~0.06873,遗传距离差距较大,部分群体的分化程度高于种群水平(0.00200~0.06500),说明该批黄尾鲴群体间存在显著的遗传分化。同时,黄尾鲴各群体间Fst均较高且差异显著,表明各群体间出现不同程度分化。除长兴与八里店群体之间的Fst(0.03782)小于0.05,属于轻度分化,其他任意2个群体间的Fst均在0.5以上,表明存在极高度的分化。可能是栖息地差异和人类繁育及增殖放流活动影响了种群遗传结构,使得群体之间出现较大差异[25]。本研究中,种群间的遗传距离分析与Fst分析结果完全一致。不同养殖群体间的极高度分化,表明在人工繁育和增殖放流时选择不同苗种作为亲本至关重要。
综上所述,本研究以mtDNA D-loop区域为依据检测了4个黄尾鲴养殖群体,湖南醴陵群体的遗传多样性相对较低,这与以往利用其他分子标记分析黄尾鲴遗传变异的研究结果一致。这可能是在人工繁育时未选择合适的亲本所导致的不良遗传状况。而其余群体均体现了高遗传多样性和高度遗传分化,这也说明了人工繁育苗种的多样性对于增殖放流的重要性。张宏等[17]对千岛湖和长江共5个地理种群共92尾黄尾鲴的线粒体COⅠ基因进行分析,仅发现了10种单倍型,且COⅠ基因核苷酸和单倍型多样性普遍低于本研究中利用D-loop区域分析的4个群体,这说明D-loop区域序列具有更高的序列多样性。因此,采用D-loop区域序列进行分析可以作为COⅠ基因分析的重要补充,为其增殖放流及种质资源保护提供技术支持。
4 结论本研究利用mtDNA D-loop区域序列对4个黄尾鲴养殖群体进行遗传多样性分析,结果表明,4个黄尾鲴养殖群体的遗传多样性存在差异,遗传分化情况与种质来源相符。其中湖南醴陵群体仅有2种单倍型,且均为该群体独有,其单倍型多样性和核苷酸多样性最低。单倍型的网络关系和系统树均说明湖南醴陵群体与浙江3个群体遗传距离较远。双浦群体具有12种单倍型,其中11种为其独有,其单倍型多样性最高。长兴群体具有5种单倍型,除包含八里店群体的4种单倍型外,独享1种单倍型。长兴和八里店群体遗传距离最近,遗传分化系数最低。本研究获得的4个黄尾鲴养殖群体的D-loop区域数据是对黄尾鲴种质数据的重要补充。研究结果可为制定合理的黄尾鲴种质资源增殖放流和物种保护政策提供初步依据。
| [1] |
李琳, 陈蔚涛, 汤永涛, 周传江, 何舜平, 冯晨光. 鲴亚科分子系统学研究[J]. 水生生物学报, 2023, 47(4): 628-636. DOI:10.7541/2023.2022.0085 LI L, CHEN W T, TANG Y T, ZHOU C J, HE S P, FENG C G. Molecular systematics of Xenocyprinae (Cypriniformes, Cyprinidae)[J]. Acta Hydrobiolog ica Sinica, 2023, 47(4): 628-636. DOI:10.7541/2023.2022.0085 |
| [2] |
BAKER H K, HANKINS D C, SHURIN J B. Introgressive hybridization erodes morphological divergence between lentic and lotic habitats in an endangered minnow[J]. Ecology and Evolution, 2021, 11(19): 13593-13600. DOI:10.1002/ece3.8086 |
| [3] |
李长春. 水库黄尾鲴(Xenocypris davidi Bleeker)种群生态学及其数量变动规律的研究[J]. 水产科技情报, 1978(11): 10-14, 32. DOI:10.16446/j.cnki.1001-1994.1978.11.003 LI C C. Study on the population ecology and quantitative variation of Xenocypris davidi Bleeker in the reservoir.[J]. Fisheries Science & Technology Information, 1978(11): 10-14, 32. DOI:10.16446/j.cnki.1001-1994.1978.11.003 |
| [4] |
郭爱环, 原居林, 练青平, 倪蒙, 刘梅, 顾志敏. 基于微卫星标记的钱塘江上游黄尾鲴增殖放流效果及其潜在遗传风险评估[J]. 水产学报, 2022, 46(12): 2349-2356. DOI:10.11964/jfc.20210112600 GUO A H, YUAN J L, LIAN Q P, NI M, LIU M, GU Z M. Evaluation of enhancement effect and potential genetic risk of Xenocypris davidi in the upper reaches of Qiantang River based on microsatellite markers[J]. Journal of Fisheries of China, 2022, 46(12): 2349-2356. DOI:10.11964/jfc.20210112600 |
| [5] |
ALLENDORF F W, LEARY R F. Conservation genetics considerations in fishery management[J]. Biological Conservation, 1993, 63(2): 190. DOI:10.1016/0006-3207(93)90518-6 |
| [6] |
李威娜, 郭美慧, 温锦添, 翁茁先, 陈洁波, 杜炳旺, 黄勋和. 基于线粒体DNA D-loop全序列的麒麟鸡遗传多样性研究[J]. 广东农业科学, 2023, 50(4): 100-107. DOI:10.16768/j.issn.1004-874X.2023.04.012 LI W N, GUO M H, WEN J T, WENG Z X, CHEN J B, DU B W, HUANG X H. Study on genetic diversity of Qilin chicken based on complete mitochondrial DNA D-loop sequences[J]. Guangdong Agricultural Sciences, 2023, 50(4): 100-107. DOI:10.16768/j.issn.1004-874X.2023.04.012 |
| [7] |
刘士力, 贾永义, 迟美丽, 蒋文枰, 程顺, 顾志敏, 王雨辰. 浙江3个河川沙塘鳢群体线粒体16S RNA基因序列的研究[J]. 基因组学与应用生物学, 2018, 37(11): 4742-4748. DOI:10.13417/j.gab.037.004742 LIU S L, JIA Y Y, CHI M L, JIANG W P, CHEN S, GU Z M, WANG Y C. Research on mitochondrial 16S rRNA genetic sequence from three populations of Odontobutis potamophila in Zhejiang Province[J]. Genomics and Applied Biology, 2018, 37(11): 4742-4748. DOI:10.13417/j.gab.037.004742 |
| [8] |
刘士力, 卞玉玲, 贾永义, 迟美丽, 李飞, 郑建波, 程顺, 顾志敏. 基于线粒体COⅠ基因序列的红螯螯虾养殖群体遗传结构分析[J]. 浙江农业学报, 2021, 33(8): 1385-1392. DOI:10.3969/j.issn.1004-1524.2021.08.05 LIU S L, BIAN Y L, JIA Y Y, CHI M L, LI F, ZHENG J B, CHEN S, GU Z M. Genetics analysis based on mitochondrial COⅠ sequences in five cultured populations of red-claw crayfish (Cherax quadricarinatus)[J]. Acta Agriculturae Zhejiangensis, 2021, 33(8): 1385-1392. DOI:10.3969/j.issn.1004-1524.2021.08.05 |
| [9] |
刘士力, 练青平, 贾永义, 迟美丽, 李飞, 姜建湖, 刘一诺, 郑建波, 程顺, 顾志敏. 基于线粒体Cyt b基因序列的浙江省3个马口鱼群体遗传多样性分析[J]. 浙江农业学报, 2023, 35(2): 293-300. DOI:10.3969/j.issn.1004-1524.2023.02.06 LIU S L, LIAN Q P, JIA Y Y, CHI M L, LI F, JIANG J H, LIU Y N, ZHENG J B, CHEN S, GU Z M. Genetic diversity analysis of three Opsariichthys bidens populations in Zhejiang Province based on mitochondrial Cyt b gene sequences[J]. Acta Agriculturae Zhejiangensis, 2023, 35(2): 293-300. DOI:10.3969/j.issn.1004-1524.2023.02.06 |
| [10] |
杨子拓, 刘丽. 线粒体分子标记技术在水产养殖行业的应用[J]. 广东农业科学, 2014, 41(22): 135-140, 145. DOI:10.16768/j.issn.1004-874X.2014.22.035 YANG Z T, LIU L. Application of mitochondrial molecular marker technology in aquaculture[J]. Guangdong Agricultural Sciences, 2014, 41(22): 135-140, 145. DOI:10.16768/j.issn.1004-874X.2014.22.035 |
| [11] |
赵文浩, 易少奎, 苏君晓, 周琼, 沈建忠, 李大鹏, 周小云. 新疆塔里木河叶尔羌高原鳅遗传多样性研究[J]. 水生生物学报, 46(3): 364-374. DOI:10.7541/2022.2021.006 ZHAO W H, YI S K, SU J X, ZHOU Q, SHEN J Z, LI D P, ZHOU X Y. Genetic diversity of Triplophysa yarkandensis populations in Tarim River basin in Xinjiang[J]. Acta Hydrobiologica Sinica, 46(3): 364-374. DOI:10.7541/2022.2021.006 |
| [12] |
XIA L M, GUO B Y, YE Y Y, LI J J, WU C W. Determination of genetic diversity of the cuttlefish Sepiella japonica inhabiting Chinese coastal waters using the mitochondrial D-loop region: The valuable inspiration to artificial releasing project[J]. Biochemical Systematics and Ecology, 2016, 69: 274-282. DOI:10.1016/j.bse.2016.05.015 |
| [13] |
GRANT W S, BOWEN B W. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. The Journal of Heredity, 1998, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
| [14] |
SHAKLEE J B, JHON C D. Speciation and evolution of marine fishes studied by electrophoresis analysis of proteins[J]. Pacific Science, 1982, 36: 141-157. DOI:10.1093/jhered/89.5.415 |
| [15] |
WRIGHT S. Evolution and the genetics of populations, Volume 4: Var iability within and among natural populations[M]. Chicago: University of Chicago Press, 1984.
|
| [16] |
张峻德, 赵良杰, 刘其根. 千岛湖细鳞鲴和黄尾鲴COI种群遗传结构比较的初步研究[J]. 淡水渔业, 2013, 43(5): 3-7. DOI:10.3969/j.issn.1000-6907.2013.05.001 ZHANG J D, ZHAO L J, LIU Q G. Primary study on comparison of population genetic structure between Xenocypris microlepis and X.davidi in Qiandao lake base on mitochondrial COI gene[J]. Freshwater Fisheries, 2013, 43(5): 3-7. DOI:10.3969/j.issn.1000-6907.2013.05.001 |
| [17] |
张宏, 赵良杰, 胡忠军, 刘军, 刘其根. 千岛湖和长江黄尾鲴种群的遗传变异研究[J]. 上海海洋大学学报, 2015, 24(1): 12-19. ZHANG H, ZHAO L J, HU Z J, LIU J, LIU Q G. Genetic variation analysis of Xenocypris davidi populations from Qiandao Lake and Yangtze River[J]. Journal of Shanghai Ocean University, 2015, 24(1): 12-19. |
| [18] |
SAMBROOK J, RUSSELL D W. Molecular cloning: a laboratory manual (Third edition)[M]. New York: Cold Spring Harbor Laboratory Press, 2001: 623-625.
|
| [19] |
HALL T A. Bioedit: A user - friendly biological sequence alignment editor and analysis program for windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1999, 41: 95-98. DOI:10.1021/bk-1999-0734.ch008 |
| [20] |
KUMAR S, ST ECHER G, TAMUR A K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33: 1870-1874. DOI:10.1093/molbev/msw054 |
| [21] |
LIBRADO P, ROZAS J. DnaSP v5: A software for comprehensive ana lysis of DNA polymor phism data[J]. Bioinfor mat ics, 2009, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 |
| [22] |
EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567. DOI:10.1111/j.1755-0998.2010.02847.x |
| [23] |
李潇轩, 郑端端, 罗明坤, 杨志强, 郭珺, 王兰梅, 董在杰. 不同体色锦鲤和" 福瑞鲤2号"mtDNA D-loop序列遗传变异分析[J]. 上海海洋大学学报, 2023, 32(2): 266-274. DOI:10.12024/jsou.20211103632 LI X X, ZHENG D D, LUO M K, YANG Z Q, GUO J, WANG L M, DONG Z J. Genetic variation analysis of mtDNA D-loop sequences in different body color koi carp and FFRC No.2 strain common carp[J]. Journal of Shanghai Ocean University, 2023, 32(2): 266-274. DOI:10.12024/jsou.20211103632 |
| [24] |
吴俊颉, 李光华, 金方彭, 赵静霞, 雷春云, 高海涛, 符世伟, 周睿, 罗永新, 薛绍伟, 张文魁, 冷云, 梁用本, 马春明, 王艳. 基于线粒体D-loop区的抚仙湖鱇浪白鱼遗传多样性分析[J]. 水生生物学报, 2022, 46(3): 385-394. DOI:10.7541/2022.2020.250 WU J J, LI G H, JING F P, ZHAO J X, LIE C Y, GAO H T, FU S W, ZHOU R, LUO Y X, XUE S W, ZHANG W K, LENG Y, LIANG Y B, MA C M, WANG Y. Genetic diversity analysis of mitochondrial D-loop region of Anabarilius graham in Fuxian Lake[J]. Acta Hydrobiologica Sinica, 2022, 46(3): 385-394. DOI:10.7541/2022.2020.250 |
| [25] |
时伟, 孔晓瑜, 江金霞, 苗宪广. 舌鳎亚科鱼类线粒体控制区快速进化及其重复序列延伸机制初步研究[J]. 中国海洋大学学报(自然科学版), 2012, 42(1/2): 81-87. DOI:10.16441/j.cnki.hdxb.2012.z1.013 SHI W, KONG X Y, JIANG J X, MIAO X G. Preliminary study on the rapid evolution of mtDNA control region and the elongated mechanism of tandem repeat units in Cynoglossinae fishes[J]. Periodical of Ocean University of China(Natural Science Edition), 2012, 42(1/2): 81-87. DOI:10.16441/j.cnki.hdxb.2012.z1.013 |
| [26] |
杨博, 陈小勇, 杨君兴. 鱇浪白鱼线粒体DNA控制区结构和种群遗传多样性分析[J]. 动物学研究, 2008, 29(4): 379-385. DOI:10.3724/SP.J.1141.2008.04379 YANG B, CHEN X Y, YANG J X. Structure of the mitochondrial DNA control region and population genetic diversity analysis of Anabarilius grahami (Regan)[J]. Zoological Research, 2008, 29(4): 379-385. DOI:10.3724/SP.J.1141.2008.04379 |
| [27] |
杨子萍, 李大命, 刘燕山, 谷先坤, 杨家新. 基于线粒体控制区D-loop序列的洪泽湖翘嘴鲌遗传多样性分析[J]. 南京师大学报(自然科学版), 2023, 46(2): 70-76. DOI:10.3969/j.issn.1001-4616.2023.02.009 YANG Z P, LI D M, LIU Y S, GU X K, YANG J X. Genetic diversity analysis of Culter alburnus in Hongze Lake based on mitochondrial control region D-loop sequence[J]. Journal of Nanjing Normal University(Natural Science Edition), 2023, 46(2): 70-76. DOI:10.3969/j.issn.1001-4616.2023.02.009 |
| [28] |
周其椿, 林艳红, 彭江涛, 王金娜, 王禄超, 安苗. 普安银鲫mtDNA D-loop序列多态性分析[J]. 广东农业科学, 2012, 39(22): 173-175, 183. DOI:10.16768/j.issn.1004-874X.2012.22.074 ZHOU Q C, LIN Y H, PENG J T, WANG J N, WANG L C, AN M. Polymorphism of D-loop sequence from mtDNA of Carassius auratus gibelio Bloch in Puan[J]. Guangdong Agricultural Sciences, 2012, 39(22): 173-175, 183. DOI:10.16768/j.issn.1004-874X.2012.22.074 |
| [29] |
BROUGHTON R E, MILAM J E, ROE B A. The complete sequence of the zebrafish (Danio rerio) mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA[J]. Genome Research, 2001, 11(11): 1958-1967. DOI:10.1101/gr.156801 |
| [30] |
GRANT W A S, BOWEN B W. Shallow population histories in deep evolutionary lineages of marine fishes: Insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity, 1998, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
| [31] |
GAO X M, MU D L, LOU J F, HOU C C, ZHU J Q, WANG J P, JIN S. Genetic diversity and structure analysis of Phascolosoma esculenta in the coastal zone of south-eastern China based on mitochondrial Cyt b gene[J]. Turkish Journal of Fisheries and Aquatic Sciences, 2018, 18(4): 519-528. DOI:10.4194/1303-2712-v18_4_03 |
| [32] |
JAMES B, SHAKLEECLYDE S, TAMARUROBIN S. Waples speciation and evolution of marine fishes studied by the electrophoretic analysis of proteins[J]. Pacific Science, 1982, 36: 141-157. DOI:10.1016/0077-7579(82)90068-0 |
(责任编辑 崔建勋)
 2023, Vol. 50
2023, Vol. 50