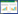文章信息
基金项目
- 广东省自然科学基金(2021A1515010948); 广东省农业科学院农业优势产业学科团队建设项目(202101TD); 广东省重点领域研发计划项目(2022B0202060002, 2020B0202090003); 广东省水稻育种新技术重点实验室项目(2023B1212060042); 广东省农业科学院水稻研究所“优谷计划(所长基金)”项目(2023YG07)
作者简介
- 李堂(1999—),女,在读硕士生,研究方向为水稻籽粒粒型控制机制和根系形态建成,E-mail:3132525673@qq.com.
通讯作者
- 梁世胡(1969—),男,研究员,研究方向为杂交水稻育种,E-mail:lisg180@sina.com.
文章历史
- 收稿日期:2023-09-20
2. 华中农业大学植物科学技术学院,湖北 武汉 430070
2. College of Plant Science and Technology, Huazhong Agricultural University, Wuhan 430070, China
水稻(Oryza sativa L.)是全球重要的谷类粮食作物之一,也是世界上约一半人口的主要食物来源。它为世界人口提供超过21% 的热量需求,而东南亚(SEA)人口高达76% 的热量摄入来自于水稻[1]。伴随着全球气候变暖、人口急速增长、耕地质量下降的一系列问题,粮食安全问题不断加剧,我国粮食安全也面临严峻的挑战[2]。因此,需进一步提高水稻单产来保障产量。水稻在我国粮食供应中占据重要地位,在过去的半个世纪里,使用矮秆育种和利用杂种优势改善了水稻产量,世界大部分地区的水稻产量增加了1倍以上,某些国家和区域甚至增加了2倍[3]。然而,矮杆育种也存在一定缺陷,大部分矮杆品种对氮肥的利用率较低,氮肥的积累使得土壤养分结构失衡、生态遭到破坏[4]。
水稻的外观品质性状大多为数量性状,主要由母体遗传效应控制,同时也受到种子、细胞质等遗传效应的影响[5]。水稻籽粒产量性状较为复杂,由每株穗数、每穗粒数和千粒重决定,水稻粒型由粒长、粒宽及粒厚组成,粒型是水稻单株产量和稻米品质的决定性因素[6]。植物育种很大程度上受到农作物性状的限制,深入了解影响水稻整体品质的因素将为高产优质稻育种奠定基础。基因编辑技术的成熟及其在水稻育种中的广泛应用、基因组作图、测序和功能基因组学的发展均为研究水稻数量性状的遗传和分子基础提供了有力的工具[7]。利用图位克隆技术克隆了许多控制粒型的数量性状基因座(QTLs),目前虽已挖掘出相关基因的调控途径,但大部分基因的作用机制还未得到深入解析[8]。本文综述了调控水稻粒型的多条通路关键基因及其互作关系,并提出结合多组学整合解析水稻粒型调控的分子机理,将基因功能研究与育种实践应用相结合,以期更好地服务于分子设计育种。
1 已克隆的水稻粒型相关基因近年来,基于图位克隆和全基因组关联分析(GWAS)技术,在水稻中分离了许多与籽粒发育有关的基因,目前已发现调控粒型的QTL超500个[9],它们分别位于水稻基因组的12条染色体上(图 1)。其中,控制粒长的有GLW7[10]、GS3[11]、GL10[12]、GL6[13],控制粒宽的有qSW5[14]、GW5[15]、GW2[16]、GS5[17]等;调节粒厚的有TGW3[18]、OsBZR1[19]、OsMADS56[20]、WTG1[21]等基因,通过调节细胞增殖来促进籽粒大小的有SLG7[22]、SMOS1[23];影响细胞分裂的有D1[24]、RGB1[25]、DEP1[26]、OsMAPK6[27]等(表 1)。大量关于粒型QTL的克隆与鉴定在很大程度上促进了水稻籽粒性状的改良。

|
| 图 1 水稻中控制粒型的主要基因/QTL及在染色体上的分布 Fig. 1 Main genes/QTLs controlling grain in rice and distribution on chromosomes |
2 水稻粒型调控分子机理研究进展
水稻种子发育从双受精开始,包括胚胎的持续生长、胚乳细胞的形成,再到种子成熟的整个过程。在对胚胎和胚乳的发育进行调控的过程中,不仅存在着数量庞大的基因,而且还具有大量调节因子形成精细复杂的网络模式协同调控种子发育[41]。调控水稻粒型有多条代谢通路,目前已知调控粒型的途径有6种,分别是E3泛素蛋白酶体降解途径、G蛋白信号通路、MAPK级联信号途径、植物激素途径以及转录因子途径和表观遗传途径等(表 2)。近年来,其他调控籽粒大小的途径相继被发现,这些调控路径的基因在水稻种子发育进程中也担当着重要角色。
2.1 泛素- 蛋白酶体降解途径
泛素- 蛋白酶体降解途径主要包括泛素(Ubiquitin)、泛素活化酶E1(Ubiquitin-Activating enzyme),泛素结合酶E2(Ubiquitin-Conjugating enzyme)和泛素连接酶E3(Ubiquitin-Protein ligase)以及26S蛋白酶体复合物[65]。
目前存在不少关于泛素化途径调控粒型的研究,其中较为典型的基因为GW2。该基因编码一种E3泛素连接酶活性蛋白,它可以通过将其底物传导到蛋白酶体以调节蛋白水解来负调控细胞分裂;GW2缺失导致其不能降解其底物,从而增加颖花外壳细胞的数量,并加快籽粒灌浆速率,促进植株粒宽、粒重增加[42]。此外,Choi等[16]研究表明GW2通过催化细胞增殖蛋白EXPLA1的泛素化,从而调控籽粒发育。TUD1是Hu等[30]首次报道的编码功能性U-box E3泛素连接酶基因,该基因对在赤霉素信号通路中起重要作用的基因D1具有上位性效应,对BR(Brassinosteroid)处理缺乏敏感性;D1直接与TUD1相互作用,从而证实D1与TUD1共同介导BR信号传导途径。Liu等[66]研究表明TUD1还参与GSK2的泛素化。WTG1是一种功能性的去泛素化酶,并且促使细胞变窄从而产生细长的籽粒[21]。Shi等[43]发现,LG 1基因编码泛素特异性蛋白酶15(OsUBP 15),在体外具有去泛素化活性;OsUBP 15表达下调产生比对照更小的籽粒,而过表达则呈现相反的性状,是水稻粒型的正调控因子。OsPUB3也是一个编码E3- 泛素连接酶的基因,其敲除突变体因具有1BP的插入引起移码突变表现出转基因阳性植株的粒重和粒长显著高于阴性植株[67]。该基因的克隆为研究水稻粒型调控的机制提供了新的基因资源。
2.2 MAPK信号途径丝裂原活化蛋白激酶MAPKs(Mitogen-Activated Protein Kinase)是一个具有11个保守结构域的丝/苏氨酸蛋白激酶家族成员,在细胞内外传递信号。MAPK信号通路是由3个层次的激酶级联反应完成:MAPKKK受到疑似类受体激酶传导的跨膜信号刺激后磷酸化激活MAPKK;活化的MAPKK再磷酸化激活MAPK;受到激活的MAPK将信号转导至细胞核内[68]。
水稻中参与MAPK级联信号途径的基因有SMG1和SMG2,SMG1编码丝裂原活化蛋白激酶OsMAKK4,smg1突变体的表型缺陷促进细胞增殖而呈现直立穗和株高较矮的形态,籽粒变小,表明SMG1参与MAPK级联信号调控粒型[44];丝裂原活化蛋白激酶OsMAPK6由DSG1编码,OsMAPK6通过调节细胞增殖、BR信号和内稳性影响水稻籽粒大小[27]。OsMAPK6与OsMKK4存在较强的相互作用,表明OsMKK4很可能是水稻OsMAPK6的上游MAPK激酶。GSN1编码双特异性磷酸酶OsMKP1,它可直接与增殖抑制蛋白OsMPK6互作使其去磷酸化,此外,GSN1可负调节OsMKKK10-OsMKK4-OsMAPK6级联途径进而负调控水稻穗型[46]。近期研究发现,OsMKKK70通过与OsMKK4-OsMAPK6的MAPK信号级联途径共同调节籽粒大小,过量表达OsMKKK70可促进颖壳细胞增殖导致籽粒长度增加[69]。OsMKKK70还可能通过OsMKK4-OsMAPK6-OsWRKY53级联途径调节BR信号传导和籽粒大小。
2.3 G蛋白信号途径G蛋白偶联受体(G protein- coupled receptors,GPCRs)是一类蛋白受体的总称,包含7个跨膜α- 螺旋结构域以及一个胞外N端和一个胞内C端尾巴,G蛋白分别由Gα(RGA1),Gβ(RGB1)和Gγ(RGG1、RGG2、GS3、DEP1/qPE-9)亚基组成[70]。在水稻中,D1编码Gα亚基(RGA1),该基因的无义突变导致d1突变体粒型变化,表现为植株矮化、叶片直立、籽粒变小和对BR敏感性下降,表明D1与G- 蛋白偶联受体相互作用来调节油菜素内酯的合成[24]。Ma等[71]通过重测序和图位克隆发现1个新等位基因RGA1-FH,由于其A-T位点突变破坏了RGA1的正常功能,导致粒长减小。Gβ亚基(RGB1)正调控ABA生物合成,Gγ亚基qPE9-1/DEP1受RGB1的调控,在ABA依赖的干旱胁迫反应中起负调控作用[25]。Gγ亚基(DEP1)已确定在水稻中具有多重作用,如正向调节籽粒大小、穗发育和对氮的响应。另外,DEP1被证明与GS3具有相反的功能,GS3是控制粒长和粒重的主效QTL,对粒重起负调控作用[11];RGG2编码B型γ亚基,过表达rgg2突变体产生株高较矮和小粒表型,表明RGG2在水稻中负调节植株生长和籽粒发育,相关研究发现RGG2参与了赤霉素信号转导,因此可以推断RGG2可能通过GA途径调控水稻粒型[47]。另有报道指出,SMOS1的等位基因GR5编码AP2转录因子,且GR5作为转录激活因子可与5个Gγ亚基(RGG1、RGG2、DEP1、GS3和GGC2)互作,通过影响细胞增殖和扩增负调控粒长。
2.4 转录因子途径转录因子是存在于植物体内、在植物生长发育的进程中发挥着关键作用的成分。研究发现,转录因子调节籽粒大小的途径包括以下几种类型:SQUAMOSA启动子结合蛋白样家族、OsmiR396-OsGRF4-OsGIFS调控网络、NGATHA-LIKE-Kluh途径、Peapod-KIX调节模型、APETALA2型(AP2)转录因子、WRKY家族转录因子、bHLH家族转录因子、染色质修饰等[72]。
OsSPL16是一个调节粒宽的SBP结构域转录因子,由GW8编码,OsSPL16直接与GW7启动子结合并抑制其表达从而负调控粒型[48];GS9编码一种未知保守功能结构域的蛋白,其可通过调节细胞分裂正调控水稻粒型,gs9缺失突变体具有细长的粒型,过表达GS9产生圆粒表型[49];Si等[10]发现一个主效数量性状基因GLW7,该基因编码植物特异性转录因子OsSPL13,GLW7正向调节颖壳中的细胞大小,从而增加水稻粒长。Hu等[50]从水稻第2条染色体上分离到控制粒型的QTL(GS2),编码1个GRF家族转录调节因子OsGRF4,GS2定位于细胞核,可能作为转录激活因子发挥作用。一种罕见的GS2突变影响microRNA的结合位点OsmiR396c,引起GS2/OsGRF4高表达。GS2基因表达增强可使细胞变大,进而增加粒重。将GS2/OsGRF4稀有等位基因导入水稻品种,可显著增加水稻粒重和产量,在水稻高产品种选育中具有潜在应用价值。SG5编码1个MYB样转录因子,其第1个外显子中的错义突变导致SG5蛋白中197位氨基酸从亮氨酸变为脯氨酸;SG5主要在幼穗和颖壳中表达,通过介导细胞扩张来调控颖壳的发育、产生短粒。转录因子HOS59为KNOX Ⅱ类亚家族的成员,hos59突变体具有更长的籽粒和颖壳细胞长度、松散的株型和下垂的叶片,而过表达株系籽粒变小、叶直立、株高降低[73]。近年来,对MiRNA调控粒型通路的研究也有一定进展,miR396家族由拟南芥中的ath-miR396a和ath-miR396b组成,在植物中相对保守。Baucher等[74]探析了miR396a-GRF网络在烟草花器官中的调控模式,研究表明,含有miR396位点的NtGRF基因表达量下调可促使叶融合和茎尖分生组织的缺失。而水稻中的miR1848可降低OsCYP51G3的转录水平,从而影响BR的生物合成,使水稻种子变小[75]。Li等[76]克隆出的主效QTL(GLW2)编码生长调节因子4(OsGRF4),同时可被miR396正调控,OsGRF4的突变干扰了OsmiR396c对OsGRF4的靶调控,从而产生大粒表型。另有研究发现,OSGIF1直接与OSGRF4互作,miR396c-OsGRF4-OsGIF1网络模式在水稻粒型中起决定性作用。Qiao等[52]在9311和日本晴的重组自交系中鉴定到1个控制粒长和粒重的数量性状基因qGl5,其精细定位于OsAUX3上,在含有osaux3的突变体中,启动子序列变异导致OsAUX3功能缺失,产生较长和较宽的粒型。OsARF6是影响OsAUX3表达的转录调控因子,其直接结合OsAUX3启动子的生长素(Auxins, IAA)响应元件,通过改变颖壳细胞扩张和生长素分布来控制粒长的形成。此外,研究发现MiR167a沉默OsARF6的MRNA调节粒长和粒宽,研究揭示了1个新调节粒型的miR167a-OsARF6-OsAUX3模式,为水稻品质改良提供了新的方向。MiR167也参与植物激素信号传导,MiR167-OsARF12模式在miR159下游促进OsCDKF; 2的表达,调控细胞分裂并介导IAA和BR信号传导,从而正向调节水稻籽粒灌浆和大小。
2.5 植物激素途径植物激素是一种微量有机分子,在植物的生长、发育、衰老、休眠和抗逆性方面发挥重要作用,根据植物激素的种类分为生长素(Auxins,IAA)、油菜素内酯(Brassinosteroid,BR)、赤霉素(Gibberellin,GA)、细胞分裂素(Cytokinin,CTK)以及脱落酸(Abscisic acid,ABA)、乙烯(Ethylene,ETH)茉莉酸、甲醋(Methyl jasmonate,MeJA)等。植物激素是调节籽粒大小的关键途径之一。水稻中常见的调控粒型的植物激素CTK、IAA和BR等。
2.5.1 CTK途径 CTK是一种抑制叶片衰老、调控水稻粒型的重要因子,但有关CTK分子机理的研究仅限于拟南芥,对其在水稻中的生理机制仍知之甚少[77]。曹煜东等[78]通过图位克隆鉴定了控制水稻粒型的基因GS2.2,该基因编码生长调节因子OsGRF4,OsGRF4的OsmiRNA396位点发生了特异性突变,导致表型差异。进一步研究发现,OsGRF 4基因可以对2个CTK脱氢酶前体基因CKX 5和CKX 1进行调节,导致细胞分裂素水平升高,进而影响穗部性状。Zhang等[79]描述了1个CTK氧化脱氢酶OsCKX11在延缓水稻叶片衰老以及增加粒数中的作用,OsCKX11蛋白可催化多种CTK的降解,在叶片衰老中对CTK和ABA起拮抗作用。相关研究表明,突变体osckx11表现出分蘖数、籽粒数明显增加的性状。Gnla编码CTK氧化脱氢酶OsCKX2,该酶降解植物激素CTK,OsCKX2表达量的减少可使花序分生组织中的CTK累积,并增加颖花数量,从而提高籽粒产量[29]。
2.5.2 IAA途径 IAA调节水稻颖壳和胚乳的发育,是调控种子发育和影响水稻产量的重要植物激素[80]。TGW6编码具有IAA- 葡萄糖水解活性的蛋白,可负调节胚乳细胞的数量和籽粒大小[53]。而与TGW6不同的是,Liu等[54]鉴定了编码植物膜定位蛋白的基因BG1,过表达BG1则导致籽粒显著增大,此突变体对IAA十分敏感,敲除BG1的植株则对IAA的敏感性降低,这说明BG1受IAA的特异性诱导,表明其在水稻IAA运输过程中发挥着关键作用。Ma等[18]发现了控制水稻粒长和产量的新基因TGW3,并筛选到其互作因子OsIAA1O,TGW3能够直接对OsIAA10蛋白的丝氨酸位点S68、S75及S97进行磷酸化修饰,调控水稻籽粒大小。遗传数据表明,存在1个TGW3-OsIAA10介导的IAA信号通路,TGW3还参与水稻植株对BR的反应,而且该效应能通过上述信号通路传递。
2.5.3 BR途径 BR是一类广泛存在于植物体内的、具有广泛生理活性的植物激素,研究BR相关基因在作物中的作用机制是其应用于农业生产的必要条件[81]。在水稻中,BR主要调控籽粒形状、株高及叶夹角等性状。目前已发现参与BR信号转导途径的基因主要有GW5、D2、SLG、OsBSK2和新克隆的ZmD11、OsBSK1-1、WG3。Liu等[15]发现编码钙调结合蛋白的基因GW5,它可与BR信号途径中的1个关键糖原合酶激酶GSK2直接互作,GW5功能的缺失导致其无法将泛素转移到靶蛋白,使得本应降解的底物不能被特异识别,进而激活颖壳细胞分裂,提高了粒重以及产量;D11是BR生物合成过程中起关键作用的基因,而ZmD11为DWARF(D11)的直系同源基因,相关实验证实ZmD11互补了cpb1突变体所表现出的异常穗型和株高的表型。该基因组成型表达可增加水稻的粒长和粒重[82];BR信号激酶OsBSK1-1可能代表水稻BR信号通路中缺失的组分。研究人员发现osbsk1-1敲除突变体对BR缺乏敏感性,并表现出多效性的表型,包括植株矮化、分蘖数减少和粒长缩短。而过表达OsBSK1-1则产生对BR高度敏感的现象,表明OsBSK1-1在调控BR信号通路中发挥一定作用[83]。Yuan等[57]鉴定了1个突变体glw10,编码BR信号激酶OsBSK2,该突变体表现出粒长和粒重显著降低,OsBSK2为质膜蛋白,可直接与BR受体激酶OsBRI1相互作用;然而,遗传学实验已证明OsBSK2可独立于BR信号通路调节籽粒大小。Du等[84]证实OsBZR1可直接结合OsBAK2的启动子,通过BR反馈抑制环抑制OsBAK2的表达,且OsBZR1受OsGSK3磷酸化,osbak2突变体产生BR缺陷的表型,粒长增加。近年来发现WG3编码植物特异性转录因子GRAS蛋白,通过影响细胞增殖调节粒宽和千粒重;WG3可与DLT互作,以相同途径调控粒型,BR敏感测试表明WG3在BR信号途径中发挥积极作用[85]。相关研究证实GRAS的成员DLT2是BR信号转导的正调因子,dlt2突变体表现出对BR较低的敏感性,且产生较宽的籽粒表型[86];SLG编码1个类BAHD酰基转移酶,可抑制细胞伸长;SLG是维持BR稳态的重要调节因子,其突变导致水稻产生长粒、叶倾角增大的表型[58]。GS5与BAK1互作,编码丝氨酸羧肽酶,通过控制颖壳细胞分裂从而调控种子生长,过表达GS5可竞争性抑制OsBAK1-7与OsMSBP1的内吞作用,致使BR信号增强、籽粒增大[17]。Jin等[87]对OsNAC129进行敲除和过表达处理,结果发现缺失突变体在未成熟种子中的表达量显著降低,株高、表观直链淀粉含量、籽粒长度和重量增加,而过表达OsNAC129的株系则呈现相反的表型且表现出对BR敏感性降低。研究表明,OsNAC129参与BR信号转导途径并负调控种子的生长和发育。参与BR信号转导的新调控因子miR168a-5p可通过靶向OsOFP3、OsNPF2.4和OsAGO1a调控BR信号通路,其转基因植株产生短粒、低千粒重的表型[88]。近两年对BR信号转导途径相关基因的研究进展迅速,这极大地丰富了控制水稻粒型的基因调控网络,利于后期分子育种的研究。
2.5.4 GA、ETH和MeJA途径GA和ETH大量 存在于模式作物中,在水稻粒型的形成中却不常见。近年来,也有不少科研人员对影响GA和ETH、MeJA生物合成的基因进行鉴定。Liu等[89]指出ETH反应因子OsERF115作为一个关键下游因子调节ETH介导的籽粒发育。OsERF115编码1个AP2/ERF型转录因子,在幼穗和发育中的颖壳中特异表达。过量表达OsERF115可显著增加粒型,而敲除则相反,表明OsERF115正向调控籽粒大小和重量。Zhan等[12]描述了正调控粒型的QTLGL10,其编码MADS56,通过纵向调节细胞伸长影响粒型,gl10的等位基因发生变异,在第一个外显子中含有1.0 kb单核苷酸的缺失,终止转录进程,从而导致植株粒长缩短、粒重降低,过表达GL10则呈现相反的表型,此外,GL10还在水稻幼穗组织中参与GA信号转导。编码己糖激酶样(HKL)蛋白的基因OsHXK3通过影响GA生物合成和体内平衡,引起表型突变,OsHXK3表达量增加,进而单株产量提升[64]。GW6编码GA调节的GAST蛋白,正向调节粒宽和粒重[59]。Glw7.1作为Ghd7的等位基因,通过上调GA合成基因的表达来提升内源GA的含量、促进细胞增殖和扩张,从而正调控水稻籽粒大小和粒重[37]。Mehra[90]发现了水稻中的MeJA信号途径的抑制因子OsJAZ11,该基因的组成型表达影响小穗形态建成,与野生型相比,过表达OsJAZ11的株系粒宽和粒重均增加,表明较高的JA在过表达植株的小穗和种子中积累,导致花器官数目异常。MeJA信号作为1个不常见信号途径的出现,为解析水稻粒型遗传机制作了较好补充。
2.6 表观修饰途径表观修饰主要指DNA序列不变而表观可稳定性遗传变化,表观修饰因子通过改变染色质状态调控基因转录,影响生长发育和对环境的适应性。表观遗传修饰主要包括组蛋白H3和H4的甲基化修饰、乙酰化修饰、非编码RNA调控等[91]。研究水稻表观遗传调控的机制对提升水稻籽粒产量具有重要意义,目前在模式作物中发现了许多与农艺性状相关的表观修饰研究,而在水稻中也发掘了一些通过表观遗传修饰途径来调控粒型的相关研究。
组蛋白修饰在种子发育过程中发挥作用[92]。GW6a编码1个拥有组蛋白乙酰转移酶的类GNAT蛋白OsglHAT1,Osg1HAT1通过增加细胞数量和加速籽粒灌浆速率来扩大颖壳,从而调节粒宽和粒重[93]。表观等位基因RAV6编码B3 DNA结合蛋白,RAV6启动子区域发生低甲基化,导致BR受体和BR生物合成基因异位表达,突变体植株产生较小籽粒[94]。水稻Harbinger转座子衍生基因PANDA被报道表观遗传调控穗粒数和籽粒大小。PANDA编码蛋白可以与复合物Polycomb2(PRC2)的组分OsMSI1和OsFIE2结合,并调节H3K27me3在靶基因中的积累,从而抑制PRC2的表达。POWA是PANDA的等位基因,通过抑制TAF2的转录活性来调节籽粒大小[95]。组蛋白H3K9、H3K27和H4K20的甲基化通常抑制基因转录,而H3K4和H3K36的甲基化与基因活化相关。水稻SDG725编码H3K36甲基转移酶,下调其表达导致植株矮化、节间缩短、直立叶片和小粒的表型。相反的是,JMJ703是水稻中的一种H3K4特异性去甲基酶,能特异地降低组蛋白H3K4的甲基化水平,参与控制转座子的活性,过表达JM703产生长粒表型。综上,一些基因可参与表观遗传途径介导水稻粒型,这为解析水稻粒型的调控网络创造了新的机制。
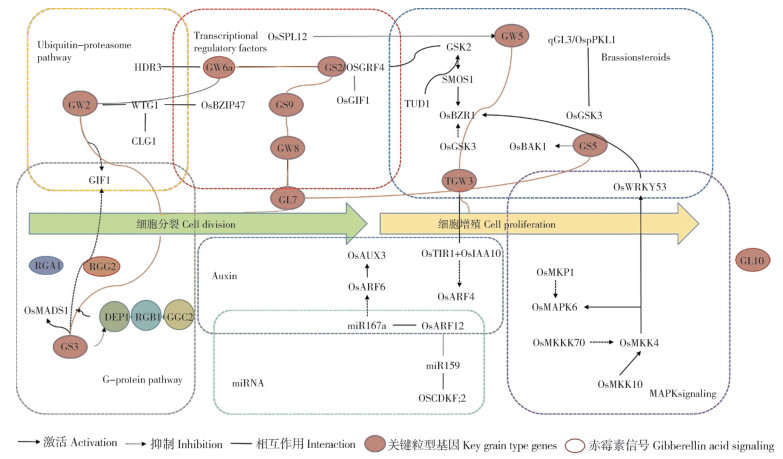
2.7 不同信号通路间的交叉互作调控粒型水稻粒型是复的杂遗传性状,其各个调控信号通路间也存在一定的交叉互作。已有研究指出,泛素化途径可能与一些转录因子共同发挥作用或参与BR信号途径调控粒型。Hao等[55]报道了GW2-WG1-OsbZIP47调控粒型的模式,其中WG1编码谷氧还蛋白,通过调节细胞增殖来促进籽粒发育;OsbZIP47减少细胞分裂限制种子生长;且WG1可与转录因子OsbZIP47互作并抑制其转录活性。进一步分析表明,E3泛素连接酶GW2泛素化WG1并介导其降解,这证实了GW2、WG1、OsbZIP47在控制籽粒生长的共同途径中起作用。与GW2相反,U-box E3泛素连接酶TUD1可能通过与G蛋白α亚基互作参与BR信号转导、增加颖壳细胞数量来正调节籽粒大小。TUD1也介导BR负调节因子GSK2的泛素化,导致其通过26S蛋白酶体途径降解,正向调节细胞扩增增加粒长[66]。Yang等[96]研究发现,编码E3泛素连接酶的基因CLG1可泛素化Gγ蛋白GS3,导致其通过蛋白内体途径降解,进而增大籽粒。SMG3和DGS1是与ERAD相关的E2E3泛素连接酶复合体,DGS 1作为有活性的E3泛素连接酶,可与SMG 3在ER中共定位,SMG 3和DGS 1被证实参与BR信号传导。DGS 1泛素化BR受体BRI 1并影响其积累。
转录因子SPL、MiR396-OsGRFs、WYKY会明显受到BR、AUX和GA等激素的影响[97]。GW5/GSE5编码具有IQ钙调蛋白结合基序结构域的膜定位蛋白,GW5/GSE5抑制GSK2的激酶活性和自磷酸化,导致不能磷酸化OsBZR1和DLT,大量非磷酸化蛋白在细胞核中积累,这些蛋白正向调节BR信号通路下游基因的表达[15]。GW5基因启动子区上游5 kb处存在一段1 212 bp的碱基缺失,而OsSPL12则发挥增强子的作用,促进GW5的表达从而抑制粒宽。这表明OsSPL12与GW5协同决定水稻粒型[98]。GS2编码OsGRF4,通过调控细胞扩张影响籽粒大小,OsGRF4中的OsmiR396靶向位点突变,阻止OsmiR396通过剪接抑制OsGRF4的表达,导致OsGRF4表达量上升。GSK2是BR的负调节因子,抑制OsGRF4的转录活性,OsGRF4的miR396靶向突变导致OsGRF4的表达增加,同时上调BR相关基因的表达,表明OsGRF4可能与BR信号通路相互作用[99]。Wang等[36]在栽培稻品种9311中鉴定到1个调控粒长和粒重的关键基因GL12,该基因被证实与GS2启动子区互作,敲除突变体植株表现出粒长、粒重减少。转录因子OsWRKY53通过BR信号传导和丝裂原活化蛋白激酶MAPKKK10-MAPKK4-MAPK6级联途径正向调节籽粒大小,遗传分析表明,GSK2磷酸化OsWRKY53, 并降低其稳定性,表明GSK2是OsWRKY53的底物,且GSK2磷酸化MAPKK4, 从而抑制MAPK6的活性[100]。
OsARF6是调控OsAUX3表达的上游转录因子,OsARF6与OsAUX3启动子的IAA响应元件结合,通过改变颖壳细胞扩张和IAA的含量从而调节粒长,此外,研究表明miR167a介导OsARF6的MRNA沉默正向调节粒宽和粒长[101]。MiR167也参与植物激素信号传导,据报道,MiR167-OsARF12模块在miR159下游通过激活OSCDKF; 2的表达,调控细胞分裂和介导IAA及BR信号转导,从而正向调节水稻籽粒灌浆和籽粒大小[101]。MADS-box家族转录因子OSMADS56由GL10编码,通过参与幼穗组织的GA信号途径促进细胞伸长,从而正调控水稻粒长,过表达gl10突变体植株产生籽粒变长、抽穗期提前的表型[12]。
此外,G蛋白信号通路的基因也可作为转录因子发挥作用。由GS3和RGB1组成的AGbc亚基二聚体激活下游关键效应因子OsMADS1的转录活性以调节籽粒长度。OsALDH2BI编码乙醛脱氢酶,其具有转录抑制和激活活性,通过直接结合GS3启动子以抑制其表达从而参与调节水稻粒型;此外,G蛋白与GA和CTK共同介导调控水稻粒型。据报道,D1/RGA1和RGG2分别编码影响GA和BR信号转导的Gα和Gγ亚基,产生小圆粒种子,从而负调控籽粒发育,G蛋白Gc/RGG2与RGB1形成G蛋白二聚体,通过介导内源GA生物合成和信号转导,负调节籽粒大小[47, 102]。G蛋白另一个亚基RGG1可能参与CTK信号途径控制籽粒大小,RGG1编码A类Gγ亚基,过表达RGG1则导致株高降低、穗长和粒长变短,研究人员对植株内源CTK水平进行测量,发现CTK在RGG1过表达系中呈现较低水平,这说明RGG1可能介导了CTK合成途径调控粒型[103]。
MAPK信号通过参与BR信号转导调节籽粒大小。SMG 1/OsMKK 4的突变导致BR信号传导反应受阻,对外源BR的敏感性降低,BR相关基因的表达降低。这些观察结果表明MAPK通路和参与调节籽粒大小的BR信号途径之间存在联系。OsMAPK 6影响BR信号转导和稳态,这种作用也受OsMKK 4磷酸化修饰的调节。OsMKK4-OsMAPK 6-OsMKKK70介导的OsWRKY 53的磷酸化对其在BR信号转导中的功能是必需的,过表达OsMAPK4、OsMAPK6和OsWRKY53可以回补osmkk62/osmkkk70双突变体所表现出的较小籽粒的表型。由此可知,BR信号通路在几条通路中均参与了对水稻粒型的调控,说明BR信号转导是大部分粒型调控通路中所必需的,且各通路间存在一定相互作用,表明控制水稻粒型的网络错综复杂,有待我们进一步解析其间的分子调控机制。
2.8 水稻关键粒型基因互作调控机制水稻粒型受多基因调控,不同粒型基因间存在不同程度的互作关系。研究水稻粒型关键基因间的互作关系对育种实践具有重要的意义。Yan等[104]为探究GS3、GW2、qSW5/GW5、GIF1基因间的关系,利用以上关键粒型基因构建了RNA干扰株系并分析了qSW5在转录水平上的表达量,结果表明,qSW5和GW2能够正调GS3的表达,且qSW5抑制GW2转录促使其表达量下降,GIF1受qSW5正调、且受GW2和GS3负调(图 2)。另一对关键粒型基因GW8和GW7也存在一定互作,GW8能够直接与GW7的启动子区结合并抑制其表达,从而负调控粒型[48]。Wu等[31]为组合gs3和dep1的优点,利用含有dep1等位基因超级稻品种SN265的基因座进行分子设计育种,结果显示,sn265/gs3基因编辑植物所表现出增加的粒长进一步提高了产量。GL3.3为TGW3的等位基因,研究表明,TGW3与GS3互作导致水稻籽粒增大[31]。郑镇武等[105]鉴定了一个新的等位基因qGL3.4,研究表明其可能调控GS3、GL3.1、GL7和GS5等关键粒型基因的表达,从而控制水稻籽粒大小。
泛素受体HDRs以泛素依赖的方式与组蛋白乙酰转移酶GW6a互作,HDR和GW6a结合到下游基因的启动子,通过相同的遗传途径共同调控水稻粒型[106]。GW2、WG1、OsbZIP47分别是水稻粒型的正调、负调因子,三者存在相互作用。OsbZIP47减少细胞分裂、产生小粒,WG1与转录因子OsbZIP47互作并抑制其转录活性,而GW2为大粒基因,GW2泛素化WG1并介导其降解,从而互补了osbzip47突变体的表型。
3 粒型基因在水稻育种上的应用目前水稻中的大部分粒型基因已得到鉴定,却很少应用到育种当中,优质稻选育中以GS3、GW5和GL7的运用较为广泛。
GS3是一个控制粒长的主效QTL,近10年来众多有关GS3基因功能的研究相继被发现,也评估了GS3基因在水稻育种上的应用价值。Fan等[107]利用180个代表性水稻品种,根据GS3基因的C-A单核苷酸多态性开发了一个CAPS标记SF28,结果证实该标记易于区分GS3的等位基因,可直接作为水稻粒长改良的功能标记;Xia等[32]利用水稻品种珍汕97(ZS97,indica)和南洋占(NYZ,japonica)构建重组自交系,得到控制粒型的QTLGL3.3,进一步分析定位到OsGSK5,由于GL3.3与之前的GS3存在互作效应导致籽粒增大,GL3与GL3.3基因组合在育种中得到推广,被广泛地应用于粳稻大粒品种选育。毛艇等[108]将gs3基因与Pita、Pib聚合,以优质粳稻嘉禾212为供体亲本进行杂交、标记辅助选择,得到籽粒长宽比大于亲本的改良系。
Sun等[109]收集3 872个重离子辐射过的CNOM2代水稻种子和单个子叶,基于靶向测序技术发现6个粒型突变体和9个粒型相关等位变异体,为分子育种和基因型突变及表型分析提供了新的功能基因位点和物质基础。Yang等[96]通过比较粒型变异的广东丝苗米群体分化指数得出GS3和GW5为控制丝苗米粒长的关键基因,而GL7则调控籽粒大小,这些丝苗米品种的遗传机制将有助于分子育种。康雪蒙[110]以南方纤细型籼稻玉针香为供体亲本、空育131为受体亲本进行分子标记筛选,最终实现来自玉针香的GS3、GL7、GW7基因对空育131粒型的改良。黄娟等[39]利用Cas9技术编辑GS3、GW8和TGW3, 导入到恢复系桂恢582(GH582),对新恢复系进行农艺性状的相关分析,结果显示GH582E与GH582相比粒长增加18.4%。温一博等[111]以籼稻品种R99和粳稻品种SN265构建重组自交系,发现Gn1aR99、DEP1SN265、qSW5SN265的组合产量表现较好,千粒重有所增加。Jiang等[112]通过将FN 60-4与亲本系Kitaake杂交并分析所得回交群体,利用GWAS技术鉴定到1个突变体gs9-1,该基因显著降低株高和千粒重,新型基因GS9是优良育种材料。韩政宏等[113]利用CRISPR/Cas9技术对东富139、龙梗31、东农427等3个品种进行GS3和GS9位点的编辑,结果表明3个品种的GS3突变体粒长、千粒重均有所增加,且GS9突变体粒长显著增加,该研究加快了长粒型粳稻品种的选育和改良进程。
4 结语与展望过去的几十年里,大量控制水稻籽粒大小的功能位点被克隆和鉴定。杂种优势的培育和分子标记技术的应用为改善水稻产量和品质创造了新模式,通过对水稻粒型多条调控通路的解析,如泛素化途径、G蛋白途径、转录因子途径、植物激素途径以及一些其他控制粒型的途径和粒型基因间的互作,丰富了水稻粒型遗传调控网络。对不同粒型调控通路的分析研究发现,许多调节控制粒型的基因网络精细复杂,各粒型基因间甚至几大通路间都均存在相互作用。不同调节因子之间的遗传关系与作用机制在极大程度上未知,一些基因以调节细胞增殖或分裂的方式参与水稻粒型的建成,但其中作用机制尚未明晰。识别和推断基因网络的结构关系,理解基因表达和功能特征,对今后水稻籽粒调控、培育优质高产的稻米具有深远影响。
目前对水稻粒型的研究在育种中的应用相对较少。在以往研究中,研究人员都是利用不同遗传背景下的等位基因变异来达到选择优良株型的目的,但也存在与后期选择不一致的情况。此外,在利用粒型基因进行性状改良时,有的基因可能会起到负调控的作用从而降低产量,这需要我们识别和鉴定出对粒型改良有利的基因,同时要考虑到粒型基因影响多个数量性状,在利用优异等位基因进行聚合选择育种过程中,需抓住关键性状,根据育种需要来选择相应的优良基因。注重经典粒型基因的应用,以传统优良基因与现代育种技术相结合,开发关键粒型基因的分子标记,从而改善水稻产量和品质。
近年来,基因编辑技术和全基因组关联分析的应用为研究粒型基因提供了方便。基因编辑技术可以敲除或插入特定DNA片段的碱基从而使突变体植株表型发生变化。Cas9技术广泛应用于基因功能鉴定、水稻产量及稻米品质、抗性的研究当中。功能基因组学的发展使得全基因组关联分析技术(GWAS)应用到相关QTL的克隆和定位的研究中,利用GWAS能够快速定位到目标位点。此外,多功能组学包括基因组学、蛋白组学、转录组学、代谢组学等整合分析技术将成为生命科学研究的必然趋势,如将转录组基因信息与代谢组当中的表型关联,结合通路富集、分子互作筛选重点基因,实现精准育种。随着粒型调控网络的深入解析,将来会有更多粒型基因应用到育种实践中,运用这一系列现代育种技术解析基因间的作用关系将有助于开发分子设计育种,为实现高产优质稻育种奠定理论基础。
| [1] |
FITZGERALD M A, MCCOUCH S R, HALL R D. Not just a grain of rice: The quest for quality[J]. Trends in Plant Science, 2009, 14(3): 133-139. DOI:10.1016/j.tplants.2008.12.004 |
| [2] |
罗雁, 毛昭庆, 陈良正. 全球粮食危机背景下中国粮食安全问题研究[J]. 江西农业学报, 2022, 34(10): 220-225. DOI:10.19386/j.cnki.jxnyxb.2022.10.035 LUO Y, MAO Z Q, CHEN L Z. Research on China's food security under background[J]. Acta Agriculturae Jiangxi, 2022, 34(10): 220-225. DOI:10.19386/j.cnki.jxnyxb.2022.10.035 |
| [3] |
何秀英, 周少川, 刘志霞, 刘传光. 广东省农业科学院常规水稻育种60年: 成就与展望[J]. 广东农业科学, 2020, 47(11): 1-8. DOI:10.16768/j.issn.1004-874X.2020.11.001 HE X Y, ZHOU S C, LIU Z X, LIU C G. Sixty years' conventional rice breeding of Guangdong Academy of Agricultural Sciences: Achievements and prospects[J]. Guangdong Agricultural Sciences, 2020, 47(11): 1-8. DOI:10.16768/j.issn.1004-874X.2020.11.001 |
| [4] |
吴比, 胡伟, 邢永忠. 中国水稻遗传育种历程与展望[J]. 遗传, 2018, 40(10): 841-857. DOI:10.16288/j.yczz.18-213 WU B, HU W, XING Y Z. The history and prospect of rice genetic breeding in China[J]. Hereditas, 2018, 40(10): 841-857. DOI:10.16288/j.yczz.18-213 |
| [5] |
张静, 李晨, 潘大建, 陈文丰, 孙炳蕊, 刘清, 吕树伟, 江立群, 毛兴学, 范芝兰. 水稻粒长遗传及其功能基因研究进展[J]. 广东农业科学, 2021, 48(3): 1-10. DOI:10.16768/j.issn.1004-874X.2021.03.001 ZHANG J, LI C, PAN D J, CHEN W F, SUN B R, LIU Q, LYU S W, JIANG L Q, MAO X X, FAN Z L. Advances in inheritance of grain length and its functional genes in rice[J]. Guangdong Agricultural Sciences, 2021, 48(3): 1-10. DOI:10.16768/j.issn.1004-874X.2021.03.001 |
| [6] |
黄勇, 胡勇, 傅向东, 邢永忠. 水稻产量性状的功能基因及其应用[J]. 生命科学, 2016, 28(10): 1147-1155. DOI:10.13376/j.cbls/2016153 HUANG Y, HU Y, FU X D, XING Y Z. Functional genes for grain yield related traits and their application in rice breeding[J]. Chinese Bulletin of Life Sciences, 2016, 28(10): 1147-1155. DOI:10.13376/j.cbls/2016153 |
| [7] |
ZHU Z, VERMA N, GONZALEZ F, SHI Z D, HUANGFU D. A CRISPR/Cas-mediated selection-free knockin strategy in human embryonic stem cells[J]. Stem Cell Reports, 2015, 4(6): 1103-1111. DOI:10.1016/j.stemcr.2015.04.016 |
| [8] |
冷春旭, 闫平, 吴立成, 王玉杰. CRISPR/Cas9系统在稻米品质改良中的应用[J]. 分子植物育种, 2021, 19(14): 4681-4689. DOI:10.13271/j.mpb.019.004681 LENG C X, YAN P, WU L C, WANG Y J. Applications of the CRISPR/Cas9 system for rice grain quality improvement[J]. Molecular Plant Breeding, 2021, 19(14): 4681-4689. DOI:10.13271/j.mpb.019.004681 |
| [9] |
WANG H, ZHU S, DANG X, LIU E, HU X, ELTAHAWY M S, ZAID I U, HONG D. Favorable alleles mining for gelatinization temperature, gel consistency and amylose content in Oryza sativa by association mapping[J]. BMC genetics, 2019, 20(1): 1-18. DOI:10.1186/s12863-019-0735-y |
| [10] |
SI L, CHEN J, HUANG X, GONG H, LUO J, HOU Q, ZHOU T, LU T, ZHU J, SHANGGUAN Y, CHEN E, GONG C, ZHAO Q, JING Y, ZHAO Y, LI Y, CUI L, FAN D, LU Y, WENG Q, WANG Y, ZHAN Q, LIU K, WEI X, AN K, AN G, HAN B. OsSPL13 controls grain size in cultivated rice[J]. Nature Genetics, 2016, 48(4): 447-456. DOI:10.1038/ng.3518 |
| [11] |
TAKANO-KAI N, JIANG H, KUBO T, SWEENEY M, MATSUMOTO T, KANAMORI H, PADHUKASAHASRAM B, BUSTAMANTE C, YOSHIMURA A, DOI K, MCCOUCH S. Evolutionary history of GS3, a gene conferring grain length in rice[J]. Genetics, 2009, 182(4): 1323-1334. DOI:10.1534/genetics.109.103002 |
| [12] |
ZHAN P, MA S, XIAO Z, LI F, WEI X, LIN S, WANG X, JI Z, FU Y, PAN J, ZHOU M, LIU Y, CHANG Z, LI L, BU S, LIU Z, ZHU H, LIU G, ZHANG G, WANG S. Natural variations in grain length 10 (GL10) regulate rice grain size[J]. Journal of Genetics and Genomics, 2022, 49(5): 405-413. DOI:10.1016/j.jgg.2022.01.008 |
| [13] |
WANG A, HOU Q, SI L, HUANG X, LUO J, LU D, ZHU J, SHANGGUAN Y, MIAO J, XIE Y, WANG Y, ZHAO Q, FENG Q, ZHOU C, LI Y, FAN D, LU Y, TIAN Q, WANG Z, HAN B. The PLATZ transcription factor GL6 affects grain length and number in rice[J]. Plant Physiology, 2019, 180(4): 2077-2090. DOI:10.1104/pp.18.01574 |
| [14] |
TIAN P, LIU J, MOU C, SHI C, ZHANG H, ZHAO Z, LIN Q, WANG J, WANG J, ZHANG X, GUO X, CHENG Z, ZHU S, REN Y, LEI C, WANG H, WAN J. GW5-like, a homolog of GW5, negatively regulates grain width, weight and salt resistance in rice[J]. Journal of Integrative Plant Biology, 2019, 61(11): 1171-1185. DOI:10.1111/jipb.12745 |
| [15] |
LIU J, CHEN J, ZHENG X, WU F, LIN Q, HENG Y, TIAN P, CHENG Z, YU X, ZHOU K, ZHANG X, GUO X, WANG J, WANG H, WAN J. GW5 acts in the brassinosteroid signalling pathway to regulate grain width and weight in rice[J]. Nature Plants, 2017, 3(5): 1-7. DOI:10.1038/nplants.2017.43 |
| [16] |
CHOI B S, KIM Y J, MARKKANDAN K, KOO Y J, SONG J T, SEO H S. GW2 functions as an E3 ubiquitin ligase for rice expansin-like 1[J]. International Journal of Molecular Sciences, 2018, 19(7): 1904. DOI:10.3390/ijms19071904 |
| [17] |
XU C, LIU Y, LI Y, XU X, XU C, LI X, XIAO J, ZHANG Q. Differential expression of GS5 regulates grain size in rice[J]. Journal of Experimental Botany, 2015, 66(9): 2611-2623. DOI:10.1093/jxb/erv058 |
| [18] |
MA M, SHEN S Y, BAI C, WANG W Q, FENG X H, YING J Z, SONG X J. Control of grain size in rice by TGW3 phosphorylation of OsIAA10 through potentiation of OsIAA10-OsARF4-mediated auxin signaling[J]. Cell Reports, 2023, 42(3): 112187. DOI:10.1016/j.celrep.2023.112187 |
| [19] |
GAO X, ZHANG J, CAI G, DU H, LI J, WANG R, WANG Y, YIN J, ZHANG W, ZHANG H, HUANG J. qGL3/OsPPKL1 induces phosphorylation of 14-3-3 protein OsGF14b to inhibit OsBZR1 function in brassinosteroid signaling[J]. Plant Physiology, 2022, 188(1): 624-636. DOI:10.1093/plphys/kiab484 |
| [20] |
ZUO Z W, ZHANG Z H, HUANG D R, FAN Y Y, YU S B, ZHUANG J Y, ZHU Y J. Control of thousand-grain weight by OsMADS56 in rice[J]. International Journal of Molecular Sciences, 2021, 23(1): 125. DOI:10.3390/ijms23010125 |
| [21] |
HUANG K, WANG D, DUAN P, ZHANG B, XU R, LI N, LI Y. WIDE AND THICK GRAIN 1, which encodes an otubain-like protease with deubiquitination activity, influences grain size and shape in rice[J]. Plant Journal, 2017, 91(5): 849-860. DOI:10.1111/tpj.13613 |
| [22] |
TAN W, MIAO J, XU B, ZHOU C, WANG Y, GU X, LIANG S, WANG B, CHEN C, ZHU J, ZUO S, YANG Z, GONG Z, YOU A, WU S, LIANG G, ZHOU Y. Rapid production of novel beneficial alleles for improving rice appearance quality by targeting a regulatory element of SLG7[J]. Plant Biotechnology Journal, 2023, 21(7): 1305-1307. DOI:10.1111/pbi.14041 |
| [23] |
QIAO S, SUN S, WANG L, WU Z, LI C, LI X, WANG T, LENG L, TIAN W, LU T, WANG X. The RLA1/SMOS1 transcription factor functions with OsBZR1 to regulate brassinosteroid signaling and rice architecture[J]. Plant Cell, 2017, 29(2): 292-309. DOI:10.1105/tpc.16.00611 |
| [24] |
PATHAK R R, MANDAL V K, JANGAM A P, SHARMA N, MADAN B, JAISWAL D K, RAGHURAM N. Heterotrimeric G-protein alpha subunit (RGA1) regulates tiller development, yield, cell wall, nitrogen response and biotic stress in rice[J]. Science Reports, 2021, 11(1): 2323. DOI:10.1038/s41598-021-81824-1 |
| [25] |
ZHANG D P, ZHOU Y, YIN J F, YAN X J, LIN S, XU W F, BALUSKA F, WANG Y P, XIA Y J, LIANG G H, LIANG J S. Rice G-protein subunits qPE9-1 and RGB1 play distinct roles in abscisic acid responses and drought adaptation[J]. Journal of Experimental Botany, 2015, 66(20): 6371-6384. DOI:10.1093/jxb/erv350 |
| [26] |
CHEN Y, CHEN Y, ZHANG Y, ZHANG D, LI G, WEI J, HUA X, LYU B, LIU L. Heterotrimeric G protein gamma subunit DEP1 is involved in hydrogen peroxide signaling and promotes aerenchyma formation in rice roots[J]. Plant Signaling & Behavior, 2021, 16(5): 1889251. DOI:10.1080/15592324.2021.1889251 |
| [27] |
LIU S, HUA L, DONG S, CHEN H, ZHU X, JIANG J, ZHANG F, LI Y, FANG X, CHEN F. OsMAPK6, a mitogen-activated protein kinase, influences rice grain size and biomass production[J]. The Plant Journal, 2015, 84(4): 672-681. DOI:10.1111/tpj.13025 |
| [28] |
FAN C, XING Y, MAO H, LU T, HAN B, XU C, LI X, ZHANG Q. GS3, a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein[J]. Theoretical and Applied Genetics, 2006, 112: 1164-1171. DOI:10.1007/s00122-006-0218-1 |
| [29] |
YAN H, WANG Y, CHEN B, WANG W, SUN H, SUN H, LI J, ZHAO Q. OsCKX2 regulates phosphate deficiency tolerance by modulating cytokinin in rice[J]. Plant Science, 2022, 319: 111257. DOI:10.1016/j.plantsci.2022.111257 |
| [30] |
HU X, QIAN Q, XU T, ZHANG Y, DONG G, GAO T, XIE Q, XUE Y. The U-box ubiquitin ligase TUD1 promotes brassinosteroid-induced GSK2 degradation in rice[J]. Plant Communications, 2023, 4(2): 100450. DOI:10.1016/j.xplc.2022.100450 |
| [31] |
WU Y, FU Y, ZHAO S, GU P, ZHU Z, SUN C, TAN L. CLUSTERED PRIMARY BRANCH 1, a new allele of DWARF11, controls panicle architecture and seed size in rice[J]. Plant Biotechnology Journal, 2016, 14(1): 377-386. DOI:10.1111/pbi.12391 |
| [32] |
XIA D, ZHOU H, LIU R, DAN W, LI P, WU B, CHEN J, WANG L, GAO G, ZHANG Q, HE Y. GL3. 3, a novel QTL encoding a GSK3/SHAGGY-like kinase, epistatically interacts with GS3 to produce extra-long grains in rice[J]. Molecular Plant, 2018, 11(5): 754-756. DOI:10.1016/j.molp.2018.03.006 |
| [33] |
CHEN G, XUAN W, ZHAO P, YAO X, PENG C, TIAN Y, YE J, WANG B, HE J, CHI W, YU J, GE Y, LI J, DAI Z, XU D, WANG C, WAN J. OsTUB1 confers salt insensitivity by interacting with Kinesin13A to stabilize microtubules and ion transporters in rice[J]. New Phytologist, 2022, 235(5): 1836-1852. DOI:10.1111/nph.18282 |
| [34] |
ZHANG B, WANG X, ZHAO Z, WANG R, HUANG X, ZHU Y, YUAN L, WANG Y, XU X, BURLINGAME A L, GAO Y, SUN Y, TANG W. OsBRI1 activates BR signaling by preventing binding between the TPR and kinase domains of OsBSK3 via phosphorylation[J]. Plant Physiology, 2016, 170(2): 1149-1161. DOI:10.1104/pp.15.01668 |
| [35] |
LIU X, TIAN Y, CHI W, ZHANG H, YU J, CHEN G, WU W, JIANG X, WANG S, LIN Z, XUAN W, YE J, WANG B, LIU Y, SUN Z, XU D, WANG C, WAN J. Alternative splicing of OsGS1; 1 affects nitrogen-use efficiency, grain development, and amylose content in rice[J]. The Plant Journal, 2022, 110(6): 1751-1762. DOI:10.1111/tpj.15768 |
| [36] |
WANG Y, YANG Z, XING M, HUANG J, DING Y, ZHANG L, LI F, NIE Y, WANG S, LI Y, ZHAO M, GE J, LOU D, LIU Z, FAN W, GUO W, ZHENG X, QIAN Q, YANG Q, QIAO W. A novel QTL GL12 from wild rice increases grain length and weight in cultivated rice[J]. Plant Biotechnology Journal, 2023, 21(8): 1513-1515. DOI:10.1111/pbi.14014 |
| [37] |
LIU R, FENG Q, LI P, LOU G, CHEN G, JIANG H, GAO G, ZHANG Q, XIAO J, LI X, XIONG L, HE Y. GLW7.1, a strong functional allele of Ghd7, enhances grain size in rice[J]. International Journal of Molecular Sciences, 2022, 23(15): 8715. DOI:10.3390/ijms23158715 |
| [38] |
SHAO G, TANG S, LUO J, JIAO G, WEI X, TANG A, WU J, ZHUANG J, HU P. Mapping of qGL7-2, a grain length QTL on chromosome 7 of rice[J]. Journal of Genetics and Genomics, 2010, 37(8): 523-531. DOI:10.1016/s1673-8527(09)60072-2 |
| [39] |
黄娟, 高利军, 李经成, 周维永, 邓国富, 潘英华, 卿冬进, 陈韦韦, 伍豪, 戴高兴. 利用CRISPR/Cas9技术改良三系杂交稻恢复系外观品质[J]. 西南农业学报, 2023, 36(9): 1835-1842. DOI:10.16213/j.cnki.scjas.2023.9.002 HUANG J, GAO L J, LI J C, ZHOU W Y, DENG G F, PAN Y H, QING D J, CHEN W W, WU H, DAI G X. Improving restorer line appearance quality of three-line hybrid rice via CRISPR/Cas9 technology[J]. Southwest China Journal of Agricultural Sciences, 2023, 36(9): 1835-1842. DOI:10.16213/j.cnki.scjas.2023.9.002 |
| [40] |
LI J, THOMSON M, MCCOUCH S R. Fine mapping of a grain-weight quantitative trait locus in the pericentromeric region of rice chromosome 3[J]. Genetics, 2004, 168(4): 2187-2195. DOI:10.1534/genetics.104.034165 |
| [41] |
胡婷婷, 王健康, 丁成伟, 郭荣良, 吴玉玲, 徐家安, 王友霜, 赵轶鹏, 何弯弯. 植物种子的发育调控研究进展[J]. 中国农业科技导报, 2021, 23(5): 27-34. DOI:10.13304/j.nykjdb.2020.0048 HU T T, WANG J K, DING C W, GUO R L, WU Y L, XU J A, WANG Y S, ZHAO Y P, HE W W. Progress on regulation mechanism of plant seed development[J]. Journal of Agricultural Science and Technology, 2021, 23(5): 27-34. DOI:10.13304/j.nykjdb.2020.0048 |
| [42] |
SONG X J, HUANG W, SHI M, ZHU M Z, LIN H X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nature Genetics, 2007, 39(5): 623-630. DOI:10.1038/ng2014 |
| [43] |
SHI C, REN Y, LIU L, WANG F, ZHANG H, TIAN P, PAN T, WANG Y, JING R, LIU T, WU F, LIN Q, LEI C, ZHANG X, ZHU S, GUO X, WANG J, ZHAO Z, WANG J, ZHAI H, CHENG Z, WAN J. Ubiquitin specific protease 15 has an important role in regulating grain width and size in rice[J]. Plant Physiology, 2019, 180(1): 381-391. DOI:10.1104/pp.19.00065 |
| [44] |
DUAN P, RAO Y, ZENG D, YANG Y, XU R, ZHANG B, DONG G, QIAN Q, LI Y. SMALL GRAIN 1, which encodes a mitogen-activated protein kinase kinase 4, influences grain size in rice[J]. The Plant Journal, 2014, 77(4): 547-557. DOI:10.1111/tpj.12405 |
| [45] |
WANG S, HAN S, ZHOU X, ZHAO C, GUO L, ZHANG J, LIU F, HUO Q, ZHAO W, GUO Z, CHEN X. Phosphorylation and ubiquitination of OsWRKY31 are integral to OsMKK10-2-mediated defense responses in rice[J]. The Plant Cell, 2023, 35(6): 2391-2412. DOI:10.1093/plcell/koad064 |
| [46] |
GUO T, CHEN K, DONG N Q, SHI C L, YE W W, GAO J P, SHAN J X, LIN H X. GRAIN SIZE AND NUMBER1 negatively regulates the OsMKKK10-OsMKK4-OsMPK6 cascade to coordinate the trade-off between grain number per panicle and grain size in rice[J]. The Plant Cell, 2018, 30(4): 871-888. DOI:10.1105/tpc.17.00959 |
| [47] |
MIAO J, YANG Z, ZHANG D, WANG Y, XU M, ZHOU L, WANG J, WU S, YAO Y, DU X, GU F, GONG Z, GU M, LIANG G, ZHOU Y. Mutation of RGG2, which encodes a type B heterotrimeric G protein gamma subunit, increases grain size and yield production in rice[J]. Plant Biotechnology Journal, 2019, 17(3): 650-664. DOI:10.1111/pbi.13005 |
| [48] |
WANG S, LI S, LIU Q, WU K, ZHANG J, WANG S, WANG Y, CHEN X, ZHANG Y, GAO C, WANG F, HUANG H, FU X. The OsSPL16-GW7 regulatory module determines grain shape and simultaneously improves rice yield and grain quality[J]. Nature Genetics, 2015, 47(8): 949-954. DOI:10.1038/ng.3352 |
| [49] |
ZHAO D S, LI Q F, ZHANG C Q, ZHANG C, YANG Q Q, PAN L X, REN X Y, LU J, GU M H, LIU Q Q. GS9 acts as a transcriptional activator to regulate rice grain shape and appearance quality[J]. Nature Communications, 2018, 9(1): 1240. DOI:10.1038/s41467-018-03616-y |
| [50] |
HU J, WANG Y, FANG Y, ZENG L, XU J, YU H, SHI Z, PAN J, ZHANG D, KANG S, ZHU L, DONG G, GUO L, ZENG D, ZHANG G, XIE L, XIONG G, LI J, QIAN Q. A rare allele of GS2 enhances grain size and grain yield in rice[J]. Molecular Plant, 2015, 8(10): 1455-1465. DOI:10.1016/j.molp.2015.07.002 |
| [51] |
PRAKASH S, RAI R, ZAMZAM M, AHMAD O, PEESAPATI R, VIJAYRAGHAVAN U. OsbZIP47 is an integrator for meristem regulators during rice plant growth and development[J]. Frontiers in Plant Science, 2022, 13: 865928. DOI:10.3389/fpls.2022.865928 |
| [52] |
QIAO J, JIANG H, LIN Y, SHANG L, WANG M, LI D, FU X, GEISLER M, QI Y, GAO Z, QIAN Q. A novel miR167a-OsARF6-OsAUX3 module regulates grain length and weight in rice[J]. Molecular Plant, 2021, 14(10): 1683-1698. DOI:10.1016/j.molp.2021.06.023 |
| [53] |
AKABANE T, SUZUK I N, TSUCHIYA W, YOSHIZAWA T, MATSUMURA H, HIROTSU N, KATOH E. Expression, purification and crystallization of TGW6, which limits grain weight in rice[J]. Protein Expression and Purification, 2021, 188: 105975. DOI:10.1016/j.pep.2021.105975 |
| [54] |
LIU L, TONG H, XIAO Y, CHE R, XU F, HU B, LIANG C, CHU J, LI J, CHU C. Activation of Big Grain1 significantly improves grain size by regulating auxin transport in rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(35): 11102-11107. DOI:10.1073/pnas.1512748112 |
| [55] |
HAO J, WANG D, WU Y, HUANG K, DUAN P, LI N, XU R, ZENG D, DONG G, ZHANG B, ZHANG L, INZE D, QIAN Q, LI Y. The GW2-WG1-OsbZIP47 pathway controls grain size and weight in rice[J]. Molecular Plant, 2021, 14(8): 1266-1280. DOI:10.1016/j.molp.2021.04.011 |
| [56] |
HONG Z, UEGUCHI-TANAK A M, UMEMURA K, UOZU S, FUJIOKA S, TAKATSUTO S, YOSHIDA S, ASHIKARI M, KITANO H, MATSUOKA M. A rice brassinosteroid-deficient mutant, ebisu dwarf (d2), is caused by a loss of function of a new member of cytochrome P450[J]. The Plant Cell, 2003, 15(12): 2900-2910. DOI:10.1105/tpc.014712 |
| [57] |
YUAN H, XU Z, CHEN W, DENG C, LIU Y, YUAN M, GAO P, SHI H, TU B, LI T, KANG L, MA B, WANG Y, WANG J, CHEN X, LI S, QIN P. OsBSK2, a putative brassinosteroid-signalling kinase, positively controls grain size in rice[J]. Journal of Experimental Botany, 2022, 73(16): 5529-5542. DOI:10.1093/jxb/erac222 |
| [58] |
FENG Z, WU C, WANG C, ROH J, ZHANG L, CHEN J, ZHANG S, ZHANG H, YANG C, HU J, YOU X, LIU X, YANG X, GUO X, ZHANG X, WU F, TERZAGHI W, KIM S K, JIANG L, WAN J. SLG controls grain size and leaf angle by modulating brassinosteroid homeostasis in rice[J]. Journal of Experimental Botany, 2016, 67(14): 4241-4253. DOI:10.1093/jxb/erw204 |
| [59] |
SHI C L, DONG N Q, GUO T, YE W W, SHAN J X, LIN H X. A quantitative trait locus GW6 controls rice grain size and yield through the gibberellin pathway[J]. The Plant Journal, 2020, 103(3): 1174-1188. DOI:10.1111/tpj.14793 |
| [60] |
RUAN B, SHANG L, ZHANG B, HU J, WANG Y, LIN H, ZHANG A, LIU C, PENG Y, ZHU L, REN D, SHEN L, DONG G, ZHANG G, ZENG D, GUO L, QIAN Q, GAO Z. Natural variation in the promoter of TGW2 determines grain width and weight in rice[J]. New Phytologist, 2020, 227(2): 629-640. DOI:10.1111/nph.16540 |
| [61] |
LUO J, LIU H, ZHOU T, GU B, HUANG X, SHANGGUAN Y, ZHU J, LI Y, ZHAO Y, WANG Y, ZHAO Q, WANG A, WANG Z, SANG T, WANG Z, HAN B. An-1 encodes a basic helix-loop-helix protein that regulates awn development, grain size, and grain number in rice[J]. The Plant Cell, 2013, 25(9): 3360-3376. DOI:10.1105/tpc.113.113589 |
| [62] |
HE Z, ZENG J, REN Y, CHEN D, LI W, GAO F, CAO Y, LUO T, YUAN G, WU X, LIANG Y, DENG Q, WANG S, ZHENG A, ZHU J, LIU H, WANG L, LI P, LI S. OsGIF1 positively regulates the sizes of stems, leaves, and grains in rice[J]. Frontiers in Plant Science, 2017, 8: 1730. DOI:10.3389/fpls.2017.01730 |
| [63] |
GAO X, ZHANG J, LI J, WANG Y, ZHANG R, DU H, YIN J, CAI G, WANG R, ZHANG B, ZHAO Z, ZHANG H, HUANG J. The phosphoproteomic and interactomic landscape of qGL3/OsPPKL1-mediated brassinosteroid signaling in rice[J]. The Plant Journal, 2022, 109(5): 1048-1063. DOI:10.1111/tpj.15613 |
| [64] |
YUN P, LI Y, WU B, ZHU Y, WANG K, LI P, GAO G, ZHANG Q, LI X, LI Z, HE Y. OsHXK3 encodes a hexokinase-like protein that positively regulates grain size in rice[J]. Theoretical and Applied Genetics, 2022, 135(10): 3417-3431. DOI:10.1007/s00122-022-04189-7 |
| [65] |
MOON J, PARRY G, ESTELLE M. The ubiquitin-proteasome pathway and plant development[J]. The Plant Cell, 2004, 16(12): 3181-3195. DOI:10.1105/tpc.104.161220 |
| [66] |
LIU D, ZHANG X, LI Q, XIAO Y, ZHANG G, YIN W, NIU M, MENG W, DONG N, LIU J, YANG Y, XIE Q, CHU C, TONG H. The U-box ubiquitin ligase TUD1 promotes brassinosteroid-induced GSK2 degradation in rice[J]. Plant Communications, 2023, 4(2): 100450. DOI:10.1016/j.xplc.2022.100450 |
| [67] |
WANG S L, ZHANG Z H, FAN Y Y, HUANG D R, YANG Y L, ZHUANG J Y, ZHU Y J. Control of grain weight and size in Rice (Oryza sativa L.) by OsPUB3 encoding a U-Box E3 ubiquitin ligase[J]. Rice, 2022, 15(1): 1-12. DOI:10.1186/s12284-022-00604-1 |
| [68] |
BENT A F. Plant mitogen-activated protein kinase cascades: Negative regulatory roles turn out positive[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(3): 784-786. DOI:10.1073/pnas.98.3.784 |
| [69] |
LIU Z, MEI E, TIAN X, HE M, TANG J, XU M, LIU J, SONG L, LI X, WANG Z, GUAN Q, XU Q, BU Q. OsMKKK70 regulates grain size and leaf angle in rice through the OsMKK4-OsMAPK6-OsWRKY53 signaling pathway[J]. Journal of Integrative Plant Biology, 2021, 63(12): 2043-2057. DOI:10.1111/jipb.13174 |
| [70] |
SUN S, WANG L, MAO H, SHAO L, LI X, XIAO J, OUYANG Y, ZHANG Q. A G-protein pathway determines grain size in rice[J]. Nature Communications, 2018, 9(1): 851. DOI:10.1038/s41467-018-03141-y |
| [71] |
MA S, ZHONG Y, ZHENG S, HE Y, YANG S, WANG L, TRAW M B, ZHANG Q, ZHANG X. Rapid identification by resequencing-based QTL Mapping of a novel allele RGA1-FH decreasing grain length in a rice restorer line 'Fuhui212'[J]. International Journal of Molecular Sciences, 2023, 24(13): 10746. DOI:10.3390/ijms241310746 |
| [72] |
LI N, XU R, LI Y. Molecular networks of seed size control in plants[J]. Annual Review of Plant Biology, 2019, 70: 435-463. DOI:10.1146/annurev-arplant-050718-095851 |
| [73] |
SHENG M, MA X, WANG J, XUE T, LI Z, CAO Y, YU X, ZHANG X, WANG Y, XU W, SU Z. KNOX Ⅱ transcription factor HOS59 functions in regulating rice grain size[J]. The Plant Journal, 2022, 110(3): 863-880. DOI:10.1111/tpj.15709 |
| [74] |
BAUCHER M, MOUSSAWI J, VANDEPUTTE O M, MONTEYNE D, MOL A, PEREZ-MORGA D, EL JAZIRI M. A role for the miR396/GRF network in specification of organ type during flower development, as supported by ectopic expression of Populus trichocarpa miR396c in transgenic tobacco[J]. Plant Biology, 2013, 15(5): 892-898. DOI:10.1111/j.1438-8677.2012.00696.x |
| [75] |
XIA K, OU X, TANG H, WANG R, WU P, JIA Y, WEI X, XU X, KANG S H, KIM S K, ZHANG M. Rice microRNA osa-miR1848 targets the obtusifoliol 14alpha-demethylase gene OsCYP51G3 and mediates the biosynthesis of phytosterols and brassinosteroids during development and in response to stress[J]. Trends in Plant Science, 2020, 25(9): 912-929. DOI:10.1111/nph.13513 |
| [76] |
LI S, GAO F, XIE K, ZENG X, CAO Y, ZENG J, HE Z, REN Y, LI W, DENG Q, WANG S, ZHENG A, ZHU J, LIU H, WANG L, LI P. The OsmiR396c-OsGRF4-OsGIF1 regulatory module determines grain size and yield in rice[J]. Plant Biotechnology Journal, 2016, 14(11): 2134-2146. DOI:10.1111/pbi.12569 |
| [77] |
ASHIKARI M, SAKAKIBARA H, LI N S, YAMAMOTO T, TAKASHI T, NISHIMURA A, ANGELES E R, QIAN Q, KITANO H, MATSUOKA M. Cytokinin oxidase regulates rice grain production[J]. Science, 2005, 309(5735): 741-745. DOI:10.1126/science.1113373 |
| [78] |
曹煜东, 肖湘谊, 叶乃忠, 丁晓雯, 易晓璇, 刘金灵, 肖应辉. 生长素调控因子OsGRF4协同调控水稻粒形和稻瘟病抗性[J]. 中国水稻科学, 2021, 35(6): 629-638. DOI:10.16819/j.1001-7216.2021.210206 CAO Y D, XIAO X Y, YE N Z, DING X W, YI X X, LIU J L, XIAO Y H. Auxin regulator OsGRF4 simultaneously regulates rice grain shape and blast resistance[J]. Chinese Journal of Rice Science, 2021, 35(6): 629-638. DOI:10.16819/j.1001-7216.2021.210206 |
| [79] |
ZHANG W, PENG K, CUI F, WANG D, ZHAO J, ZHANG Y, YU N, WANG Y, ZENG D, WANG Y, CHENG Z, ZHANG K. Cytokinin oxidase/dehydrogenase OsCKX11 coordinates source and sink relationship in rice by simultaneous regulation of leaf senescence and grain number[J]. Plant Biotechnology Journal, 2021, 19(2): 335-350. DOI:10.1111/pbi.13467 |
| [80] |
贾利霞, 齐艳华. 生长素代谢、运输及信号转导调控水稻粒型研究进展[J]. 植物学报, 2022, 57(3): 263-275. DOI:10.11983/CBB21227 JIA L X, QI Y H. Advances in regulation of auxin metabolism, transport and signal transduction on rice grain size[J]. Chinese Bulletin of Botany, 2022, 57(3): 263-275. DOI:10.11983/CBB21227 |
| [81] |
FUJIOKA S, SAKURAI A. Biosynthesis and metabolism of brassinosteroids[J]. Annual Review of Plant Biology, 2003, 54(1): 137-164. DOI:10.1146/annurev.arplant.54.031902.134921 |
| [82] |
SUN H, XU H, LI B, SHANG Y, WEI M, ZHANG S, ZHAO C, QIN R, CUI F, WU Y. The brassinosteroid biosynthesis gene, ZmD11, increases seed size and quality in rice and maize[J]. Plant Physiology and Biochemistry, 2021, 160: 281-293. DOI:10.1016/j.plaphy.2021.01.031 |
| [83] |
TIAN P, LIU J, YAN B, ZHOU C, WANG H, SHEN R. BRASSINOSTEROID-SIGNALING KINASE1-1, a positive regulator of brassinosteroid signalling, modulates plant architecture and grain size in rice[J]. Journal of Experimental Botany, 2023, 74(1): 283-295. DOI:10.1093/jxb/erac429 |
| [84] |
DU H, YONG R, ZHANG J, CAI G, WANG R, LI J, WANG Y, ZHANG H, GAO X, HUANG J. OsBAK2/OsSERK2 expression is repressed by OsBZR1 to modulate brassinosteroid response and grain length in rice[J]. Journal of Experimental Botany, 2023, 196. DOI:10.1093/jxb/erad196 |
| [85] |
CHEN W, HU X, HU L, HOU X, XU Z, YANG F, YUAN M, CHEN F, WANG Y, TU B, LI T, KANG L, TANG S, MA B, WANG Y, LI S, QIN P, YUAN H. Wide Grain 3, a GRAS protein, interacts with DLT to regulate grain size and brassinosteroid signaling in rice[J]. Rice (New York, N.Y.), 2022, 15(1): 55. DOI:10.1186/s12284-022-00601-4 |
| [86] |
ZHOU T, ZHANG K X, ZHANG J, LIU S J, LIANG J, LIU J X, ZHU J, LIANG Y Y, WANG S Q, DENG Q M, LIU H N, JIN J H, LI P, LI S C. Dwarf and low‐tillering 2 functions in brassinosteroid signaling and controls plant architecture and grain size in rice[J]. The Plant Journal, 2023. DOI:10.1111/tpj.16464 |
| [87] |
JIN S K, ZHANG M Q, LENG Y J, XU L N, JIA S W, WANG S L, SONG T, WANG R A, YANG Q Q, TAO T, CAI X L, GAO J P. OsNAC129 regulates seed development and plant growth and participates in the brassinosteroid signaling pathway[J]. Frontiers in Plant Science, 2022, 13: 905148. DOI:10.3389/fpls.2022.905148 |
| [88] |
XIA K, PAN X, CHEN H, XU X, ZHANG M. Rice miR168a-5p regulates seed length, nitrogen allocation and salt tolerance by targeting OsOFP3, OsNPF2.4 and OsAGO1a, respectively[J]. Journal of Plant Physiology, 2023, 280: 153905. DOI:10.1016/j.jplph.2022.153905 |
| [89] |
LIU C, MA T, YUAN D, ZHOU Y, LONG Y, LI Z, DONG Z, DUAN M, YU D, JING Y, BAI X, WANG Y, HOU Q, LIU S, ZHANG J S, CHEN S Y, LI D, LIU X, LI Z, WANG W, LI J, WEI X, MA B, WAN X. The OsEIL1-OsERF115-target gene regulatory module controls grain size and weight in rice[J]. Plant Biotechnology Journal, 2022, 20(8): 1470-1486. DOI:10.1111/pbi.13825 |
| [90] |
MEHRA P, PANDEY B K, VERMA L, PRUSTY A, SINGH A P, SHARMA S, MALIK N, BENNETT M J, PARIDA S K, GIRI J, TYAGI A K. OsJAZ11 regulates spikelet and seed development in rice[J]. Plant Direct, 2022, 6(5): e401. DOI:10.1002/pld3.401 |
| [91] |
BERGER S L, KOUZARIDES T, SHIEKHATTAR R, SHILATIFARD A. An operational definition of epigenetics[J]. Genes & Development, 2009, 23(7): 781-783. DOI:10.1101/gad.1787609 |
| [92] |
杨涛, 马小倩, 张全, 张洪亮. 组蛋白修饰在水稻中的研究进展[J]. 中国农业科技导报, 2022, 24(4): 11-20. DOI:10.13304/j.nykjdb.2021.0497 YANG T, MA X Q, ZHANG Q, ZHANG H L. Research progress of histone modification in rice[J]. Journal of Agricultural Science and Technology, 2022, 24(4): 11-20. DOI:10.13304/j.nykjdb.2021.0497 |
| [93] |
SONG X J, KUROHA T, AYANO M, FURUTA T, NAGAI K, KOMEDA N, SEGAMI S, MIURA K, OGAWA D, KAMURA T, SUZUKI T, HIGASHIYAMA T, YAMASAKI M, MORI H, INUKAI Y, WU J, KITANO H, SAKAKIBARA H, JACOBSEN S E, ASHIKARI M. Rare allele of a previously unidentified histone H4 acetyltransferase enhances grain weight, yield, and plant biomass in rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(1): 76-81. DOI:10.1073/pnas.1421127112 |
| [94] |
ZHANG X, SUN J, CAO X, SONG X. Epigenetic mutation of RAV6 affects leaf angle and seed size in rice[J]. Plant Physiology, 2015, 169(3): 2118-2128. DOI:10.1104/pp.15.00836 |
| [95] |
MAO D, TAO S, LI X, GAO D, TANG M, LIU C, WU D, BAI L, HE Z, WANG X, YANG L, ZHU Y, ZHANG D, ZHANG W, CHEN C. The Harbinger transposon-derived gene PANDA epigenetically coordinates panicle number and grain size in rice[J]. Plant Biotechnology Journal, 2022, 20(6): 1154-1166. DOI:10.1111/pbi.13799 |
| [96] |
YANG T, GU H, YANG W, LIU B, LIANG S, ZHAO J. Artificially selected grain shape gene combinations in Guangdong Simiao varieties of rice (Oryza sativa L.)[J]. Rice, 2023, 16(1): 1-14. DOI:10.1186/s12284-023-00620-9 |
| [97] |
REN D, DING C, QIAN Q. Molecular bases of rice grain size and quality for optimized productivity[J]. Science Bulletin, 2023, 68(3): 314-350. DOI:10.1016/j.scib.2023.01.026 |
| [98] |
ZHANG X F, YANG C Y, LIN H X, WANG J W, XUE H W. Rice SPL12 coevolved with GW5 to determine grain shape[J]. Science Bulletin, 2021, 66(23): 2353-2357. DOI:10.1016/j.scib.2021.05.005 |
| [99] |
MO Y, LI G, LIU L, ZHANG Y, LI J, YANG M, CHEN S, LIN Q, FU G, ZHENG D, LING Y. OsGRF4AA compromises heat tolerance of developing pollen grains in rice[J]. Frontiers in Plant Science, 2023, 14: 1121852. DOI:10.3389/fpls.2023.1121852 |
| [100] |
TANG J, MEI E, HE M, BU Q, TIAN X. Functions of OsWRKY24, OsWRKY70 and OsWRKY53 in regulating grain size in rice[J]. Planta, 2022, 255(4): 92. DOI:10.1007/s00425-022-03871-w |
| [101] |
ZHAO Y, ZHANG X, CHENG Y, DU X, TEOTIA S, MIAO C, SUN H, FAN G, TANG G, XUE H, ZHAO Q, PENG T. The miR167-OsARF12 module regulates rice grain filling and grain size downstream of miR159[J]. Plant Communications, 2023, 100604. DOI:10.1016/j.xplc.2023.100604 |
| [102] |
LIU Q, HAN R, WU K, ZHANG J, YE Y, WANG S, CHEN J, PAN Y, LI Q, XU X, ZHOU J, TAO D, WU Y, FU X. G-protein βγ subunits determine grain size through interaction with MADS-domain transcription factors in rice[J]. Nature Communications, 2018, 9(1): 852. DOI:10.1038/s41467-018-03047-9 |
| [103] |
TAO Y, MIAO J, WANG J, LI W, XU Y, WANG F, JIANG Y, CHEN Z, FAN F, XU M, ZHOU Y, LIANG G, YANG J. RGG1, involved in the cytokinin regulatory pathway, controls grain size in rice[J]. Rice, 2020, 13(1): 1-13. DOI:10.1186/s12284-020-00436-x |
| [104] |
YAN S, ZOU G, LI S, WANG H, LIU H, ZHAI G, GUO P, SONG H, YAN C, TAO Y. Seed size is determined by the combinations of the genes controlling different seed characteristics in rice[J]. Theoretical and Applied Genetics, 2011, 123: 1173-1181. DOI:10.1007/s00122-011-1657-x |
| [105] |
郑镇武, 赵宏源, 梁晓娅, 王一珺, 王驰航, 巩高洋, 黄金燕, 张桂权, 王少奎, 刘祖培. 水稻qGL3.4调控籽粒大小与株型[J]. 遗传, 2023, 45(9): 835-44. DOI:10.16288/j.yczz.23-064 ZHENG Z W, ZHAO H Y, LIANG X Y, WANG Y J, WANG C H, GONG G Y, HUANG J Y, ZHANG G Y, WANG S K, LIU Z P. qGL3.4 controls kernel size and plant architecture in rice[J]. Hereditas, 2023, 45(9): 835-44. DOI:10.16288/j.yczz.23-064 |
| [106] |
GAO Q, ZHANG N, WANG W Q, SHEN S Y, BAI C, SONG X J. The ubiquitin-interacting motif-type ubiquitin receptor HDR3 interacts with and stabilizes the histone acetyltransferase GW6a to control the grain size in rice[J]. The Plant Cell, 2021, 33(10): 3331-3347. DOI:10.1093/plcell/koab194 |
| [107] |
FAN C, YU S, WANG C, XING Y. A causal C-A mutation in the second exon of GS3 highly associated with rice grain length and validated as a functional marker[J]. Theoretical and Applied Genetics, 2009, 118: 465-472. DOI:10.1007/s00122-008-0913-1 |
| [108] |
毛艇, 李鑫, 刘研, 张战, 钟顺成, 王诗宇, 赵一洲, 倪善君, 李旭. 聚合gs3、Pita及Pib基因创制长粒高抗稻瘟病核心粳稻种质[J]. 分子植物育种, 2023, 21(6): 1990-1998. DOI:10.13271/j.mpb.021.001990 MAO T, LI X, LIU Y, ZHANG Z, ZHONG S C, WANG S Y, ZHAO Y Z, NI S J, LI X. Pyramiding gs3, Pita and Pib genes to create core Japonica rice germplasm with long grain shape and high blast resistance[J]. Molecular Plant Breeding, 2023, 21(6): 1990-1998. DOI:10.13271/j.mpb.021.001990 |
| [109] |
SUN K, LI D, XIA A, ZHAO H, WEN Q, JIA S, WANG J, YANG G, ZHOU D, HUANG C, WANG H, CHEN Z, GUO T. Targeted identification of rice grain-sssociated gene allelic variation through mutation induction, targeted sequencing, and whole genome sequencing combined with a Mixed-Samples Strategy[J]. Rice, 2022, 15(1): 1-15. DOI:10.1186/s12284-022-00603-2 |
| [110] |
康雪蒙. GS3、GL7、GW7基因对空育131粒型改良的研究[D]. 哈尔滨: 黑龙江大学, 2020. KANG X M. Study on the effect of GS3, GL7 and GW7 genes on the grain shape improvement of Kongyu 131[D]. Harbin: Heilongjiang University, 2020. |
| [111] |
温一博, 陈淑婷, 徐正进, 孙健, 徐铨. DEP1、Gn1a和qSW5组合应用调控水稻穗部性状[J]. 中国农业科学, 2023, 56(7): 1218-1227. DOI:10.3864/j.issn.0578-1752.2023.07.002 WEN Y B, CHEN S T, XU Z J, SUN J, XU Q. Combination of DEP1, Gn1a, and qSW5 Regulates the Panicle Architecture in Rice[J]. Scientia Agricultura Sinica, 2023, 56(7): 1218-1227. DOI:10.3864/j.issn.0578-1752.2023.07.002 |
| [112] |
JIANG L A O, LI G, CHERN M, JAIN R, PHAM N T, MARTIN J A, SCHACKWITZ W S, ZHAO J, RUAN D, HUANG R, ZHENG J, RONALD P C. Whole-genome sequencing identifies a rice grain shape mutant, gs9–1[J]. Rice, 2019, 12: 1-11. DOI:10.1186/s12284-019-0308-8 |
| [113] |
韩政宏, 段宇轩, 徐善斌, 王敬国, 刘化龙, 杨洛淼, 贾琰, 辛威, 郑洪亮, 邹德堂. 利用CRISPR/Cas9技术敲除GS3和GS9基因改良水稻粒型性状[J]. 华北农学报, 2022, 37(2): 9-17. DOI:10.7668/HBNXB.20192762 HAN Z H, DUAN Y X, XU S B, WANG J G, LIU H L, YANG L M, JIA Y, XIN W, ZHENG H L, ZOU D T. Improvement of grain shape in rice by knocking GS3 and GS9 via CRISPR/Cas9 system[J]. Acta Agriculture Boreali-Sinica, 2022, 37(2): 9-17. DOI:10.7668/HBNXB.20192762 |
(责任编辑 马春敏)
 2023, Vol. 50
2023, Vol. 50