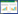文章信息
基金项目
- 国家自然科学基金(32260497);贵州省基础研究(自然科学)项目(黔科合基础-ZK〔2023〕一般178);贵州省科技计划项目(黔科合平台人才〔2018〕5263);贵州省科技计划项目(黔科合支撑〔2022〕重点028)
作者简介
-
彭强,博士,副研究员,现任职于贵州省农业科学院水稻研究所,为贵州省农业科学院科技人才“百人计划”的青年领军人才、贵州省第七批高层次创新型人才“千”层次人才。主要从事水稻分子育种与农艺性状调控基因功能研究。主持和主要参与国家自然科学基金、国家重点研发计划、省部级项目等 11 项。以第一作者发表 Q1 区 SCI 论文 1 篇( IF=21.9),在卓越期刊发表论文 1 篇,在中文核心期刊发表论文 10 篇;以通信作者在中文核心期刊发表论文 3 篇;副主编出版著作 1 部。
彭强(1986—),男,博士,副研究员,研究方向为水稻分子育种,E-mail:pengqiang_as@163.com.
通讯作者
- 朱速松(1966—),男,博士,研究员,研究方向为水稻分子育种,E-mail:susongzhu@139.com.
文章历史
- 收稿日期:2023-10-07
【研究意义】水稻(Oryza sativa L.)是我国的主要粮食作物,其产量事关国家粮食安全,与水稻产量性状相关的机制研究一直是作物研究领域的热门方向。千粒重是决定水稻单株产量的主要性状之一,而籽粒大小是决定千粒重的重要衡量指标[1]。因此,糙米籽粒大小QTL定位及遗传解析对高产优质稻品种选育具有重要的理论研究意义和应用价值。【前人研究进展】水稻籽粒大小是由主效基因和微效QTL共同控制的复杂数量性状[2]。学者们已通过不同类型的遗传作图群体,如重组自交系(Recombinant inbred line,RIL)、双单倍体(Double haploid,DH)、回交自交系(Backcross inbred line,BIL)等,在水稻全基因组范围内发掘到数量众多的籽粒大小性状QTL,有100多个水稻籽粒大小调控基因被克隆和报道[3]。研究表明,调控水稻粒长和粒重的主效基因有GS3[4]、SG3[5]、OsLG3[6]、TGW3/GL3.3[7-8]、GSA1[9]、GL4[10]、TGW6[11]、GW6a[12]和GLW7[13]等;控制水稻粒宽和粒重的主效基因有GW2[14]、TGW2[15]、GS5[16]、GW5[17]、GW6[18]和GW8[19]等;调控水稻粒厚的主效基因有BG2[20]、FUWA[21]和WTG1[22]等;调控籽粒大小和颖壳发育的主效基因有SMG12[23]、OsPEX1[24]、BSG1[25]等。GS3是第一个被克隆的调控水稻籽粒大小的主效QTL,GS3蛋白负调控粒长,功能缺失可导致籽粒粒长增长。GW5依赖蛋白酶体降解途径负调控水稻粒宽和粒重,功能缺失可促进颖壳细胞分裂,使得谷粒变宽变重。【本研究切入点】目前,尽管在水稻籽粒大小调控机制研究方面已取得一些进展,但由于SSR、InDel、RFLP等传统分子标记构建的水稻遗传连锁图谱存在标记数量少和覆盖密度低等缺陷,不能有效地筛选目标基因,因此仍有一些水稻籽粒大小QTL未被定位[26]。与传统分子标记构建的遗传图谱相比,基于高通量测序技术构建的高密度遗传图谱具有高通量、标记数量多、定位区间小等优势,有利于精确定位和筛选候选基因。【拟解决的关键问题】本研究运用籼稻品种V20B与细长型爪哇稻品种CPSLO17创制的重组自交系和SLAF-seq技术绘制的高密度遗传连锁图谱[27],在4个不同环境下定位糙米籽粒大小性状QTL,以期明确贡献率大且遗传稳定的糙米籽粒大小性状主效QTL,为籽粒大小基因挖掘和高产优质稻分子育种提供科学依据。
1 材料与方法 1.1 试验材料供试遗传群体是以籼稻品种V20B为母本、细长型爪哇稻CPSLO17为父本进行杂交,从F2代开始采用单粒传法创建的F10代RIL(150个遗传相对稳定且性状覆盖度广的家系)。
亲本材料(V20B和CPSLO17)与150份RIL群体分别在4个不同环境(2019年贵州贵阳、2020年贵州贵阳、2021年贵州贵定、2021年海南三亚)的试验田种植,每份材料种植40个单株,每行10株,种植4行,种植规格为宽窄行(宽行30 cm,窄行20 cm),株距20 cm,常规大田栽培管理。成熟后分株系混收,干燥保存备用。
1.2 糙米籽粒大小性状测定分析随机选取每份待测材料各100粒左右的饱满糙米,用万深SC-E大米外观品质检测仪精准测量糙米籽粒大小,重复3次,取算数平均值作为糙米籽粒大小性状的表型值。采用软件Excel和GraphPad Prism 7整理和统计分析RIL群体糙米籽粒大小性状的表型数据,制作分布图;采用Kolmogorov-Smirnov法进行正态分布检验;使用软件SPSS 16.0统计分析不同环境中RIL群体糙米籽粒大小的平均值、标准差、峰度、偏度及其相关系数;采用单因素方差分析评估糙米籽粒大小性状在不同环境间的差异,利用相关系数评价糙米籽粒大小性状在不同环境间的相关性。
1.3 糙米籽粒大小QTL定位本研究采用的遗传图谱为基于V20B/CPSLO17遗传背景的水稻高密度遗传连锁图谱[27]。整合RIL群体的糙米籽粒大小表型数据和基因型数据,采用IciMapping 4.0软件的ICIM-ADD方法进行QTL扫描,检测糙米籽粒大小性状相关QTL。设定全基因组扫描步长为0.1 cM、PIN值为0.001、LOD阈值为3.0,并计算每个QTL的加性效应和表型贡献率。遵循McCouch等[28]提出的QTL命名原则对检测到的QTL进行命名。增效等位基因源自亲本V20B的QTL,其加性效应>0;增效等位基因源自亲本CPSLO17的QTL,其加性效应<0。
2 结果与分析 2.1 亲本籽粒大小性状表型分析对2019年贵州贵阳试验田种植的亲本V20B和CPSLO17的籽粒大小性状(图 1)进行测量。由表 1可知,V20B谷粒和糙米粒的粒宽分别为3.233 mm和2.666 mm,均极显著大于CPSLO17的2.415 mm和2.075 mm;V20B谷粒粒长(9.532 mm)略大于CPSLO17(9.416 mm),而V20B糙米粒粒长(6.563 mm)略小于CPSLO17(6.781 mm);V20B谷粒和糙米粒的长宽比(2.952和2.465)均极显著小于CPSLO17(3.935和3.300)。谷粒和糙米粒的籽粒大小在亲本V20B和CPSLO17之间存在极显著差异。

|
| 图 1 亲本V20B和CPSLO17的籽粒大小表型 Fig. 1 Phenotypes of grain sizes from parental V20B and CPSLO17 |

|
2.2 不同环境下RIL群体的糙米籽粒大小性状表型
150份籼爪交遗传背景(V20B/CPSLO17)的RIL群体分别种植在4个不同环境(2019年贵州贵阳、2020年贵州贵阳、2021年贵州贵定、2021年海南三亚)的试验田,并考察其糙米籽粒大小性状表型。糙米籽粒大小性状在RIL群体中的分布如图 2所示,且4个环境下RIL群体糙米籽粒大小性状分布情况的Kolmogorov-Smirnov检验P值均大于0.1,表明4个环境种植RIL群体的糙米籽粒大小性状在各自的变异范围内均表现出连续的单峰分布,其中2019年贵州贵阳的RIL群体糙米籽粒大小分布在2.56~2.66 mm区间的株系数最多,2020年贵州贵阳和2021年海南三亚的RIL群体在2.46~2.56 mm区间分布的株系数最多,而2021年贵州贵定的RIL群体在2.36~2.46 mm区间分布的株系数最多,两侧的株系数逐渐减少,说明糙米籽粒大小性状易受环境影响。

|
| 图 2 4个环境下RIL群体的糙米籽粒大小性状分布 Fig. 2 Distributions of the brown rice grain size traits in RIL population under four environments |
4个不同环境下,150份RIL群体的糙米籽粒大小性状表型数据如表 2所示。2019年贵州贵阳RIL群体的糙米籽粒大小均值为2.506 mm,变异幅度为0.060,偏度和峰度分别为-0.192和-0.449;2020年贵州贵阳RIL群体的糙米籽粒大小均值为2.430 mm,变异幅度为0.038,偏度和峰度分别为-0.005和0.109;2021年贵州贵定RIL群体的糙米籽粒大小均值为2.433 mm,变异幅度为0.031,偏度和峰度分别为-0.467和-0.217;2021年海南三亚RIL群体的糙米籽粒大小均值为2.535 mm,变异幅度为0.034,偏度和峰度分别为-0.371和-0.378。

|
运用单因素方差分析对不同环境RIL群体的糙米籽粒大小性状表型数据进行统计分析,结果(表 3)显示F值(10.176)远大于F crit值(2.620),且P<0.01,表明RIL群体糙米籽粒大小在不同环境间存在极显著差异,糙米籽粒大小性状受环境影响。同年份不同种植地区间的F值(24.197)远大于F crit值(3.873),且P<0.01,表明RIL群体糙米籽粒大小在不同种植地区间存在极显著差异。同种植地区不同年份间的F值(8.675)远大于F crit值(3.873),且P<0.01,表明RIL群体糙米籽粒大小在不同年份间存在显著差异。
|
2.3 RIL群体的糙米籽粒大小QTL定位分析
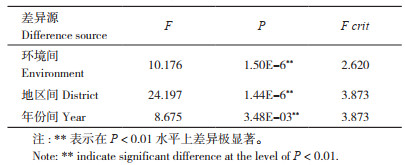
基于水稻高密度遗传图谱[26],结合4种环境种植的糙米籽粒大小性状表型数据,利用IciMapping 4.0的ICIM-ADD方法进行糙米籽粒大小QTL定位分析。结果(图 3)共检测到11个调控糙米籽粒大小的QTL,其中从2019年贵州贵阳糙米检测出3个QTL(qRGS2.1、qRGS4.1、qRGS5),从2020年贵州贵阳糙米检测出4个QTL(qRGS2.2、qRGS5、qRGS7.2和qRGS11.2),从2021年贵州贵定糙米检测出4个QTL(qRGS5、qRGS7.1、qRGS8和qRGS11.3),从2021年海南三亚糙米检测出3个QTL(qRGS5、qRGS11.1和qRGS12)。
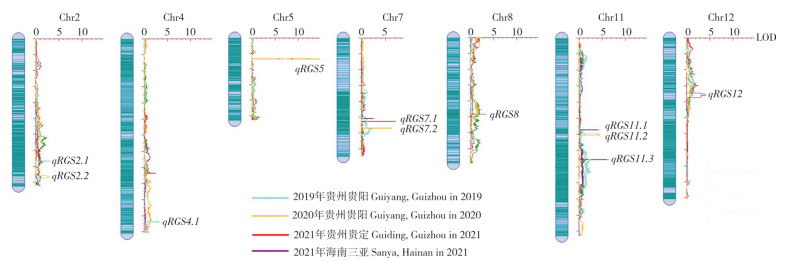
|
| 染色体中绿色线条代表上图标记 Green line in the chromosome represents the marker 图 3 糙米籽粒大小性状QTL在遗传图谱上的分布 Fig. 3 Distributions of identified QTL for brown rice grain size traits on genetic maps |
11个QTL的LOD值介于3.00~14.57之间,表型贡献率为4.84%~29.71%(表 4)。其中,表型贡献率超过10% 的QTL有4个,分别是qRGS5、qRGS7.1、qRGS7.2和qRGS11.3。定位于第5染色体M1642127~M1514505区间内的QTL qRGS5在4个环境中均被反复检测到,其对应的LOD值分别为12.61、14.57、8.88和5.87;表型贡献率分别为29.71%、28.77%、17.27% 和12.50%;加性效应分别为0.13、0.11、0.07和0.07,均大于0,表明QTL qRGS5的增效等位基因源自亲本V20B。3个QTL(qRGS2.1、qRGS2.2和qRGS12)的加性效应小于0,分别为-0.06、-0.04和-0.06,其增效等位基因源自亲本CPSLO17;其余8个QTL的加性效应均大于0,说明其增效等位基因源自亲本V20B(表 4)。

|
3 讨论
随着水稻基因组学的快速发展和新型分子标记的开发,大量的水稻籽粒大小调控基因被相继克隆。研究表明,水稻糙米籽粒大小作为受多基因控制和环境影响的数量性状,具有复杂的遗传多样性[3, 29]。本研究结果与上述观点一致,RIL群体糙米籽粒大小性状在4个环境(2019年贵州贵阳、2020年贵州贵阳、2021年贵州贵定、2021年海南三亚)下均呈现连续的正态分布,表现出受多基因控制的数量性状遗传特征;单因素方差分析结果揭示糙米籽粒大小性状受环境影响。不同种植地区间的F值(24.197)比不同年份间的F值(8.675)大,而P值比不同年份间的小,表明糙米籽粒大小性状在地区间的差异性大于年份间。
亲本间遗传背景多样性是开发多态性分子标记的前提基础,有助于挖掘数量性状QTL。目前已发现水稻12条染色体上都分布着已克隆的籽粒大小调控基因[29],本研究在4个环境中定位到11个糙米籽粒大小性状相关QTL,分布于第2、4、5、7、8、11和12染色体上,其中有6个QTL与前人结果处于相同或相近位置。2号染色体上的qRGS2.1位于SMG1基因附近,qRGS2.2与已克隆的TH1基因位置相邻;4号染色体上的qRGS4.1靠近已克隆的FLO2基因,qRGS4.2与OsBSK3基因位置临近;qRGS5与已克隆的GW5位置相近;qRGS7.1与已克隆的籽粒大小基因OsBZR1位置相临。这些QTL是否为新的调控基因还需后续研究确认。
水稻籽粒大小性状是由少数主效QTL和多个微效QTL共同调控的,表型贡献率超过10% 的主效QTL在分子育种中具有应用价值[30]。本研究共定位到4个主效QTL(qRGS5、qRGS7.1、qRGS7.2和qRGS11.3),表型贡献率超过10%,其中qRGS5的效应值最大,增效等位基因分别来自V20B。本研究基于多年多点试验进行QTL定位,位于第5染色体M1642127~M1514505区间内的qRGS5是唯一一个在4个环境中均被重复检测到的QTL,且4个环境中表型贡献率均超过10%,分别解释群体表型变异率的29.71%、28.77%、17.27% 和12.50%,表明qRGS5是贡献率高且遗传效应稳定的籽粒大小性状主效QTL,也佐证该QTL定位结果是可重复的。这些贡献率高的、遗传效应稳定的籽粒大小QTL,对高产优质稻分子育种具有重要的应用价值。在育种实践中可借助分子标记辅助选择将这些贡献率高的QTL进行聚合置换,创制高产优质新种质。本研究结果为糙米籽粒大小基因挖掘及其功能分子标记开发提供新的理论依据,对高产优质育种具有重要的实践意义。
4 结论本试验以籼稻品种V20B与细长型爪哇稻品种CPSLO17衍生的重组自交系为研究群体,在4种环境共检测到11个糙米籽粒大小性状相关QTL。4个主效QTL(qRGS5、qRGS7.1、qRGS7.2和qRGS11.3)的表型贡献率超过10%,在分子标记辅助选择育种中具有应用价值。3个QTL的增效等位基因源自亲本CPSLO17,8个QTL的增效等位基因源自亲本V20B,这些位点或片段可通过杂交、回交等手段导入改良品系中,提高水稻品种的产量和品质。在4种环境中均被重复检测到的QTL qRGS5是影响糙米籽粒大小且遗传稳定的主效QTL,其定位区间包含5个候选基因,可在DNA和RNA层面进一步开展分子鉴定和验证这些候选基因的功能,为挖掘籽粒大小基因和培育优质高产水稻品种提供新的基因资源和科学依据。
| [1] |
TAN Y F, XING Y Z, LI J X, YU S B, XU C G, ZHANG Q F. Genetic bases of appearance quality of rice grains in Shanyou 63, an elite rice hybrid[J]. Theoretical and Applied Genetics, 2000, 101: 823-829. DOI:10.1007/s001220051549 |
| [2] |
HUANG R Y, JIANG L R, ZHENG J S, WANG T S, WANG H C, HUANG Y M, HONG Z L. Genetic bases of rice grain shape: So many genes, so little known[J]. Trends in Plant Science, 2012, 18(4): 218-226. DOI:10.1016/j.tplants.2012.11.001 |
| [3] |
张健, 杨靖, 王豪, 李冬秀, 杨瑰丽, 黄翠红, 周丹华, 郭涛, 陈志强, 王慧. 基于高密度遗传图谱定位水稻籽粒大小相关性状QTL[J]. 中国农业科学, 2020, 53(2): 225-238. DOI:10.3864/j.issn.0578-1752.2020.02.001 ZHANG J, YANG J, WANG H, LI D X, YANG G L, HUANG C H, ZHOU D H, GUO T, CHEN Z Q, WANG H. QTL mapping for grain size related traits based on a high-density map in rice[J]. Scientia Agricultura Sinica, 2020, 53(2): 225-238. DOI:10.3864/j.issn.0578-1752.2020.02.001 |
| [4] |
MAO H L, SUN S Y, YAO J L, WANG C R, YU S B, XU C G, LI X H, ZHANG Q F. Linking differential domain functions of the GS3 protein to natural variation of grain size in rice[J]. Procedings of the National Acadenmy of Sciences of the United States of America, 2010, 107(45): 19579-19584. DOI:10.1073/pnas.1014419107 |
| [5] |
LI Q, LU L, LIU H, BAI X, ZHOU X, WU B, YUAN M, YANG L, XING Y. A minor QTL, SG3, encoding an R2R3-MYB protein, negatively controls grain length in rice[J]. Theoretical and Applied Genetics, 2020, 133(8): 2387-2399. DOI:10.1007/s00122-020-03606-z |
| [6] |
YU J, XIONG H, ZHU X, ZHANG H, LI H, MIAO J, WANG W, TANG Z, ZHANG Z, YAO G, ZHANG Q, PAN Y, WANG X, RASHID M A R, LI J, GAO Y, LI Z, YANG W, FU X, LI Z. OsLG3 contributing to rice grain length and yield was mined by Ho-LAMap[J]. BMC Biology, 2017, 15(1): 28. DOI:10.1186/s12915-017-0365-7 |
| [7] |
XIA D, ZHOU H, LIU R, DAN W, LI P, WU B, CHEN J, WANG L, GAO G, ZHANG Q, HE Y. GL3.3, a novel QTL encoding a GSK3/SHAGGY-like kinase, epistatically interacts with GS3 to produce extra-long grains in rice[J]. Molecular Plant, 2018, 11(5): 754-756. DOI:10.1016/j.molp.2018.03.006 |
| [8] |
YING J Z, MA M, BAI C, HUANG X H, LIU J L, FAN Y Y, SONG X J. TGW3, a major QTL that negatively modulates grain length and weight in rice[J]. Molecular Plant, 2018, 11(5): 750-753. DOI:10.1016/j.molp.2018.03.007 |
| [9] |
DONG N Q, SUN Y, GUO T, SHI C L, ZHANG Y M, KAN Y, XIANG Y H, ZHANG H, YANG Y B, LI Y C, ZHAO H Y, YU H X, LU Z Q, WANG Y, YE W W, SHAN J X, LIN H X. UDP-glucosyltransferase regulates grain size and abiotic stress tolerance associated with metabolic flux redirection in rice[J]. Nature Communications, 2020, 11(1): 2629. DOI:10.1038/s41467-020-16403-5 |
| [10] |
WU W, LIU X, WANG M, MEYER R S, LUO X, NDJIONDJOP M N, TAN L, ZHANG J, WU J, CAI H, SUN C, WANG X, WING R A, ZHU Z. A single-nucleotide polymorphism causes smaller grain size and loss of seed shattering during African rice domestication[J]. Nature Plants, 2017, 3: 17064. DOI:10.1038/nplants.2017.64 |
| [11] |
ISHIMARU K, HIROTSU N, MADOKA Y, MURAKAMI N, HARA N, ONODERA H, KASHIWAGI T, UJIIE K, SHIMIZU B, ONISHI A, MIYAGAWA H, KATOH E. Loss of function of the IAA-glucose hydrolase gene TGW6 enhances rice grain weight and increases yield[J]. Nature Genetics, 2013, 45(6): 707-711. DOI:10.1038/ng.2612 |
| [12] |
SONG X J, KUROHA T, AYANO M, FURUTA T, NAGAI K, KOMEDA N, SEGAMI S, MIURA K, OGAWA D, KAMURA T, SUZUKI T, HIGASHIYAMA T, YAMASAKI M, MORI H, INUKAI Y, WU J, KITANO H, SAKAKIBARA H, JACOBSEN S E, ASHIKARI M. Rare allele of a previously unidentified histone H4 acetyltransferase enhances grain weight, yield, and plant biomass in rice[J]. Procedings of the National Acadenmy of Sciences of the United States of America, 2015, 112(1): 76-81. DOI:10.1073/pnas.1421127112 |
| [13] |
SI L, CHEN J, HUANG X, GONG H, LUO J, HOU Q, ZHOU T, LU T, ZHU J, SHANGGUAN Y, CHEN E, GONG C, ZHAO Q, JING Y, ZHAO Y, LI Y, CUI L, FAN D, LU Y, WENG Q, WANG Y, ZHAN Q, LIU K, WEI X, AN K, AN G, HAN B. OsSPL13 controls grain size in cultivated rice[J]. Nature Genetics, 2016, 48(4): 447-456. DOI:10.1038/ng.3518 |
| [14] |
SONG X J, HUANG W, SHI M, ZHU M Z, LIN H X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nature Genetics, 2007, 39(5): 623-630. DOI:10.1038/ng2014 |
| [15] |
RUAN B, SHANG L, ZHANG B, HU J, WANG Y, LIN H, ZHANG A, LIU C, PENG Y, ZHU L, REN D, SHEN L, DONG G, ZHANG G, ZENG D, GUO L, QIAN Q, GAO Z. Natural variation in the promoter of TGW2 determines grain width and weight in rice[J]. New Phytologist, 2020, 227(2): 629-640. DOI:10.1111/nph.16540 |
| [16] |
LI Y, FAN C, XING Y, JIANG Y, LUO L, SUN L, SHAO D, XU C, LI X, XIAO J, HE Y, ZHANG Q. Natural variation in GS5 plays an important role in regulating grain size and yield in rice[J]. Nature Genetics, 2011, 43(12): 1266-1269. DOI:10.1038/ng.977 |
| [17] |
WENG J, GU S, WAN X, GAO H, GUO T, SU N, LEI C, ZHANG X, CHENG Z, GUO X, WANG J, JIANG L, ZHAI H, WAN J. Isolation and initial characterization of GW5, a major QTL associated with rice grain width and weight[J]. Cell Research, 2008, 18(12): 1199-1209. DOI:10.1038/cr.2008.307 |
| [18] |
SHI C L, DONG N Q, GUO T, YE W W, SHAN J X, LIN H X. A quantitative trait locus GW6 controls rice grain size and yield through the gibberellin pathway[J]. The Plant Journal, 2020, 103(3): 1174-1188. DOI:10.1111/tpj.14793 |
| [19] |
WANG S, WU K, YUAN Q, LIU X, LIU Z, LIN X, ZENG R, ZHU H, DONG G, QIAN Q, ZHANG G, FU X. Control of grain size, shape and quality by OsSPL16 in rice[J]. Nature Genetics, 2012, 44(8): 950-954. DOI:10.1038/ng.2327 |
| [20] |
XU F, FANG J, OU S J, GAO S P, ZHANG F X, DU L, XIAO Y H, WANG H R, SUN X H, CHU J F, WANG G D, CHU C C. Variations in CYP78A13 coding region influence grain size and yield in rice[J]. Plant Cell & Environment, 2015, 38(4): 800-811. DOI:10.1111/pce.12452 |
| [21] |
CHEN J, GAO H, ZHENG X M, JIN M N, WENG J F, MA J, REN Y L, ZHOU K N, WANG Q, WANG J, WANG J L, ZHANG X, CHENG Z J, WU C Y, WANG H Y, WAN J M. An evolutionarily conserved gene, FUWA, plays a role in determining panicle architecture, grain shape and grain weight in rice[J]. The Plant Journal, 2015, 83(3): 427-438. DOI:10.1111/tpj.12895 |
| [22] |
HUANG K, WANG D K, DUAN P G, ZHANG B L, XU R, LI N, LI Y H. WIDE AND THICK GRAIN 1, which encodes an otubain‐like protease with deubiquitination activity, influences grain size and shape in rice[J]. The Plant Journal, 2017, 91(5): 849-860. DOI:10.1111/tpj.13613 |
| [23] |
管柳蓉, 刘祖培, 徐冉, 段朋根, 张国政, 于海跃, 李静, 罗越华, 李云海. 一个新的OsBRI1弱等位突变体的鉴定及其调控种子大小的功能研究[J]. 植物学报, 2020, 55(3): 279-286. DOI:10.11983/CBB19239 GUAN L R, LIU Z P, XU R, DUAN P G, ZHANG G Z, YU H Y, LI J, LUO Y H, LI Y H. Identification of a new OsBRI1 weak allele and analysis of its function in grain size control[J]. Chin Bull Bot, 2020, 55(3): 279-286. DOI:10.11983/CBB19239 |
| [24] |
LUAN X, KE S W, LIU S C, TANG G J, HUANG D H, WEI M Y, ZHANG Y X, G Qin, ZHANG X Q. OsPEX1, a leucine-rich repeat extensin protein, functions in the regulation of caryopsis development and quality in rice[J]. The Crop Journal, 2022, 10(3): 704-715. DOI:10.1016/j.cj.2021.10.001 |
| [25] |
YAN D W, ZHOU Y, YE S H, ZENG L J, ZHANG X M, HE Z H. BEAK-SHAPED GRAIN 1/TRIANGULAR HULL 1, a DUF640 gene, is associated with grain shape, size and weight in rice[J]. Science China Life Sciences, 2013, 56(3): 275-283. DOI:10.1007/s11427-013-4449-5 |
| [26] |
胡苗, 孙志忠, 孙学武, 谭炎宁, 余东, 刘瑞芬, 袁贵龙, 丁佳, 袁定阳, 段美娟. 利用高密度SNP标记定位水稻粒形相关QTL[J]. 杂交水稻, 2015, 30(5): 54-58. DOI:10.16267/j.cnki.1005-3956.201505019 HU M, SUN Z Z, SUN X W, TAN Y N, YU D, LIU R F, YUAN G L, DING J, YUAN D Y, DUAN M J. Mapping of rice grain shape relevant QTLs using high-density SNP Ms[J]. Hybrid Rice, 2015, 30(5): 54-58. DOI:10.16267/j.cnki.1005-3956.201505019 |
| [27] |
彭强, 叶生鑫, 黄龙, 张大双, 刘颖, 吴健强, 张玉珊, 朱速松. 运用SLAF-seq技术构建水稻高密度遗传图谱[J]. 分子植物育种, 2016, 14(8): 2127-2132. DOI:10.13271/j.mpb.014.002127 PENG Q, YE S X, HUANG L, ZHANG D S, LIU Y, WU J Q, ZHANG Y S, ZHU S S. Construction of a high-density genetic map in rice by using specific-length amplified fragment sequencing (SLAF-seq) technology[J]. Molecular Plant Breeding, 2016, 14(8): 2127-2132. DOI:10.13271/j.mpb.014.002127 |
| [28] |
MCCOUCH S R. Gene nomenclature system for rice[J]. Rice, 2008(1): 72-84. DOI:10.1007/s12284-008-9004-9 |
| [29] |
刘喜, 牟昌铃, 周春雷, 程治军, 江玲, 万建民. 水稻粒型基因克隆和调控机制研究进展[J]. 中国水稻科学, 2018, 32(1): 1-11. DOI:10.16819/j.1001-7216.2018.7016 LIU X, MOU C L, ZHOU C L, CHENG Z J, JIANG L, WAN J M. Research progress on cloning and regulation mechanism of rice grain shape genes[J]. Chinese Journal of Rice Science, 2018, 32(1): 1-11. DOI:10.16819/j.1001-7216.2018.7016 |
| [30] |
闫晓霞, 朱满山, 王丰, 柳武革, 李金华, 霍兴, 黄永相, 刘迪林. 利用高密度遗传图谱定位水稻耐低氧萌发QTL[J]. 广东农业科学, 2023, 50(4): 13-21. DOI:10.16768/j.issn.1004-874X.2023.04.002 YAN X X, ZHU M S, WANG F, L IU W G, LI J H, HUO X, HUANG Y X, LIU D L. Mapping of QTLs for tolerance of hypoxia germination in rice with a high-density genetic map[J]. Guangdong Agricultural Sciences, 2023, 50(4): 13-21. DOI:10.16768/j.issn.1004-874X.2023.04.002 |
(责任编辑 崔建勋)
 2023, Vol. 50
2023, Vol. 50