文章信息
基金项目
- 广东省重点领域研发计划项目(2020B1111030002);中国水产科学研究院南海水产研究所基本科研业务费专项(2021SD02);中国水产科学研究院基本科研业务费专项(2020TD06)
作者简介
- 臧能玮(1998—),男,在读硕士生,研究方向为农业工程与信息技术,E-mail: 1017403920@qq.com.
通讯作者
- 陈丕茂(1969—),男,研究员,研究方向为海洋牧场人工鱼礁,E-mail: chenpm@scsfri.ac.cn.
文章历史
- 收稿日期:2022-09-28
2. 中国水产科学院南海水产科学研究所,广东 广州 510300;
3. 上海海洋大学海洋科学学院,上海 201306
2. South China Sea Fisheries Research Institute, Chinese Academy of Fishery Science, Guangzhou 510300, China;
3. College of Marine Sciences, Shanghai Ocean University, Shanghai 201306, China
【研究意义】海洋微生物是海洋初级生产力的代表,约占全球海洋初级生产力的一半[1],是海洋生态系统中生物要素的重要组成,在物质循环和能量流动中发挥着重要作用。海洋微生物个体或群落变化可以客观地反映水体变化[2],在生态监测实践中广泛应用[3]。海洋微生物还在环境污染的生物修复、全球气候变化调节等方面具有举足轻重的作用。研究珠海外伶仃海洋牧场微生物多样性,对于加强海洋牧场管理和维护具有重要意义。【前人研究进展】环境DNA(environmental DNA, eDNA)是采用非直接接触的方式提取环境中有机体遗留的DNA,即直接从环境样品(土壤、沉积物、水等) 获得遗传物质,是一种有效、无创且易于标准化的取样方法[4]。1990年宏基因组编码(DNA metabarcoding)由Giovannoni等[5]首次提出。1998年Handelsman等[6]提取了湖底沉积微生物的DNA片段。2000年Rondon等[7]提出“eDNA技术”概念,直到2005年Martellini等[8]从水样中提取DNA寻找地表水污染源,开启了该技术运用于水生生态系统的篇章。2008年Ficetola等[9]利用eDNA技术追踪入侵物种美国牛蛙,使eDNA技术首次应用在水生生物的监测上[10]。在2010年前后,有研究者逐渐将eDNA技术引入海洋调查中。结合海水中各种复杂的化学和物理变化过程,构建eDNA研究相关计算模型,将eDNA技术发挥更大的作用。目前,eDNA与DNA技术相结合,已成为生物多样性监测有力的新型工具。近年来,eDNA的应用经历了巨大的发展,已广泛应用于群落生态学、古环境研究、生物监测、生物保护学和入侵生态学等各个领域。珠海外伶仃国家级海洋牧场位于珠江口万山群岛中北部,海域面积9.83 km2,于2018年入选第四批国家级海洋牧场示范区[11]。目前对珠海外伶仃海洋牧场的调查研究主要集中在生物资源种类组成[12]、数量分布[13]、时空变化[14]等方面,而有关微生物研究只涉及到吖啶橙染色直接镜检计数法[15],尚未见到采用eDNA技术进行检测研究的报道。【本研究切入点】为解决珠海外伶仃海洋牧场微生物检测方法的单一性,提高检测的准确性,运用eDNA技术检测珠海外伶仃海洋牧场的微生物多样性,并与传统检测方法进行对比验证,为珠海外伶仃海洋牧场的微生物检测提供一条新的途径。【拟解决的关键问题】基于eDNA技术的检测结果,分析珠海外伶仃海洋牧场的微生物群落组成、生物多样性等特征,以期为珠海外伶仃海洋牧场生态系统管理维护提供参考。
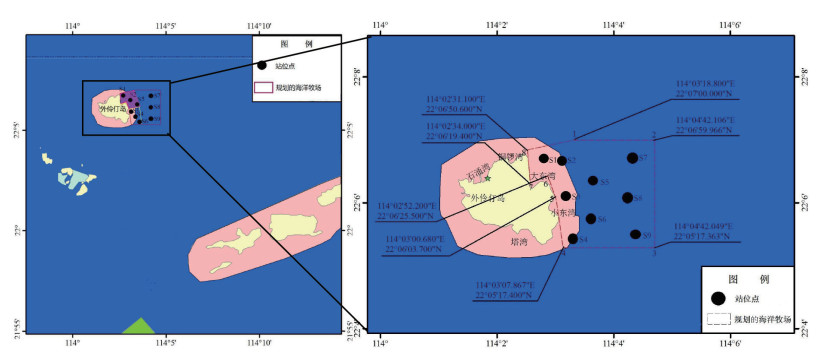
1 材料与方法 1.1 站位点设置为了解微生物多样性,本研究在广东省珠海外伶仃海域国家级海洋牧场示范区设立了S1~S9(22°05'25.107"~22°07'55.272"N、113 °59'50.170"~114°03'35.238"E)9个站位点,站位点位置分布见图 1。
|
| 图 1 9个站位点位置 Fig. 1 Location of 9 stations |
1.2 采样及样品预处理
为防止样品间出现交叉污染,在采样前对采样器皿进行消毒,以10% 次氯酸钠溶液[16-17]浸泡10 min,蒸馏水冲洗3遍,采样人员佩戴手套并及时更换。本研究于2021年9月13日在9个站位点进行样品采集,每个站位点设6个平行试验,共采集54份水样。每份水样采集2 L表层水(在水面以下约2 m处采集)[16-17],用0.45 μm孔径的混合纤维滤膜过滤[18-19],加入5 mL 95% 无水乙醇灭活,将过滤后的滤膜放置于无菌吸水纸,对折,使滤膜干燥,同一站位点的样品袋折叠放入2 mL的离心管,贴上标签纸,最后放入液氮内保存。
1.3 DNA提取DNA提取采用DNA Clean & Concentrator-25试剂盒(购于北京天漠科技开发有限公司),参考Martin-Laurent[20]的方法:洗涤后的样品加入400 μL Cell裂解液, 旋涡振荡1 min,65℃水浴30 min,每5 min振荡1次,冷却到室温。以12 000 r/min、4℃离心10 min,取上清,加入等体积的酚/氯仿/异戊醇(25 ∶ 24 ∶ 1)混合溶液,上下颠倒、混匀;继续以12 000 r/min、4℃离心10 min后取上清,加入等体积的氯仿/异戊醇(24 ∶ 1)混合溶液,上下颠倒、混匀;再以12 000 r/min、4℃离心10 min,取上清于新离心管中,加入0.6倍体积的异丙醇,室温静置1 h后,12 000 r/min、4℃离心10 min,弃上清,用70% 冰乙醇溶液洗涤沉淀,倒置,于无菌干燥沉淀中加入100 μL含20 μg/mL RNaesA的无菌TE buffer溶解DNA沉淀,于30 ℃水浴30 min后取出置于-20℃冰箱保存。
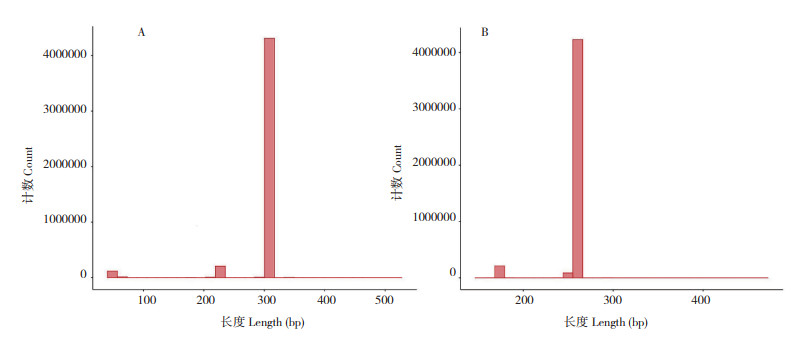
1.4 PCR扩增及高通量测序将提取好的DNA样品寄到生工生物工程(上海)股份有限公司,使用引物MF-F:5'-GTCGGTAAAACTCGTGCCAGC-3' 进行PCR扩增[21],使用Cutadapt软件[22-23]去除Read1 3' 端测序引物接头:AGATCGGAAGAGCACACGTCTG AACTCCAGTCA和Read2 3' 端测序引物接头:AG ATCGGAAGAGCGTCGTGTAGGGAAAGAGTGT,使用Prinseq软件[24-25]切除Reads尾部质量值20以下的碱基,设置10 bp的窗口,若窗口内的平均质量值低于20,则从窗口开始截去后端碱基,过滤质控后的含N序列和短序列,最终过滤掉低复杂度的序列(图 2)。
|
| 图 2 原始序列数据(A)和有效序列数据(B)长度分布 Fig. 2 Length distributions of raw sequence data (A) and valid sequence data (B) |
1.5 数据分析
1.5.1 微生物种类鉴定 序列比对使用QIIME软件中的UCLUST[26],在默认参数中将序列与Silva、NCBI、FGR数据库的模板序列相比对,利用高通量测序技术,将经过处理的序列进行OTU聚类,然后对OTU进行注释,完成物种分类。
1.5.2 多样性指数分析 (1)Chao指数[27]: 估计微生物群落中含OTU数目的指数,计算公式如下:
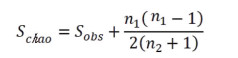
|
式中,Schao是估计的OTU数,Sobs是实际观测到的OTU数,n1是只含有1条序列的OTU数目,n2是只含有2条序列的OTU数目。
(2)Shannon多样性指数[28]: 用于估算样品中微生物多样性指数之一。Shannon值越大,说明群落多样性越高。计算公式如下:

|
式中,Sobs是实际观测到的OTU数,ni是第i个OTU包含的序列数,N是所有个体数目,此处为序列总数。
(3)Simpson指数: 用于估算样品中微生物多样性指数之一,Simpson指数值越大,说明群落多样性越低。计算公式如下:

|
式中,Sobs是实际观测到的OTU数,ni是第i个OTU包含的序列数,N是所有个体数目,此处为序列总数。
(4)物种优势度(Y):表示微生物物种所占的优势程度,计算公式为:
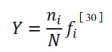
|
式中,ni是第i个OTU包含的序列数,N是所有个体数目,fi为第i个OTU在各采样点出现的频率[31]。本文将Y> 0.02的微生物确定为优势种。
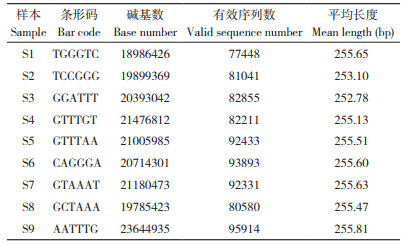
2 结果与分析 2.1 测序所得OTU测序所得有效数据见表 1,9个站位点的样品共产生779 706个OTU,平均长度255 bp,其中S9站位点样品的OTU最多、为95 914个,S1站位点的OTU最少、为77 448个。测序所得有效数据每个样品平均获得96 141条原始序列,经过拼接和质量过滤[32-33],每个样品平均得到86 634条序列,高质量数据占90.1%。
2.2 珠海外伶仃海洋牧场微生物α多样性
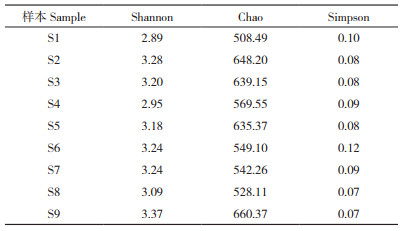
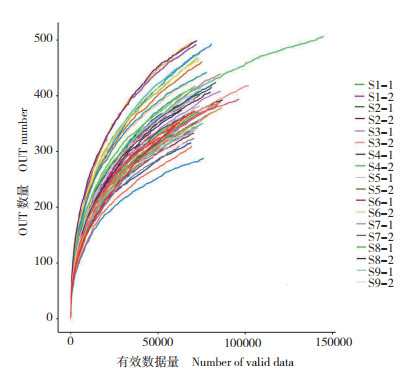
本次高通量测序有效数据与OUT数量关系如图 3,各个样本的稀释性曲线趋于平缓。其中S1-2、S4-1、S8-2等3个位点的稀释曲线趋于平缓前的斜率明显比其他曲线更高,表明3个位点的OTU数目少于其他位点,即这3个位点检测出的物种数量少于其他位点。由表 2可知,S1站位点无论是Shannon指数还是Chao指数均最低、分别为2.89和508.49,S9站位点最高、分别是3.37和660.37,表明S1站位点的微生物多样性最低,S9站位点的微生物多样性最高。
|
| 图 3 α指数稀释性曲线 Fig. 3 α index dilution curve |
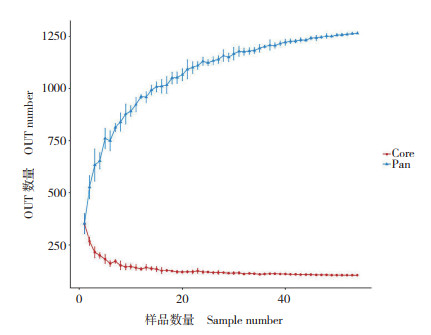
Pan/Core OTU用于描述随着样本量加大物种总量和核心物种量变化的情况。Pan OTU为泛OTU,是所有样本包含的OTU总和;Core OTU为核心OTU,是所有样本共有的OTU数目。Pan/Core曲线反映了持续抽样下新OTU出现的速率。利用物种累积曲线可以判断样品量是否充分,如图 4所示,随着样品量的加大,Pan/Core曲线趋于平缓,表示此环境中的物种并不会随样品量的增加而显著增多,证明本研究样品准备较好,准确性较高[34]。
|
| 图 4 Pan/Core曲线 Fig. 4 Pan/Core curve |
2.3 珠海外伶仃海洋牧场微生物β多样性
通过PCA分析不同样本群落组成可以反映样本间的差异和距离,PCA运用方差分解,将多组数据的差异反映在二维坐标图上,表示物种OTU在主成分上的贡献度,如样本物种组成越相似,反映在PCA图中的距离越近。图 5显示,样本分散程度较大,距离较远,说明各站位点之间的微生物物种差异较大。
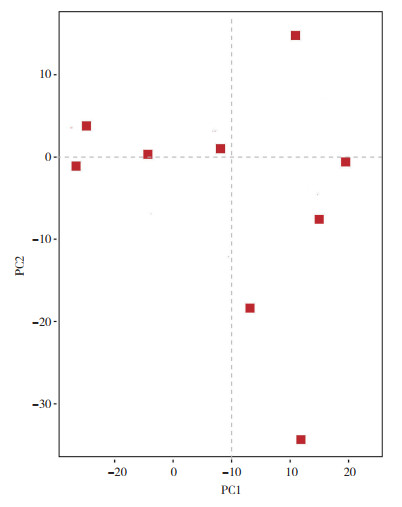
|
| 图 5 各站位点样本的PCA图 Fig. 5 PCA figure of samples from various staions |
2.4 珠海外伶仃海洋牧场微生物物种
利用eDNA技术从珠海外伶仃海洋牧场9个站位点的样本中检测到779 706个OTU,进行注释后得到的微生物物种共30种,(表 3)隶属于5门7纲14目19科26属,分为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、浮霉菌门(Planctomycetes)、厚壁菌门(Firmicutes),其中放线菌门仅有两种微生物分别是放线杆菌(Actinobacterium)和微酸菌(Ilumatobacter),在拟杆菌门中只检测到鞘氨醇杆菌(Sphingobacterium)。
2.5 珠海外伶仃海洋牧场微生物相对丰度
在珠海外伶仃海洋牧场各个站位点中变形菌门至少占61% 以上,远高于其他门,是绝对优势门(图 6)。放线菌门在各站位点分布最少,且在S1、S3、S8中未被检测到,并且各站位点有少量检测到的序列未被标注。
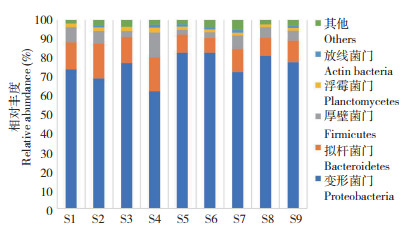
|
| 图 6 各站位点微生物相对丰度 Fig. 6 Relative abundance of microorganisms from various stations |
基于eDNA技术对珠海外伶仃海洋牧场9个站位点的样本检测到的微生物物种分为5门,其中放线菌门最少,只占总检测物种的1.06%,而变形菌门是绝对优势门,占总检测物种的75.06%,其中γ-变形菌纲(Gammaproteobacteria)最多,共5目8科11属17种;其余为α-变形菌纲(Alphaproteobacteria),共4目4科6属6种。此外,拟杆菌门占比11.35%,厚壁菌门占比5.28%,浮霉菌门占比1.91%。优势种共有7种(表 3),包含交替假单胞菌(Pseudoalteromonas)、海杆菌(Marinobacter)、莫拉克氏菌(Moraxella)、嗜冷菌(Psychrobacter)、溶藻弧菌(Vibrio alginolyticus)、嗜盐单胞菌(Halomonas)、鞘氨醇杆菌,其中溶藻弧菌优势度最高(0.191),为绝对优势种,其次是莫拉克氏菌和交替假单胞菌优势度分别达到0.14和0.127,为主要优势种。
3 讨论 3.1 eDNA方法的准确性本研究中每个样品的高质量数据占90.1%。宋伦等[35]研究表明,DNA高质量数据达到90% 以上,则测得OTU数据准确可靠。当各个样本的α指数稀释性曲线趋于平缓,表明测序数据趋于饱和,测序深度已足够反映样品中的所有OTU信息,也就可以认为测序深度已经基本覆盖到样品中所有物种[36]。且Pan/Core曲线趋于平缓,表示此环境中的物种并不会随样品量的增加而显著增多。
深入研究海洋微生物群落组成、分布及其形成机制有助于监测海洋牧场生态系统的动态变化、预测人类活动、理解气候变化等自然现象对珠海外伶仃海洋牧场的影响。eDNA技术与传统形态学方式相比,在微生物调查中更加省时省力、节约成本,并且对环境无损伤,对传统形态学方法监测结果有较高的还原性,并可检测到一些传统方法无法识别的稀有物种和隐匿物种,说明eDNA技术在生物多样性监测中具有重要应用潜力。
3.2 eDNA技术应用于珠海外伶仃海洋牧场微生物检测的不足与建议在珠海外伶仃海洋牧场的微生物多样性研究中,因受疫情影响,本研究只有2021年秋季(9月)的数据,尚未开展其他季度的调查研究,并且采集样品单一,无法较全面地掌握珠海外伶仃海洋牧场微生物群落的空间分布规律和时空变化,为提高数据结果的准确性、了解微生物群落在珠海外伶仃海洋牧场的变化趋势,应长期、定期地开展不同水深的采样研究。
本研究也揭示了eDNA技术的一些不足之处,自然界中有大量未被发现、培养和测序的物种,其基因序列在数据库中不存在,导致数据库信息与自然界实际存在的物种不对等,无法将检测到的所有OTU注释到种水平。同时,受引物的偏好性影响,有些物种无法准确定量地检测出。此外,季节、水温、流速等自然条件变化导致样品成分变化,从而影响物种的检测和相对丰度。因此,eDNA技术仍需要与传统形态学鉴定方法紧密结合,才能构建可靠的多维生物监测系统。
4 结论本研究运用eDNA检测数据和海洋生物数据库,根据微生物特征条形码序列设计扩增引物,检测到珠海外伶仃海洋牧场30种微生物。通过种类组成和优势种的对比分析,确定了变形菌门为绝对优势门,以及交替假单胞菌、海杆菌、莫拉克氏菌、嗜冷菌、溶藻弧菌、嗜盐单胞菌、鞘氨醇杆菌7种优势菌,推进了广东省珠海外伶仃海域国家级海洋牧场示范区海洋生物多样性研究,为后续海洋牧场微生物方面的研究提供基础数据和科学参考。随着eDNA技术的不断完善和发展,该方法不仅会用于大规模的生物监测,更有望融合到海洋牧场生物多样性监测的标准程序中。
| [1] |
王毅波. 渤海、南黄海、西太平洋特定海域微生物群落分布格局及驱动因素研究[D]. 北京: 中国科学院大学(中国科学院烟台海岸带研究所), 2019. WANG Y B. Distribution patterns and driving factors of microbial communities in specific areas of the Bohai Sea, the Southern Yellow Sea and the Western Pacific[D]. Beijing: University of Chinese Academy of Sciences (Yantai Institute of Coastal Zone Research, Chinese Academy of Sciences), 2019. |
| [2] |
韩宏蕾, 施心路, 白昕. 微型生物在水质监测中的作用和意义[J]. 环境科学与管理, 2007, 32(5): 147-150. HAN H L, SHI X L, BAI X. The function and significance of microscopic organisms in water quality surveillance[J]. Environmental Science and Management, 2007, 32(5): 147-150. |
| [3] |
LIU L, LIU M, WILKINSON D M. DNA metabarcoding reveals that 200 μm size fractionated filtering is unable to discriminate between planktonic microbial and large eukaryotes[J]. Molecular Ecology Resources, 2017, 17(5): 991-1002. DOI:10.1111/1755-0998.12652 |
| [4] |
DAVID J L, RACHEL G C, ROBERT H G, MIRANDA M H, JEFF R P, JOHN N K, PETER P, CLARENCE J S, JACK T T, KARI E. D. Cycling of extracellular DNA in the soil environment[J]. Soil Biology and Biochemistry, 2007, 39(12): 2977-2991. DOI:10.1016/j.soilbio.2007.06.020 |
| [5] |
GIOVANNONI S J, OLSEN G J, LANE D J. Microbial ecology and evolution: A ribosomal RNA approach[J]. Annual Review of Microbiology, 1986, 40: 337-365. DOI:10.1146/annurev.mi.40.100186.002005 |
| [6] |
HANDELSMAN J, RONDON M R, BRADY S F, CLARDY J, GOODMAN R M. Molecular biological access to the chemistry of unknown soil microbes: A new frontier for natural products[J]. Chemistry & Biology, 1998, 5(10): 245-249. DOI:10.1016/S1074-5521(98)90108-9 |
| [7] |
RONDON, MICHELLE. Molecular microbial ecology of the soil: Results from an FAO/IAEA co-ordinated research programme, 1992-1996. Gudni Hardarson, William Broughton[J]. The Quarterly Review of Biology, 2000, 75(4): 360-362. DOI:10.1016/S0016-7061(00)00033-1 |
| [8] |
MARTELLINI A, PAYMENT P, VILLEMUR R. Use of eukaryotic mitochondrial DNA to differentiate human, bovine, porcine and ovine sources in fecally contaminated surface water[J]. Water Research, 2005, 39: 541-548. DOI:10.1016/j.watres.2004.11.012 |
| [9] |
FICETOLA G F, MIAUD C, POMPANON F. Species detection using environmental DNA from water samples[J]. Biology Letters, 2008, 4(4): 423-425. DOI:10.1098/rsbl.2008.0118 |
| [10] |
HANFLING B, LAWSON H L, READ D S. Environ-mental DNA metabarcoding of lake fish communities re-flects long-term data from established survey methods[J]. Molecular Ecology, 2016, 25(13): 3101-3119. DOI:10.1111/mec.13660 |
| [11] |
袁华荣, 陈丕茂. 广东省海洋牧场发展现状、问题与对策[J]. 广东农业科学, 2022, 49(7): 141-154. DOI:10.16768/j.issn.1004-874X.2022.07.018 YUAN H R, CHEN P M. Development status, problems and countermeasures of marine ranching in Guangdong Province[J]. Guangdong Agricultural Sciences, 2022, 49(7): 141-154. DOI:10.16768/j.issn.1004-874X.2022.07.018 |
| [12] |
FICETOLA G F, MIAUD C, POMPANON F. Species detection using environmental DNA from water samples[J]. Biology Letters, 2008, 4(4): 423-425. DOI:10.1098/rsbl.2008.0118 |
| [13] |
解欣斐, 龚映雪, 胡晓娟, 杨宇峰. 外伶仃海域龙须菜栽培对水质及浮游细菌数量的影响[J]. 暨南大学学报(自然科学与医学版), 2016, 37(3): 185-192. DOI:10.11778/j.jdxb.2016.03.001 XIE X F, GONG Y X, HU X J, YANG Y F. Effect of Gracilaria lemaneiformis cultivation on water quality and bacterioplankton abundance of Wailingding Island coastal waters[J]. Journal of Jinan University(Natural Science & Medicine Edition), 2016, 37(3): 185-192. DOI:10.11778/j.jdxb.2016.03.001 |
| [14] |
黄晖, 尤丰, 练健生, 张成龙, 杨剑辉, 李秀保, 袁涛, 张浴阳. 珠江口万山群岛海域造礁石珊瑚群落分布与保护[J]. 海洋通报, 2012, 31(2): 189-197. DOI:10.11840/j.issn.1001-6392.2012.2.010 HUANG H, YOU F, LIAN J S, ZHANG C L, YANG J H, LI X B, YUAN T, ZHANG Y Y. Status and conservation strategies of the scleractinian coral community in the Wanshan Islands at Pearl River Estuary[J]. Marine Science Bulletin, 2012, 31(2): 189-197. DOI:10.11840/j.issn.1001-6392.2012.2.010 |
| [15] |
彭鹏飞, 李绪录, 蔡钰灿. 珠江口万山群岛海域秋春季浮游动物的分布特征及其与环境因子的关系[J]. 安徽农业科学, 2015, 43(18): 170-174. DOI:10.13989/j.cnki.0517-6611.2015.18.063 PENG P F, LI X L, CAI Y C. Distributing characteristics of zooplankton and its relationship to environmental factors in wanshan islands sea of the pearl river estuary in fall and spring[J]. Journal of Anhui Agricultural Sciences, 2015, 43(18): 170-174. DOI:10.13989/j.cnki.0517-6611.2015.18.063 |
| [16] |
CAMERON R T, MATTHEW A B, CHARLES C Y X, STUART E J, CHRISTOPHER L J, DAVID M L. Particle size distribution and optimal capture of aqueous macrobial eDNA[J]. Methods in Ecology and Evolution, 2014, 5(7). DOI:10.1111/2041-210X.12206 |
| [17] |
VALENTINI A, TABERLET P, MIAUD C, CIVADE R, HERDER J, THOMSEN P F, BELLEMAIN E, BESNARD A, COISSAC E, BOYER F, GABORIAUD C, JEAN P, POULE N, ROSET N, COPP G H, GENIEZ P, PONT D, ARGILLIER C, BAUDOIN J M, PEROUX T, CRIVELLI A J, OLIVIER A, ACQUEBERGE M, LE B M, MØLLER P R, W E, DEJEAN T. Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding[J]. Molecular Ecology, 2016, 25(4): 929-942. DOI:10.1111/mec.13428 |
| [18] |
BYLEMANS J, FURLAN E M, GLEESON D M, HARDY C M, DUNCAN R P. Does size matter? An experimental evaluation of the relative abundance and decay rates of Aquatic environmental DNA[J]. Environmental Science & Technology, 2018, 52(11): 6408-6416. DOI:10.1021/acs.est.8b01071 |
| [19] |
赵惠娅, 方海霞, 王燕, 刘同军, 林学政. 北极海洋沉积物可培养细菌多样性及抗植物病原真菌活性菌株筛选[J]. 广东农业科学, 2012, 39(17): 149-153. DOI:10.16768/j.issn.1004-874X.2012.17.050 ZHAO H Y, FANG H X, WANG Y, LIU T J, LIN X Z. Diversity of bacteria isolated from marine sediment of Arctic and screening of strains with antifungal activity against plant pathogenic fungi[J]. Guangdong Agricultural Sciences, 2012, 39(17): 149-153. DOI:10.16768/j.issn.1004-874X.2012.17.050 |
| [20] |
MARTIN M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet Journal, 2011, 17(1). |
| [21] |
王家昕, 谭晖, 李长影, 孔雯, 李晓华. 几种土壤微生物总DNA提取方法的比较[J]. 湖北农业科学, 2010, 49(11): 2651-2653. WANG J X, TAN H, LI C Y, KONG W, LI X H. Comparison of several sethods for the extracti on of total community DNA from soil[J]. Hubei Agricultural Sciences, 2010, 49(11): 2651-2653. |
| [22] |
陈家琪, 董丽, 麻晓梅, 田凯, 白洁, 赵彦伟. 基于eDNA技术的白洋淀微型生物群落监测[J]. 农业环境科学学报, 2021, 40(8): 1773-1786. CHEN J Q, DONG L, MA X M, TIAN K, BAI J, ZHAO Y W. Microbial community monitoring in Baiyangdian Lake based on eDNA technology[J]. Journal of Agro-Environment Science, 2021, 40(8): 1773-1786. |
| [23] |
BARNES M A, CHADDERTON W L, JERDE C L, MAHON A R, TURNER C R, LODGE D M. Environmental conditions influence eDNA particle size distribution in aquatic systems[J]. Environmental DNA, 2020, 3(3): 1-11. |
| [24] |
TONY D, ALICE V, ANTOINE D, STÉPHANIE P C, FRANÇOIS P, PIERRE T, CLAUDE M. Persistence of environmental DNA in freshwater ecosystems[J]. PLoS ONE, 2017, 6(8): 23398. DOI:10.1371/journal.pone.0023398 |
| [25] |
KATHERINE M S, ALEXANDER K F, CAREN S G. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms[J]. Biological Conservation, 2015, 183: 85-92. DOI:10.1016/j.biocon.2014.11.038 |
| [26] |
XI W H, HUI M C, WEI S, YI B C, XU X Z. Persistence of mitochondrial DNA markers as fecal indicators in water environments[J]. Science of the Total Environment, 2015, 533: 383-390. DOI:10.1016/j.scitotenv.2015.06.119 |
| [27] |
伍楚妍, 黄晓冰, 刘少君, 李洋, 谢为天, 徐春厚. 海洋源芽孢杆菌的分离鉴定及其消化酶代谢产物的测定[J]. 广东农业科学, 2021, 48(7): 137-144. DOI:10.16768/j.issn.1004-874X.2021.07.017 WU C Y, HUANG X B, LIU S J, LI Y, XIE W T, XU H C. Isolation and identification of marine bacillus and determination of its digestive enzyme metabolitesi[J]. Guangdong Agricultural Sciences, 2021, 48(7): 137-144. DOI:10.16768/j.issn.1004-874X.2021.07.017 |
| [28] |
CORINALDESI C, BEOLCHINI F, DELL ANNO A. Damage and degradation rates of extracellular DNA in marine sediments: Implications for the preservation of gene sequences[J]. Molecular Ecology, 2010, 17(17): 3939-3951. |
| [29] |
苏芯莹, 钟瑜, 李尧, 谭美婷, 黄亚东, 刘珊, 徐向荣, 宋星宇. 珠江口典型海岛周边水域浮游植物分布特征及其影响因素[J]. 热带海洋学报, 2020, 39(5): 30-42. DOI:10.11978/2020008 SU X Y, ZHONG Y, LI Y, TAN M T, HUANG Y D, LIU S, XU X R, SONG X Y. Distribution characteristics and influencing factors of phytoplankton in waters around typical islands in the Pearl River Estuary[J]. Journal of Tropical Oceanography, 2020, 39(5): 30-42. DOI:10.11978/2020008 |
| [30] |
陈锦孝. 基于环境DNA高通量条形码技术的秦岭南北两麓鱼类物种组成及多样性研究[D]. 杨凌: 西北农林科技大学, 2022. DOI: 10.27409/d.cnki.gxbnu.2022.002013. CHEN J X. Fish species composition and diversity in the northern and southern foothills of the Qinling Mountains based on environmental DNA high-throughput barcoding technology[D]. Yangling: Northwest A & F University, 2022. DOI: 10.27409/d.cnki.gxbnu.2022.002013. |
| [31] |
王晨, 陶孟, 李爱民, 施鹏, 杨江华, 王志浩, 张效伟. 基于环境DNA宏条形码技术的秦淮河生物多样性研究[J]. 生态学报, 2022, 42(2): 611-624. DOI:10.5846/stxb202007221920 WANG C, TAO M, LI A M, SHI P, YANG J H, WANG Z H, ZHANG X W. Research on the biodiversity of Qinhuai River based on environmental DNA metabacroding[J]. Acta Ecologica Sinica, 2022, 42(2): 611-624. DOI:10.5846/stxb202007221920 |
| [32] |
莫丹玫. 千岛湖浮游植物群落结构多样性及其鉴定方法的研究[D]. 上海: 上海海洋大学, 2016. MO D M. Community structures of phytoplankton and comparison on them between two identification methods in Lake Qiandaohu[D]. Shanghai: Shanghai Ocean University, 2016. |
| [33] |
MARTIN M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet Journal, 2011, 17(1): 10. DOI:10.14806/ej.17.1.200 |
| [34] |
SCHMIEDER R, EDWARDS R. Quality control and preprocessing of metagenomic datasets[J]. Bioinformatics (Oxford, England), 2011, 27(6): 863-864. DOI:10.1093/bioinformatics/btr026 |
| [35] |
宋伦, 吴景, 宋广军, 董少杰, 王志松. 基于环境DNA技术的辽东湾真核微藻群落结构特征[J]. 生态学报, 2020, 40(17): 6243-6257. DOI:10.5846/stxb201905030893 SONG L, WU J, SING G J, DONG S J, WANG Z S. Characteristics of phytoplankton community structure in Liaodong Bay based on environmental DNA technology[J]. Acta Ecologica Sinica, 2020, 40(17): 6243-6257. DOI:10.5846/stxb201905030893 |
| [36] |
王靖淇. DNA宏条形码技术对不同水环境中微型生物群落结构的研究[D]. 沈阳: 辽宁大学, 2017. WANG J Q. The research on microbial community structure in different water environment through DNA metabarcoding technology[D]. Shenyang: Liaoning University, 2017. |
(责任编辑 陈丽娥)
 2023, Vol. 50
2023, Vol. 50