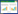文章信息
基金项目
- 广东省重点领域研发计划-重大科技专项(2022B0202110002,2018B020203003);广东省动物分子设计与精准育种重点实验室开放课题(2018A05)
作者简介
- 屈子啸(1998—),男,在读硕士生,研究方向为动物分子遗传育种,E-mail:1031516598@qq.com.
通讯作者
- 唐冬生(1962—),男,博士,教授,研究方向为动物分子遗传育种与基因编辑,E-mail:tangdsh@163.com.
文章历史
- 收稿日期:2023-01-11
【研究意义】我国是生猪养殖和猪肉消费大国,猪肉总产量和人均猪肉消费量多年来一直居于世界首位。经过数十年的选育和实践,以“杜长大”为代表的三元杂交猪(父本为杜洛克猪,母本通常是由大白公猪与长白母猪杂交所产出的二元母猪)展现出生长速度快、饲料转化率和瘦肉率高等诸多优势,逐渐在生猪生产上占据主导地位。影响母猪经济价值的生产性状很多,其中产仔数是重要指标,也是衡量猪场效益的重要因素。传统杂交改良技术及候选基因挖掘在母猪产仔数性状改良中起重要作用,目前国内外已经开展了大量对猪繁殖性状进行基因组遗传评估和鉴定关键QTL或基因的研究,发现问题主要集中在繁殖性状基因组育种值准确性低,受微效多基因影响导致被鉴定的关键QTL效应值小,造成繁殖性状遗传改良速度慢[1-2]。此外,传统QTL遗传图谱和QTL鉴定分析存在耗时长、成本高等不足,利用创新技术进一步挖掘种用母猪的遗传改良潜力具有重要的研究意义。
【前人研究进展】母猪产仔数性状的影响因素包括遗传、品种、胎次、分娩季节和营养水平等[3-4],其中遗传因素占据重要地位。目前国内外育种工作者已在母猪产仔数性状选育方面取得较多进展,特别是随着分子生物学技术的进步,分子辅助选育技术在母猪产仔数性状选育方面发挥了重要作用,相继发现了多个潜在控制该性状的候选基因。例如,赵要风等[5]发现促卵泡素基因(FSHβ)与母猪产仔数密切相关,经PCR扩增检测发现存在AA、AB、BB共3种基因型。柳淑芳等[6]在莱芜黑猪群体中通过对FSHβ基因多态性与产仔数进行关联分析,结果表明,AA基因型为优势基因型,且与高产仔数连锁显著,确定该基因为猪产仔数的主效基因。此外,ESR[7]、RBP4、PRLR[8-9]和DIS3L2[10]等基因也与母猪产仔数性状存在关联。这些母猪产仔数性状候选基因有效推进了母猪遗传选育的工作进程,提高了经济产出。但传统性状候选基因的挖掘耗时长、花费大、效率低,已严重阻碍母猪产仔数性状的进一步提升,亟需利用新技术快速高效地挖掘候选基因以进一步提高育种效率。
【本研究切入点】全基因组重测序是对已知参考序列的物种进行不同个体间的基因组测序,并在此基础上对个体或群体进行差异性分析,通过全基因组重测序以及序列比对,实现遗传进化分析和重要性状候选基因预测[11-12]。随着测序成本降低和拥有参考基因组序列物种的增多,全基因组重测序已成为生猪遗传选育研究的有效方法[13]。【拟解决的关键问题】本研究采用全基因组重测序,对极端高产与极端低产的两组大白-长白(大长)二元母猪进行比较分析,在高选择信号区域内筛选候选基因,以期为探索高产仔数种猪的选育奠定基础。
1 材料与方法 1.1 试验材料2021年6月从湖南省张家界康茜生态农业公司三岔湾生猪扩繁基地获取124头大长二元母猪的初次产仔数据。选取极端高产与极端低产母猪各10头,分别使用耳标钳采集约100 mg耳组织样本,样本保存于装有95% 酒精的EP管中,采用干冰运输带回实验室,保存在-20 ℃冰箱中备用。
1.2 试验方法1.2.1 样品基因组DNA提取与重测序 采用组织基因组DNA提取试剂盒(TIAN GEN DP304)对高产与低产母猪的耳组织样本提取基因组DNA,并委托北京诺禾致源公司进行文库构建,并基于Illumina PE150测序平台完成全基因组重测序。
对20个样本数据进行统计,包括测序数据产量、测序错误率、Q20含量、Q30含量、GC含量等。质控后的高质量数据通过BWA软件[11]比对到参考基因组(参数:mem-t 4-k 32-M),比对结果经SAMTOOLS软件[12]去除重复(参数:rmdup)。
1.2.2 SNP检测与注释 采用SAMTOOLS等软件进行群体SNP检测。利用贝叶斯模型检测群体中的多态性位点,通过过滤和筛选得到高质量的SNP,然后使用ANNOVAR软件[13]对上述过滤后的SNP进行注释。
1.2.3 选择消除分析及候选基因筛选 使用VCFtools(V0.1.16)软件计算群体分化指数(Fst)以及群体多态性(θπ)。将Fst与θπ信号区域进行交集以确定较强的选择信号,获得候选信号窗口并根据参考基因组信息进行基因注释。其中,0 ≤ Fst ≤ 1,Fst越接近1表示亚种群间的种群分化越明显;θπ分析按照100 kb为1个窗口,在基因组上滑动并对滑动窗口中群体遗传信息的差异进行分析。以Fst、θπ的Top 5%作为信号较强的区域,对此区域进行候选基因筛选。
1.2.4 GO分析与KEGG分析 利用DAVID在线数据库(https://david.ncifcrf.gov/)对筛选出的区域进行GO/KEGG富集分析,根据富集结果过滤筛选高产仔数候选基因。
2 结果与分析 2.1 样本选择与数据统计本研究共采集124头大长二元母猪的初次产仔数据,并选择极端高产和极端低产母猪各10头作比较,使用SPSS 7.16的T检验计算两组数据的差异显著性,结果(表 1)显示,两组间的总产仔数差异显著(P<0.05),健仔数差异极显著(P<0.01)。
2.2 测序数据统计及质控结果
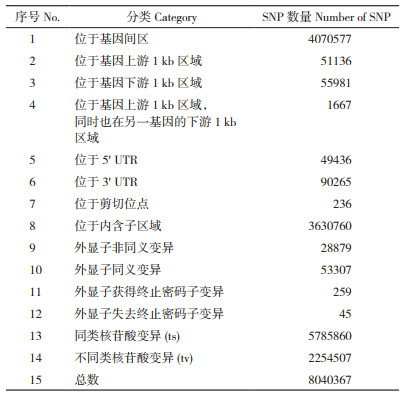
本研究基于全基因组重测序技术对20头高产母猪和低产母猪进行分析,获得高质量数据1 551 069 Gb,测序质量较高(Q20 ≥95%,Q30≥90%),GC比例为43.19%~44.14%,GC分布正常,且参考基因的比对率平均大于98%(表 2),表明建库测序成功。
|
2.3 SNP检测与注释结果
采用SAMTOOLS等软件对供试的两组猪进行SNP检测。利用贝叶斯模型检测群体中的多态性位点,通过Dp4-miss0.3-maf0.05过滤和筛选得到高质量的SNP,并使用ANNOVA软件对样本中的高质量SNP进行注释。由表 3可知,在两组样本中共发现8 040 367个SNP,50.6%的SNP位于基因间区域,45.2%的SNP位于内含子区域,在外显子区域、非编码区3' 端、5' 端的SNP分别仅占1.0%、1.1%、0.6%。其中位于基因组外显子区域的SNP中,有53 307个为同义变异,占比64.6%;有28 879个为非同义变异,占比35.0%;此外,还有304个SNP出现在终止密码子区域。
2.4 选择消除分析及差异基因筛选
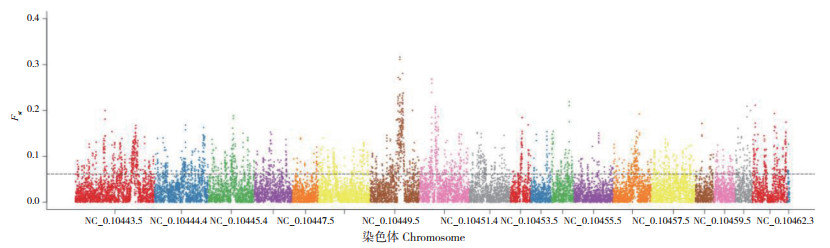
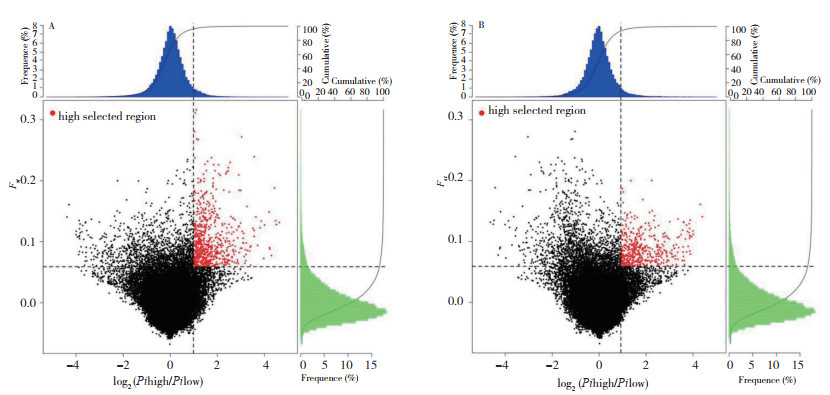
通过R语言软件包计算每个SNP位点的Fst值,以100 kb为窗口滑动计算Fst总值,除以每个窗口的SNP数,得到平均Fst值(图 1);采用滑动窗口法对每个群体分别计算θπ值(图 2),以及群体间的θπ比率。以Fst的Top5%和θπ比率的Top5%作为选择阈值,得到较强的选择信号(图 3),初步选择差异基因。根据来自于两者交集的选择信号窗口所对应的参考基因组位置信息,提取位于外显子区、内含子区、非编码区3' 端和5' 端的SNP位点进行基因注释,共筛选到两组样本间的差异基因1 136个。
|
| 虚线代表选择阈值(Fst Top 5%) The dashed line represents the selection threshold (Fst Top 5%) 图 1 群体分化Fst分布 Fig. 1 Population differentiation distribution map of Fst |
|
| 图 2 高产仔数(A)与低产仔数(B)母猪群体θπ分析 Fig. 2 Analysis of θπ between high litter size group and low litter size group of sow population |
|
|
A:低产仔数母猪群体的信号选择区域;B:高产仔数母猪群体的信号选择区域 点图横坐标为θπ比率,纵坐标为Fst值,分别对应上面的频率分布图和右侧的频率分布图;蓝色和绿色区域为θπ比率的Top 5% 区域,红色区域为Fst的Top 5% 区域 A: Signal selection regions of low litter size sow population; B: Signal selection regions of high litter size sow population The abscissa of the point graph is θπ ratio, with the vertical axis representing the Fst value, corresponding to the frequency distribution map above and the frequency distribution map on the right. The blue and green regions are the Top 5% region of the θπ ratio, and the red region represents the Top 5% region of Fst 图 3 Fst、θπ选择消除分析结果 Fig. 3 Selection elimination analysis results of Fst and θπ |
2.5 GO条目与KEGG通路富集分析
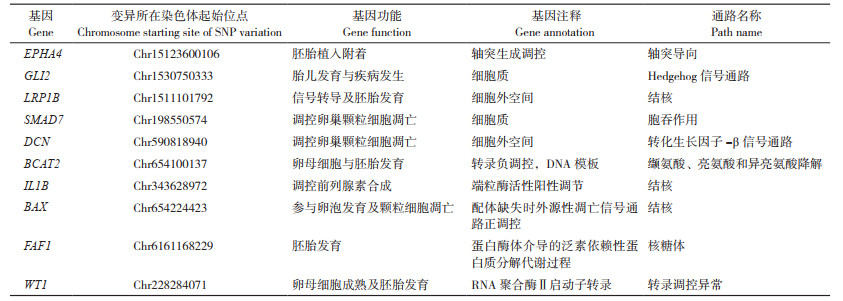
对高选择信号区域内的基因进行GO分析,图 4展示了P值最低的前20条GO条目,其中DNA物质合成过程正向调控、磷酸化调控和下丘脑发育的P值分别为0.00174、0.00274和0.00312,可能与二元母猪的繁殖性能相关,这些条目所富集到的基因包括EPHA4、SMAD7、GLI2、WT1、LRP1B、BAX等,其与猪繁殖性能和总产仔数性状的相关注释见表 4。

|
| 图 4 差异基因的GO富集条目(Top20) Fig. 4 GO enrichment entries of differential genes (Top20) |
|
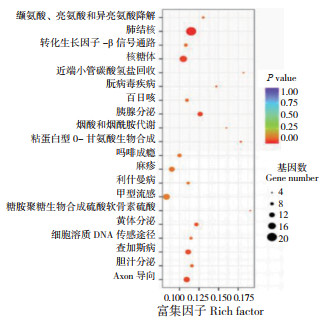
对高选择信号区域内的基因进行KEGG分析,图 5展示了P值最低的前20条KEGG通路,在显著富集的通路中筛选出SMAD7、DCN、BCAT2、IL1B、FAF1等基因,其具有参与生殖激素调控、生成、转运以及胚胎发育和卵细胞凋亡等生理功能,可能与二元母猪的繁殖性能相关,其与猪繁殖性能和总产仔数性状的相关注释见表 4。
|
| 图 5 差异基因的KEGG富集通路(Top 20) Fig. 5 KEGG enrichment pathways of differential genes (Top 20) |
3 讨论
本研究采用全基因组重测序以及GO分析初步筛选到EPHA4、SMAD7、GLI2、WT1、LRP1B、BAX等基因与二元母猪的繁殖性能和总产仔数性状密切相关,KEGG富集通路分析结果显示SMAD7、DCN、BCAT2、1L1B、FAF1等基因与各类生殖激素的调控、生成、转运以及胚胎发育和卵细胞凋亡等各项生理活动密切相关,提示可能为产仔数性状的候选基因。
胚胎植入是建立妊娠的重要环节,猪胚胎在妊娠的第13~14天开始附着于子宫,在第18~24天完成附着,已有研究报道Eph-Ephrin系统在人和小鼠的胚胎植入中发挥关键作用[14]。EPHA4基因是Eph-Ephrin家族的重要成员,负责编码多效细胞因子[15]。在有关猪产仔数性状的研究中,Fu等[16]对长白、杜洛克、大白猪种群进行EPHA4基因多态性与总产仔数和出生活仔数进行关联分析,结果表明EPHA4基因与总产仔数显著相关,AGTC、AGCC和GGCC为优势基因型。本研究的发现与该研究结论相吻合,提示EPHA4基因在猪产仔数性状方面存在潜在作用。
TGF-β通路对雌性哺乳动物主要起调节卵巢功能和卵泡发育的作用,是影响类固醇合成、黄体生成和排卵的重要通路[17]。SMAD7在哺乳动物卵巢颗粒细胞凋亡中起关键作用,其能够拮抗和微调转化生长因子b(TGFb)信号[18]。此外,SMAD7与一些微RNA(miRNA)通过中断TGFBR1和TGF-β信号通路来增强猪卵巢颗粒细胞凋亡,进而影响其繁殖生育,提示其对猪产仔数性状具有潜在作用[19-20]。
GLI2基因位于猪15号染色体(NC_010457.5),本研究发现在该基因座存在大量SNP变异。前人研究表明猪的第15号染色体存在肛门闭锁的易感基因,GLI2对远端结肠正常发育至关重要[21]。此外,GLI2是Shh(Sonic hedgehog)信号通路的响应转录因子,Shh通路在胚胎模式、细胞增殖和细胞分化中发挥重要作用[22],由此推测先天性肛门闭锁可引发胎儿死亡,从而导致产仔数降低。
LRP1B基因编码低密度脂蛋白受体相关蛋白1B,介导细胞胆固醇摄取[23]。胆固醇是一种在胎儿发育过程中起重要作用的脂质。Dietrich等[24]研究发现,小鼠LRP1B的敲除会导致早期胚胎致死性;Wang等[25]发现LRP1B与仔猪的出生体重显著相关,而仔猪的出生体重与子宫容积在很大程度上影响总产仔数。由此推测LRP1B可能在猪产仔数性状方面存在潜在作用。
卵泡发育离不开颗粒细胞参与,颗粒细胞主要参与卵泡的物质交换、信号传导等功能。99%以上的卵泡在发育和生长过程中发生“闭锁”,而滤泡闭锁主要受颗粒细胞凋亡的调控[20]。Sai等[26]证明了猪卵巢颗粒细胞是一种线粒体依赖的Ⅱ型凋亡细胞,促凋亡蛋白BAX在细胞内信号转导中起重要作用,并参与颗粒细胞凋亡,后者是猪卵巢卵泡发育的一个关键因素。多项研究指明了BAX在猪卵巢卵泡发育中的重要性,并将其作为卵巢颗粒细胞的凋亡标志物[27-28],表明BAX与母猪产仔数性状存在关联。
多项研究指出,线粒体DNA(mtDNA)的稳定性对于卵母细胞成熟和胚胎发育具有重要意义[29-30]。对携带mtDNA与缺陷mtDNA的猪卵母细胞进行线粒体基因差异表达分析,结果发现其中关键基因支链氨基酸转氨酶2(BCAT2)存在显著差异[31]。BCAT2在氨基酸代谢上发挥重要作用,提示其与mtDNA稳态存在关联,并可能影响卵母细胞质量以及产仔数性状。
前列腺素是参与母猪黄体溶解和妊娠识别的重要激素之一。Seo等[32]通过测定母猪子宫内前列腺素关键受体的表达水平变化,确定了IL1B是调控前列腺素合成的关键酶。另有研究表明,IL1B通过调节多形核嗜中性粒细胞的迁移,可加速牛输卵管上皮细胞的炎性细胞因子产生,导致慢性输卵管炎症以及生育能力减退[33]。由此可见,IL1B也可间接影响母猪的产仔数性状。
Fas为肿瘤坏死因子受体家族的成员,Fas配体(TNFSF6)与Fas抗原(TNFRSF6)的相互作用介导许多器官系统的程序性细胞死亡,Fas相关因子1(FAF1)参与细胞凋亡过程[34-35]。已有研究发现核苷酸结合寡聚化结构域样受体蛋白2(NLRP2)是卵子发生过程中转录的重要母体效应基因,而FAF1蛋白可与NLRP2蛋白共同形成FAF1-NLRP2蛋白复合物,对卵母细胞功能和胚胎植入前发挥重要作用[36]。此外,敲低小鼠NLRP2或FAF1都将导致小鼠胚胎发育异常甚至停滞[35, 37]。可见,FAF1也可能影响猪产仔数性状,与本研究的发现相吻合。
虽然本研究通过全基因组重测序分析初步挖掘了与大长二元母猪产仔数性状密切相关的基因,但鉴于样本量偏少,后续我们将进一步扩大样本量,并在更大的群体中进行基因功能鉴定。由于繁殖性状的复杂性以及微效多基因调控等因素,有必要开展更加全面的信号分析,后续研究可考虑加入CNV或Indel选择信号交集,使其数据更加全面。
4 结论本研究发现大长二元母猪在产仔数性状上经历了显著的人工选择,通过全基因组重测序分析初步挖掘出与大长二元母猪产仔数性状密切相关的基因,包括EPHA4、SMAD7、GLI2、LRP1B、WT1、BAX、BCAT2、IL1B、FAF1等,这些基因功能主要涉及胚胎发育、细胞凋亡等,在猪繁殖性能相关的生理活动中发挥作用,且调控主要发生在母猪卵细胞成熟凋亡时期及妊娠初期,可能是决定大长二元母猪产仔数性状的候选基因,其作用机制还有待进一步通过基因功能分析来确定。
| [1] |
傅衍. 猪的育种技术及其应用[J]. 猪业科学, 2021, 38(12): 39-41. FU Y. Pig breeding technology and its application[J]. Swine Industy Science, 2021, 38(12): 39-41. |
| [2] |
任慧波, 朱吉, 崔清明, 邓缘, 刘莹莹, 胡雄贵, 李华丽, 陈晨. 猪育种技术研究进展[J]. 养猪, 2020, 171(4): 61-64. DOI: 10.13257/j.cnki.21-1104/s.2020.04.023. REN H B, ZHU J, CUI Q M, DENG Y, HU X G, LI H L, CHEN C. Research progress in pig breeding technology[J]. Swine Production, 2020, 171(4): 61-64. DOI: 10.13257/j.cnki.21-1104/s.2020.04.023. |
| [3] |
刘伟明, 朱泳斯, 张健. 猪产仔数候选基因研究进展[J]. 湖南畜牧兽医, 2021(2): 48-50. DOI:10.3969/j.issn.1006-4907.2021.02.019 LIU W M, ZHU Y S, ZHANG J. Research progress on candidate genes of pig litter size[J]. Hunan Journal of Animal Science & Veterinary Medicine, 2021(2): 48-50. DOI:10.3969/j.issn.1006-4907.2021.02.019 |
| [4] |
王丽, 叶翔杨, 温晓鹿, 等. 猪营养调控技术研究进展[J]. 广东农业科学, 2020, 47(11): 114-124. DOI: 10.16768/j.issn.1004-874X.2020.11.013. WANG L, YE X Y, WEN X L, YANG X F, GAO K G, JIANG Z Y. Research progress in pig nutrition regulation technology[J]. Guangdong Agricultural Sciences, 2020, 47(11): 114-124. DOI: 10.16768/j.issn.1004-874X.2020.11.013. |
| [5] |
赵要风, 李宁, 肖璐, 曹更生, 陈怡真, 张顺, 陈永福, 吴常信, 张建生, 孙士铨, 徐学清. 猪FSHβ亚基基因结构区逆转座子插入突变及其与猪产仔数关系的研究[J]. 中国科学: C辑, 1999, 29(1): 81-86. ZHAO Y F, LI N, XIAO L, CAO G S, CHEN Y Z, ZHANG S, CHEN Y F, WU C X, ZHANG J S, SUN S Q, XU X Q. Pig FSH β study on the reverse transposon insertion mutation in the structural region of Pig FSHβ subunit gene and its relationship with the litter size of pigs[J]. Science in China (Series C), 1999, 29(1): 81-86. |
| [6] |
柳淑芳, 闫艳春, 杜立新. 莱芜黑猪FSHβ亚基基因的多态性分析[J]. 山东农业大学学报: 自然科学版, 2002, 33(4): 403-408. DOI:10.3969/j.issn.1000-2324.2002.04.002 LIU S F, YAN Y C, DU L X. Analysis on isomorphism at FSHβ subunit gene in LAIWU pigs[J]. Journal of Shandong Agricultural University (Natural Science Edition), 2002, 33(4): 403-408. DOI:10.3969/j.issn.1000-2324.2002.04.002 |
| [7] |
刘乙, 胡九英, 赵玉强, 汪德明, 刘小军, 黄涛. ESR和FSH-β基因多态性对大白猪繁殖性能的影响[J]. 中国畜牧杂志, 2017, 53(4): 44-48. DOI:10.19556/j.0258-7033.2017-04-044 LIU Y, HU J Y, ZHAO Y Q, WANG D M, LIU X J, HUANG T. ESR and FSH-β effect of gene polymorphism on reproductive performance of large white pigs[J]. Chinese Journal of Animal Science, 2017, 53(4): 44-48. DOI:10.19556/j.0258-7033.2017-04-044 |
| [8] |
孙延晓, 曾勇庆, 唐辉, 樊新忠, 陈其美, 李华, 钱源, 宋一萍. 猪PRLR和RBP4基因多态性与产仔数性能的关系[J]. 遗传, 2009, 31(1): 63-68. DOI:10.3724/SP.J.1005.2009.00063 SUN Y X, ZENG Y Q, TANG H, FAN X Z, CHEN Q M, LI H, QIAN Y, SONG Y P. The relationship between PRLR and RBP4 gene polymorphisms and litter performance in pigs[J]. Hereditas (Beijing), 2009, 31(1): 63-68. DOI:10.3724/SP.J.1005.2009.00063 |
| [9] |
刘璐, 梁永晔, 吴正常, 苏先敏, 郑先瑞, 王瑾, 吴圣龙, 包文斌. 大白猪、长白猪和杜洛克猪RBP4基因多态性及其与繁殖性能的相关分析[J]. 中国畜牧兽医, 2012, 39(10): 169-172. DOI:10.3969/j.issn.1671-7236.2012.10.040 LIU L, LIANG Y Y, WU Z C, SU X M, ZHENG X R, WANG J, WU S L, BAO W B. Association analysis on polymorphism of the RBP4 gene with reproductive performance in Yorkshire, Landrace and Duroc pigs[J]. Chinese Animal Husbandry & Veterinary Medicine, 2012, 39(10): 169-172. DOI:10.3969/j.issn.1671-7236.2012.10.040 |
| [10] |
苏先敏, 赵乔辉, 朱世平, 霍永久, 梁永晔, 包文斌, 吴圣龙. 大白猪、长白猪和杜洛克猪催乳素受体基因多态性及其与繁殖性能的关系[J]. 中国畜牧兽医, 2013, 40(8): 146-150. SU X M, ZHAO Q H, ZHU S P, HUO Y Y, LIANG Y Y, BAO W B, WU S L. The polymorphism of prolactin receptor gene and its relationship with reproductive performance in large white pigs, landrace pigs and Duroc pigs[J]. China Animal Husbandry & Veterinary Medicine, 2013, 40(8): 146-150. |
| [11] |
LI H, DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754-1760. DOI:10.1093/bioinformatics/btp324 |
| [12] |
LI H, HANDSAKER B, WYSOKER A. The Sequence Alignment/Map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078-2079. DOI:10.1093/bioinformatics/btp352 |
| [13] |
WANG K, LI M, HAKONARSON H. Annovar: functional annotation of genetic variants from high-throughput sequencing data[J]. Nucleic Acids Research, 2010, 38(16): E164. DOI:10.1093/NAR/GKQ603 |
| [14] |
FUJⅡ H, TATSUMI K, KOSAKA K, YOSHIOKA S, FUJIWARA H, FUJⅡ S. Eph-ephrin a system regulates murine blastocyst attachment and spreading[J]. Developmental Dynamics, 2006, 235(12): 3250-3258. DOI:10.1002/dvdy.20977 |
| [15] |
ZHAO J, STEVENS C H, BOYD A W, OOI L, BARTLETT PF. Role of EphA4 in mediating motor neuron death in MND[J]. International Journal of Molecular Sciences, 2021, 22(17): 9430. DOI:10.3390/ijms22179430 |
| [16] |
FU Y, FU J, WANG A. Association of EphA4 polymorphism with swine reproductive traits and mRNA expression of EphA4 during embryo implantation[J]. Molecular Biology Reports, 2012, 39(3): 2689-2696. DOI:10.1007/s11033-011-1023-8 |
| [17] |
DU X, WANG L, LI Q, WU W, SHANG P, CHAMBA Y, PAN Z, LI Q. miR-130a/TGF-β1 axis is involved in sow fertility by controlling granulosa cell apoptosis[J]. Theriogenology, 2020, 157: 407-417. DOI:10.1016/j.theriogenology.2020.08.015 |
| [18] |
LI Q. Inhibitory SMADs: potential regulators of ovarian function[J]. Biology of Reproduction, 2015, 92(2): 50. DOI:10.1095/biolreprod.114.125203 |
| [19] |
ZHANG X, CHEN Y, YANG M, SHANG J, XU Y, ZHANG L, WU X, DING Y, LIU Y, CHU M, YIN Z. MiR-21-5p actions at the Smad7 gene during pig ovarian granulosa cell apoptosis[J]. Animal Reproduction Science, 2020, 223: 106645. DOI:10.1016/j.anireprosci.2020.106645 |
| [20] |
YAO W, PAN Z, DU X, ZHANG J, LI Q. miR-181b-induced SMAD7 downregulation controls granulosa cell apoptosis through TGF-β signaling by interacting with the TGFBR1 promoter[J]. Journal of Cellular Physiology, 2018, 233(9): 6807-6821. DOI:10.1002/jcp.26431 |
| [21] |
CASSINI P, MONTIRONI A, BOTTI S, HORI T, OKHAWA H, STELLA A, ANDERSSON L, GIUFFRA E. Genetic analysis of anal atresia in pigs: evidence for segregation at two main loci[J]. Mammalian Genome, 2005, 16(3): 164-170. DOI:10.1007/s00335-004-3024-6 |
| [22] |
BAI C B, AUERBACH W, LEE J S, STEPHEN D, JOYNER A L. Gli2, but not Gli1, is required for initial Shh signaling and ecTopic activation of the Shh pathway[J]. Development, 2002, 129(20): 4753-4761. DOI:10.1242/dev.129.20.4753 |
| [23] |
HAAS J, BEER A G, WIDSCHWENDTER P, OBERDANNER J, SALZMANN K, SARG B, LINDNER H, HERZ J, PATSCH J R, MARSCHANG P. LRP1b shows restricted expression in human tissues and binds to several extracellular ligands, including fibrinogen and apoE-carrying lipoproteins[J]. Atherosclerosis, 2011, 216(2): 342-347. DOI:10.1016/j.atherosclerosis.2011.02.030 |
| [24] |
DIETRICH M F, VAN DER WEYDEN L, PROSSER H M, BRADLEY A, HERZ J, ADAMS D J. Ectodomains of the LDL receptor-related proteins LRP1b and LRP4 have anchorage independent functions in vivo[J]. PLoS One, 2010, 5(4): e9960. DOI:10.1371/journal.pone.0009960 |
| [25] |
WANG X, LIU X, DENG D, YU M, LI X. Genetic determinants of pig birth weight variability[J]. BMC Genetics, 2016, 17(Sl1): 15. DOI:10.1186/s12863-015-0309-6 |
| [26] |
SAI T, GOTO Y, YOSHIOKA R, MAEDA A, MATSUDA F, SUGIMOTO M, WONGPANIT K, JIN H Z, LI J Y, MANABE N. Bid and Bax are involved in granulosa cell apoptosis during follicular atresia in porcine ovaries[J]. Journal of Reproduction and Development, 2011, 57(3): 421-427. DOI:10.1262/jrd.11-007h |
| [27] |
FABOVÁ Z, SIROTKIN A V. Interrelationships between kisspeptin and F SH in control of porcine ovar ian cell functions[J]. Domestic Animal Endocrinology, 2021, 74: 106520. DOI:10.1016/j.domaniend.2020.106520 |
| [28] |
VINING L M, ZAK L J, HARVEY S C, HARVEY K E. The role of apoptosis in cryopreserved animal oocytes and embryos[J]. Theriogenology, 2021, 173: 93-101. DOI:10.1016/j.theriogenology.2021.07.017 |
| [29] |
CAGNONE G L, TSAI T S, MAKANJI Y, MATTHEWS P, GOULD J, BONKOWSKI M S, ELGASS K D, WONG A S, WU L E, MCKENZIE M, SINCLAIR D A, ST JOHN J C. Restoration of normal embryogenesis by mitochondrial supplementation in pig oocytes exhibiting mitochondrial DNA deficiency[J]. Scientific Reports, 2016, 6: 23229. DOI:10.1038/srep23229 |
| [30] |
KIRILLOVA A, SMITZ J E J, SUKHIKH G T, MAZUNIN I. The role of mitochondria in oocyte maturation[J]. Cells, 2021, 10(9): 2484. DOI:10.3390/cells10092484 |
| [31] |
TSAI T S, TYAGI S, ST JOHN J C. The molecular characterisation of mitochondrial DNA deficient oocytes using a pig model[J]. Human Reproduction, 2018, 33(5): 942-953. DOI:10.1093/humrep/dey052 |
| [32] |
SEO H, CHOI Y, SHIM J, CHOI Y, KA H. Regulatory mechanism for expression of IL1B receptors in the uterine endometrium and effects of IL1B on prostaglandin synthetic enzymes during the implantation period in pigs[J]. Biology of Reproduction, 2012, 87(2): 31. DOI:10.1095/biolreprod.112.099051 |
| [33] |
NAKAMURA Y, AIHARA R, IWATA H, KUWAYAMA T, SHIRASUNA K. IL1B triggers inflammatory cytokine production in bovine oviduct epithelial cells and induces neutrophil accumulation via CCL2[J]. American Journal of Reproductive Immunology, 2021, 85(5): e13365. DOI:10.1111/aji.13365 |
| [34] |
MENGES C W, ALTOMARE D A, TESTA J R. FAS-associated factor 1(FAF1): diverse functions and implications for oncogenesis[J]. Cell Cycle, 2009, 8(16): 2528-34. DOI:10.4161/cc.8.16.9280 |
| [35] |
PENG H, HUO J, GAO Y, CHEN J, YU X, XIAO T. Fas-associated protein factor 1 is involved in meiotic resumption in mouse oocytes[J]. Journal of Reproduction and Development, 2018, 64(2): 173-177. DOI:10.1262/jrd.2017-081 |
| [36] |
OHSUGI M, ZHENG P, BAIBAKOV B, LI L, DEAN J. Maternally derived FILIA-MATER complex localizes asymmetrically in cleavage-stage mouse embryos[J]. Development, 2008, 135(2): 259-269. DOI:10.1242/dev.011445 |
| [37] |
PENG H, LIU H, LIU F, GAO Y, CHEN J, HUO J, HAN J, XIAO T, ZHANG W. NLRP2 and FAF1 deficiency blocks early embryogenesis in the mouse[J]. Reproduction, 2017, 154(3): 245-251. DOI:10.1530/REP-16-0629 |
(责任编辑 崔建勋)
 2023, Vol. 50
2023, Vol. 50