文章信息
基金项目
- 广州市科技计划项目(202102010414);广东省乡村振兴战略专项资金种业振兴项目(2022-NPY-03-001)
作者简介
- 黄依琳(1998—),女,在读硕士生,研究方向为蔬菜分子遗传,E-mail:375906220@qq.com.
通讯作者
- 夏岩石(1976—),男,博士,副教授,研究方向为分子遗传育种,E-mail:xiayanshi922@163.com.
文章历史
- 收稿日期:2023-02-23
2. 广州市农业科学研究院,广东 广州 510308
2. Guangzhou Academy of Agricultural Sciences, Guangzhou 510308, China
【研究意义】菜心(Brassica campestris L. ssp. chinensis var. utilis Tsen et Lee)是十字花科芸薹属白菜亚种中以花薹为产品的一个变种,营养价值高,风味独特,已经成为华南地区人们日常饮食中必不可少的蔬菜种类[1]。随着消费者对其品质要求的提高,选育性状优良的菜心品种已成为当前主要的研究方向。通过分析候选基因的自然变异对菜心生长发育的影响,挖掘其优异等位基因,可为菜心分子辅助育种提供理论参考,加快优良菜心品种的选育。【前人研究进展】Auxin Up-regulated F-box protein(AUF)基因是一类生长素上调的F-box蛋白基因,在陆生植物中广泛分布,包含AUF1和AUF2两个同源基因,研究显示其通过调控生长素与细胞分裂素在根的分布而促进拟南芥根系的延长[2]。F-box蛋白家族是一类含有F-box基序(motif),在泛素介导的蛋白质水解过程中具有底物识别特性的蛋白质,参与植物的生长发育过程和激素的信号转导途径,也在植物的逆境和防御反应中发挥重要功能[3]。Rameneni等[4]在大白菜的蛋白数据库中鉴定出571个F-box蛋白基因,其中69个基因在非生物胁迫条件(寒冷、干旱和盐胁迫)下差异表达,446个基因在特定组织(愈伤组织、根、叶、茎、花和角果)中表达。在油菜[5-6]和芥兰[7]研究中,也证实F-box蛋白基因与作物的抗病、抗盐胁迫、器官生长和花期等性状存在显著的相关性。Ikram等[8]通过转录组分析发现,高温胁迫后菜心叶片中有27个F-box蛋白基因表达水平差异显著,其中AUF2基因表现出显著的下调,其可能对菜心的高温胁迫产生响应。目前,关于AUF2基因在菜心生长发育过程中的生物学功能未见详细的报道。【本研究切入点】检测候选基因在自然群体中具有多态性,通过关联分析有助于阐释相关基因的生物学功能,挖掘其优异等位基因可应用于作物的遗传育种[9]。Mao等[10]对262份玉米自交系材料中的ZmNAC111基因进行测序和关联分析,发现一个长度为82 bp的InDel与种子存活率显著相关,表型解释率为7.27%;唐婧泉等[11]检测了SnRK基因家族在530份油菜材料的多态性,发现14个SNP位点与种子含油量显著关联,表型解释率最高达到12.95%,构成的优异单倍型材料种子含油量平均达42%;许德蓉等[12]在110份马铃薯材料中检测到StDRO1基因的6个SNP与马铃薯根系性状显著关联,构成的优异单倍型根系性状与其他单倍型的差异更显著。【拟解决的关键问题】本研究利用156份菜心种质材料构建自然群体,通过PCR扩增和直接测序,分析AUF2基因在自然群体中变异位点,然后与菜心的农艺性状进行关联分析,确定AUF2基因在菜心生长发育中的生物功能,挖掘AUF2的优异等位位点和单倍型,为菜心品种的分子辅助选育提供理论依据。
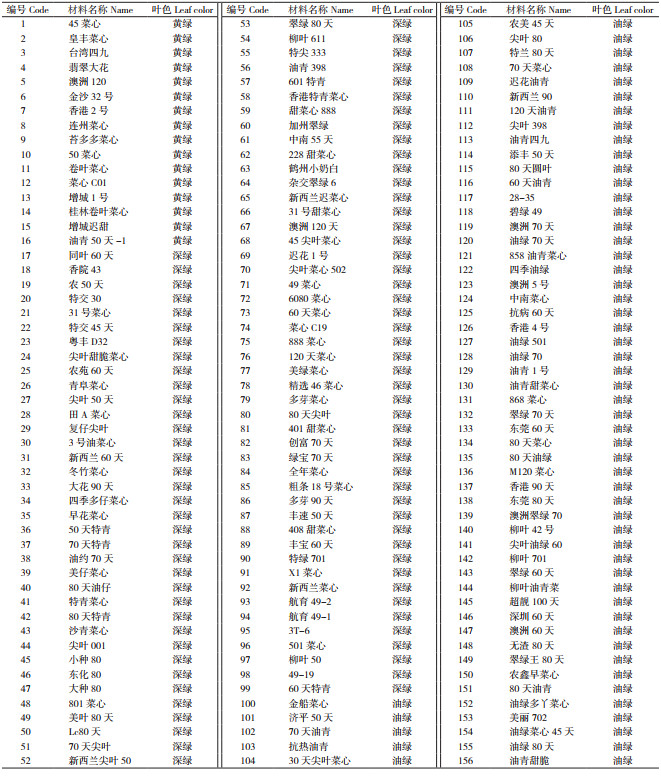
1 材料与方法 1.1 试验材料本研究选用156份菜心种质材料构建自然群体,其中黄绿叶色的材料16份、深绿叶色的材料83份和油绿叶色的材料57份,均来源于广州市农业科学研究院,部分材料为全国不同地方的商用品种,其余材料为广州市农业科学研究院在全国范围内收集的野生种并自行编号(表 1)。
所有菜心种质材料于2020年11月26日在广州大学作物抗逆国际合作研究中心的试验基地进行种植,采用随机区组设计,每个品种单独编号,均匀播种在0.5 m2的种植区域,常规水肥管理和病虫害防治,待生长至齐口花采收期。参考前人方法[13-15],每个种质材料避开边行,选取代表性的5株植株,对株高、单株质量、最大叶长、最大叶宽、最大叶柄长和叶绿素含量(SPAD值)等6个农艺性状进行测定,其中株高和单株质量是菜心优异品种选择的主要指标[14],而最大叶长、最大叶宽、最大柄长和叶绿素含量(SPAD值)能代表菜心外观品质的大部分性状[15]。
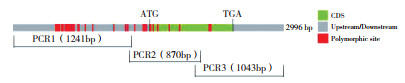
1.2 试验方法1.2.1 AUF2基因的扩增及测序 取菜心的新鲜嫩叶,利用植物基因组DNA提取试剂盒提取菜心的DNA,通过琼脂糖凝胶电泳和SmartSpec plus核酸蛋白检测仪检测所提取DNA的质量和浓度,检测合格的DNA样品稀释成10 ng/μL作为后续PCR扩增的模板。参考Brassica napus(rapa)的AUF2基因序列(XM_013866655.3),利用Primer 5.0设计3对具有重叠区域的特异性引物(表 2、图 1),扩增156份菜心种质材料的AUF2基因序列,琼脂糖凝胶电泳分离鉴定PCR扩增产物的特异性,做好相关标记后送至生工生物工程(上海)有限公司进行PCR产物双向测序。

|
|
| 图 1 AUF2基因的多态性位点及PCR扩增区域 Fig. 1 Distribution of polymorphism sites and PCR amplification region of AUF2 gene |
1.2.2 菜心群体中AUF2基因的多态性分析 使用Mega 6.0软件[16]对初始测序结果进行修剪后,拼接156份菜心种质材料的AUF2基因序列,然后与Brassica napus(rapa)的AUF2基因序列(XM_013866655.3)进行序列比对,利用Geneious 9.0.2软件[17]分析可能存在的SNP等变异位点。核苷酸多样性、单倍型多样性、Tajima’s D值、Fu and Li’s D*值及Fu and Li’s F*值等通过软件Dnasp 6[18]进行计算。
1.2.3 AUF2基因的多态性位点与菜心农艺性状的关联分析 使用Tassel 5.0软件[19]的混合线性模型(Mixed liner model,MLM)对AUF2基因的多态性位点与菜心的6个农艺性状进行关联分析,关联分析所需群体结构和亲缘关系数据来自实验室前期分析成果[20],在P<0.01水平上,统计多态性位点对表型变异的贡献率(R2)。参考王娟等[21]方法,计算等位变异的表型效应值,获得与表型性状显著关联多态位点的优异等位变异。
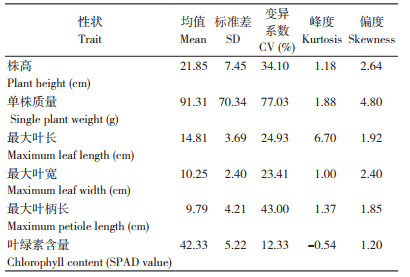
2 结果与分析 2.1 菜心群体主要农艺性状的变异特征由表 3可知,菜心的株高、单株质量、最大叶长、最大叶宽、最大叶柄长和叶绿素含量(SPAD值)等6个农艺性状在156个菜心种质材料间都存在明显差异,变异丰富,变异系数在12.30%~77.03% 之间,其中,叶绿素含量(SPAD值)的变异系数最小,单株质量的变异系数最大。6个农艺性状的偏度值分别在1.20~4.80之间,表明6个农艺性状呈现明显的右偏离,群体中低于平均值的菜心种质材料较多,其中叶绿素含量(SPAD值)偏度值最小,而单株质量的偏度值最大。
|
2.2 菜心群体中AUF2基因的核苷酸多态性
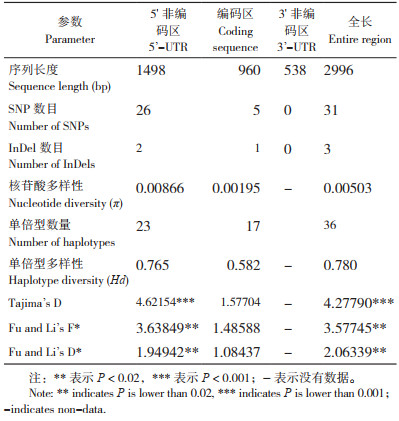
通过PCR扩增和测序分析,显示AUF2基因在菜心自然群体中存在丰富的自然变异,在2 996 bp的扩增区内共鉴定出34个变异位点,包含31个SNP和3个InDel,构成36个单倍型(图 1、表 4),其中31个SNP中碱基的转换数和颠换数分别为23、8个,核苷酸多样性(π)为0.00503,单倍型多样性(Hd)为0.780。由表 4可知,上游非编码区1 498 bp内检测到26个SNP和2个InDel(均缺失11 bp),构成23个单倍型,核苷酸多样性(π)为0.00866,单倍型多样性(Hd)为0.765;编码区960 bp内检测到5个SNP(2个SNP为同义突变,3个SNP为非同义突变)和1个InDel(缺失23 bp),构成17个单倍型,核苷酸多样性(π)为0.00195,单倍型多样性(Hd)为0.582;下游非编码538 bp范围没有检测出变异位点。
|
选用Tajima’s D、Fu and Li’s D*和Fu and Li’s F*等中性检验评价基因是否受到选择作用。在中性进化模型下,这些统计值为零,当统计值为负值通常是由于定向选择引起,而当统计值为正值时则由平衡选择引起[22]。由表 4可知,AUF2基因在菜心自然群体中Tajima’s D值为4.27790,在P<0.001水平上显著偏离中性选择,表明其存在大量中等频率的等位位点,可能的原因是群体瓶颈效应或平衡选择;Fu and Li’s D*值和Fu and Li’s F*值分别为2.06339和3.57745,在P<0.02水平上显著偏离中性检测。不同区段的中性检验显示,AUF2基因上游非编码区显著偏离中性选择,但在编码区偏离中性选择不显著。该结果说明,AUF2基因在菜心驯化过程中经受了更大的选择压力,可能是选择的靶标候选基因。
2.3 AUF2基因的多态位点与菜心农艺性状的关联分析采用Tassel 5.0软件中的混合线性模型(MLM)对AUF2基因的多态位点与菜心的株高、单株质量、最大叶长、最大叶宽、最大叶柄长和叶绿素含量(SPAD值)等6个农艺性状进行关联分析,结果显示有4个SNP分别与单株质量、最大叶长、最大叶宽和最大叶柄长等4个农艺性状显著关联(P<0.01),其对表型变异的解释率在5.04%~6.52% 之间,平均值为5.69%(表 5)。其中SNP_1267与单株质量、最大叶长、最大叶宽和最大叶柄长等4个农艺性状都显著相关,对表型变异的解释率分别为5.43%、6.42%、5.04% 和6.52%,优异等位变异为A碱基,占检测样本的频率为25.00%;SNP_1346、SNP_1461和SNP_1504等3个位点都与最大叶柄长显著相关,对表型变异的解释率分别为5.30%、5.47%和5.66%,其优异等位变异分别是C、C和T等碱基,占检测样本的频率分别为26.28%、28.85%和25.64%。SNP_1267、SNP_1346和SNP_1461 3个变异位点位于5' 端的非编码区,SNP_1504为编码区的非同义突变,导致其氨基酸由天冬氨酸突变为缬氨酸。
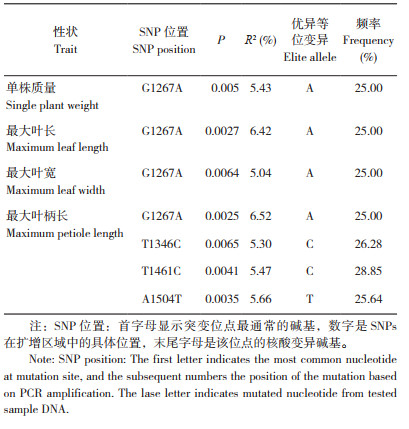
|
在156个菜心种质中,SNP_1267、SNP_1346、SNP_1461和SNP_1504等4个显著关联的SNP位点构成了7个单倍型(表 6),其中单倍型Hap1由4个最通常的碱基构成,含110个种质材料,占总数的70.51%;单倍型Hap2由4个优异等位变异构成,含35个种质材料,占总数的22.44%;其余5个单倍型为稀有单倍型,其占比均小于5%。与单倍型Hap1相比,单倍型Hap2的单株质量在P<0.05水平上显著增加,最大叶柄长在P<0.01水平上极显著提高,而株高、最大叶长、最大叶宽和叶绿素含量(SPAD值)等4个农艺性状无显著差异(表 7)。
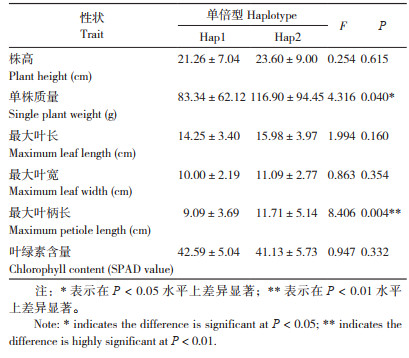
|
3 讨论
检测候选基因内的等位变异,有助于鉴定和分离遗传多样性材料中有价值的等位变异。本研究发现AUF2基因在156份菜心种质群体中存在丰富的自然变异,在2 996 bp的扩增区内共检测到34个变异位点,其中26个SNP和2个InDel分布在上游非编码区,5个SNP和1个InDel分布在编码区。前人研究证实,基因内的不同区域SNP频率不同,如编码区比非编码区存在更少的SNP和InDel[23-24]。AUF2基因在菜心群体在中的核苷酸多样性为0.00503,这一结果略高于甲基化相关基因在白菜和芥蓝群体中核苷酸多样性(0.004558和0.004352)[25],显著高于β- CT亚基编码基因accD在油菜群体中核苷酸多样性(0.00009)[26],这可能与试验材料的多样性相关,也反映了基因变异的丰富程度。中性选择性评价显示AUF2基因的Tajima’s D值、Fuand Li’s D*值和Fuand Li’s F*值均显著偏离中性检测,其可能是群体瓶颈效应或平衡选择的结果,前人通过分子标记[27-29]对菜心的多样性分析也表明菜心种质材料间的遗传背景较窄,这可能由于菜心仅是白菜的一个变种,起源于我国华南地区,栽培区域的局限及长期系统选育造成了种质资源的亲缘关系较近[29]。
对AUF2基因的多态位点与菜心6个农艺性状进行关联分析,结果发现1个SNP同时与单株质量、最大叶长、最大叶宽和最大叶柄长等4个农艺性状显著关联(P<0.01),还有3个SNP与最大叶柄长显著关联(P<0.01),4个SNP对表型变异的解释率在5.04%~6.52% 之间,平均值为5.69%,优异等位位点的频率达到25% 以上,其中3个SNP位于5’端的非编码区,1个SNP为编码区的非同义突变,导致酸性的天冬氨酸变为非极性的缬氨酸,这种单一氨基酸的变化很容易破坏分子内相互作用的复杂网络,影响蛋白质的折叠方式、稳定性、动力学,并最终影响蛋白质的功能[30]。由4个优异等位变异构成的单倍型Hap2在P<0.01水平上显著提高最大叶柄长的长度,这可能是AUF2基因促进了细胞分裂素介导的细胞在伸长和分化区扩张的结果[2]。叶柄长是白菜类蔬菜的重要农艺性状,影响作物的种植密度和外观品质。缪体云等[31]研究显示,结球甘蓝的叶柄长由2对主基因控制,其遗传率高达90.44%。李威等[32]和章云[33]利用大白菜DH群体均鉴定到2个与叶柄长相关的QTL位点,解释的遗传贡献率在6.29%~12.9% 之间,刘洋[34]在构建大白菜-菜心染色体片段替换系的基础上,共检测到8个与叶柄长相关的QTL位点,加性效应在37.32%~62.61% 之间,他们都在连锁群A08上检测到1个QTL位点,这个QTL位点可能与位于A08染色体上的AUF2基因存在相互作用来调节白菜类蔬菜叶柄的长度,而关于AUF2基因调控叶柄长的分子机理还需进一步的研究。
4 结论AUF2基因在菜心群体中存在丰富的自然变异。在2 996 bp的扩增区内共鉴定出34个变异位点,其中上游非编码区1 498 bp内含26个SNP和2个InDel,编码区960 bp内含5个SNP和1个InDel,而下游非编码区538 bp范围没有检测出变异位点。中性检测显示,AUF2基因在菜心自然群体中Tajima’s D值、Fuand Li’s D*值和Fuand Li’s F*值均显著偏离中性选择,其可能是群体瓶颈效应或平衡选择的结果。关联分析发现,4个SNP位点与菜心的单株质量、最大叶长、最大叶宽和最大叶柄长等4个农艺性状呈显著相关性(P<0.01),其表型解释率在5.04%~6.52% 范围,4个优异等位变异构成的单倍型Hap2能显著提高菜心的单株质量和最大叶柄长。这些优异等位变异和单倍型可为菜心品种的遗传改良提供参考。
| [1] |
陈汉才, 吴增祥, 林悦欣, 沈卓, 黎庭耀, 杨易, 周轩, 张艳. 广东菜心、芥蓝研究现状与展望[J]. 广东农业科学, 2021, 48(9): 62-71. DOI:10.16768/j.issn.1004-874X.2021.09.007 CHEN H C, WU Z X, LIN Y X, SHEN Z, LI T Y, YANG Y, ZHOU X, ZHANG Y. Research status and prospect of flowering Chinese cabbage and Chinese kale in Guangdong[J]. Guangdong Agricultural Sciences, 2021, 48(9): 62-71. DOI:10.16768/j.issn.1004-874X.2021.09.007 |
| [2] |
ZHENG X, MILLER N D, LEWIS D R, CHRISTIANS M J, LEE K, MUDAY G K, SPALDING E P, VIERSTRA R D. AUXIN UP-REGULATED F-BOX PROTEIN1 regulates the cross talk between auxin transport and cytokinin signaling during plant root growth[J]. Plant Physiology, 2011, 156(4): 1878-1893. DOI:10.1104/pp.111.179812 |
| [3] |
吴丹, 唐冬英, 李新梅, 李丽, 赵小英, 刘选明. F-box蛋白在植物生长发育中的功能研究进展[J]. 生命科学研究, 2015, 19(4): 362-367. DOI:10.16605/j.cnki.1007-7847.2015.04.012 WU D, TANG D Y, LI X M, LI L, ZHAO X Y, LIU X M. Progresses on F-box protein function in plant growth and development[J]. Life Science Research, 2015, 19(4): 362-367. DOI:10.16605/j.cnki.1007-7847.2015.04.012 |
| [4] |
RAMENENI J J, DHANDAPANI V, PAUL P, DEVARAJ S P, CHOI R, YI S Y, HONG S M, OH S H, OH M H, LIM Y P. F-box genes in Brassica rapa: genome-wide identification, structural characterization, expressional validation, and comparative analysis[J]. Plant Molecular Biology Reporter, 2018(36): 500-517. DOI:10.1007/s11105-018-1083-1 |
| [5] |
GAO Y, ZHAO Y, LI T T, LIU Y, REN C X, WANG M L. Molecular cloning and expression analysis of an F-box protein gene responsive to plant hormones in Brassica napus[J]. Molecular Biology Reports, 2010, 37(2): 1037-1044. DOI:10.1007/s11033-009-9822-x |
| [6] |
左蓉, 吴姗, 刘杰, 胡鸣, 程晓晖, 刘越英, 白泽涛, 刘胜毅. 油菜F-box-LRR基因全基因组鉴定与核盘菌诱导应答分析[J]. 中国油料作物学报, 2021, 44(3): 503-514. DOI:10.19802/j.issn.1007-9084.2021121 ZUO R, WU S, LIU J, HU M, CHENG X H, LIU Y Y, BAI Z T, LIU S Y. Genome-wide characterization of F-box-LRR genes and their transcript response to Sclerotinia sclerotiorum induction in Brassica napus[J]. Chinese Journal of Oil Crop Sciences, 2021, 44(3): 503-514. DOI:10.19802/j.issn.1007-9084.2021121 |
| [7] |
CAI Z M, ZENG D E, LIAO J J, CHENG C H, SAHITO Z A, XIANG M Q, CHEN Y Q, WANG D D. Genome-wide analysis of Auxin receptor family genes in Brassica juncea var. tumida[J]. Genes, 2019, 10(2): 165. DOI:10.3390/genes10020165 |
| [8] |
IKRAM M, CHEN J F, XIA Y S, LI R H, SIDDIQUE K H M, GUO P G. Comprehensive transcriptome analysis reveals heat-responsive genes in flowering Chinese cabbage (Brassica campestris L. ssp. chinensis) using RNA sequencing[J]. Frontiers in Plant Science, 2022, 13: 1077920. DOI:10.3389/fpls.2022.1077920 |
| [9] |
武晶, 黎裕. 基于作物种质资源的优异等位基因挖掘: 进展与展望[J]. 植物遗传资源学报, 2019, 20(6): 1380-1389. DOI:10.13430/j.cnki.jpgr.20190527001 WU J, LI Y. Mining superior alleles in crop germplasm resources: advances and perspectives[J]. Journal of Plant Genetic Resources, 2019, 20(6): 1380-1389. DOI:10.13430/j.cnki.jpgr.20190527001 |
| [10] |
MAO H D, WANG H W, LIU S X, LI Z G, YANG X H, YAN J B, LI J S, TASNS L P, QIN F. A transposable element in a NAC gene is associated with drought tolerance in maize seedlings[J]. Nature Communications, 2015, 6: 8326. DOI:10.1038/ncomms9326 |
| [11] |
唐婧泉, 王南, 高界, 刘婷婷, 文静, 易斌, 涂金星, 傅廷栋, 沈金雄. 甘蓝型油菜SnRK基因家族生物信息学分析及其与种子含油量的关系[J]. 作物学报, 2021, 47(3): 416-426. DOI:10.3724/SP.J.1006.2021.04108 TANG J Q, WANG N, GAO J, LIU T T, WEN J, YI B, TU J X, FU T D, SHEN J X. Bioinformatics analysis of SnRK gene family and its relation with seed oil content of Brassica napus L.[J]. Acta Agronomica Sinica, 2021, 47(3): 416-426. DOI:10.3724/SP.J.1006.2021.04108 |
| [12] |
许德蓉, 孙超, 毕真真, 秦天元, 王一好, 李成举, 范又方, 刘寅笃, 张俊莲, 白江平. 马铃薯StDRO1基因的多态性鉴定及其与根系性状的关联分析[J]. 作物学报, 2022, 48(1): 76-85. DOI:10.3724/SP.J.1006.2022.04241 XU D R, SU N C, BI Z Z, QIN T Y, WANG Y H, LI C J, FAN Y F, LIU Y D, ZHANG J L, BAI J P. Identification of StDRO1 gene polymorphism and association analysis with root traits in potato[J]. Acta Agronomica Sinica, 2022, 48(1): 76-85. DOI:10.3724/SP.J.1006.2022.04241 |
| [13] |
郭娜, 左凯峰, 张淼, 张冰冰, 秦梦凡, 马宁, 刘翔, 李青青, 黄镇, 徐爱遐. 甘蓝型油菜主要株型和产量性状的综合分析[J]. 西北农业学报, 2020, 29(6): 898-906. DOI:10.7606/j.issn.1004-1389.2020.06.010 GUO N, ZUO K F, ZHANG M, ZHANG B B, QIN M F, MA N, LIU X, LI Q Q, HUANG Z, XU A X. Comprehensive analysis of major plant-type and yield traits in Brassica napus L.[J]. Acta Agriculturae Boreali-occidentalis Sinica, 2020, 29(6): 898-906. DOI:10.7606/j.issn.1004-1389.2020.06.010 |
| [14] |
史卫东, 罗海玲, 康红卫, 农贵雄, 韦爱培. 基于主成分分析与聚类分析的菜心品种评价[J]. 安徽农业科学, 2020, 48(24): 46-49, 52. DOI:10.3969/j.issn.0517-6611.2020.24.014 SHI W D, LUO H L, KANG H W, NONG G X, WEI A P. Evaluation of Chinese flowering cabbage variety by principal component analysis and cluster analysis[J]. Journal of Anhui Agricultural Sciences, 2020, 48(24): 46-49, 52. DOI:10.3969/j.issn.0517-6611.2020.24.014 |
| [15] |
卢宇鹏, 夏岩石, 温少波, 李荣华, 李光光, 张华, 郭培国. 不同熟性菜心品质性状的多样性分析[J]. 广东农业科学, 2020, 47(5): 29-36. DOI:10.16768/j.issn.1004-874X.2020.05.004 LU Y P, XIA Y S, WEN S B, LI R H, LI G G, ZHANG H, GUO P G. Diversity of quality traits of Chinese flowering cabbage varieties with different maturity[J]. Guangdong Agricultural Sciences, 2020, 47(5): 29-36. DOI:10.16768/j.issn.1004-874X.2020.05.004 |
| [16] |
STECHER G, PETERSON D, FILIPSKI A, KUMA S. Mega6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology & Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [17] |
VENKATRAMANAN S, LSKSHMI D M, SATTAR Z A, ZAINALABIDIN N A, AZMAN N A. Case study of wildlife forensic via an integrated bioinformatics software, geneious[J]. International Journal of Medical Toxicology and Legal Medicine, 2021, 23(4): 49-52. DOI:10.5958/0974-4614.2020.00044.3 |
| [18] |
JULIO R, ALBERT F M, SÁNCHEZ-DELBARRIO J C, SARA G R, PABLO L, RAMOS-ONSINS S E, SÁNCHEZ-GRACIA A. DnaSP 6: DNA sequence polymorphism analysis of large data sets[J]. Molecular Biology & Evolution, 2017, 34(12): 3299-3302. DOI:10.1093/molbev/msx248 |
| [19] |
BRADBURY P J, ZHANG Z, KROON D E, CASSTEVENS T M, RAMDOSS Y, BUCKLER E S. TASSEL: software for association mapping of complex traits in diverse samples[J]. Bioinformatics, 2007, 23(19): 2633-2635. DOI:10.1093/bioinformatics/btm308 |
| [20] |
严少杰. 菜心重要农艺性状与SSR分子标记的关联分析[J]. 中国果菜, 2021, 41(2): 34-43. DOI:10.19590/j.cnki.1008-1038.2021.02.007 YAN S J. Association analysis of agronomic traits with SSR molecular markers in Brassica campestris L.[J]. China Fruit & Vegetable, 2021, 41(2): 34-43. DOI:10.19590/j.cnki.1008-1038.2021.02.007 |
| [21] |
王娟, 董承光, 刘丽, 孔宪辉, 王旭文, 余渝. 棉花适宜机采相关性状的SSR标记关联分析及优异等位基因挖掘[J]. 作物学报, 2017, 43(7): 954-966. DOI:10.3724/SP.J.1006.2017.00954 WANG J, DONG C G, LIU L, KONG X H, WANG X W, YU Y. Association analysis and exploration of elite alleles of mechanical harvest-related traits with SSR markers in upland cotton cultivars (Gossypium hirsutum L.)[J]. Acta Agronomica Sinica, 2017, 43(7): 954-966. DOI:10.3724/SP.J.1006.2017.00954 |
| [22] |
TANG X T, XU J, LU M X, DU Y Z. No evidence for an effect of wolbachia on mtDNA variation and evolution in natural populations of Sesamia inferens (Lepidoptera: Noctuidae)[J]. Journal of Integrative Agriculture, 2019, 18(5): 1050-1063. DOI:10.1016/S2095-3119(18)62019-2 |
| [23] |
陈发波, 姚启伦, 方平, 董二飞, 陈春. 芸薹属蔬菜Chs基因序列多态性[J]. 基因组学与应用生物学, 2015, 34(12): 2702-2715. DOI:10.13417/j.gab.034.002702 CHEN F B, YAO Q L, FANG P, DONG E F, CHEN C. Sequence polymorphism of Chs genes in Chinese Brassica vegetables[J]. Genomics and Applied Biology, 2015, 34(12): 2702-2715. DOI:10.13417/j.gab.034.002702 |
| [24] |
YANG S, GILL R A, ZAMAN Q U, ULHASSAN Z, ZHOU W J. Insights on SNP types, detection methods and their utilization in Brassica species: recent progress and future perspectives[J]. Journal of Biotechnology, 2020, 324: 11-20. DOI:10.1016/j.jbiotec.2020.09.018 |
| [25] |
AN F, ZHANG K, ZHANG L K, LI X, CHEN S M, WANG H S, CHENG F. Genome-wide identification, evolutionary selection, and genetic variation of DNA methylation-related genes in Brassica rapa and Brassica oleracea[J]. Journal of Integrative Agriculture, 2022, 21(6): 1620-1632. DOI:10.1016/S2095-3119(21)63827-3 |
| [26] |
付三雄, 张洁夫, 陈锋, 顾慧, 陈松, 戚存扣. 油菜β-CT亚基编码基因accD的SNPs分析及其分子进化[J]. 江西农业学报, 2011, 23(12): 7-11. DOI:10.3969/j.issn.1001-8581.2011.12.002 FU S X, ZHANG J F, CHEN F, GU H, CHEN S, QI C K. Single nucleotide polymorphisms analysis of β-CT subunit gene accD in Brassica napus and its molecular evolution[J]. Acta Agriculturae Jiangxi, 2011, 23(12): 7-11. DOI:10.3969/j.issn.1001-8581.2011.12.002 |
| [27] |
李桂花, 陈汉才, 张艳, 温艺敏, 陈琼贤, 张桂权. 菜心种质资源遗传多样性的SRAP分析[J]. 中国农学通报, 2012, 28(4): 110-114. DOI:10.3969/j.issn.1000-6850.2012.04.020 LI G H, CHEN H C, ZHANG Y, WEN Y M, CHEN Q X, ZHANG G Q. Genetic diversity of Brassica parachinensis germplasm revealed by SRAP analysis[J]. Chinese Agricultural Science Bulletin, 2012, 28(4): 110-114. DOI:10.3969/j.issn.1000-6850.2012.04.020 |
| [28] |
郭培国, 许兰桂, 夏岩石, 黄红弟, 张华, 郑岩松, 李荣华. 菜薹种质遗传多样性的荧光MFLP标记分析[J]. 园艺学报, 2015, 42(2): 350-360. DOI:10.16420/j.issn.0513-353x.2014-0704 GUO P G, XU L G, XIA Y S, HUANG H D, ZHANG H, ZHENG Y S, LI R H. Genetic diversity analysis for germplasm of flowering Chinese cabbage by using fluorescent microsatellite-anchored fragment length polymorphism[J]. Acta Horticulturae Sinica, 2015, 42(2): 350-360. DOI:10.16420/j.issn.0513-353x.2014-0704 |
| [29] |
史卫东, 琚茜茜, 张力, 罗海玲, 熊发前, 康红卫. 菜心种质资源遗传多样性的SCoT分析[J]. 南方农业学报, 2015, 46(8): 1350-1355. DOI:10.3969/j:issn.2095-1191.2015.08.1350 SHI W D, JU X X, ZHANG L, LUO H L, XIONG F Q, KANG H W. Analysis on genetic diversity of Chinese flowering cabbage (Brassica campestris L. ssp. chinensis Var. utilis Tsen et Lee) germplasms based on SCoT markers[J]. Journal of Southern Agriculture, 2015, 46(8): 1350-1355. DOI:10.3969/j:issn.2095-1191.2015.08.1350 |
| [30] |
RODRIGUES C H M, PIRES D E V, ASCHER D B. DynaMut2: Assessing changes in stability and flexibility upon single and multiple point missense mutations[J]. Protein Science, 2020, 30(1): 60-69. DOI:10.1002/pro.3942 |
| [31] |
缪体云, 刘玉梅, 方智远, 杨丽梅, 庄木, 张扬勇, 袁素霞, 孙培田. 一个结球甘蓝DH群体主要农艺性状的遗传效应分析[J]. 园艺学报, 2008, 35(1): 59-64. DOI:10.16420/j.issn.0513-353x.2008.01.012 MIAO T Y, LIU Y M, FANG Z Y, YANG L M, ZHUANG M, ZHANG Y Y, YUAN S X, SUN P T. Genetic analysis of the main agronomic traits of DH population in Brassica oleracea var. capitata[J]. Acta Horticulturae Sinica, 2008, 35(1): 59-64. DOI:10.16420/j.issn.0513-353x.2008.01.012 |
| [32] |
李威, 王晓武, 武剑. 大白菜叶片长宽性状的QTL定位[J]. 中国蔬菜, 2009(16): 14-19. DOI:10.19928/j.cnki.1000-6346.2009.16.003 LI W, WANG X W, WU J. QTL mapping related to leaf morphological traits in Brassica rapa L. spp. pekinensis[J]. China Vegetables, 2009(16): 14-19. DOI:10.19928/j.cnki.1000-6346.2009.16.003 |
| [33] |
章云. 大白菜分子遗传图谱构建及重要农艺性状QTL分析[D]. 沈阳: 沈阳农业大学, 2012. ZHANG Y. Construction of a molecular genetic map and QTL mapping for major agronomic traits in Chinese cabbage (Brassica campestris spp. Pekinensis)[D]. Shengyang: Shenyang Agricultural University, 2012. |
| [34] |
李洋. 菜心种子休眠和生长发育相关农艺性状的QTL定位[D]. 沈阳: 沈阳农业大学, 2018. LI Y. QTL mapping of seed dormancy and growth and development related agronomic traits in Brassica campestris L. spp. Chinensis var. [D]. Shengyang: Shenyang Agricultural University, 2018. |
(责任编辑 马春敏)
 2023, Vol. 50
2023, Vol. 50