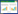文章信息
基金项目
- 国家重点研发计划(2022YFD1801000);“十四五”广东省农业科技创新十大主攻方向“揭榜挂帅”项目(2022SDZG02);广东省基础与应用基础研究基金(2022A1515110737);云南省廖明专家工作站项目(202105AF150077)
作者简介
- 曾庆航(1996—)男,在读硕士生,研究方向为H9N2亚型禽流感防控,E-mail:zengqinghangyy@163.com.
通讯作者
- 廖明(1968—),男,博士,教授,研究方向为禽流感等家禽重大疾病防控,E-mail:mliao@scau.edu.cn.
文章历史
- 收稿日期:2023-03-03
2. 广东省农业科学院动物卫生研究所/广东省畜禽疫病防治研究重点实验室/农业农村部禽流感等家禽重大疾病防控重点实验室,广东 广州 510640
2. Institute of Animal Health, Guangdong Academy of Agricultural Sciences/Key Laboratory of Livestock Disease Prevention of Guangdong Province/Key Laboratory for Prevention and Control of Avian Influenza and Other Major Poultry Diseases, Ministry of Agriculture and Rural Affairs, Guangzhou 510640, China
【研究意义】H9N2亚型禽流感病毒(Avian Influenza Virus,AIV)是目前家禽养殖中最常见的病毒之一,极易发生基因突变和基因重组,导致免疫逃逸而造成免疫失败。该病毒感染性强,传播速度快,易感染雏鸡和青年鸡群,主要侵害机体上呼吸道,可引起机体的免疫抑制,引发混合感染或继发感染,导致鸡群生长缓慢、蛋品质和产蛋量下降。由于其致病性低,导致H9N2亚型AIV在许多国家未被良好监测和有效控制,现已在全球家禽中流行;其流行广泛,危害持久,难以清除,给养禽业构成严重威胁并造成了巨大经济损失。因此,研究H9N2亚型AIV的遗传进化和生物学特征,为我国H9N2亚型AIV防控提供参考数据具有重要意义。【前人研究进展】 H9N2亚型AIV最早于1966年在美国威斯康星州北部的火鸡上发现,我国则于1992年在广东省的鸡群中首次发现该病毒,随后该病毒在我国多地出现并蔓延,经历了BJ94-like→F98-like→Y280-like的演化,当前国内流行毒株均属于h9.4.2.5分支(Y280-like)[1-3]。自1999年首次发现人感染H9N2亚型AIV以来,截至2022年3月30日全球共发现110例,我国发生97例(占比88.18%),主要集中在我国南方,而广东省报道最多[4-7]。据多项研究报道,H9N2已取代H5N6和H7N9成为在禽类中流行的优势亚型,且其作为病毒基因库为H3N8、H5N1、H5N6、H7N9、H10N8等亚型AIV提供内部基因进行基因重组,被认为可能引起下一次流感大流行[8-15]。【本研究切入点】AIV属于正黏病毒科甲型流感病毒属,基因组由8个长度在890~2 341个核苷酸的单链负义RNA片段组成,可编码病毒的17种结构蛋白,其中以HA和NA变异最大,是AIV最主要的保护性抗原,因此HA和NA基因常作为基因变异依据用于AIV遗传进化和生物学特征分析[16]。【拟解决的关键问题】本研究通过对2022年广东地区3株H9N2亚型AIV的HA和NA基因进行遗传进化和生物学特征分析,为我国H9N2亚型AIV的进化分析和防控提供参考。
1 材料与方法 1.1 试验材料1.1.1 供试毒株 H9N2亚型AIV毒株A/Chicken/Guangdong/JY01/2022、A/Chicken/Guangdong/FS03/2022、A/Chicken/Guangdong/KP03/2022(表 1),由广东省农业科学院动物卫生研究所分离鉴定和纯化保存。
1.1.2 引物 设计合成AIV-12bp随机引物、HA和NA基因PCR通用引物(表 2),引物由生工生物工程(上海)股份有限公司合成。
1.1.3 主要试剂 病毒DNA/RNA提取试剂盒(磁珠法)购自西安天隆科技有限公司,2×Taq Master Mix和DL2000 DNA Marker购自南京诺唯赞生物科技股份有限公司,PrimeScriptTM RT Master Mix、pMD19-T载体和DH5α感受态细胞购自宝生物工程(大连)有限公司,琼脂糖凝胶DNA回收试剂盒和质粒提取纯化试剂盒购自天根生化科技(北京)有限公司。
1.2 试验方法1.2.1 HA和NA基因扩增、克隆及测序 对病毒液进行总RNA提取,根据PrimeScriptTM RT Master Mix说明书,使用AIV-12bp随机引物进行反转录获得cDNA;使用HA和NA基因通用引物进行PCR扩增,将目的基因纯化回收,与pMD19-T载体连接并转入DH5α感受态细胞,挑取阳性克隆质粒送生工生物工程(上海)股份有限公司进行Sanger测序。
1.2.2 HA和NA基因遗传进化及氨基酸关键位点分析 采用SeqMan、Editseq、PhyloSuite软件对核苷酸序列进行拼接,并进行氨基酸关键位点比对分析;运用Megalign软件对核苷酸序列进行同源性分析;利用MEGA11软件采用ML法绘制HA、NA基因遗传进化树;使用NetNGlyc 1.0 Server软件对氨基酸序列N-糖基化位点进行分析。
2 结果与分析 2.1 HA和NA基因PCR扩增和测序结果3株H9N2亚型AIV的HA和NA基因PCR扩增产物分别在1 683 bp和1 401 bp的位置(图 1)。测序结果与预期大小一致,3株病毒的HA基因完整开放阅读框(ORF)各含有1 683个核苷酸,NA基因完整ORF各含有1 401个核苷酸(图 2)。
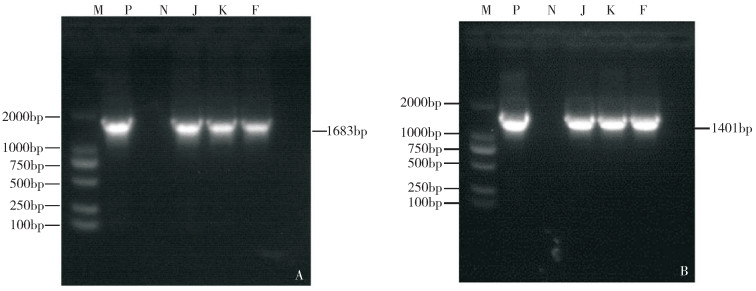
|
| M:DL2000 Marker;P:阳性对照;N:阴性对照;J、K、F:PCR产物 M: DL-2 000 Marker; N: Negative control; P: Positive control; J, K, F: PCR product 图 1 HA(A)、NA(B)基因的PCR产物 Fig. 1 PCR products of HA(A) and NA(B) genes |

|
| 图 2 3株H9N2亚型AIV的HA、NA基因ORF测序结果 Fig. 2 ORF sequencing results of HA and NA genes of three H9N2 AIV strains |
2.2 HA和NA基因系统发育树分析结果
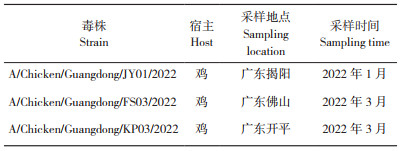
基因系统发育树分析结果显示,HA基因属于当前国内流行的h9.4.2.5分支,与经典株A/Chicken/Guangxi/55/200的遗传距离较远,已进化成新的亚群,命名为h9.4.2.5c分支(图 3A);NA基因仍属于1分支,与早期流行的经典株A/Duck/Hong Kong/Y280/97遗传距离较远,在近几年形成了一个相对稳定的新的亚分支,命名为1.2分支(图 3B)。
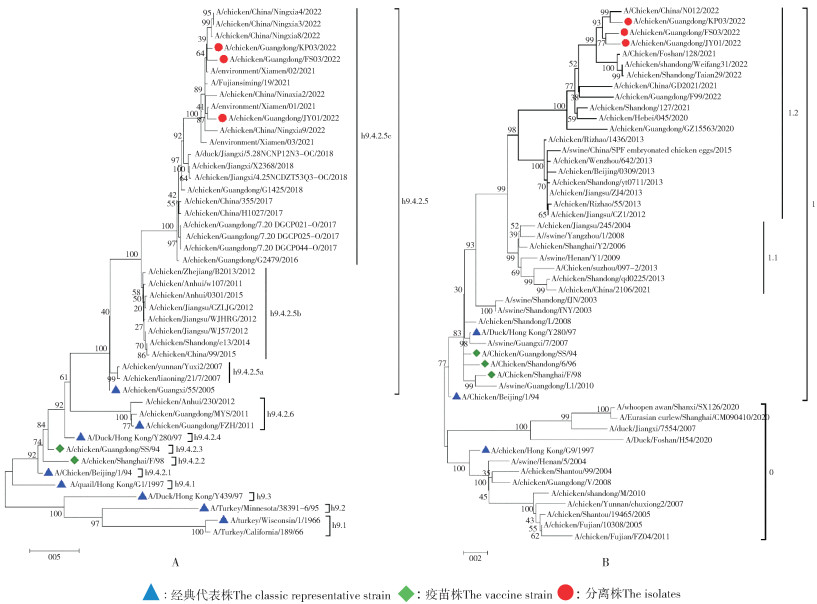
|
| 图 3 3株H9N2亚型AIV的HA(A)、NA(B)基因系统发育树 Fig. 3 Phylogenetic tree of HA(A) and NA(B) genes of three H9N2 AIV strains |
2.3 HA和NA基因核苷酸序列同源性分析结果
核苷酸序列比对分析结果(表 3)显示,3株H9N2亚型AIV之间HA基因核苷酸序列同源性为97.6%~98.3%,与h9.4.2.5分支经典代表株A/Chicken/Guangxi/55/2005的同源性最高,介于91.3%~91.7%之间;与h9.4.2.4分支经典代表株A/Duck/Hong Kong/Y280/97的同源性最低,介于81.6%~81.9%之间。3株H9N2亚型AIV之间NA基因核苷酸序列同源性为97.2%~98%,与h9.4.2.3分支疫苗株A/Chicken/Guangdong/SS/94的同源性最高,介于89.4%~89.8%之间;与h9.4.2.5经典分支代表株A/Chicken/Guangxi/55/2005同源性最低,介于86.0%~86.8%之间。
2.4 HA氨基酸序列关键位点分析结果
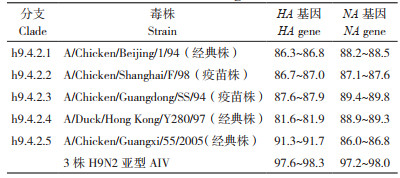
3株H9N2亚型AIV的HA基因序列完整的ORF均由1 683个核苷酸组成,编码560个氨基酸;HA裂解位点均为PSRSSR ↓ GLF,符合低致病性特征;受体结合位点均发生了I155T、H183N、A190T/V、T212I、Q226L、Q227M突变;潜在糖基化位点均只有7个,218位由NRT突变为NRI,变成非糖基化位点,313位由NCP突变为NCS,新增糖基化位点(表 4)。
2.5 NA氨基酸序列关键位点分析结果
3株H9N2亚型AIV的NA基因序列完整的ORF均由1 401个核苷酸组成,共编码466个氨基酸;与参考株A/Chicken/Beijing/1/94、A/Chicken/Guangdong/SS/94、A/Duck/Hong Kong/Y280/97相比,在茎部均缺失187~195位9个核苷酸(ACAGAGATA),导致NA蛋白缺失63~65位3个氨基酸(TEI),这与A/Duck/Hong Kong/Y280/97株为代表的国内流行毒株一致。1.2分支NA蛋白的红细胞结合位点(Hemadsorbing site,HB)366~373位产生K/E/S368N、D369N突变;与NA蛋白活性、抗流感药物奥司他韦和扎那米韦耐药性相关的E119、D151、R152、R224、E276、R292、R371位点均未发生突变(表 5)。
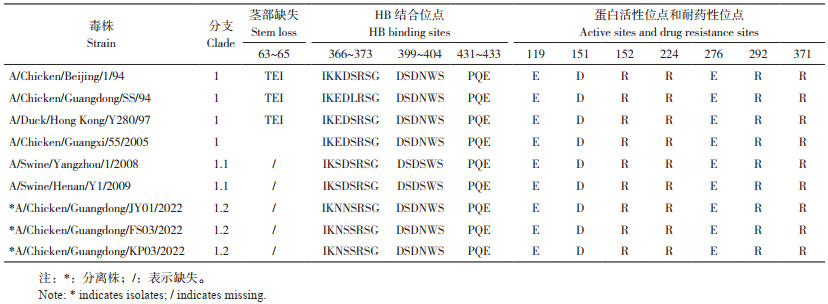
|
3 讨论
禽流感病毒属于RNA病毒,在免疫压力和外界因素影响下易发生基因突变和基因重组,进化出新的基因型。我国自1992年首次报道H9N2亚型AIV已有30年的流行历史,期间经历了BJ94-like→F98-like→Y280-like的演化,1997年报道A/Duck/Hong Kong/Y280/97毒株后,以Y280-like为代表的毒株成为我国流行的优势毒株,均属于h9.4.2.5分支[17-18]。我国于1998年开始在鸡群中使用灭活疫苗来控制该病毒引起的疾病,在家禽疫苗接种计划后,H9N2亚型AIV的演变速度加快[19-21]。AIV NA基因的0分支以A/Chicken/Hong Kong/G9/97株为代表,主要在1997—2011年流行;1分支则在1994—2006年流行,以A/Chicken/Beijing/1/94和A/Duck/Hong Kong/Y280/97株为代表;2007—2013年开始演变出稳定的1.1和1.2亚分支并流行至今[22]。本研究3株H9N2亚型AIV的基因遗传进化分析结果显示,HA、NA基因与早期的经典株A/Chicken/Beijing/1/94、A/Chicken/Guangxi/55/2005和疫苗株A/Chicken/Shanghai/F/98、A/Chicken/Guangdong/SS/94等的遗传距离较远,核苷酸同源性分别仅为81.6%~91.7%和88.2%~91.2%,已经进化成新的亚群[6-9]。因此,持续跟踪、系统分析,及时掌握流行变异情况,对H9N2亚型AIV的防控具有重要意义。
流感病毒侵染细胞的先决条件是HA裂解为HA1和HA2,HA2暴露出疏水端,HA蛋白发生融膜[23]。LPAIV HA裂解位点一般为R-S-S-R结构,只含1个或2个连续碱性氨基酸,只有精氨酸特异蛋白酶能识别并裂解这种结构,因此一般只引起局部感染。本研究3株H9N2亚型AIV HA蛋白酶解位点序列为PSRSSR ↓ GLF,符合低致病性特征。与A/Chicken/Guangxi/55/2005经典株等相比,3个分离株HA蛋白裂解位点的第334位均由A变为S,而研究表明该突变会增强病毒对家禽的致病性[24]。
流感病毒的适应性突变主要发生于病毒表面膜蛋白,包括HA蛋白受体位点的改变、糖基化修饰位点的分布和NA蛋白茎部的缺失[25]。有研究报道,HA蛋白受体结合位点155、183、190、212、226、227位与人源唾液酸受体结合的亲和力有关,I155T、H183N、A190T/V、T212I、Q226L、Q227M突变会增强与人源唾液酸受体结合的亲和力,其中190V具有高度亲和力,其次是190T[26-28]。本研究中,3个分离株HA蛋白受体结合位点分析显示,均发生I155T、H183N、A190T/V、T212I、Q226L、Q227M突变,提示表现出人源唾液酸受体结合特性。另有研究表明,HA蛋白218N突变为非糖基化位点,同时313N新增为糖基化位点,会增强病毒与受体的结合能力,阻止同源抗血清对病毒的中和作用,以及增强病毒对细胞的感染能力和在细胞中的复制能力[29-30]。本研究3个分离株HA蛋白糖基化位点分析显示,218由NRT突变为NRI成为非糖基化位点,同时313位由NCP突变为NCS,新增为糖基化位点,与前期研究相符,提示该突变同时提高了该3株病毒的致病性及改变了其抗原性。
NA蛋白头部有酶活性中心和抗原位点,茎部与蛋白锚定有关[23],茎部缺失是流感病毒毒力增强的标志[31]。本研究3株H9N2亚型AIV的NA基因序列分析显示,与参考株A/Chicken/Beijing/1/94、A/Chicken/Guangdong/SS/94、A/Duck/Hong Kong/Y280/97相比,分离株的茎部均缺失187~195位9个核苷酸(ACAGAGATA),导致NA蛋白缺失63~65位3个氨基酸(TEI),这与A/Duck/Hong Kong/Y280/97株为代表的国内流行毒株一致。茎部缺失使病毒更易脱离细胞,增强毒力、扩大宿主范围[32]。流感病毒NA蛋白上E119A/D/G/V、D151E、R152K、R224K、E276D、R292K、R371K位点突变会改变蛋白活性和增强对抗流感病毒药物奥司他韦、扎那米韦的耐药性[33-34]。本研究3株H9N2亚型AIV的以上位点均未发生此突变。研究报道,NA蛋白的红细胞结合位点366~373、399~404和431~433分别形成3个环状结构,唾液酸和氨基酸直接结合在环状结构中[35]。本研究观察到HB结合位点产生K/E/S368N、D369N的变化,该突变是否会影响唾液酸和氨基酸结合有待进一步探究。
4 结论2022年从广东部分地区分离的3株H9N2亚型AIV的HA和NA基因分别属于h9.4.2.5c和1.2分支,与以A/Chicken/Beijing/1/94为代表的经典株和A/Chicken/Guangdong/SS/94等疫苗株的遗传距离较远,核苷酸序列同源性分别仅为81.6%~91.7%和88.2%~91.2%,已经进化成新的亚群;HA氨基酸序列均发生I155T、H183N、A190T/V、T212I、Q226L、Q227M突变,218N去糖基化的同时313N糖基化,以及NA氨基酸序列63~65位(TEI)缺失,表明目前广东地区流行的H9N2亚型AIV可能增强了对哺乳动物的适应性,且其抗原性发生改变;同时,NA氨基酸序列E119、D151、R152、R224、E276、R292、R371均未发生突变,表明其尚未获得对奥司他韦和扎那米韦等抗流感病毒药物的耐药性。本研究结果表明3株H9N2亚型AIV的HA和NA部分关键氨基酸序列已发生不同程度变异,建议持续对H9N2亚型AIV的流行情况进行监测。本研究结果明确了广东省H9N2亚型AIV的感染现状及流行毒株的分子流行病学特点、遗传演化情况,可为广东地区今后更有效地预防控制H9N2亚型AIV感染及疫苗选择提供参考。
| [1] |
HOMME P J, EASTERDAY B C. Avian influenza virus infections I: Characteristics of influenza A-turkey-Wisconsin-1966 virus[J]. Avian Diseases, 1970, 14(1): 66-74. DOI:10.2307/1588557 |
| [2] |
陈伯伦, 张泽纪, 陈伟斌. 禽流感研究I: 鸡A型禽流感病毒的分离与血清学初步鉴定[J]. 中国兽医杂志, 1994(10): 3-5. CHEN B L, ZHANG Z J, CHEN W B. Bird flu research I: Isolation and preliminary serological identification of avian influenza virus type A from chickens[J]. Chinese Journal of Veterinary Medicine, 1994(10): 3-5. |
| [3] |
陈坚, 徐凌, 贺瑶, 杨青春, 李晓艳, 李超, 田志辉, 赵丽霞, 宋庆庆. 禽流感病毒在我国的流行情况及公共卫生意义[J]. 兽医导刊, 2021(7): 55-56. CHEN J, XU L, HE Y, YANG Q C, LI X Y, LI C, TIAN Z H, ZHAO L X, SONG Q Q. Epidemic situation and public health significance of avian influenza virus in China[J]. Veterinary Orientation, 2021(7): 55-56. |
| [4] |
袁世超, 张宏斌, 焦培荣, 武婕. 人感染禽流感病毒病例在我国的分布情况[J]. 中国人兽共患病学报, 2022, 38(4): 374-377. DOI:10.3969/j.issn.1002-2694.2022.00.042 YUAN S C, ZHANG H B, JIAO P R, WU J. Distribution of patients infected by avian influenza virus in China[J]. Chinese Journal of Zoonoses, 2022, 38(4): 374-377. DOI:10.3969/j.issn.1002-2694.2022.00.042 |
| [5] |
JIN Y F, CUI H, JIANG L, ZHANG C, LI J J, CHENG H L, CHEN Z H, ZHENG J, ZHANG Y D, FU Y Y. Evidence for human infection with avian influenza A (H9N2) virus via environmental transmission inside live poultry market in Xiamen, China[J]. Journal of Medical Virology, 2023, 95(1): e28242. DOI:10.1002/jmv.28242 |
| [6] |
张官婷, 梁丹, 李安安, 洪文珊, 梁玉凤, 王涛, 柯昌文. 人感染H9N2禽流感相关研究进展[J]. 中国公共卫生, 2022, 38(10): 1359-1363. DOI:10.11847/zgggws1137142 ZHANG G T, LIANG D, LI A A, HONG W S, LIANG Y F, WANG T, KE C W. Human infection of H9N2 avian influenza viruses: progress in epidemiological research[J]. Chinese Journal of Public Health, 2022, 38(10): 1359-1363. DOI:10.11847/zgggws1137142 |
| [7] |
王艳文, 王梦静, 张虹, 高鑫鑫, 郭晶, 李旭勇. 1998-2021年我国人感染H9N2亚型禽流感病毒的遗传演化规律[J]. 中国农业科学, 2022, 55(20): 4075-4090. DOI:10.3864/j.issn.0578-1752.2022.20.017 WANG Y W, WANG M J, ZHANG H, GAO X X, GUO J, LI X Y. Evolution of human H9N2 avian influenza virus in China from 1998 to 2021[J]. Scientia Agricultura Sinica, 2022, 55(20): 4075-4090. DOI:10.3864/j.issn.0578-1752.2022.20.017 |
| [8] |
BI Y H, LI J, FU G H, JIN T, ZHANG C, YANG Y C, MA Z H, TIAN W X, LI J D, XIAO S Q, LI L Q, YIN R F, ZHANG Y, WANG L X, QIN Y T, YAO Z Z, MENG F Y, HU D F, LI D L, WONG G, LIU F, NA L, WANG L, FU L F, YANG Y, PENG Y, MA J M, KIRILL S, ALEXANDER S, MARINA G, GAO F, CHEN J J, SHI Y, LIU J, CHU D, HUANG Y, LIU Y X, LIU L, LIU W J, CHEN Q J, SHI W F. Dominant subtype switch in avian influenza viruses during 2016-2019 in China[J]. Nature Communications, 2020, 11(1): 5909. DOI:10.1038/s41467-020-19671-3 |
| [9] |
SUN W Q, LI J X, HU J, JIANG D X, XING C N, ZHANG T S, LIU X F. Genetic analysis and biological characteristics of different internal gene origin H5N6 reassortment avian influenza virus in China in 2016[J]. Veterinary Microbiology, 2018, 219: 200-211. DOI:10.1016/j.vetmic.2018.04.023 |
| [10] |
PU J, YIN Y B, LIU J Y, WANG X Y, ZHOU Y, WANG Z J, SUN Y P, SUN H L, LI F T, SONG J W, QU R K, GAO W H, WANG D D, WANG Z, YAN S J, CHEN M Y, ZENG J F, JIANG Z M, SUN H R, ZONG Y N, WANG C X, TONG Q, BI Y H, HUANG Y H, DU X J, CHANG K C, LIU J H. Reassortment with dominant chicken H9N2 influenza virus contributed to the fifth H7N9 virus human epidemic[J]. Journal of virology, 2021, 95(11): e01578-20. DOI:10.1128/JVI.01578-20 |
| [11] |
刘开拓, 高如一, 王晓泉, 刘秀梵. 新型重组H10N3亚型禽流感病毒对公共卫生安全的威胁[J]. 病毒学报, 2022, 38(4): 958-964. DOI:10.13242/j.cnki.bingduxuebao.004165 LIU K T, GAO R Y, WANG X Q, LIU X F. Threat posed by a novel reassortant avian influenza virus(H10N3)to public health[J]. Chinese Journal of Virology, 2022, 38(4): 958-964. DOI:10.13242/j.cnki.bingduxuebao.004165 |
| [12] |
SONG W, QIN K. Human-infecting influenza A (H9N2) virus: A forgotten potential pandemic strain?[J]. Zoonoses Public Health, 2020, 67(3): 203-212. DOI:10.1111/zph.12685 |
| [13] |
顾敏, 刘文博, 曹军平, 曹永忠, 张小荣, 彭大新, 刘秀梵. 1株含H9N2内部基因的H5N1亚型基因重配禽流感病毒的全基因测序及遗传进化分析[J]. 病毒学报, 2010, 26(4): 298-304. DOI:10.13242/j.cnki.bingduxuebao.002093 GU M, LIU W B, CAO J P, CAO Y Z, ZHANG X R, PENG D X, LIU X F. Genome sequencing and genetic analysis of a natural reassortant H5N1 subtype avian influenza virus possessing H9N2 internal genes[J]. Chinese Journal of Virology, 2010, 26(4): 298-304. DOI:10.13242/j.cnki.bingduxuebao.002093 |
| [14] |
ZHANG P H, TANG Y H, LIU X W, LIU W B, ZHANG X R, LIU H Q, PENG D X, GAO S, WU Y T, ZHANG L Y, LU S, LIU X F. A novel genotype H9N2 influenza virus possessing human H5N1 internal genomes has been circulating in poultry in eastern China since 1998[J]. Journal of Virology, 2009, 83(17): 8428-8438. DOI:10.1128/JVI.00659-09 |
| [15] |
YANG R G, SUN H, GAO F, LUO K W, HUANG Z, YONG Q, SONG H, HANG Q Q, LIU J Y, QI J X, LI H, CHEN S L, XU M Z, QIU J S, ZENG G, ZHANG X X, HUNAG C Y, PEI R Q, ZHAN Z F, YE B W, GUO Y X, ZHOU Y Z, YE W, YAO D, REN M, LI B, YANG J Z, WANG Y N, PU J, SUN Y P, SHI Y, LIU J, OU X H, GAO F, GAO L D, LIU J H. Human infection of avian influenza A H3N8 virus and the viral origins: a descriptive study[J]. The Lancet Microbe, 2022, 3(11): e824-e834. DOI:10.1016/S2666-5247(22)00192-6 |
| [16] |
CHAUHAN R P, GORDON M L. An overview of influenza A virus genes, protein functions, and replication cycle highlighting important updates[J]. Virus Genes, 2022, 58(4): 255-269. DOI:10.1007/s11262-022-01904-w |
| [17] |
LIU Q Z, ZHAO L C, GUO Y N, ZHAO Y Z, LI Y F, CHEN N, LU Y L, YU M Q, DENG L L, PING J H. Antigenic evolution characteristics and immunological evaluation of H9N2 avian influenza viruses from 1994-2019 in China[J]. Viruses, 2022, 14(4): 726. DOI:10.3390/v14040726 |
| [18] |
SUN H L, LIN J T, LIU Z T, YU Y N, WU M H, LI S, LIU Y, FENG Y L, WU Y Q, LI M L, JIAO P R, LUO K J, LIAO M. Genetic, molecular, and pathogenic characterization of the H9N2 avian influenza viruses currently circulating in South China[J]. Viruses, 2019, 11(11): 1040. DOI:10.3390/v11111040 |
| [19] |
PARK K J, KWON H I, SONG M S, PASCUA P N Q, BEAK T H, LEE J H, JANG H L, LIM J Y, MO I P, MOON H J, KIM C J, CHOI Y L. Rapid evolution of low-pathogenic H9N2 avian influenza viruses following poultry vaccination programmes[J]. The Journal of General Virology, 2011, 92(1): 36-50. DOI:10.1099/vir.0.024992-0 |
| [20] |
LI C J, YU K Z, TIAN G B, YU D D, LIU L L, JIN B, PING J H, CHEN H L. Evolution of H9N2 influenza viruses from domestic poultry in mainland China[J]. Virology, 2005, 340(1): 70-83. DOI:10.1016/j.virol.2005.06.025 |
| [21] |
YAN W W, CUI H R, ENGELSMA M, BEERENS N, OERS M M V, JONG M C M D, LI X S, LIU Q F, YANG J M, TENG Q Y, LI Z J. Molecular and antigenic characterization of avian H9N2 viruses in Southern China[J]. Microbiology Spectrum, 2022, 10(1): e82221. DOI:10.1128/spectrum.00822-21 |
| [22] |
PU J, WANG S, YIN Y, ZHANG G, CARTER R A, WANG J, XU G, SUN H, WANG M, WEN C, WEI Y, WANG D, ZHU B, LEMMON G, JIAO Y, DUAN S, WANG Q, DU Q, SUN M, BAO J, SUN Y, ZHAO J, ZHANG H, WU G, LIU J, WEBSTER R G. Evolution of the H9N2 influenza genotype that facilitated the genesis of the novel H7N9 virus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(2): 548-553. DOI:10.1073/pnas.1422456112 |
| [23] |
LAMB R A, CHOPPIN P W. The genetic structure and replication of influenza virus[J]. Annual Review of Biochemistry, 1983, 52: 467-506. DOI:10.1146/annurev.bi.52.070183.002343 |
| [24] |
GAO Y W, ZHANG Y Z, SHINYA K, DENG G H, JIANG Y P, LI Z J, GUAN Y T, TIAN G B, LI Y B, SHI J Z, LIU L L, ZENG X Y, BU Z G, XIA X Z, KAWAOKA Y, CHEN H L. Identification of amino acids in HA and PB2 critical for the transmission of H5N1 avian influenza viruses in a mammalian host[J]. PLoS Pathogens, 2009, 5(12): e1000709. DOI:10.1371/journal.ppat.1000709 |
| [25] |
MUNOZ O, DE N M, VAN D M K, VAN R K, KOOPMANS M, HARRIS K, VON D Z S, FREIDL G, MEIJER A, BREED A, HILL A, KOSMIDER R, BANKS J, STARK K D, WIELAND B, STEVENS K, VAN D W S, ENOUF V, DAUPHIN G, DUNDON W, CATTOLI G, CAPUA I. Genetic adaptation of influenza a viruses in domestic animals and their potential role in interspecies transmission: A literature review[J]. EcoHealth, 2016, 13(1): 171-198. DOI:10.1007/s10393-014-1004-1 |
| [26] |
VINES A, WELLS K, MATROSOVICH M, CASTRUCCI M R, ITO T, KAWAOKA Y. The role of influenza A virus hemagglutinin residues 226 and 228 in receptor specificity and host range restriction[J]. Journal of Virology, 1998, 72(9): 7626-7631. DOI:10.1128/JVI.72.9.7626-7631.1998 |
| [27] |
LI X Y, SHI J Z, GUO J, DENG G H, ZHANG Q Y, WANG J L, HE X J, WANG K C, CHEN J M, LI Y Y, FAN J, KONG H H, GU C Y, GUAN Y T, SUZUKI Y S, KAWAOKA Y, LIU L L, JIANG Y P, TIAN G B, LI Y B, BU Z G, CHEN H L. Genetics, receptor binding property, and transmissibility in mammals of naturally isolated H9N2 avian influenza viruses[J]. PLoS Pathogens, 2014, 10(11): e1004508. DOI:10.1371/journal.ppat.1004508 |
| [28] |
MATROSOVICH M N, KRAUSS S, WEBSTER R G. H9N2 influenza A viruses from poultry in Asia have human virus-like receptor specificity[J]. Virology, 2001, 281(2): 156-162. DOI:10.1006/viro.2000.0799 |
| [29] |
彭欠欠. H9N2亚型流感病毒HA蛋白糖基化位点变化影响病毒生物学特性的研究[D]. 扬州: 扬州大学, 2017. PENG Q Q. Study of the biological characteristics of H9N2 virus with the variation of the glycosylation sites in HA protein[D]. Yangzhou: Yangzhou University, 2017. |
| [30] |
PENG Q Q, ZHU R, WANG X B, SHI H Y, BELLEFLEUR M, WANG S F, LIU X F. Impact of the variability in potential glycosylation sites of the hemagglutinin of H9N2 influenza virus[J]. Virus Genes, 2019, 55(2): 182-190. DOI:10.1007/s11262-018-1623-7 |
| [31] |
SPACKMAN E. A brief introduction to avian influenza virus[J]. Methods in Molecular Biology (Clifton, N. J.), 2020, 2123: 83-92. DOI:10.1007/978-1-0716-0346-8-7 |
| [32] |
SUN Y P, TAN Y Y, WEI K, SUN H L, SHI Y, PU J, YANG H C, GAO G F, YIN Y B, FENG W H, PEREZ D R, LIU J H. Amino acid 316 of hemagglutinin and the neuraminidase stalk length influence virulence of H9N2 influenza virus in chickens and mice[J]. Journal of Virology, 2013, 87(5): 2963-2968. DOI:10.1128/JVI.02688-12 |
| [33] |
YEN H L, HOFFMANN E, TAYLOR G, SCHOLTISSEK C, MONTO A S, WEBSTER R G, GOVORKOVA E A. Importance of neuraminidase active-site residues to the neuraminidase inhibitor resistance of influenza viruses[J]. Journal of Virology, 2006, 80(17): 8787-8795. DOI:10.1128/JVI.00477-06 |
| [34] |
王莹丽, 蒋艳, 朱瑞芳, 徐潜, 曹彬, 郭丽萍. 全球与中国流感病毒对NAIs耐药性趋势及特征[J]. 中华医院感染学杂志, 2020, 30(2): 272-277. DOI:10.11816/cn.ni.2020-191369 WANG Y L, JIANG Y, ZHU R F, XU Q, CAO B, GUO L P. Trend and characteristics of drug resistance of influenza viruses to neuraminidase inhibitors in China and around the glibe[J]. Chinese Journal of Nosocomiology, 2020, 30(2): 272-277. DOI:10.11816/cn.ni.2020-191369 |
| [35] |
卢秀娴. 2014—2017年我国部分地区H9N2禽流感病毒HA和NA基因分析及免疫效力评估[D]. 广州: 华南农业大学, 2017. LU X X. Evolutionary analysis and immunological evaluation of H9N2 subtype avian influenza virus HA and NA gene in parts of China from 2014-2017[D]. Guangzhou: South China Agricultural University, 2017. |
(责任编辑 崔建勋)
 2023, Vol. 50
2023, Vol. 50