文章信息
基金项目
- 国家自然科学基金(32160687、31872079);海南省重大科技专项(ZDKJ2021014);海南省自然科学基金(321RC467);海南大学科研创业基金(KYQD-ZR-20090)
作者简介
- 张红娜(1986—),博士,研究员,博士生导师,海南省高层次人才拔尖人才。现任海南大学专职教师,长期从事热带果树花果发育分子生物学研究,从物候、生理和分子生物学等多个层面对外界环境因子和外源植物激素等特异性诱导亚热带果树花芽分化的机理进行阐释,逐步形成一套亚热带果树成花机理的系统理论,为生产上采用人工调控热带果树成花技术提供理论和技术支持。主持国家自然科学基金(3项)、国家重点研发计划专项子课题(2项)、广东省自然科学基金(1项)、海南省自然科学基金(3项)、海南省重大项目子课题(2项)等11项省部级以上科研项目。以第一作者或通信作者在《 Journal of Agricultural and Food Chemistry》《 Frontiers in Plant Science》《 DNA Research》《 Scientia Horticulturae》等期刊发表学术论文20余篇,其中SCI论文13篇(中科院一区Top封面1篇、二区10篇);授权实用新型专利4件。兼任广东省园艺学会第十一届理事,获省农业科技下乡活动先进个人、园艺产业创新学术研讨会论文一等奖等荣誉称号;荣获第十届海南省青年科技奖。张红娜(1986—),女,博士,研究员,研究方向为热带果树花果发育分子生物学,E-mail:13692476979@139.com.
文章历史
- 收稿日期:2023-06-01
2. 海南大学热带农林学院,海南 儋州 571737
2. School of Tropical Agriculture and Forestry, Hainan University, Danzhou 571737, China
长链非编码RNA(Long non-coding RNA,lncRNA)是指长度大于200 nt,由RNA聚合酶转录而来但不具备或具备较弱编码蛋白能力,能够直接在表观遗传、转录、转录后、翻译和翻译后水平上调控基因的表达的RNA[1]。真核生物体中只有大约2% 的RNA能够参与蛋白编码,剩余约98% 的RNA为非编码RNA(ncRNA),lncRNA属于ncRNA的一种。由于非编码RNA不能参与蛋白质的编码,一直以来都被认为是没有作用的“基因组垃圾”,直到1984年真核生物中的第一个lncRNA——H19在小鼠中被鉴定[2]。1994年,植物中的第一个lncRNA——ENOD40 (EARLY NODULIN 40)在大豆和蒺藜苜蓿中被鉴定,并被发现在豆科植物根瘤形成中发挥作用,同时确定了lncRNA以RNA的形式发挥作用而不是蛋白质[3]。基因组学时代到来后,许多研究发现植物基因组的未注释区域广泛存在转录[4],因此开始了全基因组lncRNA的鉴定尝试。lncRNA种类繁多、功能机制复杂,在植物生长发育过程中发挥着重要作用,其中主要包括调控植物的生殖生长发育、参与植物胁迫响应等[5]。
鉴于在真核生物生长发育过程中的重要性,lncRNA成为近年研究热点。虽然相比于动物,植物lncRNA的探索仍处于初级阶段,但其基本的功能形式及机制已在多种模式植物中被了解。除拟南芥、水稻等模式植物外,lncRNA在多种园艺植物中也得到鉴定,如苹果[6]、葡萄[7]、番茄[8]、甘蓝[9] 等。园艺植物在人们生活中扮演着重要角色,园艺植物研究是农业发展重要的组成部分。园艺植物lncRNA的研究不仅可以完善植物lncRNA的调控机制,也将推动园艺植物的分子育种。近年来,园艺植物lncRNA被证明可以参与到代谢物产生、植株的生长发育以及生物、非生物胁迫响应中。本文就植物lncRNA的产生、分类、功能机制进行总结,并对目前园艺植物中所鉴定到的lncRNA及其生物学功能进行概述,以期对园艺植物中lncRNA的进一步鉴定及深入研究提供良好的基础和依据。
1 lncRNA的产生及特性 1.1 lncRNA的产生植物lncRNA的总数约占ncRNA的80%,是参与生物过程调控的关键成分[10]。虽然lncRNA没有潜在的编码能力,但其与mRNAs一样大部分由RNA聚合酶Ⅱ(Pol Ⅱ)转录而来,只有小部分由植物特有的RNA聚合酶Ⅳ或Ⅴ转录而来。植物体内的lncRNA一般有5种来源:(1)基因结构变异,导致可编码蛋白的转录本无法正常翻译而生成lncRNA;(2)染色体重组导致非转录区域合并lncRNA;(3)非编码基因复制过程中,发生反转座插入产生lncRNA;(4)串联重复序列的复制产生lncRNA;(5)转座子插入引入功能性启动子,使非转录区域具有转录活性,从而生成具有功能的lncRNA。
1.2 lncRNA的特性lncRNA在植物基因组中的数量众多,且没有共同的进化起源,因此其种类繁多,保守性也较低。研究发现,具有生物功能的lncRNA的共同特征与蛋白质类似,一般具有以下特点:(1)转录本长度较长且转录丰度低。lncRNA的转录本长度超过200 nts,但通常比mRNA编码的转录本短,且表达拷贝数较低,所以一般在转录组测序的数据中很难被鉴定,但是lncRNA的低表达确保了其对靶标的特异性。另外,部分lncRNA含有开放阅读框(ORF),且具有翻译成小肽的潜力。(2)结构与mRNA类似且具有可折叠能力。lncRNA的结构与mRNA相似,其在5’-有帽子结构,3’-有poly(A)尾巴,且可以被剪接。此外,lncRNA可以折叠成二级或更高级的结构,使其在靶向蛋白质或基因位点时更加灵活。(3)表达特异性高但保守性低。lncRNA表现出高度的组织及细胞特异性,甚至在不同生长发育时期表现出不同表达量,且其表达受到环境因素的强烈影响。lncRNA的亚细胞定位通常在细胞核中,以便与DNA、RNA、蛋白质发生相互作用,但少部分也可流出到细胞质及其他位置中。与mRNA相比,大多数lncRNA在物种间的序列保守性较低,这可能是由于其宽松的结构导致其在选择过程中快速进化的结果,而其序列变化的多样性可能是物种多样性的一个主要因素[11]。
2 lncRNA的分类 2.1 根据lncRNA与编码区域的相对位置分类根据lncRNA在基因组中相对于蛋白质编码基因的位置,可以将lncRNA分为5类:由编码基因重叠区域转录而来的正义lncRNA;由编码基因的反义链转录而来的反义lncRNA;由编码基因的启动子区域转录而来,但其转录方向与启动子方向相反的双向lncRNA;由编码基因内含子区域转录而来的基因内lncRNA;由两个编码基因之间的非编码区转录而来的基因间lncRNA。
2.2 根据lncRNA的转录位置分类根据lncRNA的转录位置,可以将lncRNA分为启动子上游型lncRNA、启动子相关型lncRNA、转录起始位点型lncRNA、非翻译区lncRNA、增强子相关lncRNA和转座子相关lncRNA。其中,增强子相关lncRNA和转座子相关lncRNA研究受到越来越多的关注,增强子相关lncRNA由于其长度较小容易被外泌体降解,因此植物大部分增强子相关lncRNA的功能是未知的,而转座子相关lncRNA主要与植物的转座子互相影响,转座子可以影响lncRNA的产生,转座子相关lncRNA也能影响转座子的活性[12]。
2.3 根据lncRNA的作用形式分类根据lncRNA在不同调控水平下的作用方式,可以将其分为4类:(1)信号分子类lncRNA,作为信号分子调控邻近基因的表达。(2)诱饵分子类lncRNA,作为诱饵招募转录因子、染色质修饰物等RNA结合蛋白(RBP)间接调控基因的表达,或者作为诱饵诱导结合miRNAs,阻断与其特异性靶基因的互作。(3)引导类lncRNA,可以引导RNA结合蛋白复合体定位到特定位置或招募染色质修饰酶到靶基因,从而调控靶基因的表达。(4)支架分子类lncRNA,通过其本身多种不同的功能结合域与不同的蛋白复合体或效应因子结合,从而实现其分子功能。
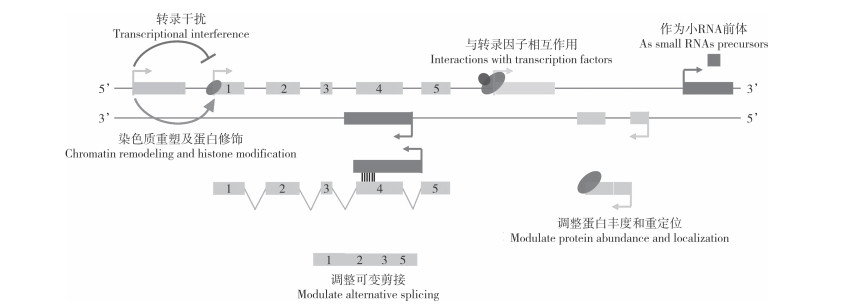
3 植物lncRNA的功能机制 3.1 干扰邻近基因的转录表达lncRNA发挥调控功能的方式繁多、调控机制复杂,可在转录前、转录后、翻译前、翻译后以及表观遗传等多个层面实现对植物生长发育过程的影响(图 1)[10]。当lncRNA邻近其下游调控基因(距离小于100 kb)时,通常会对其下游基因进行顺式调控,一般有3种方式。首先,当lncRNA结合到临邻近基因附近区域时,使得RNA pol Ⅱ由于空间阻碍而无法结合到相应区域,导致转录受抑制;第二种方式是距离相近的lncRNA和邻近基因如果同时进行转录,二者所需的转录因子及RNA pol Ⅱ等在空间中产生竞争,导致mRNA的转录效率下降;最后,如果lncRNA区含有顺式调控元件,当其开启转录时,可对下游基因进行调控。例如,尖孢镰刀菌侵染香蕉后,鉴定到的DELs通过顺式调控邻近的mRNA表达参与响应病菌胁迫,其中LNC_000607位于Ma03_t33700.1的上游,并调控参与Ma03_t33700.1水杨酸(SA)信号转导,Ma03_g02640.1受到其上游LNC_000457的调控,参与真菌感染条件下茉莉酸(JA)信号的转导,从而提高香蕉植株的抗病能力[13]。草莓lincRNA XLOC_014500顺式调控其上游邻近基因 XLOC_014501的表达,参与草莓花期及果实发育调控[14]。此外,在黄沙棘和红沙棘成熟果实中鉴定到的DELs中,XLOC_267510顺式调控红沙棘中类胡萝卜素异构酶基因CRTISO的表达[15]。当番茄晚疫病发生时,lncRNA39026通过诱导 PRs基因的表达来提高番茄对胁迫的抗性[16]。
3.2 作为miRNA的内源性靶标模拟物
内源性靶向模拟物(Endogenous target mimics, eTMs)是一类非编码的内源miRNA,它们能够与miRNA形成特定结合,从而有效阻碍miRNA的表达,同时保持其稳定性,避免其降解。LncRNA通过特异性结合位点与miRNA结合后,能够抑制胞内miRNA的积累,从而在转录后水平上增加miRNA靶标的表达,这类lncRNA被称为竞争性内源RNA(ceRNA)。番茄果实冷害处理后鉴定出的DELs中,TCONS_00364242被预测为 miRNA M00148和M00164的靶点,并在番茄遭受冷害胁迫时表达上调[17]。番茄Slylnc0195和 Slylnc1077分别作为miR166和miR399的模拟靶标,参与番茄黄花曲叶病毒TYLCV感染的响应[18]。在感染致病疫霉的番茄中,通过eTM分析鉴定到89个能够特异性结合miRNAs的lncRNAs,其中 lncRNA23468作为ceRNA竞争性结合miR482b,减轻miR482b对NBS-LRR的抑制作用,提高番茄对致病疫霉的抗性[16]。在甘蓝型油菜中,TCONS_00048391和TCONS_00010856作为miR164a的内源性靶标模拟物,参与热胁迫响应[19]。柑橘XLOC_016898和XLOC_017200作为miR166c的靶点和eTM影响果实发育[20]。
3.3 作为miRNA前体植物lncRNA除了可以作为miRNA的eTM外,还可以被直接或间接加工成更短的非编码RNA,其中与miRNA前体序列高度同源的lncRNA就以miRNA前体的角色在园艺植物中发挥作用。Boerner等[21]在玉米中鉴定到的1 802个lncRNA中,大约有60% 是miRNA的前体序列。番茄lncRNAZ114作为miRNA1919b和miRNA1919c的前体、lncRNAZ081作为miRNA6027的前体在番茄乙烯信号转导过程中发挥作用[22]。Li等[23]在木薯中鉴定出12个lncRNAs可以作为miRNA前体发挥作用。MUSA-S-NC321和MUSA-T-NC506分别作为miRNA166和miRNA156的前体在香蕉干旱胁迫条件下被鉴定[24]。
3.4 与转录因子相互作用园艺植物lncRNA在转录前水平下可以通过激活、抑制或招募转录因子到靶基因启动子区域从而调控下游基因的表达,同时,转录因子也可以靶向lncRNA诱导或抑制其表达,从而实现对下游基因的调控。HID1(HIDDEN TREASURE 1)通过靶向转录因子PIF3(PHYTOCHROME INTERACTING FACTOR3)启动子抑制其表达,参与拟南芥对红光的响应[25]。番茄中的RIN转录因子可以通过结合lncRNA2155、lncRNA1780、lncRNA3197等lncRNAs启动子区域的MADS-box元件,激活其转录,进一步调控番茄果实成熟相关基因的表达[26]。StFLORE是马铃薯StCDF1转录因子的天然反义lncRNA,StCDF1可以直接结合StFLORE启动子中的DOF基序调控其表达[27]。
3.5 调整mRNA的可变剪接选择性剪接被认为是影响mRNA命运和功能的关键机制[28]。lncRNA在转录后水平上可以通过调整mRNA的选择性剪接影响靶基因的表达。拟南芥定位于核斑点(富含mRNA前体和剪接因子的核结构域)的lncRNA ASCO介导许多mRNA前体的选择性剪接[29]。ASCO可以直接与剪接亚基PRP8a(PRE-MRNA PROCESSING 8a) 和SmD1b(Sm ring D1b)相互作用,调节剪接亚基与靶mRNA的相互作用。ASCO的缺失或过表达会导致数百个基因的选择性剪接发生显著变化。最近研究表明,ASCO还可以介导参与植物生物胁迫响应基因的剪接[30]。
3.6 调整蛋白的丰度和重定位目前为止,很少有发现lncRNA可以直接调节翻译过程。但是在水稻中,磷转运基因PHO1; 2(PHOSPHATE1; 2)的cis-NAT(天然反义转录物)在磷饥饿时显著上调并导致PHO1; 2蛋白水平增加,但是PHO1; 2的mRNA水平是稳定的[31]。在磷酸盐充足条件下,过表达PHO1; 2的cis-NAT仍然会导致PHO1; 2蛋白水平的增加,这表明通过表达长反义RNA(Long antisense RNA)可以激活蛋白翻译[32]。除直接参与mRNA翻译调控外,lncRNA也可以通过调整蛋白的重定位来发挥作用。ENOD40是最初在大豆和苜蓿中被发现的一种调控根瘤形成的lncRNA,其序列在多个物种间具有较高保守性。MtRBP1是在苜蓿中筛选到的MtENOD40互作蛋白。当在苜蓿中过表达MtENOD40后,MtRBP1会从原细胞核的核斑点中重新定位于细胞质中。随后研究又发现MtSNARP1和MtSNARP2两个RNA结合蛋白,二者也可与MtENOD40互作,且这两个蛋白的定位均受到MtENOD40的影响[33]。这表明MtENOD40通过调整蛋白的重定位发挥作用。
3.7 参与染色质重塑和组蛋白修饰染色质重塑对植物发育过程中特异基因的表达具有重要作用。LncRNA可以通过改变染色质的拓扑结构调节基因表达的变化。生长素处理后,lncRNA APOLO位点发生染色质松弛,导致APOLO及其上游调控生长素极性运输的邻近基因PID(PINOID)的RNA Pol-Ⅱ型表达。而RNA Pol-Ⅱ型APOLO的积累促进了RNA Pol-V的招募和Pol-V型APOLO变体的产生,并且RNA Pol-Ⅱ型APOLO的积累还会招募PRC1和PRC2,从而抑制该位点的H3K27me3,抑制PID基因的表达[34]。FLC(FLOWERING LOCUS C)作为开花抑制基因,通过染色质修饰调节植物开花时间。低温处理诱导VIN3表达,其招募PRC2增强FLC基因的组蛋白修饰,沉默FLC表达,促进植物开花。Swiezewski等在拟南芥中鉴定从FLC基因区域转录出的两个非编码RNA,分别为正义lncRNA COLDAIR和反义lncRNA COOLAIR,其中COOLAIR产生于FLC基因的3’ 端,其表达受低温诱导且抑制FLC转录[35]。除 COOLAIR外,COLDAIR也可以抑制FLC基因的表达,COLDAIR产生于FLC的第一个内含子区,其表达受低温处理的诱导。研究发现,COLDAIR通过与PRC2中的组分互作,影响FLC基因的组蛋白修饰过程,造成表观沉默,促进植物开花[36]。
4 园艺植物lncRNA的主要生物学功能 4.1 参与调控园艺植物的生长发育4.1.1 参与调控园艺植物的种子萌发 种子是植物的特有器官,对物种的延续具有重要意义。拟南芥lncRNA AtR8是由RNA聚合酶Ⅲ转录而来,受到水杨酸(SA)的诱导并在拟南芥幼苗根端大量表达,AtR8的缺失抑制了拟南芥对SA的响应及种子萌发[37]。AtR8在十字花科芸薹属中具有较高的序列保守性,BoNR8是拟南芥AtR8在结球甘蓝中的同源物。在甘蓝种子萌发时,BoNR8在根伸长区表皮层受脱落酸(ABA)的诱导而大量表达,在拟南芥中过表达BoNR8会导致ABA信号响应相关基因的表达上调,并抑制根的生长和种子萌发(表 1)[38]。类似地,白菜BrpR8与AtR8序列高度相似,其在白菜种子萌发时的伸长区高度表达,并参与SA信号响应[39]。

|
进一步研究发现,甘蓝BoNR8除在种子萌发中产生作用外,还参与光信号响应及下胚轴伸长[40]。lncRNA BoNR8启动子区域存在光响应G-box元件(ACGTG),暗培养条件下BoNR8的表达受到明显抑制。正常培养条件下,拟南芥中的BoNR8过表达会抑制下胚轴生长,同时显著抑制下胚轴生长相关基因PIN7和EXP3表达;暗培养条件下,BoNR8的过表达可促进PIF4和PIN7基因表达。以上结果表明BoNR8通过上调光响应基因的表达响应光信号,并影响下胚轴生长相关基因的表达,从而调节拟南芥下胚轴的生长[25]。
4.1.2 参与调控园艺植物的生殖生长 植物的生殖生长方式分为有性生殖和无性生殖,植物的生殖生长阶段是植物最重要的阶段,许多园艺植物lncRNA被证明参与生殖调控过程。Yang等[41]通过对番茄叶、花和根进行测序,共鉴定出65个DELs,其中XLOC_064364、XLOC_048112和XLOC_069054在番茄花中特异性高表达,表明其在番茄开花过程中发挥重要作用。Kang等[14]在草莓不同发育时期的花和果实中鉴定并验证了多个具有花药或成熟花粉特异性表达的lncRNAs,如XLOC_028671、XLOC_019639、XLOC_ 030226、XLOC_023242、XLOC_036386和XLOC_033366,表明这些lncRNAs在草莓花发育中的潜在作用。对龙眼花芽诱导过程中的差异表达lncRNAs进行鉴定,发现了参与不同激素信号转导的lncRNAs,并在乙烯信号传导通路中鉴定到TCONS_00025288、TCONS_00071997和TCONS_00164643,表明这3个DE-lncRNAs在龙眼花芽生理分化起到正向调控作用[42]。在甘蓝型油菜的花粉发育过程中,lncRNA可以作为miRNA的eTMs发挥调节功能,eTM160-1和eTM160-2被预测为miR160-5p的eTMs,且研究证实转基因植株花器官中的花粉粒有50% 表现出无核、皱缩、无活力或内壁缺失等败育表型[43]。
BcMF11在大白菜花粉发育和雄性育性中发挥着重要作用。抑制BcMF11的表达导致转基因植株表现出明显的形态学缺陷,并表现出花粉萌发率降低、花粉管延伸延缓、花粉粒皱缩塌陷以及绒毡层降解延迟等异常[44]。同时,白菜BrMYB80bSSLncRNA也被证实可以通过调控BrMYB80b的表达来调控花粉萌发沟的表达[45]。黄瓜lincRNA-CsM10被发现在雄性分化过程中优先表达,推测参与黄瓜花分化过程[46]。辣椒lncRNA1336、lncRNA7479及lncRNA8303在不育系中表达下调,导致花粉和绒毡层发育缺陷,最终导致辣椒败育[47]。Wu等[48] 以梅花单雌蕊品种‘青佳2号’和多雌蕊品种‘大羽’为材料,鉴定到多个DE-lncRNAs,其中XR_514690.2通过下调ppe-miR172d和上调AP2的表达,TCONS_00032517通过抑制ppe-miR160a/b并诱导细胞分裂素负调控基因A-ARR的表达促进梅花多雌蕊的形成。
大蒜是一种典型的无性生殖蔬菜,体细胞培养是大蒜脱毒快速繁殖的有效手段。植物激素在体细胞胚胎发生中起着重要作用,通过分析大蒜体细胞胚胎发生不同阶段的调控网络,发现并验证了生长素通路相关的ceRNA网络lncRNA125175-miR393h-TIR2可能通过调节生长素信号通路和改变细胞对生长素的敏感性来影响大蒜体细胞胚胎的发生[49]。
4.1.3 参与调控园艺植物的果实成熟及代谢物合成 果实成熟是一个复杂的发育过程,是一个涵盖多种植物激素和物质代谢的内外源通路整合而来的复杂网络。园艺植物的果实成熟往往伴随着果皮、果肉颜色以及口感和风味的变化,因此这一过程不仅伴随着复杂的激素信号传导,往往还涉及到色素等代谢物质的合成。lncRNA FRILAIR是从草莓成熟果实中获得的一个与果实成熟相关的lncRNA,其可与miR397竞争性结合,使其靶基因LAC11a的表达上调,从而促进花青素生物合成相关基因的表达,加速草莓的果实成熟[50]。沙棘lncRNA LNC1和LNC2是花青素生物合成的重要调节因子,二者作为miR156a和miR828a的内源性靶标模拟物分别降低SPL9和诱导MYB114的表达,导致花青素含量的增加和减少[51]。苹果ERF109是一个参与光诱导花青素生物合成的乙烯反应因子(ERF)蛋白,作为lncRNA MdLNC499的邻近基因受到其诱导表达。MdLNC499启动子中的W-box顺式元件被发现由转录因子MdWRKY1调节。因此三者形成一个调控网络,即MdWRKY1-MdLNC499-MdERF109,其中MdWRKY1被光激活增加MdLNC499的转录,从而诱导MdERF109,MdERF109蛋白诱导花青素相关基因的表达和花青素的积累[6]。同时,苹果中的MLNC3.2和MLNC4.6被发现作为miR156a的eTM在光诱导花青素生物合成过程中阻止miR156a降解SPL2和SPL33促进花青素的合成和积累[52]。Ke等[20]在柑橘中发现XLOC_017306、XLOC_017200作为csi-miR166c的eTMs,XLOC_009399、XLOC_019173作为csi-miR166c的前体形成lincRNA调控网络影响柑橘果实发育。葡萄lncRNAs鉴定及通路富集分显示部分lncRNAs参与果实类胡萝卜素的合成,共表达分析显示多个lncRNAs与编码果实成熟相关酶的mRNA表达,表明其参与葡萄果实成熟调控[53]。在番茄中,lncRNA1459影响果实中类胡萝卜素、乙烯以及番茄红素的产生和积累,敲除lncRNA1459导致番茄的成熟期滞后[54]。另外,研究发现沉默番茄lncRNA1840也可以明显延迟番茄果实的成熟[55]。Zuo等[56]通过对甜椒进行表达分析得到多个DE-lncRNAs和DE-mRNAs,发现DE-lncRNAs的靶标包括ERF、bHLH、WRKY、MYB等家族的转录因子,表明其可能参与细胞代谢、果实颜色积累、风味和香气形成以及植物激素信号转导等途径,并开发出与甜椒果实成熟相关的ncRNA和mRNA网络。Ou等[57]也在辣椒中鉴定出2 505个lncRNAs。其中一些靶基因参与植物激素信号转导、细胞壁形成和类胡萝卜素生物合成,表明这些lncRNAs在果实发育和相关代谢物调控中的作用。研究发现,胡萝卜MSTRG.2767和MSTRG.9120分别是花青素合成关键调节因子DcMYB6和DcMYB7的反义lncRNA,MSTRG.9120通过与DcMYB7相互作用激活花青素的生物合成[58]。Tian等[59]在甜瓜中鉴定到可能参与乙烯生物合成代谢以及ABA信号通路并参与果实成熟调节的相关lncRNA:LNC_000987、LNC_000693、LNC_001323、LNC_003610、LNC_00263和LNC_003380。Tang等[60]也在猕猴桃中进行了相关lncRNA的鉴定工作,为进一步研究猕猴桃的果实发育和成熟提供了基础。
除上述所鉴定到的lncRNAs外,还有部分lncRNAs不参与调控果实成熟,而是直接参与到花青素或其他代谢物质的合成过程。对苹果果皮花青苷合成相关lncRNA的表达分析研究发现,在两种不同色相‘富士’中,TCONS_00045933在片红‘富士’中表达量高、在条红‘富士’中表达量低,预测其靶基因为bHLH36,与苹果果皮花青苷的合成有关[61]。马铃薯LINC4206是从紫心马铃薯中克隆得到的lncRNA,其靶向作用于StDFR正调控马铃薯块茎花青素的生物合成[62]。牵牛花中的lncRNAs可以通过不同作用方式对其花色形成进行调控,STRG.13228.2通过顺式作用调控NHX1(INIL10g13210)的表达导致液泡中pH变化,进而改变花色;调控THF1(INIL03g17746)基因的lncRNA rna1878和rna50469在红花中高表达,THF1参与类囊体形成且具有YABBY和MYC的结合位点,YABBY能够参与JA介导的花色苷形成,因此推测rna1878和rna50469通过反式作用和YABBY共同调控THF1表达,影响类囊体发育,导致电子传递链上的光复合体发生异常,进而影响花色苷积累。同时,通过预测发现几个lncRNAs,竞争性结合miRNA157a、miRNA157b、miRNA166和miRNA167影响SBP甲基化来改变牵牛花花色[63]。
庞丹丹等[64]通过苦茶进行全长转录组测序得到778个lncRNAs,推测其在茶树次生代谢的调控中起着特殊作用。对蔗糖处理条件下的茶树进行lncRNA测序,并以MYB家族基因为靶基因筛选得到LTCONS_0005124和LTCONS_0005758,推测其参与调控茶树类黄酮代谢途径[65]。通过分析乌龙茶的转录组数据,以黄酮类代谢途径、萜类代谢途径和JA/MeJA生物合成和信号转导途径为富集途径,富集得到LTCONS_00026271、LTCONS_00020084通过促进相关靶点基因和抑制miRNA的表达从而参与相关代谢物合成及信号转导[66]。Shi等[67]通过对玫瑰RNA-Seq数据进行发掘,发现了可能参与玫瑰花香合成的候选lncRNA TCON_00008447,并使用VIGS方法在‘坦尼可’玫瑰品种中验证了TCON_00008447基因参与玫瑰花香产生的调控。
4.2 参与园艺植物的胁迫响应4.2.1 参与园艺植物的生物胁迫响应 植物在生长发育过程中常常经历生物胁迫,包括细菌、真菌、病毒或害虫等生物的侵害,因此植物自身进化出复杂的调控和防御机制来响应这些胁迫,其中包括lncRNAs参与的调控过程。对香蕉抗病和易感品种,正常及感病(叶斑病ELSD和根腐线虫)条件下的差异表达模式进行分析,发现MUSA-SS-NC945和MUSA-SS-NC913介导的抗真菌蛋白horcolin的下调可能是香蕉品种ELSD敏感性的原因[68]。
番茄晚疫病是一种毁灭性病害,在番茄各个生长阶段普遍发生。Cui等[69] 对晚疫病抗性和易感番茄进行比较转录组分析发现,lncRNA16397可作为SlGRX22的反义转录物来调节其表达,且当lncRNA16397过表达时诱导SlGRX21的表达。在感染Phytophthora infestans 后,过表达lncRNA16397和SpGRX的番茄的病症和活性氧(ROS)积累比野生型更少,表明番茄lncRNA16397通过诱导SlGRX表达,减少ROS的积累并减轻细胞膜损伤,从而增强对致病疫霉的抗性。之后其进一步发现lncRNA33732也是番茄晚疫病的正调节因子,番茄WRKY1激活lncRNA33732,lncRNA33732进一步诱导RBOH表达,增加H2O2在番茄对P. infestans攻击早期防御反应中的积累,增强对晚疫病的抗性[8]。除lncRNA16397、lncRNA33732外,Cui等[69] 又通过差异表达lncRNAs分析发现lncRNA42705和lncRNA08711可能作为ceRNA诱导结合miR159并影响其靶基因,从而提高对番茄晚疫病的抵抗力。同时,该课题组在番茄‘早粉2号’中鉴定到lncRNA39026,其在番茄中竞争性结合miR168a。在番茄中过表达lncRNA39026导致miR168a水平下调,miR168a靶基因SlAGO1水平上调,并提高番茄对致病疫霉的抗性;沉默lncRNA39026得到相反的基因表达情况,且lncRNA39026还可以诱导PRs基因的表达,因此lncRNA39026可能作为诱饵抑制miR168a的作用,并影响PRs基因的表达,增加对番茄晚疫病的抗性[70]。另外,同课题组其他成员又发现番茄miR482b负调控番茄对晚疫病的抗性,对miR482b的内源性靶标模拟物进行鉴定发现lncRNA23468、lncRNA01308和lncRNA13262含有miR482b的保守eTM位点。进一步研究发现lncRNA23468在番茄中过表达时,miR482b的表达显著降低,而靶基因NBS-LRRs的表达显著增加,对致病疫霉的抗性显著增强,表明lncRNA23468-miR482b-NBS-LRR通路参与调节番茄晚疫病抗性的调节[16]。除致病疫霉外,番茄也极易受到灰霉病菌、病毒及根结线虫等的侵害。有益根际细菌可以通过诱导系统抗性(ISR)来抑制病原体的入侵。在番茄中枯草芽孢杆菌SL18r触发ISR对叶片病原体灰葡萄孢的抵抗能力。研究发现SL18r可以诱导MSTRG18363的表达,MSTRG18363中含有miR1918的保守结合位点,其作为诱饵结合miR1918导致SlATL20表达上调,从而激发对病原体感染的ISR反应[71]。在抗病毒方面,在番茄中使用VIGS沉默lncRNA slylnc0049和slylnc0761能够显著降低对番茄黄化曲叶病毒(TYLCV)的抗性[18]。Yang等[72]发现恶臭假单孢菌Sneb821分离株能诱导相关lncRNAs的表达,增强番茄对南方根结线虫的抗性。
在南瓜白粉病(PM)抗病及易感品种中进行差异表达lncRNAs(DELs)的分析中,发现lncRNA MSTRG.28570.4作为miR171c的潜在内源性靶标模拟物响应PM,另外还有6个PM响应性的lncRNA被预测为miRNA的潜在前体[73]。Gao等[74] 在甜瓜中也进行了PM抗病和易感品种的DELs筛选,在PM抗性甜瓜中共发现了387个特异性DELs,对这些DELs及其靶基因进行共表达分析发现LNC_018800与CmWRKY21、LNC_018062与MELO3C015771、LNC_014937与 CmMLO5共表达参与甜瓜对白粉病的响应。甘蓝型油菜中lncRNA通过与mRNA形成共表达调控网络参与油菜根肿病的响应[75]。Kwenda等[76]在马铃薯中发现了17个lincRNAs与12个马铃薯防御相关基因共表达来响应胡萝卜软腐果胶杆菌的侵染。在感染高粱附球菌导致叶斑病的茶树中,通过对lncRNA进行测序得到多个DE-lncRNAs可能参与到病原菌胁迫响应[77]。
4.2.2 参与园艺植物的非生物胁迫响应 园艺植物经常处于不利于生存和生长发育甚至导致其受到伤害、破坏和死亡的环境条件,如低温、高温、干旱及高盐环境。与生物胁迫类似,大量lncRNAs参与到这些胁迫的响应中。对低温胁迫下的葡萄进行lncRNA测序,发现多个DELs靶向胁迫响应相关基因(如CBF4),这些DELs可能参与到葡萄的低温响应[7]。Wang等[17] 对受到冷害胁迫的番茄果实中的DELs进行筛选,共筛选到239个lncRNAs有显著差异表达,并鉴定了大量靶基因,其中许多基因编码冷胁迫相关蛋白,包括氧化还原反应相关酶、细胞壁降解重要酶、膜脂质过氧化相关酶、冷热休克蛋白、能量代谢相关酶、水杨酸和脱落酸代谢相关基因;同时建立了TCONS_00344785、TCONS_00223516、TCONS_00158079、TCONS_00158078、TCONS_00364242参与的番茄低温胁迫下的网络调控。甘蓝型油菜是一种重要的园艺作物,需要低温春化才能开花。通过对自交系RJKB-T24进行转录组测序,发现了549个响应低温处理的lncRNA,同时鉴定到在短期低温诱导条件下参与春化的BrFLC和BrMAF基因的NAT—— MSTRG.2765和MSTRG.14524[78]。Zuo等[79]在常温和冷藏条件下的甜椒中鉴定出380个DELs,它们顺式或反式调控多个参与冷害的靶基因,并因此建立了与甜椒冷藏相关的假定互作RNA网络,表明lncRNA在甜椒冷害过程中具有特定的调控作用。DglncTCP1是菊花DgTCP1反义链上转录而来的lncRNA。DglncTCP1可以作为支架招募组蛋白修饰蛋白DgATX到DgTCP1位点上,提高H3K4me3水平,激活DgTCP1的表达,DgTCP1的表达进一步促进DgPOD基因的上调,减少ROS积累,从而提高菊花的耐寒性[80]。
HILinc1是受到热诱导的梨基因间lncRNA,在梨的耐热性调节中起关键作用。HILinc1靶蛋白1(PbHILT1)是HILinc1的靶转录物。PbHILT1可以与PbHSFA1b结合增强其转录活性,导致热胁迫响应期间下游主要转录调节因子PbMBF1c上调。瞬时过表达HILinc1或PbHILT1均能提高梨的耐热性,表明HILinc1通过稳定PbHSFA1b转录本促进梨的耐热性[81]。对暴露于高温胁迫的黄瓜进行RNA测序,鉴定到TCONS_0031790、TCONS_0001332、TCOND_0001717、TCONS_005674、novel_circ_001543和novel_ circ_00876可能通过植物激素信号转导途径与 miR9748相互作用,以应对高温胁迫[82]。大白菜热响应lncRNA TCONS_0048391是bra-miR164a的eTM,它可能是miRNA结合的海绵(Sponge),也可能是靶基因NAC1(Bra030820)的竞争内源性RNA(ceRNA),影响bra-miR64a在大白菜中的表达,从而调控大白菜的耐热性[83]。
Muthusamy等[24] 在香蕉耐旱品种‘Saba’(ABB) 和易感品种‘Grand Naine’(AAA)中鉴定出905个新的lncRNA,其中2个转录物被鉴定为miRNA-miR156和miR166的前体在香蕉干旱胁迫中发挥关键作用。番茄lncRNA通过lncRNA-mRNA共表达在干旱应答过程中发挥关键作用。番茄潜在mRNA靶点的表达模式和初步功能分析表明,干旱响应lncRNA通过lncRNA-mRNA共表达在多种生物过程及干旱响应调控机制中发挥重要作用[84]。对白菜干旱胁迫响应差异表达lncRNAs鉴定分析中,发现部分已鉴定lncRNAs与非生物胁迫响应相关的转录因子共表达,这些lncRNAs被鉴定为miRNA的推定靶标和内源性靶标模拟物参与对高温和干旱的响应[83]。对茶树耐旱型和易感型品种的差异表达lncRNA进行分析,观察到1 395个干旱特异性lncRNAs;这些干旱特异性lncRNA一部分充当靶标模拟物,一部分作为非生物胁迫响应性miRNA的假定靶标发挥作用,并对干旱胁迫条件下茶树中对干旱响应的lncRNA及其可能功能作用进行了初步鉴定[85]。
在响应盐胁迫方面,Wan等[86]在茶树中鉴定到lncRNA MSTRG.139242.1与TEA027212.1(Ca2+-ATPase 13)基因相互作用,参与Ca2+ 转运,缓解高盐胁迫对茶树细胞的伤害作用,在茶树对高盐度的响应中发挥作用。Li等[87]对野生番茄‘潘那利’和栽培番茄‘M82’中的DELs及其靶基因进行鉴定分析,发现部分基因通过参与脱落酸(ABA)信号通路、油菜素内酯(BR)信号通路、乙烯(ETH)信号通路和抗氧化过程对盐胁迫作出积极响应。同时通过构建盐诱导的lncRNA-mRNA共表达网络,对番茄耐盐性分子机制的进一步研究提供基础。
5 结语与展望LncRNA由于不具备编码蛋白的能力,在最开始被当作基因组的“暗物质”“垃圾DNA”。但随着全基因组RNA测序技术的发展,人们对于lncRNA的认识逐渐清晰。lncRNA虽然不参与功能蛋白的编码,但其在生物体内存在广泛转录,参与生物体内各种生物过程的调控。在lncRNA鉴定初期,主要通过其长度、编码能力等特征的排除法,为更好地对其功能进行研究,研究者们专门为lncRNA开发了计算算法和方法,发明了许多新的工具和技术,创建了许多实用的数据库用于lncRNA的鉴定、功能解剖和比较分析。这些新兴工具的开发极大地推动了lncRNA在真核生物中的研究进展。在最开始的研究过程中,大部分lncRNA的鉴定工作集中在动物基因组中,随后以拟南芥、水稻等模式植物中lncRNA的鉴定工作随之开展,而园艺植物中lncRNA及其功能的鉴定起步较晚。对大部分果树品种,lncRNA的研究集中于果实成熟、花青苷合成导致的果皮颜色变化等生物过程;对更容易受到病虫害等胁迫的蔬菜,lncRNA的研究更多集中于生物及非生物胁迫响应;对花卉及茶树,更多研究则集中于花香、茶香等代谢物合成途径的调控(表 1)。虽然lncRNA在园艺植物中的调控机制已有有限了解,但对lncRNA功能的认识可能只是很小一部分,且lncRNA被错误鉴定的可能性也很高,但lncRNA在植物中悠久而传奇的历史清楚表明其研究很有价值,因此园艺植物lncRNA的鉴定及功能分析将在很长一段时间内维持研究热点。目前为止,过表达、基因沉默(VIGS)、基因敲除(CRISPR/Cas9系统)等技术已初步融入到园艺植物lncRNA的精确功能研究中,在未来,更多新的技术及研究手段,如RNA结构计算分析、单细胞测序、单分子测序等需要融合到园艺植物lncRNA的识别、鉴定及功能分析中,将lncRNA发展成精准育种的有力工具。
| [1] |
WASEEM M, LIU Y, XIA R. Long non-coding RNAs, the dark matter: an emerging regulatory component in plants[J]. International Journal of Molecular Sciences, 2020, 22(1): 86. DOI:10.3390/ijms22010086 |
| [2] |
PACHNIS V, BELAYEW A, TILGHMAN S M. Locus unlinked to alpha-fetoprotein under the control of the murine raf and Rif genes[J]. Proceedings of the National Academy of Sciences of the United States of America, 1984, 81(17): 5523-5527. DOI:10.1073/pnas.81.17.5523 |
| [3] |
VAN DE SANDE K, PAWLOWSKI K, CZAJA I, WIENEKE U, SCHELL J, SCHMIDT J, WALDEN R, MATVIENKO M, WELLINK J, VAN KAMMEN A, FRANSSEN H, BISSELING T. Modification of phytohormone response by a peptide encoded by ENOD40 of legumes and a nonlegume[J]. Science, 1996, 273(5273): 370-373. DOI:10.1126/science.273.5273.370 |
| [4] |
OKAMOTO M, TATEMATSU K, MATSUI A, MOROSAWA T, ISHIDA J, TANAKA M, ENDO T A, MOCHIZUKI Y, TOYODA T, KAMIYA Y, SHINOZAKI K, NAMBARA E, SEKI M. Genome-wide analysis of endogenous abscisic acid-mediated transcription in dry and imbibed seeds of Arabidopsis using tiling arrays[J]. Plant Journal, 2010, 62(1): 39-51. DOI:10.1111/j.1365-313X.2010.04135.x |
| [5] |
MA X, ZHAO F, ZHOU B. The characters of non-coding RNAs and their biological roles in plant development and abiotic stress response[J]. International Journal of Molecular Sciences, 2022, 23(8): 4124. DOI:10.3390/ijms23084124 |
| [6] |
MA H, YANG T, LI Y, ZHANG J, WU T, SONG T, YAO Y, TIAN J. The long noncoding RNA MdLNC499 bridges MdWRKY1 and MdERF109 function to regulate early-stage light-induced anthocyanin accumulation in apple fruit[J]. Plant Cell, 2021, 33(10): 3309-3330. DOI:10.1093/plcell/koab188 |
| [7] |
WANG P, DAI L, AI J, WANG Y, REN F. Identification and functional prediction of cold-related long non-coding RNA (lncRNA) in grapevine[J]. Scientific Reports, 2019, 9(1): 6638. DOI:10.1038/s41598-019-43269-5 |
| [8] |
CUI J, JIANG N, MENG J, YANG G, LIU W, ZHOU X, MA N, HOU X, LUAN Y. LncRNA33732-respiratory burst oxidase module associated with WRKY1 in tomato-Phytophthora infestans interactions[J]. Plant Journal, 2019, 97(5): 933-946. DOI:10.1111/tpj.14173 |
| [9] |
WU J, LIU C, LIU Z, LI S, LI D, LIU S, HUANG X, LIU S, YUKAWA Y. Pol Ⅲ -dependent cabbage BoNR8 long ncRNA affects seed germination and growth in Arabidopsis[J]. Plant and Cell Physiology, 2019, 60(2): 421-435. DOI:10.1093/pcp/pcy220 |
| [10] |
WILUSZ J E, SUNWOO H, SPECTOR D L. Long noncoding RNAs: functional surprises from the RNA world[J]. Genes & Development, 2009, 23(13): 1494-1504. DOI:10.1101/gad.1800909 |
| [11] |
MATTICK J S, AMARAL P P, CARNINCI P, CARPENTER S, CHANG H Y, CHEN L L, CHEN R, DEAN C, DINGER M E, FITZGERALD K A, GINGERAS T R, GUTTMAN M, HIROSE T, HUARTE M, JOHNSON R, KANDURI C, KAPRANOV P, LAWRENCE J B, LEE J T, MENDELL J T, MERCER T R, MOORE K J, NAKAGAWA S, RINN J L, SPECTOR D L, ULITSKY I, WAN Y, WILUSZ J E, WU M. Long non-coding RNAs: definitions, functions, challenges and recommendations[J]. Nature Reviews Molecular Cell Biology, 2023, 24(6): 430-447. DOI:10.1038/s41580-022-00566-8 |
| [12] |
王彬, 陈敏氡, 白昌辉, 林亮, 曾美娟, 叶新如, 刘建汀, 温庆放, 朱海生. 长链非编码RNA在植物生长发育和逆境胁迫响应中的研究进展[J]. 中国细胞生物学学报, 2023, 45(1): 153-163. DOI:10.11844/cjcb.2023.01.0017 WANG B, CHEN M D, BAI C H, LIN L, ZENG M J, YE X R, LIU J T, WEN Q F, ZHU H S. Research progress of long non-coding RNA in plant growth, development and response to environmental stresses[J]. Chinese Journal of Cell Biology, 2023, 45(1): 153-163. DOI:10.11844/cjcb.2023.01.0017 |
| [13] |
LI W, LI C, LI S, PENG M. Long noncoding RNAs that respond to Fusarium oxysporum infection in 'Cavendish' banana (Musa acuminata)[J]. Scientific Reports, 2017, 7(1): 16939. DOI:10.1038/s41598-017-17179-3 |
| [14] |
KANG C, LIU Z. Global identification and analysis of long non-coding RNAs in diploid strawberry Fragaria vesca during flower and fruit development[J]. BMC Genomics, 2015, 16: 815. DOI:10.1186/s12864-015-2014-2 |
| [15] |
ZHANG G, DUAN A, ZHANG J, HE C. Genome-wide analysis of long non-coding RNAs at the mature stage of sea buckthorn (Hippophae rhamnoides Linn.) fruit[J]. Gene, 2017, 596: 130-136. DOI:10.1016/j.gene.2016.10.017 |
| [16] |
JIANG N, CUI J, SHI Y, YANG G, ZHOU X, HOU X, MENG J, LUAN Y. Tomato lncRNA23468 functions as a competing endogenous RNA to modulate NBS-LRR genes by decoying miR482b in the tomato-Phytophthora infestans interaction[J]. Horticulture Research, 2019, 6: 28. DOI:10.1038/s41438-018-0096-0 |
| [17] |
WANG Y, GAO L, ZHU B, ZHU H, LUO Y, WANG Q, ZUO J. Integrative analysis of long non-coding RNA acting as ceRNAs involved in chilling injury in tomato fruit[J]. Gene, 2018, 667: 25-33. DOI:10.1016/j.gene.2018.05.030 |
| [18] |
WANG J, YU W, YANG Y, LI X, CHEN T, LIU T, MA N, YANG X, LIU R, ZHANG B. Genome-wide analysis of tomato long non-coding RNAs and identification as endogenous target mimic for microRNA in response to TYLCV infection[J]. Scientific Reports, 2015, 5: 16946. DOI:10.1038/srep16946 |
| [19] |
WANG A, HU J, GAO C, CHEN G, WANG B, LIN C, SONG L, DING Y, ZHOU G. Genome-wide analysis of long non-coding RNAs unveils the regulatory roles in the heat tolerance of Chinese cabbage (Brassica rapa ssp. chinensis)[J]. Scientific Reports, 2019, 9(1): 5002. DOI:10.1038/s41598-019-41428-2 |
| [20] |
KE L, ZHOU Z, XU X W, WANG X, LIU Y, XU Y, HUANG Y, WANG S, DENG X, CHEN L L, XU Q. Evolutionary dynamics of lincRNA transcription in nine citrus species[J]. Plant Journal, 2019, 98(5): 912-927. DOI:10.1111/tpj.14279 |
| [21] |
BOERNER S, MCGINNIS K M. Computational identification and functional predictions of long noncoding RNA in Zea mays[J]. PLoS One, 2012, 7(8): e43047. DOI:10.1371/journal.pone.0043047 |
| [22] |
WANG Y, GAO L, LI J, ZHU B, ZHU H, LUO Y, WANG Q, ZUO J. Analysis of long-non-coding RNAs associated with ethylene in tomato[J]. Gene, 2018, 674: 151-160. DOI:10.1016/j.gene.2018.06.089 |
| [23] |
LI S, CHENG Z, DONG S, LI Z, ZOU L, ZHAO P, GUO X, BAO Y, WANG W, PENG M. Global identification of full-length cassava lncRNAs unveils the role of cold-responsive intergenic lncRNA 1 in cold stress response[J]. Plant, Cell & Environment, 2022, 45(2): 412-426. DOI:10.1111/pce.14236 |
| [24] |
MUTHUSAMY M, UMA S, BACKIYARANI S, SARASWATHI M S. Genome-wide screening for novel, drought stress-responsive long non-coding RNAs in drought-stressed leaf transcriptome of drought-tolerant and -susceptible banana (Musa spp.) cultivars using Illumina high-throughput sequencing[J]. Plant Biotechnology Reports, 2015, 9(5): 279-286. DOI:10.1007/s11816-015-0363-6 |
| [25] |
WANG Y, FAN X, LIN F, HE G, TERZAGHI W, ZHU D, DENG X W. Arabidopsis noncoding RNA mediates control of photomorphogenesis by red light[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(28): 10359-10364. DOI:10.1073/pnas.1409457111 |
| [26] |
YU T, TZENG D T W, LI R, CHEN J, ZHONG S, FU D, ZHU B, LUO Y, ZHU H. Genome-wide identification of long non-coding RNA targets of the tomato MADS-box transcription factor RIN and function analysis[J]. Annals of Botany, 2019, 123(3): 469-482. DOI:10.1093/aob/mcy178 |
| [27] |
RAMÍREZ GONZALES L, SHI L, BERGONZI S B, OORTWIJN M, FRANCO-ZORRILLA J M, SOLANO-TAVIRA R, VISSER R G F, ABELENDA J A, BACHEM C W B. Potato CYCLING DOF FACTOR 1 and its lncRNA counterpart StFLORE link tuber development and drought response[J]. Plant Journal, 2021, 105(4): 855-869. DOI:10.1111/tpj.15093 |
| [28] |
PALOS K, YU L, RAILEY C E, NELSON DITTRICH A C, NELSON A D L. Linking discoveries, mechanisms, and technologies to develop a clearer perspective on plant long noncoding RNAs[J]. Plant Cell, 2023, 35(6): 1762-1786. DOI:10.1093/plcell/koad027 |
| [29] |
BARDOU F, ARIEL F, SIMPSON C G, ROMERO-BARRIOS N, LAPORTE P, BALZERGUE S, BROWN J W, CRESPI M. Long noncoding RNA modulates alternative splicing regulators in Arabidopsis[J]. Developmental Cell, 2014, 30(2): 166-176. DOI:10.1016/j.devcel.2014.06.017 |
| [30] |
RIGO R, BAZIN J, ROMERO-BARRIOS N, MOISON M, LUCERO L, CHRIST A, BENHAMED M, BLEIN T, HUGUET S, CHARON C, CRESPI M, ARIEL F. The Arabidopsis lncRNA ASCO modulates the transcriptome through interaction with splicing factors[J]. Embo Reports, 2020, 21(5): e48977. DOI:10.15252/embr.201948977 |
| [31] |
JABNOUNE M, SECCO D, LECAMPION C, ROBAGLIA C, SHU Q, POIRIER Y. A rice cis-natural antisense RNA acts as a translational enhancer for its cognate mRNA and contributes to phosphate homeostasis and plant fitness[J]. Plant Cell, 2013, 25(10): 4166-4182. DOI:10.1105/tpc.113.116251 |
| [32] |
ZUCCHELLI S, COTELLA D, TAKAHASHI H, CARRIERI C, CIMATTI L, FASOLO F, JONES M H, SBLATTERO D, SANGES R, SANTORO C, PERSICHETTI F, CARNINCI P, GUSTINCICH S. SINEUPs: A new class of natural and synthetic antisense long non-coding RNAs that activate translation[J]. RNA Biology, 2015, 12(8): 771-779. DOI:10.1080/15476286.2015.1060395 |
| [33] |
LAPORTE P, SATIAT-JEUNEMAÎTRE B, VELASCO I, CSORBA T, VAN DE VELDE W, CAMPALANS A, BURGYAN J, AREVALO-RODRIGUEZ M, CRESPI M. A novel RNA-binding peptide regulates the establishment of the Medicago truncatula-Sinorhizobium meliloti nitrogen-fixing symbiosis[J]. Plant Journal, 2010, 62(1): 24-38. DOI:10.1111/j.1365-313X.2009.04121.x |
| [34] |
ARIEL F, LUCERO L, CHRIST A, MAMMARELLA M F, JEGU T, VELUCHAMY A, MARIAPPAN K, LATRASSE D, BLEIN T, LIU C, BENHAMED M, CRESPI M. R-loop mediated trans action of the APOLO long noncoding RNA[J]. Molecular Cell, 2020, 77(5): 1055-1065. DOI:10.1016/j.molcel.2019.12.015 |
| [35] |
ZHANG J, MUJAHID H, HOU Y, NALLAMILLI B R, PENG Z. Plant long ncRNAs: a new frontier for gene regulatory control[J]. American Journal of Plant Sciences, 2013, 4(5): 1038-1045. |
| [36] |
HELLIWELL C A, ROBERTSON M, FINNEGAN E J, BUZAS D M, DENNIS E S. Vernalization-repression of Arabidopsis FLC requires promoter sequences but not antisense transcripts[J]. PLoS One, 2011, 6(6): e21513. DOI:10.1371/journal.pone.0021513 |
| [37] |
WU J, OKADA T, FUKUSHIMA T, TSUDZUKI T, SUGIURA M, YUKAWA Y. A novel hypoxic stress-responsive long non-coding RNA transcribed by RNA polymerase Ⅲ in Arabidopsis[J]. RNA Biology, 2012, 9(3): 302-313. DOI:10.4161/rna.19101 |
| [38] |
杨贺, 张楠, 刘自广, 林建辉, 孙世臣, 刘圣怡, 岑曦, 吴娟. 低温胁迫下结球甘蓝BoNR8 lncRNA过表达对拟南芥种子萌发的影响[J]. 植物研究, 2020, 40(3): 441-446. DOI:10.7525/j.issn.1673-5102.2020.03.016 YANG H, ZHANG N, LIU Z G, LIN J H, SUN S C, LIU S Y, CEN X, WU J. Expression and function of BoNR8 lncRNA in cabbage under cold stress[J]. Bulletin of Botanical Research, 2020, 40(3): 441-446. DOI:10.7525/j.issn.1673-5102.2020.03.016 |
| [39] |
岑曦, 李丹丹, 刘自广, 刘春晓, 杨贺, 黄小庆, 李爽, 汤川泰, 吴娟. 白菜中BrpR8长非编码RNA表达特性[J]. 基因组学与应用生物学, 2019, 38(10): 4641-4646. DOI:10.13417/j.gab.038.004641 CEN X, LI D D, LIU Z G, LIU C X, YANG H, HUANG X Q, LI S, TANG C T, WU J. Expression characteristics of BrpR8 long non-coding RNA in Chinese cabbage[J]. Genomics and Applied Biology, 2019, 38(10): 4641-4646. DOI:10.13417/j.gab.038.004641 |
| [40] |
张楠, 刘自广, 彭疑芳, 张晓旭, 严容, 杨东旭, 黄迪, 高新萍, 马丹阳, 吴娟. 结球甘蓝BoNR8 lncRNA光响应及调节下胚轴生长的作用[J]. 植物生理学报, 2023, 59(1): 172-178. DOI:10.13592/j.cnki.ppj.100437 ZHANG N, LIU Z G, PENG Y F, ZHANG X X, YAN R, YANG D X, HUANG D, GAO X P, MA D Y, WU J. Light response of BoNR8 lncRNA in Brassica oleracea and its role in regulating hypocotyl growth[J]. Plant Physiology Journal, 2023, 59(1): 172-178. DOI:10.13592/j.cnki.ppj.100437 |
| [41] |
YANG Z, YANG C, WANG Z, YANG Z, CHEN D, WU Y. LncRNA expression profile and ceRNA analysis in tomato during flowering[J]. PLoS One, 2019, 14(1): e0210650. DOI:10.1371/journal.pone.0210650 |
| [42] |
张艺勇. 龙眼花芽诱导过程内源激素、mRNA-lncRNA转录组分析及TFL1启动子研究[D]. 福州: 福建农林大学, 2022. ZHANG Y Y. Study on endogenous hormones, mRNA-lncRNA transcriptomes and TFL1 promoter during floral bud induction in longan (Dimocarpus longan Lour. )[D]. Fuzhou: Fujian Agriculture and Forestry University, 2022. |
| [43] |
HUANG L, DONG H, ZHOU D, LI M, LIU Y, ZHANG F, FENG Y, YU D, LIN S, CAO J. Systematic identification of long non-coding RNAs during pollen development and fertilization in Brassica rapa[J]. Plant Journal, 2018, 96(1): 203-222. DOI:10.1111/tpj.14016 |
| [44] |
SONG J H, CAO J S, WANG C G. BcMF11, a novel non-coding RNA gene from Brassica campestris, is required for pollen development and male fertility[J]. Plant Cell Reports, 2013, 32(1): 21-30. DOI:10.1007/s00299-012-1337-6 |
| [45] |
韩田雨. 白菜BrMYB80b正义lncRNA在育性调控中的作用探索[D]. 杭州: 浙江大学, 2017. HAN T Y. The exploration of BrMYB80b sense lncRNA in regulation of fertility in Brassica rapa[D]. Hangzhou: Zhejiang University, 2017. |
| [46] |
CHO J, KOO D H, NAM Y W, HAN C T, LIM H T, BANG J W, HUR Y. Isolation and characterization of cDNA clones expressed under male sex expression conditions in a monoecious cucumber plant (Cucumis sativus L. cv. Winter Long)[J]. Euphytica, 2006, 146(3): 271-281. DOI:10.1007/s10681-005-9023-1 |
| [47] |
LYU J, LIU Z, YANG B, DENG M, WANG J, LIU Y, ZHANG Z, MA Y, CHEN W, OU L, ZOU X. Systematic identification and characterization 23 of long non-coding RNAs involved in cytoplasmic male sterility in pepper (Capsicum annuum L.)[J]. Plant Growth Regulation, 2020, 91(2): 277-288. DOI:10.1007/s10725-020-00605-4 |
| [48] |
WU X, SHI T, IQBAL S, ZHANG Y, LIU L, GAO Z. Genome-wide discovery and characterization of flower development related long non-coding RNAs in Prunus mume[J]. BMC Plant Biology, 2019, 19(1): 64. DOI:10.1186/s12870-019-1672-7 |
| [49] |
BAI Y, LIU M, ZHOU R, JIANG F, LI P, LI M, ZHANG M, WEI H, WU Z. Construction of ceRNA networks at different stages of somatic embryogenesis in Garlic[J]. International Journal of Molecular Sciences, 2023, 24(6): 5311. DOI:10.3390/ijms24065311 |
| [50] |
TANG Y, QU Z, LEI J, HE R, ADELSON D L, ZHU Y, YANG Z, WANG D. The long noncoding RNA FRILAIR regulates strawberry fruit ripening by functioning as a noncanonical target mimic[J]. PLoS Genetics, 2021, 17(3): e1009461. DOI:10.1371/journal.pgen.1009461 |
| [51] |
ZHANG G, CHEN D, ZHANG T, DUAN A, ZHANG J, HE C. Transcriptomic and functional analyses unveil the role of long non-coding RNAs in anthocyanin biosynthesis during sea buckthorn fruit ripening[J]. DNA Research, 2018, 25(5): 465-476. DOI:10.1093/dnares/dsy017 |
| [52] |
YANG T, MA H, ZHANG J, WU T, SONG T, TIAN J, YAO Y. Systematic identification of long noncoding RNAs expressed during light-induced anthocyanin accumulation in apple fruit[J]. Plant Journal, 2019, 100(3): 572-590. DOI:10.1111/tpj.14470 |
| [53] |
BHATIA G, SHARMA S, UPADHYAY S K, SINGH K. Long non-coding RNAs coordinate developmental transitions and other key biological processes in Grapevine[J]. Scientific Reports, 2019, 9(1): 3552. DOI:10.1038/s41598-019-38989-7 |
| [54] |
LI R, FU D, ZHU B, LUO Y, ZHU H. CRISPR/Cas9-mediated mutagenesis of lncRNA1459 alters tomato fruit ripening[J]. Plant Journal, 2018, 94(3): 513-524. DOI:10.1111/tpj.13872 |
| [55] |
ZHU B, YANG Y, LI R, FU D, WEN L, LUO Y, ZHU H. RNA sequencing and functional analysis implicate the regulatory role of long non-coding RNAs in tomato fruit ripening[J]. Journal of Experimental Botany, 2015, 66(15): 4483-4495. DOI:10.1093/jxb/erv203 |
| [56] |
ZUO J, WANG Y, ZHU B, LUO Y, WANG Q, GAO L. Network analysis of noncoding RNAs in pepper provides insights into fruit ripening control[J]. Scientific Reports, 2019, 9(1): 8734. DOI:10.1038/s41598-019-45427-1 |
| [57] |
OU L, LIU Z, ZHANG Z, WEI G, ZHANG Y, KANG L, YANG B, YANG S, LV J, LIU Y, CHEN W, DAI X, LI X, ZHOU S, MA Y, ZOU X. Noncoding and coding transcriptome analysis reveals the regulation roles of long noncoding RNAs in fruit development of hot pepper (Capsicum annuum L.)[J]. Plant Growth Regulation, 2017, 83(1): 141-156. DOI:10.1007/s10725-017-0290-3 |
| [58] |
CHIALVA C, BLEIN T, CRESPI M, LIJAVETZKY D. Insights into long non-coding RNA regulation of anthocyanin carrot root pigmentation[J]. Scientific Reports, 2021, 11(1): 4093. DOI:10.1038/s41598-021-83514-4 |
| [59] |
TIAN Y, BAI S, DANG Z, HAO J, ZHANG J, HASI A. Genome-wide identification and characterization of long non-coding RNAs involved in fruit ripening and the climacteric in Cucumis melo[J]. BMC Plant Biology, 2019, 19(1): 369. DOI:10.1186/s12870-019-1942-4 |
| [60] |
TANG W, ZHENG Y, DONG J, YU J, YUE J, LIU F, GUO X, HUANG S, WISNIEWSKI M, SUN J, NIU X, DING J, LIU J, FEI Z, LIU Y. Comprehensive transcriptome profiling reveals long noncoding RNA expression and alternative splicing regulation during fruit development and ripening in kiwifruit (Actinidia chinensis)[J]. Frontiers in Plant Science, 2016, 7: 335. DOI:10.3389/fpls.2016.00335 |
| [61] |
盖艺. 苹果果皮花青苷合成相关lncRNA的表达分析及研究[D]. 烟台: 烟台大学, 2021. GAI Y. Expression of anthocyanin biosynthesis-related lncRNA in apple skin[D]. Yantai: Yantai University, 2021. |
| [62] |
吴万亿, 张毅, 张洁, 董静静, 唐锐敏, 贾小云, 张红梅. 马铃薯LINC4206及其靶基因StDFR的克隆与StDFR的生物信息学分析[J]. 山西农业科学, 2021, 49(6): 683-688. DOI:10.3969/j.issn.1002-2481.2021.06.01 WU W Y, ZHANG Y, ZHANG J, DONG J J, TANG R M, JIA X Y, ZHANG H M. Cloning of LINC4206 and its target gene StDFR and bioinformatics analysis of StDFR in potato[J]. Journal of Shanxi Agricultural Sciences, 2021, 49(6): 683-688. DOI:10.3969/j.issn.1002-2481.2021.06.01 |
| [63] |
周翰林. LncRNA调控牵牛花色的机制研究[D]. 宜昌: 三峡大学, 2022. ZHOU H L. Study on the mechanism of lncRNA regulation of morning glory flower color[D]. Yichang: China Three Gorges University, 2022. |
| [64] |
庞丹丹, 刘玉飞, 孙云南, 田易萍, 宋维希, 陈林波. 苦茶全长转录组测序及基因结构分析[J]. 西北农业学报, 2021, 30(4): 563-571. DOI:10.7606/j.issn.1004-1389.2021.04.011 PANG D D, LIU Y F, SUN Y N, TIAN Y P, SONG W X, CHEN L B. Full-length transcriptome and gene structure analysis of Kucha (Camellia sinensis)[J]. Acta Agriculturae Boreali-occidentalis Sinica, 2021, 30(4): 563-571. DOI:10.7606/j.issn.1004-1389.2021.04.011 |
| [65] |
韩亚惠. 参与茶树类黄酮代谢的编码和非编码RNA的研究[D]. 合肥: 安徽农业大学, 2018. HAN Y H. Study on coding and non coding RNAs involving the flavonoid metabolism in the tea plant (Camellia sinensis)[D]. Hefei: Anhui Agricultural University, 2018. |
| [66] |
ZHU C, ZHANG S, FU H, ZHOU C, CHEN L, LI X, LIN Y, LAI Z, GUO Y. Transcriptome and phytochemical analyses provide new insights into long non-coding RNAs modulating characteristic secondary metabolites of Oolong tea (Camellia sinensis) in solar-withering[J]. Frontiers in Plant Science, 2019, 10: 1638. DOI:10.3389/fpls.2019.01638 |
| [67] |
SHI S, ZHANG S, WU J, LIU X, ZHANG Z. Identification of long non-coding RNAs involved in floral scent of Rosa hybrida[J]. Frontiers in Plant Science, 2022, 13: 996474. DOI:10.3389/fpls.2022.996474 |
| [68] |
MUTHUSAMY M, UMA S, SUTHANTHIRAM B, SARASWATHI M S, CHANDRASEKAR A. Genome-wide identification of novel, long non-coding RNAs responsive to Mycosphaerella eumusae and Pratylenchus coffeae infections and their differential expression patterns in disease-resistant and sensitive banana cultivars[J]. Plant Biotechnology Reports, 2019, 13(1): 73-83. DOI:10.1007/s11816-018-00514-z |
| [69] |
CUI J, JIANG N, HOU X, WU S, ZHANG Q, MENG J, LUAN Y. Genome-wide identification of lncRNAs and analysis of ceRNA networks during tomato resistance to Phytophthora infestans[J]. Phytopathology, 2020, 110(2): 456-464. DOI:10.1094/phyto-04-19-0137-r |
| [70] |
HOU X, CUI J, LIU W, JIANG N, ZHOU X, QI H, MENG J, LUAN Y. LncRNA39026 enhances tomato resistance to Phytophthora infestans by decoying miR168a and inducing PR gene expression[J]. Phytopathology, 2020, 110(4): 873-880. DOI:10.1094/phyto-12-19-0445-r |
| [71] |
ZHOU C, ZHU J, QIAN N, GUO J, YAN C. Bacillus subtilis SL18r induces tomato resistance against Botrytis cinerea, involving activation of long non-coding RNA, MSTRG18363, to decoy miR1918[J]. Frontiers in Plant Science, 2020, 11: 634819. DOI:10.3389/fpls.2020.634819 |
| [72] |
YANG F, ZHAO D, FAN H, ZHU X, WANG Y, LIU X, DUAN Y, XUAN Y, CHEN L. Functional analysis of long non-coding RNAs reveal their novel roles in biocontrol of bacteria-induced tomato resistance to Meloidogyne incognita[J]. International Journal of Molecular Sciences, 2020, 21(3): 911. DOI:10.3390/ijms21030911 |
| [73] |
TIAN J, ZHANG G, ZHANG F, MA J, WEN C, LI H. Genome-wide identification of powdery mildew responsive long non-coding RNAs in Cucurbita pepo[J]. Frontiers in Genetics, 2022, 13: 933022. DOI:10.3389/fgene.2022.933022 |
| [74] |
GAO C, SUN J, DONG Y, WANG C, XIAO S, MO L, JIAO Z. Comparative transcriptome analysis uncovers regulatory roles of long non-coding RNAs involved in resistance to powdery mildew in melon[J]. BMC Genomics, 2020, 21(1): 125. DOI:10.1186/s12864-020-6546-8 |
| [75] |
ZHU H, LI X, XI D, ZHAI W, ZHANG Z, ZHU Y. Integrating long noncoding RNAs and mRNAs expression profiles of response to Plasmodiophora brassicae infection in Pakchoi (Brassica campestris ssp. chinensis Makino)[J]. PLoS One, 2019, 14(12): e0224927. DOI:10.1371/journal.pone.0224927 |
| [76] |
KWENDA S, BIRCH P R, MOLELEK I L N. Genome-wide identification of potato long intergenic noncoding RNAs responsive to Pectobacterium carotovorum subspecies brasiliense infection[J]. BMC Genomics, 2016, 17(1): 614. DOI:10.1186/s12864-016-2967-9 |
| [77] |
HUANG C, HUANG H, XIA Z, YANG Y, JIANG X, YANG Y, WANG D, LI X, CHEN Z. Sequence Data, functional annotation, and relationship analysis between mRNAs and long noncoding RNAs from tea leaves during infection by the fungal pathogen Epicoccum sorghinum[J]. Molecular Plant-Microbe Interactions, 2022, 35(9): 875-879. DOI:10.1094/mpmi-02-22-0048-a |
| [78] |
SHEA D J, NISHIDA N, TAKADA S, ITABASHI E, TAKAHASHI S, AKTER A, MIYAJI N, OSABE K, MEHRAJ H, SHIMIZU M, SEKI M, KAKIZAKI T, OKAZAKI K, DENNIS E S, FUJIMOTO R. Long noncoding RNAs in Brassica rapa L. following vernalization[J]. Scientific Reports, 2019, 9(1): 9302. DOI:10.1038/s41598-019-45650-w |
| [79] |
ZUO J, WANG Y, ZHU B, LUO Y, WANG Q, GAO L. Analysis of the coding and non-coding RNA transcriptomes in response to bell pepper chilling[J]. International Journal of Molecular Sciences, 2018, 19(7): 2001. DOI:10.3390/ijms19072001 |
| [80] |
LI X, YANG Q, LIAO X, TIAN Y, ZHANG F, ZHANG L, LIU Q. A natural antisense RNA improves chrysanthemum cold tolerance by regulating the transcription factor DgTCP1[J]. Plant Physiology, 2022, 190(1): 605-620. DOI:10.1093/plphys/kiac267 |
| [81] |
ZHANG Y, WANG S, LI W, WANG S, HAO L, XU C, YU Y, XIANG L, LI T, JIANG F. A long noncoding RNA HILinc1 enhances pear thermotolerance by stabilizing PbHILT1 transcripts through complementary base pairing[J]. Communications Biology, 2022, 5(1): 1134. DOI:10.1038/s42003-022-04010-7 |
| [82] |
HE X, GUO S, WANG Y, WANG L, SHU S, SUN J. Systematic identification and analysis of heat-stress-responsive lncRNAs, circRNAs and miRNAs with associated co-expression and ceRNA networks in cucumber (Cucumis sativus L.)[J]. Physiologia Plantarm, 2020, 168(3): 736-754. DOI:10.1111/ppl.12997 |
| [83] |
BHATIA G, SINGH A, VERMA D, SHARMA S, SINGH K. Genome-wide investigation of regulatory roles of lncRNAs in response to heat and drought stress in Brassica juncea (Indian mustard)[J]. Environmental and Experimental Botany, 2020, 171: 103922. DOI:10.1016/j.envexpbot.2019.103922 |
| [84] |
EOM S H, LEE H J, LEE J H, WI S H, KIM S K, HYUN T K. Identification and functional prediction of drought-responsive long non-coding RNA in tomato[J]. Agronomy, 2019, 9(10): 629. DOI:10.3390/agronomy9100629 |
| [85] |
BARUAH P M, KASHYAP P, KRISHNATREYA D B, BORDOLOI K S, GILL S S, AGARWALA N. Identification and functional analysis of drought responsive lncRNAs in tea plant[J]. Plant Genome, 2021, 27: 100311. DOI:10.1016/j.plgene.2021.100311 |
| [86] |
WAN S, ZHANG Y, DUAN M, HUANG L, WANG W, XU Q, YANG Y, YU Y. Integrated analysis of long non-coding RNAs (lncRNAs) and mRNAs reveals the regulatory role of lncRNAs associated with salt resistance in Camellia sinensis[J]. Frontiers in Plant Science, 2020, 11: 218. DOI:10.3389/fpls.2020.00218 |
| [87] |
LI N, WANG Z, WANG B, WANG J, XU R, YANG T, HUANG S, WANG H, YU Q. Identification and characterization of long non-coding RNA in tomato roots under salt stress[J]. Frontiers in Plant Science, 2022, 13: 834027. DOI:10.3389/fpls.2022.834027 |
(责任编辑 张辉玲)
 2023, Vol. 50
2023, Vol. 50