文章信息
基金项目
- 重庆市自然科学基金创新群体项目(cstc2021jcyj-cxttX0005)
作者简介
- 李晨曦(1998—),男,在读硕士生,研究方向为病原微生物学,E-mail:1303675733@qq.com.
通讯作者
- 龙梦娴(1984—),女,博士,副教授,研究方向为病原微生物学,E-mail:longmx@swu.edu.cn.
文章历史
- 收稿日期:2023-05-09
2. 西南大学蚕桑纺织与生物质科学学院,重庆 400715
2. College of Sericulture, Textile and Biomass Sciences, Southwest University, Chongqing 400715, China
【研究意义】凡纳滨对虾原产于太平洋沿岸,目前是我国主要的对虾养殖品种之一,年产量可高达百万吨[1]。但凡纳滨对虾常遭受病毒、细菌和真菌等病原感染,给全球对虾养殖业造成严重经济损失[2]。2004年泰国新发现了一种对虾病原,2009年鉴定为微孢子虫,命名为虾肝肠胞虫(Enterocytozoon hepatopenaei,EHP)。EHP主要感染对虾肝胰腺,通过影响宿主免疫和代谢,抑制对虾正常生长发育甚至危及存活率[3-5]。2013年我国首次检出EHP,至今已传播至各大养殖区,极大地威胁国内对虾养殖业的健康发展[6-8]。迄今已发现200多属、1 700多种微孢子虫[9-11],它们具有相似且特殊的侵染方式,即适宜环境条件下,刺激微孢子虫发芽,极管迅速弹出,病原性孢原质通过此中空管道运输至宿主细胞内。研究发现,水通道蛋白参与激活微孢子虫发芽[12-14]。为有效防控虾肝肠胞虫,揭示其侵染机制显得尤为重要。【前人研究进展】水通道蛋白作为细胞膜上的跨膜蛋白,参与水和小分子溶质跨膜的双向运输,几乎存在于所有生物中。水通道蛋白以四聚体形式存在,每个单体都会形成具有高度选择性的中空通道以运输合适分子。目前,已发现300多种不同的水通道蛋白,其中人的水通道蛋白有13种亚型,分别是AQP0~AQP12[15]。水通道蛋白由6个α-螺旋跨膜结构域组成,具有2个保守基序NPA(Asn-Pro-Ala)、5个环结构(A~E环),汞离子通过作用于E环上NPA基序前的半胱氨酸位点可逆地抑制水通道蛋白功能[16-17]。Frixione等[14]利用HgCl2处理按蚊微孢子虫(Nosema algerae),发现HgCl2可显著抑制按蚊微孢子虫发芽,推测水通道蛋白参与按蚊微孢子虫的发芽过程。2006年Ghosh等[18]从兔脑炎微孢子虫(Encephalitozoon cuniculi)中鉴定到首个微孢子虫水通道蛋白EcAQP,其具有保守的水通道蛋白序列特征,将其编码基因转染至非洲爪蟾卵母细胞中异源表达,发现该卵母细胞的通透性显著增加,表明EcAQP具有促进水渗透的功能。Chen等[19]从家蚕微孢子虫(Nosema bombycis)基因组中克隆得到NbAQP,发现NbAQP蛋白定位于成熟孢子的孢壁,并且NbAQP抗体能有效抑制孢子发芽率,进一步证实了水通道蛋白在微孢子虫发芽中发挥重要功能。【本研究切入点】目前虾肝肠胞虫全基因序列测序分析已经完成[20],但有关EHP水通道蛋白的研究尚未见报道。鉴于此,本研究以虾肝肠胞虫水通道蛋白EHP00_492为试验对象,对其编码基因进行克隆,预测其蛋白质的序列与结构特征,分析该蛋白在EHP成熟孢子中的表达定位特征。【拟解决的关键问题】本研究从虾肝肠胞虫EHP的cDNA中克隆EHP00_492基因;通过生物信息学方法分析其编码蛋白的序列特征、三维结构特征及其与同源蛋白间的亲缘关系;构建重组表达载体pCold-TF-EHP00_492,转化大肠杆菌异源表达,纯化重组蛋白并制备多克隆抗体;分析EHP00_492在EHP成熟孢子中的表达及亚细胞定位特征,以期为EHP水通道蛋白的功能研究奠定基础。
1 材料与方法 1.1 试验材料试验于2021—2022年在西南大学资源昆虫高效养殖与利用全国重点实验室开展。供试的感染EHP与未感染EHP的凡纳滨对虾均购自重庆市合川区和铜梁区的养殖池塘,新西兰实验兔购自重庆恩斯维尔生物科技有限公司;大肠杆菌Escherichia coli DH5α和Rosetta感受态细胞购自重庆灵精生物科技有限公司,pCold-TF表达质粒由本实验室保存;PrimeSTAR DNA聚合酶、Xho I和Pst I限制性内切酶、IPTG、脱脂奶粉购自Takara Bio公司,425-600 μm玻璃珠、弗氏佐剂、DAPI细胞核染料、HRP标记山羊抗兔IgG、Triton X-100购自Sigma-Aldrich公司,Alexa Fluor® 594山羊抗兔荧光二抗购自Invitrogen公司,TF-tag兔多克隆抗体购自迅检(重庆)生命科技有限责任公司;DNA胶回收试剂盒、Total RNA提取试剂盒、质粒抽提试剂盒购自Omega BioTek公司,一步法快速克隆试剂盒购自上海翊圣生物公司;PMSF、考马斯亮蓝染色液、Wheat Germ Agglutinin(WGA-488)购自碧云天生物技术有限公司。
1.2 试验方法1.2.1 引物设计与合成 根据虾肝肠胞虫水通道蛋白编码基因EHP00_492序列(GenBank No. MNPJ01000023.1: 89 904~90 632,729 bp),使用Primer 5.0软件设计特异性引物。用于RT-PCR的引物对,EHP00_492-F1序列:5'-ATGCAAGTCAGTAAAAAATTAATAG-3',EHP00_492-R1序列:5'-TTATTTAAACAACAAAAATACTTG-3';用于克隆的引物对,EHP00_492-F2序列:5'-GGCATATGGAGCTCGGTACCCTCGAGATGCAAGTCAGTAAAAAATTAATAG-3'(下划线为XhoI酶切位点),EHP00_492-R2序列:5'-GCAGAGATTACCTATCTAGACTGCAGTTATTTAAACAACAAAAATACTTG-3'(下划线为PstI酶切位点)。引物由生工生物工程(上海)股份有限公司合成。
1.2.2 RNA提取和RT-PCR 采用RT-PCR方法克隆EHP00_492基因。取约1×106个EHP孢子,按照RNA提取试剂盒说明书提取RNA,并利用逆转录试剂盒将总RNA逆转录为cDNA。以cDNA为模板、EHP00_492-F1/R1为引物,通过PCR扩增EHP00_492基因。PCR反应体系:PrimerSTAR DNA(2×)聚合酶25 μL,引物0.2 μmol/L,cDNA 1 µL,ddH2O补足至50 µL。PCR反应条件:98 ℃ 3 min;98 ℃ 20 s、50 ℃ 20 s、72 ℃ 10 s,35个循环;72 ℃ 10 min。采用1% 琼脂糖凝胶电泳分析PCR产物,并送生工生物工程(上海)股份有限公司进行测序。
1.2.3 生物信息学分析 通过NCBI(http://ncbi.nlm.nih.gov/)完成序列检索并利用微孢子虫数据库(https://microsporidiadb.org/micro/app/)进行基因座位分析;利用ExPASy(https://web.expasy.org/protparam/)进行分子量、氨基酸数量和等电点等特征预测;利用SignalP 5.0(http://www.cbs.dtu.dk/services/SignalP/)进行蛋白信号肽预测;利用NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/)进行蛋白潜在磷酸化位点预测;利用PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)进行蛋白二级结构预测;利用SMART(http://smart.embl-heidelberg.de/)进行蛋白质功能域预测;利用TMHMM(http://www.cbs.dtu.dk/services/TMHMM-2.0/)进行跨膜结构域预测;利用MAFFT(https://mafft.cbrc.jp/alignment/server/index.html)进行微孢子虫与不同物种之间水通道蛋白的多重序列比对;利用AlphaFold软件进行蛋白质三维结构预测与比对;利用MEGA 7.0软件基于Neighbor-Joining法构建系统发育树。
1.2.4 EHP00_492重组质粒构建 将pCold-TF载体质粒进行XhoI和PstI双酶切,酶切反应体系:限制性内切酶各3.5 µL,CutSmart buffer 5 µL,模板1~5 µg,ddH2O补足至50 µL。酶切反应条件:37 ℃下孵育20 min。将酶切产物进行琼脂糖凝胶电泳并割胶回收。使用EHP00_492-F2/R2引物扩增EHP00_492片段,反应体系:PrimerSTAR DNA聚合酶25 µL,引物0.2 µmol/L,模板1 µL,ddH2O补足至50 µL。反应条件:98 ℃ 3 min;98 ℃ 20 s、50 ℃ 20 s、72 ℃ 10 s,35个循环;72 ℃ 10 min,扩增产物进行琼脂糖凝胶电泳并割胶回收。根据一步法克隆试剂盒的操作说明将157 4 µL载体与6 µL外源片段进行连接,连接酶10 µL,50 ℃孵育20 min。将连接产物转化至E. coli DH5α感受态细胞中,并涂布于含有氨苄青霉素的LB培养基过夜培养。挑取单菌落提取质粒进行PCR和双酶切验证,并送至生工生物工程(上海)股份有限公司测序。
1.2.5 EHP00_492异源蛋白表达、纯化及其兔多克隆抗体制备 将测序正确的pCold-TF-EHP00_492重组质粒转化至表达菌株E. coli Rosetta中,添加0.5 mmol/L IPTG诱导,收集菌体进行破碎,使用Ni-NTA亲和层析柱纯化rEHP00_492重组蛋白。纯化后的rEHP00_492蛋白与佐剂混合后免疫新西兰实验兔,制备兔多克隆抗体。
1.2.6 EHP孢子总蛋白提取 取约1×106个EHP成熟孢子,加入1×PBS 300 µL、PMSF 20 µL和425~600 μm玻璃珠0.4 g混合进行破碎,破碎条件:6 m/s,30 s/轮,6轮/次,每轮间歇10 s,共破碎3次。离心后的上清即为EHP孢子总蛋白。
1.2.7 蛋白免疫印记(Western blot) 将EHP孢子总蛋白进行SDS-PAGE电泳后,电转至PVDF膜。将PVDF膜于37 ℃下封闭1~2 h;分别利用EHP00_492兔多克隆抗体和TF-tag兔多克隆抗体室温下孵育PVDF膜1 h,利用羊抗兔IgG二抗室温下孵育PVDF膜45 min。采用化学发光成像仪(Azure Biosystems C300)对PVDF膜进行曝光、成像。
1.2.8 间接免疫荧光实验(Indirect immunofluorescence assay,IFA) 将EHP孢子涂布于包被0.01%(V/V)多聚赖氨酸的载玻片上,滴加4%(V/V)多聚甲醛固定样品,加入1%(V/V)Triton X-100处理20 min;加入IFA封闭液于室温下孵育1 h,分别加入EHP00_492兔多克隆抗体和TF-tag兔多克隆抗体于室温下孵育1 h,加入Alexa Fluor® 594山羊抗兔荧光二抗于室温下避光孵育1 h;加入DAPI和Wheat Germ Agglutinin(WGA-488)于室温下避光染色20 min,使用激光共聚焦显微镜(OLYMPUS FV1000)观察结果。
2 结果与分析 2.1 EHP00_492基因座位保守性分析通过6种微孢子虫同源基因座位保守性分析发现,虾肝肠胞虫EHP00_492与毕氏肠胞虫EBI_27080、黄道蟹微孢子虫ECANGBI_2177、兔脑炎微孢子虫ECU07_0740、肠脑炎微孢子虫Eint_070680和海伦脑炎微孢子虫EHEL_070710同源基因及其邻近基因座位相当保守(图 1),推测这些基因行使相似且保守的功能。兔脑炎微孢子虫ECU07_0740已被鉴定具有水通道蛋白的功能[18],推测EHP00_492是虾肝肠胞虫水通道蛋白编码基因。
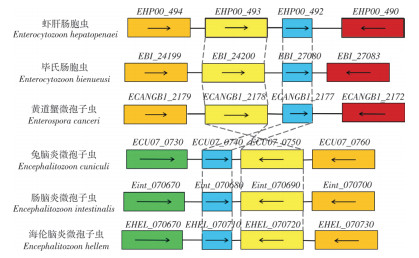
|
| 彩色矩形框代表基因;虚线连接同源基因;箭头代表ORF的方向;图中基因和间隔区域的大小按碱基数等比例(1:250)缩小 Colored rectangles represent genes; the dotted lines connect homologous genes; the arrow represents the direction of ORF; the size of genes and spacer regions in the figure is reduced in equal proportion to the number of bases (1:250) 图 1 EHP00_492同源基因座位保守性分析 Fig. 1 Conservation analysis of EHP00_492 homologous gene locus |
2.2 EHP00_492基因克隆
提取EHP总RNA,反转录cDNA,以cDNA为模板,采用EHP00_492-F1/R1引物进行RT-PCR扩增。电泳结果(图 2)显示,约750 bp处有1条特异性扩增条带;经测序可知,RT-PCR扩增条带的序列与EHP00_492全长序列一致,表明该基因无内含子。

|
| M:Trans2K Plus DNA Marker;1:PCR扩增产物 M: Trans2K Plus DNA Marker; 1: PCR amplification product 图 2 EHP00_492基因克隆 Fig. 2 Cloning of EHP00_492 gene |
2.3 EHP00_492序列特征
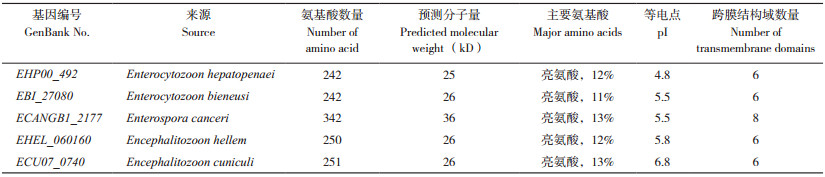
EHP00_492含有242个氨基酸,预测分子量为25 kD。经预测EHP00_492无信号肽,但具有多个丝氨酸和苏氨酸磷酸化修饰位点。利用PSIPRED对EHP00_492的二级结构进行分析,结果(图 3)表明该蛋白具有8个α螺旋和多个无规则卷曲。TMHMM分析发现,EHP00_492中12~31 aa、41~60 aa、91~113 aa、133~155 aa、176~198 aa、218~240 aa的位置存在6个跨膜结构域(图 4)。SMART预测结果(图 5)显示,EHP00_492含有1个保守的水通道蛋白家族结构域(2~236 aa)。比较EHP00_492与不同微孢子虫同源蛋白发现,它们都富含亮氨酸,等电点偏酸性,均含有6~8个跨膜结构域(表 1)。

|
| 粉色标记α螺旋,灰色标记无规则卷曲 Pink marks α helix, gray marks random curl 图 3 EHP00_492二级结构特征分析 Fig. 3 Analysis of the secondary structure of EHP00_492 |
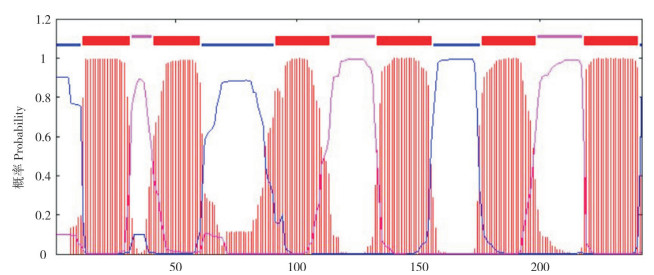
|
| 红色标记跨膜结构域,蓝色标记膜内区,粉色标记膜外区 Red marks transmembrane domains, blue marks inside, pink marks outside 图 4 EHP00_492跨膜域预测 Fig. 4 Prediction for the transmembrane domain of EHP00_492 |

|
| 图 5 EHP00_492功能结构域分析 Fig. 5 Analysis of the function domain of EHP00_492 |
|
2.4 EHP00_492序列保守性与同源蛋白亲缘进化关系分析
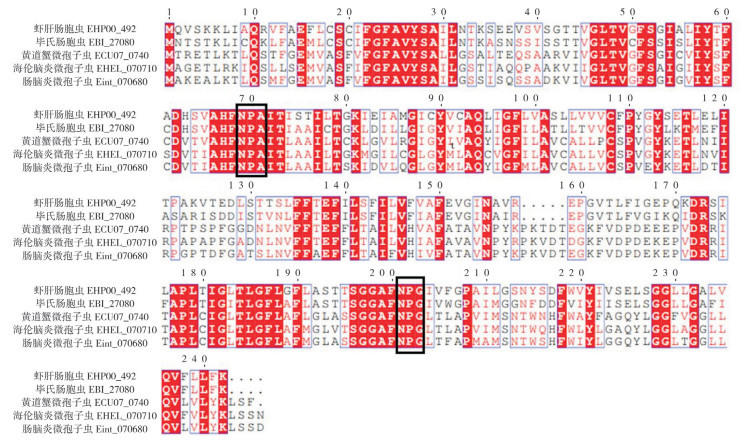
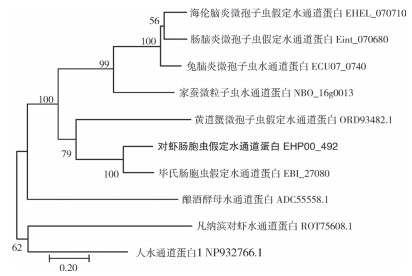
BlastP比对结果(图 6)发现,EHP00_492与毕氏肠胞虫EBI_27080序列相似性最高,为74%;与兔脑炎微孢子虫ECU07_0740、海伦脑炎微孢子虫EHEL_070710、肠脑炎微孢子虫Eint_070680序列相似性介于46%~48% 之间;多重序列比对发现,EHP00_492与其他同源蛋白均存在2个水通道蛋白的保守基序NPA/G,分别位于69~71 aa和201~203 aa处,且呈对称分布。系统进化树分析结果(图 7)发现,EHP00_492与毕氏肠胞虫水通道蛋白EBI_27080聚为一支;相比于人水通道蛋白NP932766.1和对虾水通道蛋白ROT75608.1,微孢子虫来源的水通道蛋白与酵母水通道蛋白ADC55558.1的进化关系更近。
|
| 红色背景代表相同氨基酸;黑框标记水通道蛋白保守的NPA/G基序 The red background represents the same amino acid; Black box marked the conservative NPA/G motif of aquaporin 图 6 EHP00_492同源序列比对 Fig. 6 Alignment analysis of EHP00_492 and homologous sequences |
|
| 图 7 EHP00_492系统进化树分析 Fig. 7 Phylogenetic tree analysis of EHP00_492 |
2.5 EHP00_492蛋白三维结构预测
利用AlphaFold软件预测EHP00_492蛋白的三维结构(图 8),并与已完成功能鉴定的家蚕微粒子虫水通道蛋白NbAQP(GenBank No. NBO_16g0013)和兔脑炎微孢子虫水通道蛋白EcAQP(GenBank No. ECU07_0740)的三维结构比对,发现3个蛋白质在10~32 aa、42~126 aa、129~160 aa、172~242 aa处存在重叠,暗示其蛋白功能的保守性。

|
| Merge图中的绿色区域代表NbAQP、EcAQP和EHP00_492蛋白三维结构的重叠区 The green area in the Merge diagram represents the overlap of the three-dimensional structures of NbAQP, EcAQP and EHP00_492 图 8 EHP00_492与NbAQP和EcAQP三维结构比对 Fig. 8 Comparison the three-dimensional structure of EHP00_492, NbAQP and EcAQP |
2.6 EHP00_492蛋白的表达特征
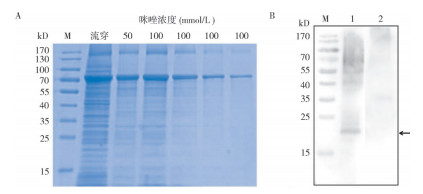
将pCold-TF-EHP00_492重组表达质粒转化E. coli Rosetta菌株中,IPTG诱导后获得大量rEHP00_492重组蛋白,利用Ni-NTA亲和层析柱纯化后,SDS-PAGE检测发现100 mmol/L咪唑能纯化出质量较高的rEHP00_492重组蛋白,分子量大小约为75 kD,与预测分子量大小一致(图 9 A)。将rEHP00_492重组蛋白免疫新西兰实验兔制备EHP00_492兔多克隆抗体。通过蛋白免疫印记分析EHP00_492在EHP中的表达特征,结果(图 9 B)显示,与阴性对照pCold-TF抗体相比,EHP00_492兔多克隆抗体可在EHP天然总蛋白中识别1条大小约21 kD的条带,表明EHP00_492在EHP中能表达,其分子量大小为21 kD。
|
|
A:rEHP00_492重组蛋白的纯化;B:Western blot分析EHP00_492蛋白的表达特征 M:低分子质量标准蛋白;1:EHP00_492兔多克隆抗体在EHP孢子总蛋白中的免疫印迹;2:TF-tag兔多克隆抗体在EHP孢子总蛋白中的免疫印迹;黑色箭头指示目标产物 A: Purification of rEHP00_492 recombinant protein; B: Expression characteristics of EHP00_492 by Western blot analysis M: Low molecular mass standard protein; 1: Western blot analysis of the total EHP proteins by EHP00_492 rabbit polyclonal antibody; 2: Western blot analysis of the total EHP proteins by TF-tag rabbit polyclonal antibody; The black arrow indicates the target product 图 9 EHP00_492蛋白的纯化与表达特征 Fig. 9 Purification and expression characteristics of EHP00_492 |
2.7 EHP00_492亚细胞定位特征
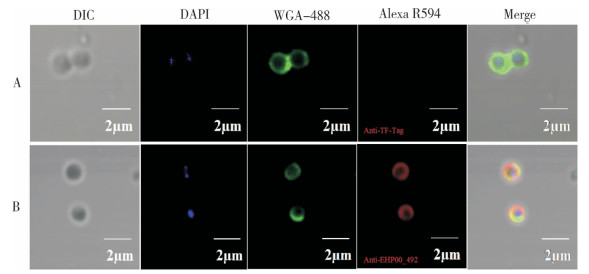
为进一步分析EHP00_492的亚细胞定位特征,利用麦胚芽凝集素WGA-488特异性标记EHP成熟孢子孢壁上的几丁质,结果(图 10)发现,与对照组相比,红色荧光标记的EHP00_492兔多克隆抗体能特异性识别EHP成熟孢子中的EHP00_492蛋白,并且红色荧光信号与绿色荧光标记的WGA-488信号存在共定位现象,表明EHP00_492蛋白定位于EHP成熟孢子的孢壁上。
|
| A:TF-Tag兔多克隆抗体标记EHP成熟孢子;B:EHP00_492兔多克隆抗体标记EHP成熟孢子绿色荧光:WGA-488标记EHP成熟孢子的孢壁,蓝色荧光:DAPI标记EHP的核 A: TF-Tag rabbit polyclonal antibody was used to label mature EHP spores; B: EHP00_492 rabbit polyclonal antibody was used to label EHP mature spores Green fluorescence: WGA-488 marked the spore wall of EHP mature spores; Blue fluorescence: DAPI marked the nuclear of EHP 图 10 IFA分析EHP00_492在EHP成熟孢子中的亚细胞定位特征 Fig. 10 Subcellular location characteristics of EHP00_492 in EHP mature spores by IFA |
3 讨论
2004年以来,EHP相继在泰国、中国、印度、印度尼西亚和委内瑞拉等对虾养殖大国被发现,由于其具有快速水平传播和垂直传播的能力,给全球对虾养殖生产造成了严重经济损失[21]。微孢子虫具有一个特殊的侵染装置,由极管、极膜层和后极泡3部分组成[22-23]。特定环境刺激孢子发芽,极管从成熟孢子内部迅速弹出并刺入宿主细胞,将具有病原性的孢原质运送至宿主细胞完成侵染[24-25]。因此,发芽是微孢子虫成功感染宿主的关键。
研究发现,微孢子虫内外渗透压的瞬间变化激活了孢子发芽,在此过程中,水通道蛋白发挥重要作用[26]。水通道蛋白属于MIP蛋白超家族,可选择性地将水、中性小分子和离子进行跨膜运输。MIP超家族分为水通道蛋白(AQPs)和甘油摄取促进剂(GlpFs)两大类,前者存在于所有生命,后者多来源于微生物[27]。作为第一亚家族,AQPs是水选择透性的水通道蛋白,包括AQP0、AQP1、AQP2、AQP4、AQP5和AQP6。第二亚家族GlpFs既能渗透水,也能运输其他不带电的小分子(如氨、尿素和甘油等)。根据氨基酸序列特征,AQP3、AQP7、AQP9和AQP10归属于这一家族[28-29]。原核生物的水通道蛋白首次发现于大肠杆菌,水通道蛋白AqpZ介导水的快速流入或流出以应对大肠杆菌细胞外渗透压变化,此外,AqpZ还有助于大肠杆菌细胞体积的增长[27]。酿酒酵母含有4个水通道蛋白基因,分别为AQY1、AQY2、AQY3和Fps1。低温使酿酒酵母细胞膜的流动性和渗透性降低,但水通道蛋白AQY1能加速胞内水的流出,避免细胞死亡[30]。水通道蛋白参与了细胞膜的渗透调节。微孢子虫也具有水通道蛋白,当其发芽激活时,水通道蛋白促进水分子迅速流入孢子内部,造成后极泡和极膜层瞬间膨胀,直至孢子前端破裂,极管被膨胀的后极泡推动并外翻弹出。此外,氯化汞能抑制水通道蛋白功能,但无法抑制EcAQP对水的渗透,推测EcAQP缺乏位于NPA保守基序旁对汞敏感的半胱氨酸位点。虾肝肠胞虫作为养殖对虾检出率最高的病原之一,目前暂无有效防治药物。因此,后续研究工作可探索靶向EHP00_492的抑制剂降低孢子发芽,以期有效控制EHP传播。
4 结论本研究从虾肝肠胞虫基因组中筛选到水通道蛋白编码基因EHP00_492,序列分析发现EHP00_492符合水通道蛋白保守的序列特征。系统进化树分析发现,EHP00_492与毕氏肠胞虫水通道蛋白EBI_27080亲缘关系最近,并且与已鉴定的家蚕微孢子虫水通道蛋白NbAQP、兔脑炎微孢子虫水通道蛋白EcAQP的三维结构高度相似。EHP00_492能表达定位于虾肝肠胞虫成熟孢子的孢壁上,推测其在EHP发芽过程中发挥重要功能。本研究初步明确了EHP00_492蛋白的序列特征、结构特征和表达定位特征,对进一步探究EHP水通道蛋白的功能具有重要意义。
| [1] |
YU D J, ZHAI Y F, HE P M, JIA R. Comprehensive transcriptomic and metabolomic analysis of the Litopenaeus vannamei hepatopancreas after WSSV challenge[J]. Frontiers in Immunology, 2022, 13: 826794. DOI:10.3389/fimmu.2022.826794 |
| [2] |
SURAWUT S, SUNTARA K, PUCKDEE W, KUNSOOK C, KANJANAVAS P, KOMPATIPARN A, LEERUKSAKIAT P. Decreased severity of acute hepatopancreatic necrosis disease in white shrimp (Litopenaeus vannamei) by mixed culture of Bacillus subtilis, Bacillus licheniformis and Bacillus megaterium[J]. Tropical Life Sciences Research, 2023, 34(1): 85-98. DOI:10.21315/tlsr2023.34.1.6 |
| [3] |
MOSER R J, FRANZ L, FIRESTONE S M, SELLARS M J. Enterocytozoon hepatopenaei real-time and shrimp MultiPathTM PCR assay validation for South-East Asian and Latin American strains of penaeid shrimp[J]. Diseases of Aquatic Organisms, 2022, 149: 11-23. DOI:10.3354/dao03655 |
| [4] |
KIM B S, JANG G I, KIM S M, JEON Y G, OH Y K, HWANG J Y, KWON M G. First report of Enterocytozoon hepatopenaei infection in pacific whiteleg shrimp (Litopenaeus vannamei) cultured in Korea[J]. Animals-Basel, 2021, 11(11): 3150. DOI:10.3390/ani11113150 |
| [5] |
WU Y J, CHEN J, LIAO G L, HU M J, ZHANG Q, MENG X Z, LI T, LONG M X, FAN X D, YU Q, ZHANG L P, PAN G Q, ZHOU Z Y. Down-Regulation of lipid metabolism in the hepatopancreas of shrimp Litopenaeus vannamei upon light and heavy infection of Enterocytozoon hepatopenaei: A comparative proteomic study[J]. International Journal of Molecular Sciences, 2022, 23(19): 11574. DOI:10.3390/ijms231911574 |
| [6] |
FAN X D, WEI C M, YANG X J, XIAO A, TAN N Q, CHEN J, LONG M X, PAN G Q, WAN Y J, ZHOU Z Y. Proteomic analysis of spore surface proteins and characteristics of a novel spore wall protein and biomarker, EhSWP3, from the shrimp microsporidium Enterocytozoon hepatopenaei (EHP)[J]. Microorganisms, 2022, 10(2): 367. DOI:10.3390/microorganisms10020367 |
| [7] |
NING M X, WEI P P, SHEN H, WAN X H, JIN M J, LI X Q, SHI H, QIAO Y, JIANG G, WANG W, WANG L, MENG Q G. Proteomic and metabolomic responses in hepatopancreas of whiteleg shrimp Litopenaeus vannamei infected by microsporidian Enterocytozoon hepatopenaei[J]. Fish Shellfish Immunology, 2019, 87: 534-545. DOI:10.1016/j.fsi.2019.01.051 |
| [8] |
WANG Y, ZHOU J Y, YIN M H, YING N, XIANG Y, LIU W C, YE J Q, LI X C, FANG W H, TAN H X. A modification of nested PCR method for detection of Enterocytozoon hepatopenaei (EHP) in giant freshwater prawn Macrobrachium rosenbergii[J]. Frontiers in Cellular and Infection Microbiology, 2022, 12: 1013016. DOI:10.3389/fcimb.2022.1013016 |
| [9] |
LYU Q, WANG L J, FAN Y P, MENG X Z, LIU K K, ZHOU B Q, CHEN J, PAN G Q, LONG M X, ZHOU Z Y. Identification and characterization a novel polar tube protein (NbPTP6) from the microsporidian Nosema bombycis[J]. Parasites & Vectors, 2020, 13(1): 475. DOI:10.1186/s13071-020-04348-z |
| [10] |
LYU Q, ZHOU B Q, LIAO H J, HE X L, CHEN Y Q, PAN G Q, LONG M X, ZHOU Z Y. Proteomic profile of polar filament and polar tube from fungal pathogen microsporidium Nosema bombycis provides new insights into its unique invasion organelle[J]. Journal of proteomics, 2022, 263: 104617. DOI:10.1016/J.JPROT.2022.104617 |
| [11] |
WANG L J, LYU Q, HE Y T, GU R C, ZHOU B Q, CHEN J, FAN X D, PAN G Q, LONG M X, ZHOU Z Y. Integrated qPCR and staining methods for detection and quantification of Enterocytozoon hepatopenaei in shrimp Litopenaeus vannamei[J]. Microorganisms, 2020, 8(9): 1366. DOI:10.3390/microorganisms8091366 |
| [12] |
JAROENLAK P, CAMMER M, DAVYDOV A, SALL J, USMANI M, LIANG F X, EKIERT D C, BHABHA G. 3-Dimensional organization and dynamics of the microsporidian polar tube invasion machinery[J]. PLoS Pathog, 2020, 16(9): e1008738. DOI:10.1371/journal.ppat.1008738 |
| [13] |
UNDEEN A H, FRIXIONE E. The role of osmotic pressure in the germination of Nosema algerae spores[J]. The Journal of Protozoology, 1990, 37(6): 561-567. DOI:10.1111/j.1550-7408.1990.tb01265.x |
| [14] |
FRIXIONE E, RUIZ L, CERBÓN J, UNDEEN A H. Germination of Nosema algerae (Microspora) spores: Conditional inhibition by D2O, ethanol and Hg2+ suggests dependence of water influx upon membrane hydration and specific transmembrane pathways[J]. Journal of Eukaryotic Microbiology, 1997, 44(2): 109-116. DOI:10.1111/j.1550-7408.1997.tb05946.x |
| [15] |
WAGNER K, UNGER L, SALMAN M M, KITCHEN P, BILL R M, YOOL A J. Signaling mechanisms and pharmacological modulators governing diverse aquaporin functions in human health and disease[J]. International Journal of Molecular Sciences, 2022, 23(3): 1388. DOI:10.3390/ijms23031388 |
| [16] |
AGGELI I K, KAPOGIANNATOU A, PARASKEVOPOULOU F, GAITANAKI C. Differential response of cardiac aquaporins to hyperosmotic stress; salutary role of AQP1 against the induced apoptosis[J]. European Review for Medical and Pharmacological Sciences, 2021, 25(1): 313-325. DOI:10.26355/eurrev_202101_24397 |
| [17] |
MEDINA Y, ACOSTA L, REPPETTI J, COROMINAS A, JUANITA B, SZPILBARG N, DAMIANO A E. Lactic acid transport mediated by aquaporin-9: implications on the pathophysiology of preeclampsia[J]. Frontiers in Physiology, 2021, 12: 774095. DOI:10.3389/fphys.2021.774095 |
| [18] |
GHOSH K, CAPPIELLO C D, MCBRIDE S M, OCCI J L, CALI A, TAKVORIAN P M, MCDONALD T V, WEISS L M. Functional characterization of a putative aquaporin from Encephalitozoon cuniculi, a microsporidia pathogenic to humans[J]. International Journal for Parasitology, 2006, 36(1): 57-62. DOI:10.1016/j.ijpara.2005.08.013 |
| [19] |
CHEN G, WANG W, CHEN H L, DAI W J, PENG X R, LI X L, TANG X D, XU L, SHEN Z Y. Functional characterization of an aquaporin from a microsporidium, Nosema bombycis[J]. PLoS One, 2017, 12(7): e0181703. DOI:10.1371/journal.pone.0181703 |
| [20] |
WIREDU BOAKYE D, JAROENLAK P, PRACHUMWAT A, WILLIAMS T A, BATEMANK S, ORNCHUMA I, SRITUNYALUCKSANA K, PASZKIEWICZ K H, MOORE K A, STRNTIFORD G D, WILLIAMS B A P. Decay of the glycolytic pathway and adaptation to intranuclear parasitism within Enterocytozoonidae microsporidia[J]. Environmental Microbiology, 2017, 19(5): 2077-2089. DOI:10.1111/1462-2920.13734 |
| [21] |
TOURTIP S, WONGTRIPOP S, STENTIFORD G D, BATEMAN K S, SRIURAIRATANA S, CHAVADEJ J, SRITUNYALUCKSANA K, WITHYACHUMNARNKUL B. Enterocytozoon hepatopenaei sp. nov. (Microsporida: Enterocytozoonidae), a parasite of the black tiger shrimp Penaeus monodon (Decapoda: Penaeidae): Fine structure and phylogenetic relationships[J]. Journal of Invertebrate Pathology, 2009, 102(1): 21-29. DOI:10.1016/j.jip.2009.06.004 |
| [22] |
KARTHIKEYAN K, SUDHAKARAN R. Experimental horizontal transmission of Enterocytozoon hepatopenaei in post-larvae of whiteleg shrimp, Litopenaeus vannamei[J]. Journal of Fish Diseases, 2019, 42(3): 397-404. DOI:10.1111/jfd.12945 |
| [23] |
CHAIJARASPHONG T, Munkongwongsiri N, STENTIFORD G D, ALDAMA-CANO D J, THANSA K, FLEGEL T W, SRITUNYALUCKSANA K, ITSATHITPHAISARN O. The shrimp microsporidian Enterocytozoon hepatopenaei (EHP): Biology, pathology, diagnostics and control[J]. Journal of Invertebrate Pathology, 2021, 186: 107458. DOI:10.1016/j.jip.2020.107458 |
| [24] |
IMURA Y, NAKAMURA H, ARAI R, HATAKEYAMA Y. Comparison of the germination conditions of two large-spore microsporidia using potassium and sodium ion solutions[J]. Insects, 2023, 14(2): 185. DOI:10.3390/insects14020185 |
| [25] |
HAN B, TAKVORIAN P M, WEISS L M. Invasion of host cells by microsporidia[J]. Frontiers in Microbiology, 2020, 11: 172. DOI:10.3389/fmicb.2020.00172 |
| [26] |
PARIA P, TASSANAKAJON A. Identification of potential druggable targets and structure-based virtual screening for drug-like molecules against the shrimp pathogen Enterocytozoon hepatopenaei[J]. International Journal of Molecular Sciences, 2023, 24(2): 1412. DOI:10.3390/ijms24021412 |
| [27] |
TONG H, HU Q, ZHU L, DONG X. Prokaryotic aquaporins[J]. Cells, 2019, 8(11): 1316. DOI:10.3390/cells8111316 |
| [28] |
LIAO S, GAN L, LYU L, MEI Z. The regulatory roles of aquaporins in the digestive system[J]. Genes and Diseases, 2020, 8(3): 250-258. DOI:10.1016/j.gendis.2019.12.011 |
| [29] |
PLUHACKOVA K, SCHITTNY V, BÜRKNER P C, SILIGAN C, HORNER A. Multiple pore lining residues modulate water permeability of GlpF[J]. Protein Science, 2022, 31(10): e4431. DOI:10.1002/pro.4431 |
| [30] |
SABIR F, LOUREIRO-DIAS M C, SOVERAL G, PRISTA C. Functional relevance of water and glycerol channels in Saccharomyces cerevisiae[J]. Fems Microbiology Letters, 2017, 364(9). DOI:10.1093/femsle/fnx080 |
(责任编辑 崔建勋)
 2023, Vol. 50
2023, Vol. 50




