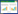文章信息
基金项目
- 国家自然科学基金面上项目(31772371);广东省自然科学基金面上项目(2021A1515011258);中国科学院华南植物园青年人才团队项目(QNXM-04)
作者简介
- 朱虹,博士,硕士生导师,中国科学院华南植物园农业与生物技术研究中心陈焕镛研究员,主要从事园艺作物采后生物学和贮运保鲜技术研究,包括园艺作物microRNA的分离鉴定及其在果实成熟衰老和抗逆过程中的功能分析,可食用植物营养成分的化学基础、调控机制及高值化利用。主持国家自然科学基金、广东省自然科学基金、教育部留学回国人员科研基金、中国科学院科技服务网络计划(STS)区域重点项目子课题、宁夏农业综合开发科技推广项目,并作为骨干成员参与国家重点研发计划项目。累计发表SCI论文40余篇,包括以第一或通信作者在《Trends in Plant Science》《New Phytologist》《Horticulture Research》《Postharvest Biology & Technology》等期刊发表论文,1篇入选ESI高被引论文,单篇最高五年影响因子22.5。获授权发明专利3件,参与制定国家农业行业标准1项,获神农中华农业科技奖和广东省科技进步二等奖。培养研究生多名,指导的毕业生曾获中国科学院“地奥”奖学金一等奖和研究生国家奖学金。朱虹(1980—),女,博士,副研究员,研究方向为果蔬采后生物学,E-mail:zhuhong@scbga.ac.cn; 朱孝扬,法国图卢兹第三大学博士,副研究员,硕士生导师,主要从事果蔬采后保鲜技术研发和品质调控机理研究。华南农业大学海外引进高层次人才,采后科学与技术系副主任,南方园艺产品保鲜教育部工程技术研究中心副主任,入选广东省高层次人才珠江人才计划 - 青年拔尖人才(2017年),硕士学位论文获得“2014年广东省优秀硕士学位论文”。主持国家自然科学基金2项,广东省自然科学基金面上项目2项,广东省创新团队岗位专家项目、广东省教育厅项目、广州市科技创新项目各1项,华南农业大学国际合作培育项目以及高层次人才引进项目各1项,作为主要成员参与国家级、省部级项目多项。以第一或通信作者在《Plant Physiology》《Trends in Plant Science》《Plant and Cell Physiology》《Journal of Agricultural and Food Chemistry》《Food Chemistry》《Horticulture Research》《Postharvest Biology and Technology》等期刊发表学术论文40多篇,授权发明专利、制定行业企业标准等多项,在果蔬成熟调控机制及技术研发中取得重要的原创性成果。现为广东省香蕉菠萝产业体系保鲜岗岗位专家,农业农村部产业技术体系香蕉采后保鲜岗核心成员。担任《Frontier in Plant Science》《Foods》客座编辑,中国热带作物学会理事,《广东农业科学》青年编委,国际园艺学会会员,担任《Plant Physiology》《 Horticulture Research》《Journal of Advanced Research》《Food Chemistry》《Postharvest Biology and Technology》《Journal of Agriculture and Food Chemistry》等重要学术期刊审稿人.
通讯作者
- 朱孝扬(1985—),男,博士,副研究员,研究方向为果实采后品质控制与机理研究,E-mail:xiaoyang_zhu@scau.edu.cn.
文章历史
- 收稿日期:2023-07-21
2. 中国科学院大学生命科学学院,北京 100049;
3. 华南农业大学园艺学院,广东 广州 510642
2. College of Life Sciences, University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Horticulture, South China Agricultural University, Guangzhou 510642, China
【研究意义】香蕉(Musa acuminata, AAA group)是芭蕉科芭蕉属植物,营养丰富,经济价值高。在我国,香蕉多以鲜食为主,而鲜食对于果实品质的要求比加工更为苛刻。一方面,香蕉属于呼吸跃变型果实,一旦启动呼吸跃变,果实会迅速完成后熟,难以继续贮藏和运输。香蕉对乙烯十分敏感,因此抑制乙烯产生和延缓呼吸跃变的到来是控制香蕉后熟及贮藏保鲜的关键[1]。另一方面,作为热带水果的典型代表,香蕉对低温十分敏感,田间生长低温或采后贮运温度过低都易发生冷害,严重影响产量和果实品质[2]。鉴于种质资源缺乏和果实成熟及冷害调控的复杂性,目前针对香蕉采后成熟和冷害的控制仍面临较大挑战。因此,深入研究其后熟和冷害发生规律及调控机制,对于维持和提升香蕉采后品质以及减轻冷害损失,具有重大的理论和实践意义。【前人研究进展】microRNA(简称miRNA)是真核生物中广泛存在的一类对基因表达调控具有重要影响的非编码小分子RNA,长度在20~24个核苷酸。一般产生于基因的间隔区域,通过与靶基因互补配对的原则在转录后水平调控基因表达。成熟miRNA序列在不同物种间表现出高度的进化保守性,而其表达则具有严格的时空特异性,并受到环境条件的影响[3]。其中,miR164最先在拟南芥和水稻中被报道,此后通过同源比对、克隆及高通量测序等技术,先后在多个植物物种中鉴定出miR164家族的成员。经miRBase 22.0版数据库检索,miR164家族目前共有70个成员,来自39个不同物种。虽然这些miR164家族成员源自不同物种或同一物种不同的基因位点,且具有各异的前体序列,但它们的成熟序列却高度同源,超过半数的成员在不同植物物种中的序列是完全相同的[4]。部分植物miR164的功能已得到解析。例如,拟南芥中miR164调控营养生长及花器官形成,且伴随叶片衰老,miR164的表达量会出现下降[5]。甜橙miR164在球形胚发育过程中特异性表达,与球形胚的形成关系密切[6]。过表达miR164的番茄出现花朵脱落延迟、果形奇特且不产种子的表型,证实miR164参与调控花器官形成及果实发育[7]。此外,环境胁迫如低氮或盐渍条件下,杨树和棉花的特定miR164家族成员均表现出早期的上调表达,通过靶基因调节根系生长以适应逆境胁迫[8-9]。尽管生信预测miR164可能作用于多种类型的靶基因,但基于现有的研究报道,miR164最主要的作用靶标为NAC家族转录因子。换言之,绝大多数植物都是通过miR164-NAC这一保守的调控模块来行使功能。例如,拟南芥miR164家族中的3个成员,分别靶向5个与分生组织形成和器官边界建成相关的NAC家族转录因子。其中CUC1和CUC2基因在拟南芥球形胚的叶原基之间表达,miR164通过负调控CUC1/2影响分生组织的发育和器官原基的边界建立。NAC基因在胚发生和花发育过程中有重要作用,其功能缺失会导致花芽发育异常[10]。Guo等[11]证实miR164-NAC调控模块和生长素信号通路存在密切的互作关系,miR164功能缺失突变体中NAC mRNA积累,生长素信号通路受阻,最终导致侧根增加。相反,miR164过表达会引发侧根数目减少。近期研究揭示了miR164-GhCUC2模块通过调控脱落酸合成影响棉花的株型[12],而番茄SlMIR164A可以通过控制SlNAM2和SlNAM3的表达调节果实的成熟及营养品质[13]。NAC家族是植物特异性转录因子超家族,广泛存在于植物界,其结构域最初的特征在于来自NAM、ATAF1/2和CUC2的共有序列[14]。大多数NACs蛋白具有保守的DNA结合域,N端包含约150个氨基酸,参与行使NAC蛋白的多种独特功能,而C端具有高度可变的转录调节区域,富含丝氨酸、苏氨酸等残基,且该区域在不同亚群中存在差异,可能赋予不同亚群NACs蛋白间的功能变异。某些NAC转录因子还在C末端含有负责锚定到质膜或内质网的跨膜基序,在需要表达时被蛋白水解切割后转移至细胞核。通过对数百个不同物种NACs基因的进化关系分析表明NAC超家族内部存在多个亚群,这些NAC家族成员在植物生长发育、果实成熟衰老以及生物和非生物胁迫响应等多个生物过程中发挥着关键作用[15]。另一方面,对NACs蛋白的结构解析也取得一些进展,如通过X光衍射分别获得ANAC019和OsNAC1保守域的晶体结构,并推断大多数NACs蛋白可能以同源二聚体的形式发挥调控作用[16-17]。【本研究切入点】解析果实成熟与抗逆调控机制一直是采后生物学的重要科学问题,目前相关研究还有待深入和拓展,特别是针对microRNA- 靶基因模块介导香蕉采后品质形成和冷害发生的调控机制尚不清楚。【拟解决的关键问题】本研究以香蕉为试验对象,对其采后成熟及低温胁迫下不同阶段的香蕉果实进行小RNA测序,筛选分析与香蕉后熟及冷害密切相关的miR164和NAC转录因子,明确香蕉中的miR164-NAC调控模块,并对该模块在香蕉后熟和冷害过程中的表达模式进行分析,以期为深入探究香蕉miR164-NAC调控模块的功能奠定基础。
1 材料与方法 1.1 试验材料及处理鉴于‘巴西蕉’高产优质,一直是我国香蕉的主栽品种,本试验选取其作为研究对象。果实于2019年8月采自广东省广州市南沙区万顷沙蕉园,采收时果实饱满度为7~8成,采收后立即运回实验室。落梳后分为单个蕉指,清水清洗后再用0.1% 施保克浸果3 min后自然晾干。挑选大小均匀、无病虫害及机械伤的单果分成4个处理:(1)对照组:自然后熟,置于23 ℃、相对湿度85%~90% 条件下贮藏;(2)实验组1:100 μL/L C2H4密闭处理18 h后,置于23 ℃、相对湿度85%~90% 条件下贮藏;(3)实验组2:0.5 μL/L 1-MCP密闭处理18 h后,置于23 ℃、相对湿度85%~90% 条件下贮藏;(4)实验组3:置于6 ℃、相对湿度85%~90% 条件下贮藏。每个处理150个果实,设置3次生物学重复。分别在贮藏第0、2、4、6、8 d取对照组与处理组的香蕉样品,分离果皮和果肉,冻存。
1.2 试验方法1.2.1 香蕉miR164家族成员的鉴定及进化分析 首先基于前期的sRNA高通量测序分析[18],获得香蕉中6条MIR164基因家族成员的前体基因组定位、长度、自由能、miRNA成熟序列及miR* star序列等信息。根据前体序列信息,利用在线RNAfold网络服务器(http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi)分析香蕉MIR164基因家族前体序列的二级茎环结构。随后从miRBase数据库(www.mirbase.org)下载所有植物物种MIR164基因家族成员的前体序列和成熟序列,按科属分类统计,利用Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)网络服务器对香蕉和其他代表性物种(选取禾本科、蔷薇科及十字花科代表)MIR164s基因的前体序列和成熟序列进行多重序列比对,分析进化特征,并在此基础上用iTOL在线软件基于参数临近法对前体序列构建系统进化树。
1.2.2 香蕉NAC基因家族成员的miR164靶位点预测及验证 从前期香蕉sRNA测序结果中获取全部的成熟miR164序列,使用Target prediction for plant microRNAs(TAPIR,http://bioinformatics.psb.ugent.be/webtools/tapir/)对香蕉NAC基因家族的蛋白编码序列进行miR164靶位点预测,Score值设为5.0,自由能比值设为0.6。预测结果使用TBtools中的Gene structure view功能进行可视化[19]。另一方面,基于香蕉降解组测序分析[18],鉴定高置信度的miR164的靶基因,以明确香蕉中特定的miR164-NAC调控模块。利用烟草瞬时表达体系进一步验证miR164与靶基因的互作需要两类载体[20]:一类为miRNA超表达载体pBI121。设计引物用PCR方法获得MIR164基因的前体,替代pBI121载体中的β- 葡萄糖醛酸酶(GUS)基因完成miR164超表达载体的构建。另一类为改造过的绿色荧光蛋白GFP超表达载体pMS4,其中GFP基因前有两个酶切位点Xho Ⅰ和Xba Ⅰ,双酶切后用于插入miR164在靶基因上的结合位点片段,该片段由两段寡核苷酸DNA退火后合成。miR164和靶位点如果能在烟草叶片上互作,则GFP表达被抑制,荧光信号减弱。
1.2.3 香蕉NAC基因家族成员的鉴定分析 从第4版香蕉基因组数据库(Version 4.3)下载全基因组文件,包括全部氨基酸序列和gff3注释文件。分别在拟南芥基因组数据库(TAIR,http://www.arabidopsis.org/)和植物转录因子数据库PlantTFDB(http://planttfdb.gao-lab.org/)下载拟南芥和水稻的NAC同源氨基酸序列,经手动去重后,共得到100个拟南芥和141个水稻的NACs蛋白,建立参考序列库。采用TBtools软件[19]的BLAST工具来检索香蕉基因组中的潜在NACs基因,其中E-value cutoff设置为e-10,其他参数默认。从PFAM蛋白数据库(http://pfam.xfam.org/)下载氨基酸序列NAM保守结构域(PF02365)的HMM文件,在E-value cutoff为0.01、其他参数默认的设置下,使用TBtools软件的Simple Hmm Search来搜索香蕉基因组中的NACs基因。将BLAST和Hmm Search结果合并,手动去重,将所有筛选出的候选NACs基因氨基酸序列使用Pfam和NCBI的CDD数据库验证蛋白序列保守结构域。含有NAM结构域的序列被确定为香蕉NAC基因家族成员,并根据其所在染色体位置的先后顺序对它们进行重命名。通过TBtools中的Gene location visualize from GTF/GFF工具,使用香蕉基因组的GFF3注释文件来创建香蕉NACs基因的染色体定位图。使用MCScanX对香蕉基因组内的同源复制事件进行分析,并通过TBtools中的Advanced Circos对香蕉NACs基因进行共线性分析。借助NCBI CDD数据库确定香蕉NACs蛋白保守结构域NAM域所在的具体位置,并使用TBtools中的Gene structure view功能对香蕉NACs基因结构进行可视化[19]。同时,利用ExPASy(www.expasy.org)网站中的ProtParam工具对香蕉NACs蛋白的理化性质进行分析。
1.2.4 香蕉NACs系统进化树的构建 使用ClustalX 2.0对拟南芥、水稻和香蕉NACs进行氨基酸多序列比对,利用TBtools中的trimAL Wrapper对多序列比对后的结果文件进行修剪,通过IQ-Tree构建系统进化树,参数默认,最后使用iTOL在线网站进行美化。
1.2.5 香蕉RNA提取及小分子RNA的Northern杂交 香蕉总RNA的提取在热硼法基础上略有改动,具体参考曾俊[21]方法。小分子RNA Northern杂交实验参考文献[20]并略作改动。配制15% PAGE工作液,充分溶解后迅速加入10% APS和凝固剂TEMED,随即灌胶插梳。等胶凝固后拔出梳子,电泳槽中加入适量1×TBE电泳液,120 V预电泳30 min。RNA样品制备:取总RNA 20 μL(浓度为1 μg/μL),加入20 μL 2×上样缓冲液,98 ℃变性2 min,快速置于冰上备用。预电泳结束后用注射器冲洗点样孔数次,点入RNA样品。60 V电压电泳30 min,接着调整为120 V电泳90 min,直至下层溴酚蓝指示剂迁移至距离胶底部约1 cm左右停止电泳。然后使用Mini垂直电转槽(Bio Rad),设置60 V电压,转膜90 min。随后紫外交联固定样品,60 ℃烘膜2 h后装袋,4 ℃保存。使用Chemiluminescent Hybridization and Detection Kit(Thermo Scientific)进行杂交及信号检测,3’端生物素标记的杂交检测探针与miR164成熟序列反向互补,选择香蕉U6片段作为内参,探针由上海生工生物合成,序列见表 1。配制杂交工作液,将膜正面朝内,贴放于玻璃杂交管中,加入杂交工作液在37 ℃预杂交30 min,随后在预杂交液中加入5 μL生物素标记的miR164探针(浓度为5 μmol/L),同样37 ℃杂交16~20 h。第2 d在37 ℃杂交炉内,滚动洗膜。弃去杂交管中的严谨洗膜液,加入5 mL显色试剂盒(Chemiluminescent Nucleic Acid Detection Module)中提供的封阻液,杂交炉温调至25 ℃,滚动封阻15 min。再取20 μL链亲和素- 辣根过氧化物酶(HRP,Streptavidin-Horseradish peroxidase conjugate),加入1 mL封阻液中,再将混合液加入上一步封阻液中,继续室温滚动15 min。弃封阻液,加入10 mL 1×检测洗液洗膜。将膜从杂交管转移至干净洗盒中,加入底物平衡液50 mL,室温匀速摇动5 min,混合250 μL鲁米诺溶液(Luminol/Enhancer solution)和250 μL H2O2溶液配制底物工作液。膜正面朝上平铺于透明夹膜上,加入底物工作液使其均匀覆盖整个膜,暗处室温孵育5 min。使用ChemiDocTM Touch Imaging System成像系统(Bio Rad)检测杂交信号。根据信号强弱调整曝光模式及时长。
1.2.6 香蕉NAC靶基因反转录及定量PCR 采用TaKaRa PrimescriptTM RT reagent Kit with gDNA Eraser试剂盒(RR047A)对质量过关的香蕉总RNA进行反转录,参照试剂盒说明书操作。将所得cDNA溶液浓度统一稀释成50 ng/μL。靶基因定量PCR采用TaKaRa TB GreenTM Premix Ex TaqTM Ⅱ(Tli RNaseH Plus)试剂盒,依次加入10 μL TB Green Premix Ex Taq Ⅱ(2×)、0.4 μL ROX Reference Dye Ⅱ(50×)、0.4 μL Forward Primer(10 μmol/L)、0.4 μL Reverse Primer(10 μmol/L)、6.8 μL超纯水以及2 μL cDNA,总体系为20 μL。使用ABI公司7500 Fast荧光定量PCR仪进行反应,程序为:95 ℃预变性5 min,95 ℃变性5 s,60 ℃退火30 s,40个循环。反应结束后,设置熔解反应95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s,60 ℃ 15 s。使用Beacon Designer 8.0软件设计靶基因定量PCR的引物,交由上海生工生物合成。定量PCR选择香蕉RPS2作为内参基因,靶基因定量PCR引物见表 1。
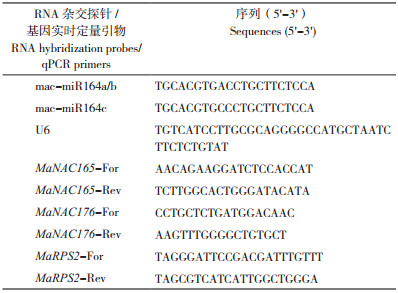
2 结果与分析 2.1 香蕉miR164家族成员的鉴定及进化分析基于前期sRNA测序结果,在香蕉中鉴定到6个MIR164基因家族成员(相应成熟miR164序列分别命名为mac-miR164 a~f),通过将这6条前体序列比对到香蕉基因组V4.3,发现它们定位在第6、8和11号3条染色体上,其中4条位于4个编码基因的内部,另外2条则位于基因间区(详见数据附表S1)。定位于第11号染色体上的2条MIR164基因前体与编码基因的外显子部分重合,而定位于第8和第6号染色体上的2条前体则完全处于编码基因的内含子区域(图 1A)。经搜索和基于植物MIRNA基因位点鉴定标准进行筛选,从miRbase数据库中共获得131条MIR164基因的前体序列,共产生70条非冗余的成熟miR164序列。依据现有数据库,miR164家族分布于18科39个植物物种中,属于保守度较高的一类MIRNA基因家族。其中,禾本科报道的MIR164最多、达30条,成熟序列有18条,分布于7个物种中;其次是蔷薇科的4个物种,有18条前体序列和8条成熟序列;排在第3位的是十字花科5个物种,共报道16条前体序列和10条成熟miR164序列。从分布上看,miR164广泛分布在单双子叶植物中,甚至也同样存在于被子植物最原始的无油樟科植物互叶梅中。将香蕉中鉴定到的miR164成熟序列与39个已报道植物物种中的非冗余miR164成熟序列进行比对,结果显示miR164成熟序列在5’端较为保守,尤其是前8位,而3’端序列呈现多变性,特别是第17、20和21位的序列,在不同物种中差异较为明显,其中香蕉miR164也贡献了部分多样性(图 1B)。比对结果表明该家族的基因扩张在进化过程中存在明显的重排和筛选,暗示其在不同物种中调控的复杂性,值得深入研究。接着分析测序鉴定到的香蕉6条miR164家族成员前体序列的二级结构。如图 1C所示,这些前体序列的长度介于100~120 nt,具有MIRNA基因前体典型的发夹茎环结构和较低的自由能(-70~-45 kcal/mol)。图中红色和蓝色标记的序列分别代表miRNA成熟序列和miR* star序列,其中有2条前体未检测到miR*star序列的表达。通过对香蕉与禾本科、蔷薇科及十字花科代表物种拟南芥的MIR164基因前体序列构建系统进化树,发现该家族能在单、双子叶植物间明显区分开来。但香蕉的6条MIR164基因前体并没有全部聚在一起,而是分布在3个亚群中,其中有5条均与热带双子叶植物番木瓜聚为一类,包括测序丰度较高的mac-miR164a/b和mac-miR164c,仅有一条低丰度的mac-miR164f与单子叶禾本科植物聚为一类(图 1D),这暗示香蕉MIR164基因家族的起源与双子叶植物更为接近。
|
| A:香蕉MIR164前体在编码基因上的定位;B:香蕉与其他植物miR164的成熟序列比对结果;C:香蕉MIR164前体的二级结构预测;D:香蕉与禾本科、蔷薇科及十字花科代表物种MIR164前体序列的系统进化树 A: Mapping of banana MIR164 precursor on coding genes; B: Comparison of miR164 mature sequences between banana and other plants; C: Prediction of secondary structure of MIR164 precursor in banana; D: A phylogenetic tree representing MIR164 precursor sequences of banana and other plants of Poaceae, Rosaceae and Cruciferae 图 1 香蕉miR164家族成员鉴定及进化分析 Fig. 1 Identification and evolutionary analysis of miR164 family members in banana |
2.2 香蕉miR164靶基因及miR164-NAC调控模块鉴定
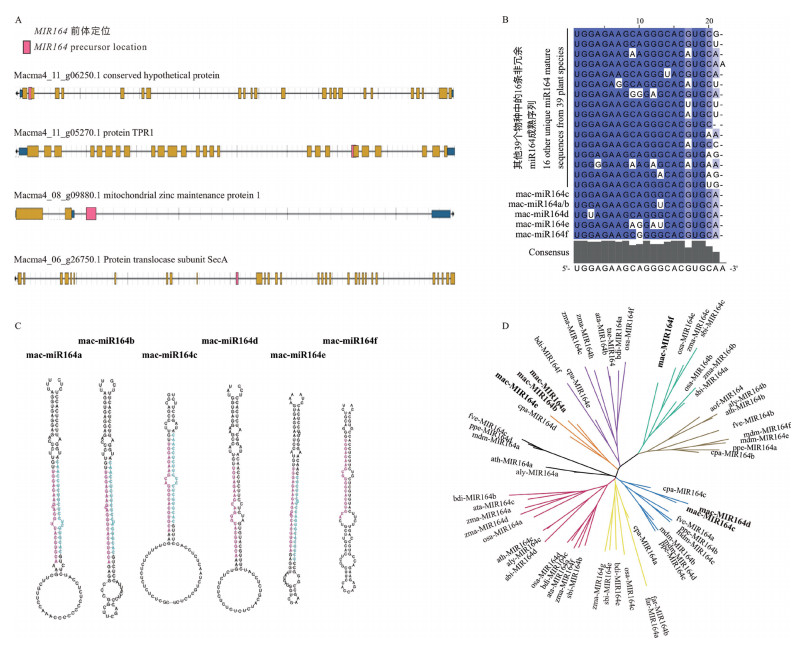
首先利用psRNATarget在线软件(http://plantgrn.noble.org/psRNATarget/)对香蕉miR164家族成员所有可能的靶基因进行预测,发现大多数是编码NAC结构域的转录因子(详见数据库附表S2)。同时,结合香蕉NACs基因家族成员的鉴定,预测miR164的结合位点。然而,在鉴定到的所有香蕉NACs基因中,仅有5% 具有miR164的靶位点,这暗示着miR164-NAC调控模块可能在香蕉物种进化过程中出现较晚,且miR164靶点均位于接近NAM保守结构域的3’UTR区域(图 2A)。另一方面,前期的香蕉降解组测序共鉴定到4条受miR164切割的NACs靶基因(均属于0类高置信靶基因,详见数据库附表S3)。降解组t-plot证实,miR164对这几个NACs靶基因都有较高的剪切效率,且切割位点较为一致(图 2B~ 图 2D)。烟草叶片的瞬转实验则进一步验证了miR164c对香蕉NAC176的抑制作用(图 2E)。而香蕉中这几个miR164的NACs靶基因均位于第9号染色体上。
|
| A:香蕉NAC基因家族成员上miR164的靶点预测;B~D:基于降解组生成的香蕉NAC165/177/176靶基因上的miR164切割图(T-plot);E:香蕉NAC176受miR164a负调控的in vivo验证 A: Target prediction of miR164 on banana NAC gene family members; B-D: miR164 cleavage map (T-plot) on the target gene of MaNAC165/177/176 generated by degradation sequencing; E: MaNAC176 is negatively regulated by miR164a in vivo 图 2 香蕉miR164靶基因及miR164-NAC调控模块鉴定 Fig. 2 Identification of miR164 target genes and miR164-NAC regulatory module in banana |
2.3 香蕉NAC基因家族成员的鉴定分析
通过同源序列比对结合手动验证蛋白保守结构域,从第4版香蕉基因组中共鉴定到222个NACs成员,比基于第2版基因组鉴定的结果多出41个[22],按照所在染色体位置分别命名为MaNAC001~MaNAC222(详见数据库附表S4)。如图 3所示,这222个香蕉NACs不均匀地分布在所有11条染色体上。其中,6号染色体编码最多、达32个NACs基因,其次是10号染色体、含28个NACs基因,8号染色体仅编码12个NACs基因。这些数量庞大的NACs基因可能源自芭蕉属谱系中发生的3轮全基因组复制(WGD)事件。此外,串联重复和区段复制在香蕉NAC家族的显著扩张中也可能起到重要作用[23-24]。
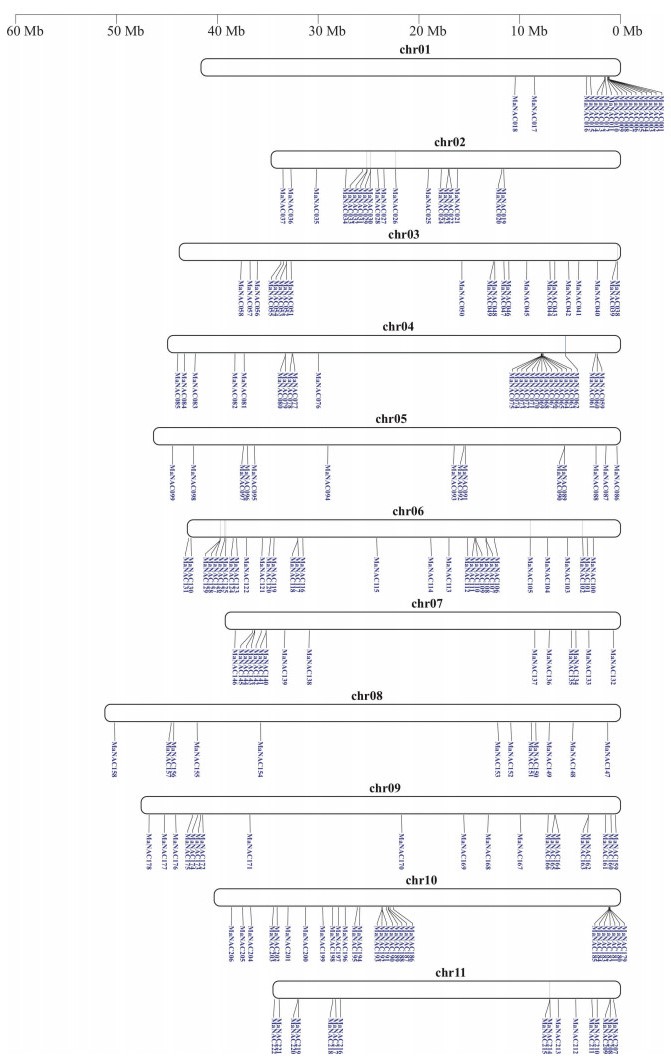
|
| 依据第4版香蕉基因组信息确定每个MaNAC的染色体位置,染色体编号标记在每条染色体的上方 Chromosomal location of each MaNAC was determined based on the banana genome information (V4.3), with the chromosome number marked above each chromosome 图 3 香蕉NAC基因家族成员的染色体定位 Fig. 3 Chromosomal locations of in NAC gene family members in banana |
使用默认参数,通过MCScanX对每个香蕉NAC基因的重复事件进行分析,在222个香蕉NACs中共鉴定出134对同源基因,其中1、2和4号染色体上共存在4对串联重复基因,其余130对同源基因均为区段复制,表明香蕉NACs基因进化的主要动力来源于基因的区段复制事件(详见数据库附表S5)。如图 4所示,香蕉NACs基因位于几乎所有染色体的同线性区块内。相应地,含有较多NACs基因的6号和10号染色体上区块间存在较多共线性关系。然而,1号染色体虽然也编码数量较多的NACs基因,但却基本聚集在染色体的末端位置,且仅存在1对串联重复和4对片段复制的同源基因,因而共线性关系也相对简单(图 4)。
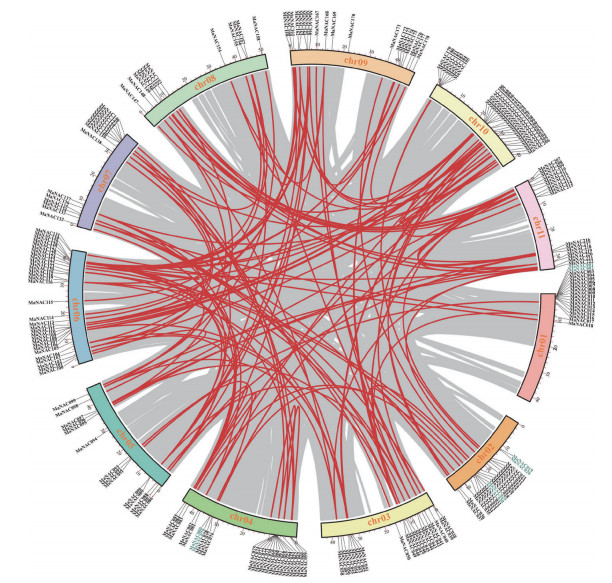
|
| 灰色线表示香蕉基因组中所有的共线性区块,红色线表示重复事件形成的NAC基因对 The gray lines represent all syntenic blocks in the banana genome and the red lines represent duplicated MaNAC gene pairs 图 4 香蕉NAC基因家族成员在染色体上的关联 Fig. 4 Interchromosomal association of NAC gene family members in banana |
2.4 香蕉NAC基因家族成员的进化分析
为研究香蕉NAC基因家族的进化特征,对香蕉、拟南芥和水稻NACs蛋白序列进行比较系统发育和亚群分布的分析。系统发育树将NAC家族蛋白划分为23个不同的亚群,比先前报道的分类[22]多出5个亚群。如图 5所示,没有任何香蕉NACs基因分布在S5、S13和S20亚群中,而另一些香蕉NACs基因则形成独立的亚群,如S1、S2、S3、S8、S21和S23。此外,绝大部分混合亚群同时含有来自单子叶植物和双子叶植物的NACs基因,表明这一超家族在单、双子叶物种中并非完全独立进化。总体而言,香蕉NACs基因在大多数进化分支中占据主导地位,尤其是S14亚群(图 5)。香蕉NACs基因在S14亚群中的数量最多达61个,其次是S15和S12亚群,分别包含36个和20个NACs基因。在香蕉NACs基因形成的6个独立亚群中,数目较多的是S1和S2亚群,分别含有24个和14个NACs基因,表明这些香蕉NACs很可能是在水稻中丢失后又在香蕉中分化获得的,暗示其可能具有特定功能。但转录组数据显示,仅有不到30% 的NACs基因在香蕉后熟过程中或低温胁迫下有表达(详见数据库附表S6),表明香蕉NACs基因高度冗余,或某些NACs基因可能只在特定条件下才表达及发挥功能。
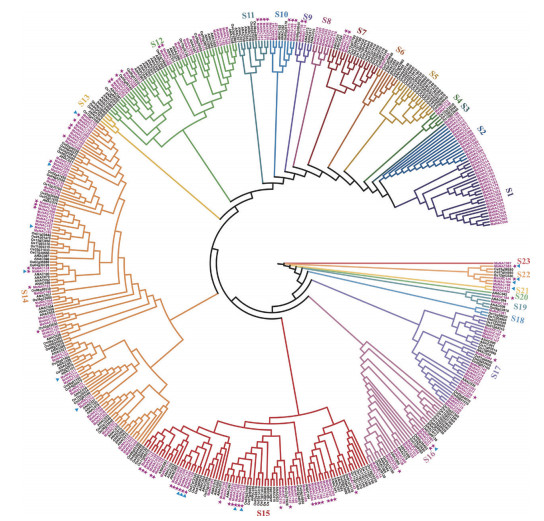
|
| 采用Clustal X比对蛋白质序列,通过IQ-Tree选择默认参数构建系统进化树;每个NAC亚族用特定颜色表示,编号在外圈;香蕉NAC蛋白名称用紫色标记,有表达和已有研究报道的香蕉NAC分别加注紫色星号和蓝色三角符号 Clustal X was used to compare protein sequences, and the phylogenetic tree was constructed by IQ-Tree in the default parameters; Each NAC subfamily is represented by a specific color and numbered; The name of MaNAC proteins is marked with purple, the expressed and previously reported MaNACs are marked with purple stars and blue triangles, respectively 图 5 香蕉与拟南芥、水稻NAC转录因子的系统发育分析 Fig. 5 Phylogenetic analysis of NAC transcription factors from banana, Arabidopsis thaliana and Oryza sativa |
2.5 香蕉NACs基因的结构与功能分析
基于蛋白比对构建系统发育树,在此背景下对有表达的66个香蕉NACs基因结构进行注释,明确其外显子- 内含子组成,并通过TBtools进行可视化。结果(图 6)表明,除MaNAC079仅含一个外显子、无内含子外,大部分香蕉NACs基因均含有3个外显子和2个内含子、占比65%。其中MaNAC062含有最多的外显子(44个)和内含子(43个),这是由于第4版香蕉基因组中的MaNAC062基因整合先前第2版基因组中的2个基因(5’端为旧版基因)的缘故。这些香蕉NACs基因中NAM结构域的长度,除MaNAC079(141 bp)外均介于315~411 bp。多数情况下,同一亚群内的NACs基因有较为相似且保守的结构,但同时也存在单个内含子丢失或获得的情况,但S7、S10、S12、S16和S22组内成员的基因结构相差较大。此外,同源基因对的外显子/ 内含子结构分析也进一步验证了系统发育树和重复事件的分析结果。另一方面,理化分析结果表明,所有香蕉NACs编码蛋白的平均长度为353个氨基酸,平均分子量为39.5 kDa,等电点范围为4.44~10.33。除MaNAC079外,所有香蕉NACs蛋白均为亲水性,且仅有不足15% 的NACs属于稳定蛋白(详见数据库附表S4)。
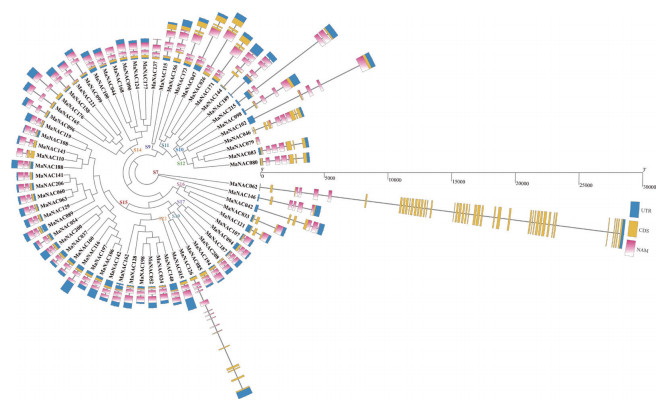
|
| 针对有表达的香蕉NACs基因进行系统进化树构建;红、黄和蓝色框分别表示NAM保守结构域、编码区CDS和非翻译区UTR;划分出的亚群编号标记在相应的分支根部 The phylogenetic tree was constructed for all expressed banana NAC genes; The red, yellow, and blue boxes represent the NAM conserved domain, CDS and UTR, respectively; The subgroup numbers are marked at the root of the corresponding branch 图 6 香蕉NAC家族蛋白聚类及基因结构 Fig. 6 Protein clustering and gene structure of NAC family members in banana |
图 5中用蓝色三角形标记了已报道的16个香蕉NACs基因(详见数据库附表S7),有5个分布在含香蕉NACs基因数目最多的S14亚群,其中3个(MaNAC177、MaNAC176、MaNAC207)被报道在乙烯诱导的香蕉后熟过程中发挥作用[25],而另外2个(MaNAC210、MaNAC217)则与次生细胞壁的形成密切相关[26]。然而,同样受乙烯上调的MaNAC121、MaNAC132和MaNAC126则分别位于S16、S21和S21亚群中。这些结果表明香蕉NACs基因在果实后熟过程中所起的作用可能是多元化的。此外,香蕉NACs还能对各类胁迫作出响应,通过多种渠道发挥抗逆作用。例如,MaNAC129通过调节气孔关闭及H2O2含量来增强香蕉的耐旱性[27],MaNAC085和MaNAC141均正向参与了香蕉对高盐和干旱胁迫的响应[28],MaNAC176可通过与WRKY家族蛋白互作,增强香蕉抵抗炭疽菌过程中病程相关基因的表达[29]。
2.6 miR164-NAC模块在香蕉后熟和冷害过程中的表达分析结合sRNA测序数据,首先对香蕉miR164家族6个成员的表达进行Northern杂交检测,分别选取后熟和冷害进程中不同时期的香蕉样品进行检测(图 7A)。其中,mac-miR164 d~f未检测到杂交信号,表明丰度极低,而另外3个丰度较高的成员均在香蕉后熟过程中上调表达,miR164a(图 7B)。此外,杂交结果显示伴随后熟进程,miR164a的表达在乙烯处理的香蕉果皮和果肉中均持续增强。1-MCP处理后,香蕉miR164a在果皮中的表达未受明显影响,但在果肉中出现先降低后上升现象(图 7B)。随后检测受miR164靶向调控的2个表达量较高的MaNAC165和MaNAC176的转录本在后熟过程中的丰度变化,图 7C显示,MaNAC176在果皮和果肉中均响应乙烯处理且随后熟进程逐渐下调表达,这与乙烯处理后果皮和果肉中miR164a的表达模式正好相反。1-MCP处理下MaNAC176的表达无明显变化,仅第8天在果皮中的表达显著升高。相比MaNAC176而言,MaNAC165在果皮和果肉中的转录本丰度更高,在乙烯处理后的果皮中其表达呈现先升高后快速下降的趋势,而在果肉中则持续下调(图 7C)。总体而言,1-MCP处理对MaNAC176和MaNAC165的表达影响不明显,特别是果肉样品。另一方面,在冷害的果皮样品中,香蕉miR164a的表达则被显著诱导,相应地,MaNAC165和MaNAC176两个靶基因的表达均出现明显下调(图 7B、图 7C)。
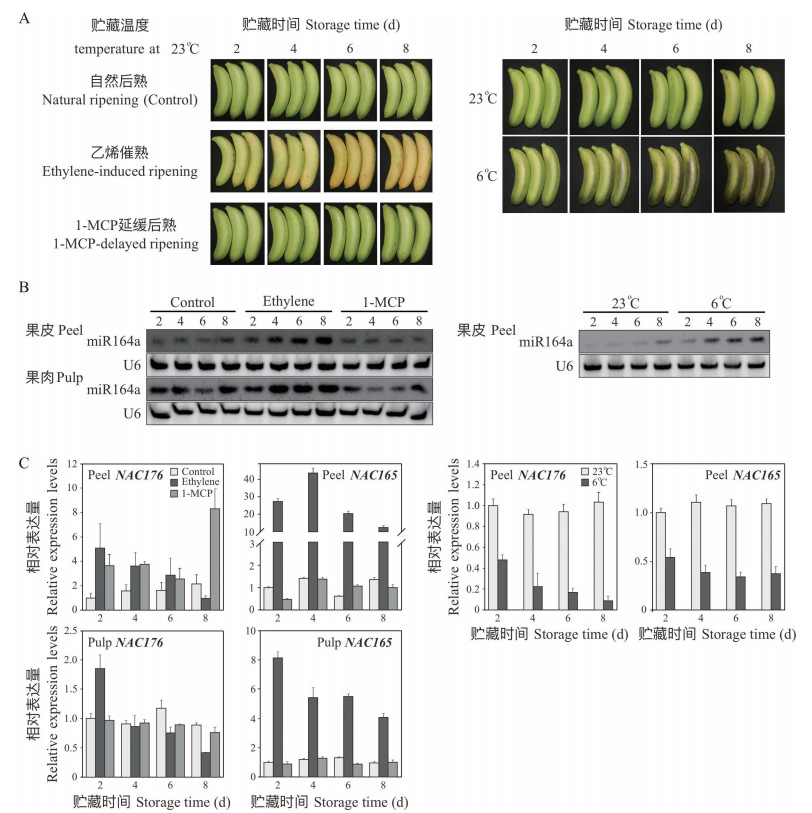
|
| A:香蕉在室温下后熟(自然后熟、乙烯催熟和1-MCP延缓后熟)及在低温胁迫下发生冷害的外观表型;B:香蕉miR164在果实后熟及冷害过程中的表达模式;C:香蕉miR164靶基因MaNAC176和MaNAC165在果实后熟及冷害过程中的表达模式 A: Phenotype of banana ripening at room temperature (natural ripening, ethylene-induced ripening and 1-MCP-delayed ripening) and chilling injury under low temperature stress; B: Expression patterns of miR164 in banana during fruit ripening and chilling injury; C: Expression patterns of miR164 target genes MaNAC176 and MaNAC165 in banana during fruit ripening and chilling injury 图 7 香蕉后熟和冷害过程中miR164-NAC模块的表达变化 Fig. 7 Expression changes of miR164-NAC module during banana ripening and chilling injury |
3 讨论
香蕉基因组共编码222个NACs成员,数量相当庞大。然而,转录组数据显示仅有不足30% 的NACs基因产生表达,特别是聚集于第1、4和10号染色体上的多个NACs基因均无表达,表明香蕉中该家族大多数成员存在功能冗余或时空表达特异性。另一方面,只有5% 的NACs基因具有miR164的靶切位点,这一比例在有表达的NACs基因中上升为20%。但相比香蕉中其他保守miRNA- 靶基因模块(如miR156-SPL、miR482-NBS-LRR)而言,香蕉miR164-NAC模块中的NAC靶标的范围仍较为局限,暗示该调控模块可能在香蕉的物种演化过程中出现较晚。
作为植物中一个高度保守的miRNA家族,miR164保守的靶基因NAC也是植物特有的一类转录因子,且miR164-NAC模块已被证实在调控植物生长发育和抗逆过程中发挥重要作用。研究发现,草莓des突变体相比野生型,其miR164a的成熟体在第19位核苷酸上存在碱基突变,miR164a通过靶向植物组织器官发育相关基因FvCUC2,影响草莓叶片和花器官的形态建成[30]。另有研究发现乙烯处理后,猕猴桃miR164的表达受到显著抑制,且与靶基因AdNAC6、AdNAC7的表达呈明显负相关。AdNAC6、AdNAC7能够结合猕猴桃乙烯生物合成、细胞壁降解和香气合成基因的启动子,进而调控猕猴桃品质形成及后熟进程[31]。在香蕉中,MaNAC089蛋白(又名MusaATAF2-like)可通过调节叶绿素降解和H2O2积累诱导香蕉叶片衰老[32],进一步研究发现,该蛋白还可以通过诱导细胞分裂素超敏和类黄酮积累,调节香蕉植株幼苗的生长[33]。
本研究基于sRNA高通量测序,发现香蕉miR164家族中共有6个成员(miR164a/b/c/d/e/f),其中miR164a/b/c的积累水平与其对应NAC靶基因的表达量呈明显负相关关系。然而,与同为呼吸跃变型果实的猕猴桃不同,香蕉中miR164的表达受到乙烯强烈诱导,且随后熟进程呈上调趋势,而其靶向的MaNAC176的表达则随香蕉后熟进程逐渐减弱。miR164另一个靶基因MaNAC165在乙烯催熟的果皮中出现先上升后下降的趋势,基本上与miR164的表达模式相反。NAC类转录因子在香蕉果实后熟调控中的作用已有不少相关报道。早期研究发现,MaNAC128、MaNAC129的表达在自然后熟、乙烯催熟和1-MCP延缓后熟的香蕉果实中,随着乙烯释放和后熟进程而不断增强[34]。作为转录激活子,MaNAC128和MaNAC129均能够结合乙烯合成关键基因MaACS1和MaACO1的启动子,激活乙烯的生物合成,加速香蕉后熟与品质形成[35]。与此相反,MaNAC197在香蕉后熟过程中明显下调,而其作为转录抑制子,通过结合MaACS1和MaACO1的启动子抑制其表达[34]。类似地,MaNAC085和MaNAC198也相继被报道是香蕉成熟衰老过程中的负调控因子[36-37]。总体来看,上述结果暗示着MaNAC176和MaNAC165很可能是香蕉后熟的负调控因子,而miR164则通过负调控这两个靶基因的表达,参与促进香蕉后熟进程中的乙烯合成。另一方面,本研究结果表明,香蕉miR164能够被低温诱导,同样可能通过负调控其靶基因MaNAC176和MaNAC165来应对冷害。前期报道多个香蕉NAC家族成员参与调控逆境胁迫响应,如MaNAC121通过与CBF1蛋白互作参与果实采后的低温胁迫应答[38],MaNAC129通过调节气孔开合度和H2O2含量增强香蕉抗旱性[27],MaNAC085和MaNAC141均能提高香蕉对高盐和干旱的耐受性[28]。近期报道指出,MaNAC127和MaNAC164可通过上调磷脂降解相关基因,促进磷脂降解积累磷脂酸,从而引发香蕉的冷害症状[39]。本研究发现的香蕉miR164-NAC165/176模块很可能是香蕉发生冷害的又一个关键调控通路,具体的分子机制还需通过转基因手段进一步证实。
4 结论本研究基于高通量测序及第4版香蕉基因组信息,鉴定出6个香蕉miR164家族成员,详细描述其染色体定位、序列同源性、前体二级结构及其与代表性物种间的系统进化关系。明确了香蕉miR164在不同NACs基因上的切割位点,并通过降解组和烟草瞬时共转化验证香蕉中多条miR164-NAC调控模块。利用更新版香蕉基因组注释,鉴定出222个香蕉NACs成员,明确其在基因组上的定位、重复事件、与拟南芥和水稻的系统发育关系,并结合转录组数据呈现部分香蕉NAC家族成员的基因结构。分别设置香蕉后熟和低温实验,通过小分子RNA杂交和qRT-PCR,分析鉴定到的香蕉miR164-NAC调控模块在后熟和冷害两种过程中的表达情况。综上,本研究系统筛选了与香蕉后熟及响应低温胁迫密切相关的miR164及其靶基因NAC转录因子,可为后续香蕉miR164-NAC模块的克隆及功能验证奠定基础。
| [1] |
朱孝扬, 李雪萍, 单伟, 邝健飞, 陈建业, 陈维信, 陆旺金. 香蕉贮运保鲜技术研究进展[J]. 热带作物学报, 2020, 41(10): 2013-2021. DOI:10.3969/j.issn.1000-2561.2020.10.008 ZHU X Y, LI X P, SHAN W, KUANG J F, CHEN J Y, CHEN W X, LU W J. Research progress of technologies in banana fruit preservation and transportation[J]. Chinese Journal of Tropical Crops, 2020, 41(10): 2013-2021. DOI:10.3969/j.issn.1000-2561.2020.10.008 |
| [2] |
李倩, 沈春生, 林启昉, 王慧, 林河通, 陈建业, 范中奇. 采后香蕉果实冷害发生与控制技术研究进展[J]. 果树学报, 2021, 38(5): 817-827. DOI:10.13925/j.cnki.gsxb.20200500 LI Q, SHEN C S, LIN Q F, WANG H, LIN H T, CHEN J Y, FAN Z Q. Advances in research on the chilling injury occurrence and control technologies of postharvest banana fruit[J]. Journal of Fruit Science, 2021, 38(5): 817-827. DOI:10.13925/j.cnki.gsxb.20200500 |
| [3] |
HE M, KONG X, JIANG Y, QU H, ZHU H. MicroRNAs: Emerging regulators in horticultural crops[J]. Trends in Plant Science, 2022, 27: 936-951. DOI:10.1016/j.tplants.2022.03.011 |
| [4] |
滕露, 于月华, 何茹月, 陈全家, 倪志勇. 大豆miR164家族的生物信息学分析[J]. 大豆科学, 2018, 37(5): 704-709. DOI:10.11861/j.issn.1000-9841.2018.05.0704 TENG L, YU Y H, HE R Y, CHEN Q J, NI Z Y. Bioinformatics analysis of soybean miR164 family[J]. Soybean Science, 2018, 37(5): 704-709. DOI:10.11861/j.issn.1000-9841.2018.05.0704 |
| [5] |
KIM J H, WOO H R, KIM J, LIM P O, LEE I C, CHOI S H, HWANG D, NAM H G. Trifurcate feed-forward regulation of age-dependent cell death involving miR164 in Arabidopsis[J]. Science, 2009, 323(5917): 1053-1057. DOI:10.1126/science.1166386 |
| [6] |
WU X M, LIU M Y, GE X X, XU Q, GUO W W. Stage and tissue-specific modulation of ten conserved miRNAs and their targets during somatic embryogenesis of Valencia sweet orange[J]. Planta, 2011, 233(3): 495-505. DOI:10.1007/s00425-010-1312-9 |
| [7] |
杨春文. 番茄miR164对花器官形成和果实发育的调控研究[D]. 重庆: 重庆大学, 2012. DOI: 10.7666/d.y2152976. YANG C W. Studies on the regulation of miR164 on flower organ formation and fruit development in tomato[D]. Chongqing: Chongqing University, 2012. DOI: 10.7666/d.y2152976. |
| [8] |
WEI H, YORDANOV Y S, GEORGIEVA T, LI X, BUSOV V. Nitrogen deprivation promotes Populus root growth through global transcriptome reprogramming and activation of hierarchical genetic networks[J]. New Phytologist, 2013, 200(2): 483-497. DOI:10.1111/nph.12375 |
| [9] |
李春贺, 阴祖军, 刘玉栋, 沈法富. 盐胁迫条件下不同耐盐棉花miRNA差异表达研究[J]. 山东农业科学, 2009(7): 12-17. DOI:10.3969/j.issn.1001-4942.2009.07.004 LI C H, YIN Z J, LIU Y D, SHEN F F. Study on miRNA differential expression of different salt tolerant cotton under salt stress[J]. Shandong Agricultural Sciences, 2009(7): 12-17. DOI:10.3969/j.issn.1001-4942.2009.07.004 |
| [10] |
MALLORY A C, DUGAS D V, BARTEL D P, BARTEL B. MicroRNA regulation of NAC-domain targets is required for proper formation and separation of adjacent embryonic, vegetative, and floral organs[J]. Current Biology, 2004, 14(12): 1035-1046. DOI:10.1016/j.cub.2004.06.022 |
| [11] |
GUO H S, XIE Q, FEI J F, CHUA N H. MicroRNA directs mRNA cleavage of the transcription factor NACl to downregulate auxin signals for Arabidopsis lateral root development[J]. Plant Cell, 2005, 17(5): 1376-1386. DOI:10.1105/tpc.105.030841 |
| [12] |
ZHAN J, CHU Y, WANG Y, DIAO Y, ZHAO Y, LIU L, WEI X, MENG Y, LI F, GE X. The miR164-GhCUC2-GhBRC1 module regulates plant architecture through abscisic acid in cotton[J]. Plant Biotechnology Journal, 2021, 19(9): 1839-1851. DOI:10.1111/pbi.13599 |
| [13] |
LIN D, ZHU X, QI B, GAO Z, TIAN P, LI Z, LIN Z, ZHANG Y, HUANG T. SlMIR164A regulates fruit ripening and quality by controlling SlNAM2 and SlNAM3 in tomato[J]. Plant Biotechnology Journal, 2022, 20(8): 1456-1469. DOI:10.1111/pbi.13824 |
| [14] |
OLSEN A N, ERNST H A, LEGGIO L L, SKRIVER K. NAC transcription factors: Structurally distinct, functionally diverse[J]. Trends in Plant Science, 2005, 10(2): 79-87. DOI:10.1016/j.tplants.2004.12.010 |
| [15] |
王芳, 孙立娇, 赵晓宇, 王婕婉, 宋兴舜. 植物NAC转录因子的研究进展[J]. 生物技术通报, 2019, 35(4): 88-93. DOI:10.13560/j.cnki.biotech.bull.1985.2018-0905 WANG F, SUN L J, ZHAO X Y, WANG J W, SONG X S. Research progresses on plant NAC transcription factors[J]. Biotechnology Bulletin, 2019, 35(4): 88-93. DOI:10.13560/j.cnki.biotech.bull.1985.2018-0905 |
| [16] |
ERNST H A, OLSEN A N, SKRIVER K, LEGGIO L L. Structure of the conserved domain of ANAC, a member of the NAC family of transcription factors[J]. EMBO Reports, 2004, 5(3): 297-303. DOI:10.1038/sj.embor.7400093 |
| [17] |
WELNER D H, LINDEMOSE S, GROSSMANN J G, MOLLEGAARD N E, OLSEN A N, HELGSTRAND C, SKRIVER K, LEGGIO L L. DNA binding by the plant-specific NAC transcription factors in crystal and solution: A firm link to WRKY and GCM transcription factors[J]. Biochemical Journal, 2012, 444(3): 395-404. DOI:10.1042/BJ20111742 |
| [18] |
ZHU H, ZHANG Y, TANG R, QU H, DUAN X, JIANG Y. Banana sRNAome and degradome identify microRNAs functioning in differential responses to temperature stress[J]. BMC Genomics, 2019, 20(1): 33. DOI:10.1186/s12864-018-5395-1 |
| [19] |
CHEN C, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [20] |
LIU Y, WANG L, CHEN D, WU X, HUANG D, CHEN L, LI L, DENG X, XU Q. Genome-wide comparison of microRNAs and their targeted transcripts among leaf, flower and fruit of sweet orange[J]. BMC Genomics, 2014, 15(1): 695. DOI:10.1186/1471-2164-15-695 |
| [21] |
曾俊. 香蕉后熟过程中相关miRNA的鉴定及转录调控初探[D]. 广州: 中国科学院华南植物园, 2020. ZENG J. Identification and transcriptional regulation of miRNAs related to banana fruit ripening[D]. Guangzhou: South China Botanical Garden, Chinese Academy of Sciences, 2020. |
| [22] |
LI B, FAN R, YANG Q, HU C, SHENG O, DENG G, DONG T, LI C, PENG X, BI F, YI G. Genome-wide identification and characterization of the NAC transcription factor family in Musa acuminata and expression analysis during fruit ripening[J]. International Journal of Molecular Sciences, 2020, 21(2): 634. DOI:10.3390/ijms21020634 |
| [23] |
D'HONT A, DENOEUD F, AURY J M, BAURENS F C, CARREEL F, et al. The banana (Musa acuminata) genome and the evolution of monocotyledonous plants[J]. Nature, 2012, 488(7410): 213-217. DOI:10.1038/nature11241 |
| [24] |
WANG Z, MIAO H, LIU J, XU B, YAO X, et al. Musa balbisiana genome reveals subgenome evolution and functional divergence[J]. Nature Plants, 2019, 5(8): 810-821. DOI:10.1038/s41477-019-0452-6 |
| [25] |
SHAN W, KUANG J F, CHEN L, XIE H, PENG H H, XIAO Y Y, LI X P, CHEN W X, HE Q G, CHEN J Y, LU W J. Molecular characterization of banana NAC transcription factors and their interactions with ethylene signalling component EIL during fruit ripening[J]. Journal of Experimental Botany, 2012, 63(14): 5171-5187. DOI:10.1093/jxb/ers178 |
| [26] |
NEGI S, TAK H, GANAPATHI T R. Functional characterization of secondary wall deposition regulating transcription factors MusaVND2 and MusaVND3 in transgenic banana plants[J]. Protoplasma, 2016, 253(2): 431-446. DOI:10.1007/s00709-015-0822-5 |
| [27] |
NEGI S, TAK H, GANAPATHI T R. A banana NAC transcription factor (MusaSNAC1) impart drought tolerance by modulating stomatal closure and H2O2 content[J]. Plant Molecular Biology, 2018, 96(4-5): 457-471. DOI:10.1007/s11103-018-0710-4 |
| [28] |
TAK H, NEGI S, GANAPATHI T R. Banana NAC transcription factor MusaNAC042 is positively associated with drought and salinity tolerance[J]. Protoplasma, 2017, 254(2): 803-816. DOI:10.1007/s00709-016-0991-x |
| [29] |
SHAN W, CHEN J Y, KUANG J F, LU W J. Banana fruit NAC transcription factor MaNAC5 cooperates with MaWRKYs to enhance the expression of pathogenesis-related genes against Colletotrichum musae[J]. Molecular Plant Pathology, 2016, 17(3): 330-338. DOI:10.1111/mpp.12281 |
| [30] |
ZHENG G, WEI W, LI Y, KAN L, WANG F, ZHANG X, LI F, LIU Z, KANG C. Conserved and novel roles of miR164-CUC2 regulatory module in specifying leaf and floral organ morphology in strawberry[J]. New Phytologist, 2019, 224(1): 480-492. DOI:10.1111/nph.15982 |
| [31] |
WANG W Q, WANG J, WU Y Y, LI D W, ALLAN A C, YIN X R. Genome-wide analysis of coding and non-coding RNA reveals a conserved miR164-NAC regulatory pathway for fruit ripening[J]. New Phytologist, 2019, 225(4): 1618-1634. DOI:10.1111/nph.16233 |
| [32] |
BHAKTA S, NEGI S, TAK H, SINGH S, GANAPATHI T R. MusaATAF2 like protein, a stress-related transcription factor, induces leaf senescence by regulating chlorophyll catabolism and H2O2 accumulation[J]. Physiologia Plantarum, 2022, 174(1): e13593. DOI:10.1111/ppl.13593 |
| [33] |
BHAKTA S, NEGI S, TAK H, SINGH S, GANAPATHI T R. Musa ATAF 2-like protein regulates shoot development and multiplication by inducing cytokinin hypersensitivity and flavonoid accumulation in banana plants[J]. Plant Cell Reports, 2022, 41(5): 1197-1208. DOI:10.1007/s00299-022-02849-y |
| [34] |
单伟. NAC类转录因子调控香蕉果实诱导耐冷性及成熟的机制分析[D]. 广州: 华南农业大学, 2016. SHAN W. Mechanism analysis of NAC transcription factors in regulation of chilling tolerance and ripening in banana fruit[D]. Guangzhou: South China Agricultural University, 2016. |
| [35] |
WEI W, YANG Y Y, CHEN J Y, LAKSHMANAN P, KUANG J F, LU W J, SHAN W. MaNAC029 modulates ethylene biosynthesis and fruit quality and undergoes MaXB3-mediated proteasomal degradation during banana ripening[J]. Journal of Advanced Research, 2022, S2090-1232(22)00270-3. DOI:10.1016/j.jare.2022.12.004 |
| [36] |
YAN H, JIANG G, WU F, LI Z, XIAO L, JIANG Y, DUAN X. Sulfoxidation regulation of transcription factor NAC42 influences its functions in relation to stress-induced fruit ripening in banana[J]. Journal of Experimental Botany, 2021, 72(2): 682-699. DOI:10.1093/jxb/eraa474 |
| [37] |
CHEN T H, WEI W, SHAN W, KUANG J F, CHEN J Y, LU W J, YANG Y Y. MaHDA6-MaNAC154 module regulates the transcription of cell wall modification genes during banana fruit ripening[J]. Postharvest Biology and Technology, 2023, 198: 112237. DOI:10.1016/j.postharvbio.2022.112237 |
| [38] |
SHAN W, KUANG J F, LU W J, CHEN J Y. Banana fruit NAC transcription factor MaNAC1 is a direct target of MaICE1 and involved in cold stress through interacting with MaCBF1[J]. Plant, Cell & Environment, 2014, 37(9): 2116-2127. DOI:10.1111/pce.12303 |
| [39] |
SONG C, WU M, ZHOU Y, GONG Z, YU W, ZHANG Y, YANG Z. NAC-mediated membrane lipid remodeling negatively regulates fruit cold tolerance[J]. Horticulture Research, 2022, 9: uhac039. DOI:10.1093/hr/uhac039 |
(责任编辑 张辉玲)
 2023, Vol. 50
2023, Vol. 50