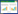文章信息
基金项目
- 广东省重点领域研发计划项目(2020B02022001);广东省乡村振兴战略专项资金种业振兴项目(2022-NJS-00-005);国家自然科学基金(32272713)
作者简介
- 金庆敏(1986—),女,硕士,助理研究员,研究方向为蔬菜分子育种与分子生物学,E-mail:dzjinqingmin@126.com; 王瑞,硕士,副研究员,华南农业大学硕士研究生校外导师。主要从事蔬菜遗传育种与分子生物学研究,特别在瓜类疫病抗性机理方面有深入研究。近5年主持国家自然科学基金面上项目、青年基金等6项,参与国家重点研发计划、省部级项目等5项。以第一或通信作者在国内外期刊发表学术论文11篇;获广东省农业技术推广奖一等奖1项;授权专利13件;获得审定或登记的蔬菜品种5个,植物新品种权3个.
通讯作者
- 王瑞(1984—),女,硕士,副研究员,研究方向为蔬菜育种与分子生物学,E-mail:wrui_1999@163.com.
文章历史
- 收稿日期:2023-07-27
2. 广东省农业科学院设施农业研究所,广东 广州 510640
2. Institute of Faclity Agriculture, Guangdong Academy of Agricultural Sciences, Guangzhou 510640, China
【研究意义】黄瓜(Cucumis sativus L.)是世界各国普遍栽培的重要蔬菜作物之一[1-2]。我国黄瓜种植面积约占世界种植总面积的50% 以上,黄瓜产量及种植规模均为世界第一[3-5]。此外,黄瓜一年内可多茬栽培,供应时间长,经济价值高,黄瓜产业在我国蔬菜中占有重要地位[1, 6]。目前市场上黄瓜品种众多、遗传背景狭窄、品种间相似性高[7-8],广泛收集黄瓜优质种质资源,并进行系统的鉴定、评价、分类和保存,对黄瓜新品种的选育、种质资源的保护及育种工作者权益的维护均具有重要意义[9]。【前人研究进展】随着生物科学的进步,SSR(Simple sequence repeat)分子标记已成为一种较为成熟的生物技术,可快捷有效地对主要作物及主要蔬菜种质资源遗传背景进行分析及鉴定[10],有助于研究植物亲缘关系[11]。SSR分子标记技术在水稻[12]、小麦[13]、玉米[14]、油菜[15]、辣椒[16-17]、茄子[18]、大白菜[19-20]、芥菜[21]、菜心[22]、黄瓜[3, 23-26]、苦瓜[27-28]、节瓜[29]、南瓜[30]、大蒜[31]等多种作物中均有广泛应用。近年来,SSR分子标记技术在黄瓜种子纯度鉴定[23, 32-34]、种质资源多样性分析[9, 35]、重要功能基因定位等方面发挥了重要作用[36-37]。Danin等[38]利用SSR标记研究了不同葫芦科蔬菜品系的多态性,发现7个SSR中有4个可用于黄瓜品系的多态性检测,并在11个黄瓜品种上进行了验证。陈劲枫等[11]综合109个SSR位点和398个RAPD条带对22份黄瓜材料进行聚类分析,结果发现可分为两类:CS群(C. hytivus、黄瓜、西南野黄瓜C. sativus var. hardwickii及野黄瓜C. hystrix)和CM群(非洲角黄瓜C. metuliferus、甜瓜、菜瓜C. melo var. conomon及野生小甜瓜C. melo ssp. agrestis)。沈镝等[39]利用13对来自黄瓜和甜瓜的SSR、ESTSSR引物,对273份西双版纳黄瓜种质群体进行遗传分析,发现遗传不同样本、不同种质以及不同来源地群体间均存在一定程度的差异。吕婧等[40]通过来自不同国家和地区的30份代表性黄瓜种质资源进行SSR扩增分析,发现23对引物可以清晰检测出亚洲、欧美和印度群体遗传多样性,较客观地反映了黄瓜群体结构和地域性差异等信息。王蕾等[3]以来源不同的32份黄瓜栽培品种为材料,利用筛选出的17对SSR引物构建指纹图谱和分析遗传多样性,结果表明这17对引物能有效区分出所有供试材料,在遗传相似系数(GS)为0.760处可将32份黄瓜品种聚为5类。【本研究切入点】目前黄瓜品种众多,但对黄瓜核心种质资源遗传多样性的了解尚不充分。为进一步了解本课题组所收集黄瓜种质资源的多样性,明确黄瓜种质资源的遗传背景、分析其多态性及各黄瓜材料之间的亲缘关系,促进黄瓜种质资源的利用,本研究通过筛选有效的SSR标记来评估和描述黄瓜核心种质资源的遗传多样性。基于筛选得到的SSR标记,对黄瓜核心种质资源进行聚类分析,探索黄瓜核心种质资源之间的遗传关系及群体结构,深入了解不同种质资源之间的相似性和差异性,并揭示潜在的优良基因组合,为黄瓜育种和种质资源保护提供重要参考和指导。【拟解决的关键问题】本研究通过黄瓜基因组共设计了995对SSR引物,从中筛选出66对引物对50份黄瓜核心种质资源的11个重要外观果实性状进行调查,并通过SSR分子标记分析对50个黄瓜核心种质进行聚类分析,以了解黄瓜种质资源间的遗传相似性和差异性,分析其相关基因型、群体结构及可能的亲缘关系,为黄瓜遗传改良和种质资源管理及保存提供科学依据,为广东乃至华南地区高品质黄瓜遗传育种组合提供重要理论参考。
1 材料与方法 1.1 试验材料供试材料为广东省农业科学院蔬菜研究所收集的来自全国各地的50份黄瓜核心种质,其中华北型40份、华南型9份、华南型与华北型杂交的中间型黄瓜材料1份。50份种质材料均挑选25粒颗粒饱满且一致的种子,于2021年2月中旬浸种催芽,播种于穴盘育苗;3月上旬(长出1~3片真叶)移栽于该所白云基地。每份材料选取生长一致的20株进行种植,采用深沟高畦,畦宽1.8~2.0 m,畦高30 cm,株距30 cm,栽培管理按常规进行。
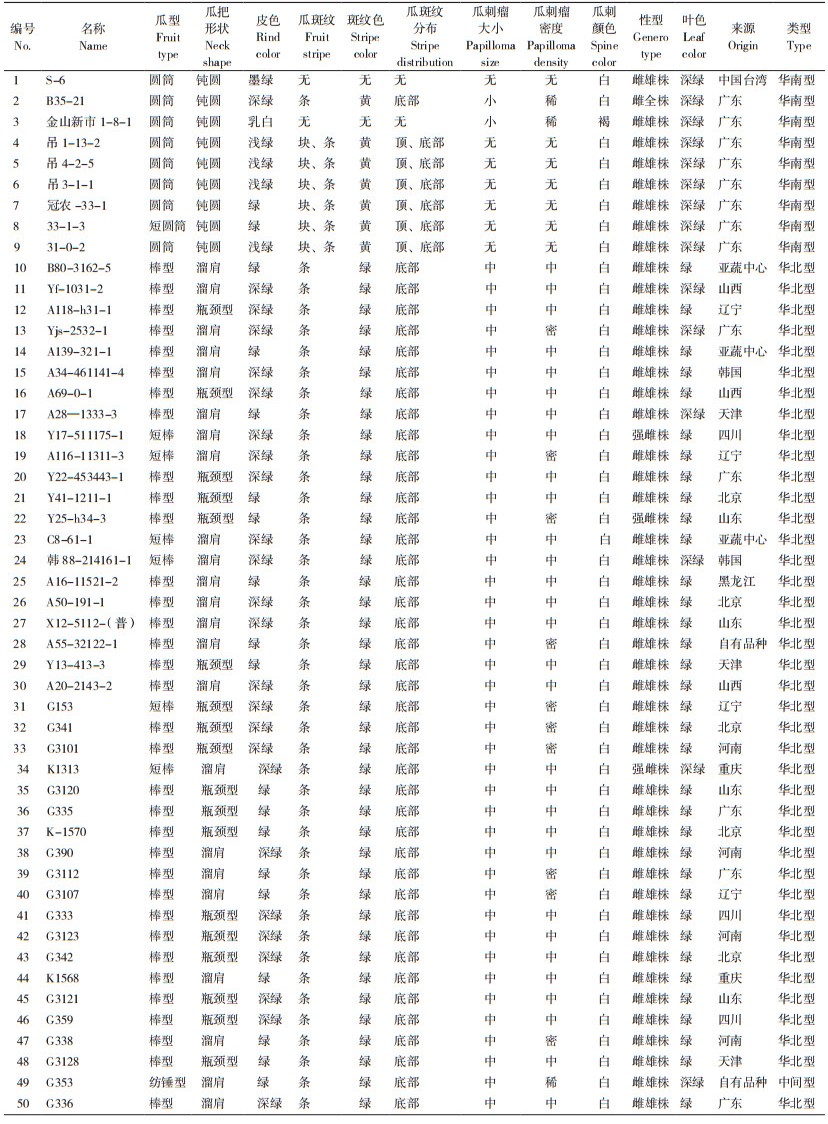
1.2 测定项目及方法1.2.1 黄瓜核心种质的农艺性状调查 在黄瓜盛果期对50份黄瓜种质材料进行性状调查统计,对瓜型、瓜把性状、皮色、瓜斑纹、刺瘤、性型、叶色等11个性状进行调查和登记。瓜型分为圆筒型、棒型、短棒型、纺锤型等;瓜把性状分为钝圆、溜肩、瓶颈型;皮色分为绿色、深绿、浅绿、乳白、墨绿;瓜斑纹分为块状、条状或无;斑纹色分为绿、黄或无;瓜斑纹分布分为顶部、底部或无;瓜刺瘤大小分为中、小或无;瓜刺瘤密度分为稀、密或无;瓜刺颜色分为白色或褐色;性型分为雌雄株、雌全株、强雌株;叶色分为绿、深绿。分类依据分参照李锡香等编著的《黄瓜种质资源描述规范和数据标准》[41]。
1.2.2 黄瓜DNA的提取 每份材料随机选取10株,每株取嫩叶一片,混合后于研钵中加液氮研磨,DNA的提取参考姚春鹏等[42]改良版的CTAB提取方法。DNA样品检测时各取DNA样品原液2 μL并稀释至100 μL,取稀释后的DNA样品1 μL,用ddH2O作为空白对照,校正零点,于核酸蛋白分析仪上测定OD260、OD280的值。取5 μL模板DNA原液,加入2 μL上样缓冲液,在加入Golden-View染料的0.8% 琼脂糖凝胶上电泳,电泳缓冲液为1×TBE,电压为5 V/cm,稳压电泳20 min,然后于凝胶成像系统下观察拍照。
1.2.3 SSR分子标记的选择 在黄瓜基因组数据库网站(http://www.cucurbitgenomics.org/)下载黄瓜已经公布的SSR分子标记序列,设计995对SSR引物送生工生物公司合成,从中筛选出66对SSR引物对50份黄瓜种质资源进行遗传多样性分析。选取表现形式差异大的10份黄瓜DNA样品用于分子标记筛选,选用稳定性好、条带清晰且多态性好的引物序列。
1.2.4 PCR反应体系和扩增程序 PCR扩增体系为20 μL反应体系:2 μL 10×buffer,2 μL dNTPs(2.5 mmol/L),1.5 μL Mg2+(20 mmol/L),0.4 μL上下游混合引物(10 mmol/μL),0.2 μL Taq DNA聚合酶(5 U/μL),2 μL DNA模板(50 ng/μL),11.9 μL灭菌H2O。扩增程序为94℃预变性3 min,94℃变性30 s,54℃退火30 s,72℃延伸30 s,循环40次,72℃延伸10 min。
1.2.5 SSR标记的凝胶电泳 扩增产物在双垂直非变性浓度为8% 的聚丙烯酰胺凝胶上电泳,120 V稳压1.5 h,电泳结束后进行0.1% AgNO3银染15 min;银染后用2% NaOH、0.4% 甲醛、0.04% Na2CO3显色,显色后在灯箱上拍照分析。
1.2.6 数据处理 根据电泳图谱进行数据统计与整理,从上往下对等位基因数量进行统计。对每个样品的条带,有条带记为“1”,无条带记为“0”,由此形成0和1原始矩阵。利用NTSYS2.10e软件进行分析得出遗传相似系数并进行聚类分析,绘制聚类图。
2 结果与分析 2.1 50份黄瓜核心种质的性状调查结果分析对50份黄瓜种质资源的11个性状调查结果(表 1)显示,40份华北型黄瓜种质材料的瓜型主要为棒型或短棒型,瓜把为溜肩或瓶颈型,条状瓜斑纹、瓜斑纹多为绿色且主要分布在瓜底部,瓜刺瘤中等大小、密度中等或密、白色瓜刺,大多为雌雄株(仅3份为强雌株),叶片大多为绿色。9份华南型黄瓜种质瓜型主要为圆筒型,只有1份为短圆筒型;瓜把较为一致,全部为钝圆型;皮色较为多样,其中墨绿1份、乳白1份、浅绿4份、绿色2份、深绿1份;瓜斑纹块状条状6份、无瓜斑纹2份、条状1份;瓜斑纹颜色黄色7份,无色2份;瓜斑纹分布在顶部及底部6份、底部1份、无瓜斑纹1份;无瓜刺瘤7份、具有小瓜刺瘤2份,且瓜刺瘤稀疏;瓜刺颜色与华北型相同,大多为白色、但有1份为褐色;大多为雌雄株,其中1份为雌全株;华南型黄瓜叶片均为深绿色。中间型黄瓜瓜型为纺锤型,瓜把形状为溜肩型,果皮绿色,条形绿色瓜斑纹、分布于底部,瓜刺瘤中等大小但较为稀疏,雌雄株,叶片深绿。
|
2.2 66对SSR引物的多态性结果分析
66对SSR引物在50份黄瓜核心种质资源进行多态性检测,共获得32对稳定性好、条带清晰、多态性高的引物(表 2)。本研究中32对SSR引物在50份种质中共扩增出280条等位基因片段,其中216条多态性片段、占片段总数的77.1%,平均每对引物产生6.75条多态性片段(图 1)。根据数据统计得到0-1矩阵,利用NTSYS2.10e软件进行分析得出50份黄瓜种质资源最大遗传相似性系数是1.00,最小遗传相似性系数为0.62。其中,与其他材料相似性最小的为3号,是广东地区的一个地方品种。相似性系数为1.00的材料有40、41、46,从来源看,41、46来自四川,40来自辽宁,虽然地区不同,但是材料相似。从图 1所示的扩增结果来看,50份黄瓜核心种质间多态性非常明显,表明32对引物可有效区分种质。在所选出的32对引物中,4B-05、6B-26、6B-27、6B-28、6B-32、6B-39、6B-48、6B-61、6B-62等9对引物扩增出的多态性片段较少,仅为1~3条,但其在材料间的差异较大,可作为种质鉴定及种子纯度鉴定的指纹标记引物。

|
| 图 1 部分SSR分子标记扩增产物电泳检测图 Fig. 1 Electrophoresis detection diagram of amplified products labeled with partial SSR markers |
2.3 50份黄瓜核心种质的聚类结果分析
利用NTSYS2.10e软件对多态性片段进行聚类分析,结果(图 2)表明,50份黄瓜种质的遗传相似系数(GS)最低为0.62,最高达1.00。

|
| 图 2 50份黄瓜核心种质的遗传多样性聚类分析 Fig. 2 Clustering analysis for genetic diversity of 50 cucumber core germplasms |
相似系数为0.620处,50份黄瓜种质在聚类图上聚为Ⅰ类。
相似系数为0.754处,50份黄瓜种质可聚为7类:(1)Ⅰ类仅包含来源于我国台湾省的S-6,果皮墨绿、无瓜斑、白刺的华南型黄瓜材料,是50份黄瓜种质材料中唯一果皮墨绿、且既无瓜斑纹也无刺瘤的材料;(2)Ⅱ类包含40份材料,分为两个亚组,包含所有华北型材料,40份材料瓜型均为棒型或短棒型、瓜把为溜肩型或瓶颈型、瓜皮为绿色或深绿色、瓜斑纹绿色且位于底部、均有瓜刺瘤、刺瘤密度中等或密、瓜刺瘤大小均为中等、叶片颜色绝大部分为绿色;(3)Ⅲ类仅包含华南型1份材料,50份材料中唯一一个性型为雌全株的是华南型黄瓜中唯一瓜皮色为深绿色的材料;(4)Ⅳ类包含华南型1份、华南型与华北型黄瓜杂交后中间型材料1份,其中中间型材料是50份材料中唯一一份瓜型为纺锤型的材料,瓜把表现为华北型黄瓜的溜肩型,瓜刺瘤大小为中型,但瓜刺瘤密度则表现为华南型的稀疏型;(5)Ⅴ类包含华南型材料1份,是50份材料中唯一一份皮色为乳白色且瓜刺颜色为褐色的黄瓜材料;(6)Ⅵ类包含华南型材料3份,分两个亚组,瓜皮色均为浅绿色;(7)Ⅶ类包含华南型材料2份,Ⅶ类与Ⅵ类的区别在于瓜的皮色,该组材料的皮色均为绿色。
相似系数为0.851处,50份黄瓜材料则可聚为9类:Ⅰ类包含华南型材料1份;Ⅱ类包含华北型材料40份,分为2个亚组;Ⅲ类包含华南型材料1份;Ⅳ类包含华南型材料1份;Ⅴ类包含中间型材料1份;Ⅵ类包含华南型材料1份;Ⅶ类包含华南型材料3份,分为2个亚组;Ⅷ类包含华南型材料1份;Ⅸ类包含华南型材料1份。
从种质来源上看,50份黄瓜种质材料并没有严格按照东北、华北、华中、华南、西南等地域因素聚为一类,同一来源的各类群种质均有一定差异,表明SSR分子标记可在一定程度上反映种质资源亲缘关系及遗传多样性。从类型上看,所有华北型黄瓜聚为一类,而华南型黄瓜则比较分散,表明华北型黄瓜材料间相似系数较高、亲缘关系较近、遗传背景较窄;华南型黄瓜材料间相似系数较低、亲缘关系较远、遗传背景较宽、遗传多样性丰富。
3 讨论种质资源是遗传育种的基础,黄瓜品种间多态性远低于同属甜瓜[43]。市面上的黄瓜栽培品种更是存在遗传背景狭窄、遗传多样性差等问题,收集、鉴定、保存多态性丰富的种质资源,拓展黄瓜的遗传多样性对黄瓜的遗传育种及生产有着极为重要的意义。本研究利用筛选出的32对SSR引物在50份黄瓜种质资源中共检测出280个基因位点,每个引物平均检测出6.75个位点,多态性位点216个,引物多态性达77.1%,均高于王蕾等[3]、李翔等[44]、苗晗等[45]研究结果,其中王蕾等[3]利用17对引物对32份黄瓜品种进行聚类分析,发现每条引物平均检测基因数仅为4.59个,引物多态性仅为71.8%,表明本研究所筛选的SSR引物具有较高的多态性,可用于50份黄瓜种质材料的遗传多样性分析和鉴定。
遗传相似系数越小,变幅越大、物种的遗传分化越大、遗传多样性越高、遗传背景越复杂[45]。本研究中50份黄瓜种质材料的遗传相似系数分布于0.62~1.00之间,遗传相似系数变化范围高于杨福强等[46]研究结果,表明其具有较为丰富的多态性。本研究中,在遗传相似系数为0.754处的40份华北型黄瓜材料聚为一类,此结果与所调查黄瓜材料的性状特征表现较为一致。40份华北型黄瓜材料性状调查结果显示,其瓜型多为棒型,瓜把多为溜肩型或瓶颈型,皮色多为绿色或深绿,瓜斑纹绿色条形,多分布在底部,瓜刺瘤大小中等,密度中等,绝大多数为雌雄株。9份华南型黄瓜材料聚为6类,具体为Ⅰ类、Ⅲ类、Ⅳ类、Ⅴ类、Ⅵ类、Ⅶ类,此结果与所调查华南型黄瓜性状表现结果也较为一致。本研究所调查华南型黄瓜材料的瓜皮颜色、瓜刺瘤、瓜皮斑纹等性状具有多样化,表明华南型黄瓜种质资源具有丰富的遗传多样性,且与史建磊等[5]、苗晗等[45]等研究结果一致。史建磊等[5]研究得出,48份华南型黄瓜材料的遗传相似系数(GS)介于0.375~1.000之间,本研究遗传相似系数结果仅为0.620~1.000之间,可能与本研究中华南型材料特异性不明显有关,本研究所用的大部分材料为华北型,华南型黄瓜材料仅有9份,同时也表明我国华北型黄瓜遗传背景狭窄。
从聚类图上看,与本研究大多数黄瓜种质材料遗传距离较远的为3号材料,这是广东省的地方品种,该材料与其他黄瓜材料基因交换较少。1号黄瓜材料来自我国台湾省,其单独聚为一类,表明1号材料与其他材料遗传距离较远。由华北型黄瓜材料与华南型黄瓜杂交所得的中间型黄瓜材料,为50份材料中唯一一份果型为纺锤型的材料,该材料瓜把表现为华北型黄瓜的溜肩型,瓜刺瘤大小为中型,但瓜刺瘤密度则表现为华南型的稀疏型,另在遗传相似系数为0.705处,49号中间型材料及1、2、9号华南型黄瓜材料与华北型黄瓜材料聚为一类,表明9份华南型黄瓜材料中,1、2、9号材料与华北型黄瓜材料的亲缘性高于3、4、5、6、7、8号材料。聚类分析结果可为利用这些种质资源进行黄瓜育种组合提供依据和参考,为品种种子纯度鉴定提供可靠的技术支撑。
4 结论本研究通过66对引物对50份黄瓜核心种质材料进行筛选,得到32对稳定性好、条带清晰、多态性高的SSR引物。50份黄瓜材料的遗传相似系数在0.620~1.000之间,在遗传相似系数为0.754处,50份黄瓜材料聚为7类;在遗传相似系数为0.851处,50份黄瓜材料聚为9类。聚类分析结果表明,50份黄瓜核心种质材料中,华北型黄瓜材料和华南型黄瓜材料间相似系数低、亲缘关系远;华北型黄瓜材料间相似系数高、亲缘关系近、遗传背景窄、遗传多样性差;而华南型黄瓜材料间相似系数较低、亲缘关系较远、遗传背景宽、遗传多样性丰富。本研究结果可为黄瓜种质材料遗传结构研究及广东乃至华南地区高品质黄瓜育种组合选育提供依据和参考。
| [1] |
吴燕, 俞治州, 程春燕, 王海平, 陈劲枫, 娄群峰. 黄瓜品种(系) 遗传多样性分析及分子身份证构建[J/OL]. 分子植物育种, 2023: 1-9. https://kns.cnki.net/kcms/detail/46.1068.S.20230417.1706.034. WU Y, YU Z Z, CHENG C Y, WANG H P, CHEN J F, LOU Q F. Genetic diversity and establishment of molecular ID of cultivars (lines) in cucumber[J/OL]. Molecular Plant Breeding, 2023: 1-9. https://kns.cnki.net/kcms/detail/46.1068.S.20230417.1706.034.html. |
| [2] |
STAUB J E, CHUNG S M, FAZIO G. Conformity and genetic relatedness estimation in crop species having a narrow genetic base the case of cucumber[J]. Plant Breeding, 2005, 124(1): 44-53. DOI:10.1111/j.1439-0523.2004.01061.x |
| [3] |
王蕾, 王福全, 尹惠萍, 赵国珍, 刘芳君, 王娟. 基于SSR标记的黄瓜栽培品种遗传多样性分析及指纹图谱构建[J]. 分子植物育种, 2021, 19(8): 2668-2677. DOI:10.13271/j.mpb.019.002668 WANG L, WANG F Q, YIN H P, ZHAO G Z, LIU F J, WANG J. Genetic diversity analysis and construction of DNA fingerprint with SSR markers for cucumber cultivars[J]. Molecular Plant Breeding, 2021, 19(8): 2668-2677. DOI:10.13271/j.mpb.019.002668 |
| [4] |
史建磊, 陈先知, 黄宗安, 王燕, 王克磊, 徐坚. 华南型黄瓜主要农艺性状遗传多样性评价[J]. 核农学报, 2016, 30(10): 1914-1924. DOI:10.11869/j.issn.100-8551.2016.10.1914 SHI J L, CHEN X Z, HUANG Z A, WANG Y, WANG K L, XU J. Genetic diversity evaluation on main agronomic traits in South China ecotypic cucumber[J]. Journal of Nuclear Agricultural Sciences, 2016, 30(10): 1914-1924. DOI:10.11869/j.issn.100-8551.2016.10.1914 |
| [5] |
史建磊, 陈先知, 王克磊, 黄宗安, 朱隆静. 华南型黄瓜种质遗传多样性的SSR分析[J]. 分子植物育种, 2017, 15(6): 2440-2449. DOI:10.13271/j.mpb.015.002440 SHI J L, CHEN X Z, WANG K L, HUANG Z A, ZHU L J. Genetic diversity analysis of South China ecotypic cucumber germplasmby SSR markers[J]. Molecular Plant Breeding, 2017, 15(6): 2440-2449. DOI:10.13271/j.mpb.015.002440 |
| [6] |
HUANG S W, LI R Q, ZHANG ZH H, LI L, GU X F, FAN W, WILLIAM J L, WANG X W, XIE B Y, NI P X, REN Y Y, ZHU H M, LI J, LIN K, JIN W W, FEI ZH J, LI G C, STAUB J, KILIAN A, EDWIN A, VOSSEN V, WU Y, GUO J, HE J, JIA ZH Q, LI S G. The genome of the cucumber[J]. Nature Genetics, 2009, 41(12): 1275-1281. DOI:10.1038/ng.475 |
| [7] |
LIANG F F, HU J B, LI J W, SI SH W. Genetic relationship of a cucumber germplasm collection revealed by newly developed EST-SSR markers[J]. Journal of Genetics, 2013, 89: 28-32. DOI:10.1007/s12041-011-0005-x |
| [8] |
HU J, WANG L, LI J. Comparison of genomic SSR and EST-SSR markers for estimating genetic diversity in cucumber[J]. Biologia Plantarum, 2011, 55(3): 577-580. DOI:10.1007/s10535-011-0129-0 |
| [9] |
司旻星, 关媛, 潘俊松, 何欢乐, 蔡润. 黄瓜(Cucumis sativus L.)种质资源遗传多样性及亲缘关系分析[J]. 上海交通大学学报(农业科学版), 2007, 25(2): 130-137. DOI:10.3969/j.issn.1671-9964.2007.02.010 SI M X, GUAN Y, PAN J S, HE H L, CAI R. Analysis of genetic diversity and genetic relationship in cucumber (Cucumis sativus L.) germplasm resources[J]. Journal of Shanghai Jiaotong University (Agricultural Science), 2007, 25(2): 130-137. DOI:10.3969/j.issn.1671-9964.2007.02.010 |
| [10] |
张广平, 李锡香, 向长萍, 沈镝, 王文玲, 宋江萍. 黄瓜种质核心样本构建方法初探[J]. 园艺学报, 2006(2): 260-265. DOI:10.16420/j.issn.0513-353x.2006.02.008 ZHANG G P, LI X X, XIANG C P, SHEN D, WANG W L, SONG J P. Studies on the methods of establishing cucumber core collection based on RAPD analysis[J]. Acta Horticulturae Sinica, 2006(2): 260-265. DOI:10.16420/j.issn.0513-353x.2006.02.008 |
| [11] |
陈劲枫, 庄飞云, 逯明辉, 钱春桃, 任刚. 采用SSR和RAPD标记研究黄瓜属(葫芦科)的系统发育关系[J]. 植物分类学报, 2003(5): 427-435. CHEN J F, ZHUANG F Y, DAI M H, QIAN C T, REN G. Phylogenetic relationships in Cucumis(Cucurbitaceae)revealed by SSR and RAPD analyses[J]. Acta Phytotaxonomica Sinica, 2003(5): 427-435. |
| [12] |
林强, 李生强, 张瑞越, 周国华, 邹杰, 黎世龄, 罗筱平. 利用SSR标记对籼稻品种(系)聚类分析的比较研究[J]. 广东农业科学, 2017, 44(1): 1-7. DOI:10.16768/j.issn.1004-874X.2017.01.001 LIN Q, LI S Q, ZHANG R Y, ZHOU G H, ZOU J, LI S L, LUO X P. Comparative study on cluster analysis of 24 indica rice varieties (lines) by SSR markers[J]. Guangdong Agricultural Sciences, 2017, 44(1): 1-7. DOI:10.16768/j.issn.1004-874X.2017.01.001 |
| [13] |
刘丽华, 刘阳娜, 张明明, 李宏博, 庞斌双, 赵昌平. 我国75份小麦品种SNP和SSR指纹图谱构建与比较分析[J]. 中国农业科技导报, 2020, 22(5): 15-23. DOI:10.13304/j.nykjdb.2019.1023 LIU L H, LIU Y N, ZHANG M M, LI H B, PANG B S, ZHAO C P. Construction and comparative analysis of SNP and SSR fingerprints of 75 wheat cultivars in China[J]. Journal of Agricultural Science and Technology, 2020, 22(5): 15-23. DOI:10.13304/j.nykjdb.2019.1023 |
| [14] |
朱志成. 辽宁省玉米品种种质SSR分子多样性分析[J]. 农业与技术, 2022, 42(2): 31-33. DOI:10.19754/j.nyyjs.20220130008 ZHU Z C. SSR molecular diversity analysis of maize germplasm in Liaoning Province[J]. Agriculture and Technology, 2022, 42(2): 31-33. DOI:10.19754/j.nyyjs.20220130008 |
| [15] |
臧珊, 张云霄, 郭媛, 胡胜武. 利用SSR和SRAP标记分析油菜骨干亲本的遗传多样性[J]. 西北农林科技大学学报(自然科学版), 2019, 47(5): 7-14. DOI:10.13207/j.cnki.jnwafu.2019.05.002 ZANG S, ZHANG Y X, GUO Y, HU S W. Genetic diversity pattern of elite lines in Brassica napus L. based on SSR and SRAP markers[J]. Journal of Northwest A & F University (Natural Science Edition), 2019, 47(5): 7-14. DOI:10.13207/j.cnki.jnwafu.2019.05.002 |
| [16] |
张强强, 梁赛, 王艳, 贾利, 方凌, 张其安, 江海坤, 隋益虎, 董言香. 基于表型性状和SSR标记的57份辣椒种质遗传多样性分析[J]. 热带亚热带植物学报, 2020, 28(4): 356-366. DOI:10.11926/jtsb.4185 ZHANG Q Q, LIANG S, WANG Y, JIA L, FANG L, ZHANG Q A, JIANG H K, SUI Y H, DONG Y X. Genetic diversity analysis of 57 germplasms of Capsicum annuum based on phenotypic traits and SSR markers[J]. Journal of Tropical and Subtropical Botany, 2020, 28(4): 356-366. DOI:10.11926/jtsb.4185 |
| [17] |
傅鸿妃, 吕晓菡, 陈建瑛, 李国景. 辣椒种质表型性状与SSR分子标记的遗传多样性分析[J]. 核农学报, 2018, 32(7): 1309-1319. DOI:10.11869/j.issn.100-8551.2018.07.1309 FU H F, LYU X H, CHEN J Y, LI G J. Genetic diversity analysis of capsicum germplasm based onphenotypic traits with SSR markers[J]. Journal of Nuclear Agricultural Sciences, 2018, 32(7): 1309-1319. DOI:10.11869/j.issn.100-8551.2018.07.1309 |
| [18] |
郭守鹏, 马村, 徐佳宁, 门洪文, 杨淑军, 王文军. 基于表型性状和SSR标记的茄子种质资源亲缘关系比对分析[J]. 农业科技通讯, 2019(9): 162-167, 296. DOI:10.3969/j.issn.1000-6400.2019.09.060 GUO S P, MA C, XU J N, MEN H W, YANG S J, WANG W J. Genetic relationship analysis of eggplant germplasm resources based on phenotypic traitsand SSR markers[J]. Bulletin of Agricultural Science and Technology, 2019(9): 162-167, 296. DOI:10.3969/j.issn.1000-6400.2019.09.060 |
| [19] |
孟艳, 卢倩倩, 刘小愿, 王亚秀, 张学芬, 和禹廷, 张妮南, 高京草, 张鲁刚. 基于SSR标记对65份大白菜自交系的聚类分析[J]. 西北农业学报, 2021, 30(11): 1656-1662. DOI:10.7606/j.issn.1004-1389.2021.11.008 MENG Y, LU Q Q, LIU X Y, WANG Y X, ZHANG X F, HE Y T, ZHANG N N, GAO J C, ZHANG L G. Cluster analysis of 65 Chinese cabbage inbred lines based on SSR markers[J]. Acta Agriculture Boreali-occidentalis Sinica, 2021, 30(11): 1656-1662. DOI:10.7606/j.issn.1004-1389.2021.11.008 |
| [20] |
段丽丽, 郑永胜, 李华, 孙加梅, 王东建, 王雪梅, 仙丽娜, 王秀娟, 张晗. 46份大白菜种质资源SSR标记的遗传多样性分析[J]. 山东农业科学, 2016, 48(5): 5-8. DOI:10.14083/j.issn.1001-4942.2016.05.002 DUAN L L, ZHENG Y S, LI H, SUN J M, WANG D J, WANG X M, XIAN L N, WANG X J, ZHANG H. Genetic diversity analysis of 46 Chinese cabbage germplasm resources with SSR markers[J]. Shandong Agricultural Sciences, 2016, 48(5): 5-8. DOI:10.14083/j.issn.1001-4942.2016.05.002 |
| [21] |
颜新林, 管中荣, 温雯, 张浙峰, 王晨宇, 沈进娟, 汪启明, 韩瑞玺, 饶力群. 基于SSR标记的芥菜品种鉴定技术体系建立及应用[J]. 植物遗传资源学报, 2021, 22(3): 758-770. DOI:10.13430/j.cnki.jpgr.20201014002 YAN X L, GUAN Z R, WEN W, ZHANG Z F, WANG C Y, SHEN J J, WANG Q M, HAN R X, RAO L Q. Establishment and application of mustard variety identification system based on SSR markers (Brassica juncea L.)[J]. Journal of Plant Genetic Resources, 2021, 22(3): 758-770. DOI:10.13430/j.cnki.jpgr.20201014002 |
| [22] |
李光光, 张华, 郑岩松, 李荣华. 利用SSR标记研究菜心资源的遗传多样性分析[J]. 基因组学与应用生物学, 2018, 37(3): 1257-1264. DOI:10.13417/j.gab.037.001257 LI G G, ZHANG H, ZHENG Y S, LI R H. The genetic diversity analysis of chinese flowering cabbage resources based on SSR marker[J]. Genomics and Applied Biology, 2018, 37(3): 1257-1264. DOI:10.13417/j.gab.037.001257 |
| [23] |
崔兴华, 赵海燕, 段骅, 刘楠, 张海朋, 苗伟利, 徐烨, 李平. SSR技术鉴定黄瓜新品种津早28的种子纯度试验[J]. 农业科技通讯, 2023(4): 110-113. DOI:10.3969/j.issn.1000-6400.2023.04.032 CUI X H, ZHAO H Y, DUAN Y, LIU N, ZHANG H P, MIAO W L, XU Y, LI P. Seed purity test of a new cucumber variety Jinzao 28 by SSR technology[J]. Bulletin of Agricultural Science and Technology, 2023(4): 110-113. DOI:10.3969/j.issn.1000-6400.2023.04.032 |
| [24] |
李春, 梁根云, 蔡鹏, 李跃建, 房超, 刘独臣, 刘小俊. 利用SSR分子标记鉴定华南型黄瓜'川绿15号'杂交种子纯度[J]. 西昌学院学报(自然科学版), 2022, 36(3): 13-17. DOI:10.16104/j.issn.1673-1891.2022.03.003 LI C, LIANG G Y, CAI P, LI Y J, FANG C, LIU D C, LIU X J. Identification of hybrid seed purity on south china type cucumber'chuanlv 15' using SSR molecular markers[J]. Journal of Xichang University (Natural Science Edition), 2022, 36(3): 13-17. DOI:10.16104/j.issn.1673-1891.2022.03.003 |
| [25] |
李利斌, 牛佳玉, 王永强, 杨宗辉, 杨桂兰, 候丽霞, 刘淑梅, 曹齐卫, 孟昭娟. 分子标记技术在黄瓜农艺性状基因定位上的应用[J]. 植物生理学报, 2019, 55(1): 1-13. DOI:10.13592/j.cnki.ppj.2018.0130 LI L B, NIU J Y, WANG Y Q, YANG Z H, YANG G L, HOU L X, LIU S M, CAO Q W, MENG S J. Application of molecular markers in gene localization of cucumber (Cucumis sativus) agronomic traits[J]. Plant Physiology Journal, 2019, 55(1): 1-13. DOI:10.13592/j.cnki.ppj.2018.0130 |
| [26] |
MASASHI H, MITSUHIRO S, NAKAO K, NOBUKO F, YOSUKE Y. Development of 101 novel SSR markers and construction of an SSR-based genetic linkage map in cucumber (Cucumis sativus L.)[J]. Theoretical and Applied Genetics, 2008, 58(1): 165-175. DOI:10.1270/jsbbs.58.475 |
| [27] |
李光光, 郑岩松, 李向阳, 黄红弟, 张华. 苦瓜种质资源SSR遗传多样性分析[J]. 分子植物育种, 2016, 14(7): 1914-1922. DOI:10.13271/j.mpb.014.001914 LI G G, ZHENG Y S, LI X Y, HUANG H D, ZHANG H. Genetic diversity of bitter gourd germplasm resources revealed by SSR analysis[J]. Molecular Plant Breeding, 2016, 14(7): 1914-1922. DOI:10.13271/j.mpb.014.001914 |
| [28] |
崔竣杰, 程蛟文, 曹毅, 胡开林. 基于SSR标记和表型性状构建苦瓜核心种质的研究[J]. 中国蔬菜, 2022(2): 25-32. DOI:10.19928/j.cnki.1000-6346.2022.1002 CUI J J, CHENG J W, CAO Y, HU K L. Studies on construction of bitter gourd core collection based on SSR markers and phenotypic traits[J]. China Vegetables, 2022(2): 25-32. DOI:10.19928/j.cnki.1000-6346.2022.1002 |
| [29] |
陈庆明, 刘政国, 王基沛, 汪春玲, 陈婕英, 马连莲. 节瓜果肉颜色遗传规律及SSR标记连锁分析[J]. 分子植物育种, 2019, 17(23): 7787-7793. DOI:10.13271/j.mpb.017.007787 CHEN Q M, LIU Z G, WANG J P, WANG C L, CHEN J Y, MA L L. Genetic law of flesh color of chieh-qua and its linkage analysis of SSR markers[J]. Molecular Plant Breeding, 2019, 17(23): 7787-7793. DOI:10.13271/j.mpb.017.007787 |
| [30] |
邹剑锋. 中国南瓜种质资源遗传多样性SSR和Indel标记分析[D]. 佛山: 佛山科学技术学院, 2020. ZOU J F. Analysis of genetic diversity of Cucurbita moschata germplasm resources by SSR and Indel marker[D]. Foshan: Foshan University, 2020. |
| [31] |
李辉玲, 张建文, 李守明. 基于SSR标记的新疆大蒜及其野生蒜种遗传多样性及聚类分析[J]. 江苏农业科学, 2022, 50(18): 158-163. DOI:10.15889/j.issn.1002-1302.2022.18.024 LI H L, ZHANG J W, LI S M. Genetic diversity and cluster analysis of Xinjiang garlic and its wild species based on SSR markers[J]. Jiangsu Agricultural Sciences, 2022, 50(18): 158-163. DOI:10.15889/j.issn.1002-1302.2022.18.024 |
| [32] |
孟淑春, 刘立功, 宋晓玉, 宋顺华, 张峰. 黄瓜新品种京研冬美9号的SSR纯度鉴定[J]. 中国瓜菜, 2023, 36(6): 23-27. DOI:10.16861/j.cnki.zggc.20230510.002 MENG S C, LIU L G, SONG X Y, SONG S H, ZHANG F. SSR purity identification of cucumber variety Jingyan Dongmei No.9[J]. China Cucurbits and Vegetables, 2023, 36(6): 23-27. DOI:10.16861/j.cnki.zggc.20230510.002 |
| [33] |
陈林, 曹振强, 梁肇均, 何晓明, 王敏, 刘文睿, 彭庆务, 林毓娥. 黄瓜种质资源农艺性状多样性分析[J]. 广东农业科学, 2022, 48(6): 15-22. DOI:10.16768/j.issn.1004-874X.2021.06.003 CHEN L, CAO Z Q, LIANG Z J, HE X M, WANG M, LIU W R, PENG Q W, LIN Y E. Diversity analysis of agronomic traits of cucumber germplasm resources[J]. Guangdong Agricultural Sciences, 2022, 48(6): 15-22. DOI:10.16768/j.issn.1004-874X.2021.06.003 |
| [34] |
孟淑春, 徐秀苹, 刘立功, 宋顺华, 张峰. 京研绿翡翠黄瓜品种纯度的SSR鉴定[J]. 湖北农业科学, 2022, 61(1): 98-101. DOI:10.14088/j.cnki.issn0439-8114.2022.01.01 MENG S C, XU X P, LIU L G, SONG S H, ZHANG F. SSR purity identification of cucumber variety Jingyan Lvfeicui[J]. Hubei Agricultural Sciences, 2022, 61(1): 98-101. DOI:10.14088/j.cnki.issn0439-8114.2022.01.01 |
| [35] |
杨瑞环, 陈德富, 闵微, 陈喜文. 利用SSR标记分析黄瓜种质资源的遗传多样性[J]. 南开大学学报(自然科学版), 2012, 45(2): 17-21. YANG R H, CHEN D F, MIN W, CHEN X W. SSR analysis of genetic diversity of cucumber germplasm[J]. Acta Scientiarum Naturalium Universitatis Nankaiensis, 2012, 45(2): 17-21. |
| [36] |
LIU C X, ZHAO Y Z, BAI Y, LU X M, SONG W B, QIN L P, CAI Y L. Molecular mapping and candidate gene analysis of the semi-dominant gene Vestigial glume1 in maize[J]. The Crop Journal, 2019, 7(5): 667-676. DOI:10.1016/j.cj.2019.04.001 |
| [37] |
PANDEY S, ANSARI W A, PANDEY M, SINGH B. Genetic diversity of cucumber estimated by morpho-physiological and EST-SSR arkers[J]. Physiology and Molecular Biology of Plants, 2017, 24(1): 135-146. DOI:10.1007/s12298-017-0489-9 |
| [38] |
DANIN Y, TZORI G, KARCHI Z, LAVI U, CREGAN P B, KATZIR N. Length polymorphism and homologies of microsatelutes in several Cucurbitaceae species[J]. Theoretical and Applied Genetics, 1996(93): 1282-1290. DOI:10.1007/BF00223461 |
| [39] |
沈镝, 李锡香, 方智远, 戚春章, 宋江萍, 王海平, 邱杨. 西双版纳黄瓜种质资源形态鉴定和亲缘关系的初步研究[J]. 中国蔬菜, 2010(2): 21-27. DOI:10.19928/j.cnki.1000-6346.2010.02.004 SHEN D, LI X X, FANG Z Y, QI C Z, SONG J P, WANG H P, QIU Y. Preliminary study on morphological identification and phylogenetic relationship of Cucumis sativus L. var. xishuangbannanesis germplasm[J]. China Vegetables, 2010(2): 21-27. DOI:10.19928/j.cnki.1000-6346.2010.02.004 |
| [40] |
吕婧, 时秋香, 任毅, 张忠华, 黄三文. 黄瓜23对高多态性SSR标记的筛选与评价[J]. 园艺学报, 2011, 38(11): 2140-2148. DOI:10.16420/j.issn.0513-353x.2011.11.015 LYU J, SHI Q X, REN Y, ZHANG Z H, HUANG S W. Screening and evaluation of 23 high polymorphism SSR markers in cucumber[J]. Acta Horticulturae Sinica, 2011, 38(11): 2140-2148. DOI:10.16420/j.issn.0513-353x.2011.11.015 |
| [41] |
李锡香, 朱德蔚. 黄瓜种质资源描述规范和数据标准[M]. 北京: 中国农业大学出版社, 2005. LI X X, ZHU D W. Descriptors and data standard for cucumber (Cucumis sativus L.)[M]. Beijng: China Agricultural University Press, 2005. |
| [42] |
姚春鹏, 张晓爱, 吴廷全, 邓洁, 金庆敏, 李海达, 王瑞娟, 张长远. 用于长绿2号苦瓜种子纯度鉴定的SSR分子标记的开发[J]. 分子植物育种, 2019, 17(11): 3660-3664. DOI:10.13271/j.mpb.017.003660 YAO C P, ZHANG X A, WU T Q, DENG J, JIN Q M, LI H D, WANG R J, ZHANG C Y. Development of SSR molecular markers for seed purity identification of bitter gourd hybrid changlü No.2[J]. Molecular Plant Breeding, 2019, 17(11): 3660-3664. DOI:10.13271/j.mpb.017.003660 |
| [43] |
REIS N, TZUR I G, KATZIR N, POLEG Y D. Development and characterization of microsatellite markers in Cucumis[J]. Theoretical and Applied Genetics, 2001, 102: 61-72. DOI:10.1007/s001220051618 |
| [44] |
李翔, 张颖, 许俊强, 宋婷, 李文璐, 董玉梅, 张应华. 基于EST-SSR标记的云南黄瓜种质资源遗传多样性分析[J]. 分子植物育种, 2016, 14(5): 1179-1188. DOI:10.13271/j.mpb.014.001179 LI X, ZHANG Y, XU J Q, SONG T, LI W L, DONG Y M, ZHANG Y H. Analysis on genetic diversity of local cucumber germplasm resources in Yunnan based on EST-SSR markers[J]. Molecular Plant Breeding, 2016, 14(5): 1179-1188. DOI:10.13271/j.mpb.014.001179 |
| [45] |
苗晗, 张圣平, 顾兴芳, 王烨, 陈磊. 中国黄瓜主栽品种SSR遗传多样性分析及指纹图谱构建[J]. 植物遗传资源学报, 2014, 15(2): 333-341. DOI:10.13430/j.cnki.jpgr.2014.02.016 MIAO H, ZHANG S P, GU X F, WANG Y, CHEN L. Genetic diversity analysis and fingerprinting construction of Chinese cucumber main cultivars by simple sequence repeat polymorphisms[J]. Journal of Plant Genetic Resources, 2014, 15(2): 333-341. DOI:10.13430/j.cnki.jpgr.2014.02.016 |
| [46] |
杨福强, 李琳, 李明远, 刘丽英, 任华中. 中国刺瘤型黄瓜种质遗传多样性和亲缘关系的AFLP分析[J]. 西北农业学报, 2009, 18(6): 205-211. YANG F Q, LI L, LI M Y, LIU L Y, REN H Z. Genetic diversity and phylogenetic relationship of Chinese warty cucumber germplasm based on AFLPs[J]. Acta Agriculturae Boreali-Occidentalis Sinica, 2009, 18(6): 205-211. |
(责任编辑 马春敏)
 2023, Vol. 50
2023, Vol. 50