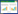文章信息
基金项目
- 广州市科技计划项目(2023B03J1322);国家重点研发计划项目(2019YFD1001003)
作者简介
- 周荣,硕士,三级教授,硕士生导师,佛山科学技术学院风景学院学科和专业带头人,主要从事园艺园林植物开发利用研究。主持广东省科技厅、省农业农村厅、佛山市等省市厅科研课题10余项,发表论文40多篇,主编专著4部、参编1部。获广东省科技进步三等奖、广东省农业技术推广奖二等奖和三等奖、佛山市科技进步二等奖、中国发明协会创业银奖等奖项10余项;授权国家发明专利1件。曾获广东省“三八红旗手”、广东省三下乡优秀教师、广东省创新创业优秀指导教师、佛山市“三八”红旗手、佛山市科技标兵等称号;指导大学生“挑战杯”创业计划竞赛获得国银和省银奖、“互联网+”大学生创新创业大赛获得省金和省铜奖;主持教育部协同育人、广东省质量工程项目各1项、校级教研课题3项;主编和参编教材2部;指导学生国家和省级大学生创业项目5项;兼任中国花卉协会兰花分会副秘书长、广东省兰花协会副会长、《佛山科学技术学院学报》编委。周荣(1966—),女,硕士,教授,研究方向为园艺园林植物开发和利用,E-mail:602597936@qq.com; 杨凤玺,博士,研究员,硕士生导师,主要从事兰花种质资源创新、重要性状功能基因挖掘、分子育种及新品种配套栽培技术研发。广东省农业科学院环境园艺研究所副所长兼兰花研究室主任、广东省园林花卉种质创新综合利用重点实验室主任,兼任中国花卉协会兰花分会副秘书长、广东省园艺学会副理事长、广东省现代农业产业技术体系花卉团队岗位专家、广东省农村科技特派员、广州市乡村振兴“百团千人科技下乡”专家等。主持国家自然科学基金面上项目、国家重点研发计划项目(子课题负责人)、广东省自然科学基金、广东省科技基础条件建设重点项目、广州市重点研发计划等30余个项目。获神农中华农业科技优秀团队奖、云南省自然科学一等奖、湖南省科技进步二等奖、广东省农业技术推广奖二等奖和汕头市科学技术二等奖等多项奖励。发表论文50余篇(SCI 30篇),参编专著2部,获授权国家发明专利8件(排名第一),培育特色兰花新品种9个.
通讯作者
- 杨凤玺(1985—),女,博士,研究员,研究方向为兰科植物遗传育种,E-mail:yangfengxi@gdaas.cn.
文章历史
- 收稿日期:2023-07-09
2. 广东省农业科学院环境园艺研究所/广东省园林花卉种质创新综合利用重点实验室,广东 广州 510640
2. Environmental Horticulture Research Institute, Guangdong Academy of Agricultural Sciences/Guangdong Key Laboratory of Ornamental Plant Germplasm Innovation and Utilization, Guangzhou 510640, China
【研究意义】墨兰(Cymbidium sinense)是我国国兰的典型代表之一,在福建、广东、台湾广泛分布。墨兰叶型独特,花姿优美,花香馥郁,花期长(9月至翌年3月),具有很高的观赏价值和经济价值[1–3],产品远销韩国、日本等地。开花性状是墨兰最重要的育种目标,而APETALA1(AP1)基因在植物成花转变和花器官发育过程中具有非常关键的作用[4]。开展墨兰AP1基因的克隆和表达分析研究,对探究墨兰开花过程中的分子调控机制具有重要意义。【前人研究进展】MADS-box基因家族是一类在植物中广泛存在的转录因子家族,可与其他转录因子和调控因子相互作用,共同在植物花器官分化、果实发育和开花调控等方面起作用。MADSbox基因家族通常分为两个亚家族(Type Ⅰ和Type Ⅱ),其中Type Ⅱ亚家族基因编码的蛋白质均有1个特征性结构域K,能够折叠形成3个α螺旋结构,介导MADS-box蛋白之间形成二聚体,通过蛋白复合体共同调控下游靶基因。以Type Ⅱ类MADS-box基因为核心构成的花发育ABCDE模型决定植物花器官的发育[5-6]。A类基因主要影响花朵的外轮花器官发育,同时与E类基因共同参与萼片的发育,并与B类基因和E类基因共同调控花瓣的形成和特化。AP1属于A类基因,其突变或过表达都会对植物的开花时间和花器官产生显著影响[7-8]。在拟南芥中,过表达AP1可使拟南芥提前1周左右开花[9]。AP1同时可以抑制最外轮花器官萼片叶腋处形成更多花原基。另外,AP1可以分别通过抑制细胞分裂素合成基因和激活细胞分裂素降解酶,减少AP1基因表达区域的细胞分裂素含量,从而维持正常花分生组织的有限生长。目前,建兰(C. ensifolium)[10]、石斛(Dendrobium Chao Praya Smile)[11]、萼脊兰(Sedirea japonica)[8]、牡丹(Paeonia suff ruticosa)[12]、荔枝(Litchi chinensis)[13]、大豆(Glycine max)[14]等多种植物的AP1基因已被成功克隆,研究发现,它们在花器官的形成和花期调控中起着非常重要的作用。【本研究切入点】目前关于墨兰AP1基因的研究报道甚少,该基因在墨兰上的功能表现也尚未清楚。因此,本研究以墨兰为研究对象,克隆并鉴定到1个墨兰AP1基因(命名为CsAP1-A),并进行基因表达模式分析[15]。【拟解决的关键问题】本研究克隆墨兰CsAP1-A基因,通过生物信息学分析其基因结构、蛋白结构域和进化关系,利用RTqPCR方法分别检测CsAP1-A在墨兰不同器官、不同花发育阶段和不同花组织部位的表达情况,通过转录组测序分析CsAP1-A在5个不同花型墨兰品种花组织部位的表达情况,并采用蛋白互作预测软件分析CsAP1-A与其他蛋白的互作关系,为进一步研究CsAP1-A在墨兰花发育过程的功能奠定基础。
1 材料与方法 1.1 试验材料基因克隆、表达分析供试的墨兰品种为‘小香’,栽培于广东省农业科学院环境园艺研究所国家墨兰种质资源圃内。挑选3株长势良好的墨兰,采集其花芽、成熟花器官及根、茎、叶和果各3 g左右,用液氮速冻,-80 ℃保存备用。
不同花型转录组测序分析供试的5个墨兰品种为‘瑞金’(WT普通花型)、‘安康梅’(花瓣蕊柱化的梅瓣花型,Gynostegium-like petal variety,GPV)、‘大圣’(重瓣花型,Multi perianth variety,MPV)、‘金凤蝶’(花瓣唇瓣化花型,Labellum -like petal variety,LaPV)和‘六瓣花’(唇瓣萼片化花型,Null-labellum variety,NLV),均来源于广东省农业科学院环境园艺研究所国家墨兰种质资源圃。采集的花朵立即用液氮速冻处理。
DH5α化学感受态细胞、荧光定量试剂盒(Taq Pro Universal SYBR qPCR Master Mix)、高保真酶试剂盒(2×Phanta Flash Master Mix)、产物纯化试剂盒(FastPure Gel DNA Extraction Mini Kit)、克隆载体试剂盒(5 min TA/Blunt Cloning Kit)和RNA提取试剂盒(FastPure Universal Plant Total RNA Isolation Kit),均购自南京诺唯赞生物科技股份有限公司。
1.2 总RNA提取及反转录用多糖多酚植物RNA提取试剂盒提取墨兰花芽的RNA,操作过程严格按照试剂盒说明书进行。使用蛋白核酸分析仪检测RNA浓度,使用1% 琼脂糖凝胶电泳检测RNA质量,将检测合格的RNA进行反转录反应得到cDNA。
1.3 目的基因克隆以前期基因组测序得到的CsAP1-A CDS为参考序列,使用NCBI设计基因克隆的PCR引物:CsAP1-A-F 5'ATGGGAAGAGGGAGGGTT3'、CsAP1-A-R 5'TTATCCATTCATATGAGTGAGC3'。以墨兰花芽cDNA为模板,按照高保真酶试剂盒说明书操作进行CsAP1-A基因克隆,PCR体系为50 μL,反应程序:98 ℃预变性30 s;98 ℃预变性10 s、58 ℃退火5 s、72 ℃延伸5 s,34个循环;72 ℃延伸1 min。扩增产物回收纯化后与TA克隆载体连接,转化到DH5α化学感受态细胞中培养,次日挑选阳性菌液送至生工生物工程(上海)股份有限公司测序,得到CsAP1-A基因完整序列。
1.4 CsAP1-A生物信息学分析使用Expasy-ProtParam(https://www.expasy.org/)分析CsAP1-A蛋白的分子量大小、亲水性等理化性质;使用NCBI(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析CsAP1-A的保守结构域;使用DNAMAN软件将CsAP1-A与6个同源蛋白进行多序列比对;使用Mega6.0软件的邻接法构建CsAP1-A系统进化树。
1.5 CsAP1-A基因表达分析使用RT-qPCR方法对CsAP1-A在墨兰不同器官(根、茎、叶、花、果)、不同花发育阶段(花芽分化初期S1、花芽分化发育期S2、花梗伸长期S3、排铃期S4和开花期S5)和不同花组织部位(萼片、花瓣、唇瓣、合蕊柱)的表达进行分析。以CsActin作为内参基因(表 1),PCR反应程序为:95 ℃ 2 min;95 ℃ 15 s、60 ℃ 30 s、72 ℃ 30 s,40个循环。基因表达试验重复3次,使用2-ΔΔCt公式计算CsAP1-A相对表达量[16]。
1.6 不同花型转录组测序分析
采集5个不同花型(WT、GPV、MPV、LaPV、NLV)墨兰品种(‘瑞金’‘安康梅’‘大圣’‘金凤蝶’‘六瓣花’)的花朵,立即液氮速冻处理,送至生工生物工程(上海)股份有限公司进行转录组测序。对CsAP1-A的FPKM值进行log2转换,并利用R语言中的Pheatmap工具包生成热图。
1.7 CsAP1-A蛋白互作关系通过STRING数据库(https://cn.string-db.org/)预测可能与CsAP1-A发生互作的蛋白质,并以水稻作为参考,构建CsAP1-A与其他蛋白的互作关系图。
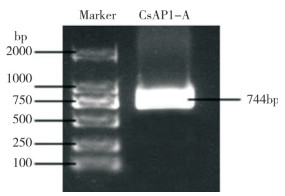
2 结果与分析 2.1 CsAP1-A基因全长克隆以墨兰花芽为材料提取RNA,并将RNA反转录为cDNA。以cDNA为模板,以墨兰基因组参考序列设计引物,扩增得到约750 bp的单一片段(图 1)。将回收的DNA片段与TA克隆载体进行连接并转化感受态细胞,最后通过测序得到CsAP1-A基因序列,全长744 bp,编码248个氨基酸(图 2)。
|
| 图 1 CsAP1-A基因克隆PCR产物 Fig. 1 PCR product of CsAP1-A gene cloning |

|
| 图 2 CsAP1-A基因的核苷酸序列和推导的氨基酸序列 Fig. 2 Nucleotide acid sequence of CsAP1-A gene and deduced amino sequence |
2.2 CsAP1-A生物信息学分析
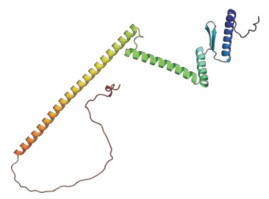
2.2.1 CsAP1-A蛋白理化性质分析 根据Expasy-ProtParam预测分析结果,CsAP1-A蛋白的分子式为C1242H2038N370O376S9,蛋白分子量为28.4 kD,总原子数为4 035,脂肪系数为83.40,平均亲水性为-0.762,说明CsAP1-A是一个亲水蛋白。选择模型一致性高达98.38% 的同源蛋白AOA1P8SCC5.1A作为模板,通过SWISS-MODEL在线软件构建CsAP1-A蛋白的三级结构,发现该结构包含4个保守的α-螺旋和2个β折叠结构(图 3)。
|
| 图 3 CsAP1-A蛋白三级结构预测 Fig. 3 Tertiary structure prediction of CsAP1-A protein |
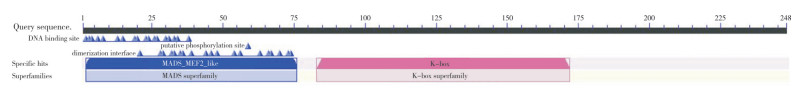
2.2.2 CsAP1-A蛋白保守域分析与同源序列比对 通过NCBI数据库分析蛋白序列可知,CsAP1-A拥有保守的MADS-box结构域和K-box结构域(图 4),属于MADS-box转录因子家族成员。此外,通过NCBI数据库得到多个与CsAP1-A同源性很高的蛋白序列,其中,CsAP1-A与春兰(C. goeringii)AP1/FUL(APY18447.1)和蕙兰(C. faberi)MADS1(AGE15496.1)的同源性均高达98.38%,与金钗石斛(D. nobile)MADSbox protein 1(ABQ08573.1)、蝴蝶兰(Phalaenopsis amabilis)AP1-related protein(AAZ76263.1)、华山姜(Alpinia oblongifolia)APETALA1-like protein(ABS83558.1)、银合欢(Nelumbo nucifera)APETALA1(AGY54940.1)的同源性分别为85.83%、82.19%、66.27% 和60.32%(图 5)。
|
| 图 4 CsAP1-A蛋白保守结构域分析 Fig. 4 Analysis of conserved domains of CsAP1-A protein |

|
| 图 5 CsAP1-A蛋白同源序列比对 Fig. 5 Homologous sequence alignment of CsAP1-A protein |
2.2.3 CsAP1-A氨基酸序列的系统进化 为进一步了解CsAP1-A蛋白的系统进化情况,从NCBI数据库中挑选20条与CsAP1-A氨基酸序列具有一定同源性的氨基酸序列,利用MEGA6.0软件的NJ法构建进化树。结果(图 6)表明,CsAP1-A的进化树可以分为单子叶植物和双子叶植物两类[17],其中在单子叶植物中,CsAP1-A与其他兰科植物亲缘关系较近,并聚类一起。

|
| 图 6 CsAP1-A蛋白NJ系统进化树 Fig. 6 Neighbor-joining phylogenetic tree of CsAP1-A protein |
2.3 CsAP1-A基因时空表达分析
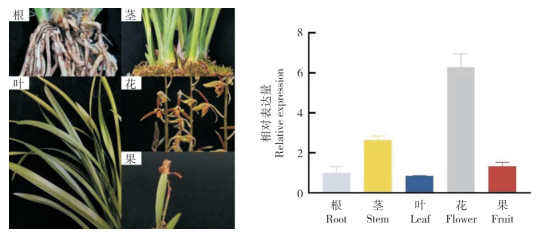
为研究CsAP1-A的表达模式,用RT-qPCR方法分别检测其在墨兰根、茎、叶、花、果不同器官,不同花发育阶段,以及萼片、花瓣、唇瓣、合蕊柱不同花朵组织部位的表达情况。结果(图 7)表明,CsAP1-A在墨兰不同器官中均有表达,在花中表达量最高,其次分别为茎、果、根和叶。
|
| 图 7 CsAP1-A在墨兰不同器官中的相对表达量 Fig. 7 Relative expression of CsAP1-A in different organs of Cymbidium sinense |
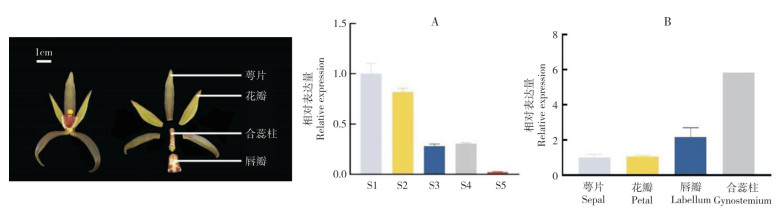
在墨兰花芽分化发育的不同阶段,CsAP1-A集中在花发育早期阶段表达,在S1阶段的表达量最高,之后逐渐下降,在S3阶段的表达量降至60%,在S5阶段的表达量降至最低。此外,对墨兰不同花组织部位的表达分析发现,CsAP1-A在合蕊柱中的表达量最高,其次分别是唇瓣、花瓣和萼片(图 8)。
|
| A:不同花发育阶段; B:不同花组织部位 A: At different stages of flower development; B: In different floral parts 图 8 CsAP1-A在墨兰花中的相对表达量 Fig. 8 Relative expression of CsAP1-A in flower of Cymbidium sinense |
2.4 不同花型墨兰CsAP1-A表达模式分析
分析墨兰不同花型转录组数据集,用热图形式显示不同花型CsAP1-A基因的表达情况。结果(图 9)表明,CsAP1-A在WT、MPV、LAPV和NLV 4种花型[18]中均以合蕊柱中的表达量最高、萼片中的表达量最低;在合蕊柱异常发育的MPV中,CsAP1-A在合蕊柱的表达量显著提高,而在GPV中的整体表达量最高,且表达区域从合蕊柱扩展到花瓣。

|
| WT:普通花型,GPV:花瓣蕊柱化的梅瓣花型,MPV:重瓣花型,LaPV:花瓣唇瓣化花型,NLV:唇瓣萼片化花型;1~4:分别代表萼片、花瓣、唇瓣和合蕊柱 WT: Common type variety; GPV: Gynostegium-like petal variety; MPV: Multi perianth variety; LaPV: Labellum-like petal variety; NLV: Null-labellum variety; 1-4: Sepal, petal, labellum and gynostemium, respectively 图 9 不同花型墨兰CsAP1-A表达模式分析 Fig. 9 Analysis of expression patterns of CsAP1-A in different floral types of Cymbidium sinense |
2.5 CsAP1-A蛋白互作预测分析
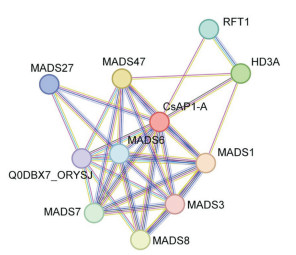
使用String在线网站预测CsAP1-A蛋白与其他蛋白之间的互作可能性,并以水稻作为参考,结果(图 10)表明,CsAP1-A可能与MADS1(A类基因)、MADS6(D类基因)、MADS47(B类基因)、MADS8(C类基因)等10个蛋白存在互作关系。MADS1与MADS47的综合得分较高,推测CsAP1-A与它们互作的可能性更大。而在水稻中,OsMADS1和OsMADS6蛋白均属于MADSbox转录因子家族成员,且这些蛋白在花器官发育和花期调控方面发挥重要作用[19]。因此,推测CsAP1-A蛋白参与墨兰花的发育。
|
| 图 10 CsAP1-A蛋白互作关系预测 Fig. 10 Prediction of CsAP1-A protein interactions |
3 讨论
MADS-box转录因子在调控植物花的形态和发育过程方面扮演着重要角色,而AP1基因是MADS-box转录因子家族的重要成员,被归类为拟南芥ABCDE开花模型中的A类基因,在许多植物中均被证实能够发挥调控植物开花、调控花器官发育的作用[20]。五峰玉兰MawuAP1基因可以使拟南芥提前开花,并在花序顶端产生顶生花[21]。过表达麻风树JcAP1基因能够显著缩短拟南芥的生长周期并导致提前开花[22]。过表达月季RcAP1也可以使拟南芥提前开花[23]。
本研究从墨兰花芽中克隆出CsAP1-A基因,该基因编码区为744 bp,编码248个氨基酸,含有MADS-box和K-box结构域,表明CsAP1-A是典型MADS-box转录因子。通过与其相似度较高的蛋白进行多序列比对,发现它们的保守结构域相同。系统进化树分析表明,CsAP1-A蛋白与春兰AP1/FUL3蛋白聚为一支,说明二者亲缘关系最近。
大多数A类基因在生殖器官的表达量均高于营养器官[24]。本研究比较了CsAP1-A基因在墨兰根、茎、叶、花、果5个器官的相对表达量,发现CsAP1-A在花中的表达量最高,在其他器官的表达量较低,说明CsAP1-A与花的发育和形成有关。在墨兰花芽分化的5个时期中,CsAP1-A在花芽分化初期(S1)的表达量最高,然后逐渐下降,在开花期(S5)的表达量降到最低,这与大部分的A类基因在不同花发育时期表达规律相似,如山茶的CjAPL1[25]、麻风树的JcAP1[22]和洋桔梗的EgAP1[26],因为S1期是花器官形成的起始阶段和关键阶段,AP1作为一个转录因子,可以在此阶段启动信号来激活其他基因促进花的形成,而随着花芽的不断发育,其他相互作用的调控因子和信号通路逐渐介入,AP1的表达量就会逐渐降低。在墨兰花朵的不同组织部位中,CsAP1-A在合蕊柱的表达量最高,其次是唇瓣,推测CsAP1-A主要参与墨兰合蕊柱的发育。对墨兰不同花型进行转录组测序分析,发现CsAP1-A在WT、MPV、LaPV和NLV 4种花型的合蕊柱中表达量均最高,这与RT-qPCR检测WT花型中CsAP1-A的表达结果相一致。AP1作为A类基因主要在第一、二轮花器官中表达,诱导花瓣和萼片的发育[7];但在兰科植物中,AP1不在第一、二轮花器官表达,而是在内轮的合蕊柱中表达[27]。
根据蛋白互作关系的预测结果可知,CsAP1-A大多与MADS-box蛋白家族存在相互作用,而这些蛋白均与花器官发育和花期相关,因此预测CsAP1-A蛋白在墨兰花器官发育过程中起重要作用,这为进一步研究墨兰花器官发育分子调控机制提供理论依据。
4 结论本研究在墨兰花芽中克隆得到CsAP1-A基因,该基因编码248个氨基酸,含有MADS-box转录因子家族的MADS-box和K-box结构域。墨兰CsAP1-A与春兰和蕙兰的亲缘关系最近,且可能与MADS1、MADS6、MADS47、MADS8等蛋白具有相互作用。CsAP1-A在墨兰花中的相对表达量显著高于其他器官,且在花芽分化初期的表达量最高,并随着花朵发育而逐渐下降;在墨兰花朵不同组织部位中,CsAP1-A在合蕊柱的表达量最高,其次是唇瓣、花瓣和萼片。通过对不同花型的墨兰品种转录组测序发现,CsAP1-A在WT、MPV、LaPV和NLV 4种花型的合蕊柱中表达量均最高。综上所述,CsAP1-A可能在墨兰花发育和合蕊柱形成中具有重要作用。
| [1] |
黄玮婷, 吴博文, 方中明. 墨兰FT同源基因的时空表达及功能分析[J]. 安徽农业大学学报, 2017, 44(1): 135-141. DOI:10.13610/j.cnki.1672-352x.20170208.011 HUANG W T, WU B W, FANG Z M. Temporal and spatial expression and functional analysis of FT homologous genes in Cymbidium sinense[J]. Journal of Anhui Agricultural University, 2017, 44(1): 135-141. DOI:10.13610/j.cnki.1672-352x.20170208.011 |
| [2] |
冯垒, 王义琴, 韩欣妤, 师凯辉, 黄鑫, 王冬梅, 孙博, 何玲, 朱永平, 杨生超, 张广辉, 和凤美. 墨兰PI基因过表达载体构建及功能初步分析[J]. 分子植物育种, 2023, 21(9): 2941-2947. DOI:10.13271/j.mpb.021.002941 FENG L, WANG Y Q, HAN X Y, SHI K H, HUANG X, WANG D M, SUN B, HE L, ZHU Y P, YANG S C, ZHANG G H, HE F M. Construction of overexpression vector of PI gene in Cymbidium sinense and preliminary analysis of its function[J]. Molecular Plant Breeding, 2023, 21(9): 2941-2947. DOI:10.13271/j.mpb.021.002941 |
| [3] |
邓乾坤, 王斌, 史学伟, 朱根发, 杨凤玺, 高洁. 墨兰精油提取工艺优化及成分分析[J]. 广东农业科学, 2021, 48(4): 131-139. DOI:10.16768/j.issn.1004-874X.2021.04.018 DENG Q K, WANG B, SHI X W, ZHU G F, YANG F X, GAO J. Optimization of extraction process and composition analysis of essential oil from Cymbidium sinense[J]. Guangdong Agricultural Sciences, 2021, 48(4): 131-139. DOI:10.16768/j.issn.1004-874X.2021.04.018 |
| [4] |
TEO Z W N, ZHOU W, SHEN L S. Dissecting the function of MADS-box transcription factors in orchid reproductive development[J]. Frontiers in Plant Science, 2019, 10: 1474. DOI:10.3389/fpls.2019.01474 |
| [5] |
王育选, 张梦超, 常丽君, 张艾丽, 张莉, 郭世华. 谷子MADS-box基因家族的鉴定和表达分析[J]. 生物信息学, 2023, 21(1): 51-59. DOI:10.12113/202106006 WANG Y X, ZHANG M C, CHANG L J, ZHANG A Y, ZHANG L, GUO S H. Genome-wide identification and expression analysis of MADSbox genes in foxtail millet[J]. Chinese Journal of Bioinformatics, 2023, 21(1): 51-59. DOI:10.12113/202106006 |
| [6] |
BECKER A, THEIBEN G. The major clades of MADS-box genes and their role in the development and evolution of flowering plants[J]. Molecular Phylogenetics and Evolution, 2003, 29: 464-489. DOI:10.1016/s1055-7903(3)002070 |
| [7] |
THEIßEN G, MELZER R, RUMPLER F. MADS-domain transcription factors and the floral quartet model of flower development: linking plant development and evolution[J]. Development, 2016, 143(18): 3259-3271. DOI:10.1242/dev.134080 |
| [8] |
张国付. 萼脊兰AP1(APETALA1)基因的序列分析及表达特性研究[D]. 郑州: 河南农业大学, 2013. ZHANG G F. Sequence analysis and expression characterization of AP1 (APETALA1) gene from Sedirea japonica[D]. Zhengzhou: Henan Agricultural University, 2013. |
| [9] |
安利忻, 刘荣维, 陈章良, 李毅. 花分生组织决定基因AP1转化矮牵牛的研究[J]. 植物学报, 2001(1): 63-66. AN L X, LIU R W, CHEN Z L, LI Y. Studies on Petunia hybrida transformed with Flower-meristem-identity gene AP1[J]. Acta Botanica Sinica, 2001(1): 63-66. |
| [10] |
吴菁华, 吴少华, 杨超, 张志忠. 建兰AP1基因的克隆、表达及其与MADS-box转录因子相互作用的分析[J]. 园艺学报, 2013, 40(10): 1935-1942. DOI:10.16420/j.issn.0513-353x.2013.10.033 WU J H, WU S H, YANG C, ZHANG Z Za. Cloning and expression analysis on AP1 homologous gene from Cymbidium ensifolium and interaction analysis between AP1 and MADS-box transcription factors[J]. Acta Horticulturae Sinica, 2013, 40(10): 1935-1942. DOI:10.16420/j.issn.0513-353x.2013.10.033 |
| [11] |
SAWETTALAKE N, BUNNAG S, WANG Y W, SHEN L S, YU H. DOAP1 promotes flowering in the orchid Dendrobium Chao Praya Smile[J]. Frontiers in Plant Science, 2017, 8: 400. DOI:10.3389/fpls.2017.00400 |
| [12] |
张璐, 李玉娥, 管世铭, 刘春英, 盖树棚, 张玉喜. 牡丹PsAP1基因的表达分析及功能鉴定[J]. 华北农学报, 2015, 30(1): 84-89. DOI:10.7668/hbnxb.2015.01.014 ZHANG L, LI Y E, GUAN S M, LIU C Y, GAI S P, ZHANG Y X. Expression analysis and functional identification of PsAP1 in tree peony[J]. Acta Agriculturae Boreali-Sinica, 2015, 30(1): 84-89. DOI:10.7668/hbnxb.2015.01.014 |
| [13] |
丁峰, 彭宏祥, 罗聪, 李冬波, 朱建华, 秦献泉, 何新华, 曹辉庆. 荔枝APETALA1(AP1) 同源基因cDNA全长克隆及其表达研究[J]. 园艺学报, 2011, 38(12): 2373-2380. DOI:10.16420/j.issn.0513-353x.2011.12.021 DING F, PENG H X, LUO C, LI D B, ZHU J H, QIN X Q, HE X H, CAO H Q. Cloning and expression analysis of the APETALA1(AP1) homologous gene cDNA from Litchi chinensis[J]. Acta Horticulturae Sinica, 2011, 38(12): 2373-2380. DOI:10.16420/j.issn.0513-353x.2011.12.021 |
| [14] |
CHEN L Y, NAN H Y, KONG L P, YUE L, YANG H, ZHAO Q S, FANG C, LI H Y, CHENG Q, LU S J, KONG F J, LIU B H, DONG L D. Soybean AP1 homologs control flowering time and plant height[J]. Journal of Integrative Plant Biology, 2020, 62(12): 1868-1879. DOI:10.1111/jipb.12988 |
| [15] |
邹冬梅, 刘月学, 张志宏, 李贺, 马跃, 代红艳. 草莓AP1同源基因的克隆、表达及启动子分析[J]. 中国农业科学, 2012, 45(10): 1972-1981. DOI:10.3864/j.issn.0578-1752.2012.10.010 ZOU D M, LIU Y X, ZHANG Z H, LI H, MA Y, DAI H Y. Cloning, expression and promoter analysis of AP1 homologous gene from strawberry (Fragaria×Ananassa)[J]. Scientia Agricultura Sinica, 2012, 45(10): 1972-1981. DOI:10.3864/j.issn.0578-1752.2012.10.010 |
| [16] |
谢炳春, 黄俊霖, 温松森, 李涛, 李颖, 徐小万, 徐晓美, 吴智明, 衡周. 中国辣椒BCAT基因家族鉴定、表达分析及克隆[J]. 广东农业科学, 2022, 49(12): 34-43. DOI:10.16768/j.issn.1004-874X.2022.12.004 XIE B C, HUANG J L, WEN S S, LI T, LI Y, XU X W, XU X M, WU Z M, HENG Z. Identification, expression analysis and cloning of Capsicum chinense BCAT gene family[J]. Guangdong Agricultural Sciences, 2022, 49(12): 34-43. DOI:10.16768/j.issn.1004-874X.2022.12.004 |
| [17] |
郑雪文, 欧阳嫣惟, 潘晓璐, 张红娜, 杨转英. 菠萝AcMADS41基因的克隆及其在花发育中的表达分析[J]. 广东农业科学, 2022, 49(1): 42-50. DOI:10.16768/j.issn.1004-874X.2022.01.005 ZHENG X W, OUYANG Y W, PAN X L, ZHANG H N, YANG Z Y. Analysis on cloning of AcMADS41 gene and its expression during flower development of pineapple[J]. Guangdong Agricultural Sciences, 2022, 49(1): 42-50. DOI:10.16768/j.issn.1004-874X.2022.01.005 |
| [18] |
YANG F X, GAO J, WEI Y L, REN R, ZHANG G Q, LU C Q, JIN J P, AI Y, WANG Y Q, CHEN L J, AHMAD S, ZHANG D Y, SUN W H, TSAI W C, LIU Z J, ZHU G F. The genome of Cymbidium sinense revealed the evolution of orchid traits[J]. Plant Biotechnology Journal, 2021, 19(12): 2501-2516. DOI:10.1111/pbi.13676 |
| [19] |
HU Y, LIANG W Q, YIN C S, YANG X L, PING B Z, LI A X, JIA R, CHEN M J, LUO Z J, CAI Q, ZHAO X X, ZHANG D B. Interactions of OsMADS1 with floral homeotic genes in rice flower development[J]. Molecular Plant, 2015, 8(9): 1366-1384. DOI:10.1016/j.molp.2015.04.009 |
| [20] |
曹智, 王艳君, 邹文桐, 丁可武, 王昌伟, 林智敏. MADS-box基因在蝴蝶兰花药中的表达谱分析及基因克隆[J]. 泉州师范学院学报, 2022, 40(2): 17-23. DOI:10.16125/j.cnki.1009-8224.2022.02.009 CAO Z, WANG Y J, ZOU W T, DING K W, WANG C W, LIN Z M. An expression profile analysis and gene cloning of MADS-box gene in anther of Phalaenopsis[J]. Journal of Quanzhou Normal University, 2022, 40(2): 17-23. DOI:10.16125/j.cnki.1009-8224.2022.02.009 |
| [21] |
LI C J, CHEN L Y, FAN X N, QI W J, MA J, TIAN T, ZHOU T, MA L Y, CHEN F J. MawuAP1 promotes flowering and fruit development in the basal angiosperm Magnolia wufengensis (Magnoliaceae)[J]. Tree Physiology, 2020, 40(9): 1247-1259. DOI:10.1093/treephys/tpaa057 |
| [22] |
TANG M Y, TAO Y B, XU Z F. Ectopic expression of Jatropha curcas APETALA1 (JcAP1) caused early flowering in Arabidopsis, but not in Jatropha[J]. PeerJ, 2016, 4: e1969. DOI:10.7717/peerj.1969 |
| [23] |
HAN Y, TANG A Y, YU J Y, CHENG T R, WANG J, YANG W R, PAN H T, ZHANG Q X. RcAP1, a homolog of APETALA1, is associated with flower bud differentiation and floral organ morphogenesis in Rosa chinensis[J]. International Journal of Molecular Sciences, 2019, 20(14): 3557. DOI:10.3390/ijms20143557 |
| [24] |
李成儒, 董钠, 李笑平, 吴沙沙, 刘仲健, 翟俊文. 兰科植物花发育调控MADS-box基因家族研究进展[J]. 园艺学报, 2020, 47(10): 2047-2062. DOI:10.16420/j.issn.0513-353x.2019-0740 LI C R, DONG N, LI X P, WU S S, LIU Z J, ZHAI J W. A review of MADS-box genes: The molecular regulatory genes for floral organ development in orchidacea[J]. Acta Horticulturae Sinica, 2020, 47(10): 2047-2062. DOI:10.16420/j.issn.0513-353x.2019-0740 |
| [25] |
SUN Y K, FAN Z Q, LI X L, LI J Y, YIN H F. The APETALA1 and FRUITFUL homologs in Camellia japonica and their roles in double flower domestication[J]. Molecular Breeding, 2014, 33(4): 821-834. DOI:10.1007/s11032-013-9995-9 |
| [26] |
CHUANG T H, LI K H, LI P F, YANG C H. Functional analysis of an APETALA1-like MADS box gene from Eustoma grandiflorum in regulating floral transition and formation[J]. Plant Biotechnology Reports, 2018, 12(2): 115-125. DOI:10.1007/s11816-018-0475-x |
| [27] |
陆顺教, 易双双, 任羽, 杨光穗, 黄少华, 尹俊梅. 兰科植物花发育分子机理研究进展[J]. 中国农学通报, 2015, 31(7): 113-124. LU S J, YI S S, REN Y, YANG G S, HUANG S H, YIN J M. Research progress on molecular mechanism in the floral development of Orchidaceae[J]. Chinese Agricultural Science Bulletin, 2015, 31(7): 113-124. |
(责任编辑 崔建勋)
 2023, Vol. 50
2023, Vol. 50