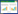文章信息
基金项目
- 广东省科技创新战略专项基金(pdjh2023b0500);惠州学院教授、博士启动项目(2021JB017)
作者简介
- 龚浩,博士,毕业于中国科学院大学,惠州学院生命科学学院讲师,惠州市食品安全协会顾问专家,主要从事不同物种群体的分化和大数据技术在临床医学的应用研究,致力于将基因组技术应用在农作物育种和分类上,并利用大数据技术挖掘农作物的优良基因资源,促进精准育种。在《Genome Biology》 《Theoretical Applied Genetics》《Nature Genetics》等期刊发表SCI论文15篇,其中第一作者7篇,论文累计被引用转载500余次;获授权国家发明专利3件。龚浩(1989—),男,博士,讲师,研究方向为植物基因组学,E-mail:mygonghao@163.com; 孙键,博士,毕业于中山大学,2017—2020年在深圳大学生命与海洋科学学院海洋研究中心进行博士后研究工作,现为惠州学院生命科学学院讲师,主要从事植物分类与植物生理生态研究,重点关注维管植物分类与红树植物生理生态过程,目前致力于粤东地区植物分类与特色植物资源开发与利用、绿色农业生产技术开发应用研究。参与国家级、省部级科研项目6项,在国内外核心期刊发表学术论文10余篇,参编专著2部.
通讯作者
- 孙键(1985—),男,博士,讲师,研究方向为植物分类学,E-mail:sunjian8@mail2.sysu.edu.cn.
文章历史
- 收稿日期:2023-07-31
2. 惠州学院经济管理学院,广东 惠州 516007
2. School of Economics and Management, Huizhou University, Huizhou 516007, China
【研究意义】茶树〔Camellia sinensis (L.) O.Kuntze〕属山茶科山茶属多年生常绿木本植物,原产于热带及亚热带,是一种喜暖喜湿的叶用植物,其嫩叶经过加工后即为茶叶。茶叶具有防辐射、提神醒脑、利尿、助消化、减肥和预防疾病的作用,因此茶叶的饮用及流传从古至今都极受重视,是中华民族的举国之饮、世界三大饮品之首[1]。但是茶树异花传粉和长期自交不育的特性,使茶树高度杂合、亲缘关系复杂,茶叶品种难以轻易分辨、分类标准难以统一、鉴别结果有误差等,这就需要对茶叶不同品种进行区分和产地溯源。SSR广泛应用于植物基因定位和QTL分析、DNA指纹和品种鉴定[2]、种质资源保存和利用、系谱分析以及标记辅助育种,通常呈共显性遗传,其多态位点丰富,实验操作简单易行[3]。基于深度神经网络的简单重复序列标记对茶叶产地的溯源研究不仅有利于茶叶的分类和产地溯源[4],还能为其他植物分类提供参考。
【前人研究进展】目前已发表相关论文的茶树测序群体一般为100~200个样本,过于零散且群体覆盖性和代表性较弱,无法用于深度的群体遗传分析[5]。目前,国内对茶叶品种、产地、产季、年份和等级等真实属性的鉴别还主要停留在传统的理化分析与感官评定相结合的水平上,例如GB/T 19598-2006《地理标志产品安溪铁观音》中评价标准是以感官为主,辅以部分理化检测。一方面,具有感官评定能力的专家非常少,特别是面对我国品种繁多的茶叶,具有特定品种茶叶感官评定能力的专家更为稀缺;另一方面,人的感官灵敏度容易受到外界因素的干扰而改变,采用感官评价方法受人为主观影响很大,可操作性较差,且目前还没有明确且易于实现的评定指标或参数,易造成判定结果的偏差[6]。基于此产生电子鼻来分类茶叶,利用气敏传感器阵列对挥发性气味物质响应,使气味成为量化指标的新技术手段,具有检测时间短、样品预处理简单、检测结果可靠等优点[7],可以高效、快速、无损检测不同种类的食品,可应用于茶叶贮藏时间[8]、加工方式、品质[9]和等级[10]等检测。但这种方法会因为检验材料部位不同而出现较大的结果误差。
【本研究切入点】近年来,分子生物学技术和生物信息学的发展有力地推动了DNA分子标记的研究。与形态学标记、细胞标记、生化标记等相比,DNA分子标记技术不易受外界环境及个体本身的影响,具有结果准确、信息量大、检测简单、重复性及稳定性较好等优点[11]。DNA分子标记技术在植物分类学[12]、遗传多样性分析[13]、遗传图谱构建[14]和辅助育种[15]等方面的研究广为应用,但在茶树种质资源方面的研究应用较少,主要集中在遗传多样性及特异标记方面。【拟解决的关键问题】本研究旨在解决茶叶主成分分析、茶叶产地溯源及品种鉴定、DNN模型构建等问题。
1 材料与方法 1.1 试验材料本研究根据Accession No. PRJNA595795和PRJNA562973,从NCBI database中下载323份茶叶的转录组数据,其中来自福建、云南、浙江、湖南省的茶叶分别有130、96、54、33份,其余10份属于外类群样本即研究类群之外亲缘关系最近的物种,这10个外类群为全国收集的茶梅Camellia sasanqua Thunb(表 1)。
1.2 试验方法
1.2.1 鉴定SSR标记位点 本研究先从323份样本中获得样本数据,使用PSR软件(Polymorphic SSR retrieval, PMID: 26428628),鉴定茶叶参考基因组(Tea tree Camellia sinensis, 舒茶早)中所有可能的SSR标记位点。首先利用PSR软件,设置参数支持的reads总数大于5,同时支持reads的比例大于10%,其他参数均为默认参数;再过滤单个位点缺失率较大的位点;最后进行线性回归分析,并结合不同SSR位点的相关性,保留最终的SSR位点[16]。
1.2.2 主成分分析 本研究将323个茶叶样本进行样本间SSR序列的相互比对,计算每个样本与其他样本的差异度,再基于样本间的差异度计算323个样本的基因差异矩阵[17]。利用PCA对323个样本基因差异矩阵进行分析,然后使用R语言中的read.table函数读入数据、ovun.sample函数清理处理数据,最后利用内置函数princomp进行PCA。
1.2.3 构建整体样本的进化树 先用perl的自编脚本获得所有个体的明氏距离矩阵,然后用PHYLIP的neighbour模块构建原始进化树,再用dendroscope对进化树进行展示和修饰。
1.2.4 建立及优化模型 本研究使用Matlab软件的神经网络工具,建立线性回归模型、随机森林模型和DNN模型。在建立线性回归模型的过程中,根据样本数据集,分别生成x矩阵和y矩阵,利用线性回归代码建模,并使用其模型进行预测。在建立随机森林模型的过程中,先将整体样本读到内存中,按照8∶2的比例分为80% 的训练集、20% 的测试集;然后将训练集的样本先分词,再转换为词向量;接着将训练集的样本和标签统一传入算法中,得到拟合后的模型;继而将测试集的样本先分词,再得到词向量;最终把测试集得出的词向量添加到拟合后的模型中,得出结果并将结果转换为准确率的形式。在建立DNN模型的过程中,本研究通过下载WeightWatcher安装包,导入样本数据,利用神经网络代码直接预测准确率。选取准确率最高的深度神经网络模型作进一步优化。
2 结果与分析 2.1 SSR标记位点鉴定结果首先需要初步鉴定SSR位点,通过PSR软件,从茶叶参考基因组数据库中获得所有可能的SSR标记,最终得到3 668个标记位点,其中,比对到染色体上的位点有3 304个(表 2)[18]。SSR标记位点的鉴定:利用PSR软件,经过筛选后得到2 924个位点;过滤单个位点缺失率大于20% 的位点后,获得2 155个多态性位点;通过线性回归分析,筛选在不同省份特异性存在的位点(P < 0.001),获得700个位点;结合不同SSR位点的相关性,在两个及其相关的位点中只保留差异性较大的位点,最终获得54个SSR位点。
2.2 不同来源茶叶样本PCA结果
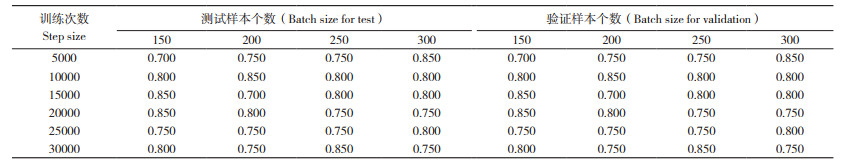
如图 1所示,图中每个点代表 1个样本,两点距离代表茶叶样品受主成分影响下的相似性距离。全部样本的PCA结果表明,Dim1(7.6%)表示第一主成分贡献率为7.6%,Dim2(4.3%)表示第二主成分贡献率为4.3%,即前两个主成分的累计贡献率为11.9%(图 1A);外类群与福建、湖南、云南、浙江4省茶叶样本差异显著,部分与云南省样品个体相近。本研究通过对4省份数据进行PCA来做进一步判断。根据4个省份间的PCA结果(图 1B),并排除外类群的影响,可以发现不同省份间的整体差异较明显,而4个省份内个体相对聚集。其中,云南省内的个体较其他省份差异大;福建、浙江、湖南的样本分别聚集,这表明福建、浙江、湖南3个省份间茶叶差异显著,但有少量交叉,具有一定相似的遗传结构特性,3个省份间的亲缘关系较近。其亲缘关系远近与地理来源并不呈现一致性,原因可能与茶叶人工驯化程度有关。PCA也存在一定的不足之处,简单的PCA只能解释部分个体的产地溯源问题,若要进一步研究溯源问题,则还需要用其他方法,如构建进化树、神经网络模型等方法,来进一步解释和验证交叉个体的溯源问题。
|
| A、C:323个样本间的差异主成分分析;B:313个样本间的差异主成分分析 A, C: PCA of variance among 323 samples; B: PCA of variance among 313 samples 图 1 主成分分析结果 Fig. 1 Principal component analysis results |
2.3 进化树构建结果
从以上PCA分析结果可以看出,不同省份的个体分别聚集,差异较为显著,但也有少量的交叉,其中福建主要与浙江、云南邻近,湖南与云南较近,外类群主要分布在云南附近,而云南个体分类较其他省份分散,由此构建不同省份茶叶的进化树(图 2),其结果与PCA结果相似。

|
| 图 2 不同省份茶叶样本的进化树 Fig. 2 Evolutionary tree of tea samples from different provinces |
2.4 模型构建与优化结果
2.4.1 不同模型预测结果 本研究利用3种不同的模型对54个SSR分子标记矩阵构建模型,再初步鉴定不同模型的差异。通过线性回归模型(81%)、随机森林模型(77%)及DNN模型(86%)对54个SSR marker矩形构建模型,发现深度神经网络模型准确率最高、为86%,故选择DNN模型进行预测[19]。
2.4.2 DNN模型的优化结果 本研究利用Matlab软件的神经网络工具对试验数据进行建模。使用54个SSR和323个样本,构建预测模型,再用Tensorflow2.0优化DNN模型的一次训练样本个数(Batch size)、训练次数(Step size)、隐藏层层数和每层节点数4个参数。
将323份样本中除了外类群以外的数据分成训练集、测试集和验证集3个部分,其中训练集、测试集、验证集的测试比例分别为0.8、0.1、0.1,即训练集273份,测试集20份,验证集20份。先用训练集训练模型,再用测试集进行最后优化,并使用验证集对优化后的模型进行验证。
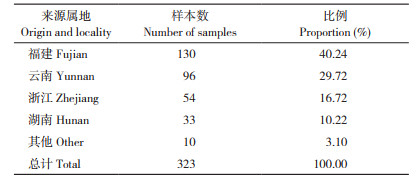
2.4.3 参数Batch size和Step的优化 本研究通过对参数Batch size和Step进行优化,测试不同参数对准确率的影响。对每次训练选取的Batch size分别设为150、200、250、300,而迭代的次数Step分别为5 000、10 000、15 000、20 000、25 000、30 000。理论上Step越高模型准确率就越高,但Step过高会导致模型过度拟合。通过对测试集10次重复验证,发现参数Batch size为150和Step为20 000综合起来表现效果最好(表 3、表 4)。
2.4.4 隐藏层层数和每层节点数的优化 (1) 隐藏层层数的优化:利用不同的随机参数模拟2~ 4层神经网络的测试集和验证集的准确率。经对比,发现神经网络为2层时验证集和测试集的准确率最高,约95%(图 2 A)。(2)每层节点数的优化:确定隐藏层为2层后,分别产生25~150间隔为5的26个可能节点数,隐藏层的两层网络组合一起是26×26共676个组合的矩阵,检测不同参数对应的准确率,每个组合进行10次重复。
然后按以下打分规则对最优准确率进行确定,通过统计不同指标对所有组合进行打分,每一种指标都能进10% 得1分:测试集和验证集准确率的平均值;验证集准确率的平均值;最优验证准确率。最后统计2分以上的次数(图 3),图 3A为在最优Batch size和Step size时不同神经层数的柱状图,对比发现神经网络为2层时验证集和测试集的准确率最高;图 3B、C、D、E为2层隐藏层神经网络参数的优化,其中B为不同维度模拟的自测数据的平均准确率,C为不同维度模拟的验证数据的平均准确率,D为不同维度模拟的自测数据的最优准确率。综合准确率方差等因素,本研究选择隐藏层第一层95、第二层40的模型为最优模型,其中自测集的平均准确率95%,自测集合验证平均值准确率89%,验证集的平均准确率75% 以上,最优准确率为100%。

|
| 图 3 深度神经网络层数和节点数的参数优化结果 Fig. 3 Oprtimization results of layer number and node for each layer for the Deep Neural Network |
3 讨论
我国是茶叶消费大国,随着人们生活水平的提高,消费者对茶品质的要求也日益提高。为了确定茶叶的真实产地,研究者运用各种方法进行研究。目前,生物信息学在基因测序分析中发挥着举足轻重的作用,国内主要是以实验为基础,通过测定农产物及其土壤中的矿质元素,再进行相关性分析、聚类分析、主成分分析等多种统计分析方法,进而对农产品进行溯源分析[20]。本研究主要以生物信息学为基础,通过分析相关的基因位点,构建模型并进行优化,最终对茶叶溯源进行分析。
本研究通过基因组的SSR位点进行基因数据分析。SSR作为第二代分子标记,具有重复性好、多态性高、变异丰富、呈共显性且广泛分布于植物基因组等优点,已被广泛应用于高粱、大麦、小麦、青稞等作物遗传多样性分析和基因研究[21]。与SNP标记相比,SSR标记的优势是成本低、试验技术简单[22]。本研究先利用PSR软件从茶叶参考基因组中鉴定所有可能的SSR标记位点,再比对到染色体上,利用PSR设置参数支持的reads总数大于5同时支持reads的比例大于10%[23],得到样本后进行位点筛选,最终获得54个SSR位点;再利用3种不同的模型对54个SSR分子标记矩阵构建模型,初步鉴定不同模型的差异[24];选择准确率最高的神经网络模型,进行人工神经网络模型的优化和参数选择、Batch size和Step size的优化、隐藏层数目和每层节点数优化、2层隐藏层神经网络参数的优化,最后选择准确率在95% 左右最优的2层神经网络模型[25]。
在研究地理溯源领域中,大部分研究都是利用分子标记或化学标记构建变异图谱,然后查看变异图谱的相似性来进行溯源。本研究使用深度学习预测方法,在研究产地溯源领域使用量较少,主要通过建立样本的基因差异矩阵,使用PCA分析323个样本间的差异度,结果非常直观。通过分析图片,发现外类群与福建、湖南、云南、浙江4省份间的差异显著,而各省份内个体相对聚集,其中云南省内的个体差异较其他省份大;4省份有部分材料重叠在一起,表明不同省份的部分茶叶也具有一定的遗传相似性[26]。构建整体样本的进化树,结果表明不同省份的茶叶个体分别聚集,差异显著,但也有少量交叉,此结果与PCA结果相似[27]。
本研究只研究福建、湖南、云南、浙江4省份和10个外类群共323个样本,茶叶转录组数据存在样本量少的局限性,后续需要增加样本容量,对茶叶溯源作进一步研究。
4 结论本研究对来自湖南、云南、福建和浙江省的313个茶叶样本的来源属地及10个外类群关系进行研究,以筛选出的54个高质量的SSR位点为基础,对样本进行主成分分析,并通过3种不同的分类模型比对及优化,得出2层神经网络模型对茶叶分析效果最佳,准确率约95%。本研究构建的分类模型也可以用于其他物种重测序数据的属地来源鉴定。
| [1] |
赵锟, 林永, 王曼丽. 茶叶中锌、铜、锰、铁、铅含量的测定[J]. 中国科技投资, 2013(S1): 165-166. ZHAO K, LIN Y, WANG M L. Determination of zinc, copper, manganese, iron and lead in tea[J]. China Science and Technology Investment, 2013(S1): 165-166. |
| [2] |
李清, 罗永坚, 吴柔贤, 贾俊婷, 张文虎, 宋松泉, 刘军. 广东省大豆种质资源遗传多样性分析及DNA分子身份证构建[J]. 广东农业科学, 2020, 47(12): 221-228. DOI:10.16768/j.issn.1004-874X.2020.12.023 LI Q, LUO Y J, WU R X, JIA J T, ZHANG W H, SONG S Q, LIU J. Genetic diversity analysis of soybean germplasm resources and construction of DNA molecular identity card in Guangdong Province[J]. Guangdong Agricultural Sciences, 2020, 47(12): 221-228. DOI:10.16768/j.issn.1004-874X.2020.12.023 |
| [3] |
刘冠群, 吴祠平, 谭礼强, 谭杰, 杨婉君, 唐茜. 利用SSR分子标记构建名山茶叶基因身份证[J]. 四川农业大学学报, 2019, 37(4): 469-474, 503. DOI:10.16036/j.issn.1000-2650.2019.04.006 LIU G Q, WU C P, TAN L Q, TAN J, YANG W J, TANG Q. Construction of SSR-based molecular IDs for tea planted in Mingshan[J]. Journal of Sichuan Agricultural University, 2019, 37(4): 469-474, 503. DOI:10.16036/j.issn.1000-2650.2019.04.006 |
| [4] |
徐晓美, 李颖, 孙启迪, 徐小万, 衡周, 李涛, 王恒明. 辣椒种质材料疫病抗性鉴评及遗传多样性分析[J]. 广东农业科学, 2022, 49(10): 19-28. DOI:10.16768/j.issn.1004-874X.2022.10.003 XU X M, LI Y, SUN Q D, XU X W, HENG Z, LI T, WANG H M. Identification of epidemic disease resistance and genetic diversity analysis of pepper germplasm materials[J]. Guangdong Agricultural Sciences, 2022, 49(10): 19-28. DOI:10.16768/j.issn.1004-874X.2022.10.003 |
| [5] |
MAS-SANDOVAL A, JIN C, FRACASSETTI M, FUMAGALLI M. ngsJulia: population genetic analysis of next-generation DNA sequencing data with Julia language[J]. F1000 Research, 2022, 11: 126. DOI:10.12688/f1000research.104368.2 |
| [6] |
周鹏. 基于多平台组学策略的茶叶溯源鉴别技术研究[D]. 南京: 南京理工大学, 2019. DOI: 10.27241/d.cnki.gnjgu.2019.001800. ZHOU P. Research on tea traceability identification technology based on multi-platform omics strategy[D]. Nanjing: Nanjing University of Science and Technology, 2019. DOI: 10.27241/d.cnki.gnjgu.2019.001800. |
| [7] |
杨雪梅, 赵建锐, 王智慧, 武珊珊, 李家华. 电子鼻技术及其在茶叶香气检测中的应用及展望[J]. 中国茶叶, 2020, 42(6): 5-9. YANG X M, ZHAO J R, WANG Z H, WU S S, LI J H. Electronic nose technology and its application and prospect in tea aroma detection[J]. China Tea, 2020, 42(6): 5-9. |
| [8] |
何鲁南, 赵苗苗, 蔡昌敏, 吕才有. 电子鼻技术对不同贮藏地的普洱茶香气分析[J]. 西南农业学报, 2018, 31(4): 717-724. DOI:10.16213/j.cnki.scjas.2018.4.013 HE L N, ZHAO M M, CAI C M, LYU C Y. Aroma analysis of pu-erh tea from different storage places by electronic nose technology[J]. Southwest China Journal of Agricultural Sciences, 2018, 31(4): 717-724. DOI:10.16213/j.cnki.scjas.2018.4.013 |
| [9] |
桂安辉, 高士伟, 叶飞, 龚自明, 郑鹏程, 王胜鹏, 刘盼盼, 王雪萍, 滕靖, 郑琳. 不同产地扁形绿茶的品质成分差异分析[J]. 食品工业科技, 2020, 41(20): 218-223, 229. DOI:10.13386/j.issn1002-0306.2020.20.035 GUI A H, GAO S W, YE F, GONG Z M, ZHENG P C, WANG S P, LIU P P, WANG X P, TENG J, ZHENG L. Analysis of quality compositional differences of flat green tea from different origins[J]. Science and Technology of Food Industry, 2020, 41(20): 218-223, 229. DOI:10.13386/j.issn1002-0306.2020.20.035 |
| [10] |
虞培力, 赵粼, 王晞丞, 张星海. 人工智能对龙井茶等级识别研究[J]. 现代农业科技, 2018(2): 260-263. YU P L, ZHAO L, WANG X C, ZHANG X H. Research on grade recognition of Longjing tea by artificial intelligence[J]. Modern Agricultural Science and Technology, 2018(2): 260-263. |
| [11] |
周超, 马银鹏, 包旭翔, 张介驰. DNA分子标记技术在黑木耳中的研究进展[J]. 北方园艺, 2023(7): 125-131. ZHOU C, MA Y P, BAO X X, ZHANG J C. Progress of DNA molecular labeling technology in Auricularia nigra[J]. Northern Horticulture, 2023(7): 125-131. |
| [12] |
高丽霞, 胡秀, 刘念, 黄邦海, 李正军, 李严. 中国姜花属基于SRAP分子标记的聚类分析[J]. 植物分类学报, 2008, 46(6): 899-905. GAO L X, HU X, LIU N, HUANG B H, LI Z J, LI Y. Cluster analysis of the genus Fluorula in China based on SRAP molecular markers[J]. Journal of Systematics and Evolution, 2008, 46(6): 899-905. |
| [13] |
周建国, 李开绵, 叶剑秋, 杨鹏雅, 王文泉. 应用SRAP标记研究木薯种质资源的遗传多样性[J]. 现代农业科学, 2009, 16(5): 45-47, 55. ZHOU J G, LI K M, YE J Q, YANG P Y, WANG W Q. Application of SRAP markers to study the genetic diversity of cassava germplasm resources[J]. Modern Agricultural Sciences, 2009, 16(5): 45-47, 55. |
| [14] |
金梦阳, 刘列钊, 付福友, 张正圣, 张学昆, 李加纳. 甘蓝型油菜SRAP、SSR、AFLP和TRAP标记遗传图谱构建[J]. 分子植物育种, 2006(4): 520-526. JIN M Y, LIU L Z, FU F Y, ZHANG Z S, ZHANG X K, LI J N. Genetic mapping of SBAP, SSR, AFLP and TRAP markers in kale-type oilseed rape[J]. Molecular Plant Breeding, 2006(4): 520-526. |
| [15] |
夏明元, 李进波, 张建华, 万丙良, 戚华雄. 利用分子标记辅助选择技术选育具有中等直链淀粉含量的早稻品种[J]. 华中农业大学学报, 2004(2): 183-186. DOI:10.13300/j.cnki.hnlkxb.2004.02.003 XIA M Y, LI J B, ZHANG J H, WAN B L, QI H X. Use of molecular marker-assisted selection technology to select early rice varieties with medium rectilinear amylose content[J]. Journal of Huazhong Agricultural University, 2004(2): 183-186. DOI:10.13300/j.cnki.hnlkxb.2004.02.003 |
| [16] |
徐放, 朱报著, 潘文, 朱政财, 钟乃盛, 李文业, 赵强民. 基于SSR标记的广东含笑等11个含笑属物种亲缘关系研究[J]. 广东农业科学, 2021, 48(1): 87-93. DOI:10.16768/j.issn.1004-874X.2021.01.011 XU F, ZHU B Z, PAN W, ZHU Z C, ZHONG N S, LI W Y, ZHAO Q M. Study on the affinities of eleven species of the genus Hibiscus in Guangdong based on SSR markers[J]. Guangdong Agricultural Sciences, 2021, 48(1): 87-93. DOI:10.16768/j.issn.1004-874X.2021.01.011 |
| [17] |
杨如兴, 何孝延, 张磊, 陈芝芝, 尤志明. 福建茶叶品种选育现状及其对茶产业的推动作用分析[J]. 福建农业学报, 2017, 32(8): 909-916. DOI:10.19303/j.issn.1008-0384.2017.08.019 YANG R X, HE X Y, ZHANG L, CHEN Z Z, YOU Z M. Analysis of the current status of tea tree variety selection in Fujian and its role in promoting the tea industry[J]. Fujian Journal of Agriculture, 2017, 32(8): 909-916. DOI:10.19303/j.issn.1008-0384.2017.08.019 |
| [18] |
陆嘉文, 陈始圆, 袁履凡, 陈扶明, 谢长勇, 李川涛. 基于Matlab深度学习的智能听诊系统应用程序开发[J]. 中国医学物理学杂志, 2023, 40(5): 602-608. LU J W, CHEN S Y, YUAN L F, CHEN F M, XIE C Y, LI C T. Application development of intelligent auscultation system based on Matlab deep learning[J]. Chinese Journal of Medical Physics, 2023, 40(5): 602-608. |
| [19] |
CHEN D, MILACIC V, CHEN M S, WAN S B, LAN W H, HUO C, LANDIS-PIWOWAR K R, CUI Q C, WALI A, CHAN T H, DOU Q P. Tea polyphenols, their biological effects and potential molecular targets[J]. Histology and Histopathology, 2008, 23(4): 487. DOI:10.14670/HH-23.487 |
| [20] |
李磊. 区县尺度下茶叶产地溯源技术研究[D]. 南京: 南京农业大学, 2018. DOI: 10.27244/d.cnki.gnjnu.2018.000514. LI L. Research on traceability technology of tea origin at district and county scale[D]. Nanjing: Nanjing Agricultural University, 2018. DOI: 10.27244/d.cnki.gnjnu.2018.000514. |
| [21] |
吴迪. 小麦全基因组抗赤霉病QTL关联标记的筛选[D]. 扬州: 扬州大学, 2018. WU D. Screening of genome-wide QTL-associated markers for resistance to downy mildew in wheat[D]. Yangzhou: Yangzhou University, 2018. |
| [22] |
李剑峰, 张博, 全建章, 王永芳, 张小梅, 赵渊, 袁玺垒, 贾小平, 董志平. 基于SSR标记的谷子主要农艺性状关联位点检测及等位变异分析[J]. 中国农业科学, 2019, 52(24): 4453-4472. DOI:10.3864/j.issn.0578-1752.2019.24.002 LI J F, ZHANG B, QUAN J Z, WANG Y F, ZHANG X M, ZHAO Y, YUAN X L, JIA X P, DONG Z P. Detection of association loci and allelic variation analysis of major agronomic traits in cereals based on SSR markers[J]. Scientia Agricultura Sinica, 2019, 52(24): 4453-4472. DOI:10.3864/j.issn.0578-1752.2019.24.002 |
| [23] |
AHMED W, FEYISSA T, TESFAYE K, FARRAKH S. Genetic diversity and population structure of date palms (Phoenix dactylifera L.) in Ethiopia using microsatellite markers[J]. Journal of Genetic Engineering and Biotechnology, 2021, 19(1): 64. DOI:10.1186/s43141-021-00168-5 |
| [24] |
KEERTHANA U, PHALGUNI M, PRABHUKARTHIKEYAN S R, NAVEENKUMAR R, YADAV M K, PARAMESWARAN C, BAITE M S, RAGHU S, REDDY M G, HARISH S, PANNEERSELVAM P, RATH P C. Elucidation of the population structure and genetic diversity of Bipolaris oryzae associated with rice brown spot disease using SSR markers[J]. 3 Biotech, 2022, 12(10): 281. DOI:10.1007/s13205-022-03347-4 |
| [25] |
孙道宗, 丁郑, 刘锦源, 刘欢, 谢家兴, 王卫星. 基于改进SqueezeNet模型的多品种茶叶叶片分类方法[J]. 农业机械学报, 2023, 54(2): 223-230, 248. DOI:10.6041/j.issn.1000-1298.2023.02.022 SUN D Z, DING Z, LIU J Y, LIU H, XIE J X, WANG W X. A multi-species tea tree leaf classification method based on improved SqueezeNet model[J]. Transactions of the Chinese Society for Agricultural Machinery, 2023, 54(2): 223-230, 248. DOI:10.6041/j.issn.1000-1298.2023.02.022 |
| [26] |
傅虹凯. 基因芯片+ 区块链的高端茶叶可追溯平台[J]. 食品安全导刊, 2023(10): 140-143. DOI:10.16043/j.cnki.cfs.2023.10.044 FU H K. Gene chip+blockchain for high-end tea traceability platform[J]. China Food Safety Magazine, 2023(10): 140-143. DOI:10.16043/j.cnki.cfs.2023.10.044 |
| [27] |
张婉, 胡文文, 叶巧茹, 林燕萍, 杨雪琪, 叶江华, 何海斌. 茶叶产地溯源研究进展[J]. 亚热带农业研究, 2021, 17(2): 137-144. DOI:10.13321/j.cnki.subtrop.agric.res.2021.02.012 ZHANG W, HU W W, YE Q R, LIN Y P, YANG X Q, YE J H, HE H B. Research progress on tea origin traceability[J]. Subtropical Agriculture Research, 2021, 17(2): 137-144. DOI:10.13321/j.cnki.subtrop.agric.res.2021.02.012 |
(责任编辑 邹移光)
 2023, Vol. 50
2023, Vol. 50