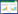文章信息
基金项目
- 安徽省农作物和畜禽良种联合攻关方案(皖农种函〔2021〕811号)
作者简介
- 黄泽珩(1999—),男,在读硕士生,研究方向为动物繁殖生物技术和生物信息学,E-mail:zuya_heng29@163.com.
通讯作者
- 杜志强(1975—),男,博士,教授,研究方向为动物遗传育种与繁殖,E-mail:zhqdu@yangtzeu.edu.cn.
文章历史
- 收稿日期:2023-11-10
【研究意义】生猪是我国最重要的家养动物之一,了解猪的早期胚胎发育规律,改善胚胎制备、培养和移植技术,有助于提高生猪扩繁效率[1-2]。依赖体内胚胎开展基础研究耗时费力,成本高且产量低[3-5],因此建立体外成熟培养技术体系尤为重要。然而,体外授精(In vitro fertilization,IVF)和孤雌激活(Parthenogenetic activation,PA)等体外胚胎激活方式制备的胚胎质量均不如体内受精(In vivo fertilization,InVivo)[6-8]。因此,解析猪早期胚胎在不同激活方式下的发育生物学和分子机制,对提高猪的繁育效率、进一步探明生殖生理和生殖疾病均具有重要意义。【前人研究进展】单细胞转录组测序(Single cell transcriptome sequencing,scRNA-seq)技术被广泛应用于生殖生物学研究,如通过比较分析猪卵巢各个成熟阶段的窦卵泡,揭示其内部复杂的细胞多样性和基因信号网络[9];分析人类和小鼠的围植期胚胎,发现体外培养或植入失败的胚胎在能量产生和甲基化等信号途径上有异常[10]。我们前期通过猪卵母细胞scRNA-seq分析,揭示了mRNA和lncRNA在卵母细胞发育成熟过程中的动态变化,并鉴定了体外成熟和孤雌胚胎间的差异表达基因;提供加权基因共表达网络分析发现IVF和PA早期胚胎的不同功能模块及其关键基因[11-13]。但是scRNA-seq数据具有大量零值,或“dropout”事件,基因不表达、RNA捕获效率不稳定,或测序建库技术等原因,均会严重阻碍下游分析。深度学习方法用于插补数据,有利于解决此类问题,并挖掘高维数据中潜在的信息[14]。AutoClass作为一种新型的深度学习模型,融合了自编码器和分类器两种组件,不依赖分布假设,可以有效捕捉基因间非线性相互作用,完成数据恢复、聚类分析及批次效应消除等任务[15-16]。【本研究切入点】前期已有体外授精和孤雌早期胚胎间差异[12]和2细胞胚胎中卵裂球异质性[17-18]的研究,需深入探讨激活方式对猪早期胚胎发育机理的影响。【拟解决的关键问题】本研究通过对3种不同激活方式的猪早期胚胎进行scRNA-seq数据采集和自编码器处理,评估自编码器对此类数据分析的有效性,揭示不同激活方式胚胎间的差异,识别影响胚胎发育的关键因子和信号通路,为深入研究猪早期胚胎发育过程中的分子生物学机制提供新视角,为进一步研究哺乳动物早期胚胎发育的调控机制提供重要的基础信息。
1 材料与方法 1.1 数据来源从受精至8细胞阶段的猪胚胎scRNA-seq数据(表 1),均从美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)和欧洲核苷酸序列库(European Nucleotide Archive, ENA)获取,包含IVF、PA、InVivo 3种不同激活方式的猪早期胚胎单细胞转录组数据,IVF、PA生物数据编号为PRJNA692116[12],InVivo生物数据编号为PRJNA580004[18]。单细胞测序平台为GPL22918平台(HiSeq X Ten),PRJNA692116和PRJNA580004的建库方式分别为Ansuper-seq和Smart-seq2。IVF和PA胚胎均包含1细胞3个、2细胞6个、4细胞9个、8细胞6个;InVivo胚胎包含1细胞3个、2细胞6个、4细胞13个、8细胞23个。
|
1.2 基因表达矩阵构建
应用fastp软件(版本0.23.0)对原始fastq文件进行质量控制。从Ensembl数据库(https://www.ensembl.org)下载猪的参考基因组和基因注释文件(Sus_scrofa.Sscrofa11.1.110.gtf),借助Hisat2(版本2.2.1)的hisat2-build命令构建基因组索引,处理过的文件被用于基因组序列比对。选择samtools(版本1.16.1)工具将格式转换为bam格式并排序。featureCounts(版本2.0.1)结合基因注释文件对排序后的bam文件进行基因表达量化,最后运用R统计软件(版本4.3.1)[19]处理量化结果,生成基因表达矩阵。
1.3 数据质控scRNA-seq数据的质量控制(Quality control,QC)和聚类处理,有助于提高数据解释的准确性。使用SCP包(版本0.5.1)(https://github.com/zhanghao-njmu/SCP)的RunCellQC函数筛选低质量细胞。QC条件包括:(1)基因表达量(log10_ nCount)和样本特征数(log10_nFeature)均需介于2.5~5.0;(2)线粒体基因表达比例不超过30%。为消除批次效应,利用Integration_SCP函数的harmony参数(版本0.1.1)进行数据整合。随后使用Standard_SCP函数对Seurat对象进行标准化、降维及聚类分析。
1.4 AutoClass数据插补AutoClass算法基于自编码器框架,使用AutoClassImpute函数执行插补运算。为得到稳定的预聚类结果,本研究调整num_cluster参数,将预聚类的数量设定为11~13。
使用Python中scikit-learn包(版本0.24.2)的函数将t-SNE结果进行K-Means聚类[15],计算选用的4个核心指标评估数据插补和聚类效果(值越高,表明聚类性能越好):(1)平均轮廓宽度(Average silhouette width, ASW)[20],计算所有数据点轮廓系数的均值,用于评价聚类一致性;(2)校正兰德指数(Adjusted Rand index, ARI)[21],比较成对聚类结果,衡量分簇准确性;(3)归一化互信息(Normalized mutual information, NMI)[22],利用两个随机变量的关联度量化聚类结果的相关性;(4)纯度分数(Purity score, PS)[23],为每个聚类簇分配主要类别并统计频数,评估聚类的统一性。
1.5 数据下游分析对AutoClass插补后的表达矩阵进行下游分析:UMAP[24] 方法降维聚类,借助SCP包的CellDimPlot函数直观可视化聚类结果;差异基因检测采用Wilcox统计检验,校正P < 0.05,avglog2FC > 1;采用clusterProfiler软件包(版本4.6.2)[25]分析Gene Ontology数据库的生物过程(Biological process, BP)中的信号通路。
2 结果与分析 2.1 AutoClass数据插补在数据预处理阶段,筛选出8个质量异常样本,包括来源于InVivo的2细胞(1个)、4细胞(1个)和8细胞(2个)胚胎,来源于IVF的4细胞(1个)胚胎,以及来源于PA的4细胞(2个)和8细胞(1个)胚胎。其中7个样本的log10nFeature < 2.5,而源自PA的8细胞胚胎样本的log10nCount < 2.5。
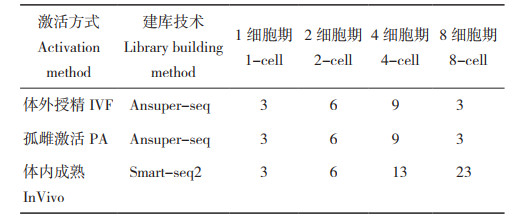
通过分析原始数据处理和AutoClass数据插补两种策略的聚类效果发现,QC处理有助于提升聚类性能,ASW指标增强尤为显著(表 2)。相比而言,采取AutoClass插补措施后,4项性能指标均明显提升(表 2,图 1B、D)。值得注意的是,8个异常样本均被正确聚类到相应簇中。此外,省略QC直接应用AutoClass可进一步提高聚类性能(图 1B)。
|
| A:原始表达矩阵未经任何处理;B:使用AutoClass进行插补但未经QC处理;C:原始表达矩阵经QC处理;D:使用AutoClass进行插补且经QC处理。A、B箭头指向8个质量异常样本 A: Raw gene expression matrix without any processing; B: Interpolation with AutoClass but without QC processing; C: Raw gene expression matrix after QC processing; D: Interpolation with AutoClass after QC processing. A and B arrows point to 8 abnormal samples 图 1 不同处理方式基因表达矩阵t-SNE的可视化结果 Fig. 1 Visualization results of t-SNE for gene expression matrix with different processing methods |
2.2 降维聚类分析
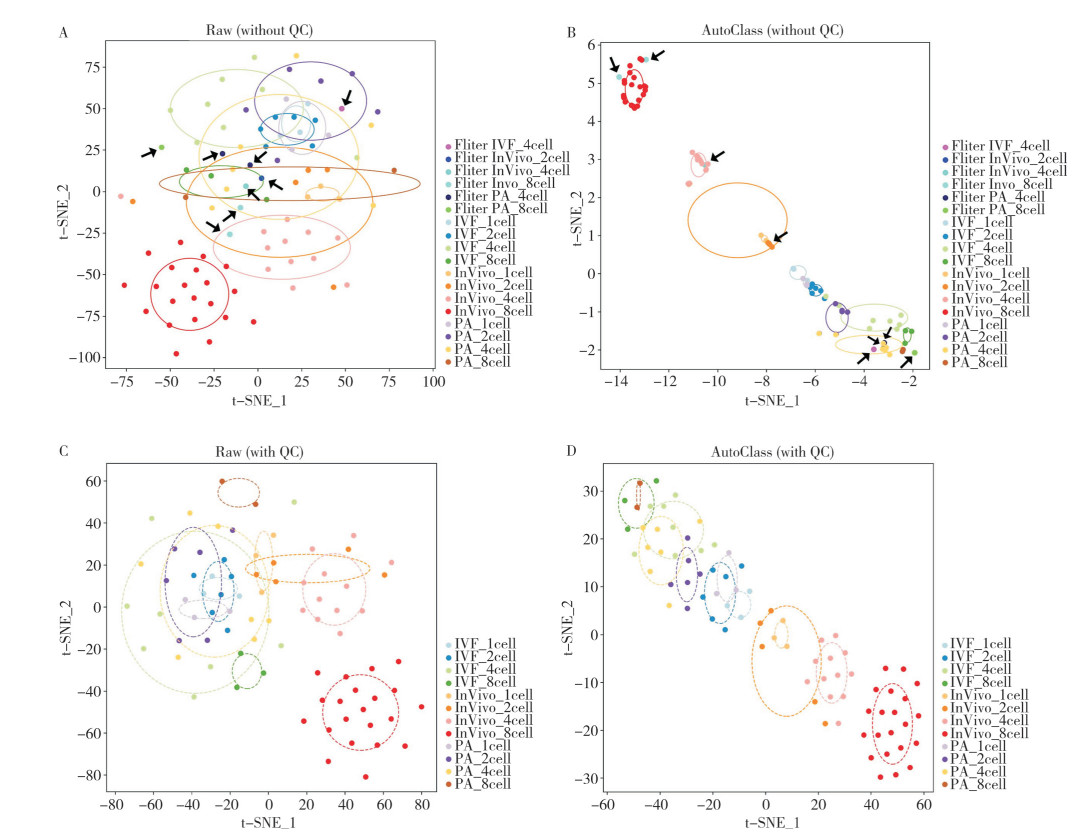
通过比较原始基因表达矩阵(仅经过QC处理)和插补后的基因表达矩阵(经QC处理、未经QC处理)的降维聚类表现,对数据插补的效果进行分析评估,基于“激活方式”(Method)和“激活方式+细胞类型”(Batch)两个标准展开UMAP降维聚类可视化。在“Method”分组下,聚类分析结果揭示了仅经QC处理和采用插补处理在分群性能上有显著差异(图 2A、C、E),插补后分界更为清晰,特别在区分InVivo激活方式与其他方式上效果明显。在“Batch”分组中,观察到不同激活方式下的胚胎发育阶段的聚类差异。仅经QC处理(图 2B)3种激活方式的1细胞期胚胎聚集在左上角,且每个类别之间呈现出更大的离散性;插补处理后(图 2D、F),IVF和PA胚胎的1~8细胞期分簇更紧密,聚集度较高,展示出聚类一致性的提升。

|
| A、B:原始表达矩阵经QC处理;C、D:使用AutoClass进行插补但未经QC处理;E、F:使用AutoClass进行插补且经QC处理 A, B: Raw gene expression matrix after QC processing; C, D: Interpolation with AutoClass but without QC processing; E, F: Interpolation with AutoClass after QC processing 图 2 不同处理方式基因表达矩阵UMAP降维聚类的可视化结果 Fig. 2 Visualization results of UMAP dimensionality reduction clustering for gene expression matrix with different processing methods |
2.3 基因表达差异分析
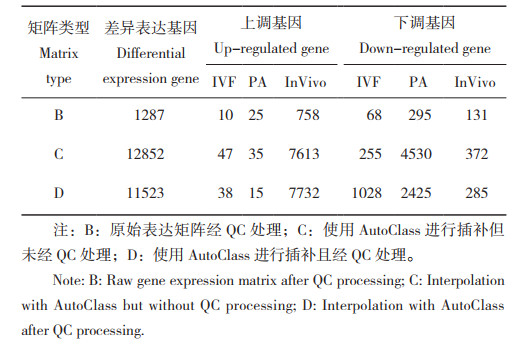
使用SCP包中的RunDEtest函数,筛选出在3种不同表达矩阵处理条件下,3种激活方式之间的显著差异表达基因(DEG)(P < 0.05)。由表 3可见,(1)仅经QC处理,发现1 287个DEG(图 3A),其中793个上调基因、494个下调基因;(2)数据插补但未经QC处理,共识别出12 852个DEG(图 3B),其中7 695个上调基因、5 157个下调基因;(3)数据插补且经QC处理,筛选出11 523个DEG(图 3C),7 785个上调基因、3 738个下调基因。由此可见,经数据插补处理大量不准确的零值后,有效挖掘出大量潜在的基因表达信息。此外,同时应用插补和QC,DEG总数相较于仅进行插补稍有下降,但下调基因数量明显减少,表明QC可以提高数据质量。
|

|
| A:原始表达矩阵经QC处理;B:使用AutoClass进行插补但未经QC处理;C:使用AutoClass进行插补且经QC处理 A: Raw gene expression matrix after QC processing; B: Interpolation with AutoClass but without QC processing; C: Interpolation with AutoClass after QC processing 图 3 3种矩阵处理方式差异基因分析火山图 Fig. 3 Volcano plot for differential gene analysis of three matrix processing methods |
本研究采用AutoClass插补和QC后的基因表达矩阵研究3种不同胚胎激活方式间的差异,显著差异前10位的基因包括:(1)InVivo上调EFNA3、HSD17B3、IQCD、GPR18、NENF和下调TMEM150B、SCN2B、IQCF6、FNDC4、KLK9;(2)IVF上调DLG3、LUZP2、NR5A1、SOX6、NFAT5和下调OLFM3、KCNC4、LYPD6B、MS4A1、DCDC2C;(3)PA上调CATSPERB、C7、CLEC12B、PRSS22和EPDR1和下调DDX39B、HNRNPK、TCP1、FLNC和GPI(表 4)。

|
2.4 功能富集分析
热图(图 4)分析揭示了InVivo的基因表达模式与体外的两种激活方式间存在明显差异,而IVF与PA表现出高度相似性。GO功能富集分析发现各激活方式活跃度排前5的BP通路。仅经QC处理,InVivo激活富集RNA加工、核糖核蛋白复合体生物合成、核糖体生物合成、核糖体RNA代谢过程、非编码RNA(Non-coding RNA, ncRNA)加工的细胞内通路;PA激活富集DNA甲基化依赖的异染色质信息、异染色质信息调节、异染色质组织调节、选择性异染色质形成的细胞内通路(图 4A)。经数据插补但未经QC,InVivo激活富集RNA加工、核糖核蛋白复合体生物合成、核糖体生物合成、ncRNA加工、ncRNA代谢过程的细胞内通路;IVF激活富集酶连接受体蛋白信号通路、跨膜受体蛋白酪氨酸激酶信号通路、性腺发育、原始性别特征发育、正向调节发育过程的细胞内通路(图 4B)。经数据插补和QC处理,InVivo激活富集RNA加工、核糖核蛋白复合体生物合成、核糖体生物合成、ncRNA代谢过程、ncRNA加工的细胞内通路;IVF激活富集性腺发育、原始性别特征发展、性别差异化、性别决定、生殖结构发育的细胞内通路;PA激活富集防御反应、对其他生物防御反应、适应性免疫反应、调节有规律的分泌途径、对其他生物反应的细胞内通路(图 4C)。

|
| 图 4 差异分析热图(左)及GO功能分析结果(右) Fig. 4 Variance analysis heat map (left) and GO function analysis results (right) |
在3种数据处理方法中,InVivo激活均显著富集在RNA加工和核糖体生物合成的细胞内通路。应用AutoClass插补后IVF中的DEG在性腺发育和性别特征发展的通路中富集。插补和QC处理后,PA的DEG主要富集在与生物防御机制相关的生物学过程通路中。虽然富集的功能条目存在相似点,但采取数据插补和QC能提供更全面的视角。
3 讨论本研究通过比较AutoClass插补前后的4项聚类指标、常规降维流程、差异分析和功能富集分析的结果,发现AutoClass有助于研究者在scRNA-seq相关研究中揭示潜在的细胞亚群和组织动态变化,降低“dropout”事件、假阳性结果带来的不利影响,使不同类型的胚胎聚类更为清晰,筛选出数量更多的差异基因和关键生物学信号通路,为探索罕见细胞亚群或状态提供支持[26-27]。
采用AutoClass挖掘出IVF中表达显著增加的NFAT5,PA中上调的CATSPERB、CLEC12B和下调的DDX39B和HNRNPK。以上5个差异基因均出现在4细胞期胚胎中[12],在胚胎发育中有重要功能。NFAT5是一种转录因子,主要在调节细胞对渗透压应激的响应中发挥作用,上调可能是胚胎适应这些外加人工条件的一种机制。该基因会以激活Wnt信号通路的方式调节小鼠卵泡中的颗粒细胞功能,对卵泡发生起到关键作用,并且在猪体外成熟后显著下调[28-29]。出现该现象可能是由于胚胎已适应体外培养环境,减少了对渗透压调节机制的依赖。CATSPERB主要与精子的运动功能有关,在PA中显著表达可能表明胚胎在无精子激活的情况下,自行调控细胞内的Ca2+通道以支持细胞活动[30]。CLEC12B是一种C型凝集素受体,可能参与调节胚胎的免疫环境,或在胚胎细胞的分化和发育中发挥作用,尤其是在没有精子基因贡献的情况下。DDX39B和HNRNPK下调可能对胚胎发育产生消极作用:DDX39B是一种RNA解旋酶,主要参与RNA的加工和转运,下调可能影响mRNA的成熟和稳定性,进而影响基因组的稳定性[31];HNRNPK是一种多功能的核糖核蛋白,该基因表达水平的降低与新生大鼠卵巢中早期卵泡的损失相关[32],其下调可能影响胚胎发育所需特定蛋白质的表达,对卵泡发育成熟具有重要性。
通过功能富集分析发现,尽管InVivo胚胎中挖掘出的差异基因具有明显的数量差异,但经过3种不同的数据预处理,均富集到与细胞基本功能和代谢过程相关的通路中[33],说明这些胚胎比IVF和PA激活的胚胎更活跃,处于高代谢状态,暗示代谢途径在胚胎发育中的重要性,同时也说明了AutoClass插补分析的可靠性。在InVivo胚胎中,上调基因EFNA3参与多种细胞间的相互作用和信号传导,能与母体共同作用,增强胚胎与子宫内膜的结合并促进发育[34];GRP18作为内源性大麻素系统配体反应的受体,调控雌性生殖周期的各个阶段[35]。此外,新发现与精卵互作相关的HSD17B3和IQCD,前者是睾酮生成的关键酶[36],而后者通过抑制顶体反应,降低精子中蛋白质酪氨酸磷酸化,特异性阻断大鼠体外授精[37]。该结果可用于在体外受精过程中模仿体内环境,提高胚胎质量和发育潜力,有助于进一步了解受精过程。而在IVF胚胎中,
富集到的主要是性腺发育和性别决定通路,可能来源于激素对体外受精的影响或细胞微环境的变化[38]。经数据插补后,还挖掘出在胚胎发育中具有重要地位的SOX6。作为一个重要的调控基因,SOX6结合其他转录因子调控特定基因的表达,进而影响组织的分化和形成[39]。研究发现NR5A1在IVF中显著表达。NR5A1是一种核受体,主要在性激素合成和性腺发育中发挥作用,但其过量表达可能会引发雄激素过多和代谢失衡等问题[40],因此其在IVF中的上调可能对胚胎发育有消极影响。PA胚胎直接富集得到与胚胎发育相关的通路,而插补后主要富集到与生物体免疫反应、对环境刺激应答和内分泌调节有关的通路,侧面反映了电刺激猪卵母细胞形成的早期胚胎可能触发了细胞防御机制[41],导致PA胚胎会表现出质量和发育潜力低下的劣势。
4 结论本研究采用AutoClass自编码器对猪早期胚胎单细胞转录组的公共数据集进行插补,成功提升了细胞分类性能。通过综合质控操作有效消除数据噪声,成功挖掘高维数据中的潜在信息,不仅显著增加了筛选出的差异基因数量,而且揭示了更全面、更具生物学意义的差异基因和生物过程信号通路,如InVivo中对生殖生物学有重要意义的EFNA3和GRP18,IVF和PA中对胚胎发育有消极影响的DDX39B、HNRNPK。此外,根据不同激活方式筛选出的差异基因各自富集到特定的生物调节通路,包括InVivo过程中的基本生物调节、InVivo中的机体防御和性腺发育、PA中的机体防御机制。其中,PA环境下的机体防御机制通路是此前研究中所未曾发现的。
| [1] |
牛安然, 张兴, 杨雨婷, 闫之春, 龚华忠, 丁偌楠, 马黎. 全基因组关联分析在猪育种中的研究进展[J]. 畜牧与兽医, 2023, 55(5): 139-147. NIU A R, ZHANG X, YANG Y T, YAN Z C, GONG H Z, DING N N, MA L. Advances in genome-wide association analysis in pig breeding[J]. Livestock and Veterinary Medicine, 2023, 55(5): 139-147. |
| [2] |
郭诗萌, 刘世超, 刘忠华. 猪胚胎发育与胚胎工程研究进展[J]. 中国畜牧杂志, 2017, 53(9): 1-3. DOI:10.19556/j.0258-7033.2017-09-001 GUO S M, LIU S C, LIU Z H. Research progress on porcine embryonic development and embryo engineering[J]. Chinese Journal of Animal Husbandry, 2017, 53(9): 1-3. DOI:10.19556/j.0258-7033.2017-09-001 |
| [3] |
张美玲, 郝鑫, 周成杰, 王璐, 宋春儒, 韩哲, 张晓洁, 张瑞丰, 梁成光. 哺乳动物雌性生殖细胞及早期胚胎基因敲除或敲减的方法[J]. 生理学报, 2020, 72(1): 17. DOI:10.13294/j.aps.2019.0081 ZHANG M L, HAO X, ZHOU C J, WANG L, SONG C R, HAN Z, ZHANG X J, ZHANG R F, LIANG C G. Deletion or reduction of female germ cells and the early embryos of mammals[J]. Physiological Journal, 2020, 72(1): 17. DOI:10.13294/j.aps.2019.0081 |
| [4] |
潘泽政, 孙梦黎, 梁夏, 李佳, 周芳月, 郑月慧. 哺乳动物雌性生殖干细胞研究的质疑、进展及挑战[J]. 生理科学进展, 2015, 46(1): 69-73. PAN Z Z, SUN M L, LIANG X, LI J, ZHOU F Y, ZHENG Y H. Questioning, progress, and challenges in mammalian female germline stem cell research[J]. Advances in Physiological Sciences, 2015, 46(1): 69-73. |
| [5] |
PATRICK, LONERGAN. Maturation of oocytes in vitro[J]. Annual Review of Animal Biosciences, 2015, 3: 255-268. DOI:10.1146/annurev-animal-022114-110822 |
| [6] |
DE VOS M, GRYNBERG M, HO T M, YE Y, ALBERTINI D F, GILCHRIST R B. Perspectives on the development and future of oocyte IVM in clinical practice[J]. Journal of Assisted Reproduction and Genetics, 2021, 38(6): 1265-1280. DOI:10.1007/s10815-021-02263-5 |
| [7] |
孙婧陶, 姜超前, 刘佳慧, 张弛, 王传玥, 边雅, 姜希卿, 张琪, 金君学. 单宁酸对猪卵母细胞体外成熟及胚胎发育能力的影响[J]. 中国畜牧兽医, 2021, 48(10): 3708-3714. DOI:10.16431/j.cnki.1671-7236.2021.10.022 SUN J T, JIANG C Q, LIU J H, ZHANG C, WANG C Y, BIAN Y, JIANG X Q, ZHANG Q, JIN J X. Effect of tannic acid on in vitro maturation of porcine oocytes and embryonic developmental capacity[J]. China Animal Husbandr y & Veterinary Medicine, 2021, 48(10): 3708-3714. DOI:10.16431/j.cnki.1671-7236.2021.10.022 |
| [8] |
相德才, 李水英, 张斌, 张彦, 梁家充, 吕春荣, 洪琼花, 权国波, 吴国权. 胎牛血清在猪孤雌囊胚玻璃化冷冻后恢复培养中的作用研究[J]. 中国畜牧兽医, 2023, 50(2): 606-615. DOI:10.16431/j.cnki.1671-7236.2023.02.018 XIANG D C, LI S Y, ZHANG B, ZHANG Y, LIANG J C, LYU C R, HONG Q H, QUAN G B, WU G Q. Study on the role of fetal bovine serum in the recovery of porcine orphan female blastocysts in culture after vitrification and freezing[J]. China Animal Husbandry & Veterinary Medicine, 2023, 50(2): 606-615. DOI:10.16431/j.cnki.1671-7236.2023.02.018 |
| [9] |
CHEN N, ZHANG Y, TIAN Y, WU S, GAO F, YUAN X. Deciphering cellular heterogeneity and communication patterns in porcine antral follicles by single-cell RNA sequencing[J]. Animals (Basel), 2023, 13(19): 3019. DOI:10.3390/ani13193019 |
| [10] |
LIU D, CHEN Y, REN Y, YUAN P, WANG N, LIU Q, YANG C, YAN Z, YANG M, WANG J, LIAN Y, YAN J, ZHAI F, NIE Y, ZHU X, CHEN Y, LI R, CHANG H M, LEUNG PCK, QIAO J, YAN L. Primary specification of blastocyst trophectoderm by scRNA-seq: New insights into embryo implantation[J]. Science Advances, 2022, 8(32): eabj3725. DOI:10.1126/sciadv.abj3725 |
| [11] |
YANG C X, WU Z W, LIU X M, LIANG H, GAO Z R, WANG Y, FANG T, LIU Y H, MIAO Y L, DU Z Q. Single-cell RNA-seq reveals mRNAs and lncRNAs important for oocytes in vitro matured in pigs[J]. Reproduction in Domest Animals, 2021, 56(4): 642-657. DOI:10.1111/rda.13901 |
| [12] |
DU ZQ, LIANG H, LIU XM, LIU YH, WANG C, YANG CX. Single cell RNA-seq reveals genes vital to in vitro fertilized embryos and parthenotes in pigs[J]. Scientific Reports, 2021, 11(1): 14393. DOI:10.1038/s41598-021-93904-3 |
| [13] |
WU Z W, GAO Z R, LIANG H, FANG T, WANG Y, DU Z Q, YANG C X. Network analysis reveals different hub genes and molecular pathways for pig in vitro fertilized early embryos and parthenogenotes[J]. Reproduction in Domest Animals, 2022, 57(12): 1544-1553. DOI:10.1111/rda.14231 |
| [14] |
XU J, CUI L, ZHAUNG J, MENG Y, BING P, HE B, TIAN G, KWOK P C, WU T, WANG B, YANG J. Evaluating the performance of dropout imputation and clustering methods for single-cell RNA sequencing data[J]. Computers in Biology and Medicine, 2022, 146: 105697. DOI:10.1016/j.compbiomed.2022.105697 |
| [15] |
BRENDEL M, SU C, BAI Z, ZHANG H, ELEMENTO O, WANG F. Application of deep learning on single-cell RNA sequencing data analysis: A review[J]. Genomics Proteomics Bioinformatics, 2022, 20(5): 814-835. DOI:10.1016/j.gpb.2022.11.011 |
| [16] |
LI H, BROUWER C R, LUO W. A universal deep neural network for in-depth cleaning of single-cell RNA-Seq data[J]. Nature Communications, 2022, 13(1): 1901. DOI:10.1038/s41467-022-29576-y |
| [17] |
FANG T, WANG F, ZHANG R, DU Z Q, YANG C X. Single-cell RNA sequencing reveals blastomere heterogeneity of 2-cell embryos in pigs[J]. Reproduction in Domest Animals, 2023, 58(10): 1393-1403. DOI:10.1111/rda.14454 |
| [18] |
KONG Q, YANG X, ZHANG H, LIU S, ZHAO J, ZHANG J, WENG X, JIN J, LIU Z. Lineage specification and pluripotency revealed by transcriptome analysis from oocyte to blastocyst in pig[J]. Federation of American Societies for Experimental Biology Journal, 2020, 34(1): 691-705. DOI:10.1096/fj.201901818RR |
| [19] |
HAO Y, HAO S, ANDERSEN-NISSEN E, MAUCK W M 3rd, ZHENG S, BUTLER A, LEE M J, WILK A J, DARBY C, ZAGER M, HOFFMAN P, STOECKIUS M, PAPALEXI E, MIMITOU E P, JAIN J, SRIVASTAVA A, STUART T, FLEMING L M, YEUNG B, ROGERS A J, MCELRATH J M, BLISH C A, GOTTARDO R, SMIBERT P, SATIJA R. Integrated analysis of multimodal single-cell data[J]. Cell, 2021, 184(13): 3573-3587. DOI:10.1016/j.cell.2021.04.048 |
| [20] |
LOVMAR L, AHLFORD A, JONSSON M. Silhouette scores for assessment of SNP genotype clusters[J]. BMC Genomics, 2005, 6: 35. DOI:10.1186/1471-2164-6-35 |
| [21] |
HUBERT L, ARABIE P. Comparing partitions[J]. Journal of Classifi cation, 1985, 2(1): 193-218. DOI:10.1007/BF01908075 |
| [22] |
ESTEVEZ P A, TESMER M, PEREZ C A, ZURADA J M. Normalized mutual information feature selection[J]. IEEE Transactions on Neural Networks, 2009(2): 20. |
| [23] |
WU T, HU E, XU S, CHEN M, GUO P, DAI Z, FENG T, ZHOU L, TANG W, ZHAN L, FU X, LIU S, BO X, YU G. clusterProfiler 4.0: A universal enrichment tool for interpreting omics data[J]. Innovation (Camb), 2021, 2(3): 11. DOI:10.1016/j.xinn.2021.100141 |
| [24] |
MANNING C D, RAGHVEN P, HINRICH S. Introduction to information retrieval[M]. London: Cambridge University Press, 2010.
|
| [25] |
BECHT E, MCINNERS L, HEALY J, DUTERE C A, KWOK I W H, NG L G, GINHOUX F, NEWELL E W. Dimensionality reduction for visualizing single-cell data using UMAP[J]. Springer Science and Business Media LLC, 2019(1): 38. DOI:10.1038/NBT.4314 |
| [26] |
姜超, 胡龙飞, 徐春祥, 葛芹玉, 赵祥伟. 单细胞转录组数据中drout的填补方法[J]. 生物医学工程学杂志, 2023, 40(4): 778-783. DOI:10.7507/1001-5515.202301009 JIANG C, HU L F, XU C X, GE Q Y, ZHAO X W. Imputation method for dropout in single-cell transcriptome data[J]. Journal of Biomedical Engineering, 2023, 40(4): 778-783. DOI:10.7507/1001-5515.202301009 |
| [27] |
GOHIL S H, IORGULESCU J B, BRAUN DA, KESKIN D B, LIVAK KJ. Applying high-dimensional single-cell technologies to the analysis of cancer immunotherapy[J]. Nature Reviews Clinical Oncology, 2021, 18(4): 224-256. DOI:10.1038/s41571-020-00449-x |
| [28] |
BRAZERT M, KRANC W, NAWROCKI M J, SUJKA-KORDOWSKA P, KONWERSKA A, JANKOWSKI M, KOCHEROVA I, CELICHOWSKI P, JESETA M, OZEGOWSKA K, ANTOSIK P, BUKOWSKA D, SKOWRONSKI MT, BRUSKA M, PAWELCZYK L, ZABEL M, PIOTROWSKA-KEMPISTY H, NOWICKI M, KEMPISTY B. New markers for regulation of transcription and macromolecule metabolic process in porcine oocytes during in vitro maturation[J]. Molecular Medicine Reports, 2020, 21(3): 1537-1551. DOI:10.3892/mmr.2020.10963 |
| [29] |
TAO H, XIONG Q, JI Z, ZHANG F, LIU Y, CHEN M. NFAT5 is regulated by p53/miR-27a signal axis and promotes mouse ovarian granulosa cells proliferation[J]. International Journal of Biological Sciences, 2019, 15(2): 287-297. DOI:10.7150/ijbs.29273 |
| [30] |
WANG H, LIU J, CHO K H, REN D. A novel, single, transmembrane protein CATSPERG is associated with CATSPER1 channel protein[J]. Biology of Reproduction, 2009, 81(3): 539-544. DOI:10.1095/biolreprod.109.077107 |
| [31] |
CARGILL M, VENKATARAMAN R, LEE S. DEAD-Box RNA helicases and genome stability[J]. Genes, 2021, 12(10): 1471. DOI:10.3390/genes12101471 |
| [32] |
WANG N, ZHANG P, GUO X, ZHOU Z, SHA J. Hnrnpk, a protein differentially expressed in immature rat ovarian development, is required for normal primordial follicle assembly and development[J]. Endocrinology, 2011, 152(3): 1024-1035. DOI:10.1210/en.2010-0797 |
| [33] |
VAN DER WEIDE RH, DE WIT E. Developing landscapes: Genome architecture during early embryogenesis[J]. Current Opinion in Genetics & Development, 2019, 55: 39-45. DOI:10.1016/j.gde.2019.04.009 |
| [34] |
WANG H Q, LIU Y, LI D, LIU J Y, JIANG Y, HE Y, ZHOU J D, WANG Z L, TANG X Y, ZHANG Y, ZHEN X, CAO Z W, SHENG X Q, YANG C F, YUE Q L, DING L J, HU Y L, HU Z B, LI C J, YAN G J, SUN H X. Maternal and embryonic signals cause functional differentiation of luminal epithelial cells and receptivity establishment[J]. Developmental Cell, 2023, 58(21): 2376-2392. DOI:10.1016/j.devcel.2023.08.004 |
| [35] |
LUSCHNIG P, SCHICHO R. Cannabinoids in gynecological diseases[J]. Medical Cannabis and Cannabinoids, 2019, 2(1): 14-21. DOI:10.1159/000499164 |
| [36] |
LAWRENCE B M, O'DONNEL L, SMITH L B, REBOURCET D. New insights into testosterone biosynthesis: Novel observations from HSD17B3 deficient mice[J]. International Journal of Molecular Sciences, 2022, 23(24): 15555. DOI:10.3390/ijms232415555 |
| [37] |
ZHANG P, JIANG W, LUO N, ZHU W, FAN L. IQ motif containing D (IQCD), a new acrosomal protein involved in the acrosome reaction and fertilisation[J]. Reproduction Fertility & Development, 2019, 31(5): 898-914. DOI:10.1071/RD18416 |
| [38] |
KIKUCHI K, KASHIWAZAKI N, NAGAI T, NAKAI M, SOMFAI T, NOGUCHI J, KANEKO H. Selected aspects of advanced porcine reproductive technology[J]. Reproduction in Domestic Animals, 2008, 43(S2): 401-406. DOI:10.1111/j.1439-0531.2008.01191.x |
| [39] |
HAGIWARA N. Sox6, jack of all trades: A versatile regulatory protein in vertebrate development[J]. Developmental Dynamics an Offi cial Publication of the American Association of Anatomists, 2011, 240(6): 1311-1321. DOI:10.1002/dvdy.22639 |
| [40] |
ROTGERS E, NICOL B, RODRIGUEZ K, RATTAN S, FLAWS J A, YAO H H. Constitutive expression of steroidogenic factor-1 (NR5A1) disrupts ovarian functions, fertility, and metabolic homeostasis in female mice[J]. Federation of American Societies for Experimental Biology Journal, 2021, 35(8): e21770. DOI:10.1096/fj.202100304R |
| [41] |
姜文杰, 李英花, 姚雪瑞, 赵予晗, 高青山, 朴春浩, 孙福亮, 许永男. 冷、热应激对猪孤雌胚胎体外发育的影响[J]. 中国畜牧兽医, 2018(9): 2507-2515. DOI:10.16431/j.cnki.1671-7236.2018.09.021 JIANG W J, LI Y H, YAO X R, ZHAO Y H, GAO Q S, PU C H, SUN F L, XIU Y N. Effects of cold and heat stress on in vitro development of solitary female embryos in pigs[J]. China Animal Husbandry & Veterinary Medicine, 2018(9): 2507-2515. DOI:10.16431/j.cnki.1671-7236.2018.09.021 |
(责任编辑 崔建勋)
 2024, Vol. 51
2024, Vol. 51